唾液酸(sialic acids,SA)是9碳氨基糖家族,目前发现大于40种形式源自神经氨酸[1]。在结构上,其2、4、5、7、8和9位上的各种类型的组合有助于SA的多样性[2]。最常见的SA分子是N-乙酰神经氨酸(N-acetylneuraminic acid,NeuAc),一种羧化的9碳单糖,5位上带有N-乙酰基[3]。NeuAc在细菌和动物中无处不在,是人类SA的主要形式。此外,NeuAc在调节生物识别、细菌免疫和疾病方面具有重要作用[4],并且是唾液酸化人乳寡糖重要单体之一,对改善婴儿发育至关重要[5]。因此,NeuAc是一种具有较大市场需求的新型生物发酵产品。
提高NeuAc的发酵产量以及降低其生产成本一直是推动NeuAc在生物发酵领域被应用的重要前提。在过去的研究中,对NeuAc代谢途径中的关键酶做理性改造以提高转化效率[6],但全细胞催化所需的底物昂贵增加了生产成本[7]。而且在微生物代谢改造中,通常利用较强的转录翻译元件以加强产物途径[8],弱化[9]甚至敲除竞争途径[10],代谢流量大量用于产物合成从而导致细胞生长受损[11]。生物传感器与适应性进化的组合策略能够缓解这种平衡失调[12],在应用选择压力时富集高产菌株[13]。进一步地,适应性进化对于稳定微生物表型以及分析进化过程中特定基因的变化情况是一种有效的办法[14]。同时,基于质粒的基因表达系统是微生物代谢途径改造的常用工具,然而质粒的不稳定往往降低了菌株产物合成效率[15]。
为了提升重组枯草芽孢杆菌(Bacillus subtilis)合成NeuAc的产量以及解决发酵过程中重组枯草芽孢杆菌质粒丢失的问题,本文在前期研究的基础上,利用已成功构建的NeuAc-Biosensor调控spc和erm抗性基因表达,实现细胞生长与产物产量偶联,进而通过适应性进化提升NeuAc合成效率。同时通过将必需基因folB插入携带NeuAc合成途径关键酶编码基因重组质粒并且敲除基因组中的folB基因,有效降低了发酵过程中的质粒丢失率。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本实验所使用的菌株及质粒如表1所示。目的基因扩增引物及敲除引物如表2所示。
表1 本研究中使用的质粒和菌株
Table 1 Plasmids and strains used in this study

质粒/菌株描述来源p136NeuAc-Biosensor质粒实验室保存[16]BS1,BS2,BS3NeuAc产量不同的B.subtilis菌株实验室保存p136-spc单抗性筛选质粒,表达spc基因本研究构建p136-erm单抗性筛选质粒,表达erm基因本研究构建p136-spc-erm双抗性筛选质粒,表达spc基因和erm基因本研究构建BS3C适应性进化筛选得到菌株,作为质粒稳定性改造出发菌株本研究构建p43 NMK-AN表达NeuAc合成相关的age基因和neuB基因实验室保存p43 NMK-folB表达必需基因folB本研究构建
表2 本研究中使用的引物
Table 1 Primers used in this study

引物名称引物序列(5’-3’)spc-F1CGTTTAAAATAGTGAGGAGGATATAATGTTTGGATCAGGAGTTGAGAGTGGACTAAAACspc-R1CATTTCACCTCCTTTTAACGCCAGGGTTTTCCCAGTCACGerm-F1CGTTTAATTAAGAAGGAGTGATTACATGAACAAAAATATAAAATATTCTCAAAACTTTTTAACGAGTGAAAAAGTerm-R1GTGTACATTTCACCTCCTTTTTATTTCCTCCCGTTAAATAATAGATAACTATTAAAAATAGACAATACTTspc-F2CGTTTAAAATAGTGAGGAGGATATAATGTTTGGATCAGGAGTTGAGAGTGGACTAAAACspc-R2CACTCCTTCTTAATTTAACGCCAGGGTTTTCCCAGTerm-F2GGCGTTAAATTAAGAAGGAGTGATTACATGAACAAAAATATAAAATATTCTCAAAACTTTTTAACGAGTerm-R2GTGTACATTTCACCTCCTTTTTATTTCCTCCCGTTAAATAATAGATAACTATTAAAAATAGACAATACTTp136-spc-FGGCGTTAAAAGGAGGTGAAATGTACACATGGGTAAGGGAGAAGAACTTTTCACTGGAGTp136-spc-RCCTCCTCACTATTTTAAACGATATACCTTTATACCTGTTATACCATTGTACAACACGAGp136-erm-FAAAGGAGGTGAAATGTACACATGGGTAAp136-erm-RCACTCCTTCTTAATTAAACGATATACCTTTATACCTGTTATACCATTGTACAACp136-spc-erm-FAAAGGAGGTGAAATGTACACATGGGTAAGGGAGAAGAACTTTTCACTGp136-spc-erm-RCCTCCTCACTATTTTAAACGATATACCTTTATACCTGTTATACCATTGTACAACACGAGp43-FTCATGATGATGAAAGCTTGGCGTAATCATGGTCATAGp43-RCCTTGATCCTTCACTTGGGGTTGACGACCGAGTAGTfolB-FGAGGGGTGCACCATGGATAAAGTTTATGTAGAAGGTATGGAGTTTTACGGATATCACGfolB-RCCAAGCTTTCATCATCATGACTTTTTTCTCGTAATTTCAATTGCTACTGATTTfolB-L-FTCATGATGATGAAAGCTTGGCGTAATCATGGTCATAGfolB-L-RGTTTCCTGTGTGAAATTGTTATCCGCTCTTGCTTAAATCCAGCTCAGCGGTfolB-R-FGACTGGGAAAACCCTGGCGTTAACCTTGAGCAAACGATCAACTATGCTfolB-R-RCTCCCGCATCGCATTTTGTAATCTRH-folB-FACTATGGAAGCCAAAGCGGAGAAGRH-folB-RGCTCAGTGGTTTCTCACGTTCATCAzeo-FGAGCGGATAACAATTTCACACAGGAAACAGCTzeo-RTAACGCCAGGGTTTTCCCAGTCACGAC
1.1.2 酶与试剂
质粒提取试剂盒、DNA片段纯化试剂盒、感受态制备试剂盒,生工生物工程(上海)有限股份公司;PrimeStar max聚合酶、限制性内切酶,TaKaRa。
1.1.3 培养基
LB培养基(g/L):胰蛋白胨10,酵母粉5,NaCl 5,pH 7.0;发酵培养基(g/L):酵母粉 12,胰蛋白胨 6,(NH4)2SO4 6,K2HPO4·3H2O 12,KH2PO4 2.5,MgSO4·7H2O 3,葡萄糖 60,尿素 6;枯草芽孢杆菌感受态培养基(均为质量分数):SPI-A:0.4% (NH4)2SO4,2.8% K2HPO4·3H2O,1.2% KH2PO4,0.2% C6H5 Na3O7·2H2O,SPI-B:0.04% MgSO4·7H2O。100×CAYE:2% 酪蛋白水解物,10% 酵母粉,100×EGTA∶10 mmol/L EGTA;SPI:980 μL SPI-A,980 μL SPI-B,20 μL 100×CAYE,20 μL 50%的葡萄糖溶液;SPII:2 mL SPI,20 μL 50 mmol/L CaCl2,20 μL 250 mmol/L MgCl2。
1.2 实验方法
1.2.1 重组抗性质粒构建方法
以质粒p136为模板,分别以引物136-spc-F/R和136-erm-F/R进行扩增得到大小为8 744 bp的线性化质粒载体;以质粒p7s6为模板,引物spc-F1/R1和spc-F2/R2进行扩增得到大小为783 bp的spc基因;以质粒p7e6为模板,引物erm-F1/R1和erm-F2/R2进行扩增得到大小为738 bp的erm基因。将扩增得到的线性DNA片段回收纯化后进行Gibson组装[17],并转化至E.coli JM109进行质粒扩增,得到正确重组质粒p136-spc、p136-erm和p136-spc-erm。
1.2.2 质粒构建与基因敲除方法
以p43 NMK-AN为模板,引物p43-F/R进行扩增得到大小为9 094 bp的线性化质粒载体,以B.subtilis 168基因组为模板,引物folB-F/R进行扩增得到大小为363 bp的folB基因。将扩增得到的线性DNA片段回收纯化后进行Gibson组装,并转化至E.coli JM109进行质粒扩增。菌落PCR和测序验证后,得到正确重组质粒p43-folB,进一步将质粒转化菌株BS3C。然后对基因组上folB进行敲除[18],以p7Z6质粒为模板,引物zeo-F/R为引物扩增得到大小为656 bp的zeo基因,以B.subtilis 168基因组为模板,分别使用引物folB-L-F/R和folB-R-F/R扩增得到大小为944和1 022 bp左右同源臂,将扩增得到的线性DNA片段回收纯化后以引物RH-folB-F/R进行PCR融合,将得到融合产物转化上述菌株BS3C,选择zeo转化子后将pTSC引入,组成型表达的Cre重组酶介导lox71和lox66之间的重组,涂布于无抗性平板并在51 ℃下培养,消除了pTSC,从而获得了目标菌株BS3B。
1.2.3 培养方法
24孔深孔板发酵:挑取单菌落接种至发酵培养基中,每孔培养基为1.5 mL,37 ℃、220 r/min培养50 h左右。上述培养基中加入相应筛选浓度的壮观霉素和红霉素。
1.2.4 感受态细胞制备
大肠杆菌感受态制备和转化:参照上海生工大肠杆菌超级感受态细胞制备试剂盒说明书。
枯草感受态制备和转化:挑取宿主菌接种于1支2 mL SPI 培养基中,于14 mL摇菌管中,37 ℃摇床培养过夜;取40 μL过夜培养的菌液,接种至用2 mL SPI 培养基中,37 ℃摇床培养5~6 h后,取200 μL接种到2 mL SPII 培养基中,37 ℃ 220 r/min培养1.5~2 h;加入20 μL 100×EGTA溶液,37 ℃ 220 r/min 摇床培养10 min,用1.5 mL离心管分装成500 μL每管;加入适量的质粒0.5~1 μg,轻轻混匀,37 ℃ 220 r/min 培养1.5~2 h;以4 000 r/min 离心2 min收集菌体,弃部分上清液,留100 μL重悬菌体,涂布于相应的抗性平板,37 ℃过夜培养。
1.2.5 分析方法
假阳性率测定:将传代结束之后的发酵液系列稀释后涂布于抗性平板上,挑取平板上全部单菌落进行发酵验证NeuAc产量,产量较出发菌株并未提高的计为假阳性细胞,计算得到假阳性率。
NeuAc浓度测定:通过HPLC测定分析产物浓度,色谱柱为Aminex HPX-87H柱(300 nm×7.8 nm);流动相为10 mmol/L H2SO4溶液,流速为0.5 mL/min,NeuAc的保留时间为9.5 min。将发酵液8 000×g离心10 min后取上清液,用30 mmol/L H2SO4溶液处理混匀后过滤以测定浓度[19]。
质粒丢失率测定:将单菌落接种至100 mL液体LB培养基中,12 h后以10% 接种量接种至LB培养基中,连续培养3代,每12 h转接1次。选择3个培养时间,随机挑取100个单菌落点板转接至LB和卡纳抗性平板,比较2种平板上菌落数,计算质粒丢失率。
上述实验均设置了3组平行。
2 结果与分析
2.1 NeuAc-Biosensor调控spc和erm基因表达效果验证
利用前期成功构建的NeuAc-Biosensor质粒p136来构建的重组质粒,调控抗性基因spc和erm表达。这里使用的NeuAc-Biosensor是基于自FadR/GntR家族的转录调控因子NanR所构建[8]。NeuAc-Biosensor由2个反向连接的启动子组成,分别调控NanR基因和GFP基因,为了将荧光蛋白的表达与细胞内NeuAc联系起来,在调控表达GFP基因的不同启动子中插入NanR结合位点,从而形成动态开关,即当细胞内NeuAc浓度低时,NanR与启动子中的结合位点结合,阻止RNA聚合酶与启动子结合并抑制下游GFP基因转录;当细胞内NeuAc浓度高时,NanR的活性被NeuAc的变构调节所消除,使得启动子与NanR结合得到释放,启动子与RNA聚合酶结合并正常开启下游基因的转录[16]。考虑到产NeuAc宿主菌株中质粒带有卡纳霉素抗性基因,因此选取枯草芽孢杆菌中另外2种常用抗性基因作为调控靶基因,即壮观霉素抗性基因spc和红霉素抗性基因erm,利用NeuAc-Biosensor调控spc和erm表达,将胞内NeuAc浓度与壮观霉素或红霉素抗性相关联。将PCR扩增后的spc基因和erm以及载体质粒通过Gibson组装构建重组质粒,如图2所示。然后将质粒p136-spc,p136-erm转化入NeuAc产量不同的枯草芽孢杆菌中,分别为BS1(0.8 g/L)、BS2(1.5 g/L)、BS3(2.4 g/L),将成功转化的上述菌株挑取单菌落后进行24孔深孔板发酵,发酵培养时培养基内添加相应抗生素,以半抑制浓度来表征调控效果,结果如表3所示。

a-p136-spc重组质粒;b-p136-erm重组质粒;c-p136-spc-erm重组质粒
图1 重组抗性质粒图
Fig.1 Map of recombinant resistance plasmid
表3 不同NeuAc产量重组枯草芽孢杆菌生长的
抗生素半抑制浓度 单位:mg/L
Table 3 IC50 of different NeuAc-producing recombinant
B.subtilis
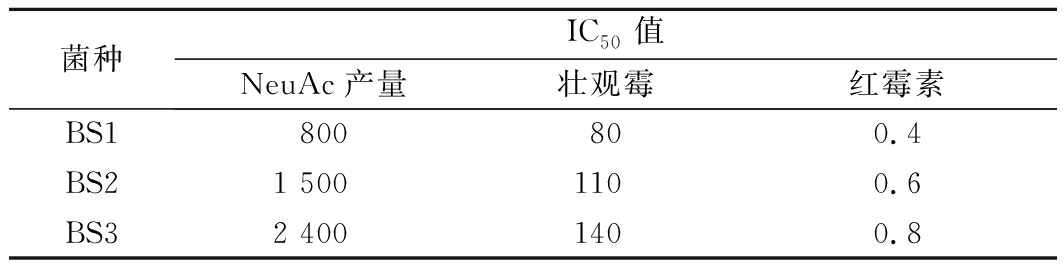
菌种IC50值NeuAc产量壮观霉红霉素BS1800800.4BS21 5001100.6BS32 4001400.8
从表3可知,高产菌株能够在较高抗生素浓度条件下生长,说明biosensor对于抗性基因有较好的调控效果。
2.2 基于适应性化的BS3菌株发酵产NeuAc
适应性进化对于提高微生物在相关培养基上的生长和抗应变能力很重要。通常,实验室中常用的定向进化方法为分批培养-连续传代,这种方法成本比较低并且可以通过深孔板等更小培养体积的设备进行大规模培养,物理化学因素也比较可控。因此在这里我们选择分批培养-连续传代培养方法。在之前的研究中,通过对2个关键前体N-乙酰氨基葡萄糖(GlcNAc)和磷酸烯醇式丙酮酸的供应途径进行模块途径工程以及平衡NeuAc的生物合成和细胞生长,NeuAc产量达到2.18 g/L [10]。为了进一步提高NeuAc产量且平衡细胞生长,我们将生物传感器与适应性进化结合,在进化过程中不断提高抗生素浓度,使得高产细胞形成生长优势,从而富集得到高产菌株。由2.1小节可知,NeuAc-biosensor对抗性基因的调控效果在菌株BS3中效果较显著,因此以带有单抗性和双抗性的BS3菌株为出发菌株,抗生素的筛选浓度梯度如表4所示,对其进行适应性进化,孔板发酵每隔24 h以10%的接种量进行取样及传代,5次传代之后结束发酵。前期研究发现,在进化过程中,小部分细胞在没有指定代谢物产生的情况下也能存活[19],这种“逃逸”是由于永久选择敏感性的突变引起的[20-21],因此我们使用了双抗性标记筛选来减少这种假阳性者的存活。经过5轮进化筛选之后,我们对3种抗性筛选平板均选取了15个单菌落进行了发酵验证,通过HPLC方法测定NeuAc浓度后发现壮观霉素和红霉素筛选分别有4个和5个菌落较出发菌株产量有提高,而双抗性筛选则有8个菌落较出发菌株产量有所提高,不同菌株的产量如图2所示。按照上述假阳性率测定方法得到单抗性筛选的假阳性率为73.3%(壮观霉素)、66.7%(红霉素),而双抗性筛选的假阳性率则为46.7%。通过单抗性标记与双抗性标记筛选的对比实验发现,双重选择能够有效减少假阳性菌株,再次体现了双抗性筛选策略的优势,同时得到1株产量为(3.16±0.19)g/L的菌株BS3C,较出发菌株产量提高了31.7%。
表4 适应性进化抗生素筛选浓度表
Table 4 Concentration of antibiotic for adaptive
evolution screening
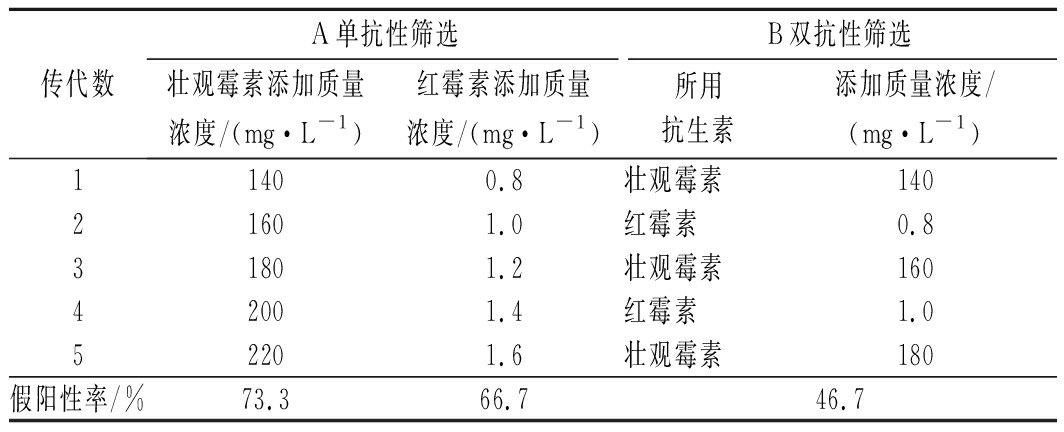
传代数A单抗性筛选B双抗性筛选壮观霉素添加质量浓度/(mg·L-1)红霉素添加质量浓度/(mg·L-1)所用抗生素添加质量浓度/(mg·L-1)11400.8壮观霉素14021601.0红霉素0.831801.2壮观霉素16042001.4红霉素1.052201.6壮观霉素180假阳性率/%73.366.746.7
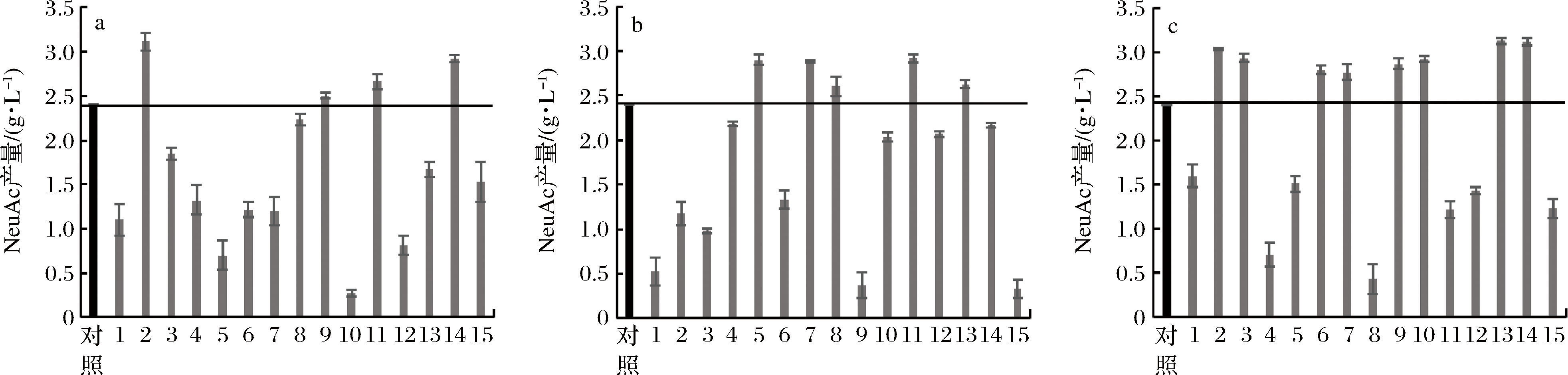
a-壮观霉素筛选产量验证;b-红霉素筛选产量验证;c-壮观霉素和红霉素筛选产量验证
图2 适应性进化后NeuAc产量验证
Fig.2 NeuAc production verification after adaptive evolution
2.3 基于对必需基因folB的改造提高质粒稳定性
抗生素抗性是重组质粒常用的筛选标记,添加抗生素可以提供选择压力,抑制质粒丢失的细胞生长。然而,抗生素在培养过程的降解会造成选择压力失效。另外,抗生素的添加还增加了成本,可能在工业环境中污染最终产品。虽然染色体整合表达具有更高的稳定性,但在许多情况下,基因整合效率低和表达强度不足阻碍了染色体整合表达的应用[22]。为了在不使用抗生素的情况下实现对质粒的选择和维持,我们敲除了枯草芽孢杆菌基因组中的必需基因folB(编码二氢喋呤醛缩酶),同时在表达NeuAc合成途径关键基因的质粒中插入必需基因folB,获得BS3C菌株,使细胞中必需基因folB的表达依赖于质粒的存在和质粒中folB表达(图3)。通过上述改造,质粒的丢失率由34.1%下降至11.8%,BS3C菌株NeuAc产量为(3.34±0.22)g/L,维持在稳定水平。上述结果说明,利用必需基因改造的方法可以有效的保证产NeuAc枯草芽孢杆菌中质粒的稳定性。

a-质粒稳定性改造策略;b-质粒稳定性改造后产量与质粒丢失率
图3 质粒稳定性改造
Fig.3 Modification of plasmid stability
3 结论
NeuAc在食品和生物医药等领域具有广泛的应用,代谢改造是提高重组枯草芽孢杆菌中NeuAc产量和降低生产成本的重要手段。本研究利用NeuAc-Biosensor调控抗生素抗性基因表达,进而通过增加抗生素浓度进行适应性进化,NeuAc合成效率提升了33.8%。进一步通过将必需基因folB插入携带NeuAc合成途径关键基因重组质粒,并且敲除基因组中的folB基因,有效降低了发酵过程中的质粒丢失率。本研究提升了重组枯草芽孢杆菌合成NeuAc的产量和稳定性,为重组枯草芽孢杆菌发酵法生产NeuAc奠定了基础。
[1] CHENG J,ZHUANG W,TANG C,et al.Efficient immobilization of AGE and NAL enzymes onto functional amino resin as recyclable and high-performance biocatalyst[J].Bioprocess and Biosystems Engineering,2017,40(3):331-340.
[2] ZHU D,ZHAN X,WU J,et al.Efficient whole-cell biocatalyst for Neu5Ac production by manipulating synthetic,degradation and transmembrane pathways[J].Biotechnology Letters,2017,39(1):55-63.
[3] LUNDGREN B R,BODDY C N.Sialic acid and N-acylsialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli[J].Organic and Biomolecular Chemistry,2007,5(12):1 903-1 909.
[4] DROUILLARD S,MINE T,KAJIWARA H,et al.Efficient synthesis of 6′-sialyllactose,6,6′-disialyllactose,and 6′-KDO-lactose by metabolically engineered E.coli expressing a multifunctional sialyltransferase from the Photobacterium sp.JT-ISH-224[J].Carbohydr Res,2010,345(10):1 394-1 399.
[5] TEN BRUGGENCATE S J,BOVEE-OUDENHOVEN I M,FEITSMA A L,et al.Functional role and mechanisms of sialyllactose and other sialylated milk oligosaccharides[J].Nutrition Reviews,2014,72(6):377-389.
[6] LIU Y,ZHU Y,MA W,et al.Spatial modulation of key pathway enzymes by DNA-guided scaffold system and respiration chain engineering for improved N-acetylglucosamine production by Bacillus subtilis[J].Metabolic Engineering,2014,24:61-69.
[7] KANG J,GU P,WANG Y,et al.Engineering of an N-acetylneuraminic acid synthetic pathway in Escherichia coli[J].Metabolic Engineering,2012,14(6):623-629.
[8] UHDE A,BRUHL N,GOLDBECK O,et al.Transcription of sialic acid catabolism genes in Corynebacterium glutamicum is subject to catabolite repression and control by the transcriptional repressor NanR[J].Journal of Bacteriology,2016,198(16):2 204-2 218.
[9] SIEDLER S,STAHLHUT S G,MALLA S,et al.Novel biosensors based on flavonoid-responsive transcriptional regulators introduced into Escherichia coli[J].Metabolic Engineering,2014,21:2-8.
[10] ZHANG X,LIU Y,LIU L,et al.Modular pathway engineering of key carbon-precursor supply-pathways for improved N-acetylneuraminic acid production in Bacillus subtilis[J].Biotechnology and Bioengineering,2018,115(9):2 217-2 231.
[11] KALIVODA K A,STEENBERGEN S M,VIMR E R,et al.Regulation of sialic acid catabolism by the DNA binding protein nanr in Escherichia coli[J].Journal of Bacteriology,2003,185(16):4 806-4 815.
[12] WANG Z,ZHUANG W,CHENG J,et al.In vivo multienzyme complex coconstruction of N-acetylneuraminic acid lyase and N-acetylglucosamine-2-epimerase for biosynthesis of N-acetylneuraminic acid[J].Journal of Agricultural and Food Chemistry,2017,65(34):7 467-7 475.
[13] SHI S,CHOI Y W,ZHAO H,et al.Discovery and engineering of a 1-butanol biosensor in Saccharomyces cerevisiae[J].Bioresource Technology,2017,245(Pt B):1 343-1 351.
[14] SANDBERG T E,SALAZAR M J,WENG L L,et al.The emergence of adaptive laboratory evolution as an efficient tool for biological discovery and industrial biotechnology[J].Metabolic Engineering,2019,56:1-16.
[15] ZHANG R,YANG Y,WANG J,et al.Synthetic symbiosis combining plasmid displacement enables rapid construction of phenotype-stable strains[J].Metabolic Engineering,2019,55:85-91.
[16] ZHANG X,CAO Y,LIU Y,et al.Development and optimization of N-acetylneuraminic acid biosensors in Bacillus subtilis[J].Biotechnology and Applied Biochemistry,2020,67(4):693-705.
[17] GIBSON D G,BENDERS G A,ANDREWS-PFANNKOCH C,et al.Complete chemical synthesis,assembly,and cloning of a mycoplasma genitalium genome[J].Science,2008,319(5867):1 215-1 220.
[18] YAN X,YU H J,HONG Q,et al.Cre/lox system and PCR-based genome engineering in Bacillus subtilis[J].Applied and Environmental Microbiology,2008,74(17):5 556-5 562.
[19] ZHAO L,TIAN R,SHEN Q,et al.Pathway engineering of Bacillus subtilis for enhanced N-Acetylneuraminic acid production via whole-cell biocatalysis[J].Biotechnology Journal,2019,14(7).DOI:10.1002/biot.201800682.
[20] RAMAN S,ROGERS J K,TAYLOR N D,et al.Evolution-guided optimization of biosynthetic pathways[J].Proceedings of the National Academy of Sciences of the United States of America,2014,111(50):17 803-17 808.
[21] NYERGES A,CSORGO B,NAGY I,et al.Conditional DNA repair mutants enable highly precise genome engineering[J].Nucleic Acids Res,2014,42(8):e62.
[22] SHUKAL S,CHEN X,ZHANG C.Systematic engineering for high-yield production of viridiflorol and amorphadiene in auxotrophic Escherichia coli[J].Metabolic Engineering,2019,55:170-178.