吡咯喹啉醌(pyrroloquinoline quinone,PQQ),作为许多细菌脱氢酶(甲醇脱氢酶,乙醇脱氢酶和葡萄糖脱氢酶等)的辅助因子于1964年首次被发现[1],它是继吡啶核苷酸和核黄素之后的第3种氧化还原酶的辅酶[2]。PQQ在生物界的分布极为广泛,在动植物和微生物细胞中均发现其存在[3],但仅有部分革兰氏阴性细菌能合成PQQ[4-6]。在具有合成PQQ能力的细菌中,绝大部分只能合成痕量的PQQ以供给细胞生长,只有少部分可以生产过量的PQQ,并将其排泄到培养基中[7-8],其中以甲基营养菌的合成能力最强,如甲基菌属(Methylobacill),甲基单胞菌属(Methylomonas),嗜甲基菌属(Methylophilus)和甲基杆菌属(Methylobacterium)等[9-12]。此外,在纳豆、青椒和猕猴桃等植物体内也含有微量的PQQ,并且牛奶和人类的母乳中也存在较高浓度的PQQ[13-16]。近几十年来的研究发现PQQ具有多种生理功能,它是许多细菌脱氢酶的辅助因子,参与细胞的电子呼吸链传递[17];PQQ可以提高微生物细胞的抗逆性,并提高宿主菌的抗氧化能力[18];PQQ还是动植物刺激或生长因子[19];此外,PQQ与葡萄糖脱氢酶(glucose dehydrogenase,GDH)结合形成PQQ-GDH全酶,可用作检测葡萄糖的生物传感器,也可用作生物燃料电池[20-21]。
由于目前PQQ化学合成步骤复杂,副产物多,而微生物发酵生产PQQ成本低,步骤少,产物分离更容易,因此微生物发酵法成为最有前景的工业化生产路线[22-23]。虽然对PQQ的研究已有几十年,但其生物合成的具体过程尚未完全阐明,从环境中筛选高产野生菌依然是必不可少的。1992年,司振军[9]筛选到1株PQQ产生菌,经鉴定为生丝微菌Hyphomicrobium sp.TK0441,优化培养基后在30 L发酵罐上发酵14 d后PQQ产量可达到1 g/L。SI等[22]从土壤中筛选到1株甲基营养菌,经鉴定为Methylobacillus sp.zju323,未经优化的PQQ摇瓶产量为23.2 mg/L,在已报道的研究中属于高水平。本研究建立了一种高效液相色谱法用来快速检测PQQ,从土壤样品中筛选得到1株PQQ高产菌,并对其进行了菌种鉴定和培养基优化,为后续进行PQQ的生产研究提供依据。
1 材料与方法
1.1 材料与试剂
细菌样品来自于无锡生产甲醇工厂附近的土壤和污水;PQQ标准品,Sigma公司;其他试剂,国药集团。
1.2 仪器与设备
G180T灭菌锅,美国致微仪器有限公司;高速冷冻离心机,美国赛默飞世尔科技公司;酶标仪,美国BioTek公司;Agilent-1260高效液相色谱仪,美国安捷伦科技公司。
1.3 培养基
筛选培养基:甲醇30 mg/L,(NH4)2SO4 3 g/L,Na2HPO4·12H2O 3 g/L,MgSO4·7H2O 1.5 g/L,KH2PO4 1.4 g/L,微量元素液 1 mL/L。固体培养基添加2%(质量分数)的琼脂粉。
种子培养基:甲醇10 mg/L,(NH4)2SO4 3 g/L,Na2HPO4·12H2O 3 g/L,MgSO4·7H2O 1.5 g/L,KH2PO4 1.4 g/L,微量元素液 1 mL/L;微量元素液配方(mg/L):CaCl2·2H2O 30,MnCl4·4H2O 5。
发酵培养基:在种子培养基的基础上加入1 mL维生素液;维生素液配方(mg/L):核黄素200,对氨基苯甲酸200,盐酸硫胺素400,烟酸400,盐酸吡哆醇400,泛酸钙400,叶酸2,生物素2,肌醇2 000。
1.4 实验方法
1.4.1 PQQ产生菌初筛方法的确立
光谱法:对PQQ标准品进行全波长扫描,发现其在249和330 nm处有2个吸收峰,将200 μL PQQ标准品溶液或发酵离心上清液转移至96孔板中,用酶标仪检测 OD330与 OD249,并将空白培养基以同样的条件处理和检测,从而减去培养基吸光值的干扰。
1.4.2 HPLC法检测PQQ
由于PQQ的结构上连有3个—COOH,因此易溶于水,极性较大,在普通的C18反相色谱柱上不保留。目前采用较多的是离子色谱对法来检测PQQ,但是离子色谱对平衡时间长,操作复杂且对柱子也有局限性;还有采用加酸调节pH来使PQQ保留在色谱柱上,但这使得峰形拖尾严重并且重现性不好。为此本实验选择亲水色谱柱来解决PQQ难以在色谱柱上保留的问题。以水为流动相,通过COSMOSIL Packed Column 5 C18-PAQ反相色谱柱,测定PQQ在249和330 nm的吸光值,根据出峰面积确定其浓度。利用安捷伦1260系列高效液相色谱仪测定PQQ含量,进样量20 μL,温度30 ℃,流动相为V(0.05 mol/L乙酸)∶V(0.05 mol/L乙酸铵)=30∶70,流速1 mL/min,在249和330 nm下检测PQQ标准品和样品含量。
1.4.3 菌株的筛选
将样品制成适当浓度梯度的稀释液,涂布于甲醇筛选培养基平板上,30 ℃培养3~5 d,分别挑取不同形态的单菌落接种到种子培养基中,30 ℃,200 r/min 培养3~5 d,将发酵液离心取上清液,用光谱法快速检测PQQ含量,再用HPLC法对PQQ产量较高的菌株进行复筛。
1.4.4 菌种的鉴定
将PQQ产量最高的菌株在固体培养基上划线,30 ℃培养3~5 d,利用普通光学显微镜观察菌体形态特征。取单菌落接种到摇瓶培养基中,30 ℃培养,当菌体OD600为0.5~1时,取3 mL发酵液,10 000 r/min 离心1 min,弃去上清液,收集菌体,按照细菌基因组DNA提取试剂盒提取DNA,利用细菌通用引物 27F:5′-AGAGTTTGATCMTGGCTCAG-3′;1492R:5′-GGTTACCTTGTTACGACTT-3′,通过PCR反应扩增该菌株的16S rDNA序列,将产物委托上海生工测序,将测序结果在NCBI网站中进行BLAST比对,用软件MEGA进行系统进化树的构建。
1.4.5 甲醇含量对菌体生长和PQQ产量的影响
对培养基中的唯一碳源甲醇进行优化,研究甲醇的不同添加量(0.5%、1%、1.5%、2%、2.5%,3%、3.5%和4%,体积分数)对菌体生长和PQQ产量的影响。
2 结果与分析
2.1 光谱法检测PQQ
研究建立了基于光谱法检测PQQ的快速筛选方法,通过全波长扫描发现PQQ在249和330 nm左右有2个吸收峰。考虑到发酵液中含有蛋白,核酸等在249 nm左右有吸收值的物质,而在330 nm处有吸收值的物质较少,因此,本实验选择研究PQQ浓度和OD330的关系。通过酶标仪检测不同浓度 PQQ标准品的OD330(检测样品时要减去空白发酵培养基的吸光值以除去干扰),发现在330 nm下PQQ浓度与OD330关系为y=0.021 8x-0.035 4(R2=0.995 7)。当PQQ质量浓度在2.5~50 mg/L时,两者呈现良好的线性关系,由于野生菌产PQQ量多在1~20 mg/L左右,因此使用本方法进行高通量筛选的初筛具有良好的可行性。
2.2 HPLC法检测PQQ
本实验首先研究了其他色谱柱检测PQQ,结果发现普通C18柱无法使PQQ有效保留,更换流动相甲醇和乙腈,或者是梯度洗脱都不能保留PQQ。另外发现改变pH可以使PQQ有所保留,但峰形较差,且拖尾严重,并且不能很好地分离样品发酵液中的PQQ。考虑到PQQ极性较大,因此采用耐100%水的COSMOSIL Packed Column 5C18-PAQ反相色谱柱来使PQQ有很好的保留。将准备好的7个不同质量浓度的PQQ标准品(100、50、25、12.5、6.125、5和2.5 mg/L)进行HPLC检测,每个质量浓度连续进样3次,每次进样20 μL。结果如图1所示,不同浓度的PQQ均在3.56 min左右出峰。随着PQQ浓度的增加,峰面积也逐渐增加。因此PQQ浓度与峰面积呈正相关的关系。以PQQ浓度为横坐标,色谱峰面积为纵坐标,绘制标准曲线,线性关系为y=30.27x+7.32,R2=0.999 8,说明线性关系良好。采用COSMOSIL Packed Column 5C18-PAQ反相色谱柱检测PQQ,运行时间短,PQQ出峰时间早,检测效率高。本实验建立的HPLC法检测PQQ具有良好的可行性,可作为菌种产PQQ的复筛方法。
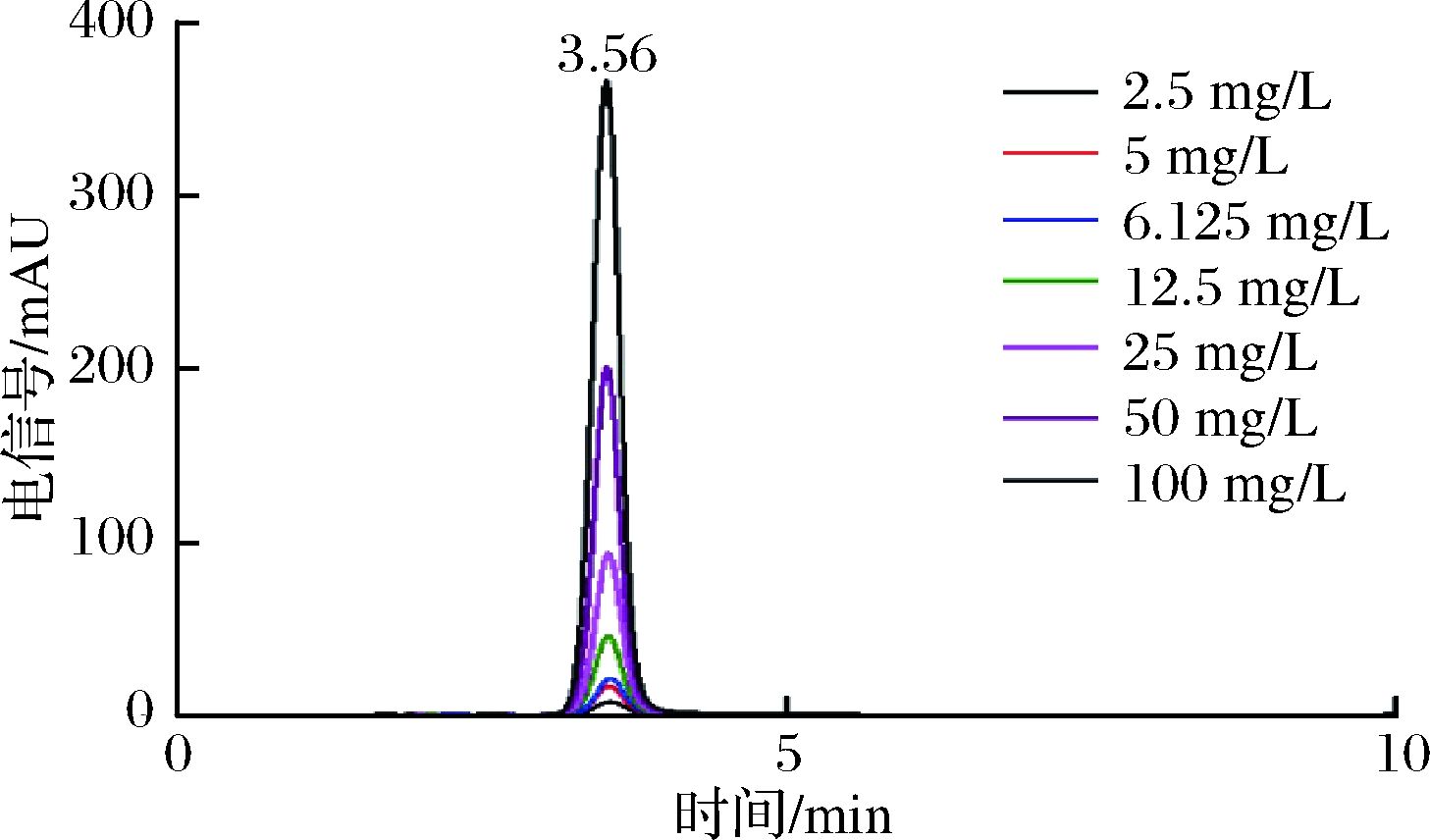
图1 不同浓度PQQ标准品的HPLC测定
Fig.1 The HPLC determination of PQQ standards with different concentrations
2.3 菌株的筛选和鉴定
经光谱法初筛,从100多个样品中筛选到50多株PQQ产生菌,大多数的产量在2~5 mg/L,少数可以达到10 mg/L左右,HPLC复筛后其中PQQ产量最高的1株为20.6 mg/L,峰图如图2所示,PQQ在3.57 min出峰,这是未经优化已报道的摇瓶较高产量。
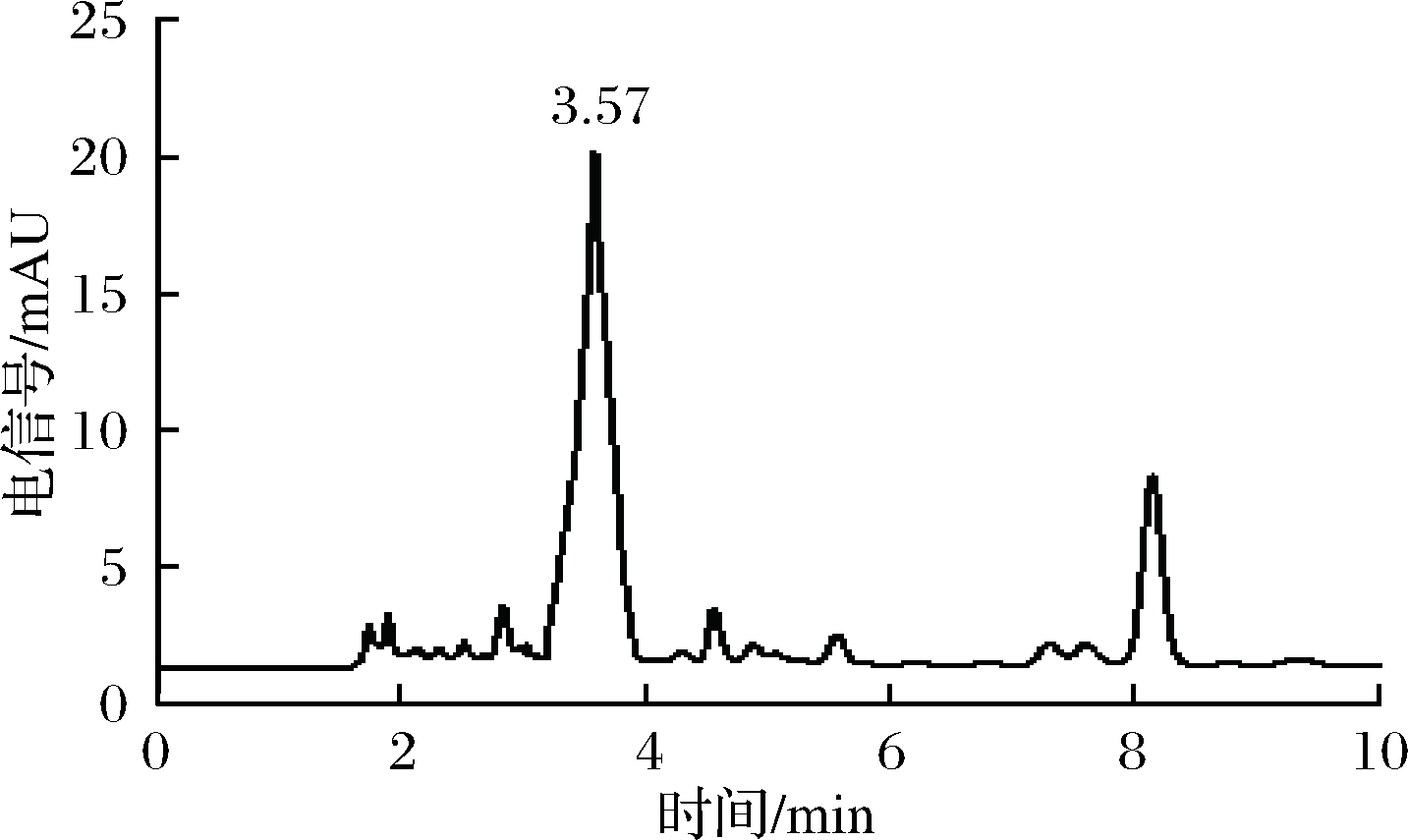
图2 Methylopila sp.Z1发酵液的HPLC图
Fig.2 The HPLC chart of the fermentation broth of
Methylopila sp.Z1
该菌株的菌落在甲醇培养基平板上为乳白色,黏稠,边缘整齐,直径较小,为1~2 mm。菌体细胞为球状,革兰氏阴性,不产芽孢,有荚膜,无鞭毛。该菌株在以甲醇和甲胺为碳源的培养基中能够良好生长,能利用D-葡萄糖,蔗糖,果糖和木糖。在以铵盐、硝酸盐、牛肉膏、蛋白胨和酵母膏为氮源的培养基中能良好生长。采用PCR技术扩增该菌株的16S rDNA 基因序列,经上海生工测序。通过在 NCBI网站中进行同源性序列搜索及比对,从中选取同源性较高菌株的16S rDNA基因序列,以集团外菌种作对照,通过MEGA软件,用邻接法(Neighbor-Joining)构建系统发育树。结果如图3所示,该菌株与 Methylopila henanensis strain LYBFD3-16A2的同源性最高,为99%。综合菌落及菌体形态特征、16S rDNA序列分析, 鉴定该菌株为Methylopila sp.菌株,命名为Methylopila sp.Z1。
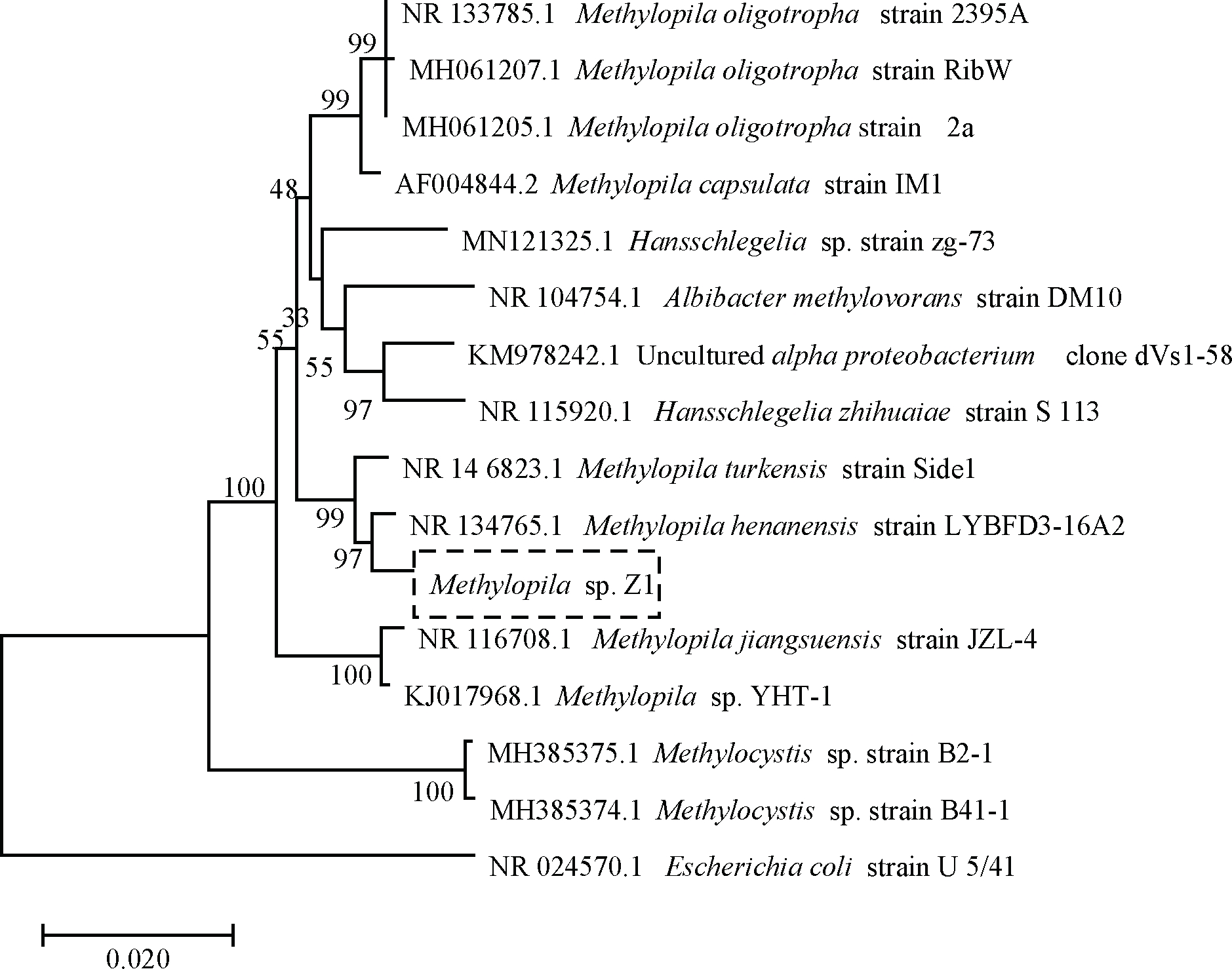
图3 Methylopila sp. Z1的系统发育树
Fig.3 The phylogenetic tree of Methylopila sp.Z1
2.4 甲醇含量的优化
研究甲醇的不同添加量(0.5%、1%、1.5%、2%、2.5%、3%、3.5%和4%,体积分数)对OD600和PQQ产量的影响,结果如图4所示。在0.5%~2.5%,随着甲醇体积分数的提高,菌体OD600和PQQ产量也逐渐提高,当甲醇体积分数为2.5%时,OD600和PQQ产量均达到最高,继续增加甲醇,OD600和PQQ产量均有明显下降。这是因为甲醇作为细菌生长的唯一碳源,并且还是细菌细胞甲醇脱氢酶的底物,在其浓度较低时,不足以提供能量使细胞生长,也无法生产过量的PQQ排泄到培养基中,导致PQQ合成效率非常低。当甲醇体积分数逐渐提高时,细胞所能利用的碳源增加,菌体OD600和PQQ产量也逐渐增加;当甲醇体积分数超过2.5%时,由于甲醇的毒性使细胞损伤,并且底物浓度过高抑制甲醇脱氢酶的活性,导致菌体OD600和PQQ产量下降。因此甲醇体积分数为2.5%对于菌体生长和PQQ合成是最佳选择。
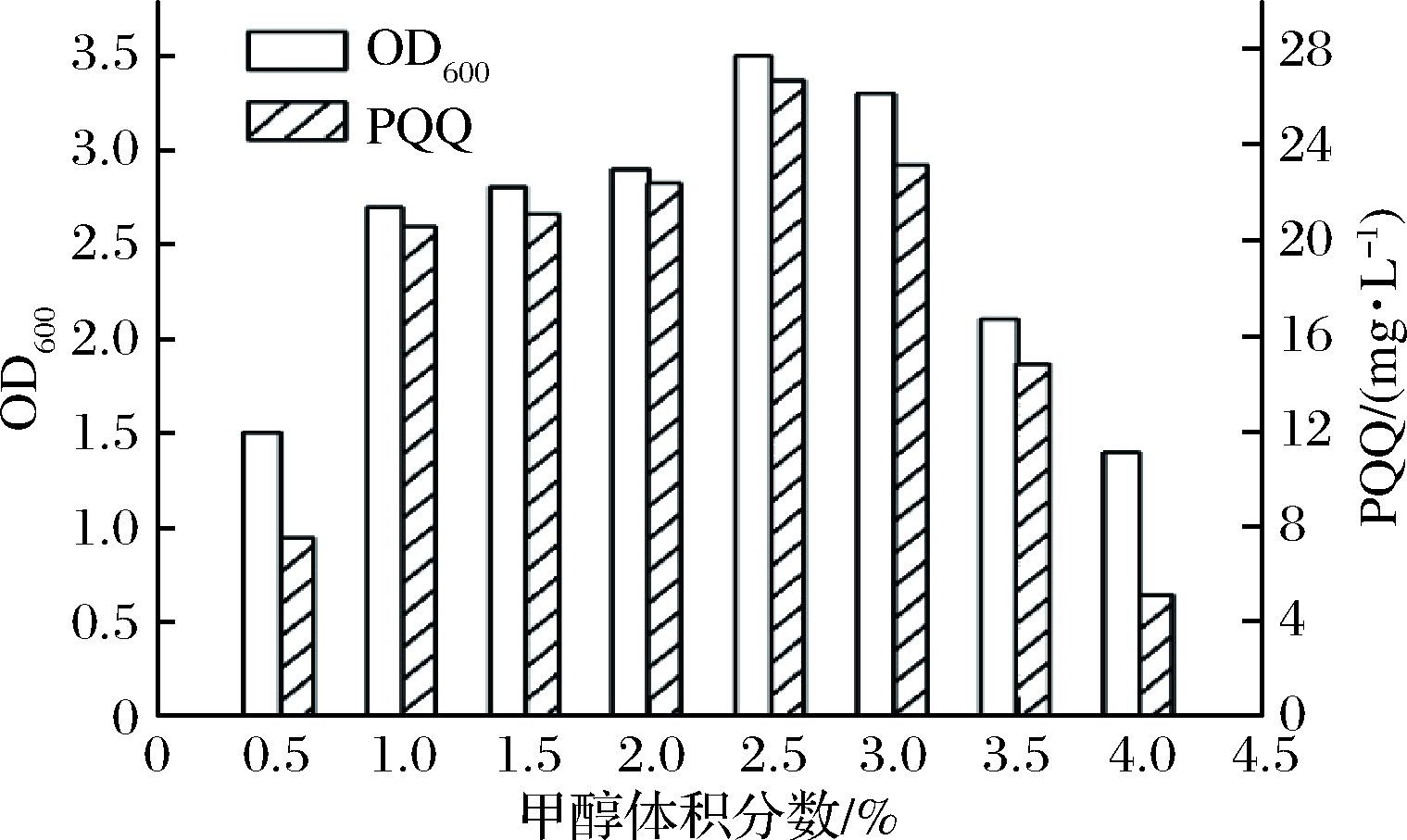
图4 不同甲醇浓度对PQQ合成和生物量的影响
Fig.4 Effect of different methanol concentration on PQQ synthesis and biomass
3 结论
目前PQQ的检测方法主要有重组酶法,氧化还原法,光谱法和HPLC[24],其中重组酶法所用到的葡萄糖脱氢酶提取繁琐,纯化过程复杂,且得到的酶活不高,不足以支持大量的菌种筛选;氧化还原法中用到的显色剂氯化硝基四氮唑蓝易受培养基中其他成分的干扰,造成阴性结果。光谱法操作简单,检测快速,但也可能受到培养基中具有相似波长吸光度的影响,但本实验采用的是甲醇无机培养基,并通过HPLC检测样品发现基本没有干扰,因此光谱法作为PQQ产生菌的初筛是高效可行的。本实验还建立了新的HPLC法检测PQQ,采用亲水色谱柱COSMOSIL Packed Column 5C18-PAQ,使得PQQ有很好的保留,并且样品运行时间短,PQQ在3.56 min出峰,出峰时间早,检测时间短,峰形无拖尾,为PQQ检测提供一种新的改进方法。
由于PQQ的生物合成机理没有得到完全阐释,通过基因工程和代谢改造提高PQQ的产量远低于野生菌的产量,因此从环境中筛选具有高产PQQ能力的菌种是十分必要的。本实验从样品中筛选到1株PQQ高产菌,经鉴定为Methylopila sp.,命名为Methylopila sp.Z1,未经优化的摇瓶产量达到20.6 mg/L,属于已报道的较高水平。Z1菌株耐受较高甲醇,在培养基甲醇体积分数为2.5%时,PQQ产量达26.7 mg/L,相比于初始浓度提高了28%。Z1是一株有高产PQQ潜力的菌株,后续发酵罐培养和优化工艺将进一步提高PQQ产量。
[1] KLINMAN J P, BONNOT F.Intrigues and intricacies of the biosynthetic pathways for the enzymatic quinocofactors:PQQ, TTQ, CTQ, TPQ, and LTQ [J].Chemical Reviews, 2014, 114:4 343-4 365.
[2] WANG C X, ZHANG B R, ZHANG H Y, et al.Effect of dietary pyrroloquinoline quinone disodium in sows on intestinal health of the offspring [J].Food & Function, 2020, 11:7 804-7 816.
[3] LISDAT F.PQQ-GDH-structure, function and application in bioelectrochemistry [J].Bioelectrochemistry, 2020, 134:107 496.
[4] MATSUTANI M, YAKUSHI T.Pyrroloquinoline quinone-dependent dehydrogenases of acetic acid bacteria [J].Applied Microbiology and Biotechnology, 2018, 102:9 531-9 540.
[5] AKAGAWA M, NAKANO M, IKEMOTO K.Recent progress in studies on the health benefits of pyrroloquinoline quinone [J].Bioscience, Biotechnology, and Biochemistry, 2016, 80:13-22.
[6] KELTIENS J T, POL A, REIMANN J, et al.PQQ-dependent methanol dehydrogenases:Rare-earth elements make a difference.[J].Applied Microbiology and Biotechnology, 2014, 98:6 163-6 183.
[7] MCINTIRE W S, WEVLER W.Factors affecting the production of pyrroloquinoline quinone by the methylotrophic bacterium W3A1[J].Applied and Environmental Microbiology, 1987, 53(9):2 183-2 188.
[8] TAKEDA K, MATSUMURA H, ISHIDA T, et al.The two-step electrochemical oxidation of alcohols using a novel recombinant PQQ alcohol dehydrogenase as a catalyst for a bioanode[J].Bioelectrochemistry, 2013, 94:75-78.
[9] 司振军. 微生物高效生产吡咯喹啉醌和聚苹果酸的研究[D]. 杭州: 浙江大学, 2017.
SI Z J. Research on the efficient production of pyrroloquinoline quinone and polymalic acid by microorganisms[D]. Hangzhou: Zhejiang University, 2017.
[10] 杨璐, 熊向华, 汪建华, 等.吡咯喹啉醌研究进展[J].生物技术通讯, 2009,20(6):874-879.
YANG L, XIONG X H, WANG J H, et al.Research progress of pyrroloquinoline quinone[J].Biotechnology Communications, 2009,20(6):874-879.
[11] ZHU W, KLINMAN J P.Biogenesis of the peptide-derived redox cofactor pyrroloquinoline quinone[J].Current Opinion in Chemical Biology, 2020, 59:93-103.
[12] YAKUSHI T, MATSUSHITA K.Alcohol dehydrogenase of acetic acid bacteria:Structure, mode of action, and applications in biotechnology[J].Applied Microbiology and Biotechnology, 2010, 86:1 257-1 265.
[13] 尹芳, 陆兵, 陈国豪, 等.甲醇利用型细菌发酵生产吡咯喹啉醌的培养条件[J].华东理工大学学报, 2004(2):227-229;233.
YIN F, LU B, CHEN G H, et al.Culture conditions for the fermentation of methanol-utilizing bacteria to produce pyrroloquinoline quinone[J].Journal of East China University of Science and Technology, 2004(2):227-229;233.
[14] 徐文, 许然, 张利平, 等.甲醇利用型吡咯喹啉醌产生菌的筛选及鉴定[J].生物技术通报,2013(1):162-165.
XU W, XU R, ZHANG L P, et al.Screening and identification of methanol-utilizing pyrroloquinoline quinone producing bacteria[J].Biotechnology Bulletin, 2013(1):162-165.
[15] 李红月, 曾伟主, 周景文, 等.高产吡咯喹啉醌扭脱甲基杆菌的高通量选育[J].生物工程学报,2018,34(5):794-802.
LI H Y, ZENG W Z, ZHOU J W, et al.High-throughput breeding of high-yield pyrroloquinoline quinone Methylobacter henryi[J].Chinese Journal of Biotechnology,2018,34(5):794-802.
[16] YAKUSHI T, TAKAHASHI R, MATSUTANI M, et al.The membrane-bound sorbosone dehydrogenase of Gluconacetobacter liquefaciens is a pyrroloquinoline quinone-dependent enzyme[J].Enzyme and Microbial Technology, 2020, 137:109 511.
[17] QIN R, SUN J Y, WU J, et al.Pyrroloquinoline quinone prevents knee osteoarthritis by inhibiting oxidative stress and chondrocyte senescence [J].American Journal of Translational Research, 2019, 11:1 460-1 472.
[18] 杨亚欣, 刘天, 李彤, 等.一株吡咯喹啉醌产生菌的筛选和初步鉴定[J].生物技术通讯,2018,29(5):609-612;624.
YANG Y X, LIU T, LI T, et al.Screening and preliminary identification of a strain producing pyrroloquinoline quinone[J].Biotechnology News,2018,29(5):609-612;624.
[19] 杨诗颖, 陈佳, 柯崇榕, 等.吡咯喹啉醌产生菌的筛选及其培养基优化[J].药物生物技术, 2014, 21(5):429-432.
YANG S Y, CHEN J, KE C R, et al.Screening of pyrroloquinoline quinone producing bacteria and optimization of its medium[J].Pharmaceutical Biotechnology, 2014, 21(5):429-432.
[20] SI Z J, MACHAKU D, WEI P, et al.Enhanced fed-batch production of pyrroloquinoline quinine in Methylobacillus sp.CCTCC M2016079 with a two-stage pH control strategy [J].Applied Microbiology and Biotechnology, 2017, 101:4 915-4 922.
[21] NAKASHIMA Y, MIZOSHITA N, TANAKA H, et al.Amphiphilic polymer mediators promoting electron transfer on bioanodes with PQQ-Dependent glucose dehydrogenase[J].Langmuir, 2016, 32:12 986-12 994.
[22] SI Z J, ZHU J Z, WANG W G, et al.Novel and efficient screening of PQQ high-yielding strains and subsequent cultivation optimization[J].Applied Microbiology and Biotechnology, 2016, 100:10 321-10 330.
[23] DUINE J A, JONGEIAN J A.Quinoproteins, enzymes with pyrrolo-quinoline quinone as cofactor[J].Annual Review of Biochemistry, 1989, 58:403-426.
[24] 杨延新, 熊向华, 游松, 等.3种检测吡咯喹啉醌的方法比较[J].生物技术通讯, 2011,22(4):544-547.
YANG Y X, XIONG X H, YOU S, et al.Comparison of three methods for detecting pyrroloquinoline quinone[J].Biotechnology Communications, 2011,22(4):544-547.