在食品加工的过程中,原料带菌和加工环境、设备染菌都可能导致食品在加工过程中污染腐败微生物从而影响食品质量和货架期品质[1-2]。腐败微生物对食品品质和食品安全的影响包括降解食品中蛋白质和脂肪等营养成分,产生气体引起包装胀起或出现异味,产生有机碱、细菌毒素等有毒物质,分解食品中淀粉类大分子物质改变食品的物理性状等。对腐败微生物进行分离培养,采用传统的分离培养方法一步分离可以获得主要的腐败菌[3-6],但是对不可培养微生物或者难培养菌,则需要全面的分析才能制定培养条件。因此,可以通过宏基因组测序技术揭示食品原料和食品加工过程中各类半成品以及成品中的微生物组成,可为识别潜在的食品腐败菌、降低腐败微生物对食品品质的影响,开发或制定有效的预防措施提供参考。通过宏基因组测序技术对奶粉加工过程物料中菌落组成的动态变化分析发现,奶粉中腐败菌最初为嗜冷的假单胞菌(Pseudomonas)和不动杆菌(Acinetobacter),然后演变为嗜热的栖热菌(Thermus)和地芽孢杆菌(Geobacillus)。这为明确奶粉加工过程不同阶段需重点监控的影响食品品质的细菌提供了依据[7]。对成品腐乳中的微生物组成结构的分析表明,假单胞菌是腐乳中普遍存在的腐败菌,而对风味有重要影响的德巴利氏酵母菌(Debaryomyces)可能会引起胀罐[8]。将宏基因组测序技术与传统的菌株分离培养技术相结合,发现破布子乳杆菌(Lactobacillus pobuzihii)是造成酱油胀罐的微生物[9]。此外,通过微生物组成分析发现,四川泡菜母水中的主要真菌哈萨克斯坦酵母菌(Kazachstania)可能对泡菜品质造成不良影响[10]。
蚝油是一种以蚝汁为基础原料,通过加入食盐、味精、酱油等调味料和增稠剂制成的复合调味品[11-12]。蚝油的生产过程主要分为4个流程:基础原料混合,加入香鲜调味剂,灭菌,灌装。淀粉液化分层是蚝油产品变质的表现形式之一,这有可能是贮存条件不当导致的水分析出,也可能是蚝油生产原料中含有产淀粉酶的耐热菌株[13],在条件合适时生长或繁殖从而水解蚝油中的淀粉,导致蚝油液化,影响产品后期品质 [14-16]。因此,本研究将通过宏基因组测序技术对蚝油、生产原料和物理状态发生改变的蚝油半成品中的细菌组成进行解析,分离并鉴定淀粉利用菌株,为蚝油及其他含淀粉调味品中腐败菌的溯源提供参考。
1 材料与方法
1.1 材料与试剂
蚝油(国内某品牌)购于超市。蚝油生产用原料蚝汁、香精和变性淀粉购于国内相应原料供应企业。
土壤基因组抽提试剂盒,美国MoBio公司;细菌基因组DNA快速抽提试剂盒、PCR引物,生工生物工程(上海)股份有限公司;pfu PCR Master Mix,上海科晴生物技术有限公司。
营养肉汤(g/100mL):蛋白胨1.0、牛肉膏0.3、氯化钠0.5,pH 7.4,用于固体培养时添加琼脂粉2.0。
1.2 仪器与设备
恒温培养箱,常州首创仪器设备公司;PCR仪,美国Bio-Rad公司;UVmini-1280分光光度计,日本Shimadzu公司;SynergyH4/Elx800 酶标仪,美国Bio Tek公司。
1.3 试验方法
1.3.1 细菌基因组DNA制备与测序
样品细菌基因的提取采用液氮研磨与试剂盒抽提相结合的方法。将样品混合均匀后,称取10.0 g至研砵中,加入液氮充分研磨、破碎细胞。使用MoBio公司的Power® Soil DNA Isolation Kit试剂盒提取并纯化宏基因组DNA[17]。将达到标准的宏基因组DNA用16S rDNA通用引物341F、908R(表1)扩增[18],扩增产物送往生工生物工程(上海)股份有限公司Illumina MiSeq平台测序。
表1 本研究所用引物序列
Table 1 Primers used in this work

引物名称序列341F5'-CCTACGGGNGGCWGCAG-3'908R5'-CCGTCAATTCMTTTGAGTTT-3'27F5'-AGAGTTTGATCCTGGCTCAG-3'1492R5'-GGTTACCTTGTTACGACTT-3'
1.3.2 测序数据处理与分析
获得测序原始数据后,先对reads拼接并去除嵌合体,根据Barcode使数据回归样品并删除片段长度小于200 bp、单碱基重复超过8个、含模糊碱基的序列,以得到高质量序列文件。再使用核糖体数据库项目(ribosomal database project,RDP)classifier贝叶斯算法对97%相似度水平的分类操作单元(operational taxonomic unit,OTU)代表序列进行分类学分析,并在门纲目科属种水平统计样品中的菌落组成[19]。最后利用MOTHUR软件对样品微生物群落进行Alpha多样性分析等[20]。
1.3.3 半成品中淀粉利用微生物的分离与初步鉴定
通过将蚝汁和香精分别与淀粉混合制备成蚝油半成品,验证原料中是否存在可使蚝油中淀粉水解的菌株。具体操作为:(1)蚝汁与淀粉混合物(mixture of oyster sauce and starch,MOS):蚝汁常压煮沸30 min,与煮沸15 min的改性淀粉(糊化淀粉)混合;(2)香精与淀粉混合物(mixture of essence and starch,MES):香精煮沸10 min,与糊化淀粉混合。将MOS和MES置于室温培养,待出现淀粉水解现象后取样。使用梯度稀释涂布平板法对水解样品中的微生物进行分离,于37 ℃培养24 h并观察菌落形态。
用生工生物工程(上海)股份有限公司的细菌基因组DNA提取试剂盒提取细菌基因组DNA。利用通用引物27F和1492R扩增其16S rDNA,PCR反应体系与反应条件具体为[21]:在50 μL体系中,含DNA模板2 μL(50 ng/μL),pfu PCR MasterMix 25 μL,ddH2O 21 μL,上下游引物各1 μL(10 μmol/L)。PCR 扩增条件:94 ℃ 3 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃ 10 min。测序所得的16S rDNA序列与美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库进行BLAST比对,用MEGA 7.0 以Neighbour-Joining法构建系统发育树。
1.3.4 样品中代表性菌株计数与淀粉水解能力分析
分别取蚝油、蚝汁、香精、MOS和MES淀粉水解样品,梯度稀释后,取10-2、10-3、10-4、10-5稀释度的菌液涂布,于37 ℃培养24 h后按照代表性菌株形态分别进行计数。
采用碘比色法测定菌株产淀粉酶水平[22]。将待检测菌株接种到营养肉汤培养基中37 ℃培养至OD600=1.0,离心取30 μL发酵上清液,加入70 μL 0.5 g/L可溶性淀粉溶液(pH 6.0、0.1 mol/L PBS溶液配制)于60 ℃下保温10 min,加入50 μL 5 mol/L HCl溶液灭活,再添加50 μL碘液显色。空白对照为加入50 μL 5 mol/L HCl灭活后再添加反应底物,其他同实验组一致。测定660 nm处吸光值。
淀粉酶活力单位定义(U/mL):在60 ℃条件下,1 min水解淀粉1 μg所用酶量为1个酶活力单位。淀粉酶酶活力根据公式(1)计算:
淀粉酶酶活力/(U·mL-1)
(1)
式中:淀粉消耗量,mg;反应时间为10 min;发酵上清液体积为30 μL。
2 结果与分析
2.1 蚝油及原料中细菌Alpha多样性分析
使用ACE、Chao1、Shannon及Simpson指数对蚝油及原料中的生物群落的丰度及多样性进行评估。由表2可知,香精中细菌种类的ACE、Chao 1、Shannon指数均为最高,且Simpson指数也较高,因此香精中细菌组成最丰富。蚝汁和成品蚝油中的微生物多样性低于香精。蚝汁与香精这2种原料分别与淀粉混合制得的蚝油半成品中物种丰度明显降低,生物群落也趋于简单,表明加工过程中的高温处理能够杀灭原料中的部分微生物。
表2 各样品菌群的Alpha多样性比较
Table 2 Alpha diversity of bacterial flora of samples
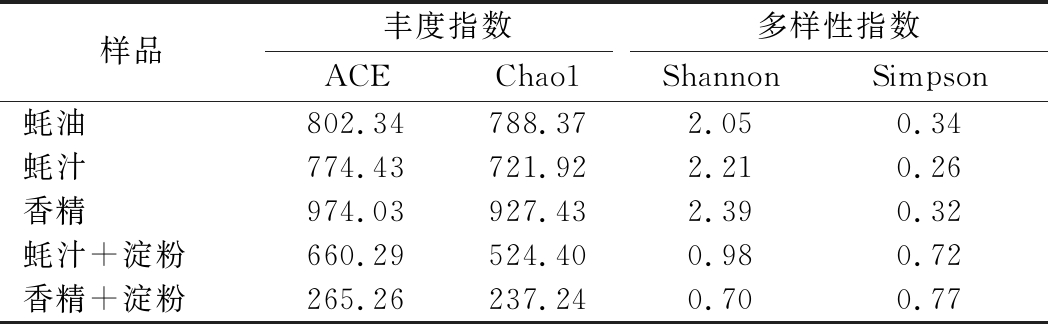
样品丰度指数多样性指数ACEChao1ShannonSimpson蚝油802.34788.372.050.34蚝汁774.43721.922.210.26香精974.03927.432.390.32蚝汁+淀粉660.29524.400.980.72香精+淀粉265.26237.240.700.77
注:“蚝汁+淀粉”、“香精+淀粉”分别指MOS、MES水解样品
2.2 蚝油及原料中细菌组成分析
成品蚝油中的细菌分布于20个门,其中厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为主要细菌,它们的相对丰度之和占总细菌的98.5%(图1-a)。蚝汁和香精中细菌种类分别包括18个门和13个门,厚壁菌门和变形菌门的微生物是蚝汁和香精中的主要细菌,它们在两类样品中的相对丰度之和>97%(图1-b)。蚝汁和香精中分别有131和144个属的细菌,其中两者相同的细菌属有82个(图2)。2种原料中优势微生物(相对丰度>5%)的组成基本相似,在属水平上相对丰度>1%的细菌包括动性微菌属(Planomicrobium)、芽孢杆菌属(Bacillus)、类芽孢杆菌属(Paenibacillus)、四联球菌属(Tetragenococcus)、乳杆菌属(Lactobacillus)和大肠埃希菌-志贺菌属(Escherichia-Shigella)(图1-b)。以上细菌属在蚝汁中的丰度占比分别为45.83%、13.63%、4.58%、15.35%、11.66%和1.67%,而在香精中分别为55.03%、20.36%、6.88%、1.99%、1.58%和1.95%。成品蚝油中有178个细菌属,其优势细菌属的种类和丰度与2种原料有较大相似性。动性微菌属、芽孢杆菌属、类芽孢杆菌、四联球菌属和大肠埃希菌-志贺菌属相对丰度占比依次为55.17%、13.78%、4.84%、1.18%和1.08%。蚝油中的独特性菌属为黄单胞菌(Xanthomonas)和微小杆菌(Exiguobacterium),分别占16.49%与1.22%。香精中独特性菌属为狭义梭菌属(Clostridium_sensu_stricto_1),相对丰度占4.79%。由此可知,假单胞菌和葡萄球菌等常见的蚝肉腐败菌在蚝汁中占比小,而具有一定耐低温能力的酸败菌——乳杆菌,是蚝汁中的优势菌属[23-24]。
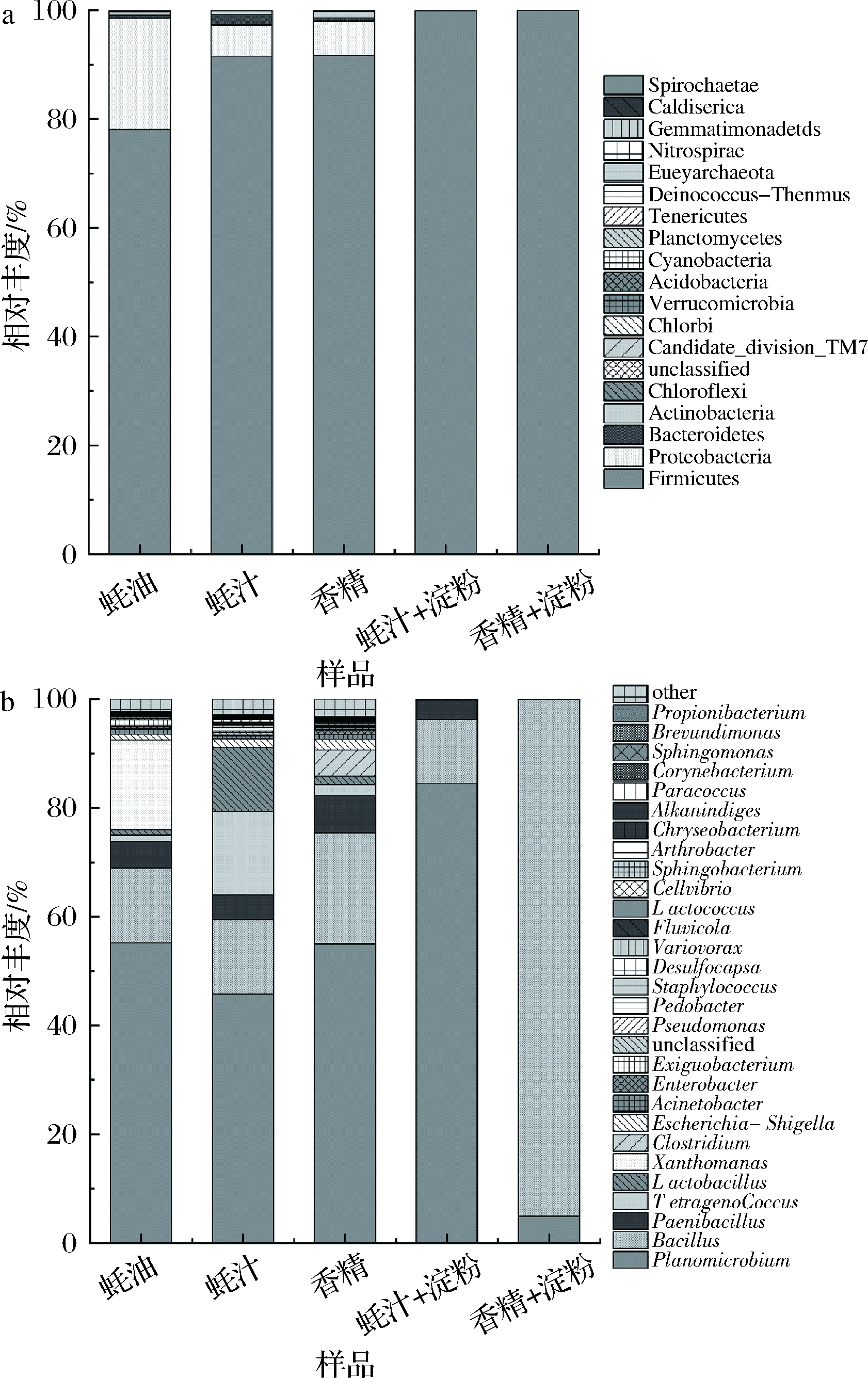
a-细菌门水平组成;b-细菌属水平组成
图1 蚝油、原料及加工后原料样品中的细菌组成
Fig.1 Bacterial composition of oyster sauce, raw materials and treated samples
注:图1-b中相对丰度(>0.01%)前29个属单独显示,其他的
合并为“other”;Clostridium为Clostridium_sensu_stricto_1;
“蚝汁+淀粉”、“香精+淀粉”分别指MOS、MES水解样品
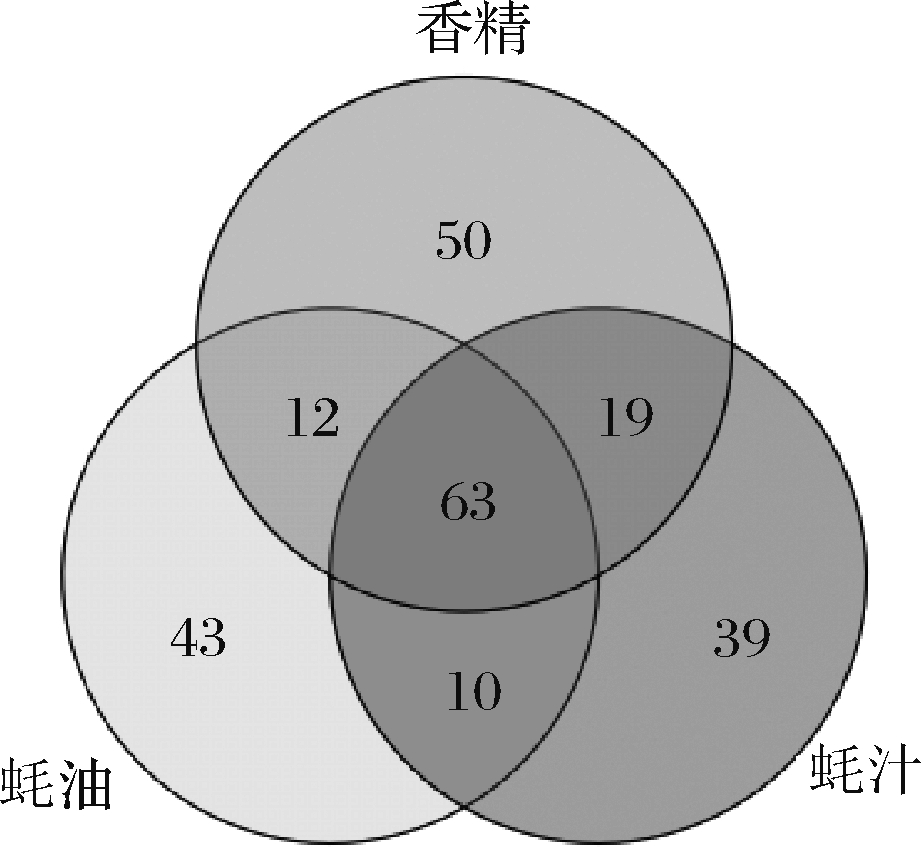
图2 蚝油及原料样品中细菌属数量的韦恩图
Fig.2 Venn diagram of phylum number of oyster sauce and raw materials
对蚝汁和香精分别与糊化淀粉混合后发生淀粉水解的混合物MOS、MES中的微生物群落结构分析表明,由于原料经过煮沸处理,原料中含有的大多数细菌已被杀灭;MOS和MES中分别仅包含12个和3个细菌属,两者中的优势细菌属为动性微菌和芽孢杆菌。MOS中动性微菌和芽孢杆菌相对丰度分别为84.46%和11.73%,在MES中这2个菌属的相对丰度分别为4.95%和95.01%。芽孢杆菌在MES中的相对丰度大幅增加并成为绝对优势细菌属,这可能对蚝油因淀粉水解而发生物理性状改变有较大影响。
2.3 代表性菌株的分离与初步鉴定
为了进一步分析在蚝油加工过程中可能污染的能水解产品中淀粉的菌株,对MOS和MES淀粉水解样品中的细菌进行了分离,共获得6株形态不同的代表性菌株S1、S2、S3、S4、S5、S6,均为革兰氏阳性菌(表3)。将6个代表性菌株的16S rDNA序列与GenBank数据库进行同源性比对[25],初步将6株菌鉴定为人葡萄球菌(Staphylococcus hominis)S1,贝莱斯芽孢杆菌(Bacillus velezensis)S2和S4,枯草芽孢杆菌(Bacillus subtilis)S3和S5, 皮特不动杆菌(Acinetobacter pitti)S6(图3)。虽然动性微菌(Planomicrobium)也是蚝油及其原料中的优势菌属之一,但由于它是严格厌氧菌且不具有淀粉水解能力[26],本研究未对其进行分离和开展后续研究。
表3 代表菌株的菌落特征及菌体形态
Table 3 Colony characteristics and morphology of representative strains

菌名菌落边缘菌落表面凸起菌落质地菌体形态菌落颜色透明度S1规则 光滑微凸起湿润球形浅黄不透明S2不规则粗糙微凸起干燥短杆浅黄透明 S3不规则粗糙扁平 干燥长杆浅黄不透明S4规则 光滑微凸起干燥短杆浅黄透明 S5不规则粗糙扁平 干燥短杆灰白不透明S6规则 粗糙微凸起湿润球形浅黄不透明

图3 基于16S rDNA序列构建的代表性菌株的系统发育树
Fig.3 Phylogenetic tree constructed based on the sequences of 16S rDNA of representative strains
2.4 代表性菌株在样品中的数量分析与淀粉利用能力分析
2.2结果表明,蚝油产品中的微生物主要来源于加工的原料蚝汁和香精。分离自MOS和MES淀粉水解样品的代表性菌株是否存在于蚝油中和具有利用淀粉的能力,还需进一步确认。根据表3代表性菌株的菌落形态特征和图3中菌种鉴定结果,选择对4个代表所分离菌株全部属种的S.hominis S1、B. velezensis S2、B. subtilis S3、A. pitti S6分析它们在蚝油、原料和淀粉水解混合物MOS与MES中的数量变化。由图4-a可以看出,蚝油和蚝汁中均含有4个代表性菌株,香精中含有除A. pitti S6外的其他3个代表性菌株。蚝油和原料中4个代表性菌株的含量均低于105 CFU/mL,当蚝汁或香精与糊化淀粉混合物出现淀粉水解现象时,葡萄球菌和芽孢杆菌的数量均显著增加,其中S.hominis S1数量增加最多,分别从2.6×103、3.6×103 CFU/mL增加至4×107、8.5×106 CFU/mL;B. velezensis S2 的数量分别从1.2×103、1.7×104 CFU/mL增加至4×106、5×105 CFU/mL;蚝汁中含有的B. subtilis S3在MOS水解样品中低于检测限,这可能是因为S3在蚝汁中含量较低,通过加工过程处理已基本杀灭,不会进一步增殖;MES中未计出A. pitti S6的数量,说明此菌与淀粉水解没有较大关系。由此可见,生产蚝油用原料中含有的葡萄球菌和芽孢杆菌可能与淀粉水解密切相关。
为了评估从蚝油半成品淀粉被水解的混合物MOS、MES中分离到的代表性菌株是否具有水解淀粉的能力,通过分析菌株产淀粉酶水平比较它们对淀粉的水解能力。由图4-b可以看出,芽孢杆菌S5和A.pitti S6淀粉酶活性极低,而葡萄球菌S1和芽孢杆菌S2、S3、S4都具有一定淀粉利用能力,其中芽孢杆菌S3产淀粉酶水平最高。依据菌株产淀粉酶活力水平,判断它们的水解淀粉能力由高到低依次为S3、S2、S4、S1、S5、S6。结合蚝油及其原料中微生物组成分析和代表性菌株数量分析的结果,推断芽孢杆菌和葡萄球菌是来源于蚝油生产原料并可使蚝油物理状态发生改变(淀粉水解后水化)的潜在危害性细菌。
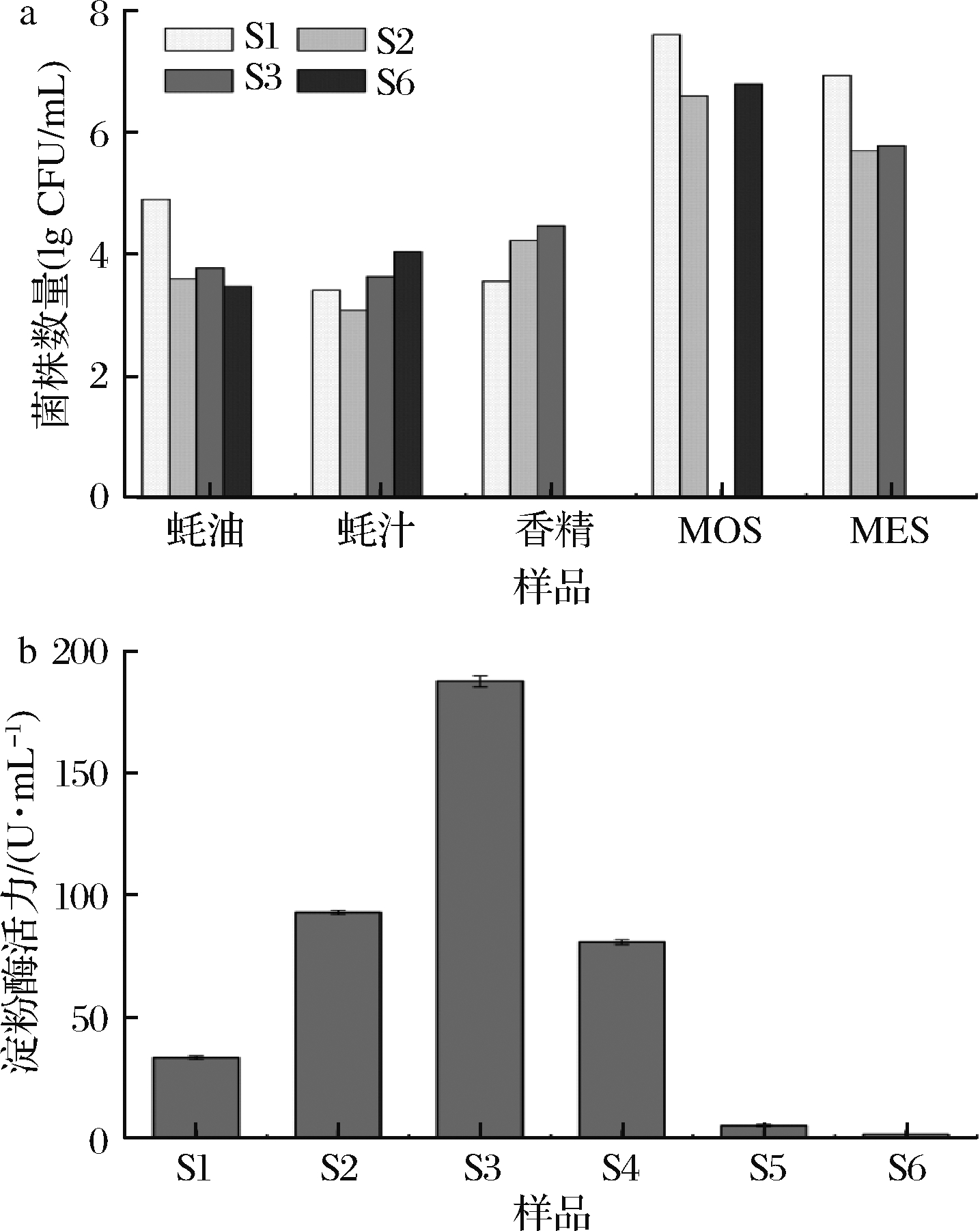
a-代表性菌株数量;b- 代表性菌株产淀粉酶能力分析
图4 蚝油原料及加工后产品中代表性菌株数量和产淀粉酶能力分析
Fig.4 Quantification and detection of amylase level of typical strains in oyster sauce raw materials and corresponding products
注:S1,人葡萄球菌;S2和S4,贝莱斯芽孢杆菌;S3和S5,枯草芽孢杆菌;S6, 皮特不动杆菌
3 结论
本研究通过16S rDNA扩增子测序分析证实,蚝油及生产原料蚝汁和香精中的优势细菌属包括动性微菌属、芽孢杆菌属、黄单胞菌属、四联球菌属和乳杆菌属,蚝油中的细菌主要来源于其生产原料蚝汁和香精。此外,对蚝油生产过程物料中代表性细菌数量变化和其利用淀粉能力的分析表明,蚝油的生产原料香精和蚝汁中含有耐热且可通过水解淀粉改变蚝油物理状态的细菌,为人葡萄球菌、贝莱斯芽孢杆菌和枯草芽孢杆菌。以上结果证实,食品原料中含有食品腐败细菌,如果在食品加工过程中对耐热或抗逆性较强的微生物的杀灭不彻底,存在影响产品品质和货架期寿命的风险。通过宏基因组测序分析样品中微生物组成与菌株分离和代谢特性分析相结合的方法,可实现对影响食品品质的微生物溯源,并为建立食品加工原料微生物控制标准以及开发含淀粉类调味品的防腐技术等方面的研究提供参考。
[1] 高磊, 谢晶, 叶藻, 等.冷鲜鸡腿肉中优势腐败菌的分离鉴定及腐败能力研究[J].食品与发酵工业, 2015, 41(8):48-53.
GAO L, XIE J, YE Z, et al.Identification of dominant spoilage bacteria isolated from leg of chilled chicken and study on its spoilage capability[J].Food and Fermentation Industries, 2015, 41(8):48-53.
[2] 蒋慧亮, 迟海, 展跃平, 等.河蚌贮藏鲜度变化及优势腐败菌分析[J].食品科技, 2019, 44(4):146-150;156.
JIANG H L, CHI H, ZHAN Y P, et al.Changes in freshness of freshwater mussels and analysis of predominant spoilage[J].Food Science and Technology, 2019,44(4):146-150;156.
[3] 谢婷婷, 王应妮,陈秋骏, 等.胀袋鲍汁中腐败菌的分离与鉴定[J].中国调味品, 2015, 40(5):27-29.
XIE T T, WANG Y N, CHEN Q J, et al.Separation and identification of spoilage bacteria in bulge bagged abalone sauce [J].China Condiment, 2015, 40(5):27-29.
[4] 冯劲, 陶宏兵, 孙廷丽, 等.番茄沙司和烧烤汁中腐败菌的分离鉴定与性质研究[J].工业微生物, 2015, 45(5):50-54.
FENG J, TAO H B, SUN T L, et al.Isolation, identification, characterization of food spoilage bacteria in tomato sauce and barbecue sauce[J].Industrial Microbiology, 2015, 45(5):50-54.
[5] 徐廷弼, 钮成拓, 王琪, 等.基于基因测序技术对辣椒酱生产设备和空气中的细菌检测[J].食品与发酵工业, 2018, 44(10):225-230.
XU T B, NIU C T, WANG Q, et al.Detection of bacteria from air and equipment of chili sauce′s production based on gene sequencing, 2018, 44(10):225-230.
[6] 蒋雪薇, 周尚庭, 叶菁, 等.成品变质酱油中微生物的分离鉴定及变质原因分析[J].食品与机械, 2016, 32(2):46-50.
JIANG X W, ZHOU S T, YE J, et al.Identification of contamination microbes separated from finished deterioration sauce and analysis of deterioration reason[J].Food & Machinery, 2016, 32(2):46-50.
[7] MHUGH A J, FEEHILY C, FENELON M A, et al.Tracking the dairy microbiota from farm bulk tank to skimmed milk powder[J/OL].mSystems, 2020.DOI:10.1128/mSystems.00226-20.
[8] 陶康,吴凌伟,金晓芳,等.基于高通量基因测序分析腐乳微生物多样性[J/OL].食品科学,2020.http://kns.cnki.net/kcms/detail/11.2206.TS.20200302.1754.050.html.
TAO K, WU L W, JIN X F, et al.Microbial diversity of Sufu was analyzed based on high-throughput gene sequencing[J].Food Science, 2020.http://kns.cnki.net/kcms/detail/11.2206.TS.20200302.1754.050.html.
[9] 杨卓, 杨欢岚, 姚泓, 等.基因测序结合传统微生物培养用于胀罐酱油中产气菌的检测[J].食品与发酵工业, 2017, 43(11):197-201.
YANG Z, YANG H L, YAO H, et al.Gene sequencing and traditional microbial culture for the detection of areogen in swelled packing soy sauce[J].Food and Fermentation Industries, 2017, 43(11):197-201.
[10] 李恒, 陈功, 伍亚龙, 等.高通量测序方法研究传统四川泡菜母水中微生物群落的动态变化[J].食品科学, 2018, 39(24):131-138.
LI H, CHEN G, WU Y L, et al.Analysis of microbial community dynamics of traditional Sichuan Paocai brine by high-throughput sequencing[J].Food Science, 2018, 39(24):131-138.
[11] 江雄辉, 廖国洪.蚝油的生产技术[J].食品与机械, 2002(1):30-33.
JIANG X H, LIAO G H.Productive techniques of oyster sauce[J].Food and Machinery, 2002 (1):30-33.
[12] 巩发永, 李静.四种液体调味料的生产工艺[J].江苏调味副食品, 2011, 28(2):34-37.
GONG F Y, LI J.Processing technology of four liquid condiment[J].Jiangsu Condiment and Subsidiary Food, 2011, 28(2):34-37.
[13] 刘仁杰, 梁珊, 李哲, 等.杀灭芽孢杆菌的方法及机理的研究综述[J].食品与发酵工业, 2019, 45(13):257-261.
LIU R J, LIANG S, LI Z, et al.Methods and mechanisms for killing Bacillus:A review[J].Food and Fermentation Industries, 2019, 45(13):257-261.
[14] 章薇, 熊国远, 董井云, 等.符离集烧鸡加工过程中微生物危害的关键控制点分析及控制[J].肉类工业, 2016(1):41-45.ZHANG W, XIONG G Y, DONG J Y, et al.Microbial hazard analysis and critical control points in processing of Fuliji red- cooked chicken[J].Meat Industry, 2016(1):41-45.
[15] 汤水粉. 即食海蜇加工过程关键控制点研究[J].渔业研究, 2019, 41(4):333-338.
TANG S F.Summary of critical control points in processing of ready-to-eat jellyfish[J].Journal of Fisheries Research, 2019, 41(4):333-338.
[16] 任伟. 影响白干货架期的主要因素及控制措施[D].无锡:江南大学, 2009.
REN W.Influencing factors of shelf life for Baigan and control measures[D].Wuxi:Jiangnan University, 2009.
[17] ANTONY-BABU S, MURAT C, DEVEAU A, et al.An improved method compatible with metagenomic analyses to extract genomic DNA from soils in Tuber melanosporum orchards[J].Journal of Applied Microbiology, 2013, 115(1):163-170.
[18] FROSSARD A, RAMOND J B, SEELY M, et al.Water regime history drives responses of soil Namib Desert microbial communities to wetting events[J].Scientific Reports, 2015, 5:12 263.
[19] LI J, HU H W, MA Y B, et al.Long-term nickel exposure altered the bacterial community composition but not diversity in two contrasting agricultural soils[J].Environmental Science and Pollution Research International, 2015, 22(14):10 496-10 505.
[20] KEMP P F, ALLER J Y.Bacterial diversity in aquatic and other environments:What 16S rDNA libraries can tell us[J].FEMS Microbiology Ecology, 2004, 47(2):161-177.
[21] 胡传旺. 酱油发酵过程微生物群落解析及功能研究[D].无锡:江南大学, 2017.
HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains[D].Wuxi:Jiangnan University, 2017.
[22] 何伟, 李菁菁, 包可翔, 等.高效表达淀粉酶Bacillus koreensis的培养基响应面优化[J].天然产物研究与开发,2019, 31(8):1 425-1 433.
HE W, LI J J, BAO K X, et al.Optimization of culture medium for a high performance amylase-producing Bacillus koreensis by response surface methodology[J].Natural Product Research and Development, 2019, 31(8):1 425-1 433.
[23] 陈慧斌. 导致牡蛎腐败的鳃部菌群及其致腐调控机制研究[D].福州:福建农林大学, 2013.
CHEN H B.Study on the rot-causing microflora in oyster gill and its regulating mechanism for spoilage[D].Fuzhou:Fujian Agriculture and Forestry University, 2013.
[24] 曹荣.太平洋牡蛎在冷藏和冻藏过程中细菌菌相及品质变化[D].青岛:中国海洋大学, 2006.
CAO R.Changes in the Microflora and quality of pacific oysters during ice and frozen storage[D].Qingdao:Ocean University of China, 2006.
[25] VANDAMME P, POT B, GILLIS M, et al.Polyphasic taxonomy, a consensus approach to bacterial systematics[J].Microbiological Reviews, 1996, 60 (2):407-438.
[26] YOON J H, KIM B C, PARK Y H.Planomicrobium:Bergey′s Manual of Systematics Bacteriology[M].2nd ed.New York:Springer-Verlag, 2004:373-374.