高级醇是3个及3个C以上一元醇的总称,是小曲清香型白酒重要的风味物质之一,对酒的品质有很大的影响,但高级醇含量过高会引起白酒冲辣以及消费者头痛,对白酒品质产生不利影响[1-2]。针对小曲清香型白酒中高级醇含量过高的问题,目前主要采用基因工程的手段,改造Saccharomyces cerevisiae获得低产高级醇的酵母[3],或高产酯类酵母将体系中的高级醇转化为酯类物质[4]。但这种单菌改造的方法对于自然发酵、互作复杂的白酒而言作用极为有限,并且工程菌在食品中应用的安全性仍存争议。因此,对于自然接种发酵的白酒酿造体系,有必要揭示微生物菌群发酵及高级醇的代谢特征,为生产控制高级醇提供理论基础。
本研究以春秋两季小曲清香型白酒发酵过程为研究对象,重点分析了2种含量较高的高级醇(异丁醇、异戊醇)的差异及变化规律。通过高通量扩增子测序揭示发酵过程中微生物的群落结构及变化规律。由随机森林算法判定春秋季发酵的差异微生物,相关性分析算法判定发酵过程中与高级醇产生相关的微生物。通过固态模拟发酵验证了微生物菌群互作对高级醇合成代谢的调控作用,这对白酒以及其他食品发酵的定向调控具有一定的理论意义。
1 材料与方法
1.1 实验材料
1.1.1 样品采集
本研究所用酒醅样本均来源于小曲清香型白酒厂劲牌公司的手工生产车间,于2018年春季(5月)、秋季(10月)取样。发酵周期为7 d,酒醅分别取入池发酵0、1、2、3、5、7 d的样本。每个时间点样本取3个平行窖池,用于高通量测序的样品于-80 ℃保存,用于微生物筛选的样品于4 ℃保存。
1.1.2 菌株及活化
本研究使用菌株:短乳杆菌(Lactobacillus brevis JN-JB-2)、酿酒酵母(Saccharomyces cerevisiae JN-JY-1)、库德里阿兹威氏毕赤酵母(Pichia kudriavzevii JN-JY-2),均来源于酒醅。乳酸菌使用MRS培养基活化,37 ℃静置培养3 d;酵母使用YPD培养基活化,30 ℃,200 r/min摇床培养2 d。
1.1.3 主要试剂及仪器
2×SG Fast qPCR Master Mix,生工生物工程股份有限公司;L-薄荷醇,北京百灵威科技有限公司;异丁醇、异戊醇,西格玛奥德里奇贸易有限公司。
高速离心机(Eppendorf centrifuge 5804R),艾本德国际贸易有限公司;荧光定量PCR仪(Applied Biosystems StepOnePlus),爱普拜斯应用生物系统贸易有限公司;气相色谱-氢离子火焰检测器(Agilent 7890 GC-FID),安捷伦科技有限公司。
1.2 实验方法
1.2.1 基因组DNA提取方法
样本前处理及基因组DNA提取方法参照文献[5]:利用无菌0.1 mol/L PBS缓冲液与无菌玻璃珠洗涤样本并收集细胞。用酚-氯仿法抽提宏基因组,真空干燥处理后,加入40 μL无菌ddH2O溶解DNA,作为高通量测序及qPCR定量的模板。
1.2.2 高通量扩增子测序结果的处理
高通量扩增子测序:通过扩增子测序技术分析发酵过程中微生物群落结构。对于真菌,选用ITS3/ITS4引物对ITS2区域进行扩增[6];对于细菌,选用338 F/806 R对16S rRNA的V3-V4区域进行扩增[7]。用PCR纯化试剂盒纯化PCR产物并测定浓度。使用MiSeq台式测序仪进行高通量测序[8],用于2×300 bp配对末端测序。
序列处理:通过QIIME处理所有从MiSeq生成的原始序列。经质量控制,去除不合要求的序列[9],通过UCLUST将修整后的序列以97%的相似性聚类为OUT(operational taxonomic unit)[10]。
1.2.3 生物量的测定
采用实时荧光定量PCR(quantitative real-time PCR,qPCR)技术定量样品中总细菌和总真菌的生物量。反应体系:10 μL 2×SG Fast qPCR Master Mix(High Rox),上、下游引物各0.4 μL(20 μmol/L),1 μL DNA模板(10~50 ng/μL),用ddH2O补齐至20 μL。引物、扩增条件和标曲构建条件如文献报道[11]。
1.2.4 高级醇检测方法
浸提液制备:称取10.0 g酒醅并加入25 mL超纯水,混匀后于4 ℃过夜浸提。浸提后的样本冰浴超声30 min,在8 000×g条件下离心5 min,吸取上清液,即为样本浸提液。
液液微萃取[12]:取1 mL浸提液,加入1 mL乙酸乙酯、10 μL 10.10 g/L L-薄荷醇内标化合物、0.4 g NaCl,充分混匀,收集上清液,用0.22 μm有机滤膜过滤备用。
色谱分析:气相色谱火焰离子化检测器(GC-FID)测定高级醇含量,色谱柱为CP-Wax(30 m×0.25 mm,0.25 μm),进样口、检测器温度均为250 ℃,分流比为5∶1。升温程序参照文献[13]进行设置。
1.2.5 固态模拟发酵
参照文献的方法进行糖化,至糖含量为50 g/kg[14]。依照小曲清香型白酒的酿造工艺,如表1所示的接种量接入微生物,拌2倍质量的酒糟,装入烧杯,严格密封,20 ℃静置发酵。
表1 固态模拟发酵初始接种量
Table 1 The inoculation conditions of simulated solid state fermentation

1.2.6 统计学分析方法
主成分分析:为揭示不同季节小曲清香型白酒发酵过程中微生物群落的演替轨迹,通过SPSS 22.0对微生物属的相对丰度进行主成分分析。
随机森林算法:利用R程序的Random forest包进行计算,以平均下降精度(Mean decrease accuracy)表示变量的差异程度[15]。
相关性分析:为解析调控高级醇产生的关键微生物,计算小曲清香型白酒发酵过程微生物属水平的相对丰度与异丁醇、异戊醇含量的Pearson相关系数(ρ),选取|ρ|>0.6且P<0.05的数据作为有效网络连接[16],通过Gephi进行可视化。微生物属要求至少在一个平行样品中相对丰度≥1.0%。
2 结果与分析
2.1 不同季节小曲清香型白酒高级醇的变化规律
对不同季节发酵过程中高级醇含量进行比较分析,春、秋两季发酵过程中异丁醇和异戊醇的变化规律如图1所示。发酵终点春季样本的异丁醇、异戊醇含量分别为(54.78±13.54)和(124.66±21.04) mg/kg,高级醇总量(179.43±9.57) mg/kg;而秋季分别可达(88.86±15.43)和(188.00±17.40) mg/kg,总量达(276.86±26.80) mg/kg。秋季样本的高级醇含量显著高于春季。
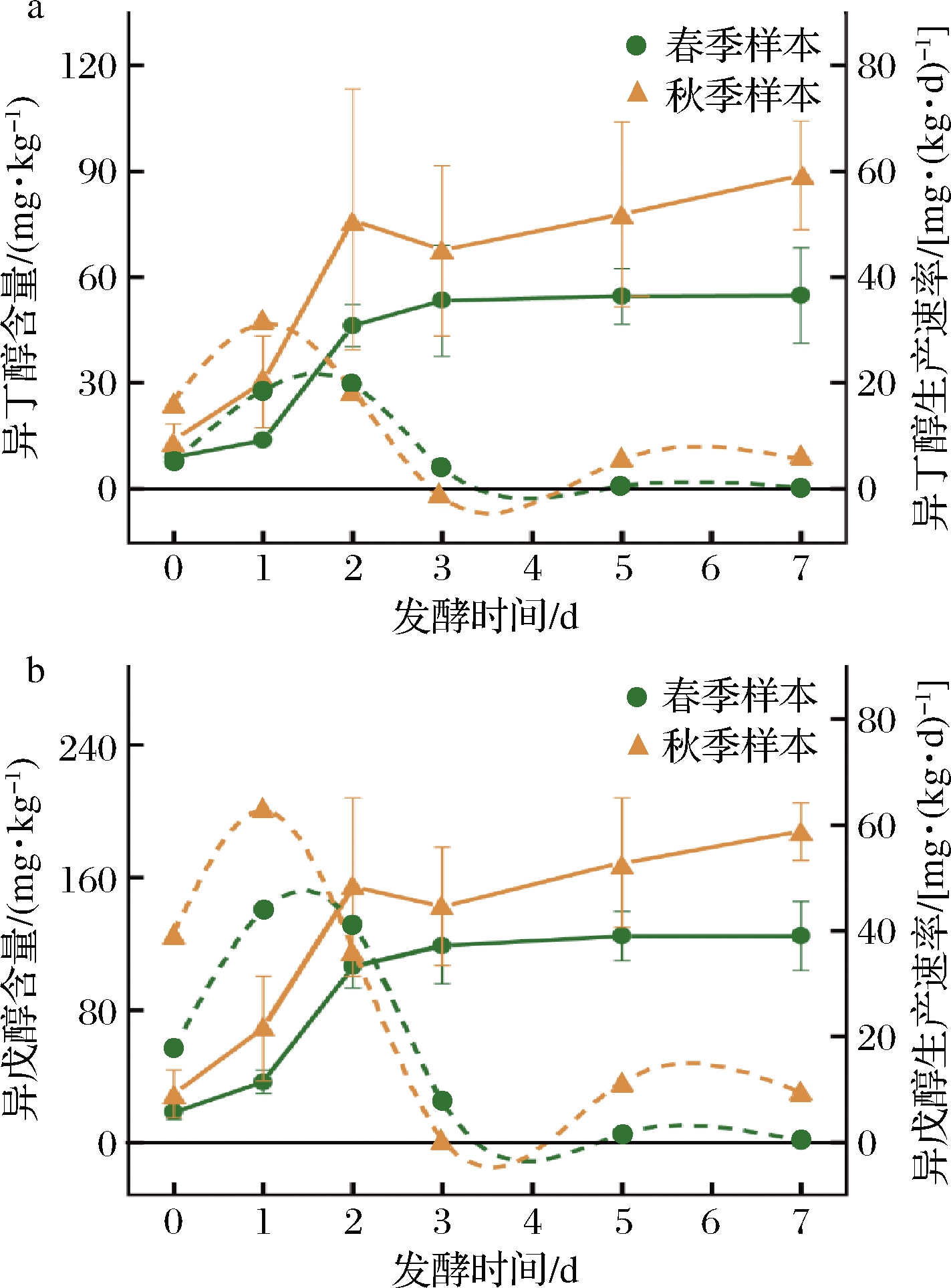
a-异丁醇;b-异戊醇
图1 春秋季样本中高级醇代谢规律
Fig.1 Mechanisms of higher alcohols synthesized in fermented grains of spring and autumn
注:实线表示高级醇含量,虚线表示代谢速率
春季样本异丁醇、异戊醇的最大产生速率分别为19.76和43.92 mg/(kg·d),秋季样本分别为31.27和62.78 mg/(kg·d);秋季异丁醇、异戊醇的最大产生速率分别为春季的1.58和1.43倍。不同季节发酵过程中2种高级醇代谢均可分为2个阶段:(1)发酵前期(0~3 d),高级醇快速累积,说明该阶段微生物代谢合成高级醇最为迅速;(2)发酵中后期(3~7 d),2种高级醇含量变化缓慢甚至趋于稳定。高级醇的产量与最大产生速率在不同季节均有较大的差异。
2.2 不同季节小曲清香型白酒高级醇的变化规律
2.2.1 菌群差异是高级醇差异的潜在影响因素
通过Illumina Miseq测序揭示发酵过程中的微生物群落的组成。对于细菌,质控后共得到1 293 003条高质量的序列,平均每个样本具有(35 917±11 946)条;共得到5 428个OTU,平均每个样品得到(150±36)个OTU。对于真菌,质控后共得到1 944 028条高质量的序列,平均每个样本具有(54 001±21 014)条;并得到2 150个OTU,平均每个样品得到(60±14)个OTU。每个样品的覆盖率均在99%以上,表明测序深度足够和数据质量可靠。
发酵过程中的高级醇由微生物代谢产生,因此进一步分析了不同季节发酵过程中微生物的演替情况。如图2所示,由主成分分析比较2个季节酿造微生物的差异,发现春秋季发酵微生物的组成从发酵起始点开始已具有差异;同时,两季样本中酿造微生物的演替方向差异显著,真菌和细菌在秋季发酵过程都比春季演替更剧烈。
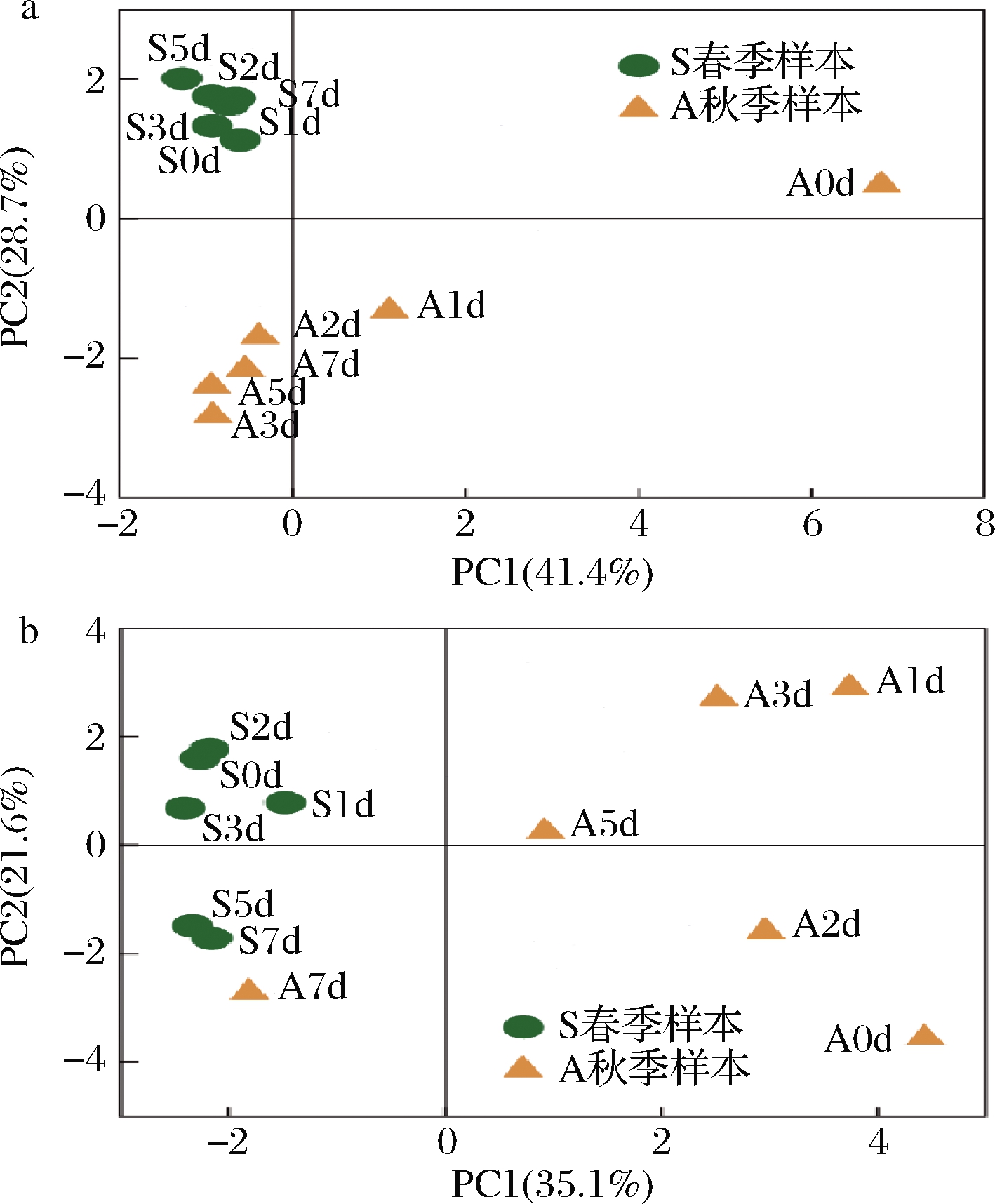
a-真菌;b-细菌
图2 春秋季发酵过程中微生物菌群结构主成分分析
Fig.2 Principal components analysis of the microbiota structure
注:绿色与橙色实心点分别表示不同取样时间点春秋季的样本
2.2.2 微生物群落演替规律分析
将平均相对丰度>1%的属定义为优势微生物,平均相对丰度>10%的属定义为绝对优势微生物[17]。微生物群落结构的变化规律如图3所示。由测序结果分析得到优势微生物中真菌属有11个,细菌属有14个,并通过绝对定量方法测定了细菌、真菌的总量。发酵过程中,两季真菌的绝对丰度均呈现出先增加后下降的趋势,其中绝对优势真菌属为Saccharomyces、Pichia和Rhizopus。在高级醇快速增加的发酵前期(0~3 d),春季样本优势属的丰度变化平缓。秋季发酵前期Rhizopus迅速降低;Saccharomyces丰度迅速升至峰值;Pichia的丰度变化幅度也较大。因为Saccharomyces是产高级醇的主要微生物[13],Saccharomyces随时间的变化规律如图3-c所示,秋季样本发酵前期Saccharomyces的快速增殖和较高丰度可能是秋季样本中高级醇含量较高的原因之一。
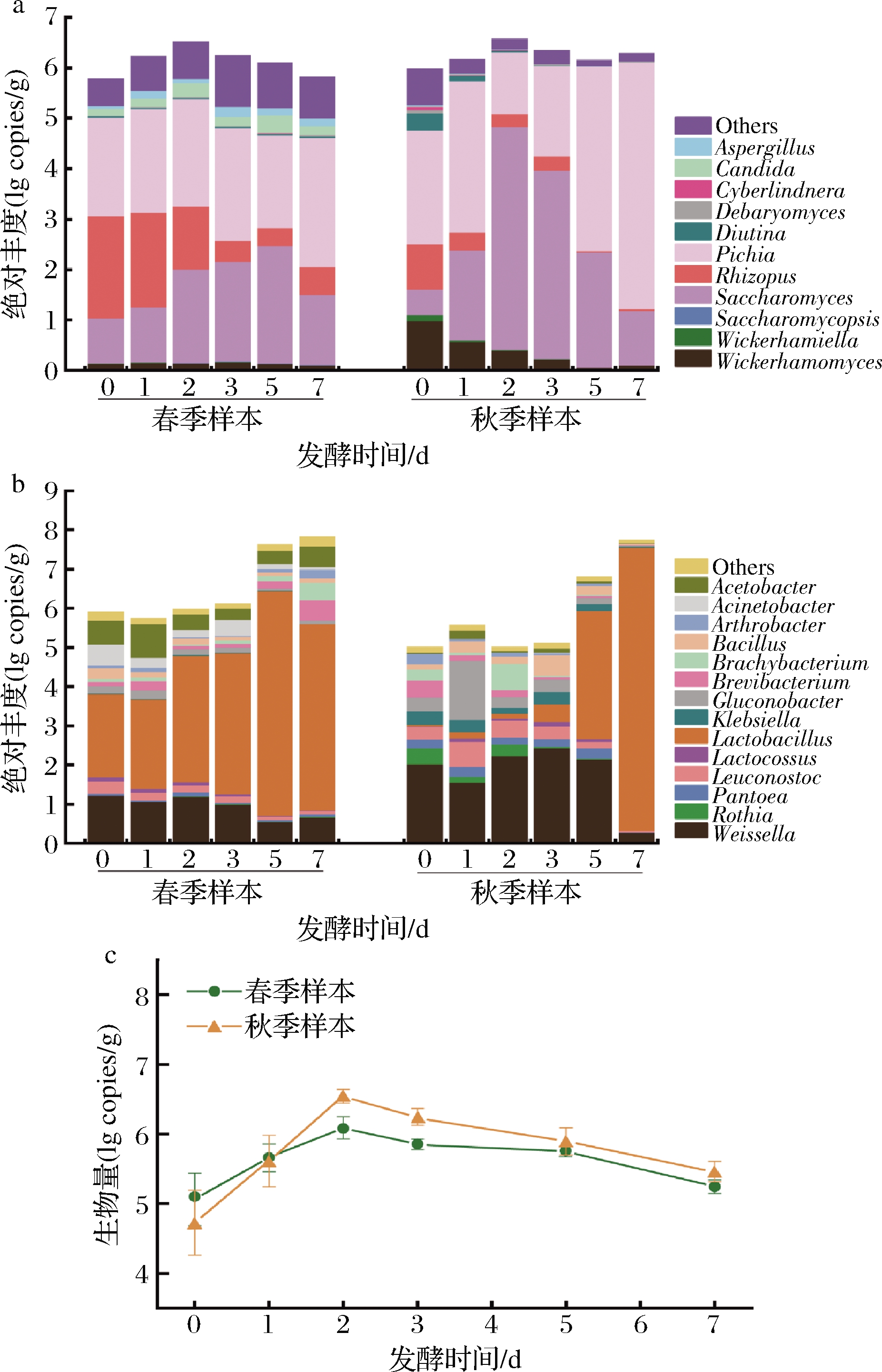
a-春秋季真菌;b-春秋季细菌;c-春秋季Saccharomyces生物量的动态变化
图3 春秋季发酵过程群落结构(属水平)分析
Fig.3 The microbial community structure(genera) during fermentation in spring and autumn
注:平均相对丰度<1%的属被合并划分为“other”
发酵过程中,细菌绝对丰度整体呈现上升的趋势。绝对优势细菌为Weissella和Lactobacillus。春季样本中,发酵前期Lactobacillus相对丰度较高,5 d达到峰值;而秋季样品发酵前期Weissella相对丰度较高,Gluconobacter、Leuconostoc等其他细菌的丰度同样较高,Lactobacillus在发酵前期丰度较低,变化缓慢,直到发酵7 d才达到峰值。Lactobacillus是酿造系统中重要的功能微生物,与春季样本相比,秋季样品中Lactobacillus的相对丰度偏低,且到达峰值的时间较晚,这可能是秋季样本高级醇含量高的原因之一,Lactobacillus可能对于抑制高级醇的产生有一定的作用。
2.2.3 关键差异微生物引起高级醇的差异
采用随机森林的算法,对发酵过程群落中绝对优势属进行差异分析。如图4-a所示,微生物对群落差异的贡献程度由平均下降精度表示,Lactobacillus、Weissella、Rhizopus、Saccharomyces、Pichia属是春秋季样本中的显著差异微生物。

a-随机森林计算绝对优势微生物在两季样本中的平均下降精度;
b-相关性网络分析与高级醇合成显著相关的微生物
图4 关键差异微生物的判定
Fig.4 Identification of key differential microorganisms
注:图4-b中绿色、橙色和蓝色的节点分别表示真菌、细菌和高级醇,数字(ρ值)及连线粗细表示相关性大小,橙色的连线表示正相关关系,灰色的连线表示负相关关系;*,P<0.05;**,P<0.01
春秋样本差异微生物中,由相关性分析解析调控高级醇产生的潜在关键微生物。有效网络连接(|ρ|>0.6,P<0.05)如图4-b所示,在小曲清香型白酒发酵过程中,与异丁醇、异戊醇显著相关的绝对优势属共有3个,其中Saccharomyces与2种高级醇产量成正相关关系,Pichia和Lactobacillus均与2种高级醇产量成负相关关系,说明Pichia和Lactobacillus可能对于高级醇的降低有所贡献。
2.3 微生物相互作用对高级醇产生的影响
利用模拟小曲清香型白酒酿造工艺的固态发酵分析Saccharomyces、Pichia与Lactobacillus相互作用对高级醇的调控作用。选择3个属中相对丰度最高的种,分别对S.cerevisiae JN-JY-1、P.kudriavzevii JN-JY-2、L.brevis JN-JB-2进行固态发酵,并检测高级醇含量,如图5-a所示,P.kudriavzevii与L.brevis单培养时产高级醇的能力均较低(<3 mg/kg)。如图5-b所示,异丁醇、异戊醇在只接种S.cerevisiae的对照组产量均为最高,总高级醇产量可达(43.52±2.43) mg/kg;成对共培养的发酵实验组高级醇产量有一定程度下降;而接种S.cerevisiae、P.kudriavzevii、L.brevis的实验组总高级醇产量最低,只有(35.0±1.64) mg/kg,比S.cerevisiae单菌发酵组降低了8.49 mg/kg(P=0.02)说明3株菌在高级醇的调控上存在着密切的相互作用关系,P.kudriavzevii与L.brevis的添加对于高级醇的调控有一定的贡献。
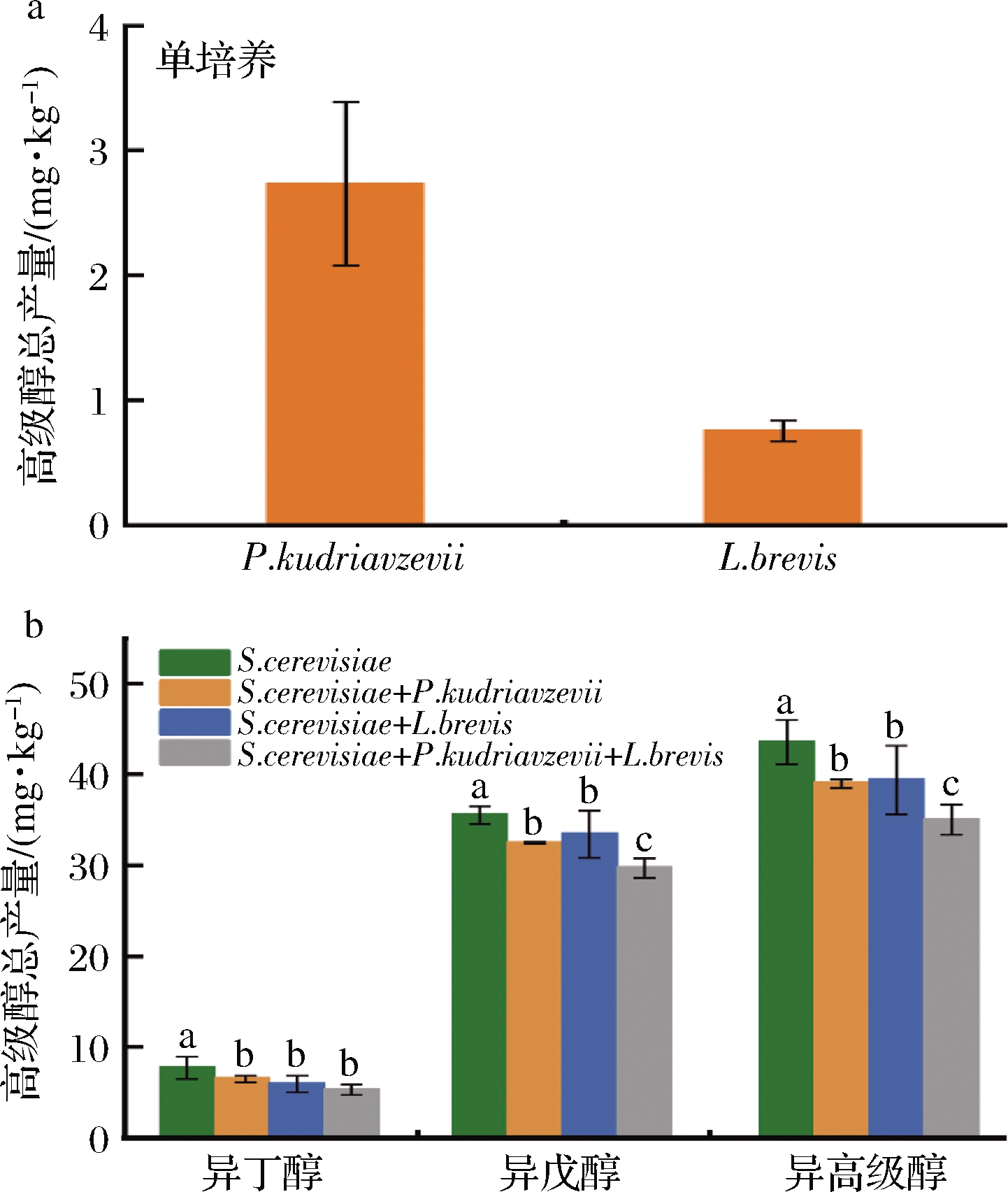
a-P.kudriavzevii与L.brevis单培养发酵时高级醇的产量;
b-高级醇相关微生物属固态模拟共培养发酵时高级醇的产量
图5 固态模拟发酵高级醇的产量
Fig.5 The yield of higher alcohols in simulated solid fermentation
注:不同字母表示差异显著性
3 结论与讨论
近年来,对于发酵食品风味或功能性代谢产物调控的研究越来越多,同时测序技术及分析手段的不断进步,为解析酿造微生物群落的结构和演替规律提供了更好的平台[18],但通过菌群来调控高级醇的有效策略仍为空缺。本研究创新性地通过分析高级醇的代谢规律、菌群的变化差异,成功确定了影响高级醇产生的微生物。通过这种分析思路及统计学方法来进一步确定风味调控微生物对于大多数自然接种的传统发酵食品都是适用的。此外,微生物之间具有密切的相互作用,多种风味调控微生物的添加会改变微生物群落中原有的部分代谢特征,可能达到更好的风味调控效果。
发酵食品微生物群落组成与相互作用复杂,本研究发现了微生物菌群自然发酵过程中对高级醇合成代谢具有显著影响的微生物,表明通过风味调控微生物对菌群进行物质代谢调控与其他研究中单菌改造的方式相比具有一定的优势。多数情况下,风味调控微生物包含多个种或属,明确风味调控微生物后,可以进一步理性设计,利用不同的菌株组合对目标物质达到更好的调控效果[19],为食品发酵过程中的风味代谢调控提供了一种有效的策略。然而,目前仍存在一些问题有待揭示,白酒等诸多传统发酵食品酿造微生物群落复杂、功能多样、相互作用密切[20],在发酵过程中微生物群落的动态规律揭示尚不完善,代谢物在酶、基因等分子水平如何受到影响及调控,其深层机制仍有待研究。
综上所述,本研究通过深入分析不同季节样本高级醇的变化规律和微生物群落的结构及演替规律的差异,明确了春秋两季高级醇产量有差异的原因以及与高级醇合成相关的微生物,验证了相关微生物对于发酵产生高级醇的调控作用,进一步说明了风味调控微生物对于食品发酵过程中代谢产物具有重要的影响,可以基于此来进行代谢产物的调控,并用于构建食品发酵合成微生物组,为不同季节稳定生产白酒提供理论指导。
[1] YANG D S,LUO X Q,WANG X G.Characteristics of traditional Chinese shanlan wine fermentation[J].Journal of Bioscience and Bioengineering,2014,117(2):203-207.
[2] MCKARNS S C,HANSCH C,CALDWELL W S,et al.Correlation between hydrophobicity of short-chain aliphatic alcohols and their ability to alter plasma membrane integrity[J].Fundamental and Applied Toxicology,1997,36(1):62-70.
[3] MA L J,HUANG S Y,DU L P,et al.Reduced production of higher alcohols by Saccharomyces cerevisiae in red wine fermentation by simultaneously overexpressing BAT1 and deleting BAT2[J].Journal of Agricultureal and Food Chemistry,2017,65(32):6 936-6 942.
[4] LI W,WANG J H,ZHANG C Y,et al.Regulation of Saccharomyces cerevisiae genetic engineering on the production of acetate esters and higher alcohols during Chinese Baijiu fermentation[J].Journal of Industrial Microbiology and Biotechnology,2017,44(6):949-960.
[5] 李小龙. 芝麻香型白酒发酵过程微生物群落演替及其驱动因素[D].无锡:江南大学,2018.
LI X L.Microbial community succession and its drivers during roasted sesame-flavor liquor fermentation[D].Wuxi:Jiangnan University,2018.
[6] HONG X T,CHEN J,LIU L,et al.Metagenomic sequencing reveals the relationship between microbiota composition and quality of Chinese rice wine[J].Scientific Reports,2016,6.DOI:10.1038/srep26621.
[7] DEFILIPPIS F,GENOVESE A,FERRANTI P,et al.Metatranscriptomics reveals temperature-driven functional changes in microbiome impacting cheese maturation rate[J].Scientific Reports,2016,6.DOI:10.1038/srep21871.
[8] LEMOS L N,FULTHORPE R R,TRIPLETT E W,et al.Rethinking microbial diversity analysis in the high throughput sequencing era[J].Journal of Microbiological Methods,2011,86(1):42-51.
[9] CAPORASO J G,KUCZNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[10] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2 460-2 461.
[11] 王鹏. 地衣芽孢杆菌强化对浓香型白酒酿造微生物群落结构和代谢的影响[D].无锡:江南大学,2017.
WANG P.Effects of Bacillus licheniformis inoculation on the microbial community structure and metabolism during strong flavored liquor brewing process[D].Wuxi:Jiangnan University,2017.
[12] GAO W J,FAN W L,XU Y.Characterization of the key odorants in light aroma type Chinese liquor by gas chromatography-olfactometry,quantitative measurements,aroma recombination,and omission studies[J].Journal of Agricultural and Food Chemistry,2014,62(25):5 796-5 804.
[13] JIANG J,LIU Y C,LI H H,et al.Modeling and regulation of higher alcohol production through the combined effects of the C/N ratio and microbial interaction[J].Journal of Agricultureal and Food Chemistry,2019,67(38):10 694-10 701.
[14] WANG S L,WU Q,NIE Y,et al.Construction of synthetic microbiota for reproducible flavor compound metabolism in Chinese light-aroma-type liquor produced by solid-state fermentation[J].Applied and Environmental Microbiology,2019,85(10):e03 090-03 018.
[15] ZHANG J Y,LIU Y X,ZHANG N,et al.NRT1.1B is associated with root microbiota composition and nitrogen use in field-grown rice[J].Nature Biotechnology,2019,37:676-684.
[16] BARBER N A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].The ISME Journal,2012,6(2):343-351.
N A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].The ISME Journal,2012,6(2):343-351.
[17] WANG X S,DU H,XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor[J].International Journal of Food Microbiology,2017,244:27-35.
[18] 王鹏, 吴群,徐岩.中国白酒发酵过程中的核心微生物群及其与环境因子的关系[J].微生物学报,2018,58(1):142-153.
WANG P,WU Q,XU Y.Core microbiota in Chinese liquor fermentation and associations with environmental factors[J].Acta Microbiologica Sinica,2018,58(1):142-153.
[19] SMID E J,LACROIX C.Microbe-microbe interactions in mixed culture food fermentations[J].Current Opinion in Biotechnology,2013,24(2):148-154.
[20] 王慧琳,周炜城,任聪,等.传统发酵食品微生物学研究进展[J].生物学杂志,2018,35(6):1-5.
WANG H L,ZHOU W C,REN C,et al.Recent advances in the study of microbes in traditional fermented foods[J].Journal of Biology,2018,35(6):1-5.