葡萄酒自然发酵过程中微生物因葡萄园管理、土壤类型、光照、降水量等自然因素的影响而存在显著差异[1-3],导致不同产区葡萄酒口感、风味不同。国内对酿酒葡萄表皮微生物的研究日益增多[2,4-6]。高通量测序技术又称下一代测序(next-generation sequencing,NGS)技术,是目前应用最普遍的测序技术,对微生物的群落结构分析具有独特的优势[7],一次可以对十几万到几百万条DNA分子进行序列测定,克服了传统培养技术及分子鉴定方法的缺陷,可为微生物群落组成提供更准确和详细的分析,并在改善微生物资源开发和食品安全方面作出了巨大的贡献。
目前,相较于国际上著名葡萄酒产地微生物的研究而言,我国虽然葡萄栽培区域广阔,但对于不同产区葡萄表面微生物群落的分析研究一直处于空白。随着中国葡萄酒产业的发展,探究不同产区酿酒葡萄表皮微生物的多样性,对选育产区特色发酵菌株,提升中国葡萄酒的品质及国际影响力具有深远意义。本研究利用NGS技术对比研究了宁夏贺兰山、河北怀来、山东蓬莱、山东青岛4个产区赤霞珠葡萄表皮微生物的组成和多样性,以期为我国著名产区酿酒微生物资源库的建设以及提高葡萄酒品质等方面提供理论依据。
1 材料与方法
1.1 材料与试剂
葡萄:宁夏贺兰山(nxcs)、山东蓬莱(plcs)、山东青岛(qdcs)、河北怀来(hbcs) 产区葡萄园中的赤霞珠(Cabernet Sauvignon) 酿酒葡萄品种,样品分别于2019年9月采样。从1株葡萄树的上、中、下3个部位采样。
聚乙烯吡咯烷酮(polyvinyl pyrrolidone,PVP)、三羟甲基氨基甲烷(Tris)、乙二胺四乙酸 (ethylene diamine tetraacetic acid,EDTA)、氯化钠及其他试剂均为分析纯。
TENP缓冲液:50 mmol/L的Tris,20 mmol/L的EDTA,100 mmol/L的氯化钠,0.01 g/mL的PVP(pH 10),调pH至10。
磷酸盐缓冲液(phosphate buffered saline,PBS,pH7.4):在800 mL蒸馏水中溶解8 g NaCl、0.2 g KCl、1.44 g Na2HPO4和0.24 g KH2PO4,用HCl溶液调节溶液的pH值至7.4,加水定容至1 L,在121 ℃下高压蒸汽灭菌20 min。保存于室温,待用。
1.2 实验方法
1.2.1 样品预处理[8]
(1)样品由于自然环境,葡萄表面复杂成分的影响,在微生物总DNA提取前需要对样品进行预处理,分离多糖、色素、多酚类等杂质。取适量葡萄原料,在无菌条件下将葡萄果皮取下,准确称取不同产区的酿酒葡萄果皮8 g,分别加入到50 mL无菌离心管中并标记A,加入12 mL TENP缓冲液,涡旋振荡5 min后3 000×g离心5 min,转移上清液至新的离心管中并标记B。(2)吸取12 mL TENP缓冲液到离心管A,涡旋振荡5 min,3 000×g离心5 min后转移上清液至离心管B中。(3)在离心管A中加入6 mL PBS溶液,涡旋振荡5 min,3 000×g离心5 min,转移上清液至离心管B中,将离心管B以9 000×g离心10 min,弃去上清液,沉淀物于-20 ℃保存备用。
1.2.2 DNA的提取
酿酒葡萄表皮微生物DNA采用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒进行提取。
1.2.3 样品PCR
第一轮扩增前利用Qubit3.0 DNA检测试剂盒对基因组DNA精确定量,以确定PCR反应体系中应加入的 DNA量。真菌DNA进行PCR所用引物是已经融合了测序平台的通用引物ITS1(5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2(5′-GCTGCGTTCTTCATCGATGC-3′);细菌DNA 进行PCR所用的引物为测序通用引物Nobar_341F(5′-CCTACGGGNGGCWGCAG-3′)和Nobar_805R(5′-GACTACHVGGGTATCTAATCC-3′)。
取2×Hieff® Robust PCR 预混液15 μL、正向测序引物和反向测序引物各1 μL、10~20 ng模板DNA、9~12 μL H2O,配制总体积为30 μL的 PCR反应体系。PCR反应程序为:94 ℃预变性3 min;94 ℃变性30 s,45 ℃退火20 s,65 ℃延伸30 s,反应5个循环;94 ℃变性20 s,55 ℃ 退火20 s,72 ℃延伸30 s,反应20个循环;72 ℃延伸5 min。
对第一轮PCR扩增质检合格的产物进行第二轮Illumina桥式PCR,用于测序文库的构建,反应体系为:15 μL 2×Hieff® Robust PCR预混液、1 μL Index-PCR Primer F、1 μL Index-PCR Primer R、20~30 ng PCR 扩增产物、9~12 μL H2O,总体积为30 μL。
扩增样品振荡混匀后短暂离心将反应液离心至管底。将PCR管置于PCR仪中进行扩增,PCR反应条件:95 ℃预变性3 min;94 ℃变性20 s,55 ℃ 退火20 s,72 ℃延伸30 s,反应5个循环;72 ℃延伸 5 min,得到的产物于-20 ℃保存备用。
1.2.4 高通量测序[9]
PCR扩增产物送至生工生物工程(上海)股份有限公司进行真菌ITS序列的ITS1-ITS2区间、细菌16S rRNA基因 V3-V4区域文库的构建,之后应用Illumina Misq测序平台进行分析。
1.3 数据预处理
将测序得到的原始数据进行预处理,过滤去除引物和接头序列、非特异性扩增序列及嵌合体,以保证最终用于操作分类单元(operational taxonomic units,OTU)分析的数据质量可靠。
1.4 数据分析
1.4.1 OTU聚类分析
采用Usearch软件,将所有样本序列按照序列间的距离进行聚类,后根据序列之间的相似性将序列分成不同的OTU。通常在低于97%的相似水平的OTU进行生物信息统计分析。
在OTU聚类结果的基础上,获取每1个OTU聚类中的代表性序列。选择丰富度最高的序列作为代表性序列,使用R软件的VennDiagram package数据包绘制韦恩图。
1.4.2 Alpha多样性分析
通过对样品的多样性分析能够反映微生物群落的丰度和多样性,主要包括Chao1、Shannon和Simpson;并通过计算Coverage指标检测各样品文库的覆盖率,其数值越高,则样本中序列没有测出结果的概率越低,反映本次测序结果具有代表性。
1.4.3 微生物分类及差异性分析
对OTU数据进行整理,在门、属的水平上对各个样本中的真菌、细菌进行物种丰度柱形图统计,分析每一个样本物种占比并对优势菌属进行分析,通0.01 g/mL过主坐标分析图及聚类图分析样本间相似度。
2 结果与分析
2.1 稀疏曲线
稀疏曲线用于比较不同测序数据量的样本中物种的丰富度,同时可以说明样本的测序数据量是否合理。当曲线趋向平坦时,说明测序数据量合理,增大数据量只会产生少量新的OTU,反之则表明继续测序还可能产生较多新的OTU。如图1显示,细菌样品的OTU稀疏曲线在小于12 000个序列、真菌样品的OTU稀疏曲线在小于60 000个序列的条件下均没有进入平台期,继续增加测序量,其OTU数量也会随着增加。但随着测序量的增加,细菌、真菌的Shannon指数稀疏曲线在测序量到达一定数值之后趋于平稳进入平台期。由此可知,随着测序量的增加有可能会发现新的微生物种类,但微生物多样性已经不再随测序量的增加而发生变化,说明测序数量足够充分合理。因此,本研究中细菌、真菌的测序量满足后续的生物信息学分析。
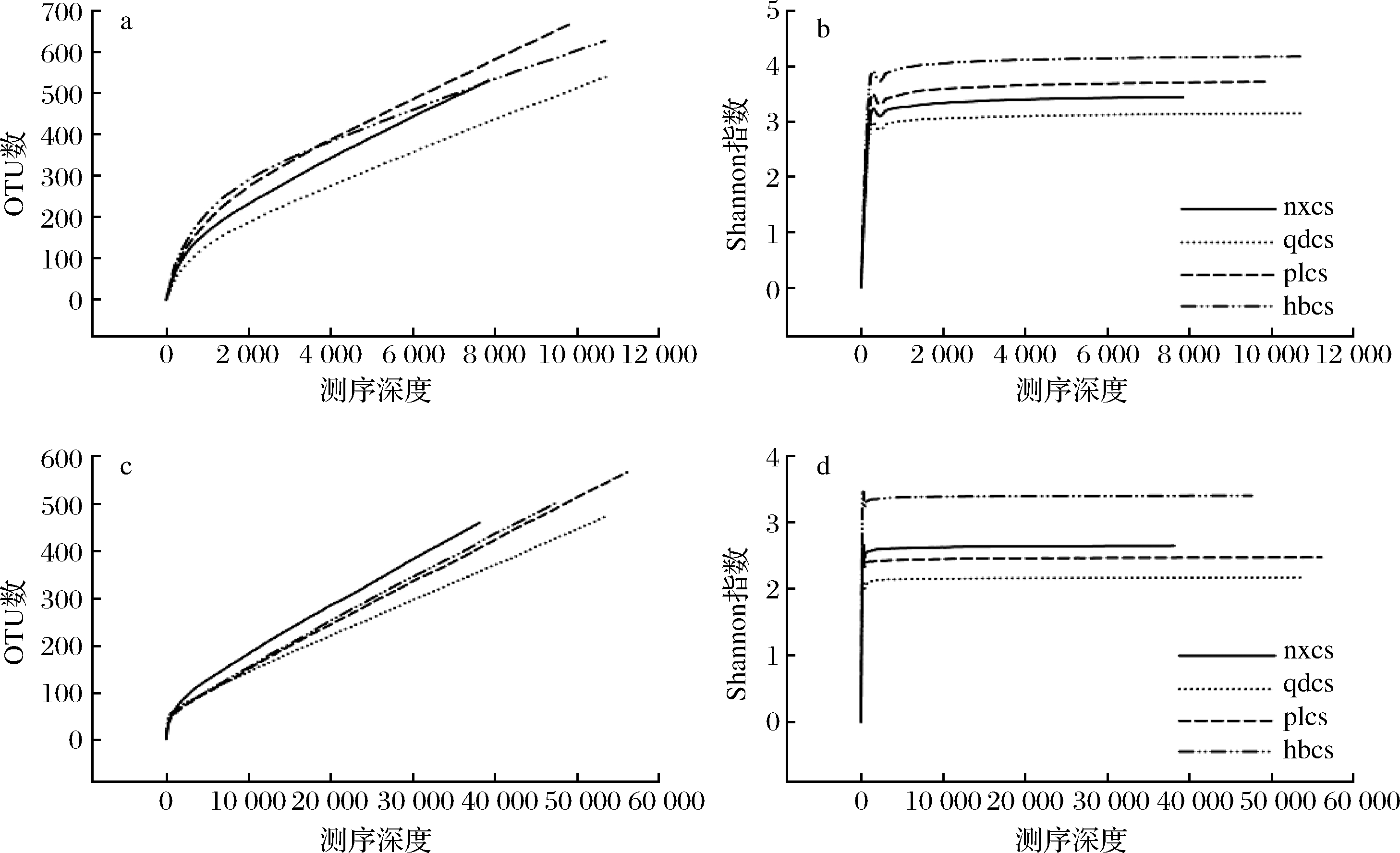
a-细菌群落OTU稀疏曲线;b-细菌群落Shannon指数稀疏曲线;c-真菌群落OTU稀疏曲线;d-S真菌群落hannon指数稀疏曲线
图1 OTU稀疏曲线和Shannon指数稀疏曲线
Fig.1 OTU sparse curve and Shannon exponential sparse curve
2.2 样品中所含OTU数目分析
由图2可知,共检测到2 364个细菌OTU,其中,宁夏贺兰山、山东蓬莱、河北怀来和山东青岛产区的赤霞珠表皮细菌种类分别为531、666、626、541种;上述4个产区中特有的细菌种类分别为422、523、481、463种,共有的细菌种类有38种。由此可知,4个产区中葡萄样品中细菌菌群结构差异明显,其中山东蓬莱赤霞珠葡萄表皮细菌菌种最为丰富。另外,山东蓬莱和河北怀来产区的赤霞珠果实表面共有的OTU数目有117个,远高于其他产区间的共有细菌菌种数,说明山东蓬莱与河北怀来赤霞珠表皮细菌种类相似度较大。
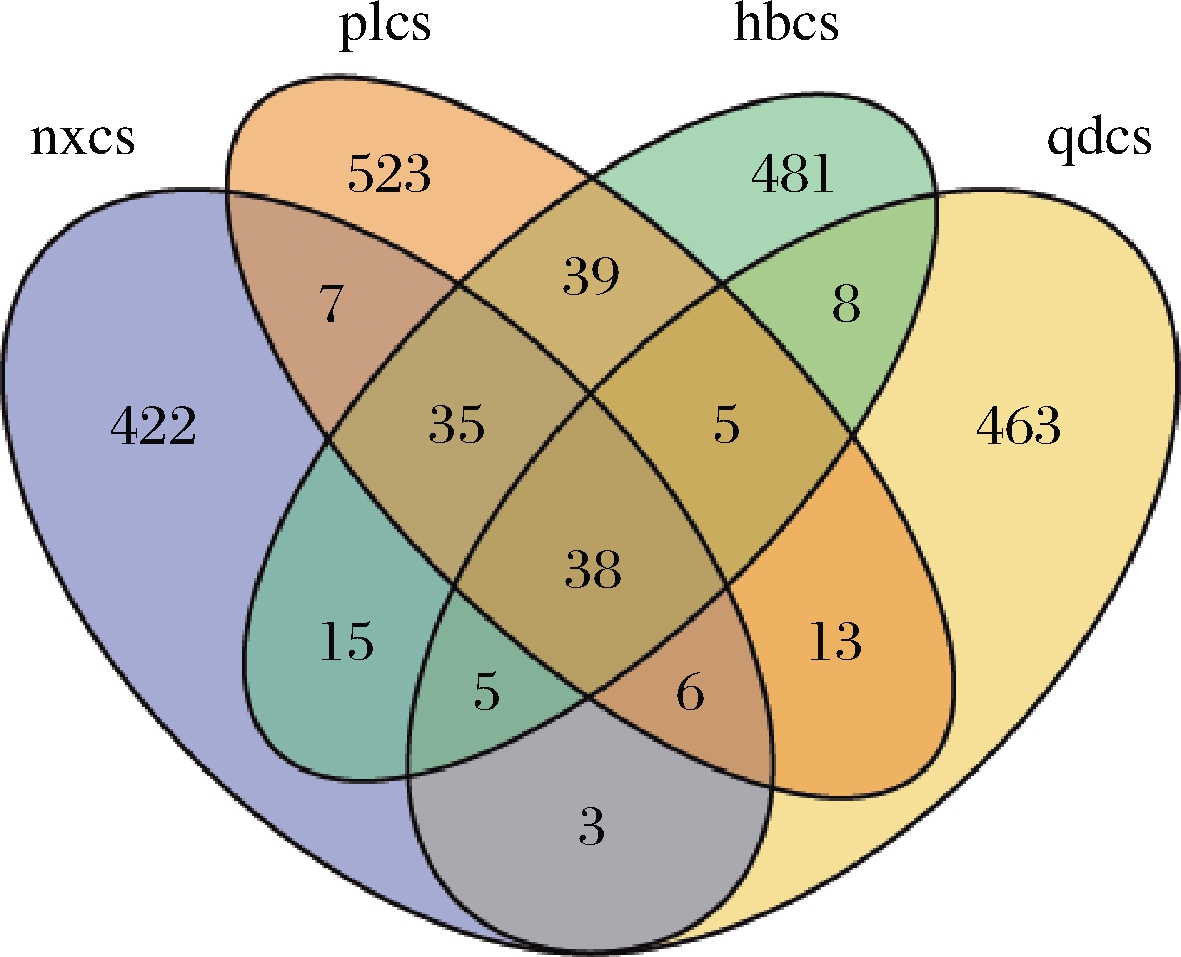
图2 细菌群落韦恩图
Fig.2 Venn diagram of bacterial community
由图3所示,共检测到2 007个真菌OTU,其中,宁夏贺兰山、山东蓬莱、河北怀来和山东青岛产区的赤霞珠表皮真菌种类分别为460、567、504、476个;上述4个产区特有真菌种类分别为403、505、446、424个,共有的真菌种类有12个。由此可知,4个产区赤霞珠表皮真菌种类总数相差不大,但真菌群落结构差异较大。
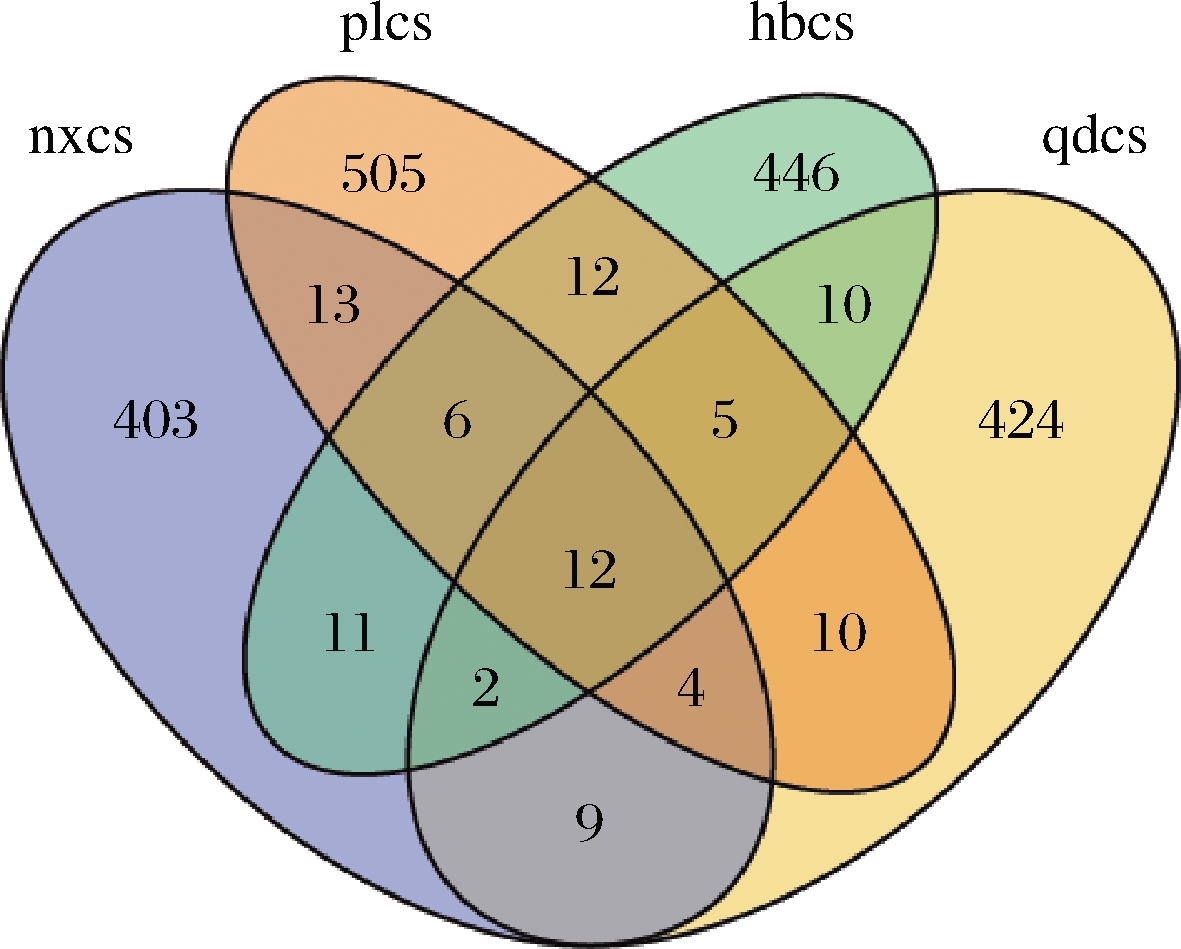
图3 真菌群落韦恩图
Fig.3 Venn diagram of fungal community
2.3 细菌和真菌群落多样性分析
由表1可知,经测序后4个产区有效序列数分别为hbcs 10 698条、nxcs 7 846条、plcs 9 828条和qdcs 10 747条,并且所有样品Coverage指数覆盖率达到95%~97%,说明所测的OTU数基本能反映样品的真实情况。Shannon值越大,说明微生物的多样性越高,其中河北怀来赤霞珠表皮微生物的Shannon指数最大(4.18),青岛样品的Shannon指数最小(3.15),证实河北怀来赤霞珠葡萄表皮的细菌微生物多样性最高,而青岛产区的多样性最小。Chao1用来估计物种总数,由数据可知,hbcs、nxcs、plcs、qdcs的数值分别为4 934、5 460、7 979和9 902,表明河北怀来赤霞珠表皮细菌包含的物种总数最少,青岛赤霞珠表皮细菌总数最多。Simpson指数也是表征样品微生物多样性的指标,数值越大,反而多样性越低,表1数据显示河北怀来葡萄样品的Simpson指数最小,其余3个产区的样品Simpson指数没有显著差异(P<0.05),再次验证河北怀来葡萄表面微生物多样性显著高于其他3个产区。
表1 细菌群落Alpha多样性指数
Table 1 Alpha diversity index of bacterial community
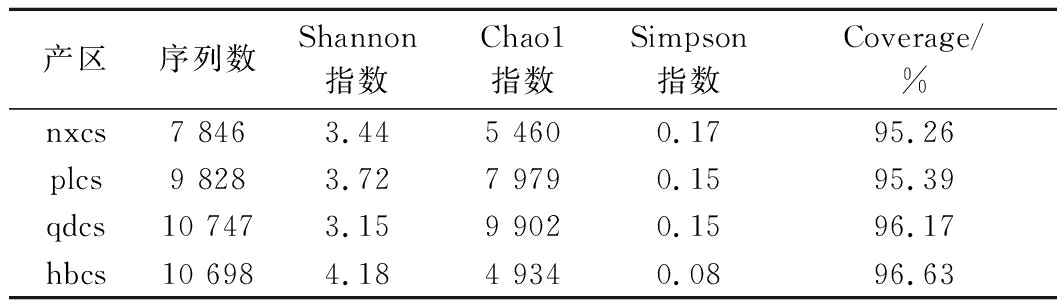
产区序列数Shannon指数Chao1指数Simpson指数Coverage/%nxcs7 8463.445 4600.1795.26plcs9 8283.727 9790.1595.39qdcs10 7473.159 9020.1596.17hbcs10 6984.184 9340.0896.63
真菌菌群Alpha多样性分析结果如表2所示,测序数据经过质控、过滤后,各产区样品得到的有效序列数分别为38 019(nxcs)、55 957(plcs)、53 676(qdcs)、47 464(hbcs),且4个产区Coverage指数的覆盖率都高达99%以上,说明所测的OTU数基本能反映样品的真实情况。其中,河北怀来产区样品的Shannon指数值最大(3.40),Simpson指数最小(0.07),且Chao1指数值亦最大(6 596),说明河北怀来赤霞珠表皮真菌微生物丰富度最大且微生物物种总数最多;而青岛地区赤霞珠葡萄表皮微生物菌群的Shannon指数数值最小(2.17)且Simpson 指数为0.30,说明山东青岛赤霞珠表皮真菌微生物丰富度最小;宁夏贺兰山产区样品的Shannon指数为2.64,但Chao1仅为4 744,说明该地区的赤霞珠葡萄真菌多样性较为丰富,但所含微生物总数较其他产区要少。
表2 真菌群落Alpha多样性指数
Table 2 Alpha diversity index of fungal community
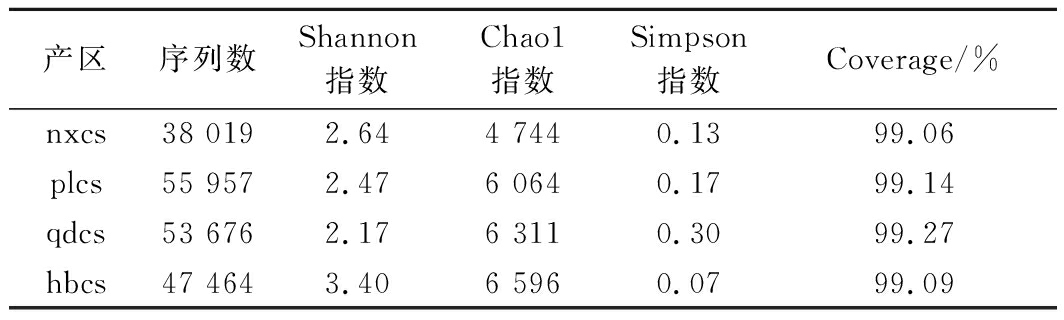
产区序列数Shannon指数Chao1指数Simpson指数Coverage/%nxcs38 0192.644 7440.1399.06plcs55 9572.476 0640.1799.14qdcs53 6762.176 3110.3099.27hbcs47 4643.406 5960.0799.09
综上所述,由Alpha多样性指数分析可知,河北怀来赤霞珠表皮微生物多样性最为丰富且所含真菌微生物数量最多;山东青岛赤霞珠表皮的细菌总数最多但微生物多样性却最低;宁夏贺兰山地区的赤霞珠葡萄真菌多样性较为丰富,但葡萄表面所含的微生物总数较少。
2.4 微生物丰度分析
2.4.1 细菌丰度
由于从样品中检测出的细菌种类高达200多种。为了便于统计,本研究将物种的丰度大于1%作为划分依据,在属水平上选择了排名前20的优势细菌。由图4-a可知,除去未分类的菌属,苯基杆菌属(Phenylobacterium)为河北怀来、山东青岛、宁夏贺兰山产区赤霞珠表皮菌群中含量最高的优势细菌属,相对丰度分别为6%、15%、11%。苯基杆菌属细菌属于典型的土壤微生物,其特征为营养谱单一,不能利用大多数糖、醇,可降解除草剂Pyramin®中的成效成分氯吡嗪(Chloridazon)[10]。上述3个产区的葡萄园中有机农药的使用可能较为频繁,经过长期的自然选择造就了苯基杆菌属这一优势细菌属。而从蓬莱地区样品检测到的优势的菌属为劳尔氏菌属(Ralstonia),相对丰度为8%。该菌能够导致番茄、烟草、桑树等植物枯萎死亡,俗称“青枯病”[11]。噬几丁质菌属(Chitinophaga)在4个产区的葡萄表面细菌群中大量存在,该菌属为一种土壤微生物,有利于植物固氮[12]。不动杆菌属(Acinetobacter)是一类异养硝化细菌,具有较强的脱氮能力,其只在河北怀来的葡萄样品中大量存在,其他3个产区几乎检测不到。这是由于河北怀来近几年为促进葡萄产业发展,推广了一系列促早优质栽培技术,使用含氮磷钾肥料较多,从而促进了该菌属的生长[13]。总体而言,4个产区的葡萄样品中细菌微生物菌群结构相差不大,但特定菌属的相对丰度差异较大,这与气候、田间管理有直接关系[3]。
在“门”水平上,将物种的丰度大于1%作为划分依据,选择排名前7的优势细菌进行分析。由图4-b可知,变形菌门(Proteobacteria)在4个产区的赤霞珠葡萄表皮上均检测到,且丰度在各样品中均最高(约50%~70%)。此外,拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)在4个产区也均可检出。由此可见,4个产区优势菌种在门水平上依旧相差不大,而相对丰度有一定差异,与前面细菌属水平的结果保持一致。张世伟[14]研究表明,酿酒葡萄表面的优势菌门有变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes),本实验结果与其大致相同。相关研究表明,厚壁菌门中大部分细菌属于发酵型细菌,拟杆菌门是一类具有碳水化合物发酵等诸多功能的菌群[15]。绿弯菌门是一类利用CO2,通过光合作用产生能量的细菌,这种菌在有机质含量较低的土壤中很有优势[16]。本实验中河北怀来赤霞珠表皮中绿弯菌门相比于其他产区含量较高,可能是因为河北多沙质土壤,有机质含量低。
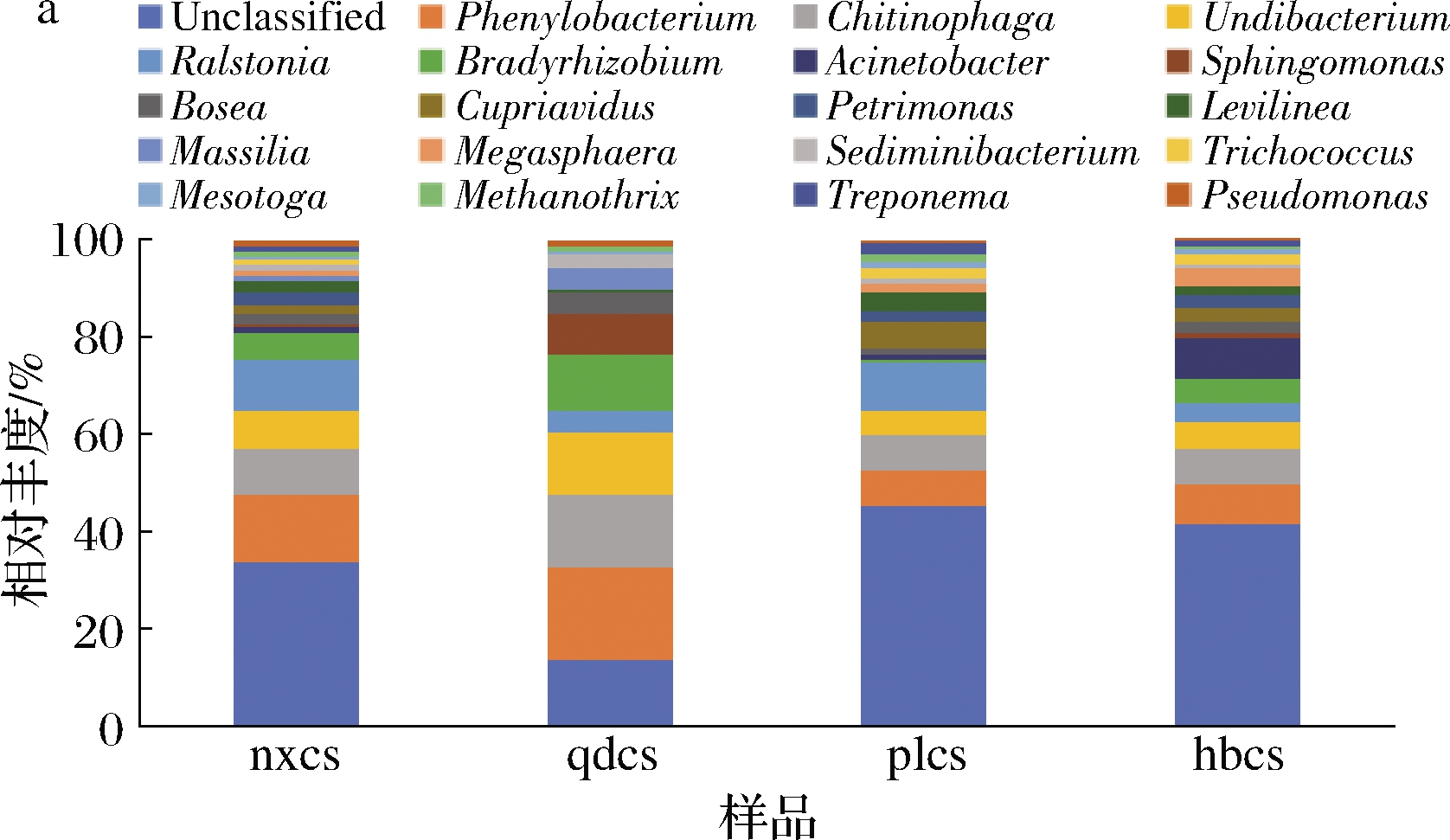
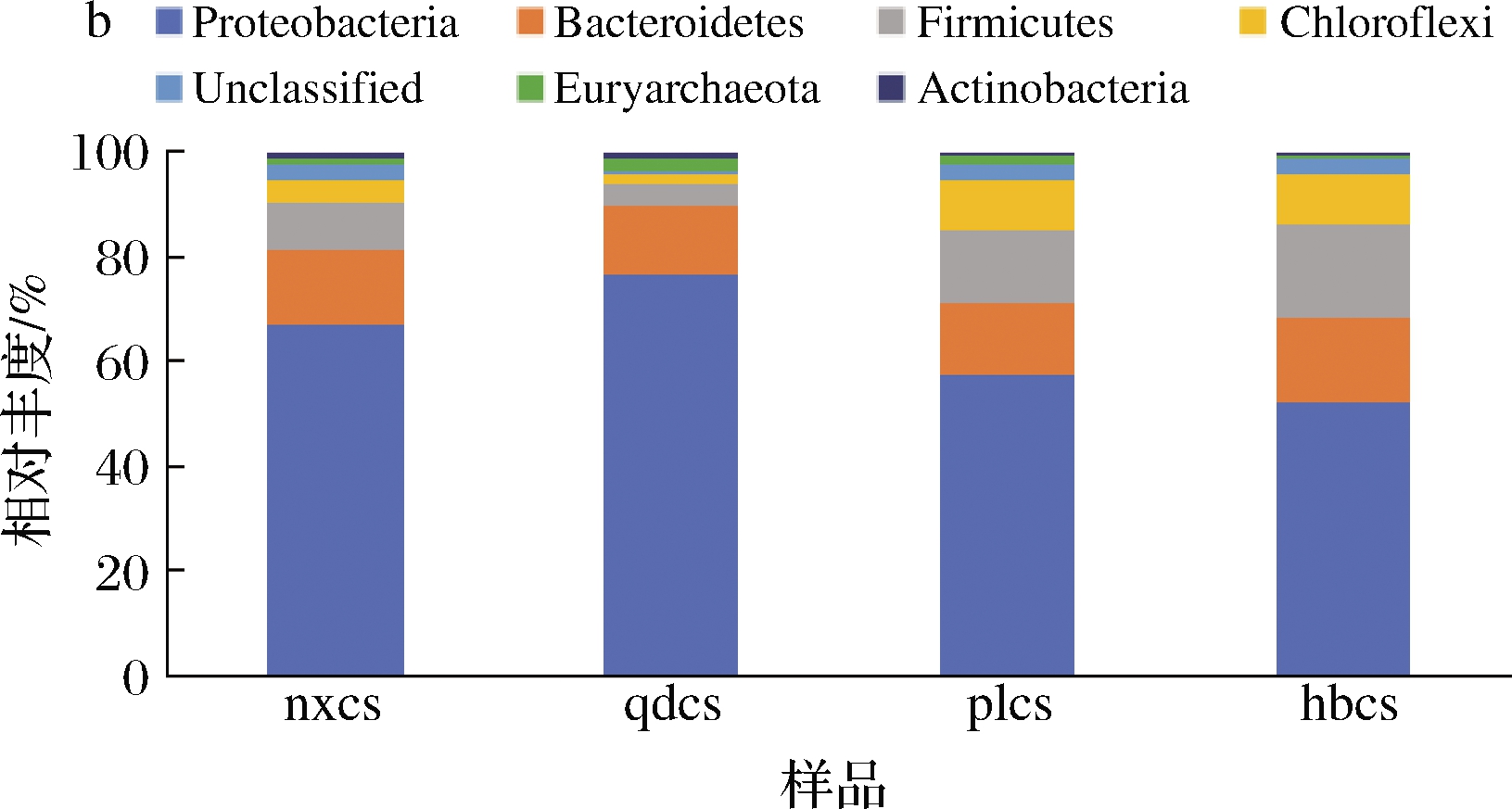
a-属水平;b-门水平
图4 细菌群落属水平和门水平相对丰度
Fig.4 Relative abundance of bacteria community at genus and phylum level
2.4.2 真菌丰度
从样品中检测出的真菌种类繁多,为了便于统计,本研究将物种的丰度大于1%作为划分依据,选择排名前18的优势真菌属进行分析。由图5-a可知,在属水平4个产区赤霞珠葡萄表皮的真菌菌群结构存在一定的差异。宁夏贺兰山产区含量较高的优势菌属为短梗霉属(Aureobasidium),相对丰度为44%。山东青岛和蓬莱产区含量较高的短梗霉属(Aureobasidium)和红酵母属(Rhodotorula),相对丰度之和达到60%。河北怀来产区葡萄表皮真菌菌群结构最为复杂,且各真菌微生物的丰度分布较平均。魏玉洁等[2]利用高通量测序的方法发现葡萄果实和叶片中最丰富的真菌属为短梗霉属,这与本文的研究结果一致。目前,已报到与葡萄酒相关的酵母菌中红酵母属(Rhodotorula)容易在未成熟的浆果表面产生[17]。酿酒酵母属(Saccharomyces)的微生物在4个产区的样品中均出现,但相对丰度并不高,说明葡萄果皮并非酿酒酵母属菌株良好的生活环境。
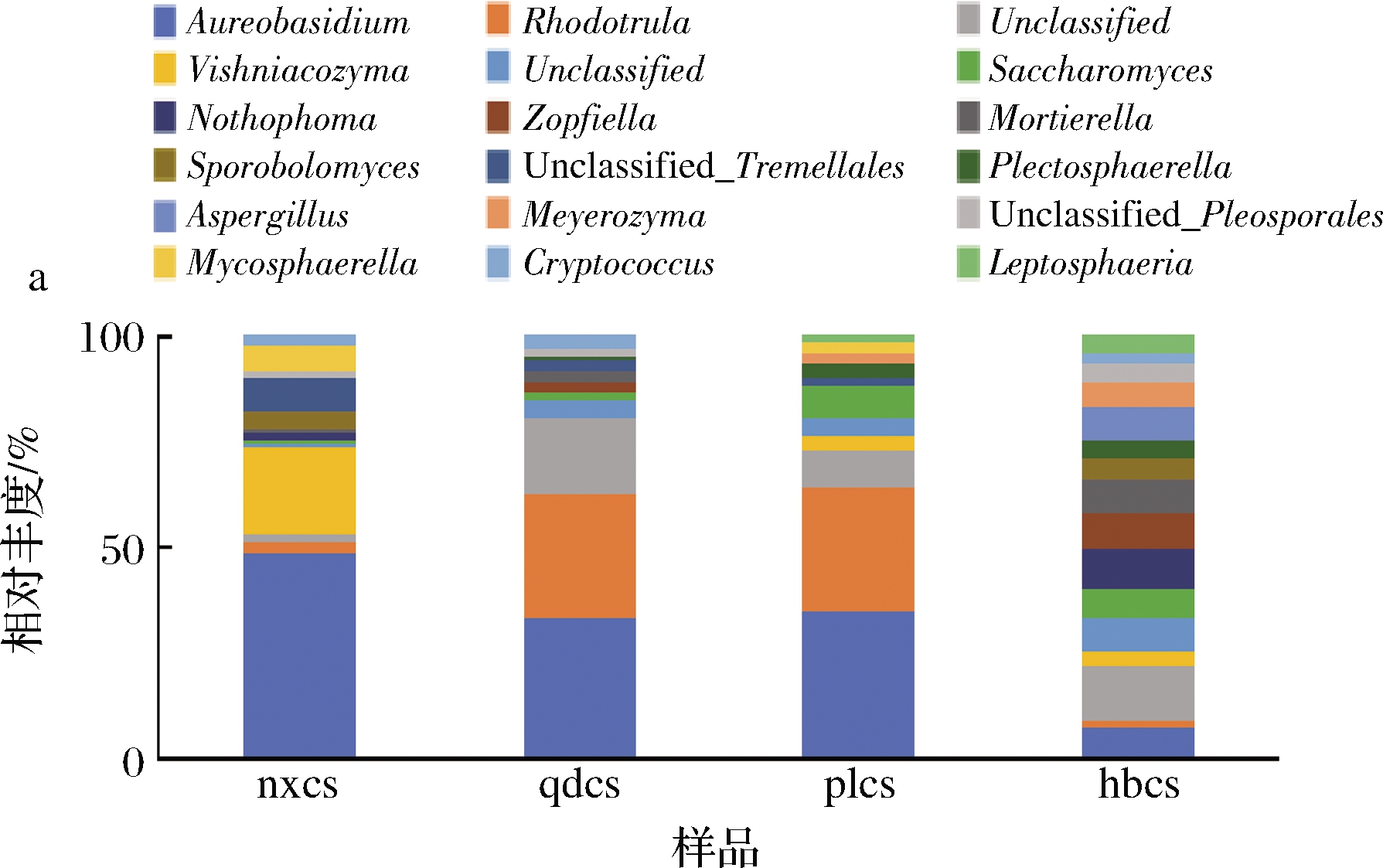

a-属水平;b-门水平
图5 真菌群落属水平和门水平相对丰度
Fig.5 Relative abundance of fungal community at genus and phylum level
图5-b显示,将物种丰度大于1%作为划分依据,4个产区最具有优势的菌门为子囊菌门(Ascomycota),宁夏贺兰山、山东青岛、蓬莱和河北怀来的相对含量分别为59%、68%、88%、70%。其次,担子菌门(Basidiomycota)在4个产区的菌群中含量亦较高。研究表明,子囊菌门和担子菌门中的微生物均为土壤中的优势菌种,能够分解大多数有机物质和植物残骸[18],这与本文的研究结果一致。另外,本研究发现部分真菌菌门独立存在于特定产区,如在青岛样品中检测出特有的丝竹虫门(Cercozoa);河北怀来产区样品中检测到特有的球囊菌门(Glomeromycota),这可能与河北产区的砂质土壤有关。被孢霉门(Mortierellomycota)未在蓬莱产区样品检出,其他3个产区的葡萄表皮样品上均检测到。由此说明在以上4个产区中的葡萄果实上优势真菌群落结构在菌门水平差异不大但相对丰度有明显差异。
2.5 微生物群落结构相似性分析
对4个产区的酿酒葡萄果皮上细菌群落进行基于OTU的主成分分析(principal component analysis,PCA),样本间相似度越高则在图中表现出越聚集。如图6-a显示,当PCA1为97%,PCA2为2%时,4个样品点没有重叠,说明4个产区葡萄皮细菌菌群结构具有一定的差异性。其中,通过各产区样品在PCA1上的分布来看,蓬莱、青岛2个产区的样本距离更为接近,均与宁夏、河北产区的样本相距较远,尤其是与河北产区的样品之间的距离差异最为明显,这说明4个产区中山东青岛、蓬莱的菌群结构较为相似,均与宁夏贺兰山、河北产区的葡萄在细菌群落结构上差异较明显。
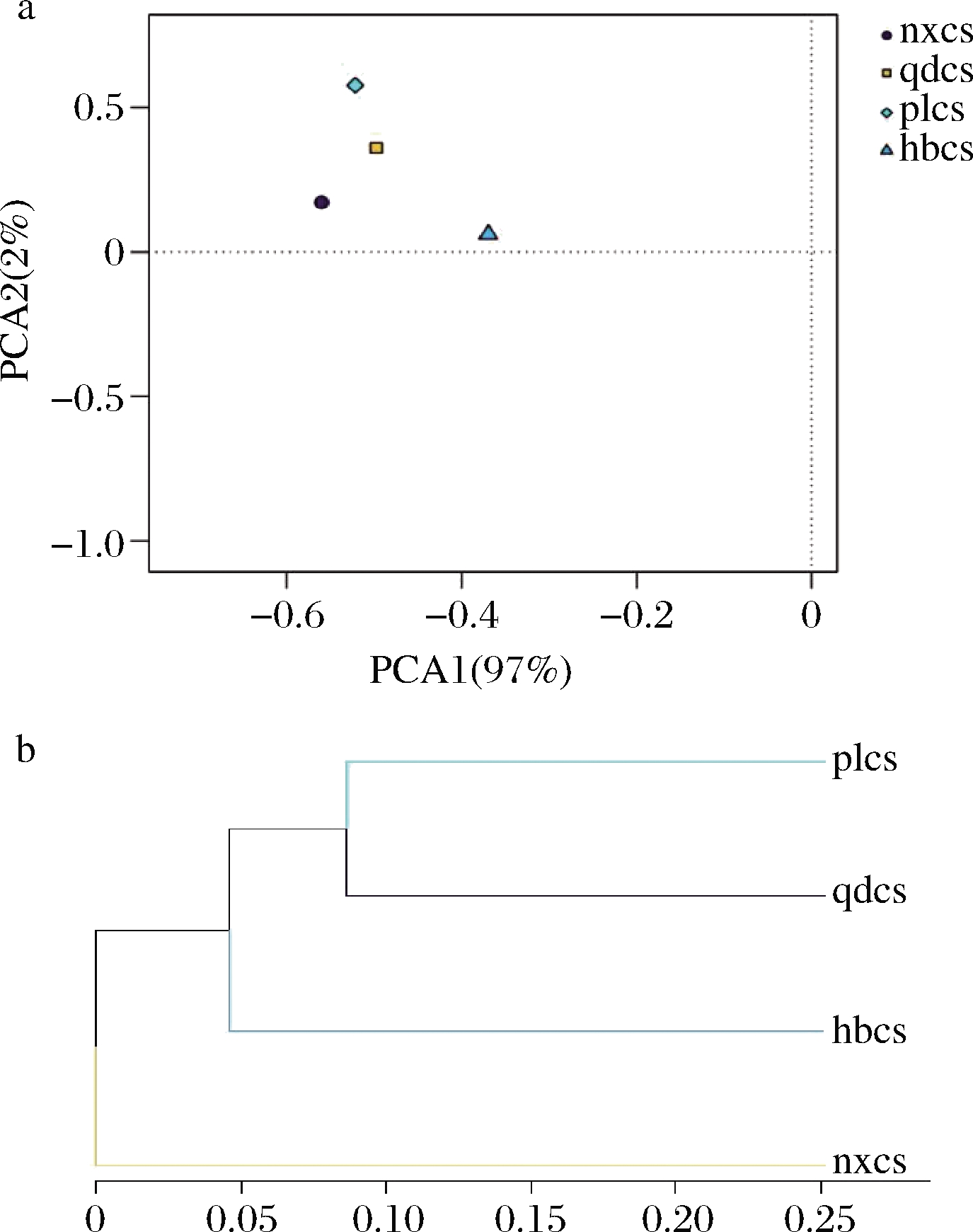
a-主成分分析图;b-聚类分析图
图6 基于OTU的细菌群落主成分分析图和聚类分析图
Fig.6 OTU-based principal component analysisand cluster analysis of bacterial community
样本聚类树图可以通过树枝结构反映出多个样品间的相似性和差异关系。对4个产区细菌群落进行了基于OTU的聚类分析,如图6-b所示。聚类树图树枝的长度代表样本间的距离,越相似的样本会越靠近,图中同一颜色的树枝代表来源于同一组。结果显示,4个样品共聚为三大支,山东青岛和蓬莱产区2个样本归为一类,河北怀来、宁夏贺兰山产区的样品各另聚为一类。说明4个产区赤霞珠表皮微生物除部分差异微生物群落外,山东青岛赤霞珠和山东蓬莱赤霞珠表皮在微生物群落上更为相似,差异较小。宁夏贺兰山产区葡萄表皮微生物群落组成与山东产区样品差异较大,这与PCA结果基本一致。
真菌群落PCA结果如图7-a所示,当PCA1为76%,PCA2为18%时,4个样品点没有重叠,有明显差距,说明4个产区葡萄皮真菌菌群结构存在一定的差异性;从PCA1上各样品点的分布来看,4个产区距离分别都较为接近,说明山东青岛与蓬莱产区、河北怀来与宁夏贺兰山产区的葡萄果皮上大部分真菌微生物相似性较高。同细菌聚类分析的结果类似,如图7-b所示,山东蓬莱、青岛产区的样本中真菌菌群结构聚为一类,说明山东青岛和蓬莱2个地区的葡萄果实上的微生物群落结构差异不明显,这可能与空间距离、气候特征有直接关系。
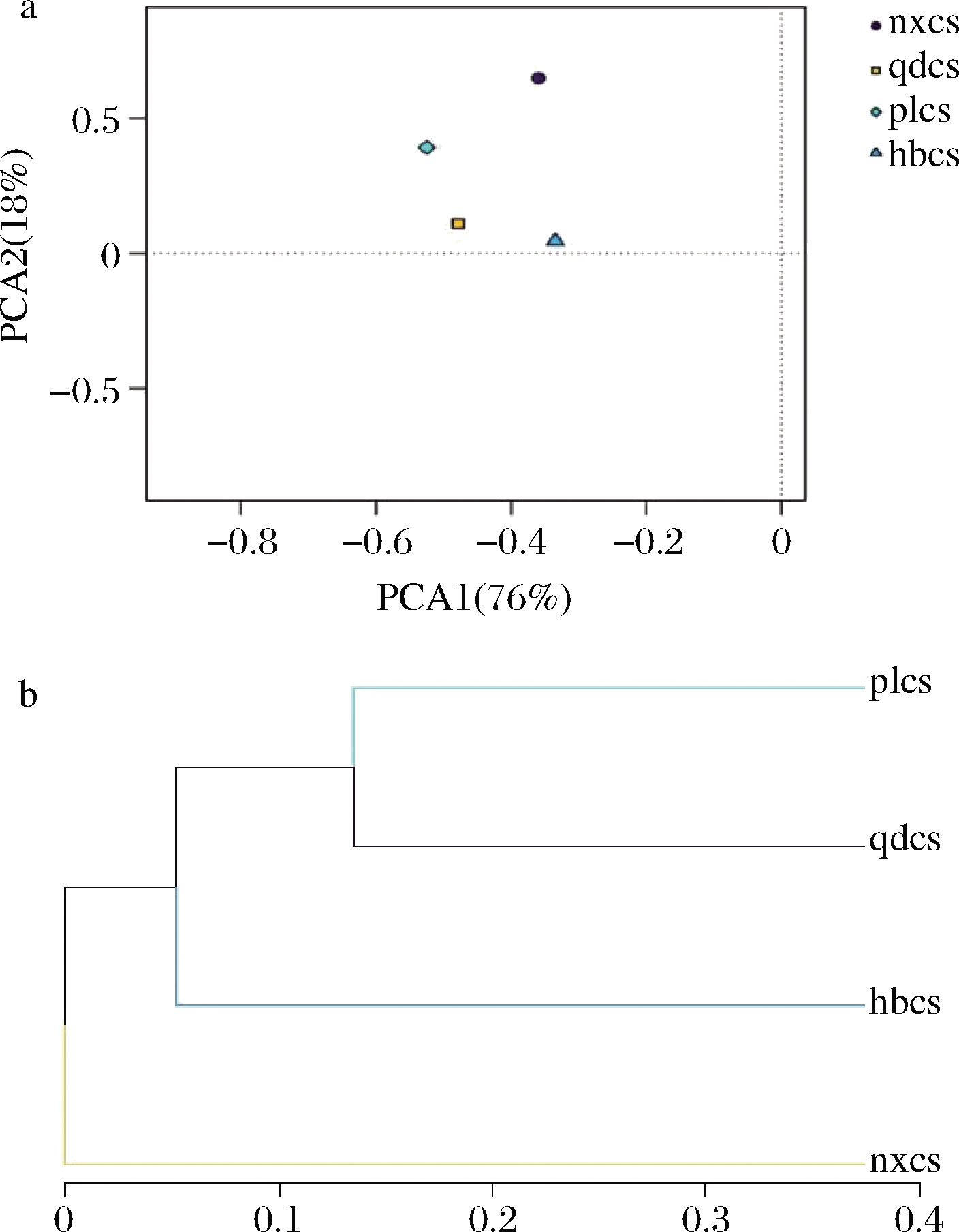
a-主成分分析图;b-聚类分析图
图7 基于OTU的真菌群落主成分分析图和聚类分析图
Fig.7 OTU-based principal component analysis and cluster analysis of fungal community
综上,山东蓬莱、山东青岛的赤霞珠果实表皮微生物的菌群结构较为相似;河北怀来和宁夏贺兰山产区的葡萄样品菌群结构与山东两地的有一定差异,分别各聚为一类。研究表明,酿酒葡萄表皮细菌群落多样性受葡萄园土壤微生物群落的影响[14]。山东蓬莱和山东青岛产区大多为棕壤土,河北怀来土壤为褐土,质地偏砂,宁夏贺兰山土壤以灰钙土为主,疏松多孔。因此,4个产区葡萄表皮微生物菌群结构的不同与各产区不同的土壤条件密切相关。
3 讨论
LEVEAU等[19]发现,霞多丽葡萄上53.5% 的细菌为变形菌门,其他主要有厚壁菌门(15.1%)、拟杆菌门(10.1%)和放线菌门(Actinobacteria,8.0%);MARTINS等[20]发现美乐葡萄上的优势细菌也主要归属于变形菌门。而本研究通过高通量测序分析宁夏贺兰山、河北怀来、山东蓬莱、山东青岛产区的赤霞珠表皮微生物的群落结构及多样性,检测到优势细菌门有变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和绿弯菌门(Chloroflexi)。通过文献对比发现,虽然葡萄品种、所属地区不同,但葡萄果实表面的优势菌种在门水平的分布基本一致。在属水平,苯基杆菌属(Phenylobacterium)为河北怀来、山东青岛、宁夏贺兰山产区赤霞珠表皮上的优势细菌菌种。而蓬莱地区样品检测到的最优势菌属为劳尔氏菌属(Ralstonia)。
葡萄酒酿造相关细菌主要为乳酸菌,起到将苹果酸转化为乳酸的作用,如检测到的明串珠菌属(Leuconostoc)[21]。另外,本研究检测到的厚壁菌门的微生物,发生苹果酸-乳酸发酵后可以有效地降低葡萄酒酸度,避免葡萄酒在装瓶、贮存期间发生二次发酵,增加葡萄酒的微生物学稳定性,从而影响葡萄酒的感官品质。除此之外,大多数细菌微生物对于葡萄酒来说都属于有害菌,会在不同程度上对葡萄酒的品质造成影响,例如检测到的变形菌门(Proteobacteria),是葡萄酒酿造中典型的有害菌[22],导致葡萄酒变质。
4个产区的葡萄表皮微生物共检测到11种真菌菌门,其中优势菌门为子囊菌门(Ascomycota)和担子菌门(Basidiomycota)。在属水平4个产区的真菌菌群结构有一定的差异。宁夏贺兰山产区优势菌属为短梗霉属(Aureobasidium);山东青岛和蓬莱产区含量较高的2个真菌属为短梗霉属(Aureobasidium)和红酵母属(Rhodotorula)。BOKULICH等[23]研究发现增芳德葡萄表面上的优势真菌属为假丝酵母(Candida zemplinina),与本文的研究结果相差较大,可能由于葡萄的栽培方式不同造成。
本研究4个产区的样品中均检测到霉菌,霉菌是一种丝状真菌,通过侵染葡萄的茎、叶、花、果实等影响果实品质。发酵过程若霉菌侵染会产生霉味,造成葡萄酒病害。有研究指出由曲霉属(Aspergillus)产生的赭曲霉素是全球葡萄酒都存在的问题[24],但其存在于葡萄酒中的可能性风险不高,在本研究中仅在河北怀来产区的葡萄表面检出该菌属。酵母菌是葡萄酒酿造的主体,决定葡萄酒的品质,本研究检测到Saccharomyces、Pichia、Candida、Hanseniaspora、Dekkera、Rhodotorula 这6个菌属的菌株,其中,Saccharomyces是酒精发酵的主要完成者,将葡萄中的糖转化成乙醇、CO2及其他代谢产物[25]。
4 结论
本试验通过对我国中、东部地区4个产地赤霞珠葡萄表面微生物多样性进行研究,发现在门水平,4个区域葡萄果皮上的优势细菌均属于变形菌门(Proteobacteria),优势真菌为子囊菌门(Ascomycota)和担子菌门(Basidiomycota)。在属水平上,河北怀来、山东青岛和宁夏贺兰山产区的葡萄样品上的优势细菌为苯基杆菌属(Phenylobacterium);山东蓬莱产区优势菌属为劳尔氏菌属(Ralstonia),宁夏贺兰山地区葡萄的优势真菌为短梗霉属(Aureobasidium),而山东青岛和蓬莱地区丰度较高的真菌属于短梗霉属和红酵母属(Rhodotorula)。结果证实产区是影响葡萄表皮微生物菌群结构的重要因素之一,这可能与气候条件、土壤类型等因素有关。
[1] CAPECE A,ROMANIELLO R,SIESTO G,et al.Selection of indigenous Saccharomyces cerevisiae strains for Nero d’Avola wine and evaluation of selected starter implantation in pilot fermentation[J].International Journal of Food Microbiology,2010,144(1):187-192.
[2] 魏玉洁, 邹弯,马文瑞,等.应用高通量测序技术研究新疆产区葡萄果实、叶片及果园土壤微生物多样性[J].食品科学,2018,39(6):162-170.
WEI Y J,ZOU W,MA W R,et al.Microbial diversity of berries,leaves and soil of grapevine plants grown in Xinjiang analyzed by high-throughput sequencing[J].Food Science,2018,39(6):162-170.
[3] 许维娜, 刘希萌,孔祥君,等.田间施药对自然发酵葡萄酒酵母菌群落结构的影响[J].微生物学通报,2019,46(4):721-728.
XU W N,LIU X M,KONG X J,et al.Effect of pesticide application on the community structure of wine yeasts during spontaneous fermentation[J].Microbiology Sinica,2019,46(4):721-728.
[4] 张世伟, 陈曦,钟其顶,等.不同品种酿酒葡萄表皮微生物群落多样性分析[J].生物技术通报,2017,33(3):128-137.
ZHANG S W,CHEN X,ZHONG Q D,et al.Microbial communities on the wine grape surfaces of different cultivars[J].Biotechnology Bulletin,2017,33(3):128-137.
[5] 王晓雯. 贵州地区葡萄园土壤微生物多样性研究[D].广州:华南理工大学,2017.
WANG X W.Study on microbial diversity in vineyard soil from regions of Guizhou[D].Guangzhou:South China University of Technology,2017.
[6] 张俊杰, 郭晨,尚益民,等.市售夏黑无核葡萄果表微生物多样性研究[J].轻工学报,2017,32(6):20-26.
ZHANG J J,GUO C,SHANG Y M,et al.Study on the microbial diversity on the grape surface of marketed summer black seedless variety[J].Journal of Light Industry,2017,32(6):20-26.
[7] WANG X H,XU X,BAO Z,et al.High-throughput sequencing analysis of microbial diversity in the combined fermentation stages of cow dung and corn straw[J].Food and Fermentation Industries,2019,45(3):47-55.
[8] BOKULICH N A,THORNGATE J H,RICHARDSON P M,et al.Microbial biogeography of wine grapes is conditioned by cultivar,vintage,and climate[J].Proceedings of the National Academy of Sciences of the United States of America,2014,111(1):E139-E148.
[9] MORGAN H H,DU T M,SETATI M E.The grapevine and wine microbiome:Insights from high-throughput amplicon sequencing[J].Frontiers in Microbiology,2017,8:820.
[10] INAM U K,HABIB N,XIAO M,et al.Phenylobacterium terrae sp.nov.,isolated from a soil sample of Khyber-Pakhtun-Khwa,Pakistan[J].Antonie van Leeuwenhoek,2018,111(10):1 767-1 775.
[11] CHANDRASEKARAN M,SUBRAMANIAND,YOON E,et al.Meta-analysis reveals that the genus Pseudomonas can be a better choice of biological control agent against bacterial wilt disease caused by Ralstonia solanacearum [J].The Plant Pathology Journal,2016,32(3):216-227.
[12] SIROVA D,SANTRUCEK J,ADAMEC L,et al.Dinitrogen fixation associated with shoots of aquatic carnivorous plants:Is it ecologically important?[J].Annals of Botany,2014,114(1):125-133.
[13] YANG J R,WANG Y,CHEN H,et al.Ammonium removal characteristics of an acid-resistant bacterium Acinetobacter sp.JR1 from pharmaceutical wastewater capable of heterotrophic nitrification-aerobic denitrification[J].Bioresource Technology,2019,274:56-64.
[14] 张世伟.酿酒葡萄微生物群落多样性及其氮代谢通量研究[D].北京:中国矿业大学(北京),2018.
ZHANG S W.Study on microbial community diversity and the nitrogen metabolism flux of wine grapes[D].Beijing:China University of Mining and Technology(Beijing),2018.
[15] 杨敏, 殷绒,张国涛,等.基于高通量测序技术的香格里拉葡萄酒产区根际微生物多样性研究[J].云南农业大学学报(自然科学版),2020,35(3):392-400.
YANG M,YIN R,ZHANG G T,et al.Study on the grape rhizosphere microbial diversity in Shangri-La wine region by high-throughput sequencing technology[J].Journal of Yunnan Agricultural University(Natural Science Edition),2020,35(3):392-400.
[16] 刘思. 贺兰山东麓葡萄园行内覆盖对微域生态环境、葡萄及葡萄酒品质的影响[D].银川:宁夏大学,2019.
LIU S,Effects of within-row mulching on micro-ecological environment grape and wine quality in vineyards of eastern foothill of Helan mountain[D].Yinchuan:Ningxia University,2019.
[17] 焦红茹. 不同酿酒葡萄品种相关酵母菌的分离及分类鉴定[D].杨凌:西北农林科技大学,2008.
JIAO H R.Isolation and identification of wine-related yeast associated to different varieties[D].Yangling:Northwest A&F University,2008.
[18] 侯芳彬, 王蕊,SALMAN A,等.黄土区沟道泥沙微生物群落变化特征及其影响因素[J].中国环境科学,2019,39(10):4 350-4 359.
HOU F B,WANG R,SALMAN A,et al.Variations of soil microbial communities along a valley bottom of the loess plateau and the influencing factors[J].China Environmental Science,2019,39(10):4 350-4 359.
[19] LEVEAU J H J,TECH J J.Grapevine microbiomics:Bacterial diversity on grape leaves and berries revealed by high-throughput sequence analysis of 16S rRNA amplicons[J].Acta Horticulturae,2011(905):31-42.
[20] MARTINS G,LAUGA B,MIOT-SERTIER C,et al.Characterization of epiphytic bacterial communities from grapes,leaves,bark and soil of grapevine plants grown,and their relations[J].PLoS One,2013,8(8):e73 013.
[21] 金刚, 王华,张昂,等.中国葡萄酒产区酒酒球菌种质资源遗传多样性分析[J].食品科学,2015,36(17):134-139.
JIN G,WANG H,ZHANG A,et al.Genetic diversity analysis of Oenococcus oeni strains isolated from Chinese wines[J].Food Science,2015,36(17):134-139.
[22] 屈慧鸽.葡萄酒生产过程中醋酸菌的危害及影响因素分析[J].酿酒科技,2009(2):43-46.
QU H G.Analysis of the hazard and the influencing factors of acetic acid bacteria in the production of grape wine[J].Liquor-Making Science & Technology,2009(2):43-46.
[23] BOKULICH N A,JOSEPH C M L,ALLEN G,et al.Next-generation sequencing reveals significant bacterial diversity of botrytized wine[J].PLoS One,2012,7(5):e36 357.
[24] GARMENDIA G,VERO S.Occurrence and biodiversity of Aspergillus section Nigri on ‘Tannat’ grapes in Uruguay[J].International Journal of Food Microbiology,2016,216:31-39.
[25] HONG Y A,PARK H D.Role of non-Saccharomyces yeasts in Korean wines produced from Campbell early grapes:Potential use of Hanseniaspora uvarum as a starter culture[J].Food Microbiology,2013,34(1):207-214.