嘧啶核苷包括尿嘧啶核苷(尿苷,uridine)和胞嘧啶核苷(胞苷,cytidine),广泛应用在食品、保健品、化妆品和医药等诸多行业。作为抗病毒抗肿瘤药物的前体[1],嘧啶核苷是生物医药研究必不可少的原料。随着抗病毒抗肿瘤药物研发的深入以及嘧啶核苷功能性研究的不断拓展,嘧啶核苷的市场需求不断扩大,迫切需要开发廉价的可规模化应用的嘧啶核苷生产工艺。微生物发酵法具有原料来源广泛、生产效率高和环境友好可持续等诸多优势[2],是当前嘧啶核苷生产研究的重点。
嘧啶核苷的生物合成可分为3部分(图1):首先谷氨酰胺、碳酸氢盐、腺嘌呤核苷三磷酸(adenosine triphosphate,ATP)、天冬氨酸和5-磷酸核糖-1-焦磷酸(5-phosphoribosyl 1-pyrophosphate,PRPP)等前体物质经6步酶促反应生成尿苷酸(uridylic monophosphate,UMP),然后一部分UMP直接脱去磷酸基团生成尿苷,一部分UMP则经过一系列的磷酸化,胺化再去磷酸后生成胞苷[3]。该合成途径会受到严谨的调控,如在大肠杆菌中,嘧啶核苷操纵子基因转录会受到尿苷酸或者胞苷酸的反馈阻遏,且催化第一步反应的氨甲酰磷酸合成酶的活性还受到UMP的反馈抑制[4-5]。另外,胞苷合成的关键酶UMP激酶和三磷酸胞苷(cytidine triphosphate,CTP)合酶的活性还分别受到三磷酸尿苷(uridine triphosphate,UTP)和CTP的反馈抑制[5]。严谨的反馈调控机制,冗长复杂的反应过程以及对多种重要前体物的需求是限制嘧啶核苷生物合成的主要因素。早期研究中,通过诱变筛选结构类似物抗性菌株是选育嘧啶核苷生产菌的主要方法,但是诱变菌株往往存在负向突变的累积,菌株的持续改良也受限制[6-7]。随着系统生物学的发展,代谢工程等理性构建的方法逐渐成为工业菌株从头构建和持续改良的首选方法[8-9]。WU等[4]和FAN等[10]综合运用高通量筛选和代谢工程的方法构建了1株产尿苷的菌株UR6,5 L罐上发酵64 h,可生产70.3 g/L的尿苷。YANG等[11]通过阻断降解途径,促进关键酶的表达,增强前体物的供应等代谢工程策略的运用,成功构建产胞苷的工程菌株。但是现存胞苷工程菌株的发酵性能还较低,很难应用于胞苷的工业化生产。
优化胞苷合成途径,构建高产胞苷的微生物细胞工厂,逐步提高菌株的胞苷发酵指标,对胞苷发酵生产意义重大。课题组前期构建的尿苷高产菌UR6的UMP从头合成途径已经得到了有效强化,本研究接着对UR6的胞苷合成途径进行了系统的代谢工程改造(图1),期望通过强化胞苷合成途径,将UR6逐步改造为胞苷的高产菌株。主要考察了阻断胞苷及其前体物的降解途径,过表达关键酶增强胞苷合成途径,以及融合蛋白表达改变UMP分别到胞苷和尿苷的流量分配对大肠杆菌嘧啶核苷发酵的影响,为胞苷高产菌株的构建提供新的借鉴和参考。
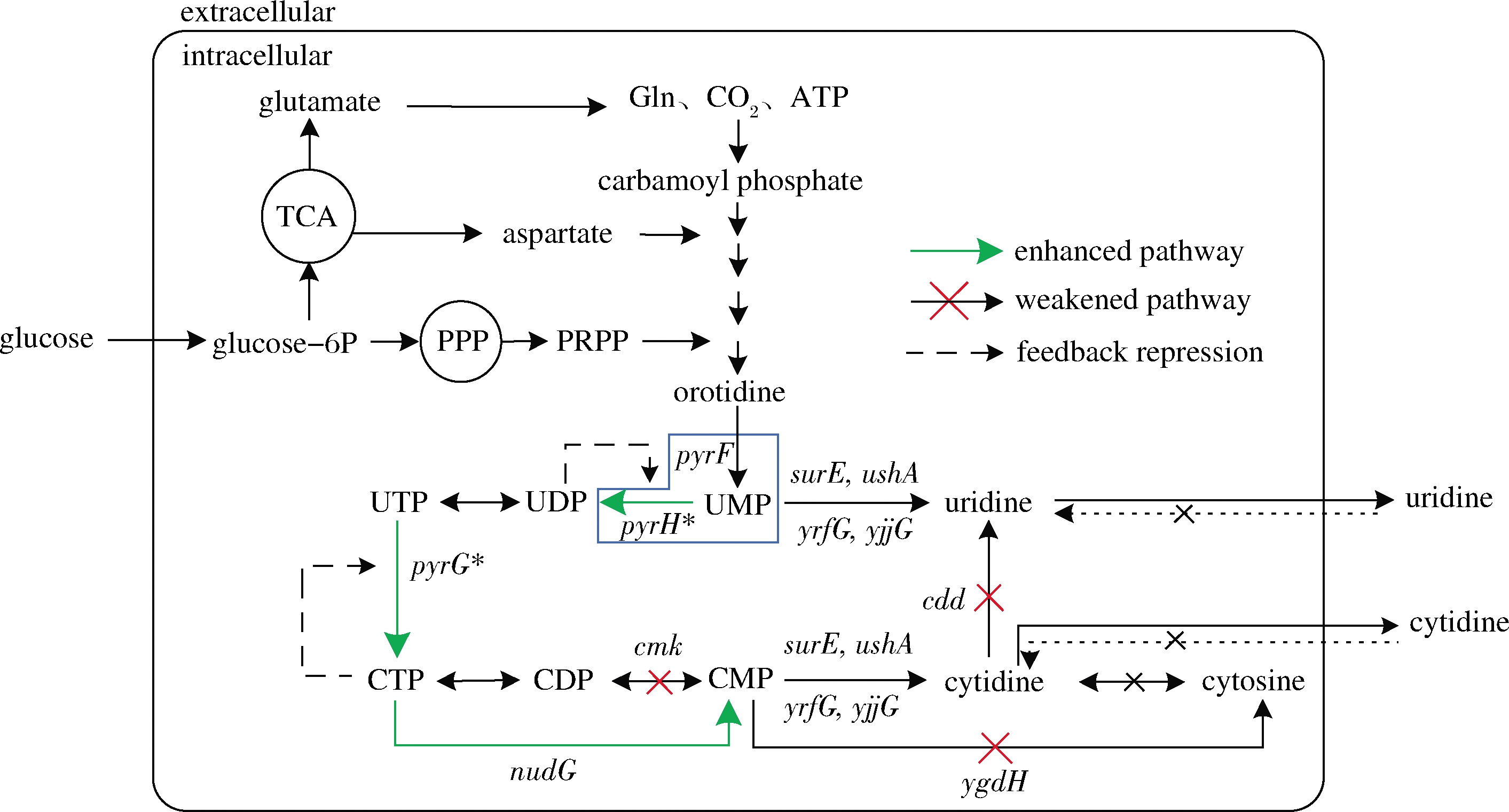
图1 嘧啶核苷生物合成途径和本研究的代谢工程改造策略
Fig.1 Biosynthetic pathway of pyrimidine nucleoside and the metabolic engineering strategies used in this study 注:CMP为胞嘧啶核苷酸(cytidine monophosphate);CDP为二磷酸胞苷(cytidine diphosphate);UDP为二磷酸尿苷(uridine diphosphate); TCA为三羧酸循环(tricarboxylic acid cycle);PPP为磷酸戊糖途径(pentose phosphate pathway)
1 材料与方法
1.1 菌株和质粒
本研究所涉及的菌株和质粒见表1。
表1 本研究中涉及的菌株和质粒
Table 1 Bacterial strains and plasmids used in this study.

菌株/质粒基因型来源E.coli DH5αthe cloning host本研究室UR6pyrBCAKDFEbsu, trpR∶prseco, tyrR∶Ptrc-pyrAA/AB(E949∗), tdcD∶Ptrc-pyrB,ΔnupC/G, Δupp,Δudp,ΔpepA,ΔpurR,ΔargR,ΔrihA/B/C,ΔargF,Δudk本研究室CYT1-1UR6,Δcdd本研究CYT1-2UR6, Δcmk本研究CYT1-3UR6, ΔygdH本研究CYT1-4UR6, Δcdd, Δcmk本研究CYT1-5UR6, Δcdd, Δcmk, ΔygdH本研究CYT2-1CYT1-5, ilvG∶Ptrc-pyrH∗本研究CYT2-2CYT1-5, tehB∶Ptrc-pyrG∗本研究CYT2-3CYT1-5, yeep∶Ptrc-nudG本研究CYT2-4CYT1-5, ilvG∶Ptrc-pyrH∗, tehB∶Ptrc-pyrG∗本研究CYT2-5CYT1-5, ilvG∶Ptrc-pyrH∗, tehB∶Ptrc-pyrG∗, yeep∶Ptrc-nudG,本研究CYT3-1CYT2-5, ΔsurE, ΔushA,ΔyjjG本研究CYT3-2CYT2-5, ΔushA, ΔyjjG,ΔyrfG本研究CYT3-3CYT2-5, ΔyjjG, ΔsurE, ΔyrfG本研究CYT3-4CYT2-5, ΔyrfG, ΔsurE, ΔushA本研究CYT3-5CYT2-5, mbhA∶Ptrc-phm8本研究CYT4-1CYT3-5, ΔpyrFbsu本研究CYT4-2CYT3-5, ΔpyrFecj本研究CYT4-3CYT4-2, yjiT∶Ptrc-pyrFecj-[(GGT)4(TCA)]3-pyrH∗本研究CYT4-4CYT4-2, yjiT∶Ptrc-pyrH∗-[(GGT)4(TCA)]3-pyrFecj本研究CYT4-5CYT3-5, yjiT∶Ptrc-pyrH∗本研究pREDCas9Sper, Cas9 and λ-Red recombinase expression vecto[12]pGRBAmpr, gRNA expression vector[12]
1.2 主要试剂
引物及基因,苏州金唯智科技有限公司;Primer STAR HS DNA聚合酶,大连宝生物科技有限公司;2×Rapid Taq Mix、ClonExpress® Ⅱ One Step Cloning Kit,南京诺唯赞生物科技有限公司;质粒快速提取试剂盒、DNA凝胶纯化回收试剂盒、胞苷和尿苷标准品,美国Omega公司;其余生化试剂为进口或国产分析纯试剂。
1.3 培养基
斜面培养基(g/L):葡萄糖 1,蛋白胨 10,牛肉膏 10,酵母粉 5,NaCl 2.5,琼脂 20。
种子培养基(g/L):酵母粉 5,蛋白胨 3,KH2PO4·3H2O 1.2,MgSO4·7 H2O 0.5,FeSO4·7 H2O 0.01,MnSO4·H2O 0.01,苯酚红 0.008,VB1、VB3、VB5、VB12、VH各0.001。
发酵培养基(g/L):酵母粉 4,蛋白胨 5,柠檬酸钠 2,KH2PO4·3 H2O 2,MgSO4·7 H2O 2, FeSO4·7 H2O 0.02,MnSO4·H2O 0.02,苯酚红 0.008,VB1、VB3、VB5、VB12、VH各0.002。
1.4 基因编辑方法
本研究采用LI等[12]报道的CRISPR/Cas9介导的基因组编辑方法进行工程菌株的构建。该方法中CRISPR-Cas9系统包括pGRB和pREDCas9两个质粒,其中pREDCas9携带pGRB的消除系统、λ噬菌体的Red重组系统及Cas9蛋白表达系统,携带奇霉素抗性(工作质量浓度为100 mg/L),32 ℃培养。pGRB以pUC18为骨架,包含gRNA-Cas9结合区域序列和终止子序列,携带氨苄青霉素抗性(工作质量浓度为100 mg/L),37 ℃培养。
1.4.1 pGRB质粒和重组片段的构建
为构建含有不同靶位点序列的重组pGRB质粒,首先使用工具CRISPR RGEN Tools选择靶序列(PAM:5′-NGG-3′),设计合成2条完全反向互补的单链DNA,然后通过退火形成双链DNA,该双链DNA中间序列是靶位点的特定gRNA间隔序列,两端序列与pGRB具有同源序列。利用ClonExpress® Ⅱ One Step Cloning Kit,将双链DNA与线性化pGRB通过同源重组构建出含有靶点序列的重组pGRB质粒。
构建重组片段时,先利用引物设计软件primer 5,以待敲除基因或待整合位点的上下游序列为模板,设计上下游同源臂引物(扩增长度约400~500 bp)。以待整合基因为模板,设计整合基因的扩增引物,扩增上下游同源臂和目的基因片段后,通过重叠PCR的方法连接成重组片段。
1.4.2 DNA片段重组
制备出发菌株的感受态细胞,先将pREDCas9质粒化转至感受态细胞,平板培养并筛选阳性转化子。将阳性菌株接种到含LB培养基的摇管中培养12 h,再接种于2 XYT培养基(蛋白胨16 g/L,酵母粉10 g/L,NaCl 5 g/L,奇霉素100 mg/L)中培养,待OD600值为0.1~0.2时加入异丙基-β-D-硫代半乳糖苷,OD600增至0.3~0.4时收集菌体,用10%(体积分数)的甘油洗涤,制备电转化感受态。将重组pGRB质粒和重组片段同时电转化至感受态细胞中,复苏2 h后涂布在含氨苄青霉素和奇霉素抗性的LB平板上,32 ℃培养。待长出单菌落后,通过菌落PCR筛选阳性转化子,再消除pGRB和pREDCas9质粒,获得目标菌株。
1.5 摇瓶培养方法
用接种环刮取1环斜面种子接种于装有30 mL种子培养基的500 mL三角瓶中,9层纱布封口,37 ℃,200 r/min振荡培养8 h;按10%接种量接种到装有发酵培养基的500 mL三角瓶中(终体积为30 mL),9层纱布封口,37 ℃,200 r/min振荡培养;发酵过程中通过补加氨水维持pH值为7.0~7.2;初始葡萄糖耗尽后,补加600 g/L葡萄糖溶液维持发酵进行,发酵周期为24 h。
1.6 检测方法
利用分光光度计测量发酵液在600 nm处的吸光度(OD600)表征生物量;采用HPLC对尿苷和胞苷浓度进行定量分析:所用仪器为Thermo U3000,色谱柱为phenomenex Gemini 5u C18 110A(150 mm×4.6 mm);流动相为V(乙腈)∶V(水)=2∶98;流速为1.0 mL/min;柱温为30 ℃;进样量为20 μL;紫外检测器波长为270 nm。
1.7 数据分析方法
发酵数据代表3组平行发酵数据的平均值和标准差。利用T检验双尾分布对改造菌和对应出发菌的2组发酵参数进行单向方差分析。“*”表示P<0.05,说明结果具有显著的统计学差异;“**”表示P<0.01,说明结果具有极显著的统计学差异。
2 结果与分析
2.1 阻断胞苷及前体物降解途径对嘧啶核苷发酵的影响
阻断降解途径是促使目标产物过量积累的重要方法。胞苷或尿苷可由核苷水解酶(rihA/B/C,UR6中此3个基因已被敲除)水解为胞嘧啶或尿嘧啶,也可被胞苷/尿苷激酶(udk,UR6中此基因也已敲除)磷酸化为CMP和UMP,同胞苷或者尿苷的合成方向相悖[4]。此外,胞苷还可被胞苷脱氨酶(cdd)降解为尿苷[13]。另外,胞苷合成的直接前体物CMP可在核苷酶(ygdH)的作用下降解为胞嘧啶[14],也可在胞苷酸激酶(cmk)的作用下磷酸化成CDP[15],是胞苷合成的逆向反应。
分别敲除UR6中的cdd,cmk和ygdH基因,构建了菌株CYT1-1,CYT1-2和CYT1-3。摇瓶发酵结果如图2所示,摇瓶发酵24 h后,4株菌的尿苷产量基本相同,且只有CYT1-1积累了0.2 g/L胞苷,说明胞苷脱氨酶的敲除是胞苷积累的关键因素,而cmk基因和ygdH基因的单敲除对发酵结果基本无影响。逐一敲除CYT1-1的cmk和ygdH基因后,所得菌株CYT1-4和CYT1-5的胞苷产量分别提高1.3和2.8 g/L,尿苷产量变化不明显。结果表明,胞苷脱氨酶催化的反应是胞苷的主要降解途径,同先前的研究报道相符[11]。而cmk基因和ygdH基因的敲除,切断胞苷合成主要前体物质CMP的主要分支代谢,有利于提高胞内CMP的浓度,进而促进胞苷的生成。
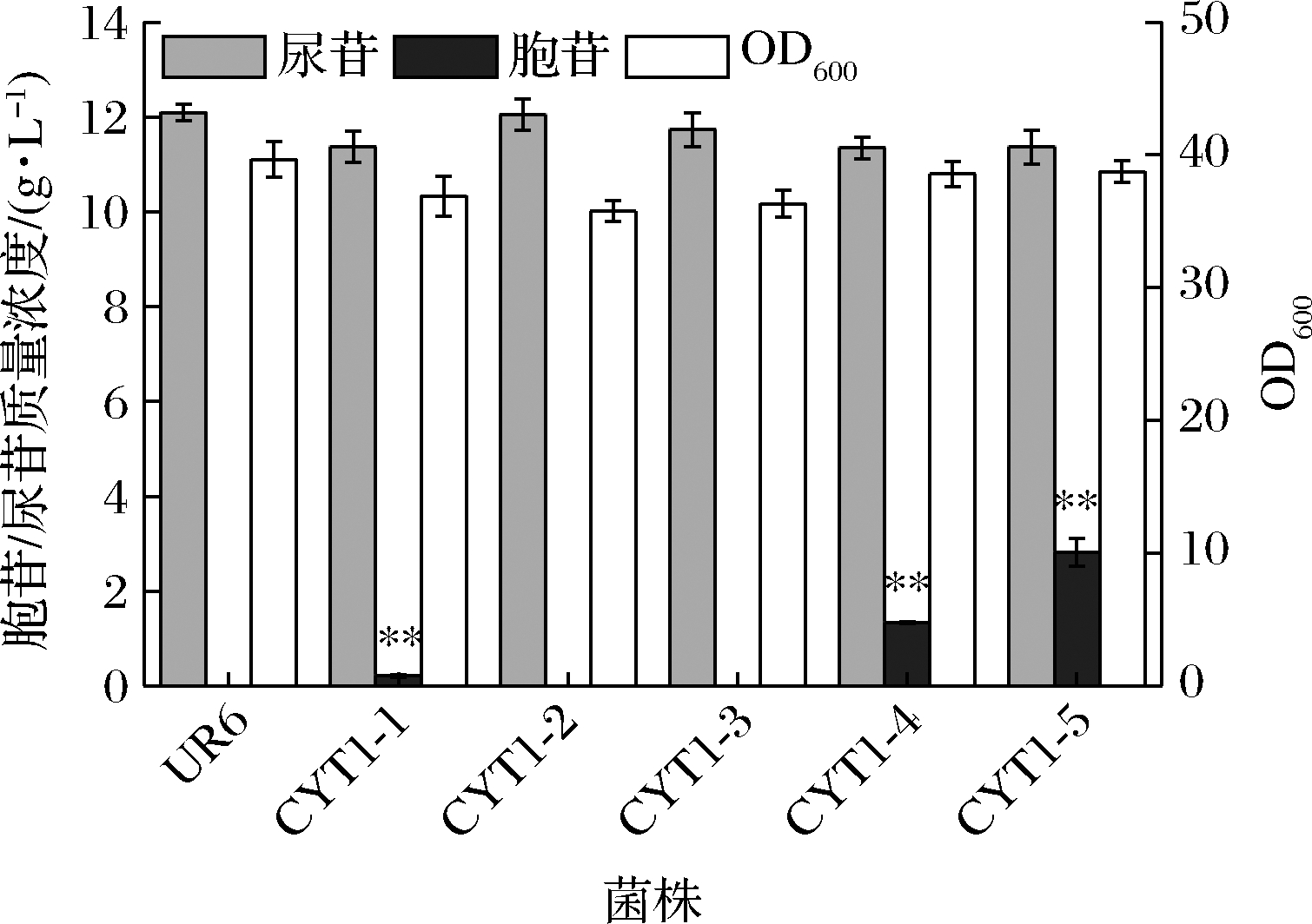
图2 cdd、cmk和ygdH基因的敲除对嘧啶核苷 发酵的影响
Fig.2 Effect of knockout of cdd, cmk and ygdH genes on pyrimidine nucleoside fermentation 注:“*”代表差异显著(P<0.05);“**”代表差异极显著(P<0.01)(下同)
2.2 加强胞苷合成途径对嘧啶核苷发酵的影响
为进一步提高胞苷产量,本研究接着对CYT1-5胞苷合成途径的主要限速步骤进行了强化。UMP由尿苷酸激酶(pyrH基因编码)催化生成UDP是UMP合成胞苷的第一个限速步骤,而尿苷酸激酶的催化活性受三磷酸鸟苷 (guanosine triphosphate, GTP)的变构激活和UTP的变构抑制。研究表明,将大肠杆菌尿苷酸激酶第93位的天冬氨酸替换为丙氨酸可降低该酶与UTP的结合从而减弱尿苷酸激酶所受的反馈抑制作用[16]。胞苷合成途径的第二个限速步骤是pyrG基因编码的CTP合酶所催化的反应,该反应受到CTP的反馈抑制。据报道,在谷氨酸棒状杆菌的CTP合酶中引入3个位点的突变(D160E,E162A,E168K),所得CTP合酶突变体所受CTP的抑制作用大幅减弱[17]。另外,由nudG编码的CTP二磷酸酶可催化CTP至CMP的反应。强化nudG基因的转录表达理论上也有利于胞苷的生成。
本研究首先在CYT1-5中的ilvG、tehB和yeeP 3个假基因位点分别整合了PyrHecj(D93A)、PyrGcgl(D160E、E162、E168K)的编码基因pyrH*和pyrG*,以及nudG基因,并都由启动子Ptrc启动,构建了菌株CYT2-1、CYT2-2和CYT2-3。摇瓶发酵结果如图3所示,CYT2-1的胞苷和尿苷产量分别为4.2和7.4 g/L,相比于CYT1-5,胞苷产量提高了50%,尿苷产量降低了35.1%。另外,CYT2-1的OD600值为27.5,较CYT1-5降低了29%;CYT2-2的发酵结果同CYT1-5基本无变化;CYT2-3的胞苷产量为4.4 g/L,较CYT1-5提高了57.1%,而尿苷产量和OD600值与CYT1-5相比基本无变化。结果表明,PyrHecj(D93A)的表达有效促进了胞苷的积累,但是中间代谢物如UDP或UTP的浓度也可能提高,从而造成代谢不平衡妨碍了菌体的生长;PyrGcgl(D160E、E162A、E168K)在CYT1-5中的表达对其嘧啶核苷的发酵基本无影响,分析可能是底物UTP供应不足,于是本研究对PyrHecj(D93A)和PyrGcgl(D160E、E162A、E168K)进行了组合表达,构建了菌株CYT2-4,摇瓶发酵结果显示,CYT2-4的胞苷和尿苷产量分别为4.9和8.4 g/L,相较于CYT2-2,胞苷产量有了显著提升,而尿苷产量和菌体生长情况也得以恢复,说明这2个酶的组合过表达更有利于胞苷合成途径的强化,有效促进了胞苷的积累。最后在CYT2-4上对nudG基因进行了双拷贝,构建了菌株CYT2-5,摇瓶结果显示CYT2-5的胞苷和尿苷产量分别为5.6和9.3 g/L,OD600值为34.5。相较于CYT2-4,CYT2-5的胞苷产量提升了14.3%,尿苷产量提升了12.0%,菌体生长基本无变化。
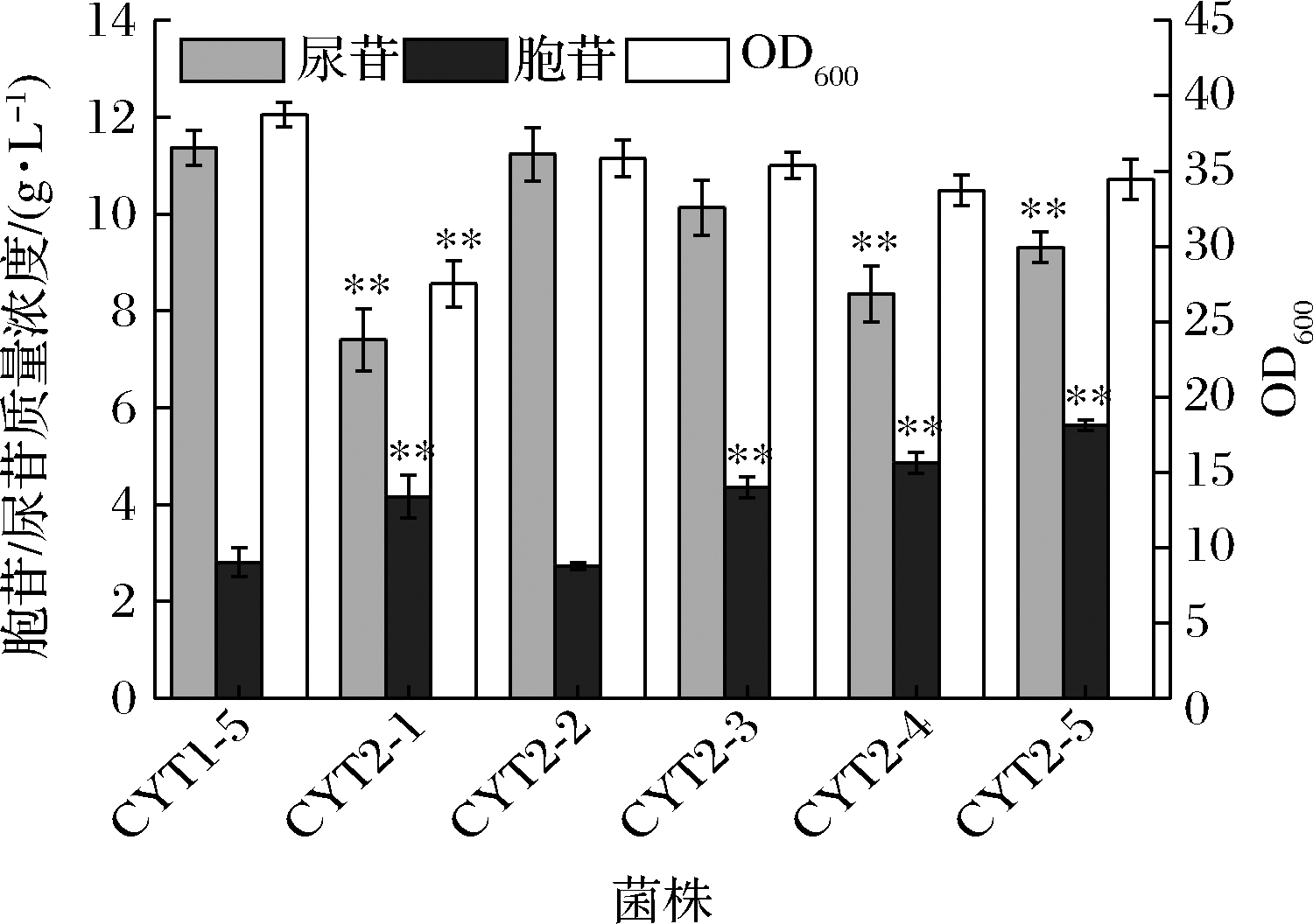
图3 pyrH*、pyrG*和nudG基因的整合对 嘧啶核苷发酵的影响
Fig.3 Effect of integration of pyrH*、pyrG* and nudG genes on pyrimidine nucleoside fermentation
突变体PyrHecj(D93A)和PyrGcgl(D160E、E162A、E168K)的组合表达以及内源nudG基因表达的强化有效促进了胞苷的积累。但是尽管胞苷产量得到了显著提升,CYT2-5中主要产物仍旧是尿苷,需进一步改变UMP的流量分配,将更多地UMP导向胞苷的合成。
2.3 5′-核苷酸酶对嘧啶核苷发酵的影响
从前述结果来看,在嘧啶核苷合成代谢中,尿苷是优先合成的,原因可能有两方面:一是因为胞苷合成的直接前体CMP来源于尿苷合成的直接前体UMP,且UMP向CMP的合成还受到严谨的调控,因此相比CMP,胞内更容易积累UMP,从而有利于尿苷的合成;二是大肠杆菌中,5′-核苷酸酶的专一性较弱,嘌呤和嘧啶核苷酸都可被5′-核苷酸酶催化生成相应的核苷,而且相对于胞苷酸,大肠杆菌中的5′-核苷酸酶可能对UMP的亲和力更强,因此在前体物UMP充足的条件下,细胞更容易积累尿苷。选择对UMP亲和力较弱,而对CMP具有更高亲和力,甚至专一性结合CMP的5′-核苷酸酶会更有利于胞苷的合成。
大肠杆菌中已被注释的编码5′-核苷酸酶的基因有4个,分别是ushA、yrfG、yjjG和surE。为了验证这4个基因所编码的4种5′-核苷酸酶对UMP和CMP的亲和力,本研究首先将CYT2-5中ushA、yrfG、yjjG和surE 4个基因进行了组合敲除,构建了只含1种5′-核苷酸酶的菌株CYT3-1、CYT3-2、CYT3-3和CYT3-4。摇瓶结果如图4所示,由于3个5′-核苷酸酶基因的敲除会导致胞内5′-核苷酸酶活力大幅降低,所以相对CYT2-5,4株菌的胞苷和尿苷产量都显著降低,而且4株菌的胞苷产量在总的嘧啶核苷产量的占比也都呈现了明显的降低趋势,说明菌株自身的4种5′-核苷酸酶都对UMP表现出了更高的亲和力。
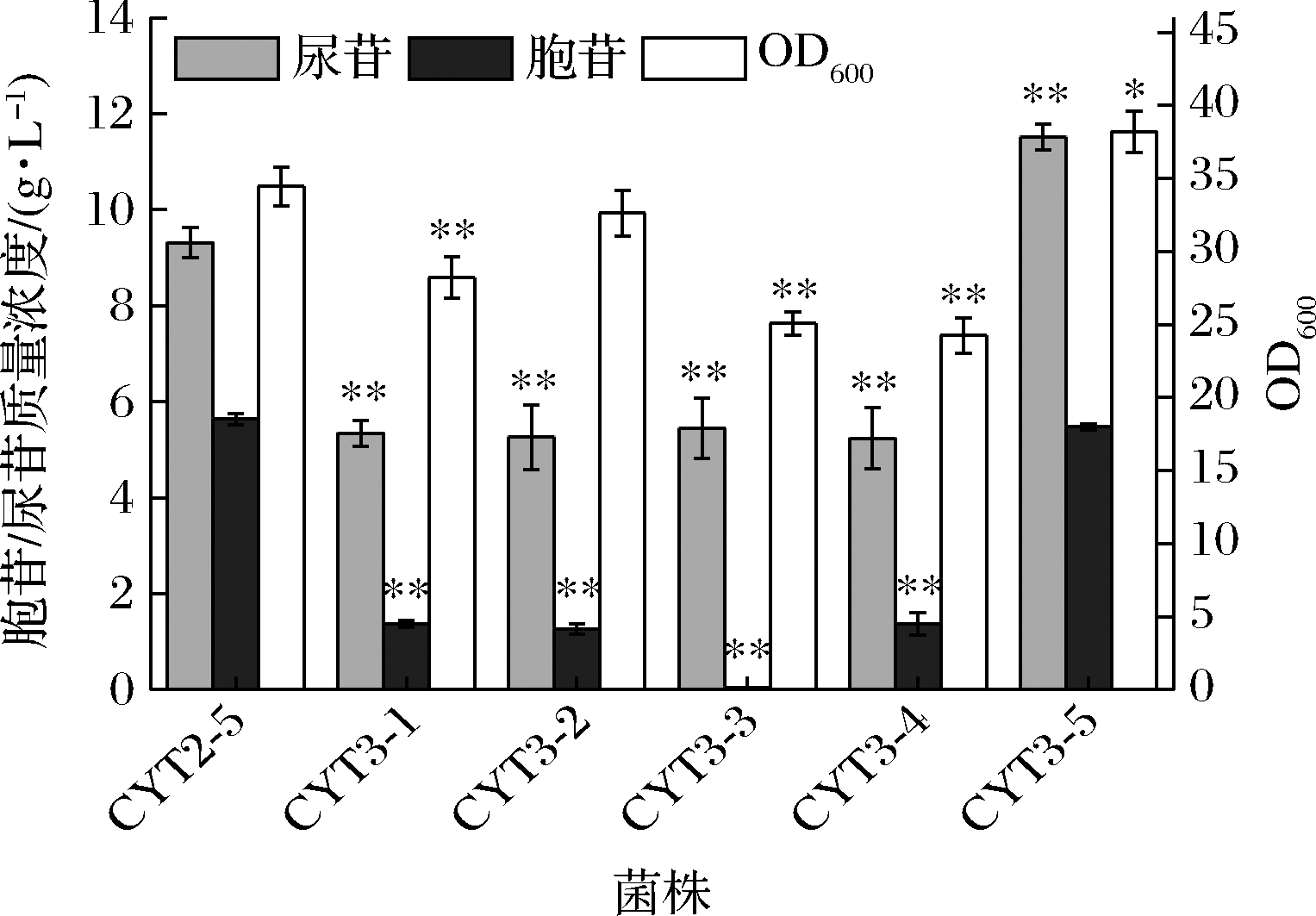
图4 不同5′-核苷酸酶对嘧啶核苷发酵的影响
Fig.4 Effect of different 5′-nucleotidases on pyrimidine nucleoside fermentation
XU等[18]的研究报道指出,来源于酵母菌的5′-核苷酸酶(phm8基因编码)对CMP的亲和力更强,于是本研究在CYT2-5的假基因mbhA位点上整合了酵母菌的phm8基因,构建了菌株CYT3-5,发酵结果显示,CYT3-5的尿苷产量为11.5 g/L,较CYT2-5提高了23.7%,但是胞苷产量变化不大。和XU等[18]的研究报道不同,5′-核苷酸酶PHM8在大肠杆菌中的表达显著提高了尿苷的产量,可能的原因是CYT3-5胞内UMP的浓度远高于CMP的浓度,UMP会竞争性地与PHM8结合,从而有利于尿苷的生成。
2.4 融合蛋白的表达对嘧啶核苷发酵的影响
融合蛋白是将2个甚至多个不同基因相互串联表达或不同蛋白通过特定支架融合的产物[19]。对于催化连续反应的2种或几种酶,融合蛋白可产生“邻近效应”,降低中间产物的扩散,提高整体的催化效率[20-21]。2个功能性蛋白的融合需要连接肽,目前常用的连接肽是由甘氨酸和丝氨酸残基组成的,比如![]() 本研究尝试利用连接肽(Gly4Ser)3将催化UMP从头合成最后一步的乳清酸核苷-5′-磷酸脱羧酶(PyrF)同催化UMP向胞苷合成途径第一步反应的尿苷酸激酶(PyrH)进行融合表达,以促使UMP流向胞苷的合成途径。
本研究尝试利用连接肽(Gly4Ser)3将催化UMP从头合成最后一步的乳清酸核苷-5′-磷酸脱羧酶(PyrF)同催化UMP向胞苷合成途径第一步反应的尿苷酸激酶(PyrH)进行融合表达,以促使UMP流向胞苷的合成途径。
由于本研究所用出发菌株含有2个PyrF编码基因,来源于枯草芽孢杆菌的pyrFbsu基因以及本源的pyrFecj基因,所以分别敲除CYT3-5中的pyrFbsu基因和pyrFecj基因,构建了菌株CYT4-1和CYT4-2,以验证这2个基因对嘧啶核苷合成所起的作用。摇瓶结果如图5所示,3株菌的生长情况基本相同;相比于CYT3-5,CYT4-1的胞苷和尿苷产量变化不大,而CYT4-2的胞苷产量和尿苷产量都有了显著下降,表明CYT3-5自身的PyrF对嘧啶核苷合成起主要的作用。于是本研究选择将PyrFecj同尿苷酸激酶突变体PyrHecj(D93A)进行了融合表达。考虑到融合蛋白中2个功能蛋白在表达过程中的先后顺序对整体催化效率的影响,本研究构建了2个融合蛋白:PyrFecj-(Gly4Ser)3-PyrHecj(D93A)和PyrHecj(D93A)-(Gly4Ser)3-PyrFecj。对应的编码基因分别为pyrFecj-[(GGT)4(TCA)]3-pyrH*和pyrH*-[(GGT))4(TCA)]3-pyrFecj(前一个蛋白编码基因的终止密码子已去除)。将这2个基因分别整合在CYT4-2的假基因yjiT位点,并由启动子Ptrc启动,构建了菌株CYT4-3和CYT4-4。另外在CYT3-5中的相同位点整合了pyrH*基因,构建菌株CYT4-5。将CYT3-5、CYT4-3、CYT4-4和CYT4-5进行摇瓶发酵,结果如图5所示,这几株菌的生长情况基本无差别;CYT4-3的胞苷和尿苷产量分别为5.9和11.9 g/L,和CYT3-5相比,胞苷产量提高了7.2%,尿苷产量降低了6.1%;CYT4-4的胞苷和尿苷产量分别为7.2和8.2 g/L,和CYT3-5相比,胞苷产量提高了30.9%,尿苷产量降低了28.7%;CYT4-5的胞苷和尿苷产量分别为6.2和8.8 g/L,较CYT3-5相比,胞苷产量提高了14.8%,尿苷产量降低了23.5%。
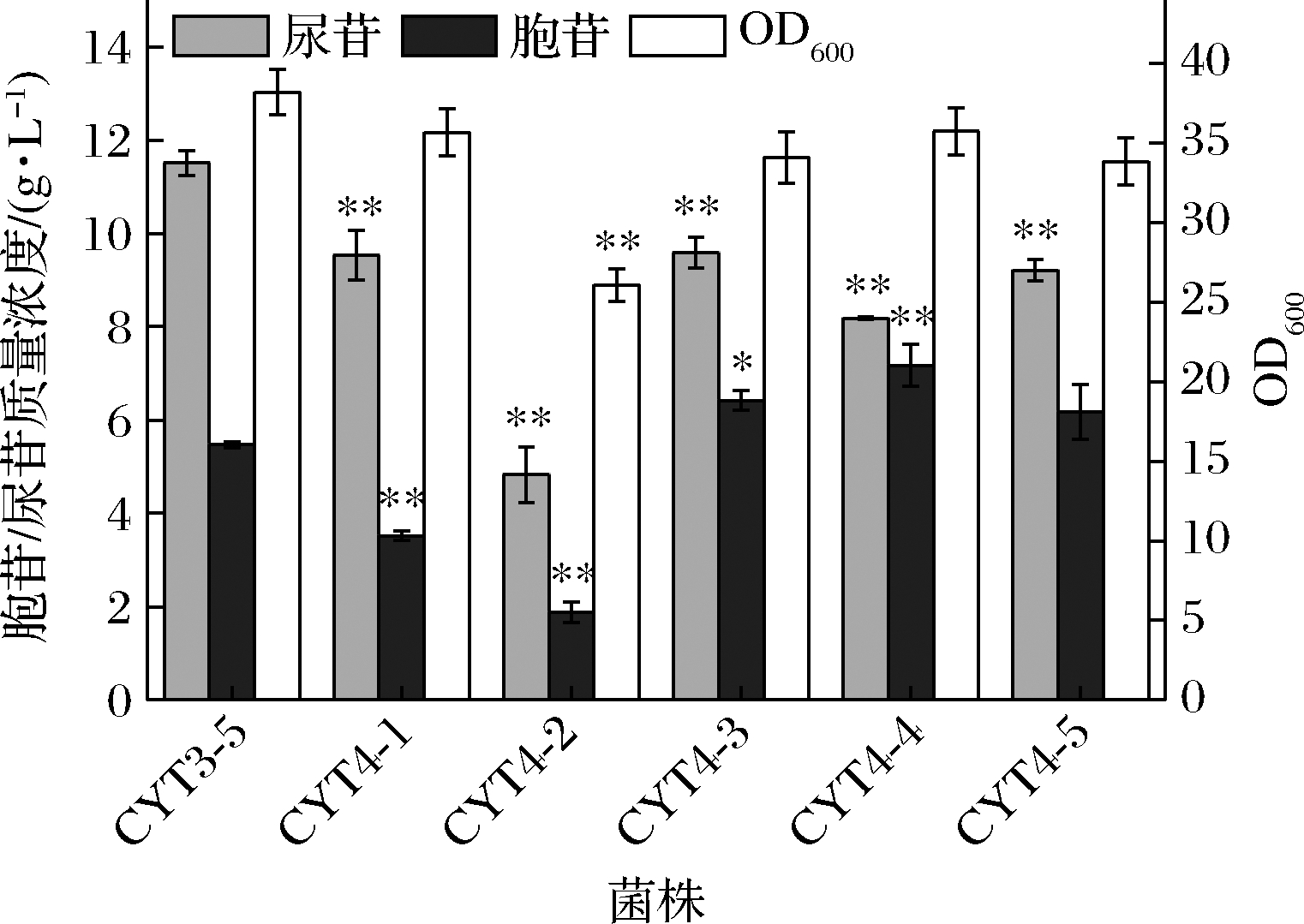
图5 PyrF和PyrH融合表达对嘧啶核苷发酵的影响
Fig.5 Effect of fusion expression of PyrF and PyrH on pyrimidine nucleoside fermentation
结果表明,pyrH*基因表达的强化和融合蛋白的表达,都有利于UMP流向胞苷的合成,从而促进胞苷的积累,降低尿苷的合成。比较而言,融合蛋白的表达对胞苷的合成更有利,且PyrHecj(D93A)-(Gly4Ser)3-PyrFecj的表达效果更好。融合蛋白的表达之所以比pyrH*基因的单独过表达效果更为显著,原因可能在于pyrH*基因的单独过表达只是通过提高胞内PyrHecj(D93A)的酶量,促进UMP流向胞苷的合成,而融合蛋白的表达不仅增加了胞内PyrHecj(D93A)的酶量,还通过融合蛋白所具有的“邻近效应”使PyrHecj(D93A)与其相融合的PyrFecj所催化的产物UMP有更高的碰撞几率,从而更有效地促进了胞苷合成前体物UTP的合成,显著促进了胞苷的积累,相应地,UMP流向尿苷的合成通量显著降低。
3 讨论与结论
UR6是1株经过系统代谢工程改造所得的尿苷高产菌株,其UMP合成途径较强,适合作为构建胞苷高产菌株的出发菌株。为将UR6由只产尿苷的菌株改造为胞苷高产菌,本研究从4个方面入手,对UR6的胞苷合成途径进行了系统改造。
(1)阻断胞苷及其前体物的降解途径。cdd基因的敲除切断了胞苷的主要降解途径,使菌株开始积累胞苷。cmk基因和ygdH基因的敲除切断了胞苷合成途径中的主要支路途径,减少了胞苷合成前体物的额外消耗,也促进了胞苷的积累。(2)组合强化胞苷合成关键酶的表达。本研究引入了调控作用减弱的UMP激酶和CTP合酶突变体,并对本源的CTP二磷酸酶的编码基因进行了双拷贝,通过关键酶的组合表达有效地加强了胞苷的合成途径,使得胞苷的产量大幅提升。(3)比较不同5′-核苷酸酶对嘧啶核苷发酵的影响。尿苷和胞苷合成途径的竞争主要在于5′-核苷酸酶所催化的反应。本研究构建了只含其中1种5′-核苷酸酶的菌株,考察了大肠杆菌自身的4种核苷酸酶分别在嘧啶核苷发酵中所起的作用。结果证实这4种核苷酸酶的专一性较差,并且相对于CMP,4种核苷酸酶都更倾向于和UMP结合,从而催化其合成尿苷。PHM8是来源于酵母菌的5′-核苷酸酶,先前有报道称其对CMP有更高的亲和力。但是和报道的结果不同,该酶在大肠杆菌的表达,显著促进了尿苷的积累,而对胞苷积累的影响不大。说明在UMP相对过量的条件下,PHM8还是优先结合UMP,催化其生成尿苷。(4)通过蛋白的融合表达增强胞苷合成。本研究将乳清酸核苷-5-磷酸脱羧酶同尿苷酸激酶进行融合表达,利用融合蛋白的“邻近效应”,促进前体物UMP流向胞苷的合成,使胞苷的产量得到了显著提升。
最终所得菌株CYT4-4摇瓶发酵24 h可积累7.2 g/L胞苷和8.2 g/L尿苷,说明本研究通过对胞苷合成途径的一系列改造,成功改变了UMP向胞苷和尿苷合成的流量分配,使约46.7%的UMP流向了胞苷的合成。多种可分离产物的联产是提高原料利用率、降低发酵生产成本的有效方法[24]。胞苷和尿苷可以通过色谱分离等技术进行分离纯化,因此该菌可用来联产尿苷和胞苷,具有一定的工业化应用价值。为进一步提升胞苷产量,最终获得只高产胞苷的工程菌,需要继续强化菌株的胞苷合成途径,如筛选对CMP有更高亲和力甚至专一亲和CMP的5′-核苷酸酶和完全解调控的UMP激酶和CTP合酶等。
[1] KUMAR S, DEEP A, NARASIMHAN B.A review on synthesis, anticancer and antiviral potentials of pyrimidine derivatives[J].Current Bioactive Compounds, 2019, 15(3):289-303.
[2] 吴鹤云, 张悦, 蒋帅, 等.木糖和葡萄糖共发酵生产L-组氨酸[J].食品与发酵工业, 2020, 46(16):115-120.
WU H Y, ZHANG Y, JIANG S, et al.Co-fermentation of xylose and glucose for L-histidine production[J].Food and Fermentation Industries, 2020, 46(16):115-120.
[3] ZHU H, YANG S M, YUAN Z M,et al.Metabolic and genetic factors affecting the productivity of pyrimidine nucleoside in Bacillus subtilis[J].Microbial Cell Factories, 2015, 14(1):1-12.
[4] WU H Y, LI Y J, MA Q, et al.Metabolic engineering of Escherichia coli for high-yield uridine production[J].Metabolic Engineering, 2018, 49:248-256.
[5] 范晓光, 李燕军.基于分子生物学技术的胞苷生产菌的育种研究进展[J].发酵科技通讯, 2015, 44(1):53-57.
FAN X G, LI Y J.Research advance in cytidine production strain by molecular biology technology[J].Bulletin of Fermentation Science and Technology, 2015, 44(1):53-57.
[6] ASAHI S, TSUNEMI Y, IZAWA M, et al.Cytidine production by mutants of Bacillus subtilis[J].Bioscience, Biotechnology, and Biochemistry, 1994, 58(8):1 399-1 402.
[7] DOI M, TSUNEMI Y, ASAHI S.Opimization of conditions for production of uridine by a mutant of Bacillus subtilis[J].Bioscience, biotechnology, and biochemistry, 1994, 58(9):1 608-1 612.
[8] KO Y S, KIM J W, LEE J A, et al.Tools and strategies of systems metabolic engineering for the development of microbial cell factories for chemical production[J].Chemical Society Reviews, 2020, 49(14):4 615-4 636.
[9] YANG D, PARK S Y, PARK Y S, et al.Metabolic engineering of Escherichia coli for natural product biosynthesis[J].Trends in Biotechnology, 2020, 38(7):745-765.
[10] FAN X G, WU H Y, LI G L, et al.Improvement of uridine production of Bacillus subtilis by atmospheric and room temperature plasma mutagenesis and high-throughput screening[J].PloS One, 2017, 12(5):e0 176 545.
[11] YANG K, LI Z M.Multistep construction of metabolically engineered Escherichia coli for enhanced cytidine biosynthesis[J].Biochemical Engineering Journal, 2020, 154:107 433.
[12] LI Y F, LIN Z Q, HUANG C, et al.Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J].Metabolic Engineering, 2015, 31:13-21.
[13] DONG H N, LIU Y F, ZU X, et al.An enzymatic assay for high-throughput screening of cytidine-producing microbial strains[J].PloS One, 2015, 10(3):e0 121 612.
[14] SÉVIN D C, FUHRER T, ZAMBONI N, et al.Nontargeted in vitro metabolomics for high-throughput identification of novel enzymes in Escherichia coli[J].Nature methods, 2017, 14(2):187-194.
[15] 姜巨全, 杨立娜, 刘艳双, 等.肇东盐单胞菌中胞苷酸激酶基因cmk的克隆与功能分析[J].东北农业大学学报, 2017, 48(7):33-40.
JIANG J Q, YANG L N, LIU Y S, et al.Cloning and functional analysis of cmk encoding cytidylate kinase in Halomonas zhaodongensis[J].Journal of Northeast Agricultural University, 2017, 48(7):33-40.
[16] MEYER P, EVRIN C, BRIOZZO P, et al.Structural and functional characterization of Escherichia coli UMP kinase in complex with its allosteric regulator GTP[J].Journal of Biological Chemistry, 2008, 283(51):36 011-36 018.
[17] ZHU M Z, SUN W J, WANG Y, et al.Engineered cytidine triphosphate synthetase with reduced product inhibition[J].Protein Engineering Design & Selection, 2014, 27(7):225-233.
[18] XU Y F, LÉTISSE F, ABSALAN F, et al.Nucleotide degradation and ribose salvage in yeast[J].Molecular Systems Biology, 2013, 9(1):665.
[19] KANG W, MA T, LIU M, et al.Modular enzyme assembly for enhanced cascade biocatalysis and metabolic flux[J].Nature Communications, 2019, 10(1).DOI:10.1038/S416467-019-12247-W.
[20] MYHRVOLD C, POLKA J K, SILVER P A.Synthetic lipid-containing scaffolds enhance production by colocalizing enzymes[J].ACS Synthetic Biology, 2016, 5(12):1 396-1 403.
[21] LIN J L, ZHU J, WHEELDON I.Synthetic protein scaffolds for biosynthetic pathway colocalization on lipid droplet membranes[J].ACS Synthetic Biology, 2017, 6(8):1 534-1 544.
[22] 于健, SARRA S, 徐寒梅.连接肽在融合蛋白设计中的选择及应用[J].药物生物技术, 2016, 23(3):260-263.
YU J, SARRA S,XU H M.Selection and application of linker peptide in the design of fusion protein[J].Pharmaceutical Biotechnology, 2016, 23(3):260-263.
[23] KHADEMI F, YOUSEFI-AVARVAND A, DERAKHSHAN M, et al.Mycobacterium tuberculosis HspX/EsxS fusion protein:Gene cloning, protein expression, and purification in Escherichia coli[J].Reports of Biochemistry & Molecular Biology, 2017, 6(1):15-21.
[24] FAN X G, WU H Y, JIA Z F, et al.Metabolic engineering of Bacillus subtilis for the co-production of uridine and acetoin[J].Applied Microbiology and Biotechnology.2018, 102(20):8 753-8 762.