脂肪酶(EC.3.1.1.3)是自然界中最有潜力的工业酶之一,属于α/β水解超家族,具有多种催化能力,催化中心由亲核基团丝氨酸、2个残基天冬氨酸和组氨酸形成的催化三联体和阴离子氧洞组成[1],在脂水界面存在的情况下,活性位点被激活,产生构象变化,使底物进入活性位点[2],不仅能催化甘油三酯水解反应,而且可以催化酯合成、酯交换、醇解、酸解等反应[3],在生物清洗剂、手性药物拆分、生物传感器和生物柴油合成等方面引起了人们的广泛关注[4-5]。
酶在工业过程中的应用要求具有良好的稳定性[6],嗜热脂肪酶通常在较高温度下仍能比较稳定,并且大多是从嗜热细菌中分离出来的,具有催化效率高、冷却能耗低等优点,可以完成许多高温反应过程[7],然而天然酶生长缓慢、生长条件苛刻,限制了其产量,直接利用脂肪酶往往不实际,科研工作者利用基因工程、蛋白质工程等方法异源表达重组酶已成为研究热点,但现有报道中的嗜热脂肪酶耐受温度不是太高[8],无法在工业上大范围应用,因而需要寻找一种更稳定的嗜热脂肪酶。本实验室早期的研究集中在嗜热玫瑰红球菌Thermomicrobium roseum DSM 5159,这是1株从美国黄石温泉筛选出的嗜热古细菌,适宜生长温度为70~75 ℃[9],根据其全基因组注释,预测该古细菌产生一种322个氨基酸组成的脂肪酶,将这段氨基酸序列应用到网站(https://swissmodel.expasy.org)同源建模发现,T.roseum DSM 5159脂肪酶与海洛因酯酶(PDB ID:1lzk.1)相似度为45.19%,因而可能被认为是生产耐热脂肪酶的良好候选者。
高效异源表达嗜热脂肪酶,提高生产效率使其应用到工业上具有重要意义。本研究克隆了一个预测的T.roseum DSM 5159 liP基因到pTIG质粒中,转化至大肠杆菌BL21(DE3)中表达,经镍柱亲和层析纯化得到纯酶,基质辅助激光解析电离串联飞行时间质谱(matrix-assisted laser desorption ionization time of flight,MALDI-TOF)进一步鉴定表达结果,并考察了热稳定性、酸碱性、金属离子、有机溶剂和表面活性剂对重组酶的影响,较全面系统地表征该酶的酶学性质。
1 材料与方法
1.1 质粒和菌株
Escherichia coli JM109、Escherichia coli BL21(DE3)、质粒pTIG均为本实验室保存。
1.2 试剂、培养基和仪器
LB培养基(g/L):酵母粉5,胰蛋白胨10,NaCl 10,115 ℃灭菌20 min。
主要试剂:PrimerSTAR Max DNA聚合酶、同源重组酶、限制性内切酶Hind Ⅲ、EcoR Ⅰ,TaKaRa公司;对硝基苯酚棕榈酸酯等系列底物,SIGMA公司;其他试剂均为国产分析纯。
仪器:S100D型PCR仪,美国BIO-RAD公司;V-1200分光光度计,上海美普达仪器公司;SCG蛋白纯化系统,苏州赛谱仪器有限公司;MOS-450圆二色光谱仪,法国Bio-Logic公司;NANO DSC差示扫描微量热仪,美国Waters公司。
1.3 实验方法
1.3.1 重组菌的构建及诱导表达
从NCBI下载预测脂肪酶氨基酸序列(ACM04789.1),送生工生物工程(上海)股份有限公司合成基因,在引物上设计载体pTIG同源臂,以上、下游引物GGATCCGGTAAGGAGGAATTCTAAATGTCTGTTTTCGCTCGTCTTG和CTCGAGTGCGGCCGCAAGCTTAAGAGCACGTTGAAGAACA进行PCR扩增liP片段,对获得的片段采用胶回收后,用同源重组酶将其与载体pTIG连接,得到的连接产物pTIG-liP转化至E.coli JM109感受态中,提取质粒双酶切和测序验证正确后,转化至E.coli BL21(DE3)表达。将重组菌pTIG-liP/BL21(DE3)、对照菌pTIG/BL21(DE3)分别划线于氨苄抗性平板上37 ℃倒置培养12 h,挑取单菌落接种于15 mL LB液体培养基中,在37 ℃,200 r/min的条件下培养12 h,以2%体积分数的转接量转接至50 mL LB培养基中,培养条件不变,继续培养至OD600=0.6~0.8,添加终浓度为0.1 mmol/L的异丙基硫代半乳糖苷(IPTG)诱导表达,在20 ℃,200 r/min条件下,诱导发酵24 h,离心弃上清液收集菌体,用20 mmol/L Tris-HCl缓冲液(pH 8.0)洗涤3次,破碎细胞收集上清液即为粗酶液。
1.3.2 重组脂肪酶纯化及SDS-PAGE分析
将粗酶液用1 mL镍柱纯化,收集纯化的酶液(Lip),透析去除高浓度咪唑,蛋白浓度的测定采用考马斯亮蓝法,用SDS-PAGE分析检测。
1.3.3 MALDI-TOF分析
回收10% SDS-PAGE凝胶重组脂肪酶条带于1.5 mL EP管中,然后对凝胶进行脱色、溶化、消化等预处理[10],用MALDI-TOF仪器分析。
1.3.4 圆二色谱(circular dichroism,CD)分析
通过CD分析估计了各种二级结构的占比,将纯酶液(0.1 mg/mL)置于1 mm的比色皿中,在190~250 nm波长处扫描[11],3次测量的数据取平均值。
1.3.5 差示扫描量热法(differential scanning calorimetry,DSC)分析
通过差示扫描量热仪测定Lip的变性温度(Tm),在测定样品前,先用透析液重复测定3次,以平衡基线。取500 μL Lip放入样品池中扫描,设置温度为70~130 ℃,升温速率为2 ℃/min[10],实验结果用NanoAnalyze软件进行分析。
1.3.6 脂肪酶活力测定
参考SHA等[12]的方法略有改动,底物1∶15 mmol/L对硝基苯酚棕榈酸酯溶于二甲基亚砜中;底物2∶50 mmol/L Tris-HCl、0.55 g/L阿拉伯胶、1.2 g/L脱氧胆酸钠,pH 8.0;按体积比1∶100混合,现配现用,反应前将混合物置于45 ℃孵育5 min。反应体系:取上述混合物2.4 mL,加入0.1 mL适当稀释的酶液,于70 ℃准确反应5 min,加入0.1 mL浓度为1 mol/L Na2CO3溶液终止反应,空白对照为不加酶液的缓冲液,于410 nm波长处测定吸光值。酶活力定义:每分钟产生1 μmol对硝基苯酚所需要的酶量为1个酶活单位(U)。酶活力计算如公式(1)所示:
![]()
(1)
式中:V1,反应总体积,mL;ε,摩尔消光系数,mL/mmol;t,反应时间,min。
以上反应均设置3组平行实验,没有特殊说明酶活力检测方法均按此实验。
1.3.7 重组脂肪酶酶学性质测定
1.3.7.1 最适反应pH及pH稳定性测定
按1.3.6的方法,在pH为5.5~11.0的缓冲液中测定酶活力,将实验中所测得的最高酶活力记作100%,并计算其他pH值条件下的相对酶活力,确定最适pH值。pH稳定性测定:将适量稀释的Lip置于pH为5.5~11.0的不同缓冲液中,于4 ℃下,2 h后测定残留酶活力。各pH缓冲体系分别为:Na2HPO4-NaH2PO4(pH=5.5~7.5)、Tris-HCl(pH=7.5~9.0)、Gly-NaOH(pH=9.0~11.0)。
1.3.7.2 最适反应温度及温度稳定性测定
将Lip在40~100 ℃范围内测定酶活力,以最高酶活力作为100%,计算其他温度的相对酶活力,确定最适反应温度。温度稳定性测定:在0~90 ℃的梯度温度范围内孵育12 h后,以未处理酶的酶活力作为100%,测定残留脂肪酶相对酶活力。
1.3.7.3 有机溶剂对重组脂肪酶的影响
将Lip在不同的有机溶剂V(Lip)∶V(有机溶剂)=1∶1中孵育,以不加有机溶剂的酶活力作为100%,分别于1、12、24 h取样测定酶活力。有机溶剂包括二甲基甲酰胺、二甲基亚砜、甲醇、乙醇、乙腈、甘油、正己烷、环己烷、异丙醇、甲苯、正丁醇、乙酸乙酯、氯仿和四氢呋喃。
1.3.7.4 金属离子、表面活性剂和抑制剂对重组脂肪酶的影响
向反应体系中分别加入不同金属离子(Al3+、K+、Ca2+、Zn2+、Co2+、Mn2+、Mg2+、Fe3+、Cu2+和Ni2+)至终浓度为0.1、1.0、3.0 mmol/L,表面活性剂[十二烷基硫酸钠(SDS),曲拉通100,吐温40,吐温80]和抑制剂[乙二胺四乙酸(EDTA),苯甲基磺酰氟(PMSF),L-半胱氨酸和二硫苏糖醇(DTT)]至终浓度为0.1、0.5、1.0 mmol/L,以未处理酶的酶活力为对照,测定脂肪酶相对酶活力。
1.3.7.5 底物特异性及动力学研究
选择不同浓度的底物(对硝基苯酚乙酸酯、对硝基苯酚丁酸酯、对硝基苯酚辛酸酯、对硝基苯酚月桂酸酯和对硝基苯酚棕榈酸酯),在70 ℃下与Lip反应,测定初始反应速度,利用Origin软件中的Michaelis-Menten方程进行非线性曲线拟合,得到米氏常数Km和最大反应速率Vm值,并计算kcat和kcat/Km值,确定脂肪酶对不同碳链长度底物的特异性。kcat计算如公式(2)所示:
(2)
![]()
(3)
式中:kcat,催化常数,s-1;Vm,最大反应速率,μmol/min;[E],蛋白物质的量,μmol;c,蛋白浓度,mg/mL;V,酶液体积,μL;M,蛋白分子质量,Da。
2 结果与分析
2.1 重组脂肪酶的构建、表达和纯化
如图1-a,1-b所示,设计分子伴侣硫氧还蛋白TrxA和脂肪酶共表达,成功构建了重组质粒。SDS-PAGE(图1-c)分析表明重组蛋白表达量高,纯化条带单一,分子质量大小约为38 kDa,与氨基酸序列计算的理论分子质量36.7 kDa基本一致,首次实现了该脂肪酶可溶性表达。测定粗酶和纯酶酶活力发现,纯酶的比酶活为3.23 μmol/(min·mg),较之粗酶的0.38 μmol/(min·mg)提高了8.5倍。
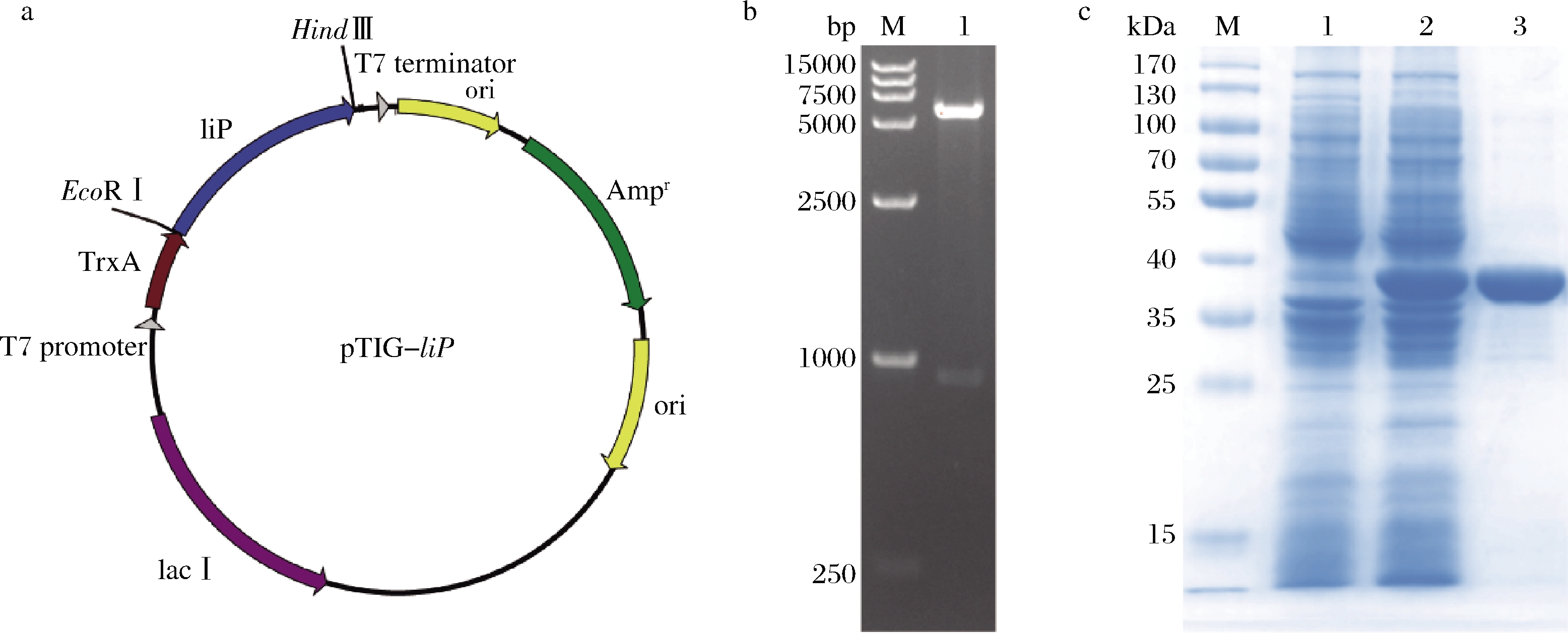
a-pTIG-liP质粒图;b-重组质粒双酶切鉴定;c-SDS-PAGE分析在b图中,M-Marker;1-5 727和966 bp;在c图中,M-Marker;1-空载;2-重组脂肪酶粗酶液;3-纯酶
图1 重组脂肪酶的构建及诱导表达
Fig.1 Construction and induced expression of the recombinant lipase
2.2 重组脂肪酶Tm和二级结构分析
如图2所示,5个肽段匹配得分有4个可信度单独超过阈值分数,蛋白得分357分,认为质谱结果基本可信,约38 kDa大小的条带确实是脂肪酶(gi|506402911)。CD检测结果如图3所示,并进一步将数据在网站(http://dichroweb.cryst.bbk.ac.uk/html/home.shtml)分析,结果表明重组脂肪酶由56.3% α-螺旋、4.3% β-折叠、19.9% β-转角和20.3%无规则卷曲组成,α-螺旋结构在增强稳定性方面发挥了重要作用,这与CAO等[13]报道的脂肪酶一致。在Nano-DSC分析中,纯酶的Tm和ΔH分别为97.53 ℃和1 637 kJ/mol(图4),Tm值是蛋白质的解链温度即变性温度,数值越大越稳定,且远高于表1已报道的耐热脂肪酶,表明该酶具有非常优异的热稳定性。
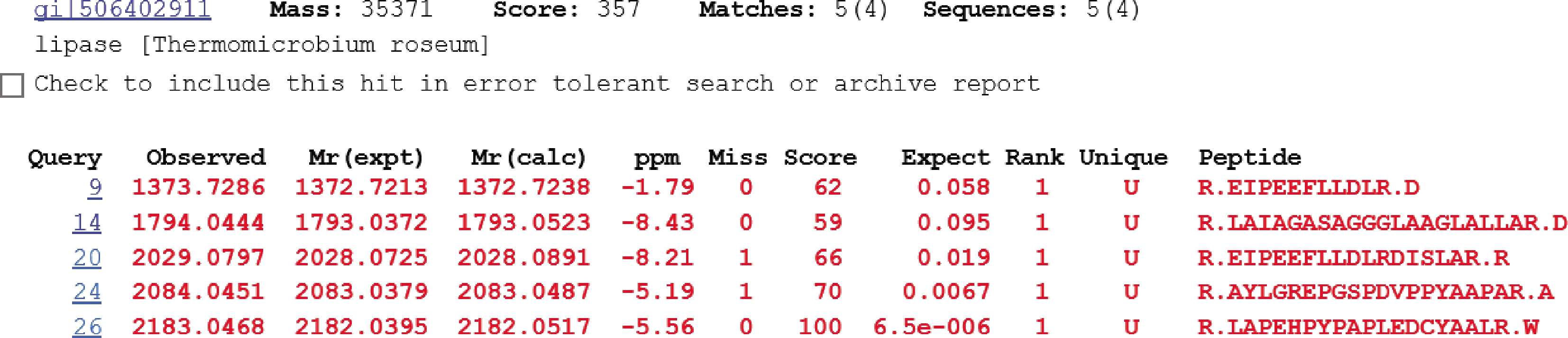
图2 重组脂肪酶的MALDI-TOF分析结果
Fig.2 MALDI-TOF analysis of the recombinant lipase
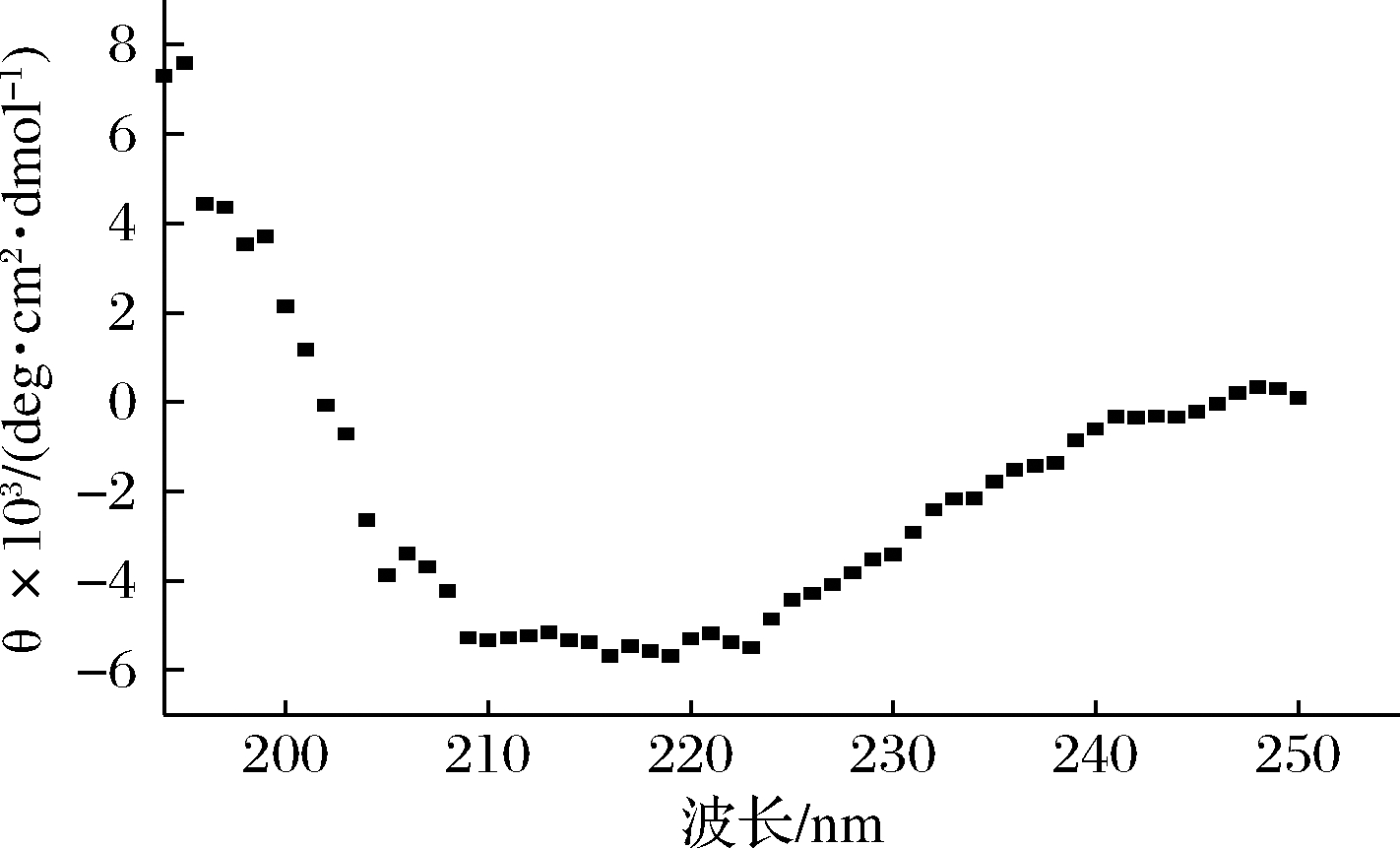
图3 重组脂肪酶的二级结构分析
Fig.3 Secondary structure analysis of the recombinant lipase
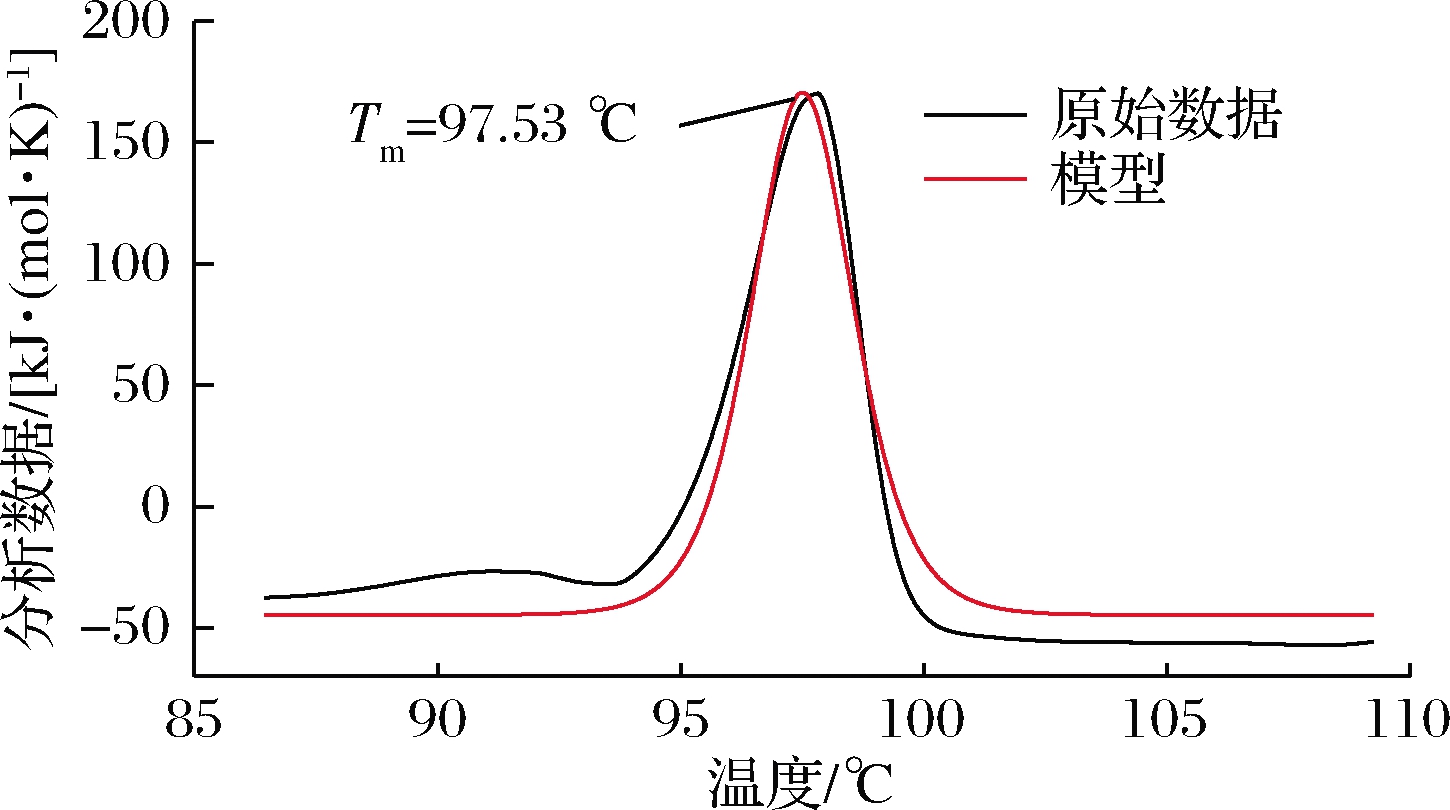
图4 重组脂肪酶熔融温度(Tm)分析
Fig.4 Melting temperature(Tm)analysis of the recombinant lipase
表1 不同来源脂肪酶的热稳定性
Table 1 Thermal stability of lipases from different sources
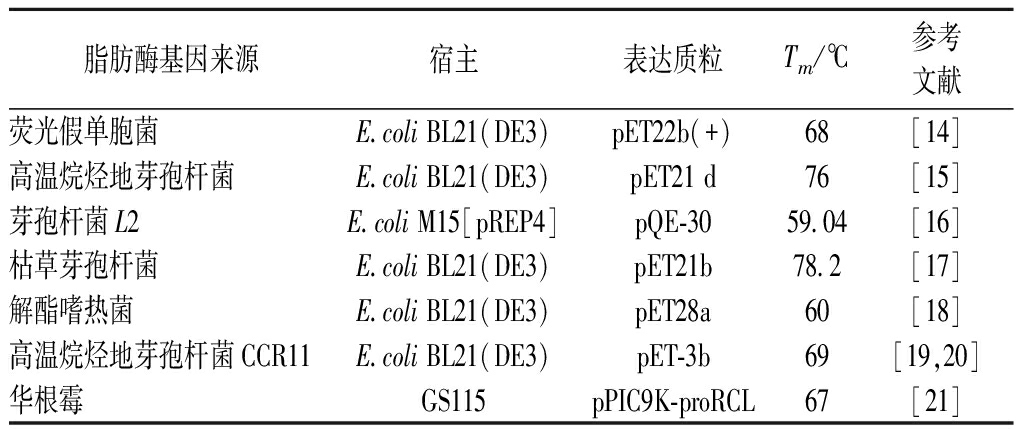
脂肪酶基因来源宿主表达质粒Tm/℃参考文献荧光假单胞菌E.coliBL21(DE3)pET22b(+)68[14]高温烷烃地芽孢杆菌E.coliBL21(DE3)pET21d76[15]芽孢杆菌L2E.coliM15[pREP4]pQE-3059.04[16]枯草芽孢杆菌E.coliBL21(DE3)pET21b78.2[17]解酯嗜热菌E.coliBL21(DE3)pET28a60[18]高温烷烃地芽孢杆菌CCR11E.coliBL21(DE3)pET-3b69[19,20]华根霉GS115pPIC9K-proRCL67[21]
2.3 重组脂肪酶酶学性质分析
2.3.1 温度及pH对重组脂肪酶的影响
如图5-a,5-b所示,最适反应温度为85 ℃,0~90 ℃孵育12 h后,0~80 ℃的酶活力保留率在80%以上,而由于酶的构象在温度过高时被破坏[2],90 ℃时仅有40%。酶的最适反应pH为8.5(图5-c),属于碱性脂肪酶。在不同pH于4 ℃孵育2 h,酶活力残留率均在80%以上,pH 8.5~9.5酶活力仍有90%左右,表现出较好的耐碱性(图5-d)。超嗜热耐碱脂肪酶鲜有报道,因此该脂肪酶有望在工业上发挥重要作用。
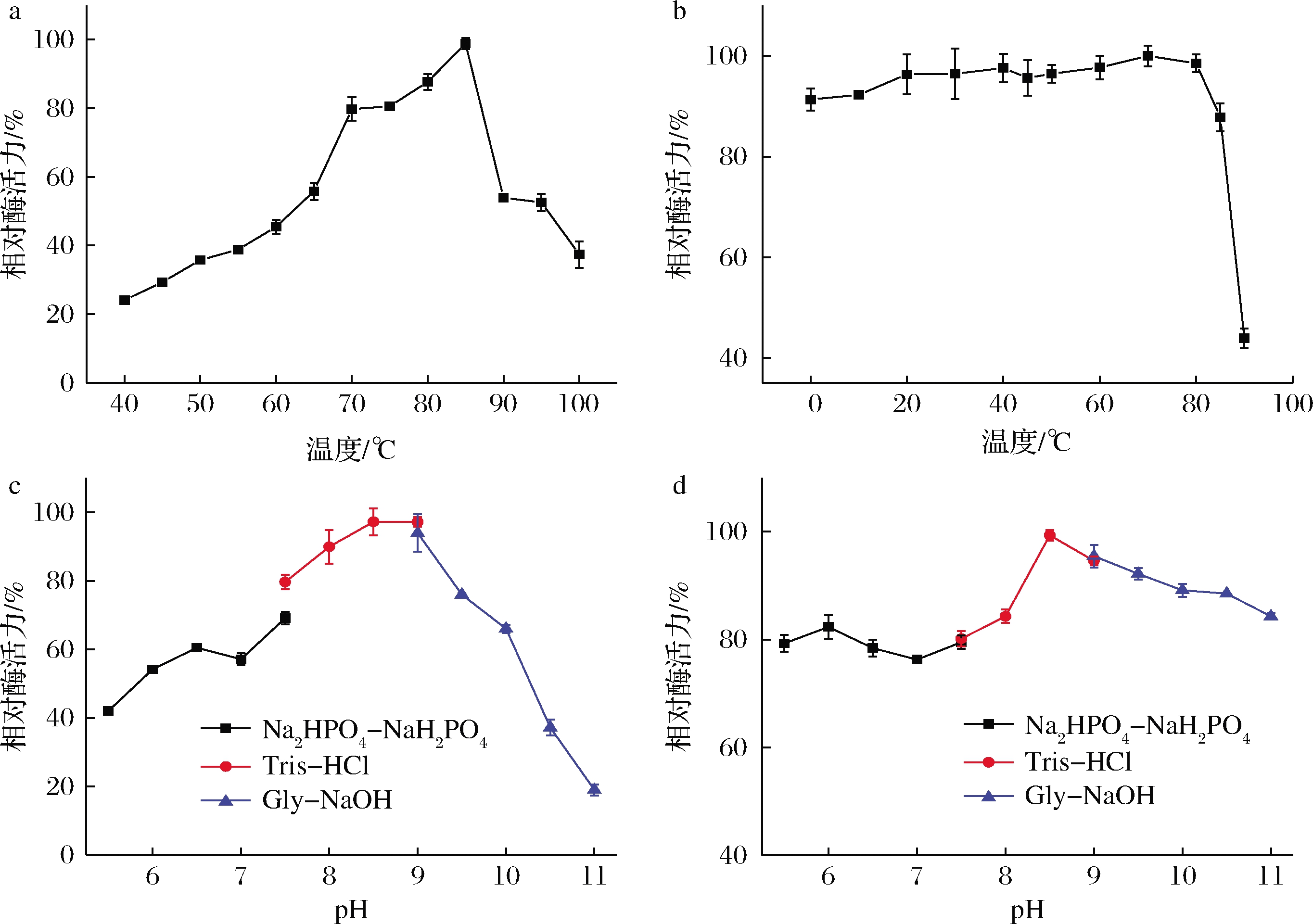
a-最适反应温度;b-温度稳定性;c-最适反应pH;d-pH稳定性
图5 温度和pH对重组脂肪酶的影响
Fig.5 Effect of temperature and pH on the recombinant lipase
2.3.2 有机溶剂对重组脂肪酶的影响
如图6所示,除氯仿和四氢呋喃对酶活力有强烈抑制外,其他有机溶剂均只有轻微波动,说明重组脂肪酶在大多数有机溶剂中相对稳定。随着孵育时间的延长,酶活力保留率略有下降,脂肪酶在1 h时对log P值小的有机溶剂耐受能力较强,如:二甲基甲酰胺(121.10%)、二甲基亚砜(110.23%)、甲醇(105.83%)、乙醇(96.77%)、乙腈(105.03%)。可能原因是log P值小的有机溶剂更亲水,会降低环境的极性,使酶分子能够正确折叠[22]。此外,乙酸乙酯显著抑制酶活力(18.81%),猜测可能是乙酸乙酯与底物竞争性结合脂肪酶,后续以乙酸乙酯为底物,采用氢氧化钠滴定法[23]进行验证实验,脂肪酶对乙酸乙酯表现出较强的水解作用。综上所述,Lip对有机溶剂具有较强的耐受能力,为其在有机相中实现酯化和酯交换反应的广泛应用奠定了基础。
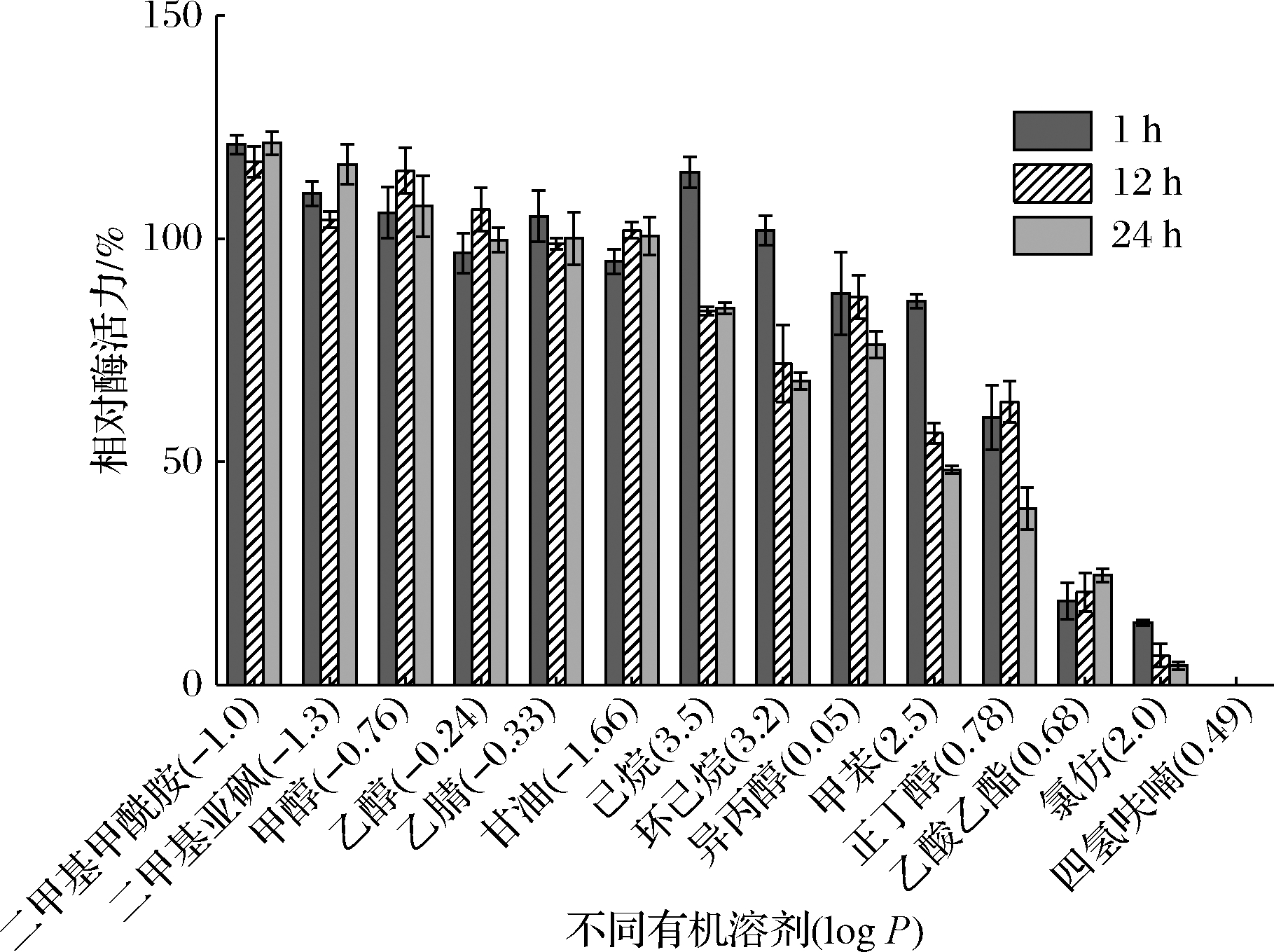
图6 有机溶剂对重组脂肪酶酶活力的影响
Fig.6 Effect of organic solvents on the enzyme activity of the recombinant lipase
2.3.3 金属离子、表面活性剂及抑制剂对重组脂肪酶的影响
如图7-a所示,随着离子浓度的增加,K+和Ca2+对Lip有激活作用,Mg2+、Mn2+和Ba2+的加入对Lip几乎没有影响。高浓度的Cu2+和Fe3+对Lip有明显的抑制作用,Co2+、Zn2+和Ni2+的存在使Lip的相对活性降低到50%。图7-b展示了表面活性剂和抑制剂对Lip的影响,阴离子表面活性剂SDS(2.67%)对Lip活性有明显的抑制作用,SDS破坏了蛋白质分子内和分子间的氢键,使酶失活,同样的现象也发生在热链状地杆菌的嗜热脂肪酶中[24]。曲拉通 100(91.77%)对脂肪酶的影响较小,而高浓度的非离子表面活性剂吐温 80和吐温 40抑制了脂肪酶的活性,1.0 mmol/L 吐温 80甚至使酶活力降至39.77%,EDTA、L-半胱氨酸和DTT等抑制剂对其活性影响不大,均在85%以上,1.0 mmol/L PMSF对Lip活性有轻微抑制作用,说明活性位点上存在丝氨酸残基[22]。所以,该酶不含二硫键,也不是金属依赖型酶,在反应体系中加入适当浓度的金属离子和表面活性剂可以调节Lip的水解活性。

a-金属离子;b-抑制剂
图7 金属离子、表面活性剂和抑制剂对重组脂肪酶酶活力的影响
Fig.7 Effect of metal ions,surfactants and inhibitors on the enzyme activity of the recombinant lipase
2.4 重组脂肪酶的动力学研究
Km表示酶和底物之间的亲和能力,Km值越小,亲和能力越强,以对硝基苯棕榈酸酯为底物时,该酶的Km=0.01 mmol/L;以对硝基苯基月桂酸酯为底物时,该酶的Km=0.047 mmol/L;以对硝基苯基丁酸酯为底物时,该酶的Km=9.67 mmol/L,说明Lip对中长链底物具有较好的亲和力。以对硝基苯酚棕榈酸酯为底物时,Lip的Km值低于文献中已报道的黏质沙利菌(0.39 mmol/L)[25]、类芽孢杆菌(0.34 mmol/L)[22]和真菌蛹虫草(0.162 mmol/L)的脂肪酶[26],表现出更强的亲和力。kcat/Km被认为能最全面的衡量酶催化能力的指标,数值越大催化效率越高,以对硝基苯棕榈酸酯为底物时,该酶的kcat/Km为299.0 L/(s·mmol),比其他底物都大,也比NORO等[27]报道的疏绵状嗜热丝孢菌重组脂肪酶高[约2 000 L/(s·mol)]。这一动力学研究证实,Lip不仅底物范围广,而且催化效率高。
表2 重组脂肪酶动力学参数
Table 2 Recombinant lipase kinetic parameters
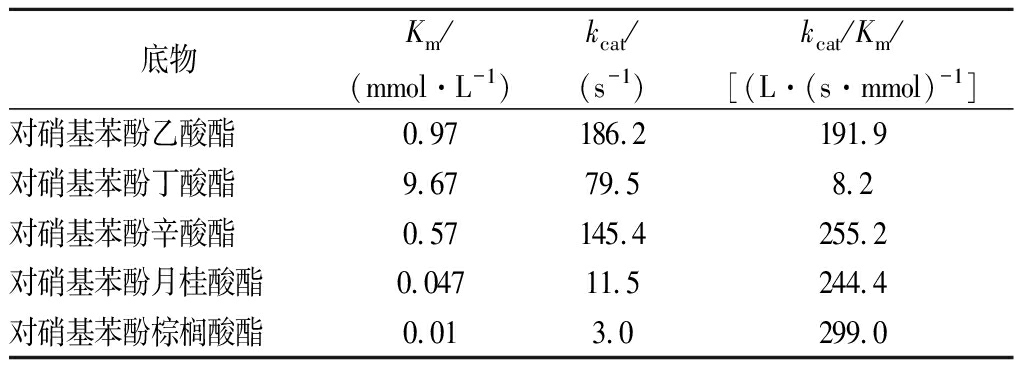
底物Km/(mmol·L-1)kcat/(s-1)kcat/Km/[(L·(s·mmol)-1]对硝基苯酚乙酸酯0.97186.2191.9对硝基苯酚丁酸酯9.6779.58.2对硝基苯酚辛酸酯0.57145.4255.2对硝基苯酚月桂酸酯0.04711.5244.4对硝基苯酚棕榈酸酯0.013.0299.0
3 结论与讨论
本研究克隆并表达了T.roseum DSM 5159的预测脂肪酶基因,对纯酶进行了生物化学表征,有助于更好地了解嗜热细菌脂肪酶的基因和酶学性质。获得的重组脂肪酶具有较高的产量和催化活力,最适反应温度85 ℃,Tm为97.53 ℃,比其他大部分嗜热脂肪酶都高,在80 ℃孵育12 h后仍有80%以上的酶活保留率,最适反应pH为8.5,在8.5~9.5范围内孵育2 h酶活力在90%以上,具有非常优越的稳定性。该酶对一系列有机溶剂耐受力较强,金属离子Ca2+和K+对纯化的脂肪酶有激活作用,表面活性剂SDS对该酶有强烈的抑制效果,而抑制剂对酶活力没有明显影响。脂肪酶对不同底物的动力学参数表明,最适底物是对硝基苯酚棕榈酸酯,Km为0.01 mmol/L,kcat/Km为299.0 L/(s·mmol),底物范围广且对中长链底物亲和力更高,这与文献中脂肪酶性质一致[1],但相较于商业脂肪酶,酶活力仍有待提高,后续研究将通过同源建模,针对催化中心周围氨基酸理性改造,期望获得高催化效率的耐高温脂肪酶,使其能够在非水相催化、洗涤和高温环境工业等领域得到广泛应用。
[1] CASAS-GODOY L,GASTEAZORO F,DUQUESNE S,et al.Lipases:An overview[J].Methods Molecular Biology,2018,1 835(1):3-38.
[2] VAREJAO N,DE-ANDRADE R A,ALMEIDA R V,et al.Structural mechanism for the temperature-dependent activation of the hyperthermophilic Pf2001 esterase[J].Structure,2018,26(2):199-213.
[3] PALOMO J M.Synthetic complexity created by lipases[J].Nature Catalysis,2020,3(4):335-336.
[4] ZHANG H X,LIU T S,ZHU Y W,et al.Lipases immobilized on the modified polyporous magnetic cellulose support as an efficient and recyclable catalyst for biodiesel production from yellow horn seed oil[J].Renewable Energy,2020,145(1):1 246-1 254.
[5] PHUKON L C,CHOURASIA R,KUMARI M,et al.Production and characterisation of lipase for application in detergent industry from a novel Pseudomonas helmanticensis HS6[J].Bioresource Technology,2020,309(2):1-32.
[6] SAMOYLOVA Y V,SOROKINA K N,ROMANENKO M V,et al.Cloning,expression and characterization of the esterase estUT1 from Ureibacillus thermosphaericus which belongs to a new lipase family XVIII[J].Extremophiles,2018,22(2):271-285.
[7] MAHESHWARI R,BHARADWAJ G,BHAT M K.Thermophilic fungi:Their physiology and enzymes[J].Microbiology and Molecular Biology Reviews,2000,64(3):461-488.
[8] 彭燕鸿, 苏爱秋,黄伟文,等.微生物嗜热脂肪酶研究进展[J].食品与发酵工业,2021,47(6):289-294.
PENG Y H,SU A Q,HUANG W W,et al.Research progress on microbial thermophilic lipase[J].Food and Fermentation Industries,2021,47(6):289-294.
[9] JACKSON T J,RAMALEY R F,MEINSCHEIN W G.Thermomicrobium,a new genus of extremely thermophilic bacteria[J].International Journal of Systematic Bacteriology,1973,23(1):28-36.
[10] XIN Y,HAO M Y,FAN G M,et al.Soluble expression of Thermomicrobium roseum sarcosine oxidase and characterization of n-demethylation activity[J].Molecular Catalysis,2019,464(1):48-56.
[11] XIN Y,ZHENG M L,WANG Q,et al.Structural and catalytic alteration of sarcosine oxidase through reconstruction with coenzyme-like ligands[J].Journal of Molecular Catalysis B:Enzymatic,2016,133(2):250-258.
[12] SHA C,YU X W,LIN N X,et al.Enhancement of lipase r27RCL production in Pichia pastoris by regulating gene dosage and co-expression with chaperone protein disulfide isomerase[J].Enzyme and Microbial Technology,2013,53(6-7):438-443.
[13] CAO Y P,XIA Y P,GU X F,et al.PEI-crosslinked lipase on the surface of magnetic microspheres and its characteristics[J].Colloids and Surfaces B:Biointerfaces,2020,189(2):6-14.
[14] WILLEMS N,LELIMOUSIN M,KOLDSO H,et al.Interfacial activation of M37 lipase:A multi-scale simulation study[J].Biochimica et Biophysica Acta-Biomembranes,2017,1 859(3):340-349.
[15] MOHARANA T R,PAL B,RAO N M.X-ray structure and characterization of a thermostable lipase from Geobacillus thermoleovorans[J].Biochemical and Biophysical Research Communications,2019,508(1):145-151.
[16] SHARIFF F M,ABD RAHMAN R N Z R,BASRI M,et al.a newly isolated thermostable lipase from Bacillus sp[J].International Journal of Molecular Sciences,2011,12(5):2 917-2 934.
[17] KAMAL M Z,AHMAD S,MOLUGU T R,et al.In vitro evolved non-aggregating and thermostable lipase:Structural and thermodynamic investigation[J].Journal of Molecular Biology,2011,413(3):726-741.
[18] 许蕊, 张昕怡,潘悦,等.来源于解酯菌的嗜热耐碱脂肪酶的表达纯化及其酶学性质研究[J].林业工程学报,2020,5(3):108-114.
XU R,ZHANG X Y,PAN Y,et al.Expression,purification and characterization of lipase from Thermosyntropha lipolytica[J].Journal of Forestry Engineering,2020,5(3):108-114.
[19] OZTUG M,KILINC E,AKGOZ M,et al.Thermal proteome profiling and meltome analysis of a thermophilic bacterial strain,Geobacillus thermoleovorans ARTRW1:Toward industrial applications[J].Omics:A Journal of Integrative Biology,2020,24(12):756-765.
[20] ESPINOSA-LUNA G,SANCHEZ-OTERO M G,QUINTANA-CASTRO R,et al.Gene cloning and characterization of the Geobacillus thermoleovorans CCR11 carboxylesterase CaesCCR11,a new member of family XV[J].Molecular Biotechnology,2016,58(1):37-46.
[21] YU X W,WANG R,ZHANG M,et al.Enhanced thermostability of a Rhizopus chinensis lipase by in vivo recombination in Pichia pastoris[J].Microbial Cell Factories,2012,11(1):102-113.
[22] GAO J,OU X,XU P,et al.Cloning,overexpression,and characterization of a novel organic solvent-tolerant lipase from Paenibacillus pasadenensis CS0611[J].Chinese Journal of Catalysis,2018,39(5):937-945.
[23] 中华人民共和国国家质量监督检验检疫局, 中国国家标准化管理委员会.GB/T 23535—2009 脂肪酶制剂[S].北京:中国标准出版社,2009.
State Administration of Quality Supervision,Inspection and Quarantine of the People's Republic of China,China National Standardization Administration Committee.GB/T 23535—2009 Lipase preparations[S].Beijing:China Standards Press,2009.
[24] ZHANG J,TIAN M,LV P,et al.High-efficiency expression of the thermophilic lipase from Geobacillus thermocatenulatus in Escherichia coli and its application in the enzymatic hydrolysis of rapeseed oil[J].3 Biotech,2020,10(12):523-532.
[25] MOHAMMADI M,SEPEHRIZADEH Z,EBRAHIM-HABIBI A,et al.Enhancing activity and thermostability of lipase a from Serratia marcescens by site-directed mutagenesis[J].Enzyme and Microbial Technology,2016,93-94(2):18-28.
[26] PARK J H,PARK K M,CHANG Y,et al.Cloning and protein expression of the sn-1(3)regioselective lipase from Cordyceps militaris[J].Enzyme and Microbial Technology,2018,119(3):30-36.
[27] NORO J,CASTRO T G,CAVACO-PAULO A,et al.Substrate hydrophobicity and enzyme modifiers play a major role in the activity of lipase from Thermomyces lanuginosus[J].Catalysis Science & Technology,2020,10(17):5 913-5 924.