苯乳酸(phenyllactic acid,PLA),即2-羟基-3-苯基丙酸[1],是一种小分子天然有机酸,广泛存在于干酪、天然蜂蜜中。溶解性良好,具有耐酸、耐碱、耐高温等特性[2],并能有效的抑制食腐性微生物[3]的生长,在食品工业中具有广阔的应用前景。L-苯乳酸(L-PLA)作为PLA两种对映异构体中的一种,是合成许多药物制剂的前体[4]。
合成L-PLA方法主要为化学合成法、微生物发酵和生物催化合成。化学合成法[5]技术路线复杂,反应条件苛刻,易产生环境污染,后续分离困难,不易纯化。微生物发酵法[6]通过微生物分解与合成代谢生产PLA。由于其发酵周期长,培养基利用率不高,生产效率低,不具工业利用价值。基因工程和蛋白质工程[7]的发展使得生物催化技术得到广泛的运用。XU等[8]通过在大肠杆菌中共表达源于干酪乳杆菌(Lactobacillus casei)的酮酸还原酶(LcKAR)和葡萄糖脱氢酶(glucose dehydrogenase,GDH),以苯丙酮酸(phenylpyruvic acid,PPA)为底物不对称合成PLA,时空产率达到10.12 g/(L·h)。LUO等[9]研究中以PPA为底物目前产量达到20.5 g/L。近几年以PPA为底物合成PLA的研究较多,关于苯丙氨酸(phenylalanine,Phe)一步合成PLA的报道相对较少,且PPA价格偏高,挖掘能够成本低廉并高效转化的生物催化合成PLA方法是未来研究亟待解决的问题。
L-氨基酸脱氨酶(L-amino acid deaminase,L-AAD)可以用来催化Phe生成PPA,L-AAD有广泛的底物特异性[10],对疏水性氨基酸有显著催化特性,L-AAD是膜蛋白[11],更有利于制备全细胞催化剂。苯丙酮酸还原酶(phenylpyruvate reductase,LaPPR)是一种还原型辅酶Ⅰ(nicotinamide adenine dinucleotide health,NADH)依赖型的氧化还原酶,具有对芳香族和脂肪族酮酸的底物偏好[12]。本研究首次将L-AAD和LaPPR用于催化L-苯丙氨酸(L-Phe)合成L-PLA。考虑到还原性辅酶NADH稳定性差,价格昂贵,提高成本,不适合外源添加[13],其中,GDH和甲酸脱氢酶(formate dehydrogenase , FDH)是构建辅酶再生体系最常用的酶[14],通过对辅酶再生酶的筛选,制备全细胞催化剂转化合成PLA。旨在为L-PLA生产建立一种简洁、高效、经济的生物催化合成方法,为其工业化生产奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
大肠杆菌Escherichia coli BL21(DE3)及质粒pET-28a均为本实验室保藏。
1.1.2 酶和试剂
Hind Ⅲ、NotⅠ、XhoⅠ限制性核酸内切酶与DNA聚合酶,TaKaRa公司;小量质粒提取试剂盒、同源重组试剂盒和胶回收试剂盒,南京诺维赞生物科技有限公司;L-Phe、L-苯丙酮酸、L-PLA,国药集团;酵母提取物和胰蛋白胨,英国Oxoid 公司。所有实验试剂如果无特殊说明均为分析纯。
1.1.3 培养基
LB种子培养基(g/L)[15]:蛋白胨10,酵母粉5,NaCl 10,pH 7.0。
TB发酵培养基(g/L)[15]:蛋白胨 12,酵母粉 24,甘油5,KH2PO4 2.31, K2HPO4 12.54。
1.2 实验方法
1.2.1 引物设计
通过NCBI数据库获得编码普通变形杆菌Proteus vulgaris来源的L-AAD基因、乳酸杆菌Lactobacillus sp. CGMCC 9967苯丙酮酸还原酶的基因序列,选择酶切位点,设计并合成多对引物,序列如表1所示。
表1 本研究中使用的引物
Table 1 Primers used in this study

注:斜体序列为酶切位点
1.2.2 重组质粒的构建
根据NCBI中公布的普通变形杆菌P.vulgaris来源的L-AAD基因laad(GenBank登录号:AB030003.1)、乳酸杆菌Lactobacillus sp.CGMCC 9967来源的LaPPR基因[16]lappr(GenBank登录号:KP735960.1)、枯草芽孢杆菌Bacillus subtilis来源的GDH基因[16]gdh(GenBank登录号:938261)以及博伊丁假丝酵母Candida boidinii 的FDH基因[16]fdh(GenBank登录号:7657866)序列设计引物,分别以各自基因组为模板,进行PCR扩增, PCR产物laad、lappr、gdh和fdh经过试剂盒纯化后,连接载体pET-28a,转化E.coli BL21(DE3)中。
将gdh基因克隆到重组表达载体pET-28a-lappr的MCS位点上,引入限制性酶切位点NcoⅠ,用各自的T7启动子进行表达,构建pET-28a-t7-gdh-t7-ppr共表达质粒。选择限制性酶切位点XhoⅠ,将laad基因克隆到重组质粒上,构建pET-28a-t7-gdh-t7-lappr-t7-laad共表达质粒,转化E.coli BL21(DE3)中,涂布平板,并过夜培养,挑取阳性转化子,送至测序,若正确,即重组菌株E.coli BL21/pET28a-laad-lappr-gdh构建成功。
1.2.3 重组菌的培养及诱导
重组菌E.coli BL21/pET28a-laad、E.coli BL21/pET28a-lappr、E.coli BL21/pET28a-gdh、E.coli BL21/pET28a-fdh、E.coli BL21/pET28a-laad-lappr-gdh,1%的接种量转接入含Kan 50 μg/mL的种子培养基,37 ℃、180 r/min,培养12 h。再按2%的接种量接入发酵培养基,37 ℃,菌体OD600值为0.4~0.5时,加入异丙基-β-D-硫代半乳糖苷至终浓度为0.5 mmol/L,24 ℃、180 r/min诱导12 h[15]。
1.2.4 酶活测定
诱导后的大肠杆菌发酵液于4 ℃离心(8 000 r/min,5 min)收集菌体,用PB缓冲液(0.1 mol/L,pH为8.0)重悬菌体沉淀,低温超声破碎,并在4 ℃条件下高速离心后,获得破碎上清液,即为粗酶液,进行SDS-PAGE分析及酶活力测定[17]。酶活力测定分别参考HOU等[12]、XU等[8]的报道。
1.2.5 酶学性质
酶最适反应温度和最适反应pH测定:在25、30、35、40、45、50 ℃下测定酶活力,分析确定酶反应的最适温度,设置酶活力最高组为100%对照[15]。在pH 4.0、5.0、6.0、7.0、8.0、9.0、10.0的条件下测定酶活力,分析确定酶反应的最适温度,设置酶活力最高组为100%对照[18]。
酶温度和pH稳定性测定:取适量纯化的酶置于不同温度(25~45 ℃)下,在最适pH条件下定时取样测定残余酶活力,以最高残余酶活力为100%计算相对酶活力。取适量纯化的酶置于不同pH(4.0~10.0)条件下,在最适温度条件下定时取样测定残余酶活力,以最高残余酶活力为100%计算相对酶活力。
1.2.6 L-PLA的合成及条件优化
利用重组菌E.coli/pET28a-laad-lappr-gdh进行全细胞转化环合成L-PLA,以 L-Phe作为底物,同时加入辅底物葡萄糖及5%(体积分数)的甘油,转化体系为20 mL,转速220 r/min进行反应,反应过程中通过添加适量的NaOH来调节反应体系的pH。
全细胞转化体系的优化:在不同的温度(25~45 ℃)、初始pH(6.0~9.0)、菌体量OD600(10~50)、底物质量浓度(20~50 g/L)、辅底物与底物摩尔质量比(0.5∶1~4∶1)的条件下进行L-PLA的转化实验。HPLC法检测产物L-PLA的产量的具体方法参考黄国昌等[19]的检测方法。
2 结果与分析
2.1 laad,lappr,gdh及fdh基因的克隆与表达
从 NCBI 数据库中检索到L-AAD、LaPPR、GDH、FDH的序列,长度分别为1 416、1 047、789、1 197 bp,分别作为模板,进行 PCR 扩增,电泳结果如图1所示,可见特异性条带位置与目标基本一致。
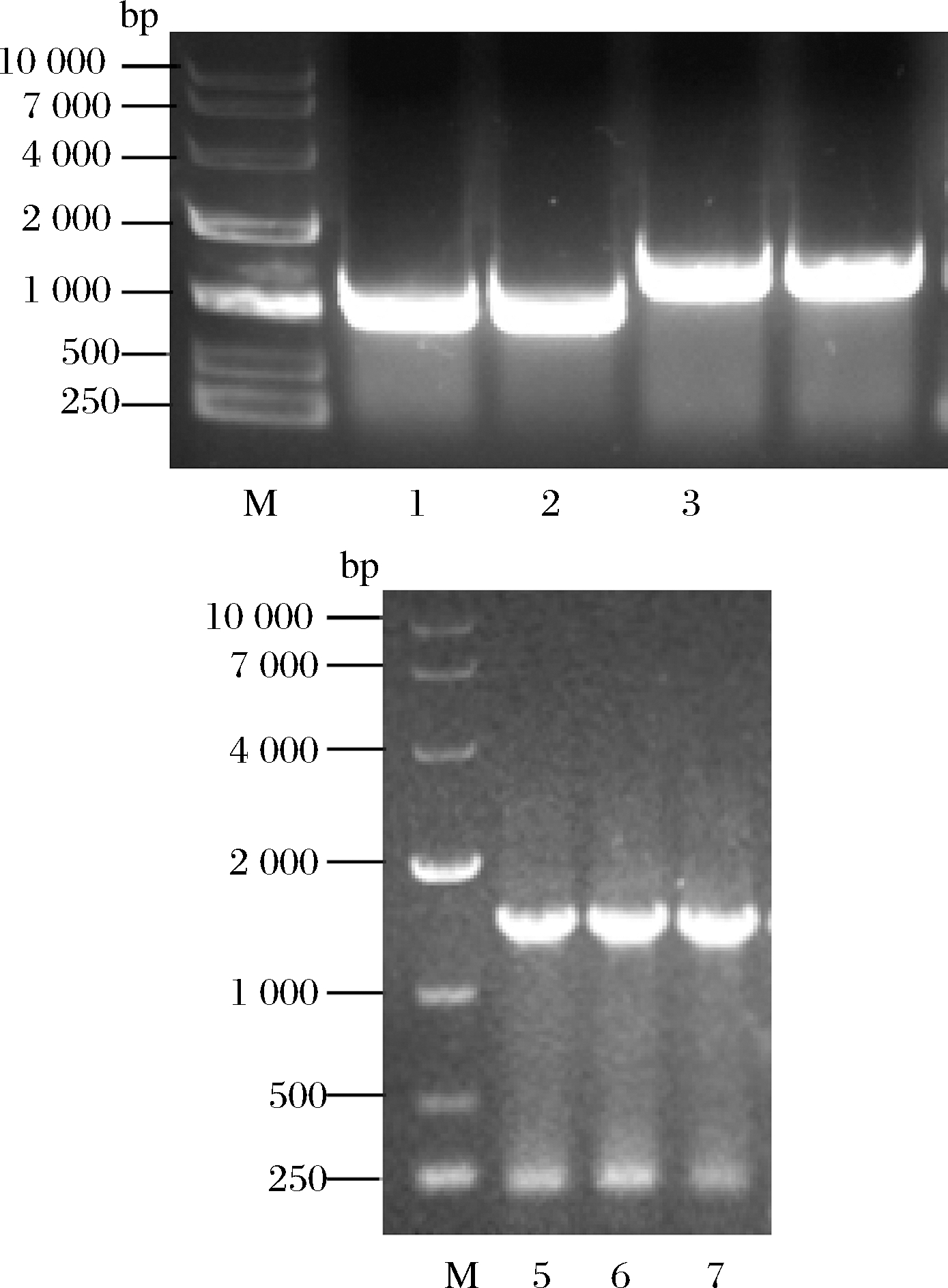
M-DL 10 000 marker;1、2-基因 gdh;3、4基因 lappr;5~7-基因laad
图1 基因的克隆
Fig.1 The result of gene cloning
2.2 温度和pH对L-AAD及LaPPR酶活力和稳定性的影响
2.2.1 温度对L-AAD和LaPPR酶活力和稳定性的影响
在不同反应温度下测定L-AAD酶活力,如图2-a,L-AAD的最适反应温度为35 ℃,存在较高酶活力,温度高于40 ℃,酶活力下降。图2-b中显示,L-AAD在30~40 ℃较稳定,酶活力保持在75%以上,而在45、50 ℃条件下,则迅速降到40%以下,可见在30~40 ℃有较好的稳定性。
在不同反应温度下测定LaPPR酶活力,如图2-c,LaPPR的最适反应温度为30 ℃,并具有较高酶活力,温度高于35 ℃,酶活力下降。图2-d中显示,LaPPR在25~35 ℃比较稳定,酶活力保持在80%以上,而在40~45 ℃条件下,相对酶活力迅速降到20%以下,这表明在25~35 ℃有较好的催化性能和热稳定性。
2.2.2 pH对L-AAD和LaPPR酶活力和稳定性的影响
在不同pH条件下测定L-AAD酶活力,结果如图3-a所示,随着反应体系pH的逐渐提高,L-AAD 的相对酶活力也逐渐提高,L-AAD的最适反应pH值为8.0,在pH 7.0~9.0具有较高酶活力。图3-b中显示,当处于偏酸性环境中时,L-AAD稳定性较差,相对酶活力降到50%以下;在pH 7.0~9.0偏中性范围内比较稳定,酶活力保持在80%以上,有利于其在生物催化工业上的应用。
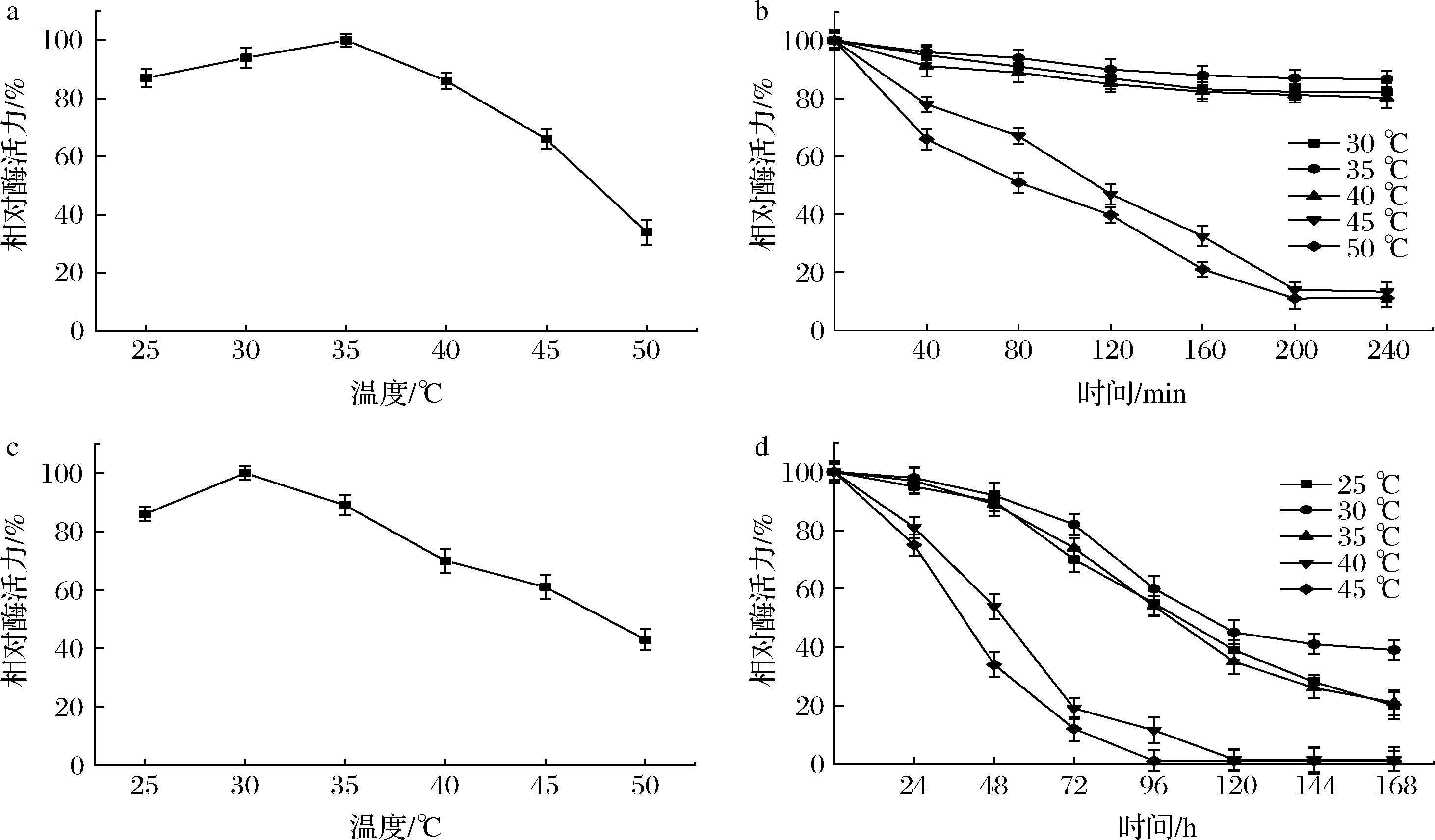
a-L-AAD的最适温度;b-L-AAD温度稳定性;c-LaPPR的最适温度;d-LaPPR温度稳定性
图2 温度对L-AAD和LaPPR的影响
Fig.2 The influence of temperature on L-AAD and LaPPR
在不同反应pH下测定LaPPR酶活力,结果如图3-c所示,LaPPR的最适反应pH为7.0,在pH 7.0~8.0具有较高酶活力,当pH值低于7.0,酶活力迅速下降。图3-d中显示,在pH 7.0~8.0比较稳定,酶活力保持在80%以上,而偏酸性条件下,相对酶活力迅速降到40%以下,这表明在pH 7.0~8.0有较好的稳定性。本研究中使用的L-AAD和LaPPR具有良好的兼容性,为后续多酶级联奠定基础。

a-L-AAD的最适pH;b-L-AAD pH稳定性;c-LaPPR的最适pH;d-LaPPR pH稳定性
图3 pH对L-AAD和LaPPR的影响
Fig.3 The effect of pH on L-AAD and LaPPR
2.3 辅酶自循环体系的筛选及优化
2.3.1 辅酶自循环体系的优化
LaPPR催化PPA过程中需要消耗NADH,而NADH稳定性差,成本高[18],在工业生产中添加昂贵的辅因子会大大增加生产成本,不符合绿色经济。辅酶再生是解决其价格昂贵的有效方法,现主要有葡萄糖脱氢酶和甲酸脱氢酶再生系统,通过分别催化葡萄糖和甲酸来再生NADH。
首先对来源于Lactobacillus sp.CGMCC 9967的LaPPR转化能力进行验证,并对GDH和FDH进行筛选比较。20 mL的转化体系中包含PPA 10 g/L、1.5倍摩尔量的辅底物,分别加入LaPPR和GDH,以及LaPPR和FDH的粗酶液,并以LaPPR和原始菌株粗酶液组作为对照,各粗酶液在转化体系中的终浓度均为1 U/mL,转化反应于pH值为7.5, 220 r/min、30 ℃ 条件下进行[16]。结果如表2所示,通过比较两者酶活力以及计算其初始反应速率,GDH初始反应速率为16.16 mg/(L·min),FDH初始反应速率为8.78 mg/(L·min),因此选择GDH为下一步构建重组菌作铺垫。
2.3.2 LaPPR和GDH的酶量配比优化
在多酶级联催化反应中,酶的添加比例能够影响催化反应速率的平衡[20]。LaPPR和GDH作为参与还原反应中的重要酶,为了保证催化速率达到平衡,减少中间产物PPA积累,须对LaPPR和GDH的添加比例进行优化。
20 mL的反应体系中包含10 g/L的底物PPA,保持LaPPR终浓度为1 U/mL,GDH的添加量高于LaPPR,设定GDH与LaPPR不同添加比例,酶活力优化范围设定为1∶1~5∶1,按照不同添加比例催化底物,计算产物L-PLA的生成速率[16]。如图4所示,随着GDH的逐步增加,初始反应速率不断增加,当m(GDH)∶m(LaPPR)=4∶1时,初始反应速率达到最大值,为27.63 mg/(L·min)。

图4 LaPPR和GDH添加比例优化
Fig.4 Optimization of dosage of LaPPR and GDH
2.4 共表达重组菌的构建及优化
按照方法1.2.2构建重组菌株E.coli BL21/pET28a-laad-lappr-gdh,超声破碎并离心,取上清液进行SDS-PAGE 分析,结果见图5,在约52.05、37、29.8 kDa位置处有明显的蛋白质特征条带,大小一致,各基因成功在大肠杆菌中表达。
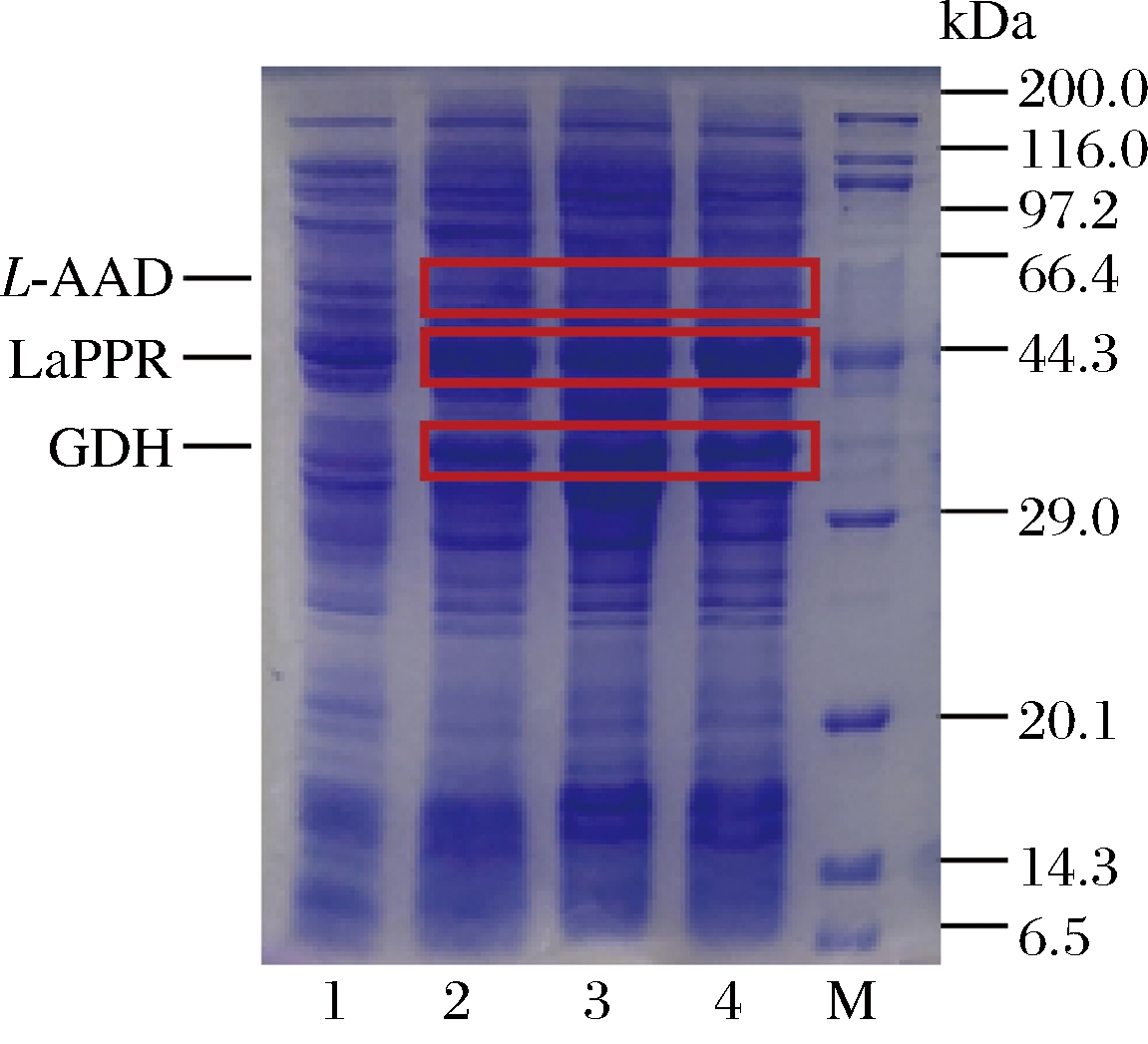
M-蛋白质marker;1- E.coil BL21/pET-28a crude enzyme;
2~4-E.coil BL21/pET-28a-laad-lappr-gdh
图5 基因的表达及分析
Fig.5 The result of gene expression and purification
利用上述共表达重组菌进行全细胞转化,结果如图6所示,当提供足够的Phe和葡萄糖时,底物Phe可大量被消耗,此时中间产物PPA大量积累,质量浓度达到27.89 g/L,而最终产物L-PLA产生速率较为缓慢。中间产物积累浓度较高,影响整个反应顺利进行。可能是由于LaPPR和GDH两个关键酶转化过程不平衡,从而影响PPA转化至L-PLA的催化速率。经酶活力测定,结果如表2所示,在重组菌中,LaPPR酶活力为12.89 U/mL,GDH为7.64 U/mL,GDH酶活力较低,从而影响反应使其不能相互协调,导致PPA不能被快速催化,造成中间产物的积累,影响L-PLA的生产。这一限速步骤对L-PLA产量的影响是亟需解决的问题。

图6 重组菌E.coli BL21/pET28a-laad-lappr-gdh全细胞转化合成L-PLA
Fig.6 The recombinant E.coli BL21/pET28a-laad-lappr-gdh whole cells catalyze the synthesis of L-PLA
2.5 限速步骤的解除
为了协调具有一个载体的共表达菌株中LaPPR和GDH的表达水平,通过对LaPPR和GDH的表达量进行调节优化,增加GDH的拷贝量,其酶活力情况如表2所示。通过调节级联全细胞生物催化剂中酶的表达,当GDH为2个拷贝量时,中间产物PPA的积累减少,减少了84.60%,L-PLA产量有较大的提高,产量增加了3倍多。
表2 LaPPR、FDH及GDH在大肠杆菌中的酶活力 单位:U/mL
Table 2 The activities of LaPPR, FDH and GDH in E.coli

注:—表示未检测出酶活力
2.6 重组菌全细胞转化合成L-PLA的条件优化
2.6.1 温度对全细胞转化合成L-PLA的影响
反应体系的温度会直接影响酶的催化活性和稳定性[15],从而影响最终产物PLA的产量。例如L-AAD和LaPPR最适反应温度分别为37、30 ℃,为了能够达到最佳的转化效果,需要对转化的温度进行优化。控制初始反应pH值为8.0,底物质量浓度30 g/L,菌体量OD600为30,辅底物葡萄糖1倍摩尔当量,在不同温度(25~45 ℃)下进行全细胞转化反应。
如图7-a所示,温度为30 ℃时,L-PLA产量达到最高,20.24 g/L,转化率为67.49%,而当温度增加到45 ℃时,L-PLA产量显著下降,说明此时某种酶活力受到影响,无法维持反应的进行。
2.6.2 初始pH对全细胞转化合成L-PLA的影响
在多酶催化反应体系中,pH通过影响菌体状态和酶促反应的效率而影响L-PLA的合成,例如L-AAD和LaPPR最适反应pH值分别为8.0和7.0,为了使反应高效进行,对转化初始pH进行了优化。维持反应温度30 ℃,底物质量浓度30 g/L,菌体量OD600为30,辅底物葡萄糖1倍摩尔当量,在初始反应体系pH值分别为6.0、7.0、8.0、9.0、10.0条件下进行全细胞转化反应。
如图7-b所示,随着pH的升高,L-PLA产量增加,且8.0时达到最高21.02 g/L,转化率为70.09%,偏碱性环境可以实现3种酶的良好催化。
2.6.3 菌体量对全细胞转化合成L-PLA的影响
实际应用中为了满足生产效率和降低生产成本,控制菌体浓度在适宜范围内也是至关重要的。控制初始反应pH值为8.0,反应温度30 ℃,底物质量浓度30 g/L,辅底物葡萄糖1倍摩尔当量,菌体OD600为10~50,进行催化反应。
如图7-c所示,转化体系菌体OD600为30时,L-PLA产量达到最高,为21.20 g/L,转化率为70.66%。
2.6.4 底物浓度对全细胞转化合成L-PLA的影响
为了提高L-PLA的产量,在前期优化的最佳转化条件下(温度30 ℃,初始pH值为8.0,菌体量OD600,辅底物葡萄糖添加量为1倍摩尔当量)合成L-PLA,底物Phe质量浓度分别为10、20、30、40、50 g/L 进行催化反应。
如图7-d所示,底物质量浓度为30 g/L时,产量达到最大值,为21.36 g/L,转化率为71.22%,当底物质量浓度为50 g/L时,转化率也能维持在40.29%左右。可能由于Phe溶解度低,反应体系中还有其他物质,体系无法承载过多底物,从而产量有所下降[19]。
2.6.5 辅底物与底物比例对全细胞转化合成L-PLA的影响
葡萄糖在GDH作用下生成辅酶NADH,随着NADH的增加,重组菌合成L-PLA产量越高,对反应体系中葡萄糖的添加量进行了优化。控制反应温度30 ℃,初始反应pH值为8.0,菌体量OD600为30,底物Phe质量浓度30 g/L,添加0、0.5、1、1.5、2、2.5倍摩尔当量的葡萄糖进行全细胞催化。
如图7-e示,未添加葡萄糖时,由于大肠杆菌本身含有一定的辅酶NADH,但不能支撑整个反应的进行,可生成少量L-PLA。当葡萄糖添加量为1、1.5、2、2.5倍摩尔当量时,L-PLA产量无明显差异变化,表明辅底物添加量≥1倍摩尔量时即可满足反应需求,选择葡萄糖添加量为1倍摩尔当量最为合适。

a-温度;b-初始pH;c-菌体浓度;d-底物浓度;e-辅底物添加量
图7 转化条件对重组菌E.coli BL21/pET28a-laad-lappr-gdh-gdh全细胞转化合成L-PLA的影响
Fig.7 Effect of transformation conditions on the transformation and synthesis of L-PLA by recombinant E.coli BL21/pET28a-laad-lappr-gdh-gdh
2.7 1 L放大全细胞转化L-PLA
为利用重组大肠杆菌E.coli BL21/pET28a-laad-lappr-gdh-gdh高效转化生产L-PLA,按照上述优化后全细胞催化条件研究重组菌在大体系中的转化水平。用1 L 0.1 mol/L 的PB缓冲液悬浮菌体,控制反应温度30 ℃,初始pH值为8.0,底物Phe 30 g/L,甘油2.5 mol/L,辅底物葡萄糖1倍摩尔当量,菌体OD600为30的条件下进行转化,液相检测转化液中各组分含量情况。
如图8所示,30 g/L底物质量浓度下,共生成21.39 g/L L-PLA,转化率为71.33%。反应后期延长转化时间,转化率变化不明显,可能是反应趋于平衡。

图8 重组菌E.coli BL21/pET28a-laad-lappr-gdh-gdh全细胞转化合成L-PLA
Fig.8 The recombinant E.coli BL21/pET28a-laad-lappr-gdh-gdh whole cells catalyze the synthesis of L-PLA
3 结论与讨论
本研究通过构建高效便捷的多酶级联催化体系,利用重组大肠杆菌全细胞催化Phe合成L-PLA。首先分别对L-AAD和LaPPR分别进行酶学性质研究,以此为基础筛选比较构建辅酶自循环体系,解决中间产物积累问题,从而实现Phe到L-PLA的直接转化,最终产量达到21.39 g/L,摩尔转化率为71.33%。然而本研究所制备的产量仍有提升空间,导致产量无法进一步提高的原因是由于Phe溶解度低,反应体系无法承载过多底物,且有结晶存在,因此转化率无法进一步提高,增加了转化体系的传质阻力[21],同时影响酶的催化特性。本研究提供了一种新型的L-PLA合成途径,因PPA价格较高,由Phe为底物,大大减少了生产成本,缩短了工艺流程,采用全细胞催化合成L-PLA,操作工艺便捷,转化效率高。对提供挖掘成本低廉并能高效转化的生物催化合成L-PLA的方法具有借鉴作用,同时对其在食品及合成药物行业生产具有广泛的指导意义。
[1] ARMAFORTE E, CARRI S, FERRI G, et al.High-performance liquid chromatography determination of phenyllactic acid in MRS broth[J].Journal of Chromatography A, 2006, 1 131(1-2):281-284.
[2] PENG Q, ZENG X F, ZHU J L, et al.Effects of dietary Lactobacillus plantarum B1 on growth performance, intestinal microbiota, and short chain fatty acid profiles in broiler chickens[J].Poultry Science, 2016, 95(4):893-900.
[3] SCHNRER J, MAGNUSSON J.Antifungal lactic acid bacteria as biopreservatives[J].Trends in Food Science & Technology, 2005, 16(1-3):70-78.
[4] HASHIMOTO Y, KOBAYASHI E, ENDO T, et al.Conversion of a cyanhydrin compound into S-(-)-3-phenyllactic acid by enantioselective hydrolytic activity of Pseudomonas sp.BC-18[J].Bioscience, Biotechnology, and Biochemistry, 1996, 60(8):1 279-1 283.
[5] BUSTOS A Y, VALDEZ G F, GEREZ C L.Optimization of phenyllactic acid production by Pediococcus acidilactici CRL 1753.application of the formulated bio-preserver culture in bread[J].Biological Control, 2018, 123(3):137-143.
[6] LEE H S, PARK J, YOO Y J, et al.A novel d-2-hydroxy acid dehydrogenase with high substrate preference for phenylpyruvate originating from lactic acid bacteria:Structural analysis on the substrate specificity[J].Enzyme and Microbial Technology, 2019, 125:37-44.
[7] LIU S P, ZHANG L, MAO J, et al.Metabolic engineering of Escherichia coli for the production of phenylpyruvate derivatives[J].Metabolic Engineering, 2015, 32(1):55-65.
[8] XU G C, ZHANG L L, NI Y.Enzymatic preparation of D-phenyllactic acid at high space-time yield with a novel phenylpyruvate reductase identified from Lactobacillus sp.CGMCC 9967[J].Journal of Biotechnology, 2016, 222(1):29-37.
[9] LUO X, ZHANG Y, YIN F, et al.Enzymological characterization of a novel d-lactate dehydrogenase from Lactobacillus rossiae and its application in d-phenyllactic acid synthesis[J].3 Biotech, 2020, 10(3):101.
[10] COBAN H B, DEMIRCI A, PATTERSON P H, et al.Screening of phenylpyruvic acid producers and optimization of culture conditions in bench scale bioreactors[J].Bioprocess and Biosystems Engineering, 2014, 37(11):2 343-2 352.
[11] ZHANG L, LIU Q, PAN H, et al.Metabolic engineering of Escherichia coli to high efficient synthesis phenylacetic acid from phenylalanine[J].AMB Express, 2017, 7(1):105.
[12] HOU Y, HOSSAIN G S, LI J H, et al.Combination of phenylpyruvic acid (PPA) pathway engineering and molecular engineering of L-amino acid deaminase improves PPA production with an Escherichia coli whole-cell biocatalyst[J].Applied Microbiology and Biotechnology, 2016, 100(5):2 183-2 189.
[13] LI J F, LI X Q, LIU Y, et al.Directed modification of l-LcLDH1, an l-lactate dehydrogenase from Lactobacillus casei, to improve its specific activity and catalytic efficiency towards phenylpyruvic acid91[J].Journal of Biotechnology, 2018, 281(1):193-198.
[14] HOU Y, GAO B, CUI J, et al.Combination of multi-enzyme expression fine-tuning and co-substrates addition improves phenyllactic acid production with an Escherichia coli whole-cell biocatalyst[J].Bioresource Technology, 2019, 287(2):121 423.
[15] 段培峰, 尤甲甲, 徐美娟, 等.重组枯草芽孢杆菌全细胞催化合成 2-O-α-D-甘油葡糖苷展[J].生物工程学报, 2020, 36(8):1-11.
DUAN P F, YOU J J, XU M J, et al.Whole-cell biosynthesis of 2-O-α-D-glu-copyranosyl-sn-glycerol by recombinant Bacillus subtilis[J].Chinese Journal of Biotechnology, 2020, 36(8):1-11.
[16] 刘巧利, 杨套伟, 周俊平, 等.酶法高效转化苯乙酮酸合成L-苯甘氨酸[J].应用与环境生物学报.2019, 25 (2):451-156.
LIU Q L, YANG T W, ZHOU J P, et al.Efficient enzymatic synthesis of L-phenylglycine from benzoylformic acid[J].Chinese Journal of Applied & Environmental Biology, 2019, 25 (2):451-456.
[17] KAWAGUCHI H, MIYAGAWA H, NAKAMURA T S, et al.Enhanced phenyllactic acid production in Escherichia coli via oxygen limitation and shikimate pathway gene expression[J].Biotechnology Journal, 2019, 14(6):1 800 478.
[18] ZHAO W, DING H, LYU C J, et al.Two-step biocatalytic reaction using recombinant Escherichia coli cells for efficient production of phenyllactic acid from L-phenylalanine[J].Process Biochemistry, 2018, 64(2):7-31.
[19] 黄国昌, 顾斌涛, 黄朝, 等.苯乳酸生产菌的筛选及鉴定[J].食品与发酵工业, 2018, 44(3):97-100.
HUANG G C,GU B T, HUANG Z, et al.Screening and identification of phenyllactic acid-producing bacteria[J].Food and Fermentation Industries,2018, 44(3):97-100.
[20] WU S, CHEN Y, XU Y, et al.Enantioselective trans-dihydroxylation of aryl olefins by cascade biocatalysis with recombinant Escherichia coli coexpressing monooxygenase and epoxide hydrolase[J].ACS Catalysis, 2014, 4(2):409-420.
[21] DAO Y, ZHANG K, LU X F, et al.Role of glucose and 2-oxoglutarate/malate translocator(OMT1) in the production of phenyllactic acid and p-hydroxyphenyllactic acid, two food-borne pathogen inhibitors[J].Journal of Agricultural and Food Chemistry, 2019, 67(20):5 820-5 826.