藏东南地区主要包括林芝和昌都地区,其独特的地理位置形成了特殊的气候类型,适合葡萄种植和酿造葡萄酒,素有“中国野生红葡萄之乡”和“水果之乡”之称。微生物在葡萄种植和酿酒过程中扮演着养分吸收和能量转化的重要角色,是葡萄生态系统的重要组成部分[1]。有研究表明土壤微生物在促进植物生长、提高土壤肥力、植物健康和物质运输及转化过程中发挥重要作用[2],CHAUDHRY等[3]研究发现较高丰度的变形菌门(Proteobacteria)有助于土壤养分积累和植物生长。由于酿造葡萄酒是一个复杂的微生物发酵过程,其微生物主要来源于葡萄表面、种植土壤和发酵设备[4-5]。MARTINS等[6]研究葡萄表皮附着微生物时发现假单胞杆菌属(Pseudomonas)普遍存在于葡萄中,同时还发现土壤微生物与葡萄上的微生物存在一定的联系;BOKULICH等[7]在研究葡萄汁微生物时发现糖杆菌属(Gluconobacter)具有促进脂类物质合成、改善酒的风味和质量的功效。因此,葡萄表皮及根际土壤存在与酿酒及种植相关的重要微生物类群。
葡萄表皮和土壤微生物种类与其生长环境息息相关,有研究表明,不同气候类型或地理位置其葡萄表皮和土壤微生物种类存在显著差异[8-10],藏东南独特的地理气候环境势必蕴藏着丰富的微生物资源。目前中国各地葡萄产区的微生物群落已被广泛深入研究,但西藏藏东南葡萄产区微生物群落结构及多样性尚未见报道。
近年来,高通量测序技术由于其准确性高、通量大的优点,在微生物学领域得到了广泛的应用和发展[11-12]。为了研究藏东南葡萄产区细菌种类和丰度,本文采用高通量测序技术,对藏东南2个地区葡萄园中的葡萄和土壤细菌群落组成和多样性进行了分析,以期分析不同产区,不同生态系统中细菌群落间的生态联系和相互作用,为藏东南优良酿酒细菌的筛选和利用提供理论依据。
1 材料与方法
1.1 材料与试剂
林芝葡萄(LP)、林芝葡萄根际土壤(LS)、芒康葡萄(MP)和芒康葡萄根际土壤(MS);DNeasy Plant Mini Kit(Qiagen,Germantown,MD,USA);Thermus Aquaticus(Taq),天根生化科技(北京)有限公司;胶回收试剂盒,美国Omega公司;琼脂糖、Qubit2.0 DNA检测试剂盒,美国Invitrogen公司。
1.2 仪器与设备
NanoDrop2000微量分光光度计,美国Thermo公司;5418R高速冷冻离心机,德国Eppendorf公司;PCR仪、凝胶成像系统,美国Bio-rad公司;电泳仪,北京六一;Hiseq测序平台,美国Illumina公司。
1.3 材料与方法
1.3.1 样品采集及处理
新鲜葡萄和土壤样品来自昌都芒康(纬度29°1′53.1″N,经度98°36′21.77″E)和林芝(纬度29°25′33.37″N,经度94°26′50.93″E),于2019年8月采集,每组3个重复。将采集到的样品装入无菌袋中,低温保存并带回实验室,24 h内处理。根际土壤过2 mm的过滤筛以除去残枝落叶并自然风干,4 ℃保存备用。
1.3.2 不同种源地葡萄根际土壤样品理化性质分析
土壤理化性质测定采用常规检测方法[13],测定指标主要包括pH、总磷(total phosphorus,TP)、总钾(total potassium,TK)、总氮(total nitrogen,TN)、速效氮(available nitrogen,AN)、速效磷(available phosphorus,AP)、电导率(conductivity,EC)和有机质(soil organic matter,SOM)含量。土壤pH检测采用电位法,总磷和速效磷检测采用钼锑抗比色法,总钾和速效钾检测采用火焰光度计法,总氮检测采用凯氏定氮法,速效氮检测采用碱解扩散法,电导率测定采用电极法,有机质测定采用重铬酸钾水合热法。
1.3.3 样品总DNA的提取
组织和土壤样品总DNA的提取使用DNeasy Plant Mini Kit(Qiagen,Germantown,MD,USA),用1%的琼脂糖凝胶电泳检测所提取DNA的质量并用NanoDrop ND-2000紫外分光光度计检测DNA的浓度和纯度。
1.3.4 样品PCR
利用338F(ACTCCTACGGGAGGCAGCA)和806R(GGACTACHVGGGTWTCTAAT)对细菌16S rDNA进行PCR扩增,引物由上海美吉生物有限公司合成。PCR扩增采用25 μL反应体系:10×PCR Buffer 2.5 μL,338F和806R引物各1 μL,5 mmol/L dNTPs 1 μL,5.0 U/μL rTaq酶0.25 μL,DNA模板1 μL,ddH20 18.25 μL。PCR反应程序如下:95 ℃ 5 min;35个循环(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s),72 ℃ 10 min。2%的琼脂糖凝胶电泳检测并纯化PCR产物,将纯化的PCR产物送至上海美吉生物医药有限公司进行测序。
1.3.5 数据统计与分析
原始数据经数据过滤除去低质量的reads以获得 Clean data;通过reads间的Overlap将reads拼接成Tags;在97%相似水平下对Tags进行可操作分类单元(operational taxonomic units,OTUs)聚类,采用RDP classifier贝叶斯算法对OTUs序列进行分类学分析。采用Mothur 1.30.2对样品的Alpha多样性进行分析,包括群落覆盖度、Chao1指数、香农指数、辛普森指数和Shannoneven等指数,Chao1指数值越大,说明群落丰富度越高,利用Qiime计算Beta多样性和主坐标分析(principal coordinates analysis,PCoA)[14];Venn图利用R语言工具统计和作图;用R语言vegan包进行冗余分析(redundancy analysis,RDA)和热图制作;利用PICRUSt软件包对所有样本的微生物群落进行功能预测。
2 结果与分析
2.1 不同种源地葡萄根际土壤理化性质分析
葡萄根际土壤理化性质如表1所示,MS样品中TP、TK、TN、AN、EC和SOM含量均显著高于LS,但AP含量显著低于LS,两地根际土壤pH在6.753~7.183,呈中性。总体而言,MS样品中土壤营养成分明显高于LS,LS样品以沙土为主,而MS样品主要以腐殖土为主,不同根际土壤理化性质差异可能与两地根际土壤性质和施肥管理措施有关。
表1 不同样品根际土壤理化性质
Table 1 Rhizosphere soil physical and chemical properties of different samples

样品TP/(g·kg-1)TK/(g·kg-1)TN/(g·kg-1)AN/(g·kg-1)pHEC/(μs·cm-1)AP/(mg·kg-1)SOM/(g·kg-1)LS0.779±0.0085.830±0.0350.811±0.0410.153±0.0066.753±0.09459.833±3.37430.853±1.0297.980±0.425MS1.025±0.0556.227±0.2283.208±0.2610.243±0.0157.183±0.03867.167±0.93916.829±1.26776.193±1.503
注:LS为林芝葡萄根际土壤;MS为芒康葡萄根际土壤(下同)
2.2 实验样品细菌序列统计及多样性分析
高通量测序结果如表2所示,LP、LS、MP和MS分别获得有效序列数为38 986、26 430、29 267和27 556,产生的OTUs分别为463、1 247、310和1 448。相对而言,葡萄中获得的有效序列数最多,产生的OTUs个数最少,而根际土壤中获得的有效序列数最少,但产生的OTUs个数最多,说明葡萄中大部分有效序列被聚类到个别OTU。Alpha多样性分析表明,MS中细菌均匀度指数、丰富度指数和多样性指数最高,葡萄样品中LP的均匀度指数和多样性指数显著高于MP,而丰富度指数低于MP。以上结果表明,不同种源地葡萄和根际土壤细菌多样性指数差异显著,根际土壤细菌群落种类最多、最为复杂。覆盖度检测结果代表了每个样本的测序深度,反映了样本中细菌的真实情况。LP、LS、MP和MS的测序覆盖度均超过99%,说明此次测序结果基本能反映样品中细菌的真实情况。
表2 不同样品中细菌群落多样性指数分析
Table 2 Diversity index of bacteria community in the different samples

样品Chao1ShannonSimpson均匀度指数覆盖度/%有效序列数 OTUsLP231.44±4.793.87±0.230.055±0.0140.72±0.0499.9838 986463LS1 207.54±22.024.86±0.070.030±0.0020.70±0.0199.1126 4301 247MP235.77±4.383.13±0.120.089±0.0150.58±0.0399.8729 267310MS1 373.97±12.825.96±0.050.006±0.000 50.84±0.00599.2627 5561 448
注:LP为林芝葡萄;MP为芒康葡萄(下同)
2.3 实验样品细菌群落结构分析
细菌在门分类水平的相对丰度如图1-a所示,Proteobacteria是葡萄表皮的优势门,其相对丰度为52.34%~69.08%,次优势菌是放线菌门(Actinobacteria),其相对丰度为21.31%~26.99%;而根际土壤中优势菌门则相反,为Actinobacteria,次优势菌门是Proteobacteria,其相对丰度分别为44.18%~44.60%和21.41%~36.22%,说明不同样品间群落组成相似但相对丰度差异较大。
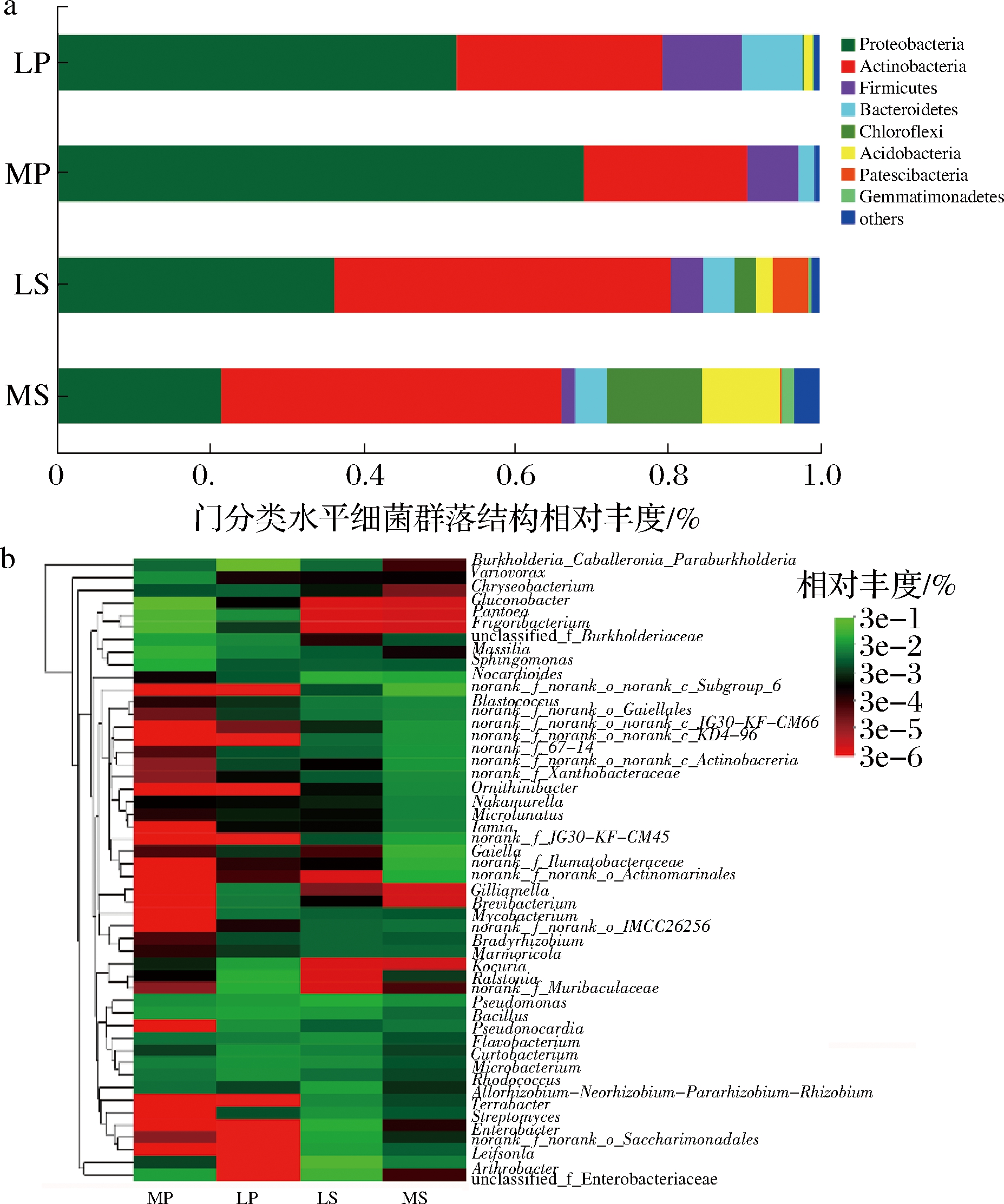
a-门水平;b-属水平
图1 不同样品中细菌在门水平和属水平的优势类群相对丰度
Fig.1 Relative abundance of dominant groups of bacteria
in different samples at phylum level and genus level
注:LP-林芝葡萄;MP-芒康葡萄;LS-林芝葡萄根际土壤;
MS-芒康葡萄根际土壤(下同);others为相对丰度低于0.5%
或未被分类的门或属
在属分类水平上,样品中细菌隶属713属,在相对丰度≥1.00%条件下,LP样品中有9个属,其优势属是伯克氏菌属(Burkholderia)(27.60%),其次是脆弱拟杆菌(Muribaculaceae)(5.34%),而MP样品中有10个属,葡萄糖杆菌属(Gluconobacter)(22.53%)是优势属,其次是冰冻小杆菌属(Frigoribacterium)(14.14%)和泛菌属(Pantoea)(13.52%);在相对丰度≥1.00%下, LS样品中有10个属,节杆菌属(Arthrobacter)(11.49%)是优势属,其次是肠杆菌属(Enterobacteriaceae)7.43%和类诺卡氏菌属(Nocardioides)5.15%,而MS样品中有12个属,其优势属是Gaiella(5.17%),其次是Llumatobacteraceae(4.43%)(图1-b)。Venn分析结果表明,根际土壤细菌种类最多(图2-a),各样品间共有64个属,不同种源地间葡萄样品共有133个属,LP特有属55个,MP特有属32个;不同种源地间土壤共有310个属,LS中有118个特有属,而MS中有98个特有属。LP和LS共有137个属,而MP与MS共有110个属(图2-b),说明不同种源葡萄和根际土壤细菌群落结构既存在共性又存在差异。
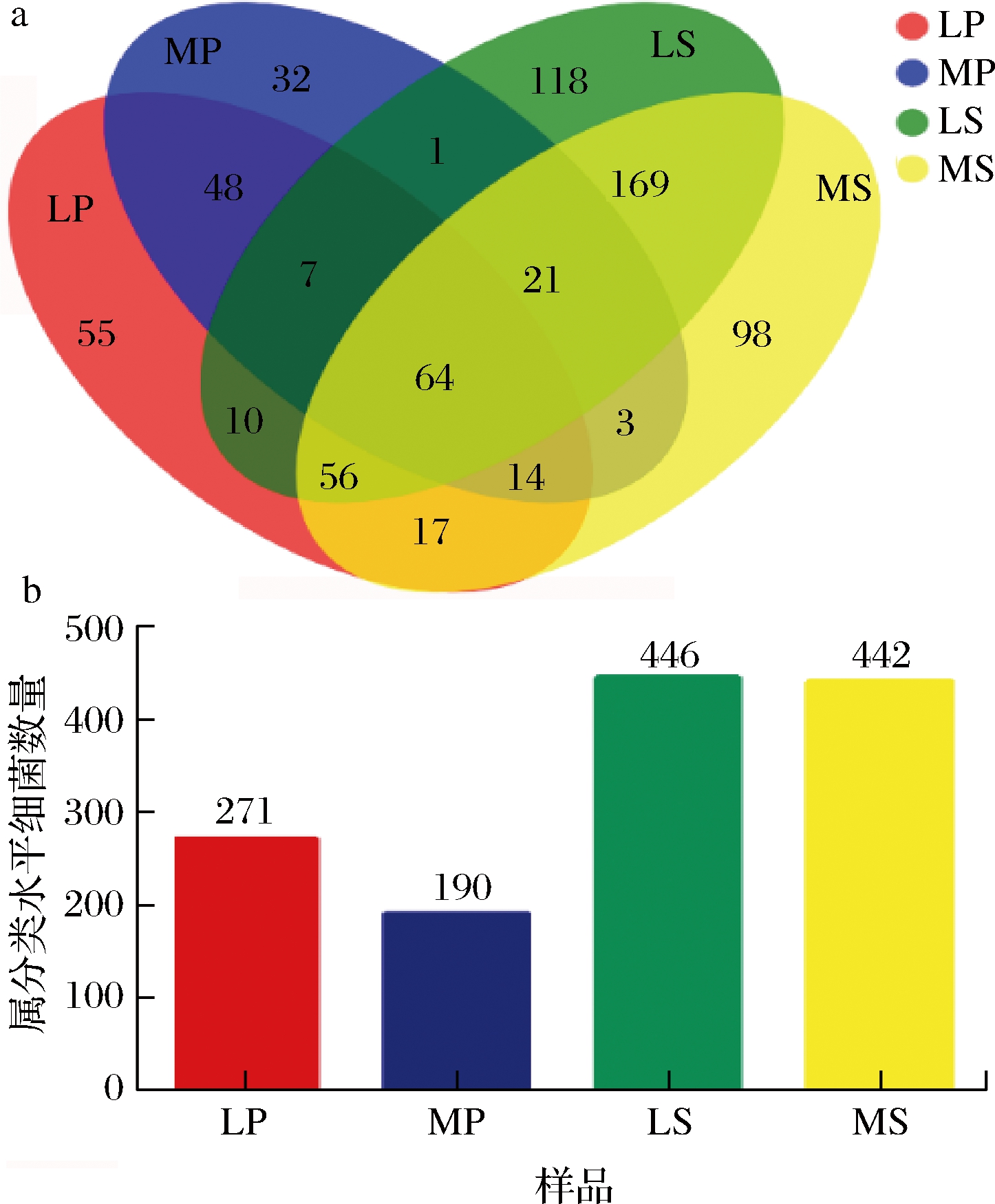
a-Venn图;b-细菌数量
图2 不同样品中细菌群落在属分类水平Venn图和数量
Fig.2 Veen diagrams and the number of bacteria in the different samples at genus level
2.4 不同样品中细菌多样性分析
为了分析不同样品中细菌群落结构特征,对不同样品进行了主坐标分析(principal coordinate analysis, PCoA)。如图3所示,PC1(41.62%)能够很好的区分葡萄和土壤样品,而不同种源地根际土壤样品更易被PC2(23.17%)区分,不同样品点之间距离有明显分离,说明不同样品中细菌群落结构存在差异。相比之下,LP和MP距离较近,说明细菌群落结构相似性较高,而LS和MS距离较远,说明相似性较低且组间差异大于组内差异。结合土壤理化性质分析结果来看,土壤营养成分的差异可能是导致根际土壤细菌群落结构差异的主要原因。
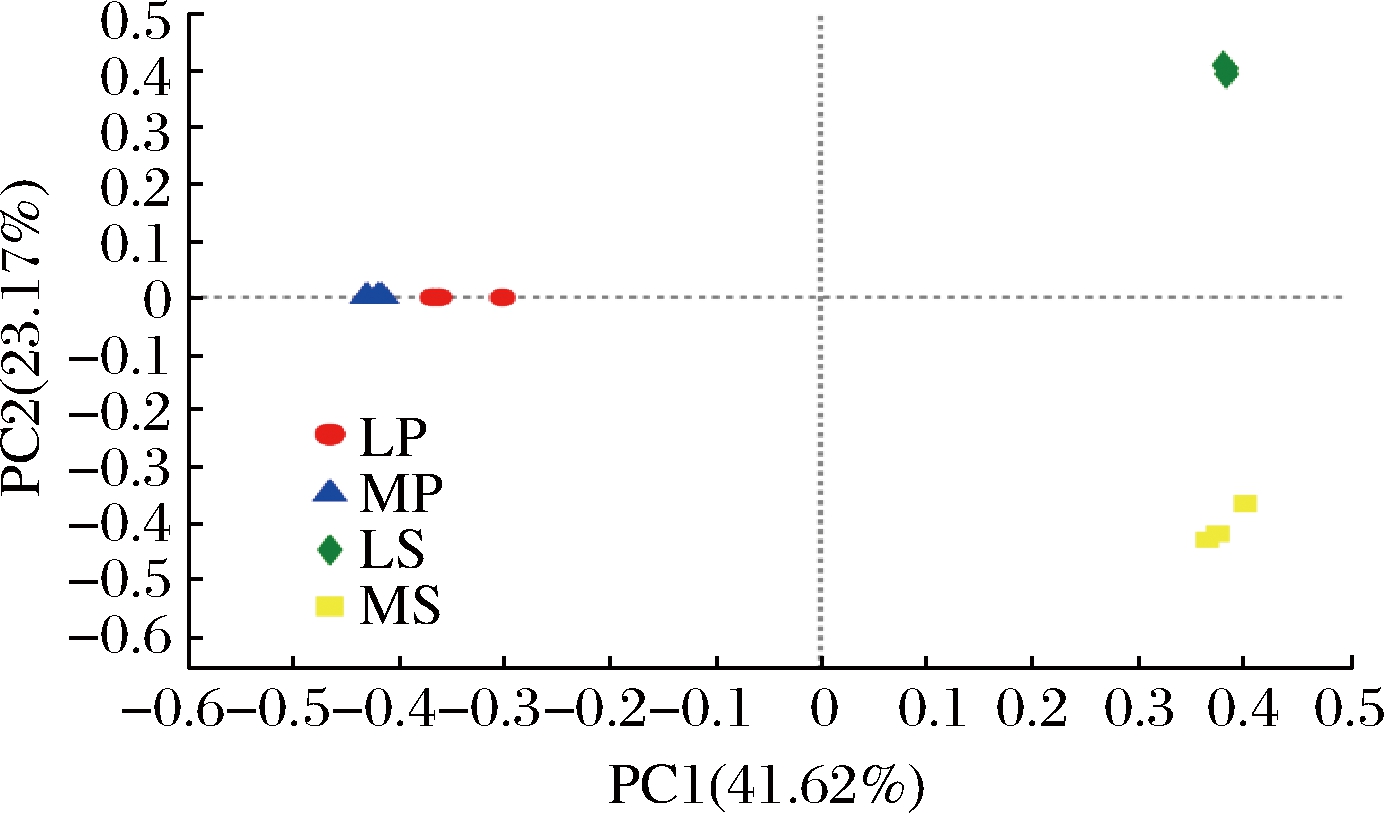
图3 细菌群落主坐标分析
Fig.3 Principal coordinate analysis of bacterial community
2.5 细菌群落结构与环境因子的冗余分析
为了分析环境因子对细菌群落结构的影响,对环境因子和不同样品进行了冗余分析(redundancy analysis,RDA)。结果如图4所示,TN、TP、TK,AN和pH对样品中细菌群落结构影响较大,而AP,EC和SOM对细菌群落结构影响不明显,其中TN含量对细菌群落结构影响最大,TK含量对细菌群落结构影响最小,MP、MS和LS与TN、TP、TK、AN和pH呈正相关,而LP呈负相关,说明氮、磷、钾和pH是影响葡萄和根际土壤细菌群落结构的关键因素。伯克氏菌科(Burkholderiaceae)、微球菌科(Micrococcaceae)细菌与TN、TP、TK、AN和pH呈负相关,而醋酸菌科(Acetobacteraceae)细菌相反,Enterobacteriaceae与pH呈负相关,而与其他土壤成分呈正相关,说明土壤营养成分含量决定了细菌群落的相对丰度。
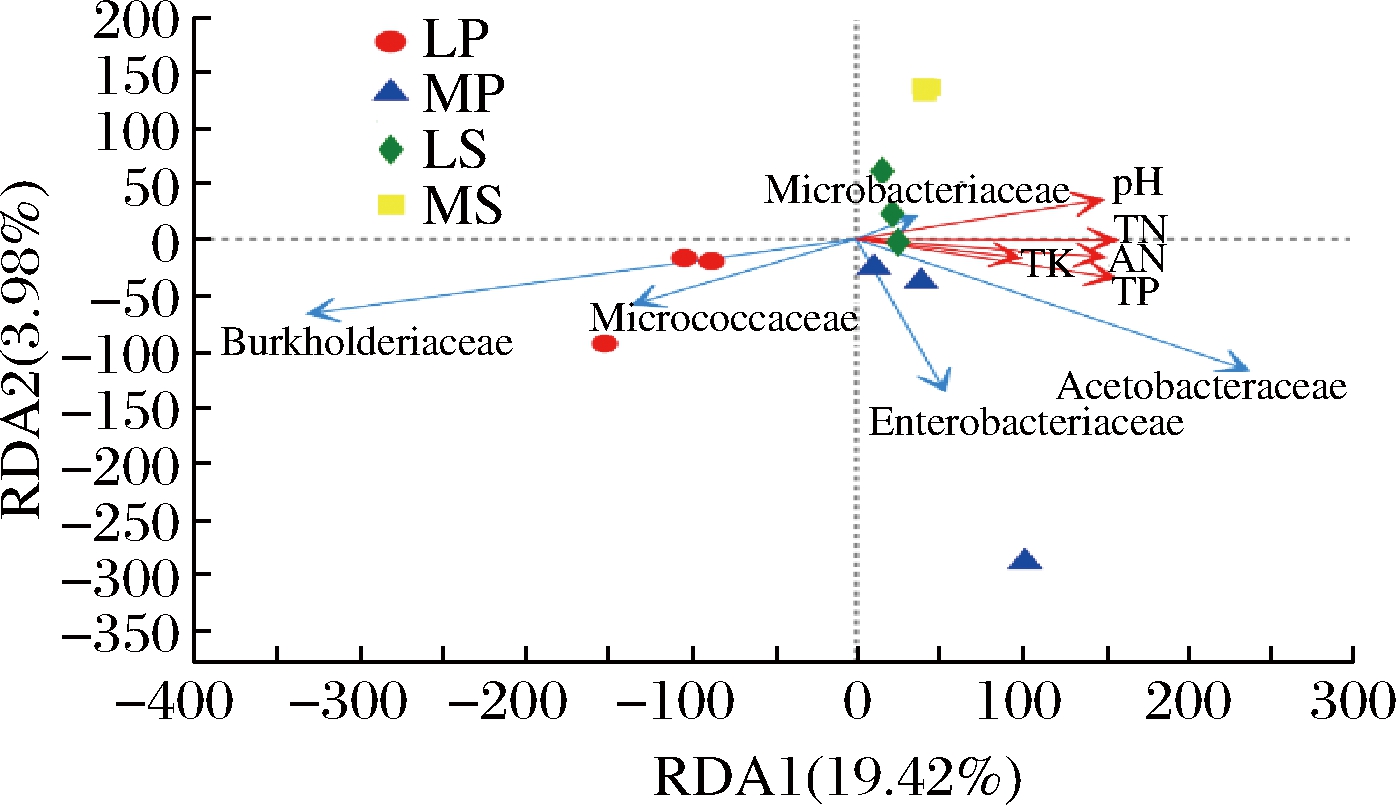
图4 细菌群落与环境因子间的冗余分析
Fig.4 Redundancy analysis between the bacterial community and environment factor
2.6 细菌群落功能预测分析
为了分析2个种源地葡萄和土壤细菌的功能组成变化,本文利用PICRUSt软件对细菌功能进行预测分析。结果如图5所示,共获得24项COG功能分组,其相对丰度随样品的不同而不同,主要参与氨基酸转运和代谢、转录、碳水化合物转运和代谢及能量生产和转换。KEGG(Kyoto Encyclopedia of Genes and Genomes)分析结果如表3所示,在第2等级功能上,样品细菌群落主要参与氨基酸代谢、碳水化合物代谢、能量代谢、脂类代谢和辅助因子、维生素代谢且葡萄表皮细菌相对丰度均高于根际土壤微生物,有研究表明,脂类物质的合成有助于改善葡萄酒的口感[7],

图5 细菌PICRUSt功能基因预测
Fig.5 PICRUSt functional gene prediction of bacterial community
表3 第2等级功能水平上细菌群落的KEGG代谢通路丰度
Table 3 KEGG metabolic pathway abundance of bacterial community at the pathway level 2
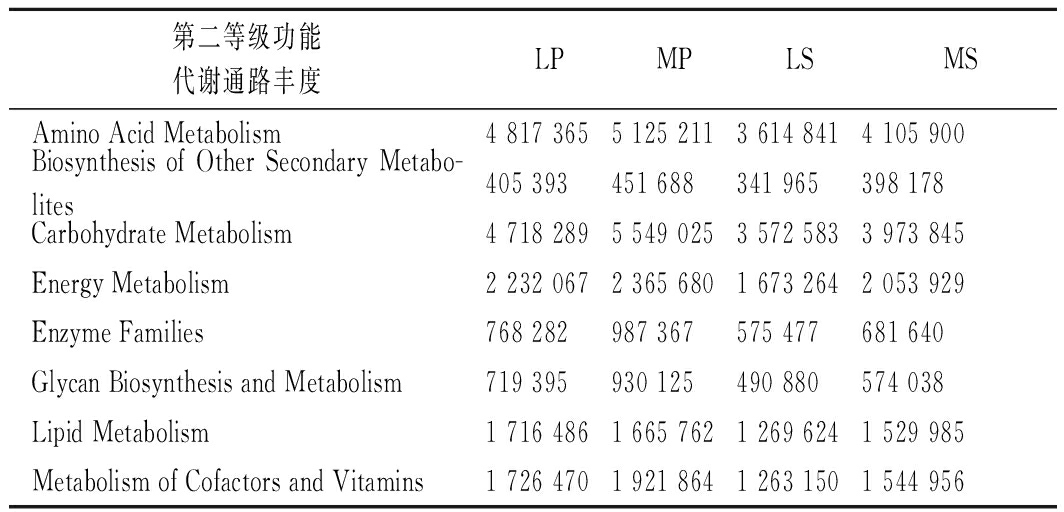
第二等级功能代谢通路丰度LPMPLSMSAmino Acid Metabolism4 817 3655 125 2113 614 8414 105 900Biosynthesis of Other Secondary Metabo-lites405 393451 688341 965398 178Carbohydrate Metabolism4 718 2895 549 0253 572 5833 973 845Energy Metabolism2 232 0672 365 6801 673 2642 053 929Enzyme Families768 282987 367575 477681 640Glycan Biosynthesis and Metabolism719 395930 125490 880574 038Lipid Metabolism1 716 4861 665 7621 269 6241 529 985Metabolism of Cofactors and Vitamins1 726 4701 921 8641 263 1501 544 956
在本文中也发现具有较高相对丰度的细菌参与脂类代谢,说明葡萄表皮微生物在葡萄酒酿造过程中扮演着重要角色。同时还发现部分细菌参与抗生素和植物激素类物质的生物合成,说明其可能参与植物生长和病害防治。
3 讨论
酵母菌和细菌在葡萄酒发酵过程中扮演者重要角色,其主要来源于葡萄表面和种植土壤[4-5],因此为提高葡萄酒品质,分离本地酿酒微生物成为人们研究的热点。本研究利用高通量测序技术对藏东南2个种源地葡萄和土壤细菌群落结构和多样性进行了分析。土壤作为一个天然的微生物资源库,为微生物提供了必需的营养物质(如碳源、氮源和生长因子),致使其微生物种类复杂多样[6,15]。魏玉洁等[8]研究表明无论是细菌还是真菌,土壤中微生物种类和数量最多,在本研究中得到了类似的实验结果,2个种源地根际土壤中所获得的OTUs最多,同时其多样性指数、均匀度指数和丰富度指数最高且MS大于LS。本研究发现根际土壤细菌的优势门是变形菌门和放线菌门,研究表明,土壤微生物在门分类水平上优势细菌门基本相似,主要包括10种左右的细菌类群[16],如变形菌门、放线菌门和酸杆菌门,杨敏等[17]通过研究香格里拉葡萄酒产区葡萄根际微生物时发现根际土壤的优势门是变形菌门、放线菌门和酸杆菌门,而本研究发现酸杆菌门(Acidobacteria)在MS(10.28%)中的相对丰度显著高于LS(2.22%),这可能与MS中含有较高的氮源有关[18];张世伟[1]在研究不同品种酿酒葡萄表皮微生物时发现变形菌门和放线菌门是葡萄表皮的优势细菌门,这与本研究结果一致。
HIRANO等[19]和PARK等[20]研究表明,凯氏杆菌(Kaistobacter)、Arthrobacter和鞘氨醇单胞菌 (Sphingomonas)是土壤中的优势类群,而杨敏等[17]研究表明,香格里拉葡萄园根际土壤的优势细菌属是红游动菌属(Rhodoplanes)、拟杆菌属(Bacteroides)、硝化螺旋菌门(Nitrospira)和类固醇杆菌(Steroidobacter),魏玉洁等[8]研究发现吐哈盆地葡萄园土壤中优势属是Kaistobacter、Arthrobacter和 克氏菌(Skermanella);上述研究结果表明,不同地区根际土壤细菌优势类群存在明显的差异,可能与地理环境和土壤理化性质差异有关[8-10]。张世伟[1]研究不同品种葡萄表皮微生物群落结构时发现,欧文氏菌属(Erwinia)、假单胞菌(Pseudomonas)和芽孢杆菌(Bacillus)是优势属,而新疆葡萄表皮细菌的优势属以Pseudomonas、Sphingomonas和杆菌属(Adhaeribacter)为主[8],但本研究发现LP样品中的优势属是Burkholderia 和Muribaculaceae,而MP样品中的Gluconobacter,Frigoribacterium和Pantoea是优势属,这与前人研究结果差异较大,藏东南独特的地理环境和气候类型可能是造成葡萄表皮微生物类群差异的关键因素。
根际土壤细菌以放线菌门和变形菌门为主,其中变形菌门具有促进土壤氮循环和植物生长的功能[3],研究结果表明根际土壤中含有较多的Nocardioides,Enterobacteriaceae和少量的中生根瘤菌(Mesorhizobium),有研究表明Nocardioides具有抑制病原菌和降解有毒物质的作用[21],Enterobacteriaceae具有抑制黄曲霉生长和减缓黄曲霉毒素的作用[22],而Mesorhizobium具有固氮和促进植物生长的作用[23],说明LS和MS根际土壤状态良好。参与葡萄酒酿造过程中的细菌主要是乳酸菌,如乳杆菌、片球菌、酒类酒球菌和植物乳杆菌等[24-25],本文在葡萄表皮检测到了一定数量的乳杆菌(Lactobacillus)、乳球菌(Lactococcus)、Leuconosto和Weissella,其中Lactobacillus和Lactococcus是决定发酵成功的关键,具有产酸、细菌素、维生素和香气物质的功能,这些物质直接影响了发酵食品的营养成分和口感[26],而Leuconosto和Weissella在异乳酸发酵过程中产生醇类等风味物质[27-28]。本研究还发现了厚壁菌门(Firmicutes),Firmicutes具有二次发酵,提升葡萄酒品质的功能[29]。以上结果表明葡萄表皮含有丰富的酿酒微生物且对葡萄酒的品质和口感至关重要。
综上所述,本文首次通过高通量测序技术对林芝、芒康两地葡萄及根际土壤细菌群落结构及多样性进行了分析。发现土壤细菌种类和数量最多,不同种源地葡萄和土壤中细菌群落结构相似,同时还发现葡萄表皮存在较多与酿酒相关的细菌。本文对西藏葡萄和土壤细菌有了深入的了解,为西藏设施葡萄种植、酿酒细菌的筛选和利用提供了理论依据。
[1] 张世伟. 酿酒葡萄微生物群落多样性及其氮代谢通量研究[D].北京:中国矿业大学(北京), 2018.
ZHANG S W.Study on microbial community diversity and the nitrogen metabolism flux of wine grapes[D].Beijing:China University of Mining & Technology (Beijing), 2018.
[2] ALEKLETT K, LEFF J W, FIERER N, et al.Wild plant species growing closely connected in a subalpine meadow host distinct root-associated bacterial communities[J].Peer J, 2015, 3(8):e804.
[3] CHAUDHRY V, REHMAN A, MISHRA A, et al.Changes in bacterial community structure of agricultural land due to long-term organic and chemical amendments[J].Microbial Ecology, 2012, 64(2):450-460.
[4] RENOUF V, CLAISSE O, LONVAUD-FUNEL A.Numeration, identification and understanding of microbial biofilm on grape berry surface[J].Australian Journal of Grape and Wine Research, 2005, 11:316-327.
[5] OC N E, GUTIÉRREZ A R, GARIJO P, et al.Presence of non-Saccharomyces yeasts in cellar equipment and grape juice during harvest time[J].Food Microbiology, 2010, 27(8):1 023-1 027.
N E, GUTIÉRREZ A R, GARIJO P, et al.Presence of non-Saccharomyces yeasts in cellar equipment and grape juice during harvest time[J].Food Microbiology, 2010, 27(8):1 023-1 027.
[6] MARTINS G, LAUGA B, MIOT-SERTIER C, et al.Characterization of epiphytic bacterial communities from grapes, leaves, bark and soil of grapevine plants grown, and their relations[J].PLoS One, 2013, 8(8):e73 013.
[7] BOKULICH N A, THORNGATE J H, RICHARDSON P M, et al.Microbial biogeography of wine grapes is conditioned by cultivar, vintage, and climate[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(1):e139-e148.
[8] 魏玉洁, 邹弯, 马文瑞, 等.应用高通量测序技术研究新疆产区葡萄果实、叶片及果园土壤微生物多样性[J].食品科学, 2018, 39(6):162-170.
WEI Y J, ZOU W, MA W R, et al.Microbial diversity of berries, leaves and soil of grapevine plants grown in Xinjiang analyzed by high-throughput sequencing[J].Food Science, 2018, 39(6):162-170.
[9] RIBÉREAU-GAYON P, DUBOURDIEU D, DON CHE B.Handbook of Enology:The Microbiology of Wine and Vinifications[M].New York:John Wiley and Sons, 2006:18-37.
CHE B.Handbook of Enology:The Microbiology of Wine and Vinifications[M].New York:John Wiley and Sons, 2006:18-37.
[10] 王伟, 布丽根·加冷别克, 胡晓东, 等.基于高通量测序技术的酿酒葡萄产区土壤微生物多样性[J].新疆农业科学, 2020, 57(5):859-868.
WANG W, BULIGEN J L B K, HU X D, et al.Analysis of the microbial community diversity of soil from wine grape producting area in Xinjiang based on high-throughput sequencing[J].Xinjiang Agricultural Sciences, 2020, 57(5):859-868.
[11] WU L Y, WEN C Q, QIN Y J, et al.Phasing amplicon sequencing on Illumina MiSeq for robust environmental microbial community analysis[J].BMC Microbiology, 2015, 15(1):125.
[12] YOU J, WU G, REN F P, et al.Microbial community dynamics in Baolige oilfield during MEOR treatment, revealed by Illumina MiSeq sequencing[J].Applied Microbiology & Biotechnology, 2016, 100(3):1 469-1 478.
[13] 鲍士旦. 土壤农化分析[M].第三版.北京:中国农业出版社, 2000.
BAO S D.Soil and Agricultural Chemistry Analysis[M].3rd ed.Beijing:China Agricultural Press, 2000.
[14] GREGORY CAPORASO J, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[15] WAWRIK B, KERKHOF L, KUKOR J, et al.Effect of different carbon sources on community composition of bacterial enrichments from soil[J].Appl Environ Microbiol, 2005, 71(11):6 776-6 783.
[16] PHILIPPOT L, ANDERSSON S G E, BATTIN T J, et al.The ecological coherence of high bacterial taxonomic ranks[J].Nature Reviews Microbiology, 2010, 8(7):523-529.
[17] 杨敏, 殷绒, 张国涛, 等.基于高通量测序技术的香格里拉葡萄酒产区根际微生物多样性研究[J].云南农业大学学报(自然科学), 2020, 35(3):392-400.
YANG M, YIN R, ZHANG G T, et al.Study on the grape rhizosphere microbial diversity in Shangri-La wine region by high-throughput sequencing technology[J].Journal of Yunnan Agricultural University (Natural Science), 2020, 35(3):392-400.
[18] FIERER N, BRADFORD M A, JACKSON R B.Toward an ecological classification of soil bacteria[J].Ecology, 2007, 88(6):1 354-1 364.
[19] HIRANO S S, UPPER C D.Bacteria in the leaf ecosystem with emphasis on Pseudomonas syringae-a pathogen, ice nucleus, and epiphyte[J].Microbiology and Molecular Biology Reviews, 2000, 64(3):624-653.
[20] PARK J Y, HAN S H, LEE J H, et al.Draft genome sequence of the biocontrol bacterium Pseudomonas putida B001, an oligotrophic bacterium that induces systemic resistance to plant diseases[J].Journal of Bacteriology, 2011, 193(23):6 795-6 796.
[21] 雒晓芳, 陈俊楠, 田丹妮, 等.白色类诺卡氏菌的分离鉴定及其抗菌活性初探[J].中国酿造, 2015, 34(10):58-61.
LUO X F, CHEN J N, TIAN D N, et al.Separation and identification of Nocardioides albus and preliminary research on its antibacterial activity[J].China Brewing, 2015, 34(10):58-61.
[22] GONG A D, DONG F Y, HU M J, et al.Antifungal activity of volatile emitted from Enterobacter asburiae Vt-7 against Aspergillus flavus and aflatoxins in peanuts during storage[J].Food Control, 2019, 106:106 718.
[23] 姜怡, 杨颖, 陈华红, 等.植物内生菌资源[J].微生物学通报, 2005(6):146-147.
JIANG Y, YANG Y, CHEN H H, et al.Plant endophyte resources[J].Microbiology China, 2005(6):146-147.
[24] MAICAS S.The use of alternative technologies to develop malolactic fermentation in wine[J].Applied Microbiology and Biotechnology, 2001, 56(1-2):35-39.
[25] CLAUDIO D, FORMICA J V.Wine Microbiology[M].Taylor and Francis, 2012-06-15.
[26] ZABAT M A,SANO W H,WURSTER J I, et al.Microbial community analysis of sauerkraut fermentation reveals a stable and rapidly established community[J].Foods, 2018, 7(5):77.
[27] RHEE S J, LEE J E, LEE C H.Importance of lactic acid bacteria in Asian fermented foods[J].Microbial Cell Factories, 2011, 10(S1):5.
[28] KIM M J, LEE H W, LEE M E, et al.Mixed starter of Lactococcus lactis and Leuconostoc citreum for extending kimchi shelf-life[J].Journal of Microbiology, 2019, 57(6):479-484.
[29] 周利国. 宁夏银川葡萄酒产区酒酒球菌分离筛选及鉴定[D].杨凌:西北农林科技大学, 2009.
ZHOU L G.The isolation and identification of Oenococcus oeni from Ningxia Yinchuan wine production region[D].Yangling:Northwest A&F University, 2009.