魏斯氏菌(Weissella)是乳酸菌的一种,在酱油、泡菜、豆豉和香肠等发酵食品中广泛存在[1],不仅能够提高发酵食品产品质量或者缩短发酵周期还可以赋予食品独特的风味[2],并且具有合成细菌素[3]、产胞外多糖[4]、降胆固醇[5]、降甘油三酯[6]和抑制炎症水平[7]等益生特性。
转录组测序(RNA-Seq)技术通过对细胞转录产物进行测序,统计测得细胞在某一特定时间特定条件下每个转录本的表达量[8],有助于精准评估细胞表型和对目标基因进行改良[9],有低成本、高通量、准确、快速的特点[10]。通过转录组测序,比较菌体在不同环境、不同刺激物作用下基因表达量的差异,并对其进行注释和富集分析,可以研究环境及刺激物对菌体的影响及作用机理[11]。近年来,随着RNA-Seq技术和生物信息学分析方法的不断成熟,转录组测序因其检测动态范围宽、数据重复性好而被广泛应用于乳酸菌研究领域,逐渐成为基因表达差异研究、功能注释和代谢机制分析的重要手段,为深入理解微生物蕴含的分子机制提供了可信的证据[11-15]。陈兴麟等[16]通过转录组测序发现新琼四糖和新琼六糖在促进乳酸菌生长的作用机理上具有极大的相似性。田源等[17]利用宏转录组揭示了在浓香型白酒窖内发酵过程中与正丙醇合成有关的微生物与代谢途径。目前对魏斯氏菌转录组方面的研究仍然比较匮乏。本实验通过转录组测序技术分析比对了魏斯氏菌10d-17在饥饿及正常状态下RNA转录表达情况,筛选差异基因,对差异基因进行GO(Gene Ontology)注释和代谢路径KEGG(Kyoto Encyclopedia of Genes and Genomes)注释,从而阐明在关键代谢路径中基因转录水平的变化,探究关键基因和蛋白参与代谢模式的调控及其调控方式。
1 材料与方法
1.1 供试菌株
魏斯氏菌(Weissella)由本课题组从前期实验的发酵剁辣椒中分离得到。
1.2 试剂盒及仪器
Qubitarna分析试剂盒,美国加利福尼亚州生命技术公司;RNA提取试剂盒,Clontech(USA);RNA Nano6000检测试剂盒、安捷伦生物分析仪2100,安捷伦技术公司(美国);RNA保护剂,ZYMO RESEARCH(USA);Qubit®RNA分析试剂盒,IMPLEN(USA);Qubi 2.0荧光计,Invitrogen(USA)。
1.3 实验方法
1.3.1 菌种的活化
将保藏的菌种从-80 ℃冰箱取出,于4 ℃解冻30 min,吸取适量菌液于MRS液体培养基中于恒温培养箱中37 ℃培养12 h,然后划线分离,挑取单菌落液体培养,如此反复操作3次,得到活化后的种子液。
1.3.2 样品的制备
饥饿处理组:按1.3.1方法活化后,参考ERCAN等[18]研究方法,取10 mL菌液,于4 ℃ 8 000 r/min离心后去上清液,0.9%生理盐水洗脱2次,离心后取菌泥静置菌2 h。后加入RNA保护剂(保护剂添加比例为1∶1),4 ℃ 8 000 r/min离心后去上清液,迅速放入液氮中冷冻(后续测序结果中以J-菌种编号标记)。
正常培养组:菌种按1.3.1的方法活化后,饥饿处理2 h后,按1%接种量接入10 mL已杀菌培养基中,培养8 h。后4 ℃ 8 000 r/min离心取菌泥,添加RNA保护剂,离心后去上清液,放入液氮中冷冻保藏(后续测序结果中以Z-菌种编号标记)。每1株菌株每1种处理做3份平行。
1.3.3 RNA的质量检测
使用1%的琼脂糖凝胶检测提取的RNA样品是否降解或被污染,然后用Nano光度计®分光光度计检查RNA纯度,Qubit®2.0荧光计测量RNA浓度[19]。质量检测最后采用安捷伦生物分析仪评估RNA完整性。
1.3.4 建立测序文库及测序
RNA的质量检测后,使用Ribo-zero试剂盒和fragmentation buffer去除rRNA,使被打断成短片段的mRNA富集。以mRNA为模板,在反转录酶试剂盒作用下,用六碱基随机引物合成第1条cDNA链。利用Qia Quick PCR试剂盒和缓冲液合成纯化第2条cDNA链,并加EB缓冲液洗后进行末端修复,添加poly A连接测序接头,用AMPure XP beads进行片段大小选择,进行PCR扩增,得到最终的测序,并利用Ilumina hiseq 2000平台对样品进行高通量测序。然后去除原始数据中不合格的Reads,得到高质量的Clean Reads,并利用Bowtie2软件将高质量的Clean Reads进行基因比对分析。通过HT seq将比对基因组的Reads进一步定位到基因上,统计每个基因比对的Reads数,通过FPKM值表示基因的表达量,并对基因结构和表达水平进行分析。在有生物学重复的条件下,以P<0.05作为阈值筛选差异基因,并对其进行GO富集分析及KEGG富集处理。
2 结果与分析
2.1 样品RNA质量检测结果
魏斯氏菌做饥饿和全营养2种处理,每组3个平行,共6个样本。由表1可知,每个样品RNA总量均≥6 g(3 g为合格),质量浓度≥200 g/L。同时OD260/280均>1.5,RNA完整值(RNA integrity number,RIN)≥8(≥6为合格),且total RNA条带清晰,说明RNA质量符合后续测序要求。
表1 样品RNA质量情况
Table 1 RNA quality of samples

样品名称质量浓度/(g·L-1)体积/L总量/gOD260/280OD260/230RINJ10d-17-129432.009.4082.231.799.8J10d-17-233232.0010.6242.161.409.8J10d-17-333432.0010.6882.232.179.6Z10d-17-135432.0011.3282.272.069.8Z10d-17-223732.007.5842.251.849.5Z10d-17-320832.006.6562.420.628.7
2.2 RNA-seq测序质量结果
由表2可知,魏斯氏菌做饥饿和全营养2种处理,每组3个平行,每1组高通量测序获得的原始测序数据量都在1.7 GB以上,单个碱基位置的测序错误率为0.02%,其中Q20(Phred数值大于20的碱基占总体碱基的百分比)均在98.36%以上,质量值在Q30也都高于94.65%。去除含有带接头的、低质量的序列后,各组得到的高质量reads数为983万~1 359万条。从RNA-seq测序质量结果来看,本次测序的数据饱和度高,符合开展后续转录本拼接实验的要求。
表2 不同样品RNA-seq数据产出质量情况表
Table 2 Output quality of RNA-seq data of different samples
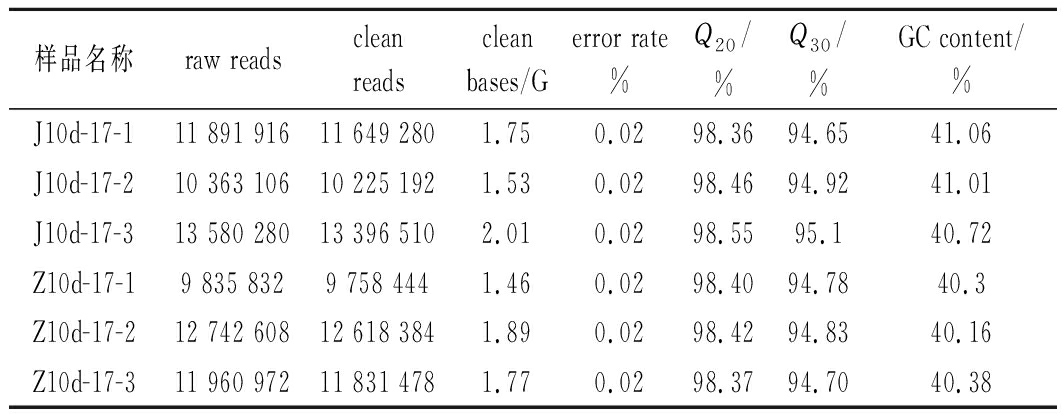
样品名称raw readsclean readsclean bases/Gerror rate%Q20/%Q30/%GC content/%J10d-17-111 891 91611 649 2801.750.0298.3694.6541.06J10d-17-210 363 10610 225 1921.530.0298.4694.9241.01J10d-17-313 580 28013 396 5102.010.0298.5595.140.72Z10d-17-19 835 8329 758 4441.460.0298.4094.7840.3Z10d-17-212 742 60812 618 3841.890.0298.4294.8340.16Z10d-17-311 960 97211 831 4781.770.0298.3794.7040.38
注:raw reads-统计原始序列数据,以4行为1个单位,统计每个文件的测序序列的个数;clean reads-过滤后的测序数据;clean bases-Clean reads的个数乘以长度,并转化为以G为单位;Q20、Q30-分别计算 Phred 数值大于20、30的碱基占总体碱基的百分比;GC content-计算碱基G和C的数量总和占总的碱基数量的百分比
2.3 魏斯氏菌10d-17 RNA-seq相关性检查
转录组测序结果要求生物学实验操作是可重复的,并且变异不大。同时为确保后续的差异性基因分析结果的准确性,可以通过绘制皮尔逊相关系数图,来检验样品间的基因表达水平的相关性。由图1可知,各个样品间生物学重复皮尔逊相关系数均≥0.97,实验重复性好。

图1 样品皮尔逊相关系数图
Fig.1 Pearson correlation coefficient of sample
2.4 魏斯氏菌转录结果分析
2.4.1 组间差异表达基因分析
为了使不同的基因和实验之间估计的基因表达水平有可比性,本实验除了用reads计数来估计基因的表达水平外还加入了FPKM(fragments per kilobase of exon model per million mapped fragments)的概念,用FPKM值是否≥1作为判断基因表达的标准。这株魏斯氏菌注释到结构中73%以上的基因的FPKM值>1[20]。本实验为了揭示饥饿处理对魏斯氏菌的基因表达的影响,通过DE seq软件对read count标准化,再根据负二项分布P模型计算得出假设检验概率(P),最后进行多重假设检验校正,得到错误发现率(false discovery rate,FDR)值,以P<0.05作为筛选的差异表达基因的标准,若差异基因log2fold change>0,则该差异基因是上调,反之则下调。
本实验一共筛选得到1 018个显著性差异基因,509个显著表达上调差异基因,509个显著表达下调基因。因此,饥饿胁迫可引起魏斯氏菌代谢过程中基因的显著变化。了解魏斯氏菌在饥饿胁迫下的转录组差异,对更好的利用乳酸菌具有实际的指导意义。
2.4.2 魏斯氏菌10d-17差异基因GO富集
使用软件GO seq对差异基因进行GO基因功能国际标准分类体系注释后,将基因和蛋白质功能进行限定和描述分为分子功能、生物过程和细胞组成3个部分。本研究挑选了富集最显著的30个GO term,得到了饥饿处理后与正常状态下的差异基因GO富集柱状图。如图2所示,最显著的30个GO term中,有14个term与RNA的活动相关,并且没有term涉及细胞组成。由此可见,魏斯氏菌的生命活动与RNA的活动息息相关,主要通过调整与转录、翻译相关基因的表达来调控蛋白,从而提高环境适应性,而营养限制对魏斯氏菌细胞组成方面的影响不大。
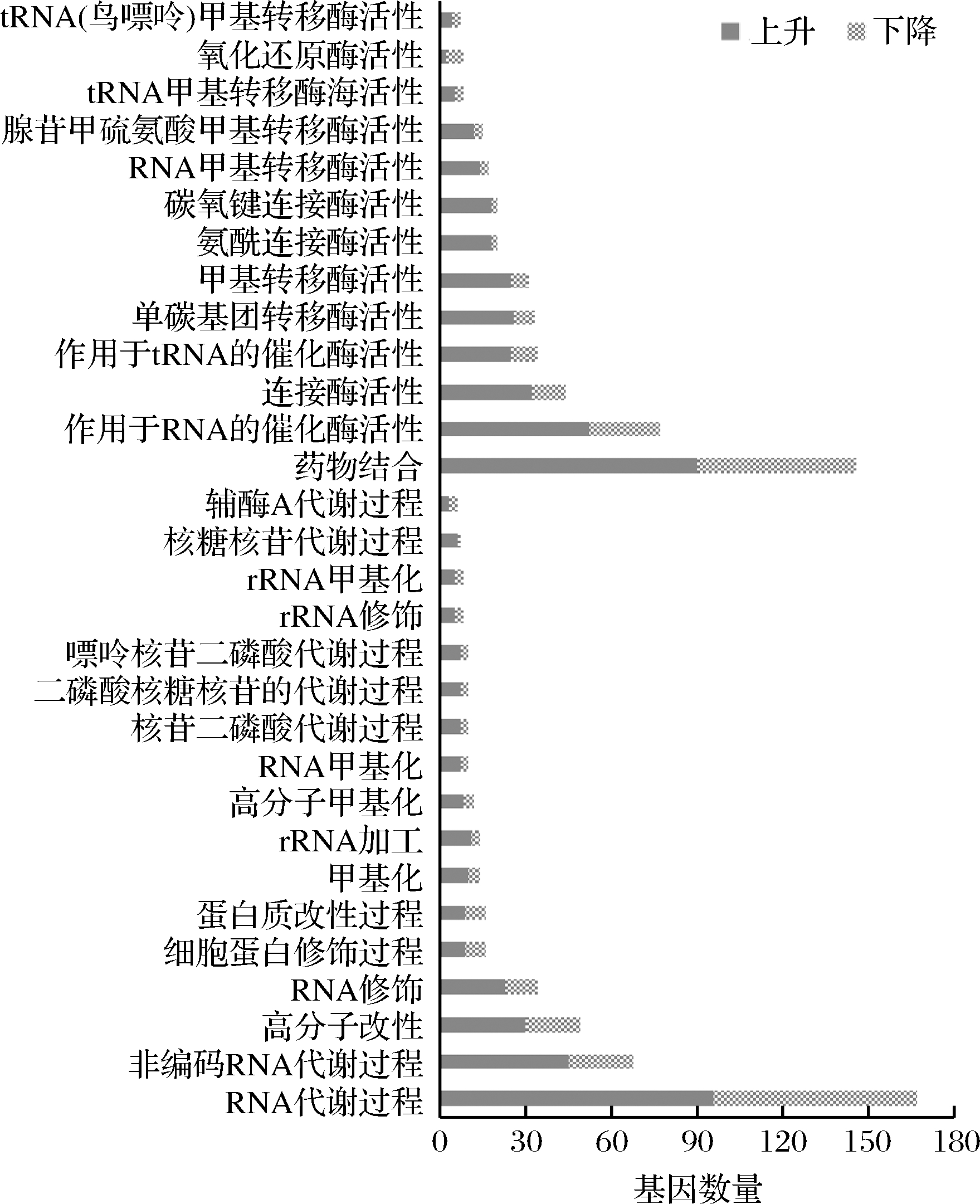
图2 GO富集柱状图
Fig.2 Histogram of go enrichment
GO功能注释分析结果显示,在筛选得到的1 018 个显著性差异基因中,有772个基因获得GO功能注释信息,其中上调基因有380个,下调基因有392个。有667个基因至少能关联到MF的1个GO功能条目,有309个基因至少能关联到CC的1个GO功能条目,以及639个基因至少能关联到BP的1个GO功能条目。生物过程二级分类中基因数目最多的类别是RNA代谢过程,该分类包括了167个差异表达基因,其中96个上调基因,61个下调基因,生物过程二级分类中基因数目最多的类别是药物结合,包含了146个差异表达基因,其中90个上调基因,56个下调基因。富集于药物结合的差异基因大多数与ATP结合盒式蛋白(ATP-binding cassette,ABC)转运系统和氨酰-tRNA的合成有关,RNA代谢过程的差异基因与氨酰-tRNA的合成有关。ABC转运是实现物质跨膜运输的重要途径,能够将发酵液中的无机离子、单糖等物质运输至细胞内。全营养状态下,细胞生命活动活跃,物质转运加快。氨酰tRNA是一种与之对应的氨基酸相结合的tRNA(也被称作“转移核糖核酸”)。它能够将氨基酸传递到核糖体中,并且把氨基酸加入正在延伸中的多肽链。氨酰-tRNA的合成收到氨酰-tRNA合成酶的调控,每1个氨酰-tRNA合成酶可识别一个特定的氨基酸与此氨基酸对应的tRNA的特定部位。与组氨酸、颉氨酸、苏氨酸等16种氨基酸合成相应氨酰-tRNA的酶的调控基因均表达上调。这可能是因为在全营养状态下,魏斯氏菌生长和繁殖速度加快,需要更多的蛋白质来支持它生长,而该菌可以通过调控氨酰-tRNA合成酶的表达来加速蛋白质的合成。
2.4.3 KEGG通路富集分析
KEGG是系统分析基因功能、基因组信息的数据库,通过KEGG通路富集和注释能确定差异表达基因参与的最主要生化代谢途径和信号转导途径[21]。魏斯氏菌共有290个差异基因被注释到了43条通路,上调基因158个,下调基因132个。其中富集差异基因数量较多的通路为:生物化学代谢富集了132个差异基因,次生代谢物的生物合成富集了51个差异基因,微生物在不同环境中的代谢富集了40个差异基因,ABC转运蛋白富集了31个差异基因。
2.4.4 魏斯氏菌10d-17糖酵解和丙酮酸代谢途径差异基因表达分析
如表3所示,通过对2种不同状态魏斯氏菌差异基因的分析发现,差异倍数大的基因集中于糖代谢和丙酮酸代谢途径中,故选取这2个代谢途径绘制通路图进行分析。如图3所示,糖酵解通路共注释到20个差异基因,其中5个显著性差异基因。葡萄糖发生酵解的第1步就是葡萄糖分子在己糖激酶和镁离子的作用下发生磷酸化生成葡萄糖-6-磷酸。该菌的葡萄糖-6-磷酸主要有2种去向:(1)是进入磷酸戊糖途径,磷酸戊糖途径中的相关酶的表达呈现上调趋势,从而促进PRPP的生成,为嘌呤核苷酸的合成提供重要的中间产物;(2)是在葡萄糖激酶和葡萄糖磷酸异构酶生成果糖-6-磷酸,葡萄糖激酶和葡萄糖磷酸异构酶编码基因有上调趋势,可能是因为底物浓度增加以后,高浓度的果糖-6-磷酸加速了果糖-2,6-二磷酸的合成,该化合物又进一步激活了果糖磷酸激酶的活性。果糖-1,6-二磷酸生成三磷酸甘油醛及后续代谢途径相关编码基因表达均下调或无明显差异。
表3 魏斯氏菌10d-17丙酮酸代谢显著差异基因表
Table 3 Gene table of phosphotransferase system difference of Weissella 10d-17

酶编号名称基因IDlog2fold change(Z-10d-17vsJ-10d-17)padj(Z-10d-17vsJ-10d-17)EC2.7.1.2葡萄糖激酶TY24_RS022951.447 54.81E-66TY24_RS04475-2.880 91.654 8E-55EC5.3.1.1磷酸丙糖异构酶TY24_RS00735-3.058 21.661 7E-23EC5.4.2.12磷酸甘油酸变位酶TY24_RS060151.882 92.056 1E-15TY24_RS07015-2.455 05.011 7E-49EC1.1.1.28D-乳酸脱氢酶TY24_RS02350-2.177 58.599 2E-23EC1.2.4.1丙酮酸脱氢酶组分E1TY24_RS02990-3.924 89.062 3E-44TY24_RS02995-3.570 04.81E-66EC1.8.1.4二氢脂酰胺脱氢酶TY24_RS03005-2.908 91.497 5E-70EC2.3.1.12二氢脂酰胺S-乙酰转移酶TY24_RS03000-3.168 02.389 7E-61EC1.2.3.3丙酮酸氧化酶TY24_RS02455-3.100 32.799 1E-24EC1.1.1.38苹果酸酶TY24_RS04845-1.972 94.064 8E-40TY24_RS037651.207 20.004 270 8

图3 魏斯氏菌10d-17糖酵解代谢通路图
Fig.3 Pathway map of glycolysis metabolism of Weissella 10d-17
丙酮酸代谢通路共注释到13个差异基因,其中8个显著性差异基因,丙酮酸核心代谢通路相关基因表达量均显著下调,差异倍数为2~16。该菌的丙酮酸主要有3个去向:(1)在苹果酸脱氢酶的作用下合成苹果酸;(2)在乳酸脱氢酶的作用下合成乳酸;(3)变为乙酰辅酶A进入丁酸代谢。但是该菌未注释到合成苹果酸和合成乳酸的后续相关基因,苹果酸脱氢酶和乳酸脱氢酶的相关基因表达量差异不显著。丙酮酸脱氢酶的编码基因变化最为显著,在饥饿条件下,分别上调了15.1、11.9、7.5倍,说明丙酮酸脱氢酶可能是该魏斯氏菌能量代谢中的关键酶,且在饥饿状态具有对抗胁迫意义。丙酮酸脱氢酶(pyruvate dehydrogenase,E1)编码基因分别下调了15.1、11.9、7.5倍。可能还原型辅酶Ⅰ(nicotinamide adenine dinucleotide,NADH)和乙酰-辅酶A这2种产物的累积使二氢硫锌酰转乙酰基酶(dihydrolipoyl transacetylase,E2)停留在与乙酰基结合的形式,不能接受在E1酶上与焦磷酸硫胺素(thiamine pyrophosphate,TPP)结合的乙酰基,从而抑制了丙酮酸脱羧作用。
3 结论与讨论
本研究为揭示饥饿胁迫对魏斯氏菌代谢的影响,对魏斯氏菌的转录组进行测序和分析。结果显示,每一组高通量测序获得的原始测序数据量都在1.7 GB以上,其中Q20均在98.36%以上,高质量reads数在983万~1 359万条。鉴别出个显著性1 018差异表达基因,其中实验组中有509个基因表达上调,509个基因表达下调。对这些差异表达基因进行GO功能注释和通路富集分析,结果发现,有772个基因获得GO功能注释信息,主要富集于RNA代谢过程和药物结合。这些差异表达基因在KEGG生物通路中,以生物化学代谢、次生代谢物的生物合成等代谢途径富集显著。并且差异倍数较大的基因都富集于糖酵解和丙酮酸代谢通路。丙酮酸代谢途径共注释到8个显著差异基因。丙酮酸在丙酮酸脱氢酶的作用下转化生成乙酰辅酶A,进入丁酸代谢,它的核心代谢通路相关基因表达量均显著下调。
在面临饥饿胁迫时,细胞繁殖生长速度减慢,与蛋白质合成相关氨酰-tRNA合成酶表达下调。为了维持基本的能量供给,该菌提高了糖酵解和丙酮酸代谢相关酶的表达。魏斯氏菌作为乳酸菌的一种,在产胞外多糖、抗炎症、促进乳杆菌生长等益生作用方面显示出一定的优势,具有极高的生产和应用价值。并且近年来高通量测序技术的不断发展,转录组学研究已经广泛地应用于乳酸菌研究领域,并且成为研究乳酸菌的一项重要技术手段。但是目前对魏斯氏菌转录组相关研究还比较少。这株魏斯氏菌来源于传统发酵辣椒,了解魏斯氏菌在面临饥饿胁迫时的响应机制,为研究乳酸菌胁迫应答机制及其工业化应用提供新思路。
[1] 李巧玉, 陈坚, 曾伟主, 等.酱醪中魏斯氏菌的分离及特性分析[J].微生物学通报, 2018, 45(11):2 449-2 462.
LI Q Y, CHEN J, ZENG W Z, et al.Isolation and characterization of Weissella strains from soy sauce moromi mash[J].Microbiology China, 2018, 45(11):2 449-2 462.
[2] AMMOR M S, MAYO B.Selection criteria for lactic acid bacteria to be used as functional starter cultures in dry sausage production:An update[J].Meat Science, 2007, 76(1):138-146.
[3] PAPAGIANNI M, PAPAMICHAEL E M.Purification, amino acid sequence and characterization of the class IIa bacteriocin weissellin A, produced by Weissella paramesenteroides DX[J].Bioresource Technology, 2011, 102(12):6 730-6 734.
[4] LYNCH K M, MCSWEENEY P L H, ARENDT E K, et al.Isolation and characterisation of exopolysaccharide-producing Weissella and Lactobacillus and their application as adjunct cultures in Cheddar cheese[J].International Dairy Journal, 2014, 34(1):125-134.
[5] PARK J A, TIRUPATHI PICHIAH P B, YU J J, et al. Anti-obesity effect of kimchi fermented with Weissella koreensis OK1-6 as starter in high-fat diet-induced obese C57BL/6 J mice[J].Journal of Applied Microbiology, 2012, 113(6):1 507-1 516.
[6] 李巧玉, 方芳, 堵国成, 等.魏斯氏菌在发酵食品中的应用[J].食品与发酵工业, 2017, 43(10):241-247.
LI Q Y, FANG F, DU G C, et al.The application of Weissella strains in fermented food[J].Food and Fermentation Industries, 2017, 43(10):241-247.
[7] 李文, 陈银元, 陈立明, 等.两株魏斯氏菌的益生特性比较[J].食品与发酵工业, 2019, 45(15):37-43.
LI W, CHEN Y Y, CHEN L M, et al.Comparative analysis of probiotic properties of two Weissella strains[J].Food and Fermentation Industries, 2019, 45(15):37-43.
[8] 雷忠华, 陈聪聪, 陈谷.基于宏基因组和宏转录组的发酵食品微生物研究进展[J].食品科学, 2018, 39(3):330-337.
LEI Z H, CHEN C C, CHEN G.Metagenomic and metatranscriptomic analysis of microbiota in fermented foods:Review of recent advances[J].Food Science, 2018, 39(3):330-337.
[9] DEUTSCH S M, MARIADASSOU M, NICOLAS P, et al. Identification of proteins involved in the anti-inflammatory properties of Propionibacterium freudenreichii by means of a multi-strain study[J]. Scientific reports, 2017, 7(1): 1-13..
[10] 彭勇, 陈尚武, 马会勤.黑果枸杞果实成熟发育过程表达谱差异分析[J].生物技术通报, 2016, 32(11):144-151.
PENG Y, CHEN S W, MA H Q.Differential expression analysis of gene expression profiles during fruit development and ripening of Lycium ruthenicum[J].Biotechnology Bulletin, 2016, 32(11):144-151.
[11] ESCOBAR-ZEPEDA A, SANCHEZ-FLORES A, QUIRASCO BARUCH M.Metagenomic analysis of a Mexican ripened cheese reveals a unique complex microbiota[J].Food Microbiology, 2016, 57:116-127.
[12] XU Q, LI S, HUANG H, et al.Key technologies for the industrial production of fumaric acid by fermentation[J].Biotechnology Advances, 2012, 30(6):1 685-1 696.
[13] BRANTON D, DEAMER D W, MARZIALI A, et al. The potential and challenges of nanopore sequencing[J]. Nature, 2008, 26(10): 1 146-1 153.
[14] CAO N, DU J, GONG C S, et al.Simultaneous production and recovery of fumaric acid from immobilized Rhizopus oryzae with a rotary biofilm contactor and an adsorption column[J].Applied and Environmental Microbiology, 1996, 62(8):2 926-2 931.
[15] FOSTER J W, WAKSMAN S A.The production of fumaric acid by molds belonging to the genus Rhizopus[J].Journal of the American Chemical Society, 1939, 61(1):127-135.
[16] 陈兴麟, 李丽, 林河通.两种海藻寡糖促乳酸菌增长的转录组比较研究[J].应用海洋学学报, 2020, 39(1):1-11.
CHEN X L, LI L, LIN H T.Comparative study of transcriptomes from Lactobacillus promoted by two algal oligosaccharides[J].Journal of Applied Oceanography, 2020, 39(1):1-11.
[17] 田源, 孔小勇, 方芳.浓香型白酒发酵过程微生物合成正丙醇途径解析[J].微生物学报, 2020, 60(7):1 421-1 432.
TIAN Y, KONG X Y, FANG F.Microbial n-propanol synthesis during Luzhou-flavor liquor fermentation[J].Acta Microbiologica Sinica, 2020, 60(7):1 421-1 432.
[18] ERCAN O, WELS M, SMID E J, et al.Genome-wide transcriptional responses to carbon starvation in nongrowing Lactococcus lactis[J].Applied & Environmental Microbiology, 2015, 81(7):2 554-2 561.
[19] MARIONI J C, MASON C E, MANE S M, et al.RNA-seq:An assessment of technical reproducibility and comparison with gene expression arrays[J].Genome Research, 2008, 18(9):1 509-1 517.
[20] 李欣,钟永亮,刘云宏,等. 胱氨酸/半胱氨酸调控活性氧代谢影响大肠杆菌耐药性[J].微生物学通报, 2019, 46(10):2 710-2 718.
LI X,ZHONG Y L, LIU Y H, et al.Impact of cystine/cysteine on drug resistance of Escherichia coli by regulating ROS metabolisms[J].Microbiology China, 2019, 46(10):2 710-2 718.
[21] ROBINSON M D, MCCARTHY D, SMYTH G K.Edger:A bioconductor package for differential expression analysis of digital gene expression data[J].Biogeosciences, 2010, 26:139-140.