微生物谷氨酰胺转氨酶(transglutaminase,TGase)是工业中常用的工具酶,能够催化酰基供体进行脱酰胺和酰基转移,进而形成共价键以完成分子交联[1]。目前市场上食品级TGase来源于茂原链霉菌(Streptomyces mobaraenesis),因具有非钙离子依赖、发酵成本低等优势而备受市场青睐[2]。近些年随着对TGase催化特异性的研究,其在纺织、制药和材料加工等非食品领域的应用也广受关注[3-5]。
随着S.mobaraenesis TGase在工业领域的广泛应用,其较差的热稳定性也成为亟待解决的问题。目前,已有随机突变、DNA改组和饱和突变为基础的定向进化提高TGase热稳定性的报道[6-8]。其中,突变体MS(S2P-S23V-Y24 N-S199A-K294L)是目前热稳定性较优的TGase突变体[9]。与氢键等因素相比,二硫键可以降低蛋白质中未折叠多肽链的熵以提升稳定性,是影响热稳定性的最重要因素[10]。理性设计形成蛋白质分子内部二硫键也是提高酶稳定性的重要手段。ZHANG等[11]对一种嗜热植酸酶理性设计引入了8组二硫键,其中最佳一组突变体60 ℃半衰期提升了3.8倍,同时表现出更强的抗胰蛋白酶及胃蛋白酶降解能力;朱方剑[12]通过理性设计在角质酶中引入二硫键成功获得一个热稳定性显著提高的突变体,该突变体70 ℃处理10 min残余酶活是野生型的1.5倍,80 ℃半衰期可达16 h;王睿等[13]通过理性设计在脂肪酶中引入二硫键,最佳一组突变体60 ℃半衰期较原始酶延长了3.5倍,Tm提高4.2 ℃。
本研究以突变体MS为基础,通过对野生型TGase结构进行虚拟突变和能量优化获得了MS的模拟构象。应用Disulfide by design 2.0线上分析软件对潜在的二硫键成键突变体进行预测,选择12对二硫键突变体,利用大肠杆菌(Escherichia coli)进行表达、纯化及酶学性质分析,并对相关突变体蛋白机理进行了探讨。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
所用大肠杆菌E.coli JM109和E.coli BL21(DE3)由本实验室保存。E.coli JM109作为克隆宿主,E.coli BL21(DE3)作为构建质粒的表达宿主。编码突变体MS(S2P-S23V-Y24 N-S199A-K294L)基因由苏州金唯智(GENEWIZ)合成,并通过酶切位点BlpI、NdeI克隆至的pET-22b(+),得到质粒pET-22b(+)/pro-MS。编码硫氧还原蛋白TrxA的质粒pET-48b由本实验室保存。
1.1.2 实验试剂
DNA工具酶及磷酸化试剂盒(Blunting Kination Ligation Kit),Takara大连宝生物公司;中性蛋白酶,北京索莱宝科技有限公司;5 mL镍离子亲和层析柱、5 mL G-25凝胶层析柱,江苏千纯生物科技有限公司;Bradford蛋白浓度测定试剂盒,碧云天生物技术公司;CBZ-Gln-Gly,Sigma-Aldrich中国;一步克隆试剂盒(Vazyme ClonExpress II One Step Cloning Kit C112),诺唯赞;其他分子生物学试剂,大连宝生物公司。
1.1.3 培养基与缓冲液
LB培养基:酵母提取物5 g/L、胰蛋白胨10 g/L、NaCl 10 g/L,固体培养基添加质量分数2%的琼脂粉。
TB培养基:酵母提取物24 g/L、胰蛋白胨12 g/L、K2HPO4·3H2O 12.84 g/L、KH2PO4 2.31 g/L,甘油4 mL/L。
TGase活化液:50 mmol/L Tris-HCl,100 mmol/L NaCl,2 mmol/L CaCl2,1 mmol/L还原型谷胱甘肽、200 g/L中性蛋白酶,调节pH至7.0。
纯化缓冲液A液:50 mmol/L Tris-HCl,100 mmol/L NaCl,25 mmol/L咪唑,调节pH至7.8。
洗脱缓冲液B液:50 mmol/L Tris-HCl,100 mmol/L NaCl,500 mmol/L咪唑,调节pH至7.8。
脱盐缓冲液C液:50 mmol/L Tris-HCl,调节pH至7.8。
二硫键还原缓冲液:0.1 mol/L Tris-HCl,8 mol/L脲素,调节pH至7.0。
酶活测试底物:200 mmol/L Tris-HCl,100 mmol/L羟胺,10 mmol/L还原型谷胱甘肽,30 mmol/L CBZ-Gln-Gly,调节pH至6.0。
酶活力测试终止液:将120 g/L三氯乙酸、3 mmol/L HCl、50 g/L FeCl3·6H2O(溶于0.1 mol/L HCl)等体积混合均匀,避光保存。
1.2 实验方法
1.2.1 软件分析
通过I-TASSER (https://zhanglab.ccmb.med.umich.edu/I-TASSER) 线上服务器根据野生型成熟TGase构象(PDB:1iu4)对突变体MS构象建模[14]。在此基础上,通过Rosetta-StructureRelax模块对MS构象进行优化。将获得的最终构象上传至Disulfide by design 2.0(http://cptweb.cpt.wayne.edu/DbD2/)线上服务器进行二硫键突变预测[15]。对预测得到的55个突变体根据能量分组,即能量由高到低每9个分为一组(最后一组10个)。在各分组中抽取2个作为实验对象,进行突变后验证。
1.2.2 突变体的构建
通过一步克隆构建表达载体pET-22b(+)/TrxA-pro-MS(图1-a)。以trxa-f和trxa-r为引物,通过PCR从质粒pET-48b扩增得到TrxA片段。以vec-f和vec-r为引物,通过PCR从pET-22b/pro-MS扩增得到pET-22b/pro-MS开环片段。开环片段溶液经Dpn I消化后,与TrxA片段通过一步克隆试剂盒连接,产物转化E.coli JM109,提取质粒测序,筛选得到序列正确的表达载体pET-22b(+)/TrxA-pro-MS。
以ms-s1-f/ms-s1-r、ms-s2-f/ms-s2-r、ms-s3-f/ms-s3-r、ms-s4-f/ms-s4-r、ms-s5-f/ms-s5-r、ms-s6-f/ms-s6-r、ms-s7-f/ms-s7-r和ms-s8-f/ms-s8-r为引物对pET-22b(+)/TrxA-pro-MS进行PCR扩增,分别得到含有MS-P244C-E249C、MS-S293C-N297C、MS-E182C-G185C、MS-A136C-N149C、MS-D118C-K121C、MS-A136C-L150C、MS-L154C-R162C和MS-D86C-R89C的突变基因片段。PCR产物经Dpn I消化后回收,用磷酸化试剂盒连接后转化E.coli JM109并提取质粒测序验证。以pET-22b(+)/TrxA-pro-MS为模板,通过一步克隆法构建二硫键突变体MS-Q206C-I240C、MS-G110C-Y217C、MS-P22C-Q328C和MS-A130C-A195C。胶回收PCR产物并将含有同源臂的片段试剂盒连接,转化E.coli JM109并提取质粒测序验证。测序正确的质粒转化E.coli BL21(DE3),获得突变体生产菌株。本研究所使用的引物见表1。
表1 本研究所使用的引物
Table 1 Primers for this study
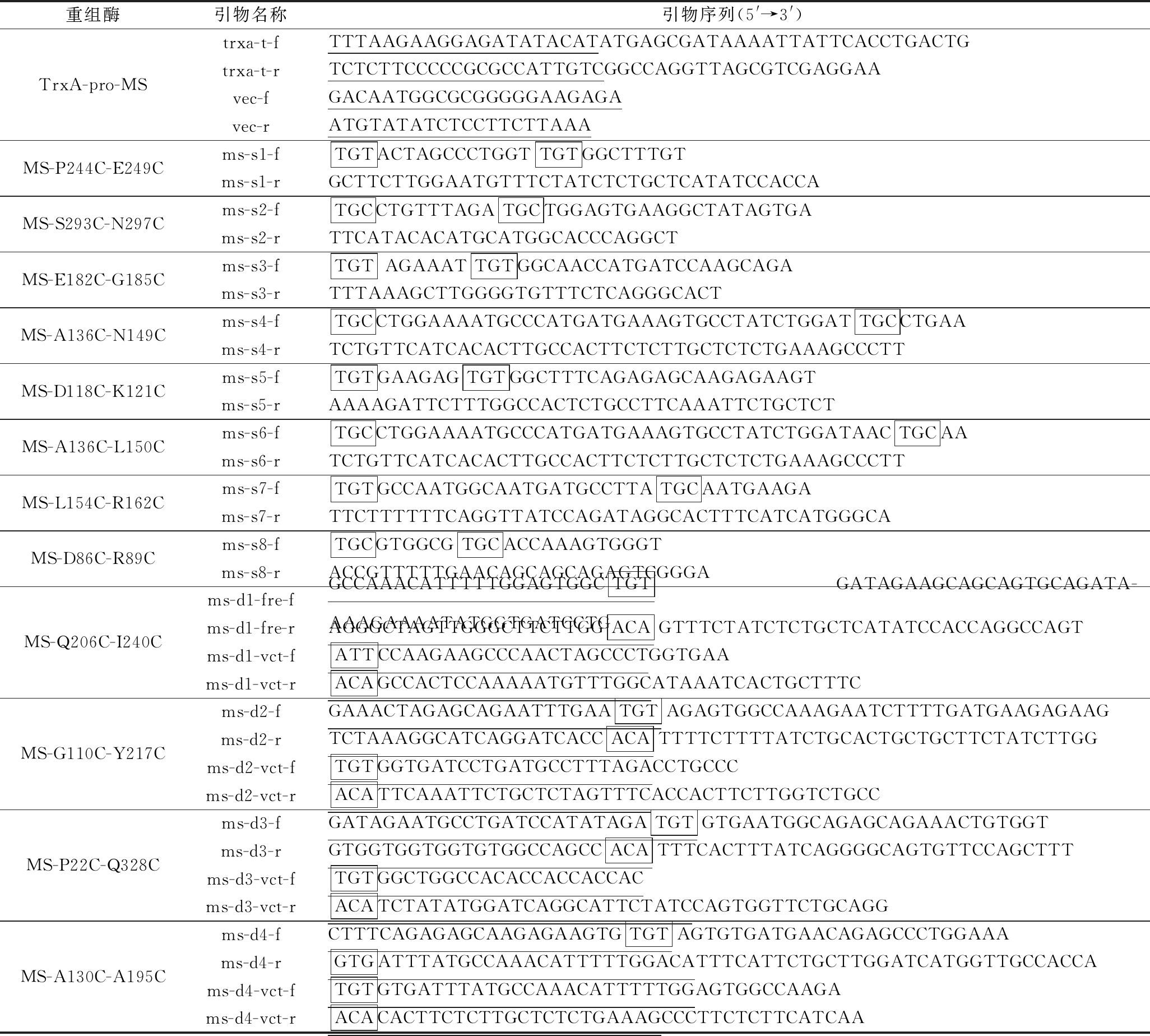
注:下划线表示同源片段,方框中为突变位点
重组酶引物名称引物序列(5'→3')TrxA-pro-MStrxa-t-fTTTAAGAAGGAGATATACATATGAGCGATAAAATTATTCACCTGACTGtrxa-t-rTCTCTTCCCCCGCGCCATTGTCGGCCAGGTTAGCGTCGAGGAAvec-fGACAATGGCGCGGGGGAAGAGAvec-rATGTATATCTCCTTCTTAAAMS-P244C-E249Cms-s1-fTGTACTAGCCCTGGTTGTGGCTTTGTms-s1-rGCTTCTTGGAATGTTTCTATCTCTGCTCATATCCACCAMS-S293C-N297Cms-s2-fTGCCTGTTTAGATGCTGGAGTGAAGGCTATAGTGAms-s2-rTTCATACACATGCATGGCACCCAGGCTMS-E182C-G185Cms-s3-fTGTAGAAATTGTGGCAACCATGATCCAAGCAGAms-s3-rTTTAAAGCTTGGGGTGTTTCTCAGGGCACTMS-A136C-N149Cms-s4-fTGCCTGGAAAATGCCCATGATGAAAGTGCCTATCTGGATTGCCTGAAms-s4-rTCTGTTCATCACACTTGCCACTTCTCTTGCTCTCTGAAAGCCCTTMS-D118C-K121Cms-s5-fTGTGAAGAGTGTGGCTTTCAGAGAGCAAGAGAAGTms-s5-rAAAAGATTCTTTGGCCACTCTGCCTTCAAATTCTGCTCTMS-A136C-L150Cms-s6-fTGCCTGGAAAATGCCCATGATGAAAGTGCCTATCTGGATAACTGCAAms-s6-rTCTGTTCATCACACTTGCCACTTCTCTTGCTCTCTGAAAGCCCTTMS-L154C-R162Cms-s7-fTGTGCCAATGGCAATGATGCCTTATGCAATGAAGAms-s7-rTTCTTTTTTCAGGTTATCCAGATAGGCACTTTCATCATGGGCAMS-D86C-R89Cms-s8-fTGCGTGGCGTGCACCAAAGTGGGTms-s8-rACCGTTTTTGAACAGCAGCAGAGTCGGGAMS-Q206C-I240Cms-d1-fre-fGCCAAACATTTTTGGAGTGGCTGTGATAGAAGCAGCAGTGCAGATA-AAAGAAAATATGGTGATCCTGms-d1-fre-rAGGGCTAGTTGGGCTTCTTGGACAGTTTCTATCTCTGCTCATATCCACCAGGCCAGTms-d1-vct-fATTCCAAGAAGCCCAACTAGCCCTGGTGAAms-d1-vct-rACAGCCACTCCAAAAATGTTTGGCATAAATCACTGCTTTCMS-G110C-Y217Cms-d2-fGAAACTAGAGCAGAATTTGAATGTAGAGTGGCCAAAGAATCTTTTGATGAAGAGAAGms-d2-rTCTAAAGGCATCAGGATCACCACATTTTCTTTTATCTGCACTGCTGCTTCTATCTTGGms-d2-vct-fTGTGGTGATCCTGATGCCTTTAGACCTGCCCms-d2-vct-rACATTCAAATTCTGCTCTAGTTTCACCACTTCTTGGTCTGCCMS-P22C-Q328Cms-d3-fGATAGAATGCCTGATCCATATAGATGTGTGAATGGCAGAGCAGAAACTGTGGTms-d3-rGTGGTGGTGGTGTGGCCAGCCACATTTCACTTTATCAGGGGCAGTGTTCCAGCTTTms-d3-vct-fTGTGGCTGGCCACACCACCACCACms-d3-vct-rACATCTATATGGATCAGGCATTCTATCCAGTGGTTCTGCAGGMS-A130C-A195Cms-d4-fCTTTCAGAGAGCAAGAGAAGTGTGTAGTGTGATGAACAGAGCCCTGGAAAms-d4-rGTGATTTATGCCAAACATTTTTGGACATTTCATTCTGCTTGGATCATGGTTGCCACCAms-d4-vct-fTGTGTGATTTATGCCAAACATTTTTGGAGTGGCCAAGAms-d4-vct-rACACACTTCTCTTGCTCTCTGAAAGCCCTTCTCTTCATCAA
1.2.3 突变体的诱导表达
阳性转化子接入氨苄青霉素浓度为0.1 g/L的LB培养基中,37 ℃过夜培养10 h后以2%接种量接于相同质量浓度抗性的TB培养基中,37 ℃培养至OD600=1.0时加入终浓度为0.1 mmol/L IPTG诱导表达,20 ℃培养40 h。
1.2.4 发酵产物回收
发酵液6 000×g离心10 min,收集沉淀以脱盐溶液C液重悬,冰上预冷后超声破碎20 min,10 000×g离心20 min,取上清液添加1/5体积TGase活化液,37 ℃水浴孵育30 min后10 000×g离心25 min,收集上清液用于纯化。
1.2.5 TGase的纯化、脱盐及蛋白浓度测定
蛋白纯化采用镍柱通过纯化缓冲液A液进行平衡,以V(纯化缓冲液A)∶V(洗脱缓冲液B液)=7∶3的比例对蛋白样品进行洗脱。回收蛋白后通过G-25凝胶柱使用脱盐溶液C液对样品进行脱盐。蛋白浓度采用Bradford(碧云天)试剂盒进行测定。
1.2.6 酶活测定
比色法测定TGase酶活力[16],定义1单位TGase酶活力为37 ℃条件每下1 min生成1 μmol L-谷氨酸 γ-单羟肟酸[17]的酶量。
1.2.7 酶半衰期测定
酶溶液稀释至0.5 g/L,60 ℃水浴,0~10 min内每1 min取样,10~40 min内每隔2 min取样,取出的样品置于10 ℃水浴冷敷。对所取样品进行酶活力测定,通过Original 2018-Exponential-ExpDec1对残余酶活力百分比进行非线性拟合,获得拟合公式(1)后计算出酶活力下降至初始的50%所对应的时间即为半衰期。
残余酶活力![]()
(1)
1.2.8 巯基滴定法检测二硫键生成
将蛋白样品(非还原态TGase)用二硫键还原缓冲液稀释至0.3 g/L,取2 mL TGase样品与50 μL β-巯基乙醇混合,37 ℃热处理1 h得到还原态TGase样品,加入2 mL 三氯乙酸继续孵育1 h后14 000×g离心10 min,取沉淀加入2 mL缓冲液溶液复溶得到还原态TGase。取1 mL还原态及非还原态TGase加入15 μL 2-对硝基苯甲酸,室温放置20 min检测412 nm处吸收光。根据公式(2)计算二硫键数量:
(2)
式中:n,二硫键数量;Ncys,半胱氨酸残基数量;A412reduced,还原态TGase样品在412 nm处吸光值;A412non-reduced,非还原态TGase样品在412 nm处吸光值。
1.2.9 熔解温度分析
稀释TGase纯蛋白溶液(溶剂为Tris-HCl,pH=8.0)至质量浓度为2 g/L,通过差示扫描量热法进行熔解温度测试。以蛋白所在溶剂作为内参,扫描温度从40~90 ℃、升温速率为1 ℃/min,压力为303.975 kPa。
1.2.10 动力学参数测定
TGase样品液稀释至0.05 g/L,配制10份含不同浓度CBZ-Gln-Gly的底物溶液A(CBZ-Gln-Gly含量分别为3、6、9、12、15、18、21、24、27、30 mmol/L)。测定37 ℃反应10 min后各底物溶液A中CBZ-Gln-Gly的转化量,通过Origin 2018-Growth/Sigmodial-Hill非线性拟合获得Km和Vmax值,以酶浓度换算获得kcat值及Km/kcat值获得酶催化效率。
1.2.11 酶-底物分子对接
以S.mobaraenesis TGase晶体结构(PDB:1iu4)为模板,通过Discovery studio 2016 Macromolecule-Design Protein-Build Mutants模块分别构建MS及MS-P22C-Q328C结构,利用Minimization模块中的Smart minimizer对模拟结果进行能量最小化处理。底物CBZ-Gln-Gly构象从PubChem(https://pubchem.ncbi.nlm.nih.gov/compound/Z-Gln-Gly)获得,并通过Discovery studio 2016转为pdb文件。应用AutodockTools-1.5.6(ADT)分别对底物与酶进行柔性对接,对接盒子设定为50×50×50 ang。采用ADT对结果进行聚类分析,最终对接构象选择依据如下:(1)对接构象来源于大类;(2)底物CBZ-Gln-Gly的4-氨基甲酰基团需与酶分子中β-SH基团满足距离小于3;(3)对接构象在对应聚类中能量最低。
1.2.12 分子动力学模拟
应用Gromacs-2020分别对能量优化后的MS和MS-P22C-Q328C构象进行力场化和溶剂化处理,应用溶剂为水、力场为54a7、分子边界为1.2 nm[18]。之后对体系进行能量最小化、升温平衡(达到330 K),并进行10 ns的动力学模拟,温度设定为330 K,对动力学模拟过程中的蛋白总体RMSD和各氨基酸RMSF进行分析,以获得突变位点对蛋白结构的影响。
2 结果与分析
2.1 突变位点的选择及突变体的表达
使用I-TASSER对MS突变体建模,通过Rossetta的StructureRelax进行结构优化,最终获得的MS构象与野生型TGase构象RMSD相差为0.813。通过Disulfide by design 2.0对该结构进行二硫键突变模拟,结果给出55组可能形成二硫键的突变对。一般认为,二硫键成键自由能在一定程度上影响突变体的热稳定性[19]。基于二硫键成键自由能的大小,将预测的55个二硫键突变体排序分为6组(表2)。前5组每组包含9个突变体,第6组包含10个突变体。每组抽取2个,共12个二硫键突变体(图1-b),以保证其代表性。
表2 基于成键能量对预测的二硫键突变体进行分组
Table 2 Classification of predicted disulfide bonds mutations based on predicted Gibbs energy changes

分组突变体二硫键成键自由能/(kJ·mol-1)分组突变体二硫键成键自由能/(kJ·mol-1)第1组22PRO-27ALA34.4143SER-50GLN15.8671ASN-321THR33.7417PRO-36ARG15.32280ALA-283GLY29.76113ALA-220PRO15.32206GLN-240ILE29.47140ALA-146TYR15.1944HIS-320ASN27.96第4组106ALA-213ASP15.03302TYR-304ASP27.7922PRO-328GLN14.90164GLU-168SER26.9284SER-121LYS14.65185GLY-316PRO26.29130ALA-195ALA13.60244PRO-249GLU25.1690PHE-116SER12.68第2组79ARG-169PRO23.82272TRP-298TRP12.0181ALA-232VAL23.36279HIS-288MET11.89266ASP-269LYS22.81118ASP-121LYS11.80110GLY-217TYR22.7778ASN-234MET11.5561SER-279HIS22.10第5组281PRO-287ALA11.3499PRO-108PHE22.06204SER-306ASP10.3829THR-33ASN21.35136ALA-150LEU10.00293SER-297ASN20.80126ALA-310TYR9.29201HIS-305PHE20.13103GLU-107GLU9.25第3组179SER-185GLY19.97140ALA-145ALA8.46241PRO-253ASN19.8426ARG-293SER8.29182GLU-185GLY19.51207ASP-210SER7.2031VAL-288MET17.54154LEU-162ARG7.16136ALA-149ASN17.25第6组219ASP-222ALA6.57261ALA-322ALA17.12215ARG-243SER5.69177THR-180PHE16.99119GLU-299SER5.4499PRO-103GLU16.3234TYR-290VAL4.2384SER-118ASP15.9186ASP-89ARG3.73205GLY-214LYS1.97
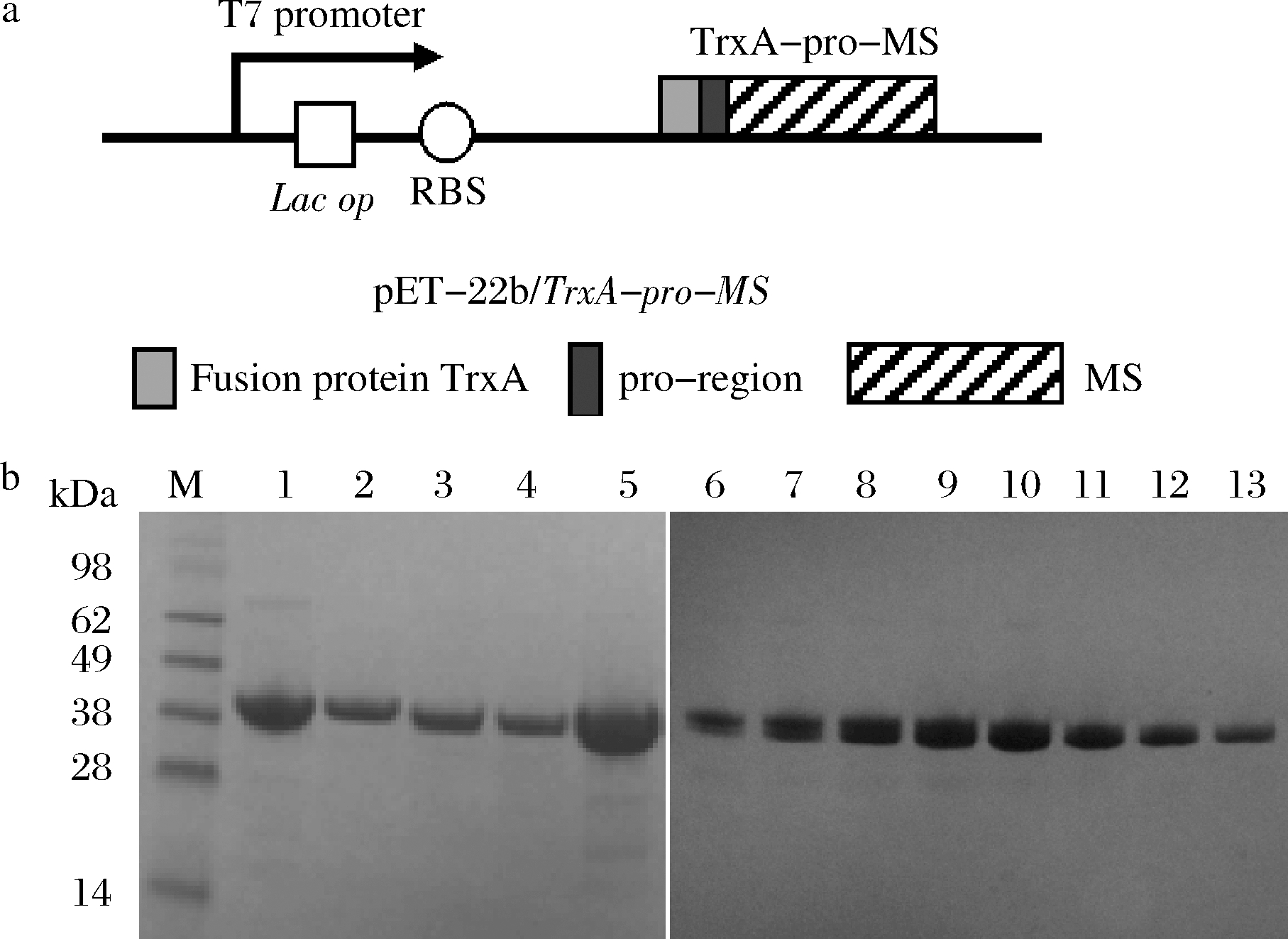
a-突变体pET-22b/TrxA-pro-MS表达框;b-含二硫键突变体纯化后电泳图
注:1-MS;2-MS-GLN206-ILE240;3-MS-PRO244-GLU249;4-MS-GLY110-TYR217;5-MS-SER293-ASN297;6-MS-GLU182-GLY185;7-MS-ALA136-AS-N149;8-MS-PRO22-GLN328;9-MS-ALA130-ALA195;10-MS-ASP118-LYS121;11-MS-ALA136-LEU150;12-MS-LEU154-ARG162;13-MS-ASP86-ARG89
图1 TGase突变体的表达及纯化
Fig.1 TGase variants expression and purification
由于大肠杆菌E.coli BL21不利于二硫键的形成,故本研究在构建TGase表达框时选择硫氧还原蛋白TrxA与pro-TGase进行共表达以促进二硫键的形成及TGase的正确折叠(图1-a)。对挑选出的12对突变体进行构建、转化及摇瓶发酵,对获得的重组菌胞内可溶TGase进行体外活化及镍柱纯化得到TGase纯蛋白。SDS-PAGE分析显示,纯化后的TGase蛋白条带均一(图1-b)。
2.2 高热稳定型TGase 突变体的初筛及二硫键验证
热稳定性分析显示,突变体MS于60 ℃水浴处理20 min后残余酶活力32.9%。对12对突变体进行热稳定性实验,共有6对突变体残余酶活力有所升高。其中,突变体D118C-K121C、P244C-E249C和P22C-Q328C残余酶活力分别为58.36%、56.45%和62.86%,相较于MS分别提升了77.39%、71.58%和91.06%。突变体MS-P244C-E249C和MS-P22C-Q328C的比酶活略有下降(图2)。

图2 TGase突变体初始比酶活力及热处理 (60 ℃ 20 min)后残余酶活力
Fig.2 Residual activity after heat treatment (60 ℃,20 min) and specific activity of TGase mutants
对热稳定性提升最明显的3对突变体,进行二硫键还原反应测定。结果如图3所示,横坐标表示突变体,纵坐标表示二硫键的数量,3种突变体均成功形成了分子内二硫键。后续实验中,以热稳定性提升最高的突变体MS-P22C-Q328C为研究对象,研究其稳定性提升状态、催化效率变化及稳定性提升机制。
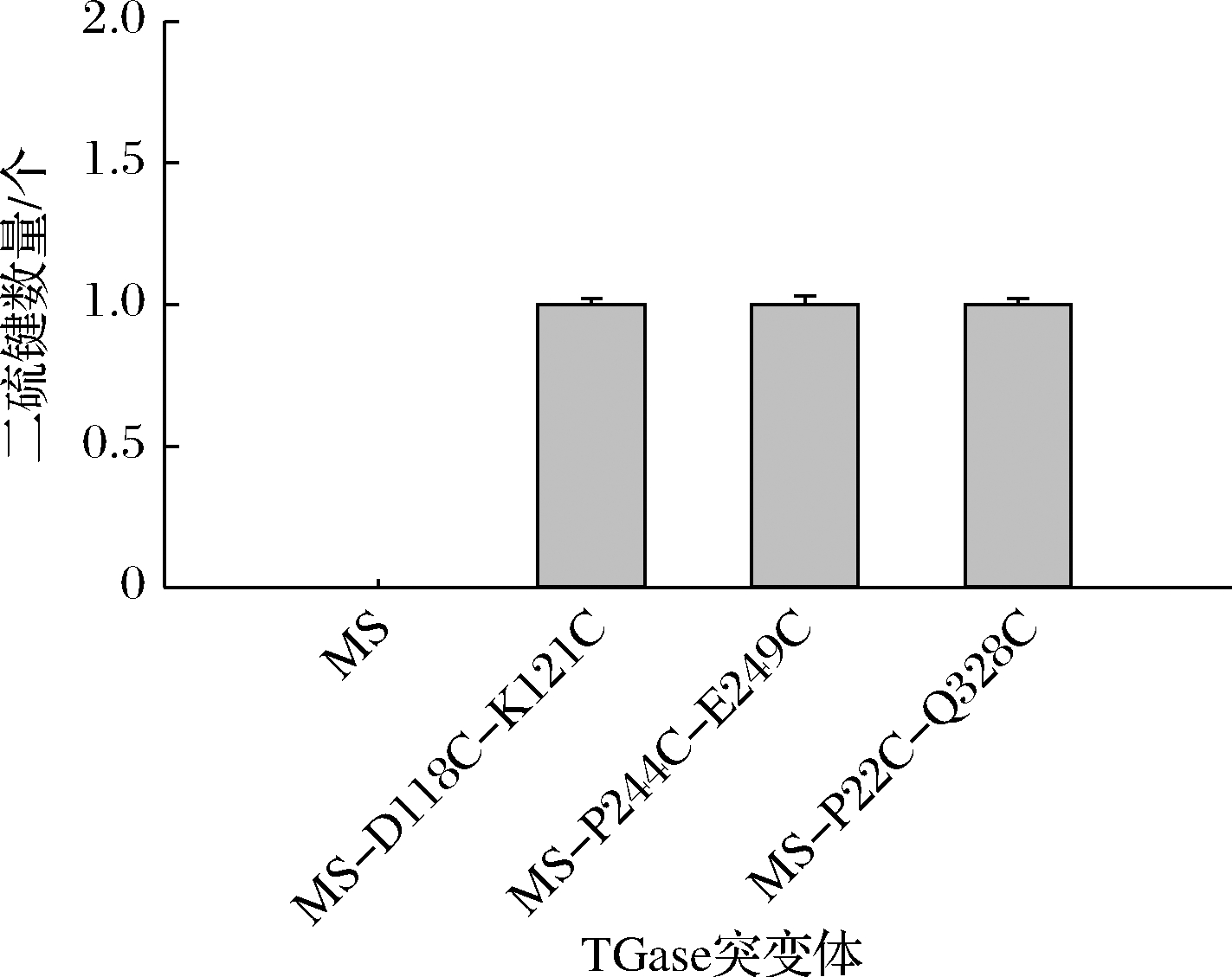
图3 突变体二硫键测定
Fig.3 Disulfide bond number of TGase mutants
2.3 TGase突变体MS-P22C-Q328C酶学性质分析
2.3.1 突变体MS-P22C-Q328C半衰期和温度稳定性测定
为进一步研究TGase突变体MS-P22C-Q328C的稳定性提升效果,进行半衰期及熔解温度测定。2种蛋白在60 ℃水浴前5 min内残余酶活力下降均比较快,之后残余酶活力百分比差距逐渐增大(图4-a)。经分析发现,突变体MS-P22C-Q328C的t1/2(60 ℃)达到了34.61 min,较MS(11.31 min)提升了2.06倍。差示扫描热量测定,发现MS-P22C-Q328C的tm相较于MS提高了1.06 ℃(图4-b)。

a-TGase突变体的半衰期曲线;b-TGase突变体的熔解温度曲线
图4 TGase突变体的半衰期及熔解温度测定
Fig.4 Half-life and melting temperature measurement for TGase mutants
2.3.2 突变体MS-P22C-Q328C酶促反应动力学测定
本研究分别测试了TGase突变体MS及MS-P22C-Q328C的酶促反应动力学参数。通过梯度稀释得到含不同浓度CBZ-Gln-Gly的底物溶液A,测试TGase突变体对不同浓度CBZ-Gln-Gly的转化率。结果发现,MS对于低浓度CBZ-Gln-Gly具有较高的转化效果,即Km值较低,而在实际催化效率上,即kcat差距不是很明显(表3)。结果表明突变体的底物结合能力受到了一定的影响。
表3 TGase突变体的酶促反应动力学参数测定
Table 3 Kinetic parameters of TGase mutants
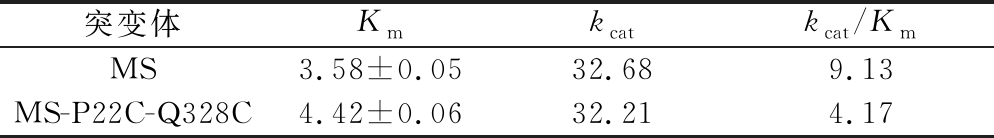
突变体Kmkcatkcat/KmMS3.58±0.0532.689.13MS-P22C-Q328C4.42±0.0632.214.17
2.3.3 突变体MS-P22C-Q328C突变位点分析
基于S.mobaraenesis TGase晶体结构,分别构建了MS和MS-P22C-Q328C的模拟结构。如图5所示,MS-P22C-Q328C分子中的二硫键位于酶分子长Loop结构上。研究表明,强化蛋白质分子的loop区域的刚性可显著提高其热稳定性[20]。因此,在突变体MS-P22C-Q328C中,二硫键可能限制了Loop环摆动,从而提高了其热稳定性[21]。值得注意的是,该Loop结构位于底物进入酶分子的通道[1],其刚性的增强可能影响了酶与底物结合,导致了MS-P22C-Q328C底物亲和力和活性的下降的下降(表3)。
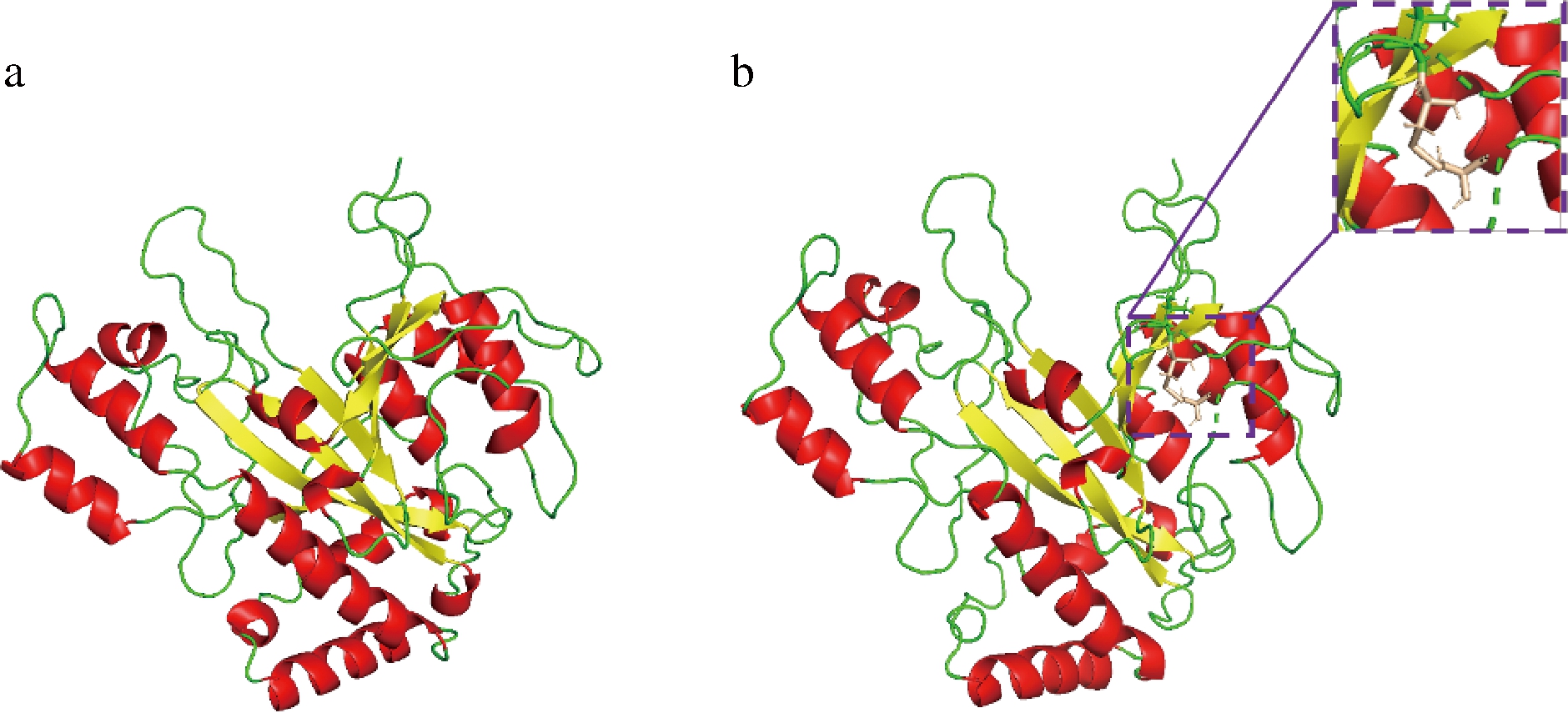
a-MS结构;b-MS-P22C-Q328C结构
图5 MS及MS-P22C-Q328C分子结构
Fig.5 Structure of MS and MS-P22C-Q328C
为进一步解释突变体MS-P22C-Q328C较MS活性下降的原因,以CBZ-Gln-Gly为底物进行对接分析。如图6-a和图6-b所示,MS及MS-P22C-Q328C与底物的结合能分别为-17.96和-17.50 kJ/mol,表明前者较后者与底物结合更紧密。事实上,MS与CBZ-Gln-Gly对接时CBZ基团指向酶分子的催化三联体(C64-D255-H274)(图6-c)。在MS-P22C-Q328C中,底物分子的CBZ基团则指向催化三联体(图6-d)。因此,MS-P22C-Q328C中二硫键可能改变了其底物结方式,降低了结合力。

a-MS与底物对接聚类分析;b-MS-P22C-Q328C与底物对接聚类分析;c-MS与底物对接最终构象;d-MS-P22C-Q328C与底物对接最终构象
图6 TGase突变体与底物CBZ-Gln-Gly对接结果展示
Fig.6 Molecular docking of TGase mutants with CBZ-Gln-Gly
2.3.4 突变体MS-P22C-Q328C分子动力学模拟
分子动力学模拟作为反映蛋白分子运动的一个重要方法,可以用来解释蛋白稳定性成因、蛋白与底物对接机制等。通过分子动力学中的升温动力学分析TGase突变体在体系中运动的情况,基于MS对突变体MS-P22C-Q328C进行建模,并对两者同时进行能量最小化处理和热量平衡。后在330 K条件下进行动力学模拟10 ns。经动力学模拟后观察TGase突变体的RMSD变化,发现突变体MS-P22C-Q328C的RMSD整体变化低于MS,说明其在高温状态下刚性更强,不易于变性(图7-a)。通过对动力学模拟过程中,TGase突变体的各氨基酸位移变化分析,发现突变体MS-P22C-Q328C蛋白中区域15-32及311-330中氨基酸整体位移降低,即RMSF低于MS(图7-b)。因此,可以推断突变氨基酸对P22C-Q328C可有效地提高区域的刚性程度,从而提高蛋白整体稳定性。

a-TGase突变体动力学模拟过程中RMSD变化曲线;b-TGase突变体动力学模拟过程中RMSF变化曲线
图7 TGase突变体的动力学模拟
Fig.7 MD simulations on TGase mutants
通过对突变体MS-P22C-Q328C进行的酶学性质测定、分子结构分析及分子动力学模拟,可以认为发生在突变体MS结构中Loop环上P22C-Q328C两处突变形成了二硫键,从而在一定程度上提升了TGase的刚性,有效提高了蛋白质的热稳定性,同时保留了大部分的催化活性(MS-P22C-Q328C比酶活力为43.36 U/mg,相比于MS的50.01 U/mg仅降低13.29%)。
3 结论
为进一步提高TGase的热稳定性,基于高稳定性TGase突变体MS构建了酶分子内二硫键,共获得了3个稳定性明显提升的二硫键突变体。60 ℃处理20 min,P244C-E249C、D118C-K121C和P22C-Q328C残余酶活力分为56.45%,58.36%和62.86%。其中,MS-P22C-Q328C较MS t1/2(60 ℃)提高2.06倍,tm提高1.06 ℃。值得注意的是,P244C-E249C、D118C-K121C和P22C-Q328C成键自由能分别为25.16、11.80和14.90 kJ/mol。突变体二硫键成键自由能大小与蛋白质热稳定性的相关性不明显。
在理性设计二硫键的过程中,除了考虑二硫键成键自由能的大小,还需要结合可能形成二硫键的突变位点空间位置的距离(即成键后侧链偏转角度)以及原始氨基酸的柔性[15]。在预测二硫键过程中,成键自由能并不是影响突变体热稳定性的主要因素。随机抽取不同成键自由能的二硫键突变体进行构建后,发现最终对于TGase稳定性提高较明显的二硫键突变体成键能适中,后续的基于构建分子内二硫键提升蛋白稳定性过程中还有其他重要因素有待挖掘。
[1] KASHIWAGI T,YOKOYAMA K I,ISHIKAWA K,et al.Crystal structure of microbial transglutaminase from Streptoverticillium mobaraense[J].Journal of Biological Chemistry,2002,277(46):44 252-44 260.
[2] ANDO H,ADACHI M,UMEDA K,et al.Purification and characteristics of a novel transglutaminase derived from microorganisms[J].Agricultural and Biological Chemistry,1989,53(10):2 613-2 617.
[3] STROP P.Versatility of microbial transglutaminase[J].Bioconjugate Chemistry,2014,25(5):855-862.
[4] LESIOW T,RENTFROW G K,XIONG Y L.Polyphosphate and myofibrillar protein extract promote transglutaminase-mediated enhancements of rheological and textural properties of PSE pork meat batters[J].Meat Science,2017,128:40-46.
[5] SERNA N,LAURA S G,UNZUETA U,et al.Protein-based therapeutic killing for cancer therapies[J].Trends in Biotechnology,2018,36(3):318-335.
[6] MARX C K,HERTEL T C,PIETZSCH M.Random mutagenesis of a recombinant microbial transglutaminase for the generation of thermostable and heat-sensitive variants[J].Journal of Biotechnology,2008,136(3-4):156-162.
[7] BUETTNER K,HERTEL T C,PIETZSCH M.Increased thermostability of microbial transglutaminase by combination of several hot spots evolved by random and saturation mutagenesis[J].Amino Acids,2012,42(2-3):987-996.
[8] LIU Y H,HUANG L,SHAN M Y,et al.Enhancing the activity and thermostability of Streptomyces mobaraensis transglutaminase by directed evolution and molecular dynamics simulation[J].Biochemical Engineering Journal,2019,151:107 333.
[9] MU D D,LU J J,SHU C,et al.Improvement of the activity and thermostability of microbial transglutaminase by multiple-site mutagenesis[J].Bioscience,Biotechnology,and Biochemistry,2018,82(1):106-109.
[10] MATSUMURA M,SIGNOR G,MATTHEWS B W.Substantial increase of protein stability by multiple disulphide bonds[J].Nature,1989,342(6247):291-293.
[11] ZHANG Z J,YANG J,XIE P J,et al.Characterization of a thermostable phytase from Bacillus licheniformis WHU and further stabilization of the enzyme through disulfide bond engineering[J].Enzyme and Microbial Technology,2020,142:109 679.
[12] 朱方剑. 二硫键理性设计提高Thermobifida fusca角质酶热稳定性研究[D].无锡:江南大学,2020.
ZHU F J.Study on rational design of disulfide bonds to improve thermal stability of Thermobifida fusca cutinase[D].Wuxi:Jiangnan University,2020.
[13] 王睿, 喻晓蔚,徐岩.理性设计二硫键提高华根霉脂肪酶热稳定性[J].微生物学通报,2018,45(11):2 311-2 319.
WANG R,YU X W,XU Y.Rational design of disulfide bond in Rhizopus chinensis lipase to improve thermostability[J].Microbiology China,2018,45(11):2 311-2 319.
[14] YANG J Y,ZHANG Y.I-TASSER server:New development for protein structure and function predictions[J].Nucleic Acids Research,2015,43(W1):W174-W181.
[15] CRAIG D B,DOMBKOWSKI A A.Disulfide by Design 2.0:a web-based tool for disulfide engineering in proteins[J].BMC Bioinformatics,2013,14:1 471-2 105.
[16] 杜坤, 周丽,堵国成,等.内含肽介导谷氨酰胺转胺酶酶原的活化[J].食品科学,2013,34(9):90-94.
DU K,ZHOU L,DU G C,et al.Intein-mediated activation of transglutaminase from Streptomyces hygroscopicus[J].Food Science,2013,34(9):90-94.
[17] FOLK J E,COLE P W.Transglutaminase:Mechanistic features of the active site as determined by kinetic and inhibitor studies[J].Biochimica et Biophysica Acta (BBA)-Enzymology and Biological Oxidation,1966,122(2):244-264.
[18] MARK J A,TEEMU M,ROLAND S,et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX,2015,1-2:19-25.
[19] PEZESHGI M H,MOFRAD M R,SANATI N A.H.Protein thermostability engineering[J].RSC Advances,2016,6(116):115 252-115 270.
[20] 郭超, 王志彦,甘一如,等.理性设计改造牛肠激酶的热稳定性[J].中国生物工程杂志,2016,36(8):46-54.
GUO C,WANG Z Y,GAN Y R,et al.Engineering thermostability of bovine enterokinase by rational design method[J].China Biotechnology,2016,36(8):46-54.
[21] MASAZUMI M,WAYNE J B,MICHAEL L,et al.Stabilization of phage T4 lysozyme by engineered disulfide bonds[J].Proceedings of the National Academy of Sciences of the United States of America,1989,86(17):6 562-6 566.