广陈皮为芸香科植物橘的栽培变种茶枝柑的干燥成熟果皮[1],有理气健脾、燥湿化痰之功效。其药用历史悠久,是岭南地区著名道地中药材[2]。同时可制成陈皮茶、饮料、添加剂和香料等,具有很高的食用价值[3]。传统理论认为陈皮“陈久者良”[4],现代主要从不同贮藏年限的陈皮化学成分变化、药理作用的强弱比较等方面对其进行探讨,而对陈皮陈化机理的现代研究相对薄弱[5-6]。本课题组前期研究发现,陈皮中黄酮类成分的增长与药材贮藏过程中表面污染真菌黑曲霉等的代谢活动相关[7]。相关文献也报道微生物参与了陈皮的陈化过程,能够转化陈皮中化合物从而使活性物质含量增加[8]。
既往基于传统的分离培养方法对广陈皮中微生物多样性进行研究,但广陈皮中微生物菌群种类繁多,采用传统的方法无法检测出所有微生物,导致低估了菌群数量和多样性[9]。随着现代生物技术的发展和测序成本的降低,高通量测序技术越来越受到国内外学者的关注[10]。如陈聪聪[11]对3个不同产地的广陈皮进行高通量分子测序,发现广陈皮表面存在大量的Bacillus、Pseudomonas、Lactococcus、Enterococcus、Mycobacterium、Arthrobacter;何静[12]对不同产地陈皮表面细菌群落多样性进行研究,发现主要分布有Bacillus、Pseudomonas、Sphingomonas、Methylobacterium、Acinetobacter、Enhydrobacter、Enterobacter;刘丽娜[8]研究4个不同年份广陈皮表面微生物群落多样性,能准确检测到的细菌属有Pseudomonas、Sphingomonas、Methylobacterium、Lactobacillus等,检测到的真菌属有Xeromyces、Alternaria、Symmetrospora、Xerochrysium、Cladosporium、Mortierella、Pleurotus、Chaetomium、Gibberella;张鑫等[13]分析了不同产地陈皮表面真菌群落多样性,发现主要优势真菌是Erythrobasidium、Penicillium、Aspergillus、Rhodotorula和Mycosphaerella。不同学者研究结果不太一致,究其原因是由于这些研究实验材料不一。但未见对陈化过程中陈皮表面细菌、真菌结构的分析报道。
本研究首次采用高通量测序技术对固定采收地点、采样地点、果树树龄、贮藏养护条件下,陈化1~5年的广陈皮表面微生物群落结构及多样性变化进行分析,以期揭示广陈皮陈化过程中微生物群落变化特征及变化规律,为适合陈皮陈化微生物的筛选提供基础,同时为后续优势功能菌的分离筛选,深入研究广陈皮陈化机理提供理论依据。
1 材料与方法
1.1 广陈皮样品的收集
于广东省江门市新会区三江镇新马单村收集陈化1、2、3、4、5年的广陈皮样品(均在同一环境下贮存)各1批,编号依次为Y1~Y5。采集后迅速保存于-80 ℃。实验所用样品经成都中医药大学严铸云教授鉴定为芸香科植物橘的栽培变种茶枝柑(Citrus reticulata cv. ‘Chachi’)的干燥成熟果皮。
1.2 主要试剂和仪器
SW-CJ-2FD型洁净工作台,苏州安泰空气技术有限公司;DZX-50KBS型立式高压灭菌锅,上海申安医疗器械厂;GeneJET 胶回收试剂盒,Thermo Scientific公司;Phusion®High-Fidelity PCR Master Mix with GC Buffer、高效高保真酶,New England Biolabs 公司;Bio-rad T100 梯度 PCR 仪,美国Bio-rad公司;TruSeq DNA PCR-Free Library Preparation Kit建库试剂盒、NovaSeq 6000测序系统,Illumina公司。
1.3 实验方法
1.3.1 基因组DNA提取
用棉签在广陈皮表面擦拭多次,擦拭面积5 cm×5 cm共计用20支棉签。擦拭后,将棉签部分剪下。采用CTAB法提取基因组DNA,琼脂糖凝胶电泳检测DNA浓度,取适量样品入离心管,使用无菌水稀释样品至10- 6 g/L[13]。
1.3.2 PCR扩增与测序
以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物。细菌主要针对16S rDNA的V4区进行扩增,引物为515F(GTGCCAGCMGCCGCGGTAA)和806R(GGACTACHVGGGTWTCTAAT)[14]。真菌扩增区域为ITS1区,引物为ITS1-1F-F (CTTGGTCATTTAGAGGAAGTAA)和ITS1-1F-R(GCTGCGTTCTTCATCGATGC)[14]。使用Phusion® High-Fidelity PCR Master Mix with GC Buffer和高效高保真酶进行PCR,确保扩增效率和准确性[15]。PCR反应体系(30 μL):Phusion Master Mix(2×)15 μL,正反引物(2 μmol/L)各 1.5 μL,gDNA(1 mg/L)10 μL,ddH2O 2 μL。细菌PCR 反应参数:98 ℃预变性1 min;98 ℃ 10 s,50 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。真菌PCR 反应参数:98 ℃ 30 s;98 ℃ 10 s,35个循环;56 ℃ 30 s;72 ℃ 15 s;72 ℃ 7 min。
1.3.3 PCR产物的混样和纯化
PCR产物使用质量分数2%的琼脂糖凝胶进行电泳检测;根据PCR产物浓度进行等浓度混样,充分混匀后使用质量分数2%的琼脂糖凝胶电泳纯化PCR产物,割胶回收目标条带并用胶回收试剂盒回收目标条带。
1.3.4 文库构建和上机测序
使用Illumina 公司的TruSeq DNA PCR-Free Library Preparation Kit建库试剂盒进行文库构建,构建好的文库经Qubit定量和文库检测合格后,用NovaSeq 6000 测序系统进行上机测序。
1.4 数据分析
去除Barcode序列和引物序列,使用FLASH v1.2.7软件进行拼接,得到原始Tags 数据(raw tags)。Raw tags进行严格的过滤处理[16]后参照QIIME v1.9.1[17]的Tags质量控制流程,与物种注释数据库比对剔除嵌合体,得到有效序列。利用UPARSE v7.0.1001软件[18]以97%的一致性将序列聚类为操作分类单元(operational taxonomic units,OTUs)。16S:用MOTHUR方法与SSUrRNA 数据库对OTUs代表序列进行物种注释分析(设定比对阈值为0.8~1)[19]。ITS:用QIIME软件中的BLAST方法与Unit数据库对OTUs代表序列进行物种注释分析。获得分类学信息,对各个分类水平上的群落结构进行统计分析。通过QIIME软件进行Alpha多样性指数分析。利用R软件v2.15.3进行Alpha多样性指数组间差异分析及绘制主成分分析(principal component analysis,PCA)图。利用LEfSe软件对样品进行线性判别分析(linear discriminant analysis,LDA)。
2 结果分析
2.1 样本数据统计
对不同陈化时间广陈皮样品进行微生物群落结构和多样性测序分析质控后细菌共得到1 790 539条有效数据,对有效序列进行聚类,以97%的一致性将序列聚类为OTUs,共得到可分类OTUs数目约为90 033个,范围为1 769~3 616。真菌共得到1 997 205条有效数据,以97%的一致性将序列聚类为OTUs,累计得到可分类OTUs数目约为7 886个,范围在126~435。
2.2 不同陈化时间广陈皮表面微生物多样性分析
Alpha 多样性用于分析样品内微生物群落丰富度和多样性。常用的分析指数包括Shannon、Simpson、Chao1、ACE和Coverage[20]。其中ACE指数和Chao1指数表示群落物种丰富度,Simpson指数和Shannon指数表示群落多样性,Coverage指数表征测序得到的物种覆盖率[21]。样本中细菌的覆盖率均在0.977以上,真菌覆盖率均在0.999以上,表明测序深度均已覆盖到测试样本中的大部分物种,具有可靠的数据质量。
不同陈化时间广陈皮表面细菌的Shannon、Chao1指数相对真菌而言普遍较大,说明广陈皮陈化过程中细菌的物种总数和种类多于真菌。在表1中,陈化2年样品具有最高的Shannon指数(7.67)和Chaol指数(3 632),此时细菌物种总量最高,群落组成最复杂。再者,Shannon指数和Chao1指数在5年陈皮样品中数值最低,分别为5.78和2 082,明显低于其余样品。说明在广陈皮陈化过程中细菌的物种总数和种类呈降低趋势,细菌物种趋向单一化。
表1 不同陈化时间广陈皮表面细菌Alpha多样性指数(n=6)
Table 1 Alpha diversity index of bacterial community on the surface of Citrus reticulata ‘Chachi’ with different aging time (n=6)
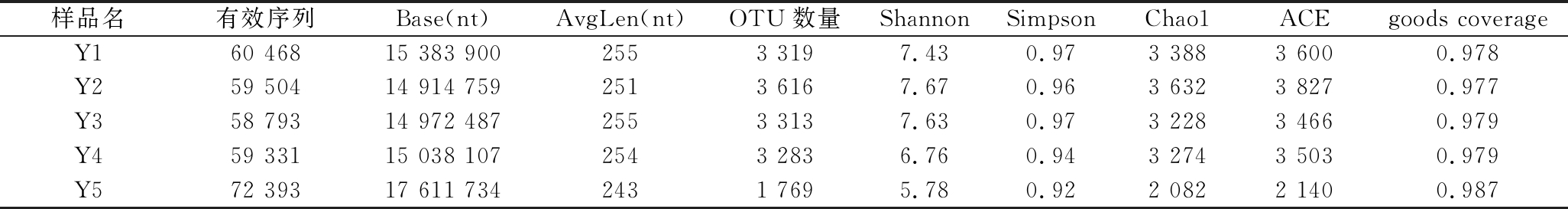
样品名有效序列Base(nt)AvgLen(nt)OTU数量ShannonSimpsonChao1ACEgoods coverageY160 46815 383 9002553 3197.430.973 3883 6000.978Y259 50414 914 7592513 6167.670.963 6323 8270.977Y358 79314 972 4872553 3137.630.973 2283 4660.979Y459 33115 038 1072543 2836.760.943 2743 5030.979Y572 39317 611 7342431 7695.780.922 0822 1400.987
对于真菌,由表2可知,陈化3年样品具有最高的Shannon指数(4.14)和Chao1指数(439),此时真菌物种总量最高,群落组成最复杂。Shannon指数和Chao1指数在2年陈皮样品中数值最低,分别为0.93和129,低于其余样品。在广陈皮陈化过程中,真菌的Shannon指数和Chao1指数变化趋势没有细菌明显,但总体上呈降低趋势。具体见图1。
表2 不同陈化时间广陈皮表面真菌 Alpha 多样性指数(n=6)
Table 2 Alpha diversity index of fungal community on the surface of Citrus reticulata ‘Chachi’ with different aging time (n=6)
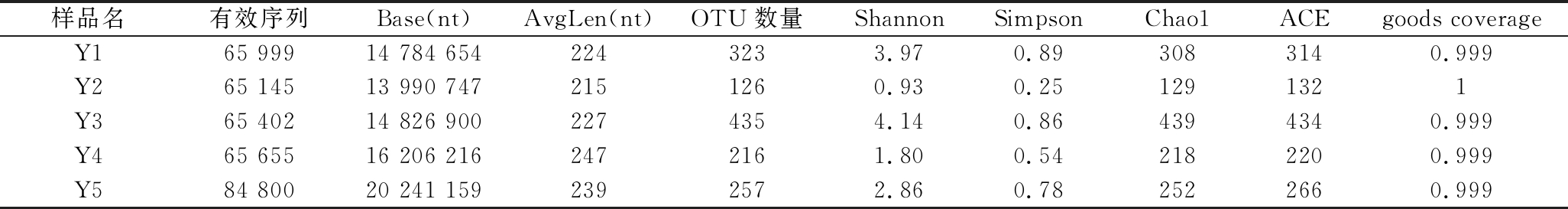
样品名有效序列Base(nt)AvgLen(nt)OTU数量ShannonSimpsonChao1ACEgoods coverageY165 99914 784 6542243233.970.893083140.999Y265 14513 990 7472151260.930.251291321Y365 40214 826 9002274354.140.864394340.999Y465 65516 206 2162472161.800.542182200.999Y584 80020 241 1592392572.860.782522660.999
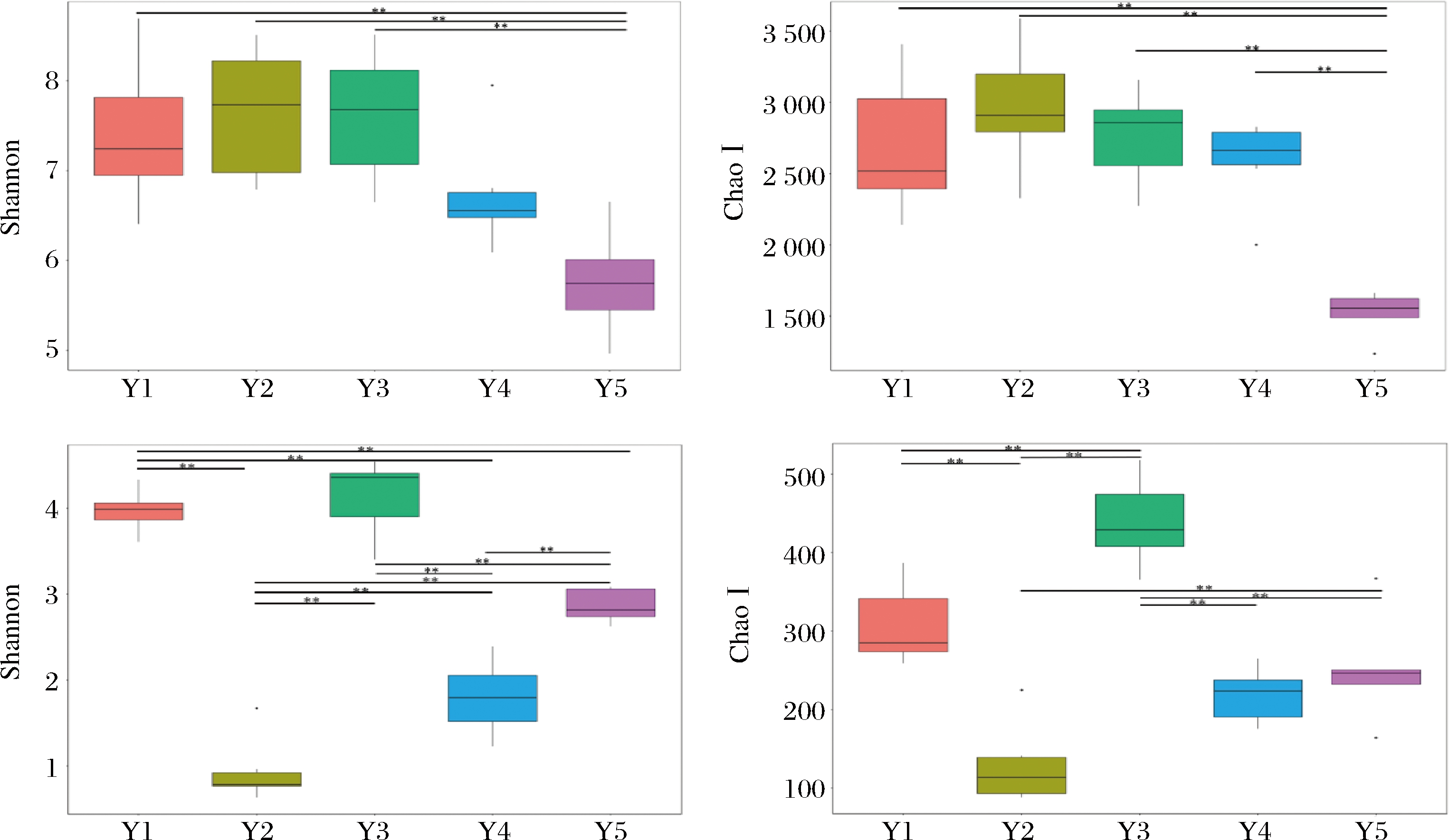
图1 不同陈化时间广陈皮表面细菌和真菌群落多样性(Shannon)及相对丰度(Chao 1)分析**P<0.01
Fig.1 Estimated values of bacterial and fungal community Shannon diversity index (Shannon) and relative abundances (Chao 1) on the surface of Citrus reticulata ‘Chachi’ with different aging time
注:**表示存在极显著差异(P<0.01)
2.3 微生物群落结构组成及其动态变化
2.3.1 不同陈化时间广陈皮微生物门水平群落组成特征
不同陈化时间广陈皮门水平细菌群落组成如图2-a所示,Proteobacteria、Firmicutes、Actinobacteria、Cyanobacteria等为优势细菌。Proteobacteria为广陈皮陈化过程中的绝对优势菌门,不同陈化时间广陈皮所有样本相对丰度均在70%以上。Firmicutes随陈化时间的增加相对丰度总体呈增长趋势,并在陈化2年时相对丰度最高(10.22%)。Cyanobacteria 在广陈皮陈化过程中相对丰度呈波动变化(0.77%~6.22%)。Actinobacteria随陈化时间的增加总体呈降低趋势,在陈化1年时相对丰度最高(7.21%),说明Actinobacteria的细菌较少或基本没有参与陈皮陈化过程。根据高通量测序结果,在门的分类水平上,不同陈化时间广陈皮样品间细菌群落组成基本一致。
不同陈化时间广陈皮门水平真菌群落组成如图2-b所示,Ascomycota、Basidiomycota等为优势真菌。Ascomycota为陈化1~5年广陈皮中主要优势真菌,相对丰度分别为62.17%、8.89%、46.11%、86.06%和52.13%。其次为Basidiomycota,相对丰度分别为13.13%、1.3%、17.94%、0.57%和1.49%。不同陈化时间广陈皮中真菌群落结构一致,只是不同陈化时间广陈皮的群落组成比例有差异。
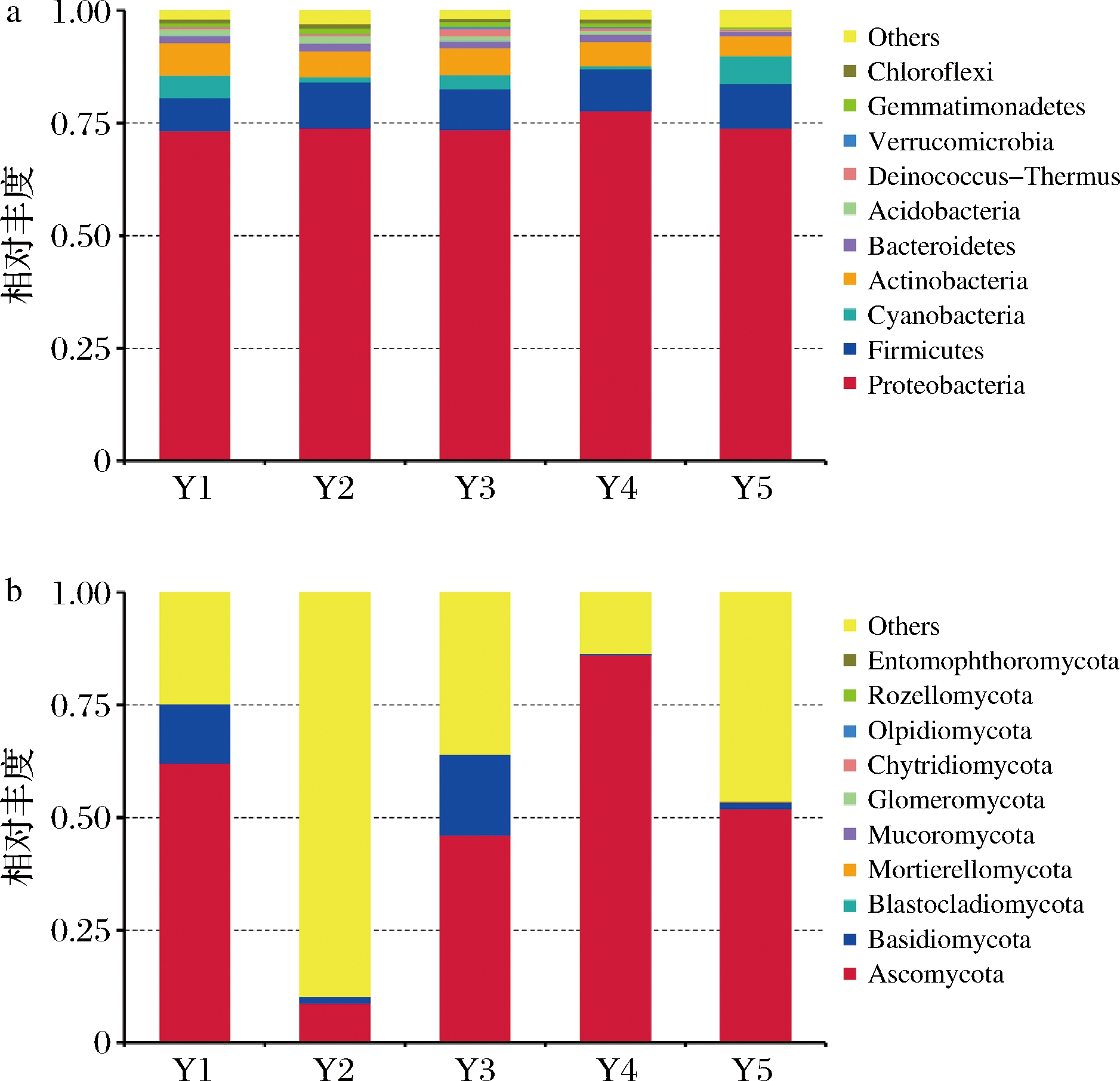
图2 门水平上细菌(a)及真菌(b)样本的群落组成分析
Fig.2 The community abundance analysis of bacterial (a) and fungal (b) samples at phylum level
2.3.2 不同陈化时间广陈皮微生物属水平群落组成
由图3-a可知,在细菌属水平上,主要分布为unidentified_Rickettsiales、Phyllobacterium、Methylobacterium、unidentified_Cyanobacteria、Pseudomonas、Sphingomonas、Stenotrophomonas、Lactococcus、Bacillus等。在已知细菌中,Phyllobacterium、Stenotrophomonas在陈化1~4年时相对丰度呈增加趋势,在陈化4年相对丰度最高,分别为19.58%和10.21%,至陈化5年迅速下降。Pseudomonas随陈化时间的增加相对丰度总体呈增加趋势,在陈化4年时相对丰度最大(13.02%)。Methylobacterium随陈化时间增加相对丰度总体呈降低趋势,在陈化1年时相对丰度最高,为19.56%。Sphingomonas、Bacillus在陈化过程中相对丰度皆呈波动变化,其波动范围分别在3.04%~7.02%、0.71%~1.24%。Lactococcus仅在陈化5年的广陈皮样本中存在,其相对丰度为3.14%。值得关注的是,陈皮表面Methylobacterium、Pseudomonas、Sphingomonas、Stenotrophomonas、Lactococcus、Bacillus与多种挥发油相关[11-12];在对烟叶微生物结构多样性分析中发现,Pseudomonas、Sphingomonas、Stenotrophomonas为广泛分布的优势细菌属[22]。烟叶陈化过程中,菌属间呈此消彼长的关系,细菌优势功能菌群变化与化学成分逐级降解间呈显著相关[23],能够有效降低烟叶中尼古丁等有害成分的含量[24],增加烟叶香气[25]。
在真菌属水平上,主要包括Xeromyces、Cladosporium、Zasmidium、Symmetrospora、Aureobasidium、Fusarium、Aspergillus等(图3-b)。其中Xeromyces在广陈皮陈化过程中相对丰度总体呈增长趋势,陈化4年时相对丰度最高(49.72%)。Zasmidium、Fusarium在陈化1年时相对丰度最高,分别为22.58%和11.53%,随陈化时间的增加,相对丰度迅速下降,至陈化5年时丰度一直较低,说明Zasmidium、Fusarium的真菌菌落较少或基本没有参与陈皮陈化过程;Cladosporium、Symmetrospora、Aureobasidium在陈化1年和3年样本中相对丰度较高,其中Cladosporium、Aureobasidium在陈化3年丰度最高,分别为13.78%、8.48%,Symmetrospora在陈化1年丰度最高(10.87%)。Aspergillus在陈化3年丰度最高,为0.92%。值得注意的是,本课题组前期在陈皮表面鉴定出Aspergillus niger、Aspergillus flavus、Aspergillus fumigatus 3种真菌,属于Aspergillus。可见Aspergillus在陈皮中检出率较高。Aspergillus虽是陈皮在陈化过程中常见的致病菌和霉菌,但大量文献报道[7-8,26] Aspergillus niger等真菌广泛应用于微生物转化,影响陈皮药效物质基础的变化。
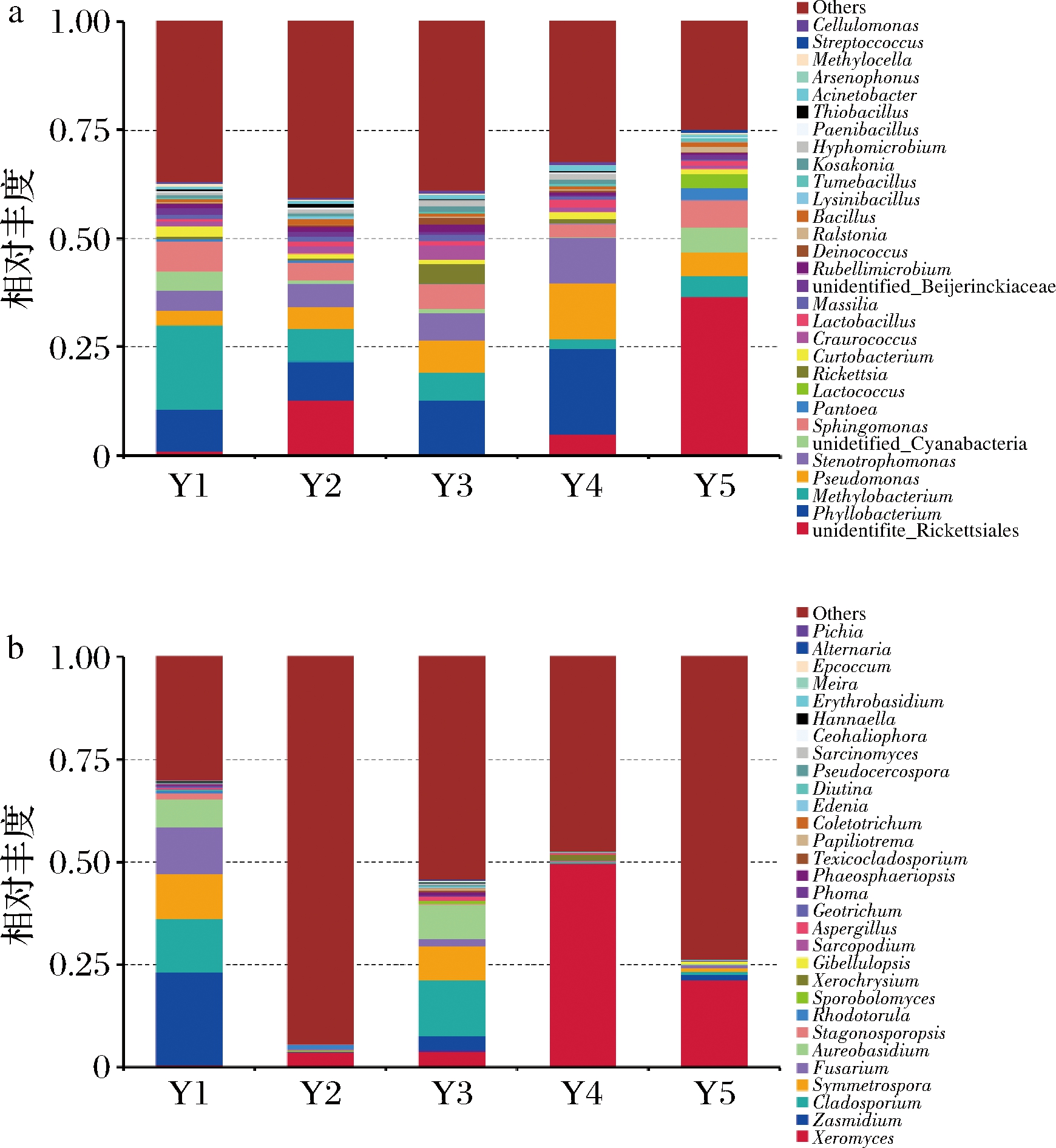
图3 属水平上细菌(a)及真菌(b)样本的群落组成分析
Fig.3 The community abundance analysis of bacterial (a) and fungal (b) samples at the genus level
2.4 不同陈化时间广陈皮样本微生物属水平丰度聚类分析
选取丰度排名前35的细菌属和真菌属,根据每个样本各属的丰度均值,分别对样本之间细菌、真菌相对丰度进行聚类分析,得到热图。热图可以通过板块颜色的相似性及差异性来反映多个样本在各分类水平上群落组成,这样能直观地比较分析不同样本在同一个分类学水平上的的异同[27]。将细菌属水平群落结构热图(图4-a)中Z>1的物种定为优势细菌属,绘制成表(表3)。结合表3和图4-a可知,Methylobacterium,Sphingomonas等在陈化1年样本中聚集较多;Bacillus,Lysinibacillus等在陈化2年样本中分布最为丰富;Rickettsia,Craurococcus等主要分布在陈化3年样本中;Phyllobacterium,Pseudomonas等分布在陈化4年的样本;Pantoea,Lactococcus等在陈化5年时分布较多。
表3 不同陈化时间广陈皮表面优势细菌属分布情况
Table 3 Distribution of dominant bacterial genera in Citrus reticulata ‘Chachi’ with different aging time

样品编号优势细菌属Y1Methylobacterium,Sphingomonas,Curtobacterium,un-identified_Beijerinckiaceae,Methylocella,AcidiphiliumY2Bacillus,Lysinibacillus,Paenibacillus,ThiobacillusY3Rickettsia,Craurococcus,Massilia,Rubellimicrobium,Deinococcus,Kosakonia,Cellulomonas,GemmatirosaY4Phyllobacterium,Pseudomonas,Stenotrophomonas,Lactobacillus,Hyphomicrobium,Acinetobacter,Rhodo-coccusY5unidentified_Rickettsiales,unidentified_Cyanobacteria,Pantoea,Lactococcus,Ralstonia,Tumebacillus,Ar-senophonus,Streptococcus,unidentified_Enterobacteri-aceae
将真菌属水平群落结构热图(图4-b)中Z>1的物种定为优势真菌属,绘制成表(表4)。结合表4和图4-b可知,Zasmidium,Cladosporium等在陈化1年样本中聚集较多;Rhodotorula在陈化2年样本中分布最为丰富;Cladosporium,Aureobasidium等主要分布在陈化3年样本中;Xeromyces,Xerochrysium分布在陈化4年的样本;Gibellulopsis,Erythrobasidium在陈化5年时分布较多。

a-属水平上细菌群落结构热图;b-属水平上真菌群落结构热图
图4 属水平上细菌(a)及真菌(b)群落结构热图
Fig.4 The heatmap of bacterial (a) and fungal (b) community at the genus level
注:横向为样本信息,纵向为物种注释信息,图中左侧的聚类树为物种聚类树;中间热图对应的值为每一行物种相对丰度经过标准化处理后得到的Z值,即一个样品在某个分类上的Z值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
表4 不同陈化时间广陈皮表面优势真菌属分布情况
Table 4 Distribution of dominant fungal genera in Citrus reticulata ‘Chachi’ with different aging time

样品编号优势真菌属Y1Zasmidium,Cladosporium,Symmetrospora,Fusarium,Stagonos-poropsis,Sarcopodium,Geotrichum,Phoma,Pseudocercospora,Hannaella,Meira,Pichia,Setophaeosphaeria,CryptococcusY2RhodotorulaY3Cladosporium,Aureobasidium,Sporobolomyces,Aspergillus,Pho-ma,Phaeosphaeriopsis,Toxicocladosporium,Papiliotrema,Colle-totrichum,Edenia,Diutina,Sarcinomyces,Cephaliophora,Han-naella,Epicoccum,Alternaria,Cercospora,Curvularia,BoeremiaY4Xeromyces,XerochrysiumY5Gibellulopsis,Erythrobasidium
2.5 不同陈化时间广陈皮物种组成相似度分析
应用PCA分析对不同陈化时间广陈皮样品物种组成相似度进行评价,若样品的物种组成越相似,则在PCA图中距离越相近。对不同陈化时间广陈皮样本进行属水平上细菌的PCA分析,研究细菌样本的相似性和差异性。结果显示第1主坐标(PC1)和第2主坐标(PC2)分别解释65.08%和12.02%的群体遗传变异。PCA分析结果显示,陈化5年的样本距离其他样本较远,说明陈化5年的样本群落组成与其他样本相比具有一定的菌群差异(图5-a)。对不同陈化时间广陈皮样本进行属水平上真菌的PCA分析,第1主坐标(PC1)和第2主坐标(PC2)分别占所有差异的44.13%和38.69%。根据距离远近,陈化1年与陈化3年品的距离较小,物种组成相似。陈化2、4、5年样本间距离较小,物种组成接近(图5-b)。
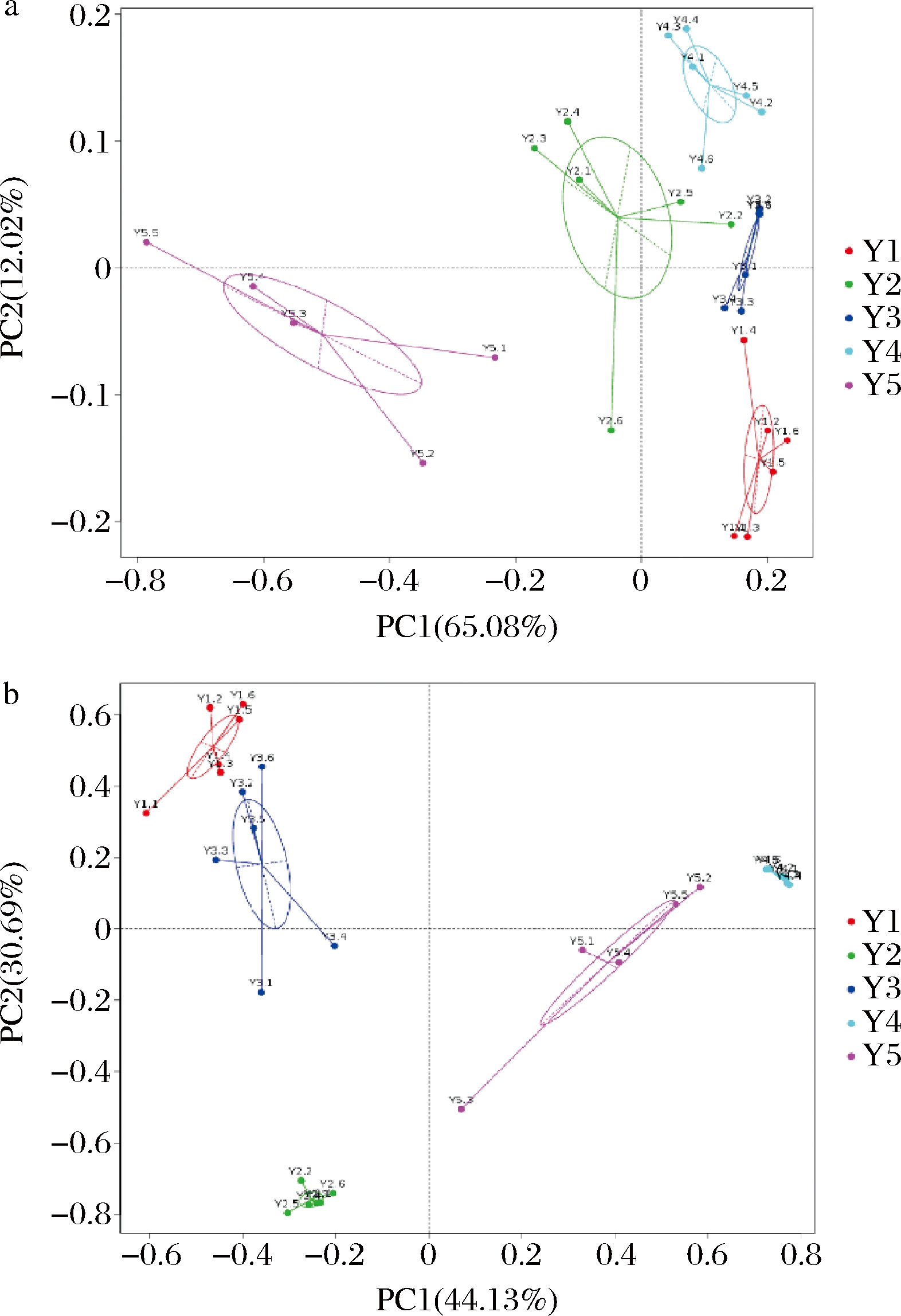
a-细菌样本;b-真菌样本
图5 属水平上细菌及真菌样本的主成分分析
Fig.5 Principal component analysis of bacterial and fungal genesat the genus level
2.6 不同陈化时间广陈皮微生物差异物种分析
LEfSe分析能够在组与组之间寻找具有统计学差异的Biomarker,柱状图种展示了LDA Score>4的物种,即组间具有统计学差异的Biomarker。柱状图的长度代表差异物种的影响大小。在柱状图中,分析了不同陈化时间广陈皮表面由门至属(或种)分类级别的微生物。由图6-a可知,陈化1年广陈皮中细菌丰度差异显著的物种有3种,主要是Rhizobiales、Beijerinckiaceae、Methylobacterium;陈化2年时细菌差异显著物种主要是unidentified Gammaproteobacteria;陈化3年细菌差异显著物种主要是Rickettsiaceae、Rickettsia等7种;陈化4年为Rhizobiaceae、Gammaproteobacteria等11种。陈化5年为unidentified Rickettsiales、Rickettsiales 等16种。可以看出在陈化5年广陈皮中细菌差异显著物种最多。
由图6-b可知,陈化1年广陈皮中真菌丰度差异显著的物种有15种,主要是Dothideomycetes、Capnodiales、Mycosphaerellaceae、Zasmidium等;陈化3年时真菌差异显著物种主要为Basidiomycota、Cystobasidiomycetes等9种;陈化4年真菌显著差异物种主要是Eurotiales、Eurotiomycetes、Aspergillaceae等6种。可以看出在陈化1年广陈皮中真菌差异显著物种最多。

a-细菌样本;b-真菌样本
图6 LDA值分布柱状图
Fig.6 A histogram documenting the distribution of LDA values
3 结论
既往报道主要在陈皮表面致病菌的分离鉴定和生物学特征上,对陈化过程中微生物群落结构及多样性变化研究鲜有报道。结合历代本草对于陈皮陈化时间“隔年”、“2、3年”记载和地理标志产品新会陈皮规定自然陈化3年以上者才为陈皮,该实验首次通过高通量测序技术对固定采收地点、采样地点、果树树龄、贮藏养护等条件陈化1~5年的广陈皮表面微生物群落结构及多样性变化进行研究。
结果表明不同陈化时间广陈皮样本表面细菌、真菌组成结构基本一致,但相对丰度差异明显,优势菌属不同。Proteobacteria、Firmicutes、Actinobacteria、Cyanobacteria是不同陈化时间广陈皮表面主要优势细菌菌门。细菌属水平上,主要分布有unidentified_Rickettsiales、Phyllobacteriu、Methylobacterium、unidentified_Cyanobacteria、Pseudomonas、Sphingomonas、Stenotrophomonas、Lactococcus、Bacillus。随陈化时间的增加,从以Methylobacterium、Sphingomonas为优势转变为以Bacillus, Pseudomonas,Stenotrophomonas,Lactococcus为主。就真菌而言,Ascomycota、Basidiomycota是不同陈化时间广陈皮表面主要优势真菌菌门。真菌属水平上,主要分布有Xeromyces、Cladosporium、Zasmidium、Symmetrospora、Aureobasidium、Fusarium、Aspergillus。随陈化时间的增加,从以Zasmidium、Cladosporium、Symmetrospora、Fusarium为优势转变为以Cladosporium、Aureobasidium、Aspergillus、Xeromyces为主。
本实验通过高通量测序技术,不仅全面揭示参与广陈皮自然陈化过程中的微生物群落及其动态变化规律,并检测出不同陈化时间广陈皮表面优势菌属,为后续优势功能菌的分离筛选及探究陈皮陈化机理提供科学依据。陈皮陈化对提升陈皮质量至关重要,然而陈皮自然陈化过程时间长、成本高。目前,利用微生物转化增加有效成分,改善品质研究较为广泛。因此可以在明确陈皮陈化机理后可以探索是否有“人工催陈”方法来缩短陈化时间、减少经济的损失、资源的浪费[5]。
[1] 国家药典委员会. 中华人民共和国药典:一部[M].北京:中国医药科技出版社,2020:199-200.
China Pharmacopoeia Committee.Pharmacopoeia of the People’s Republic of China (Edition one)[M].Beijing:Chinese Medical Science and Technology Press,2020:199-200.
[2] 鲍倩, 夏荃,潘超美,等.基于Delphi法对广陈皮商品规格等级划分[J].中国实验方剂学杂志,2017,23(22):48-54.
BAO Q,XIA Q,PAN C M,et al.Commercial specification of Citrus reticulata ‘Chachi’ based on delphi method[J].Chinese Journal of Experimental Traditional Medical Formulae,2017,23(22):48-54.
[3] 席秀利, 黄海波,楼步青,等.SCoT分子标记对茶枝柑及近缘种遗传多态性分析[J].中草药,2018,49(10):2 426-2 431.
XI X L,HUANG H B,LOU B Q,et al.Genetic polymorphism analysis of Citrus reticulata Blanco cv.Chachiensis Tanaka and its relatives by SCoT molecular marker[J].Chinese Traditional and Herbal Drugs,2018,49(10):2 426-2 431.
[4] 陶弘景. 本草经集注[M].上海:群联出版社,1955:360-362.
TAO H J.Collective commentaries on the classic of materia medica[M].Shanghai:Group Press,1955:360-362.
[5] 王智磊, 张鑫,刘素娟,等.陈皮“陈久者良”历史沿革和研究现状[J].中华中医药学刊,2017,35(10):2 580-2 584.
WANG Z L,ZHANG X,LIU S J,et al.Historical evolution and research status of Citri reticulatae pericarpium[J].Chinese Archives of Traditional Chinese Medicine,2017,35(10):2 580-2 584.
[6] 杨放晴, 何丽英,杨丹,等.不同陈化时间广陈皮中黄酮类成分的UPLC-Q-Orbitrap HRMS分析[J].中国实验方剂学杂志,2021,27(12):125-132.
YANG F Q,HE L Y,YANG D,et al.Analysis and identification of flavonoids in Citri reticulatae pericarpium with different aging time by UPLC-Q-Orbitrap HRMS[J].Chinese Journal of Experimental Traditional Medical Formulae,2021,27(12):125-132.
[7] 王福, 张鑫,卢俊宇,等.陈皮“陈久者良”之黄酮类成分增加原因探究[J].中国中药杂志,2015,40(24):4 890-4 896.
WANG F,ZHANG X,LU J Y,et al.Study of reason of “the older,the better” of pericarpium Citri reticulatae (PCR) which contributed increase of flavonoids[J].China Journal of Chinese Materia Medica,2015,40(24):4 890-4 896.
[8] 刘丽娜. 基于微生物代谢对陈皮陈化活性物质转化机制的研究[D].湛江:广东海洋大学,2019.
LIU L N.Study on the mechanism of the active substance transformation of Citri reticulatae pericarpium aging based on microbial metabolism[D].Zhanjiang:Guangdong Ocean University,2019.
[9] 叶雷, 闫亚丽,陈庆森,等.高通量测序技术在肠道微生物宏基因组学研究中的应用[J].中国食品学报,2016,16(7):216-223.
YE L,YAN Y L,CHEN Q S,et al.Application of high-throughput sequencing technology in studying matagenomics of intestinal microbiota[J].Journal of Chinese Institute of Food Science and Technology,2016,16(7):216-223.
[10] TURCATTI G,ROMIEU A,FEDURCO M,et al.A new class of cleavable fluorescent nucleotides:Synthesis and optimization as reversible terminators for DNA sequencing by synthesis[J].Nucleic Acids Research,2008,36(4):25.
[11] 陈聪聪. 广陈皮陈化过程中微生物群落多样性解析及代谢物成分变化分析[D].广州:华南理工大学,2017.
CHEN C C.Characterization of microbial community diversity and metabolites changes of pericarpium Citri reticulatae ‘Chachiensis’ during aging process[D].Guangzhou:South China University of Technology,2017.
[12] 何静. 广陈皮细菌群落和植物化学物独特性及其相关性分析[D].广州:华南理工大学,2019.
HE J.The correlation of bacterial community and phytochemical peculiarities of Citri reticulatae pericarpium[D].Guangzhou:South China University of Technology,2019.
[13] 张鑫, 刘素娟,王智磊,等.橘皮表面真菌群落结构多样性分析[J].微生物学通报,2017,44(5):1 089-1 098.
ZHANG X,LIU S J,WANG Z L,et al.Community structure analysis of fungi isolated from citrus surfaces[J].Microbiology China,2017,44(5):1 089-1 098.
[14] WANG X Y,ZHU X G,BI Y L,et al.Dynamics of microbial community and changes of metabolites during production of type Ι sourdough steamed bread made by retarded sponge-dough method[J].Food Chemistry,2020,330:127 316.
[15] AWASTHI M K,CHEN H,WANG Q,et al.Succession of bacteria diversity in the poultry manure composted mixed with clay:studies upon its dynamics and associations with physicochemical and gaseous parameters[J].Bioresource Technology,2018,267:618-625.
[16] BOKULICH N A,SUBRAMANIAN S,FAITH J J,et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nature Methods,2013,10(1):57-59.
[17] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[18] HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Research,2011,21(3):494-504.
[19] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[20] 刘素娟. 陈皮陈化过程“环境因子—真菌—药效物质基础”相关性研究[D].成都:成都中医药大学,2018.
LIU S J.Correlation research of environmental factors,fungi and effective substance in the aging process of Citri reticulatae pericarpium[D].Chengdu:Chengdu University of Traditional Chinese Medicine,2018.
[21] 黎瑶依, 胡小霞,黄永光.茅台镇酱香型白酒酿造环境中真菌菌群多样性分析[J/OL].食品科学:1-11[2021-04-19].http://kns.cnki.net/kcms/detail/11.2206.TS.20200831.0939.012.html.
LI Y Y,HU X X,HUANG Y G.Analysis on the diversity of fungi flora in Maotai-flavor Liquor brewing environment in Maotai Town[J].Food Science:1-11[2021-04-19].http://kns.cnki.net/kcms/detail/11.2206.TS.20200831.0939.012.html.
[22] 伍雪莹. 陈化烤烟烟叶细菌群落鉴定及应用研究[D].广州:华南理工大学,2014.
WU X Y.Identification and application study of bacterial communities on flue-cured tobacco leaves[D].Guangzhou:South China University of Technology,2014.
[23] 周家喜, 喻理飞,张健,等.烟叶陈化过程细菌群落演替特征[J].生态学报,2018,38(21):7 739-7 748.
ZHOU J X,YU L F,ZHANG J,et al.Study on the characteristics of bacterial community succession in tobacco aging[J].Acta Ecologica Sinica,2018,38(21):7 739-7 748.
[24] WANG F,MEN X,ZHANG G,et al.Assessment of 16S rRNA gene primers for studying bacterial community structure and function of aging flue-cured tobaccos[J].AMB Express,2018,8(1):182.
[25] 赵铭钦, 刘云,李芳芳,等.陈化烤烟叶面优势菌的筛选鉴定与其增香效应[J].微生物学报,2009,49(5):625-631.
ZHAO M Q,LIU Y,LI F F,et al.Identification of dominant and fragrance-enhancing microorganisms of tobacco leaves during ripening[J].Acta Microbiologica Sinica,2009,49(5):625-631.
[26] 张鑫, 贺仪,刘素娟,等.陈皮“陈久者良”原因探究[J].食品科技,2017,42(1):90-95.
ZHANG X,HE Y,LIU S J,et al.The reason of “the older,the better” of Citri reticulatae pericarpium (PCR)[J].Food Science and Technology,2017,42(1):90-95.
[27] LI K K,ZHUO C,TENG C Y,et al.Effects of Ganoderma lucidum polysaccharides on chronic pancreatitis and intestinal microbiota in mice[J].International Journal of Biological Macromolecules,2016,93:904-912.