当前,人们对化石燃料的过度需求和开发引起了许多问题,从能源短缺、环境污染再到不可持续性发展,因此,亟需寻找绿色、清洁、可再生能源作为替代[1]。生物质资源是指利用环境中大气、土壤、水等进行光合作用而产生的可再生有机物质,包括农作物、树木等植物及其废弃物、畜禽粪便等有机废弃物,例如草木、秸秆。生物质中含有丰富的纤维素、半纤维素、木质素等有机物[2],是生产清洁能源的主要原料[3]。因此,生物质资源将成为石油工业的重要替代品。我国作为农业大国,粮食作物产量大,而农业废弃物(如秸秆)年产量高达7亿t[4]。但目前,大部分秸秆废弃物被就地焚烧,这不仅对环境造成了严重的污染,同时也是生物质资源的极大浪费[5]。
在生物质资源的利用过程中发现,木质素作为天然的物理屏障,将纤维素紧紧包裹,不利于纤维素的开发和利用,极大的降低纤维素生产能源的效率。近年来,学者们开始关注植物生物质中木质素的降解。目前,木质纤维素生物质的降解方法主要包括物理处理(机械粉碎,热解等)、物理化学处理(二氧化碳爆炸,蒸汽爆炸等)、化学处理(臭氧分解,碱水解等)以及生物法处理(微生物、酶的降解)。但物理和化学法降解木质纤维素通常需要昂贵的设备,消耗更多的能量甚至会产生有毒有害的废弃物。与此相比微生物介导的生物催化过程因其反应条件温和、设备简单,在降解木质素方面具有较高的优势,为木质素的利用提供了更具体有效的替代方法[6]。本文主要对木质素降解酶系及其作用机制进行综述,尤其总结了基于组学技术挖掘的木质素降解酶,旨在全面深入的了解木质素降解酶系,对生物质资源的高效利用奠定基础。
1 木质素的结构和性质
木质纤维素是植物生物质中细胞壁的主要成分,其结构如图1所示,包括木质素、纤维素、半纤维素、果胶等[7]。木质素由复杂的芳烃组成,其单元结构是愈创木基(G型)、对羟基苯基(H型)和丁香基(S型),3种结构单元通过不同的化学键相连构成无规则的3维网状结构,因此木质素结构复杂,难溶于水,不易降解。木质素降解的意义在于:(1)木质素包裹在纤维素和半纤维素的外部,降解木质素可以增加纤维素和半纤维素的利用率,因此木质素的有效解聚是打破木质纤维素物理屏障对其进行生物利用的关键过程[8];(2)木质素是生物圈中含量最丰富的芳香生物聚合物,降解后可用来生产香草酸等芳香族化合物类高附加值化学品[9];(3)木质素结构中含有甲氧基、羰基、醇羟基等,使其具有抗菌、吸附、抗氧化等潜力,可用来生产木质素基树脂材料、木质素基吸附材料。对木质纤维素的生物降解进行有效了解和控制,将减轻因秸秆焚烧或副产物丢弃造成的环境污染,同时木质素也可作为生产可再生化合物和高级生物燃料的原料,以满足对化石燃料衍生能源和材料日益增长的需求。
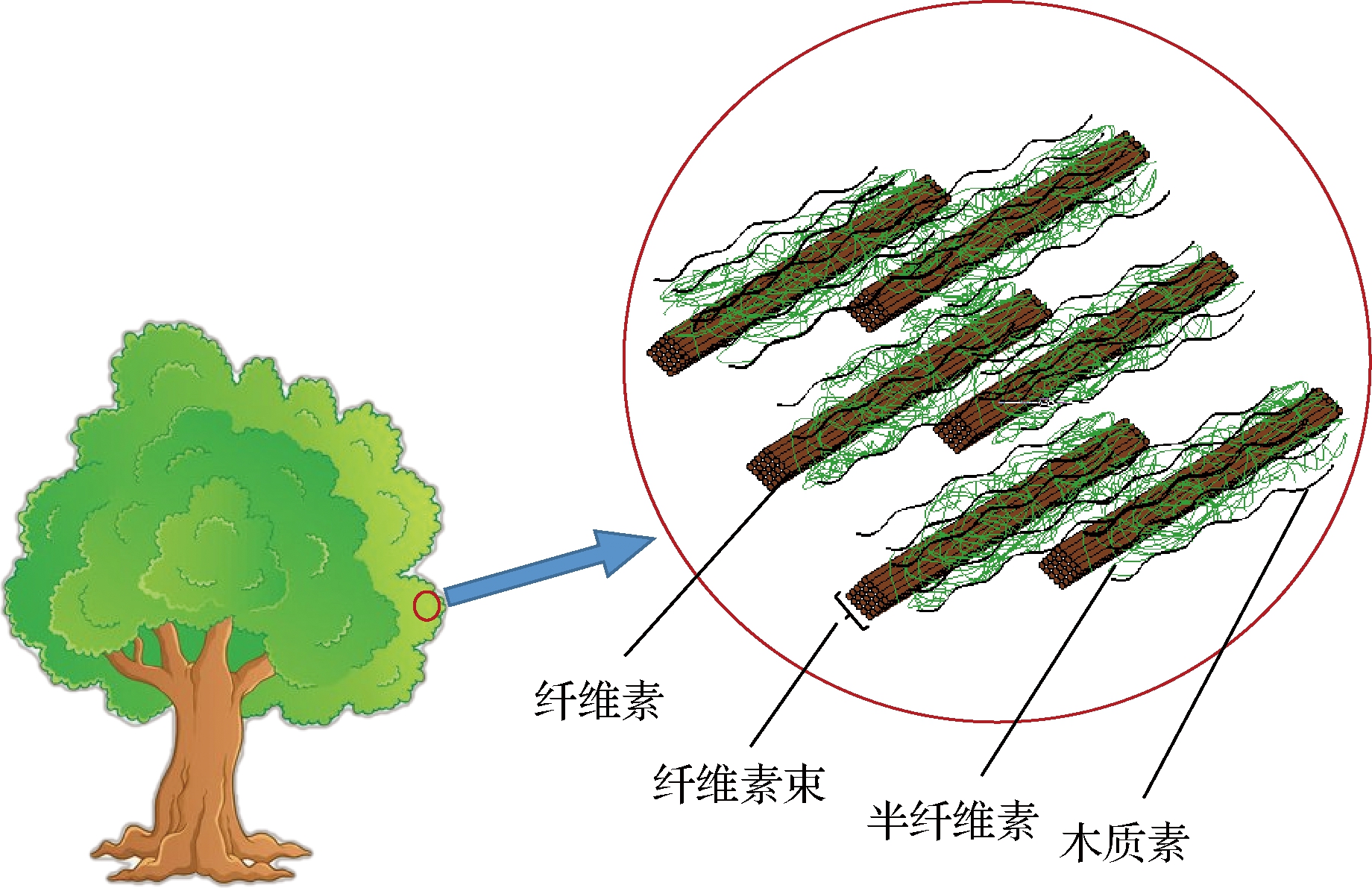
图1 植物生物质结构
Fig.1 Structure of plant biomass
2 木质素降解酶系的研究
细菌、真菌和放线菌是自然界中能够降解木质纤维素的微生物[10]。在木质素的降解中,真菌因其具有强大的木质素降解酶系统而起主要作用,例如可产生胞外木质素降解酶的白腐菌[11]。与真菌相比,细菌因其生长快、结构简单、具有较强的适应性,分泌的酶热稳定性强以及pH耐受范围广等优点,引起了越来越多研究者的注意[12]。除有氧环境外,木质纤维素还被厌氧细菌降解和转化。低氧浓度真菌降解木质素具有抑制作用,但是细菌表现出对厌氧条件较强的适应性,这也是细菌降解木质素成为近年来研究热点的原因之一[13]。
木质素降解通常可以得到高产率的芳香族化合物,尤其是木质素单体。木质素解聚过程主要包括2个阶段:天然木质素的解聚和所得异质芳烃的矿化,在不同的降解阶段起主要作用的酶不同[14]。
2.1 木质素解聚过程相关酶
木质素过氧化物酶(lignin peroxidase,LiP)、锰过氧化物酶(Mn-dependent peroxidase,MnP)和多功能过氧化物酶(versatile peroxidase,VP)在木质素解聚过程中发挥着重要的作用[15]。3种木质素分解过氧化物酶具有相当高的序列同一性,并与二硫键和钙离子共享相似的螺旋形折叠,以实现结构完整性。LiP具有高氧化还原电位,可氧化高达90%的木质素非酚结构。它还具有氧化多种芳香族化合物的能力,因此具有在木质素酶促降解中的作用。除了非酚类底物的特征性氧化外,LiP还显示出氧化多种酚类化合物的能力。此外,VP结合了MnP和LiP的催化特性,具有广泛的木质素分解能力。VP活性的多功能性可能归因于其独特的分子结构。VP氧化高氧化还原电位化合物的能力与暴露的催化色氨酸有关,该色氨酸通过远端电子转移至血红素而在酶表面形成自由基[16]。MnP分子结构与LiP相似,也是一种糖蛋白,大多数的白腐菌和土壤垃圾降解菌具有合成MnP的能力,MnP具有底物特异性,主要催化酚醛类的Cα—Cβ键裂解,Cα—H键氧化和烷基-芳基的C—C键裂解。MnP表现出对Mn2+的依赖,MnP催化通常是将Mn2+转化为Mn3+,高氧化活性的Mn3+可将大量的酚类底物氧化[17]。
与上述木质素分解过氧化物酶不同,漆酶(laccase,Lac)是一种氧化酶,可以促进木质素分子的裂解,包括芳香环的打开,烷基-芳基的破坏和酚羟基的增加,完整的催化反应是电子转移氧化,涉及4个连续的单电子氧化[18]。一般而言,漆酶蛋白质分子中均含有4个铜离子,分为3种不同类型:1个Ⅰ型铜,1个Ⅱ型铜,2个Ⅲ型铜[19-20]。漆酶氧化木质素时首先从木质素中提取1个电子,利用双氧作为电子受体,导致底物分子产生自由基,产生的自由基不稳定,并且会发生聚合或解聚反应,从而导致木质素降解。产生的大量自由基经历非酶反应[21],最终,木质素大分子解聚为单体,产生了大量的芳香族化合物,漆酶可以将其用作底物以继续进行酶促水解[22]。
在细菌中,最早被鉴定的木质素降解酶是来自红球菌Rhodococcus jostii RHA1的过氧化物酶DyPB[23]。DyP型过氧化物酶具有使染料脱色和氧化多种酚类底物的活性[24]。基于序列比对建立系统发育树后,根据一级结构的同源性将DyPs分为ABCD 4个亚型,其中A~C型DyP主要在细菌中发现,D型大多存在于真菌中。DyPs结构通常表现出二聚体铁氧还蛋白样折叠,构成具有由α-螺旋外围连接的4个β链的反平行β-折叠[3, 25]。DyPs的特异性比较低,主要在酸性pH下具有活性,显示出非常广泛的底物分布,比如一些典型的过氧化物酶底物:ABTS、刚果红;DyPs能够降解不同的染料,尤其是一些不易降解的蒽醌染料;此外还能降解单酚化合物、β-胡萝卜素,木质素模型化合物[26-27]。YU等[28]从Saccharomonospora viridis中克隆了编码SviDyP的基因,在大肠杆菌中异源表达,然后纯化,根据系统发育分析,SviDyP属于DyPs的A型,与来自A型的其他DyPs相比,纯化的SviDyP具有显著的广泛底物特异性,SviDyP重组蛋白显示出显著的耐热性和耐碱性,并且可以直接有效地降解木质素。
2.2 木质素矿化过程相关酶
木质素解聚衍生的异质低分子质量芳烃的矿化作用主要受细菌的控制[14]。降解木质素的细菌通常形成聚合体共同降解木质素,Sphingobium sp.SYK-6具有同化各种联芳基的能力[29],包括β-芳基醚,联苯甲基以及单芳基如阿魏酸酯、香兰素,在细菌分解代谢途径中,各种芳香族化合物通过各种酶促反应例如醚裂解、双键裂解、侧链裂解、脱羧和氧化作用分别转移到香草醛、丁香酸酯和对羟基苯甲酸酯中,然后这些化合物都转化为儿茶酚衍生物,芳环被裂解产物通过几种不同的途径进入三羧酸循环[30-31]。
细菌β-醚酶系统可以降解木质素中的β-芳基醚结构(β-O-4)。β-醚酶家族首先在Sphingobium sp.SYK-6中鉴定出来,该细菌具有降解含有β-O-4结构的木质素二聚体的能力,并生成香草酸、丁香酸等中间体化合物[32]。Sphingobium sp.SYK-6对木质素中β-O-4键的降解包括:Cα脱氢酶(LigD、LigL、LigO、LigN)、醚酶(LigE、LigF、LigP)和谷胱甘肽裂解酶(LigG)。LigDFG系统催化以下反应主要分为3步:(1)在NAD+存在下,LigD将底物中Cα的羟基氧化形成酮基,使木质素底物由醇氧化为相应的酮;(2)LigF通过还原型谷胱甘肽攻击Cβ而破坏醚键;(3)谷胱甘肽转移酶LigG在另一种还原型谷胱甘肽的辅助下,从底物中释放出氧化的二聚体谷胱甘肽和芳香族单体,生成最终产物。在β-芳基醚催化途径中,需要多种Lig酶共同作用才能将木质素中的β-醚键断裂。其中,LigL、LigO、LigN和LigD作用相似,LigE、LigP也能催化与LigF相同的反应。
芳香族裂解双加氧酶代表一组细胞内酶,主要将木质素衍生的芳香族化合物解构为线性形式,可以进一步代谢为三羧酸循环中间体。因此,双加氧酶被认为是酶降解木质素利用芳香族化合物作为碳源和能源的重要组成部分。儿茶酸双加氧酶和儿茶酚双加氧酶是最常见的催化双氧依赖性环裂变的酶。
2.3 其他相关酶类
除Lip、MnP和漆酶以外,也有一些其他酶类与木质素降解过程密切相关。芳醇氧化酶(aryl-alcohol oxidase,AAO)是一种提供细胞外H2O2的酶,在酶促木质素解聚,特别是在木质素聚合物的氧化中起着重要作用。AAO底物可包括木质素衍生的化合物和芳香族真菌代谢产物,还可减少木质素降解过程中漆酶产生的苯氧基,防止木质素在酶的作用下聚合[33]。AAO能够将芳香族醇转化为相应的醛,并提供细胞外H2O2来降解木质素。在几种降解木质素的真菌中,从1株Trichoderma reesei中检测到了AAO活性[34]。RASHID等[35]从Sphingobacterium sp.T2 鉴定出2种细胞外锰超氧化物歧化酶(superoxide dismutase,SOD)(MnSOD1和MnSOD2),催化双氧与活性氧超氧化物和过氧化物的相互转化。它们以重组形式对有机溶剂木质素的氧化均具有高活性,产生多种反应产物,并且在存在双氧而不是H2O2的情况下Sphingobacterium sp.T2具有更高的木质素氧化活性。
3 组学在木质素生物降解酶系挖掘中的应用
3.1 基因组学在木质素降解过程中的应用
基因组学是应用DNA的重组、测序以及生物信息学的组装对基因的功能和结构进行分析。全基因组测序可以将木质素降解体系中含有的多种微生物进行分析[36]。LIU等[37]通过PacBio测序技术对Trometes trogii S0301基因组进行了测序,并进行高质量注释,确定T.trogii S0301具有潜在的环境木质素降解和适应能力,为研究Trametes属真菌对木质素的降解提供有价值的基因组资源。KUMER等[38]对Pandoraea sp.ISTKB进行基因组学分析揭示了负责木质素降解和聚羟基脂肪酸酯(polyhydroxyalkanoates,PHA)合成的各种潜在基因的存在,确定了木质素降解的主要途径,包括芬顿反应以及辅酶A介导的苯乙酸和苯甲酸酯的降解。RIYADI等[39]测定Streptomyces sp.S6的基因组草图显示细菌过氧化物酶的存在(例如过氧化氢酶、谷胱甘肽过氧化物酶和DyP型过氧化物酶),并且可参与在木质素的初始解聚阶段,基因特征表明Streptomyces sp.S6具有碱木素降解能力,同时可生产各种低分子质量木质素衍生的化合物,具有明显的酶促活性(LiP、Lac和AAO)。宏基因组是将环境中的所有木质素降解微生物的遗传信息作为一个整体直接提取基因组,将基因组学原本对于单个木质素降解微生物的研究扩大到与环境的研究。MORAES等[40]进行了LigMet的基因组组装和整体功能注释,从宏基因组数据集重建基因组草图,在LigMet中鉴定出与木质素降解代谢途径相关的几种微生物和酶,并且分析了木质素降解和香兰素生产所涉及的生物催化剂和途径。ZHU等[41]使用PacBio RS II平台通过SMRT方法获得了Comamonas serinivorans SP-35的全基因组序列,鉴定出94个可能与木质素降解和代谢有关的基因,并且发现可能与β-芳基醚的裂解有关的酶基因,预测了菌株C.serinivorans SP-35的芳香族中间代谢物的代谢途径(表1)。
3.2 转录组学在木质素降解过程的应用
转录组学在木质纤维素降解中的应用主要通过先提取能够有效降解木质纤维素的微生物样品的总RNA,将其反转录成cDNA后进行高通量测序与生物信息学分析[42]。转录组学技术已经在堆肥、青贮、动物瘤胃及肠道等环境微生物研究领域加以应用。LIU等[37]从T.trogii S0301菌丝体中提取总RNA进行转录组分析,发现大量的CAZymes编码基因显示出下调趋势,结合基因组与酶活分析探索木质纤维素降解酶及其编码基因对木质纤维素底物的反应,发现在葡萄糖耗尽的同时,木质素降解酶与MnP、LiP,纤维素酶和木聚糖酶活性同时增加。吴小峰[43]以麝牛为试验动物,采用转录组学技术去除rRNA的Ribozero RNA进行高通量测序,研究真菌、原虫、细菌和古细菌等微生物的编码基因表达以及对小黑麦秸秆中木质纤维素的降解作用,探讨了秋季麝牛瘤胃对小黑麦秸秆木质纤维素的降解机制(表1)。
利用转录组测序技术对木质纤维素降解微生物代谢进行研究,可以说明其基因是否表达以及表达量的多少,确定木质纤维素降解微生物是否能产生所需的降解酶,填补了宏基因组学中仅能说明木质纤维素降解微生物中是否存在产生木质纤维素降解酶的基因,而不能对基因是否转录情况进行分析的研究空间。
3.3 蛋白质组学在木质素降解过程的应用
对木质纤维素降解的传统研究是先筛选出降解能力较强的菌株,再对培养基进行优化,使相关基因过表达得到足量的目标产物,最终有效降解木质素,但这种方法无法全面了解和掌握木质素降解过程的酶系组成以及变化情况。蛋白质组学可以分析木质素降解过程中的酶系组成和表达量,为全面深入地研究酶对木质素的降解途径和降解机制提供了新方法。
在木质素降解方面可以利用生物质谱分析及绝对定量技术(isobaric tag for relative and absolute quantification,iTRAQ)找差异蛋白表达,并对其功能进行分析,例如对细菌、真菌的分泌蛋白组进行分析以确定木质素降解过程中的相关酶类以及模型化合物。KUMER等[38]应用无标记的定量蛋白质组学方法来鉴定香草酸和碱木素的表达谱,利用蛋白质组学分析发现了DyP型过氧化物酶、乙醇酸氧化酶、醛氧化酶、漆酶、双加氧酶、谷胱甘肽依赖性醚酶、脱氢酶以及各种还原酶等功能活跃,确定它们在木质素或芳香族化合物的降解中起重要作用。冯茜[44]采用同位素的相对和绝对定量技术探究Aspergillus fumigatus G-13降解3种木质素模型化合物的产酶规律,鉴定得到了41个参与木质纤维素降解的酶,筛选出酶活表达差异最大的两组进行蛋白质组学分析,确定差异蛋白主要富集于NADH-泛醌氧化还原酶、菌丝过氧化氢酶相关蛋白质,且主要富集于碳源和能量、核苷酸代谢相关途径。ADAV等[45]利用不同的天然木质纤维素生物质作为主要碳源,以基于相对和绝对定量的蛋白质组学方法,以及高通量等压标记进行纤维素水解蛋白、半纤维素水解蛋白、木质素解聚氧化还原酶蛋白、肽酶和其他蛋白质的鉴定和绝对定量的分析,以量化比较木质纤维素分解蛋白及其可变的底物诱导的表达。RASHID等[35]对来自Sphingobacterium sp.T2细胞外部分的蛋白质组学分析鉴定出2种细胞外锰超氧化物歧化酶,它们以重组形式对有机溶剂木质素的氧化具有很高的活性,产生多种反应产物,据此提出了锰超氧化物歧化酶与木质素相互作用的2种可能假设(表1)。
表1 组学技术在研究木质素降解酶中的进展
Table 1 Progress of omics technology in the study of lignin degrading enzymes
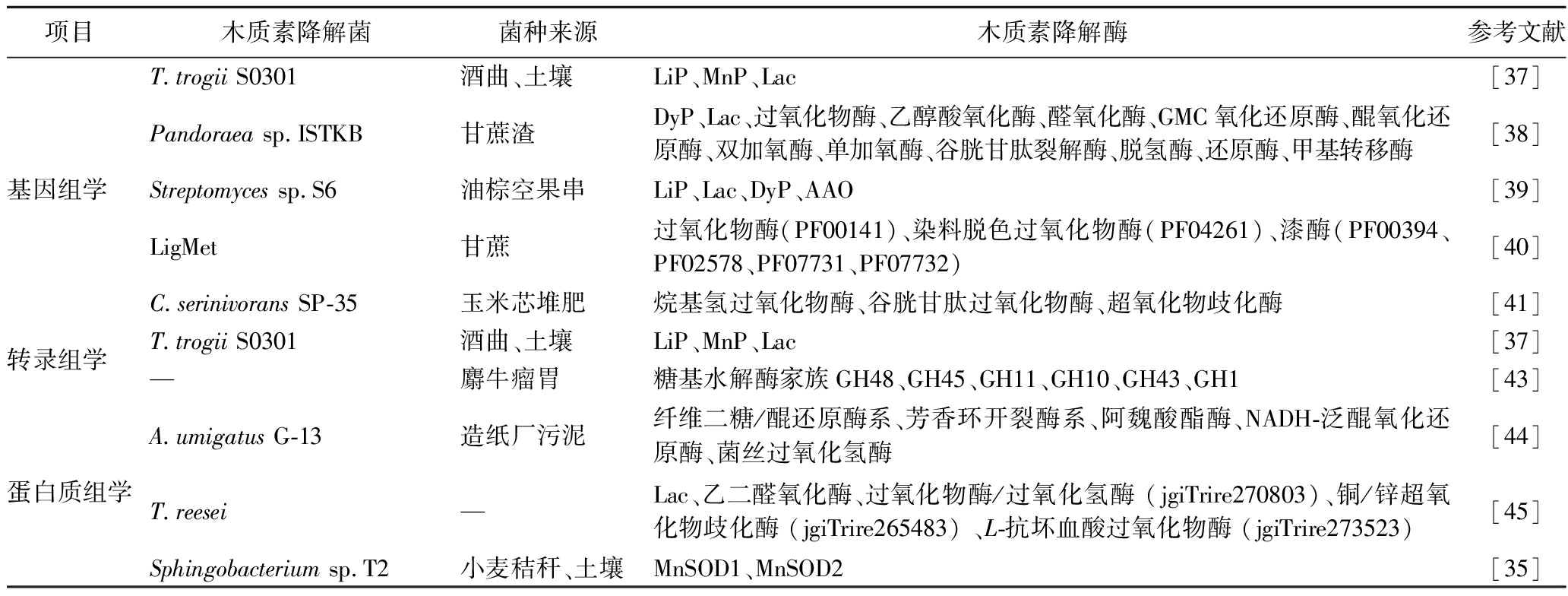
项目木质素降解菌菌种来源木质素降解酶参考文献基因组学 T.trogii S0301酒曲、土壤LiP、MnP、Lac[37]Pandoraea sp.ISTKB甘蔗渣DyP、Lac、过氧化物酶、乙醇酸氧化酶、醛氧化酶、GMC氧化还原酶、醌氧化还原酶、双加氧酶、单加氧酶、谷胱甘肽裂解酶、脱氢酶、还原酶、甲基转移酶[38]Streptomyces sp.S6油棕空果串LiP、Lac、DyP、AAO[39]LigMet甘蔗过氧化物酶(PF00141)、染料脱色过氧化物酶(PF04261)、漆酶(PF00394、PF02578、PF07731、PF07732)[40]C.serinivorans SP-35玉米芯堆肥烷基氢过氧化物酶、谷胱甘肽过氧化物酶、超氧化物歧化酶[41]转录组学 T.trogii S0301酒曲、土壤LiP、MnP、Lac[37]—麝牛瘤胃糖基水解酶家族GH48、GH45、GH11、GH10、GH43、GH1[43]蛋白质组学A.umigatus G-13造纸厂污泥纤维二糖/醌还原酶系、芳香环开裂酶系、阿魏酸酯酶、NADH-泛醌氧化还原酶、菌丝过氧化氢酶[44]T.reesei—Lac、乙二醛氧化酶、过氧化物酶/过氧化氢酶 (jgiTrire270803)、铜/锌超氧化物歧化酶 (jgiTrire265483) 、L-抗坏血酸过氧化物酶 (jgiTrire273523)[45]Sphingobacterium sp.T2小麦秸秆、土壤MnSOD1、MnSOD2[35]
注:-表示该项在对应文献中未注明
4 总结与展望
木质纤维素通过酶的降解可以产生一系列芳香族化合物,能够取代诸如多种石油基化合物高价值药物和化妆品等贵重物品的新型平台化学品。通过对木质纤维素降解体系的研究,可以探索木质素衍生芳香族化合物生产的新途径及关键酶类,扩大木质素的应用范围,为实现木质纤维素生物降解和发酵的产业化奠定基础。
[1] WANG W Y, ZHANG C, SUN X X, et al.Efficient environmentally-friendly and specific valorization of lignin:Promising role of non-radical lignolytic enzymes[J].World Journal of Microbiology & Biotechnology, 2017, 33(6):1-14.
[2] 郑娟善, 张剑搏, 梁泽毅, 等.瘤胃微生物对木质纤维素降解的研究进展[J].动物营养学报, 2020, 32(5):2 010-2 019.
ZHENG J S, ZHANG J B, LIANG Z Y, et al.Research progress on the degradation of lignocellulose by rumen microorganisms[J].Chinese Journal of Animal Nutrition, 2020, 32(5):2 010-2 019.
[3] LI C, CHEN C, WU X F, et al.Recent advancement in lignin biorefinery:With special focus on enzymatic degradation and valorization[J].Bioresource Technology, 2019, 291:121 898.
[4] 杜洋. 秸秆焚烧现状和秸秆利用途径探讨及前景展望[J].现代农业, 2020(7):90-91.
DU Y.Discussion on the status quo of straw burning and the ways of straw utilization and prospects[J].Modern Agriculture, 2020(7):90-91.
[5] 钟平, 张超旭, 王丽, 等.秸秆资源综合利用研究[J].现代农业, 2020(6):4-5.
ZHONG P, ZHANG C X, WANG L, et al.Research on comprehensive utilization of straw resources[J].Modern Agriculture, 2020(6):4-5.
[6] 李凤莲. 农用有机肥料资源分析与开发利用[J].农业技术与装备, 2017(5):33-35.
LI F L.Analysis and development of agricultural organic fertilizer resources[J].Agricultural Technology and Equipment, 2017(5):33-35.
[7] KRICKA W, FITZPATRICK J, BOND U.Metabolic engineering of yeasts by heterologous enzyme production for degradation of cellulose and hemicellulose from biomass:A perspective[J].Frontiers in Microbiology, 2014, 5:174.
[8] YADAV M, PARITOSH K, PAREEK N, et al.Coupled treatment of lignocellulosic agricultural residues for augmented biomethanation[J].Journal of Cleaner Production, 2019, 213:75-88.
[9] WAN C X, LI Y B.Fungal pretreatment of lignocellulosic biomass[J].Biotechnology advances, 2012, 30(6):1 447-1 457.
[10] 谢长校, 孙建中, 李成林, 等.细菌降解木质素的研究进展[J].微生物学通报, 2015, 42(6):1 122-1 132.
XIE C X, SUN J Z, LI C L, et al. Exploring the lignin degradation by bacteria[J]. Microbiology China, 2015, 42(6):1 122-1 132.
[11] 乔悦, 甘洪宇, 李响, 等.木质素降解技术研究进展[J].化工科技, 2019, 27(4):84-88.
QIAO Y, GAN H Y, LI X, et al.Research on degradation of lignin[J].Science & Technology in Chemical Industry, 2019, 27(4):84-88.
[12] 熊乙, 杨富裕, 倪奎奎, 等.微生物在木质纤维素降解中的应用进展[J].草学, 2019(5):1-7.
XIONG Y, YANG F Y, NI K K, et al. Progress in application of microorganisms in lignocellulosic degradation[J]. Journal of Grassland and Forage Science, 2019(5):1-7.
[13] 杨茜, 李维尊, 鞠美庭, 等.微生物降解木质纤维素类生物质固废的研究进展[J].微生物学通报, 2015, 42(8):1 569-1 583.
YANG Q, LI W Z, JU M T, et al. Advances in microbial degradation of lignocelluloses biomass solid waste-a review[J]. Microbiology China, 2015, 42(8):1 569-1 583.
[14] KAMIMURA N, SAKAMOTO S, MITSUDA N, et al.Advances in microbial lignin degradation and its applications[J].Current Opinion in Biotechnology, 2019, 56:179-186.
[15] HE J, HUANG C X, LAI C H, et al.The effect of lignin degradation products on the generation of pseudo-lignin during dilute acid pretreatment[J].Industrial Crops & Products, 2020, 146:112 205.
[16] S EZ-JIMÉNEZ V, BARATTO M C, POGNI R, et al.Demonstration of lignin-to-peroxidase direct electron transfer.A TRANSIENT-STATE KINETICS, DIRECTED MUTAGENESIS, EPR AND NMR STUDY.[J].Journal of Biological Chemistry, 2015, 290(51):30 268.
EZ-JIMÉNEZ V, BARATTO M C, POGNI R, et al.Demonstration of lignin-to-peroxidase direct electron transfer.A TRANSIENT-STATE KINETICS, DIRECTED MUTAGENESIS, EPR AND NMR STUDY.[J].Journal of Biological Chemistry, 2015, 290(51):30 268.
[17] 洪伟杰. 木素过氧化物酶和乙二醛氧化酶的共固定化及在手性合成中的应用[D].杭州:浙江工业大学,2006.
HONG W J.Study on coimmobilization of lignin peroxidase and glyoxal oxidase and application in asymmetric synthesis[D].Hangzhou:Zhejiang University of Technology, 2006.
[18] SU J, FU J J, WANG Q, et al.Laccase:A green catalyst for the biosynthesis of poly-phenols[J].Critical Reviews in Biotechnology, 2018, 38(2):294-307.
[19] 孙正茂, 肖克宇.真菌木质素降解酶系的研究进展[J].广东饲料, 2006,15(2):41-43.
SUN Z M, XIAO K Y.Research progress of fungal lignin degrading enzyme system[J].Guangdong Feed, 2006,15(2):41-43.
[20] LIU C J, ZHANG W J, QU M R, et al.Heterologous expression of laccase from Lentinula edodes in Pichia pastoris and its application in degrading rape straw[J].Frontiers in Microbiology, 2020, 11:1 086.
[21] 鲁伦慧. 农业废物堆肥中木质素降解功能微生物群落结构研究[D].长沙:湖南大学,2014.
LU L H.Researches on the ligninolytic functional microbial communities constructions during agricultural waste composting[D].Changsha:Hunan University, 2014.
[22] ZHANG S T, XIAO J L, WANG G, et al.Enzymatic hydrolysis of lignin by ligninolytic enzymes and analysis of the hydrolyzed lignin products[J].Bioresource Technology, 2020, 304:122 975.
[23] BUGG T D H, WILLIAMSON J J, RASHID G M M.Bacterial enzymes for lignin depolymerisation:New biocatalysts for generation of renewable chemicals from biomass[J].Current Opinion in Chemical Biology, 2020,55:26-33.
[24] YOSHIDA T, SUGANO Y.A structural and functional perspective of DyP-type peroxidase family[J].Archives of Biochemistry and Biophysics, 2015, 574:49-55.
[25] LINDE D, RUIZ-DUE AS F J, FERN
AS F J, FERN NDEZ-FUEYO E, et al.Basidiomycete DyPs:Genomic diversity, structural-functional aspects, reaction mechanism and environmental significance[J].Archives of Biochemistry and Biophysics, 2015, 574:66-74.
NDEZ-FUEYO E, et al.Basidiomycete DyPs:Genomic diversity, structural-functional aspects, reaction mechanism and environmental significance[J].Archives of Biochemistry and Biophysics, 2015, 574:66-74.
[26] DE GONZALO G, COLPA D I, HABIB M H M, et al.Bacterial enzymes involved in lignin degradation[J].Journal of Biotechnology, 2016, 236:110-119.
[27] SILVEIRA C M, MOEE L, FRAAIJE M, et al.Resonance Raman view of the active site architecture in bacterial DyP-type peroxidases[J].RSC Advances, 2020, 10(19):11 095-11 104.
[28] YU W N, LIU W N, HUANG H Q, et al.Application of a novel alkali-tolerant thermostable DyP-type peroxidase from Saccharomonospora viridis DSM 43017 in biobleaching of Eucalyptus kraft pulp[J].PLoS One, 2014, 9(10):e110 319.
[29] TSUJI Y, VANHOLME R, TOBIMATSU Y, et al.Introduction of chemically labile substructures into Arabidopsis lignin through the use of LigD, the Cα-dehydrogenase from Sphingobium sp.strain SYK-6[J].Plant Biotechnology Journal, 2015, 13(6):821-832.
[30] BUGG T D H, WILLIAMSON J J, RASHID G M M.Bacterial enzymes for lignin depolymerisation:New biocatalysts for generation of renewable chemicals from biomass[J].Current Opinion in Chemical Biology, 2020, 55:26-33.
[31] CHAUHAN P S.Role of various bacterial enzymes in complete depolymerization of lignin:A review[J].Biocatalysis and Agricultural Biotechnology, 2020, 23:101 498.
[32] FUJITA M, SAKUMOTO T, TANATANI K, et al.Iron acquisition system of Sphingobium sp.strain SYK-6, a degrader of lignin-derived aromatic compounds[J].Scientific Reports, 2020, 10(1):12 177.
[33] MATHIEU Y, PIUMI F, VALLI R, et al.Activities of secreted aryl alcohol quinone oxidoreductases from Pycnoporus cinnabarinus provide insights into fungal degradation of plant biomass[J].Applied and Environmental Microbiology, 2016, 82(8):2 411-2 423.
[34] LIU E S, LI M X, ABDELLA A, et al.Development of a cost-effective medium for submerged production of fungal aryl alcohol oxidase using a genetically modified Aspergillus nidulans strain[J].Bioresource Technology, 2020, 305:123 038.
[35] RASHID G M M, TAYLOR C R, LIU Y, et al.Identification of manganese superoxide dismutase from Sphingobacterium sp.T2 as a novel bacterial enzyme for lignin oxidation[J].ACS Chemical Biology, 2015, 10(10):2 286-2 294.
[36] 田朝光, 马延和.真菌降解木质纤维素的功能基因组学研究进展[J].生物工程学报, 2010, 26(10):1 333-1 339.
TIAN C G, MA Y H.Progress in lignocellulose deconstruction by fungi[J].Chinese Journal of Bioengineering, 2010, 26(10):1 333-1 339.
[37] LIU Y, WU Y Y, ZHANG Y, et al.Lignin degradation potential and draft genome sequence of Trametes trogii S0301[J].Biotechnology for Biofuels, 2019, 12:256.
[38] KUMAR M, VERMA S, GAZARA R K, et al.Genomic and proteomic analysis of lignin degrading and polyhydroxyalkanoate accumulating β-proteobacterium Pandoraea sp.ISTKB[J].Biotechnology for Biofuels, 2018, 11(1):1-23.
[39] RIYADI F A, TAHIR A A, YUSOF N, et al.Enzymatic and genetic characterization of lignin depolymerization by Streptomyces sp.S6 isolated from a tropical environment[J].Scientific Reports, 2020, 10:7 813.
[40] MORAES E C, ALVAREZ T M, PERSINOTI G F, et al.Lignolytic-consortium omics analyses reveal novel genomes and pathways involved in lignin modification and valorization[J].Biotechnology for Biofuels, 2018, 11:75.
[41] ZHU D C, SI H B, ZHANG P P, et al.Genomics and biochemistry investigation on the metabolic pathway of milled wood and alkali lignin-derived aromatic metabolites of Comamonas serinivorans SP-35[J].Biotechnology for Biofuels, 2018, 11:338.
[42] 魏雯丽, 宫尾茂雄, 吴正云, 等.基于宏转录组学技术解析工业豇豆泡菜发酵过程中活性微生物群落结构变化[J].食品与发酵工业, 2020, 46(10):60-65.
WEI W L, MIYAO Shigeo, WU Z Y, et al.Analysis of active microbial community structure changes in industrial cowpea pickle fermentation based on meta-transcriptomics technology[J].Food and Fermentation Industries, 2020, 46(10):60-65.
[43] 吴小峰. 基于宏转录组学技术研究饲草类型和季节对麝牛瘤胃微生物组的影响[D].雅安:四川农业大学,2017.
WU X F.The effect of forage type and season on the microbiota of musk ox rumen through macrotranscriptomics approaches[D].Ya′an:Agricultural University, 2017.
[44] 冯茜. 基于蛋白质组学的烟曲霉G-13木质素降解机理研究[D].哈尔滨:哈尔滨理工大学,2018.
FENG Q.Proteomics analysis of lignin degradation machanism of lignindagrading fungal Aspergillus fumigatus G-13[D].Harbin:Harbin University of Science and Technology, 2018.
[45] ADAV S S, CHAO L T, SZE S K.Quantitative secretomic analysis of Trichoderma reesei strains reveals enzymatic composition for lignocellulosic biomass degradation[J].Molecular & Cellular Proteomics, 2012, 11(7):DOI:10.1074/mcp.M111.012419.