透明质酸是由N-乙酰氨基葡萄糖和葡萄糖醛酸为双糖单位聚合而成的直链酸性多糖[1]。透明质酸的功能和分子质量相关,分子质量>1 MDa的高分子透明质酸主要应用于临床和美容领域[2]。与低分子透明质酸相比,高分子质量透明质酸具有更好的保湿和抵抗炎症的功能,可保护软骨免于降解[3-6]。
动物组织提取法和微生物发酵法是目前高分子质量透明质酸的主要生产方法。动物组织提取法是通过物理化学方法处理富含透明质酸的动物组织如鸡冠等,获得高分子透明质酸,主要应用于医药和临床治疗[7]。然而动物组织原材料有限,该提取方法难以应用到工业生产中[8]。微生物发酵法最为常见的生产菌株是兽疫链球菌[9]。但该菌具有致病性,在工业生产中存在安全隐患。同时该菌遗传操作十分困难[10],因而近年来兽疫链球菌生产高分子透明质酸的研究没有有效进展[7]。2016年,全球透明质酸的市场规模达到72亿美元[11],而高分子透明质酸因难以大规模安全高效生产,价格十分昂贵。
近年来,如何通过基因工程改造菌株安全高效地生产高分子质量透明质酸引发了广泛关注。目前已经在枯草芽孢杆菌[12-13]、乳酸乳球菌、巴斯德毕赤酵母和谷氨酸棒杆菌[14]等不同宿主中,通过表达透明质酸合酶基因合成高分子质量透明质酸,多数重组菌中采用I型透明质酸合酶。JEEVA等[15]在乳酸乳球菌中表达来源于兽疫链球菌的透明质酸编码基因sehasA,通过发酵条件的优化最终合成透明质酸分子质量达到1.4 MDa。JEONG等[9]在毕赤酵母中表达来源于Xenopus laevis的透明质酸合酶编码基因xhasA2,通过启动子优化以及低温发酵最终合成透明质酸分子质量达到2.5 MDa,产量为0.8~1.7 g/L。
本研究首先在谷氨酸棒杆菌中表达多杀性巴斯德杆菌(Pasteurella multocida)来源的Ⅱ型透明质酸合酶,优化密码子和胞内透明质酸提取方法后,实现了高分子质量透明质酸的合成。其次,对关键合酶基因采取截短分析和定点突变的策略,并强化表达透明质酸的合成前体内源途径酶基因。在此基础上对重组谷氨酸棒杆菌的培养条件进行了优化。最后通过3 L发酵罐分批补料发酵,实现了高分子质量透明质酸的安全、高效合成。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
研究所用的质粒和菌株如表1、表2所示。
表1 研究中所用质粒
Table 1 Plasmids used in this study

质粒描述来源pXMJ19E.coli-C.glutamicum shuttle expression vector,Cmr本实验室pEC-XK99EE.coli-C.glutamicum shuttle expression vector,Kmr本实验室pXMJ19-pmHasA-0pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA-0 本研究pXMJ19-pmHasApXMJ19 plasmid expressing codon-optimized hyaluronic acid synthase gene of P.multocida,pmhasA本研究pXMJ19-pmHasAΔ2-45pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA Δ2-45本研究pXMJ19-pmHasAΔ2-71pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmHasA Δ2-71本研究pXMJ19-pmHasAΔ2-95pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmHasA Δ2-95本研究pXMJ19-pmHasAΔ2-117pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmHasA Δ2-117本研究pXMJ19-pmHasAΔ704-972pXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmHasAΔ704-972本研究pXMJ19-pmHasA T40LpXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA T40L本研究pXMJ19-pmHasA V59MpXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA V59M本研究pXMJ19-pmHasA T104ApXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA T104A本研究pXMJ19-pmHasA V59M T104ApXMJ19 plasmid expressing hyaluronic acid synthase gene of P.multocida,pmhasA V59M T104A本研究pEC-XK99E-galUpEC-XK99E plasmid expressing UTP-glucose-1-phosphate uridylyltransferase gene of C.glutamicum ATCC13032,galu本研究pEC-XK99E-ugdApEC-XK99E plasmid expressing UDP-glucose 6-dehydrogenase gene of C.glutamicum ATCC13032,ugdA 本研究pEC-XK99E-glmSpEC-XK99E plasmid expressing L-glutamine-D-fructose-6-phosphate aminotransferase gene of C.glutamicum ATCC13032,glmS 本研究pEC-XK99E-glmMpEC-XK99E plasmid expressing phosphoglucosamine mutase gene of C.glutamicum ATCC13032,glmM 本研究pEC-XK99E-glmUpEC-XK99E plasmid expressing UDP-N-acetylglucosamine pyrophosphorylase gene of C.glutamicum ATCC13032,glmU 本研究
表2 研究中所用菌株
Table 2 Strains used in this study

菌株描述来源E.coli JM109 E.coli K12 JM109 本实验室C.glutamicumC.glutamicum ATCC 13032 本实验室pXMJ C.glutamicum containing pXMJ19,Cmr 本实验室pmHasA-0C.glutamicum containing pXMJ-pmHasA-0,Cmr本研究pmHasAC.glutamicum containing pXMJ-pmHasA,Cmr本研究pmHasAΔ2-45C.glutamicum containing pXMJ-pmHasAΔ2-45,Cmr本研究pmHasAΔ2-71C.glutamicum containing pXMJ-pmHasAΔ2-71,Cmr本研究pmHasAΔ2-95C.glutamicum containing pXMJ-pmHasAΔ2-95,Cmr本研究pmHasAΔ2-117C.glutamicum containing pXMJ-pmHasAΔ2-117,Cmr本研究pmHasAΔ704-972C.glutamicum containing pXMJ-pmHasAΔ704-972,Cmr本研究pmHasAT40LC.glutamicum containing pXMJ-pmHasA T40L,Cmr本研究pmHasAV59MC.glutamicum containing pXMJ-pmHasA V59M,Cmr本研究pmHasA T104AC.glutamicum containing pXMJ-pmHasA T104A,Cmr本研究pmHasAV59M T104AC.glutamicum containing pXMJ-pmHasA V59M T104A,Cmr本研究galUC.glutamicum containing pXMJ-pmHasA T104A and pEC-XK99E-galU,Cmr and Kmr本研究ugdAC.glutamicum containing pXMJ-pmHasA T104A and pEC-XK99E-ugdA,Cmr and Kmr本研究glmMC.glutamicum containing pXMJ-pmHasA T104A and pEC-XK99E-glmM,Cmr and Kmr本研究glmSC.glutamicum containing pXMJ-pmHasA T104A and pEC-XK99E-glmS,Cmr and Kmr本研究glmUC.glutamicum containing pXMJ-pmHasA T104A and pEC-XK99E-glmU,Cmr and Kmr本研究
1.1.2 引物
该文中的引物如表3所示。
表3 研究中所用引物
Table 3 Primers used in this study
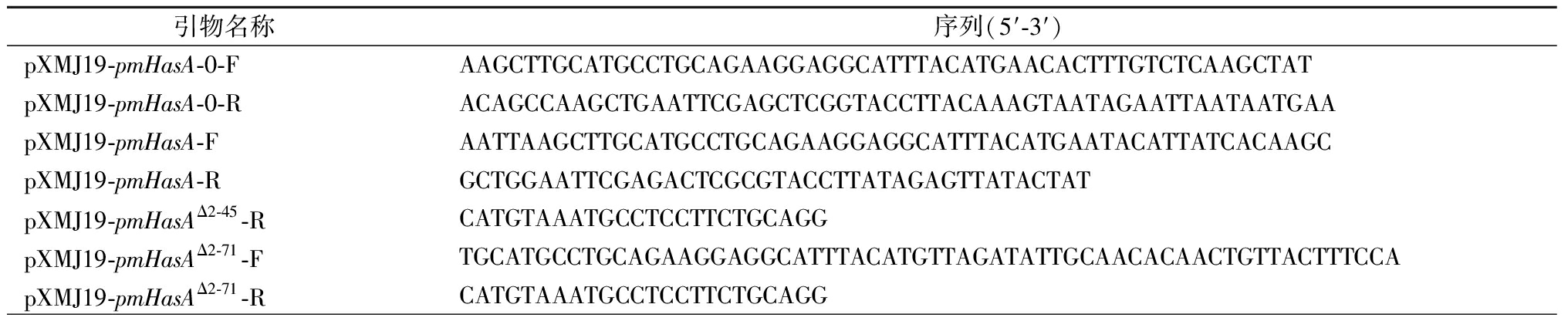
引物名称序列(5′-3′)pXMJ19-pmHasA-0-FAAGCTTGCATGCCTGCAGAAGGAGGCATTTACATGAACACTTTGTCTCAAGCTATpXMJ19-pmHasA-0-RACAGCCAAGCTGAATTCGAGCTCGGTACCTTACAAAGTAATAGAATTAATAATGAApXMJ19-pmHasA-FAATTAAGCTTGCATGCCTGCAGAAGGAGGCATTTACATGAATACATTATCACAAGCpXMJ19-pmHasA-RGCTGGAATTCGAGACTCGCGTACCTTATAGAGTTATACTATpXMJ19-pmHasAΔ2-45-RCATGTAAATGCCTCCTTCTGCAGGpXMJ19-pmHasAΔ2-71-FTGCATGCCTGCAGAAGGAGGCATTTACATGTTAGATATTGCAACACAACTGTTACTTTCCApXMJ19-pmHasAΔ2-71-RCATGTAAATGCCTCCTTCTGCAGG
续表3

引物名称序列(5′-3′)pXMJ19-pmHasAΔ2-45-FTGCATGCCTGCAGAAGGAGGCATTTACATGCTCTCAGCACATCCTTCTGTTAATTCpXMJ19-pmHasAΔ2-95-FCCTGCAGAAGGAGGCATTTACATGTTAAAAAATAAATGGAAATTGCTCACTGAGAAGAApXMJ19-pmHasAΔ2-95-RCATGTAAATGCCTCCTTCTGCAGGpXMJ19-pmHasAΔ2-117-FCATGCCTGCAGAAGGAGGCATTTACATGCTTGTACCAAAAGATTTTCCCAAAGATCTGGpXMJ19-pmHasAΔ2-117-RGTAAATGCCTCCTTCTGCAGGCATGpXMJ19-pmHasAΔ704-972-FCAAGAAGAGATTGATATCTTAAAAGATATTTAAGGTACCGAGCTCGAATTCAGCTpXMJ19-pmHasAΔ704-972-RAATATCTTTTAAGATATCAATCTCTTCTTGpXMJ19-pmHasA T40L-FCTCAAATGCAAAGAAAAACTCTCAGCACATpXMJ19-pmHasA T40L-RAATTTGAAATTCAACAATTTTCCGTCCATAGATTTCCpXMJ19-pmHasA V59M-FATGAATAAAGAAGAAAAAGTCAATGTTTGCGATAGTCpXMJ19-pmHasA V59M-RAGAAAGATGTGCTGAATTAACAGAAGGATGTpXMJ19-pmHasA T104A-FGCAGAGAAGAAATCTGAAAATGCGGAGpXMJ19-pmHasA T104A-RGAGCAATTTCCATTTATTTTTTAACGTGTTTTTTTCCGApEC-XK99E-galU-F AGCTCAGGTACCCGGGGGATCCAAGGAGCGGTTTGCCTATCGATGAGCpEC-XK99E-galU-RAAGCTTTTGCCTGCACCTATTTTACTTGATAATCGTCTGCpEC-XK99E-ugdA-FCAGGAAGACCATG GAATTCAAGAGCATTTACATGCGGGACAGTGATTGGpEC-XK99E-ugdA-RGTCGACTCTAGAGGATCCCCGGGTACCCTAAAGGTTGCGGCCGAGCGCTpEC-XK99E-glmS-FAGCTCGGTACCCGGGGATCCAAGGAGCGTATATCATGCGCATGTGTGGAATTGTTGpEC-XK99E-glmS-RAAGCTTGCATGCCTGCAGTTATTCGACGGTGACAGACpEC-XK99E-glmM-FAGCTCGGTACCCGGGGATCCAAGGAGCGTATATCATGACTCGACTATTTGGAACTGpEC-XK99E-glmM-RAAGCTTGGCCTGCAGTTAGATCTTCTGCAACCCACTGpEC-XK99E-glmU-FAGCTCGGTAACCCGGGGATCCAAGGAGCGTTCTTGAGCGCAAGCGATTTCTCGpEC-XK99E-glmU-RAAGCTTGGGCCTGCAGTTAGCCTTCCTGGTTGTGGAC
1.2 实验方法
1.2.1 试剂与培养基
1.2.1.1 试剂
蛋白胨、酵母粉、脑心浸出液,OXIOD 公司。其余试剂均为分析纯试剂。
1.2.1.2 培养基
BHI培养基(g/L):脑心浸出液37,山梨醇91;固体培养基另外添加琼脂糖15。
发酵培养基(g/L):(NH4)2SO4 20,玉米浆干粉20,K2HPO4 1,KH2PO4 1,MgSO4 0.25,3-(N-吗啉基)丙磺酸钠盐 4.2,葡萄糖 20;pH 7.0。
1.2.2 培养条件
摇瓶发酵培养:将种子液转接至摇瓶中,使初始OD600为0.1,28 ℃,200 r/min培养3 h后添加0.25 mmol/L IPTG诱导,培养48 h。
1.2.3 重组菌株的构建
根据报道的P.multocida基因序列,通过GENEWIZ(苏州)合成透明质酸合酶基因pmHasA(Gene ID:11805012)序列。利用引物pXMJ-pmHasA-0-F和pXMJ-pmHasA-0-R进行PCR扩增,再通过T5 exonuclease-dependent assembly(TEDA)[16]方法将pmHasA连接至质粒pXMJ,构建得到重组表达质粒pXMJ-pmHasA-0。通过电转将重组表达质粒转入谷氨酸棒杆菌中,构建得到重组谷氨酸棒杆菌pmHasA-0。利用引物pXMJ-pmHasA-F和pXMJ-pmHasA-R进行PCR扩增,通过TEDA方法构建重组质粒pXMJ-pmHasA;并以该重组质粒为模板,使用引物pXMJ19-pmHasAΔ2-45-F、pXMJ19-pmHasAΔ2-45-R等用TEDA构建得到重组质粒pXMJ19-pmHasAΔ2-45、pXMJ19-pmHasAΔ2-71、pXMJ19-pmHasAΔ2-95、pXMJ19-pmHasAΔ2-117、pXMJ19-pmHasAΔ704-972、pmHasA T40L、pmHasA V59M、pmHasA T104A和pmHasA V59M T104A。以谷氨酸棒杆菌基因组为模板,以pEC-XK99E-galU-F、pEC-XK99E-galU-R等引物扩增基因galU (Gene ID:1021411)、ugdA(Gene ID:23502230)、glmS(Gene ID:1020224)、glmM(Gene ID:23502018)和glmU(Gene ID:1018935),通过TEDA方法构建得到重组质粒。通过电转将重组质粒转入谷氨酸棒杆菌中,得到对应重组菌;同时通过电转将空质粒pXMJ转入谷氨酸棒杆菌中,构建得到重组菌pXMJ作为阴性对照。
1.2.4 检测方法
1.2.4.1 胞内透明质酸碱裂解提取法
取10 mL发酵样品,离心去除上清液保留菌体,加入5 mL PI溶液轻轻重悬菌体,放于37 ℃水浴1 h;加入5 mL PⅡ溶液裂解菌体溶液,上下轻轻颠倒至溶液澄清使细胞完全裂开;加入7.5 mL PⅢ溶液使蛋白与基因组共沉淀;离心取上清液,加入4倍体积无水乙醇,反复醇沉复溶2次;加入质量浓度为20 mg/mL DNase和100 μg/mL RNase,37 ℃水浴1 h,去除溶液内残余DNA和RNA,醇沉复溶后得到高纯度透明质酸溶液。(PI:25 mmol/L Tris-HCl,10 mmol/L EDTA,20 mg/mL Lyzsome,pH 8.0;PⅡ:0.2 mol/L NaOH,1% SDS;PⅢ:3 mol/L KoAC,pH 6.0;DNase/RNase溶液缓冲液:10 mmol/L Tris-HCl pH 7.5,2.5 mmol/L MgCl2,0.5 mmol/L CaCl2)
1.2.4.2 透明质酸定量测定
采用Bitte-Muir法[17]。在500 mL浓硫酸溶液中加入4.77 g四硼酸钠,超声溶解,配置成硼砂硫酸溶液;在500 mL无水乙醇溶液中加入1.25 g咔唑,配置成咔唑溶液。配置1 g/L的葡萄糖醛酸溶液,稀释成0、10、20、30、40和50 mg/L,在硫酸硼砂与咔唑反应后,置于530 nm条件下测定吸光度,将吸光度与葡萄糖浓度绘制成标准曲线。
将提取纯化的透明质酸溶液根据浓度稀释成相应倍数(5~1 000),取200 μL与1 mL硫酸混合于比色管中,在沸水中煮10~15 min后,放在冰上冷却至室温。加入50 μL咔唑后,在沸水中煮10~15 min,于530 nm条件下测定吸光度。根据标准曲线计算对应样品的葡萄糖醛酸浓度,再根据公式(1)计算出相应的透明质酸质量浓度。
(1)
注:ρ(HA),透漏质酸质量浓度,g/L;ρ(葡萄糖醛酸),葡萄糖醛酸质量浓度,g/L;n,稀释倍数。
1.2.4.3 透明质酸分子质量测定
将发酵样品通过反复醇沉获得纯度较高的透明质酸,经过水系膜过滤除去颗粒杂质后,通过高效尺寸排除色谱(high performance size exclusion chromatography,HPSEC)测定透明质酸分子质量[18]。仪器型号Agilent 1260 Infinity,液相色谱柱Waters WAT011545,流动相0.1 mol/L NaNO3,流速0.6 mL/min,控制柱温25 ℃。
2 结果与分析
2.1 P.multocida来源透明质酸合酶基因密码子优化
将重组菌pmHasA-0在摇瓶中进行培养,按如上所述方法检测所得产物,结果如图1-a所示。对照菌株pXMJ和重组菌株pmhasA-0均未能检测到透明质酸的合成。考虑到谷氨酸棒杆菌有较强的密码子偏好性,根据密码子优化网站http://www.jcat.de/将透明质酸合酶基因pmhasA密码子优化后,重新构建质粒并转入谷氨酸棒杆菌中,得到重组菌pmHasA。按相同的方法对菌株进行培养并进行产物提取纯化及检测,结果如图1-a所示,重组菌pmHasA能实现透明质酸的合成,产量达到0.14 g/L。

a-P.multocida来源的透明质酸合酶基因密码子优化; b-比较分析不同方法提取胞内透明质酸对分子质量的影响
图1 重组谷氨酸棒杆菌合成透明质酸
Fig.1 Hyaluronic acid synthesis in recombinant C.glutamicum
2.2 胞内透明质酸提取方法的优化
P.multocida来源的透明质酸合酶为Ⅱ型合酶,依赖宿主自身的ABC转运体系实现透明质酸的分泌[19-20]。而谷氨酸棒杆菌没有该转运体系,所以会导致透明质酸积累在细胞内部,无法正常分泌。为了提取胞内的透明质酸且对其分子质量不造成较大的影响,需要开发一种相对温和的细菌破壁方法。为此探究比较了传统的透明质酸提取方法——高温高压破壁法和高压匀浆法与碱裂解法所提取的透明质酸分子质量上的差异。结果如图1-b所示,采用高温高压破壁法和高压匀浆法提取的透明质酸分子质量分别为0.21 M和0.72 MDa,而该研究中采用优化的碱裂解法提取的透明质酸分子质量达到1.51 MDa。结果表明,采用高温高压和高压匀浆条件下会破坏透明质酸原有的链长,造成长链断裂,影响最终透明质酸分子质量测定的结果,而采用更为温和的方法破壁处理菌体,更有利于获得高分子质量的透明质酸。
2.3 透明质酸合酶(pmHasA)的理性改造
2.3.1 透明质酸合酶(pmHasA)截短分析
透明质酸合酶(pmHasA)不同氨基酸区域对于透明质酸的合成有不同的影响[21]。为了研究透明质酸合酶(pmHasA)各区域氨基酸对透明质酸分子质量的影响,对透明质酸合酶基因pmhasA从N端和C端进行不同长度的截短分析,构建合酶截短突变体重组菌pmHasAΔ2-45、pmHasAΔ2-71、pmHasAΔ2-95、pmHasAΔ2-117和pmHasAΔ704-972。结果如图2-a所示,重组菌合成的透明质酸分子质量分别为1.42 M、1.36 M、1.26 M、0.80 M和1.41 MDa,产量分别为0.13、0.11、0.12、0.02和0.11 g/L。其中,重组菌pmHasAΔ2-117合成的透明质酸分子质量相比重组菌pmHasA下降了48%,产量下降81%。此外重组菌pmHasAΔ2-45、pmHasAΔ2-71和pmHasAΔ2-95合成的透明质酸分子质量和产量出现了一定的下降,且截短越多分子质量下降越多。而在截短C段268个氨基酸后,重组菌pmHasAΔ704-972合成的透明质酸分子质量仅下降7%。因此,推测透明质酸合酶pmHasA氨基酸2~117位点对合成透明质酸的分子质量和产量有重要作用。
2.3.2 透明质酸合酶(pmHasA)定点突变改造
为进一步探究透明质酸合酶pmHasA N端关键位点对透明质酸的分子质量的重要影响,在全长透明质酸合酶基因pmhasA中选取氨基酸位点40、59和104,构建重组菌pmHasA T40L,pmHasA V59M,pmHasA T104A和pmHasA V59M T104A进一步证明[22]。结果如2-b所示,合成的透明质酸分子质量分别约为1.34 M、1.53 M、1.82 M和1.34 MDa。其中,重组菌pmHasA T104A分子质量提高最为明显,较重组菌pmHasA提高了约22%。为了探究该位点对于合成透明质酸分子质量的影响,构建了该104位点其余的饱和突变体。重组菌表达其余突变体透明质酸分子质量结果如图2-c所示,其中合成的透明质酸分子质量最高的为重组菌表达突变体pmHasA T104H,为1.25 MDa,分子质量低于重组菌pmHasA T104A所产透明质酸。同时,该结果也表明在合酶截短分析中,透明质酸合酶pmHasA N端2~117位点中存在氨基酸位点显著影响透明质酸分子质量。

a-P.multocida来源的透明质酸合酶基因截短突变体对分子质量和产量的影响;b-透明质酸合酶pmhasA基因点突变体对分子质量和产量影响; c-透明质酸合酶pmhasA基因氨基酸T104位点饱和突变体对分子质量和产量影响
图2 P.multocida来源的透明质酸合酶基因理性改造
Fig.2 Genetic modification of hyaluronic acid synthase derived from P.multocida
2.4 透明质酸合成途径的表达强化
强化透明质酸合成途径常用于提高透明质酸合成链长[9]。在过表达透明质酸合酶突变体pmhasA T104A的基础上,通过质粒pEC-XK99E分别单独强化表达谷氨酸棒杆菌内源代谢途径基因galU、ugdA、glmS、glmM和glmU[23]。结果如图3所示,当分别强化UDP-GlcUA前体物质的途径基因galU、ugdA时,合成透明质酸的分子质量相比重组菌T104A提高了41%和84%;而强化UDP-GlcNAc前体合成的途径基因glmS、glmU时,合成透明质酸的分子质量相比重组菌T104A降低了11%和16%。这表明在谷氨酸棒杆菌中,前体物质UDP-GlcUA显著影响透明质酸链长,其中过表达ugdA效果最为明显,透明质酸产量达到0.54 g/L,分子质量达到3.31 MDa。
2.5 重组菌株摇瓶培养温度优化
在前期的研究中,重组菌培养温度为28 ℃。温度会显著影响重组菌合成透明质酸的分子质量[24],设计5种不同温度(20,25,30,37和40 ℃)摇瓶培养重组菌株ugdA,来确定合成高分子质量透明质酸的最佳培养温度。结果如图4所示,培养温度为20~40 ℃,透明质酸分子质量随温度的升高而下降。表明重组菌ugdA合成高分子质量透明质酸的最优培养温度为20 ℃,此时透明质酸分子质量为4.8 MDa,产量达到0.52 g/L。
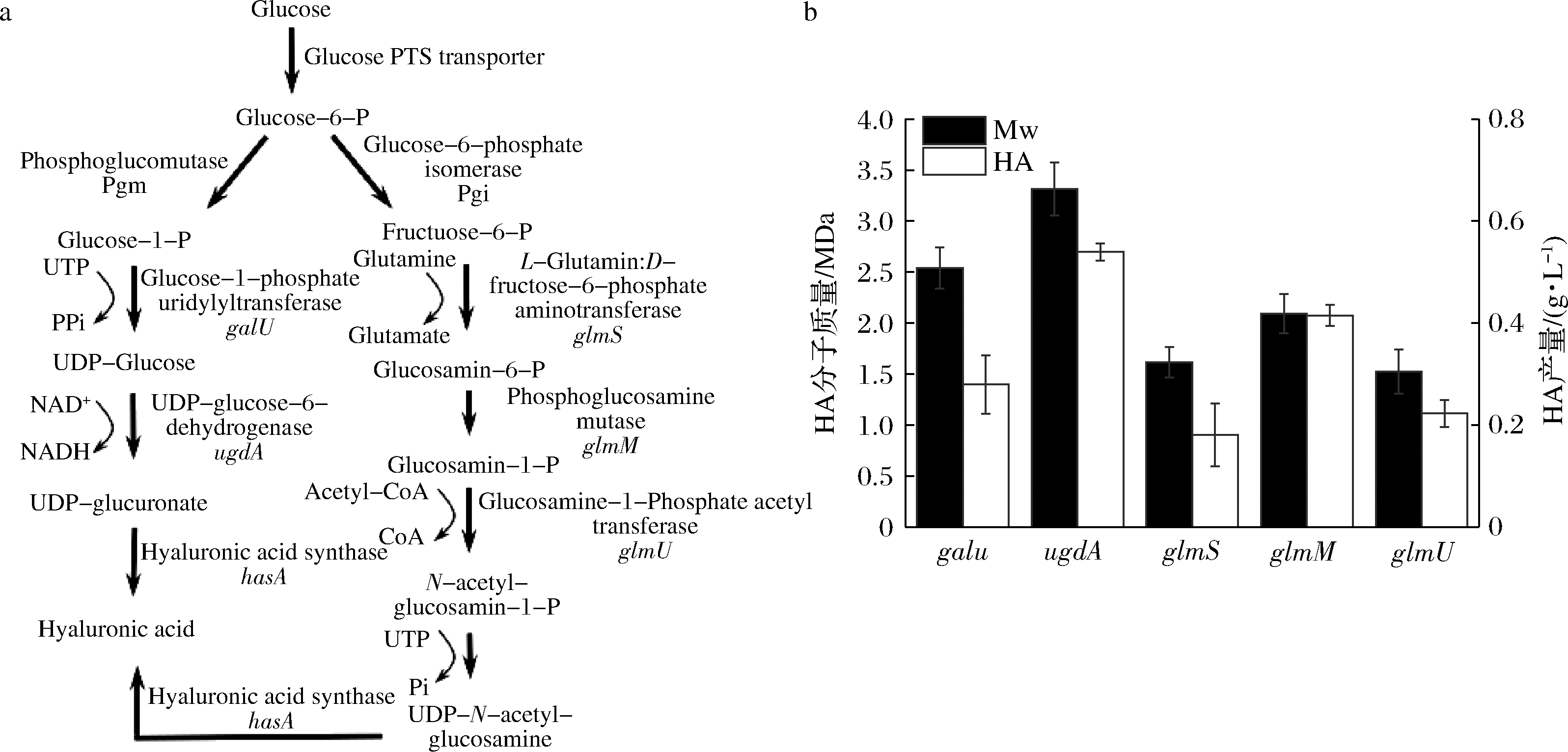
a-谷氨酸棒杆菌中透明质酸的代谢途径;b-在谷氨酸棒杆菌中过表达途径基因galU、ugdA、glmS、glmM和glmU促进透明质酸合成
图3 透明质酸合成途径基因的表达强化
Fig.3 Overexpression of pathway genes in hyaluronic acid biosynthesis
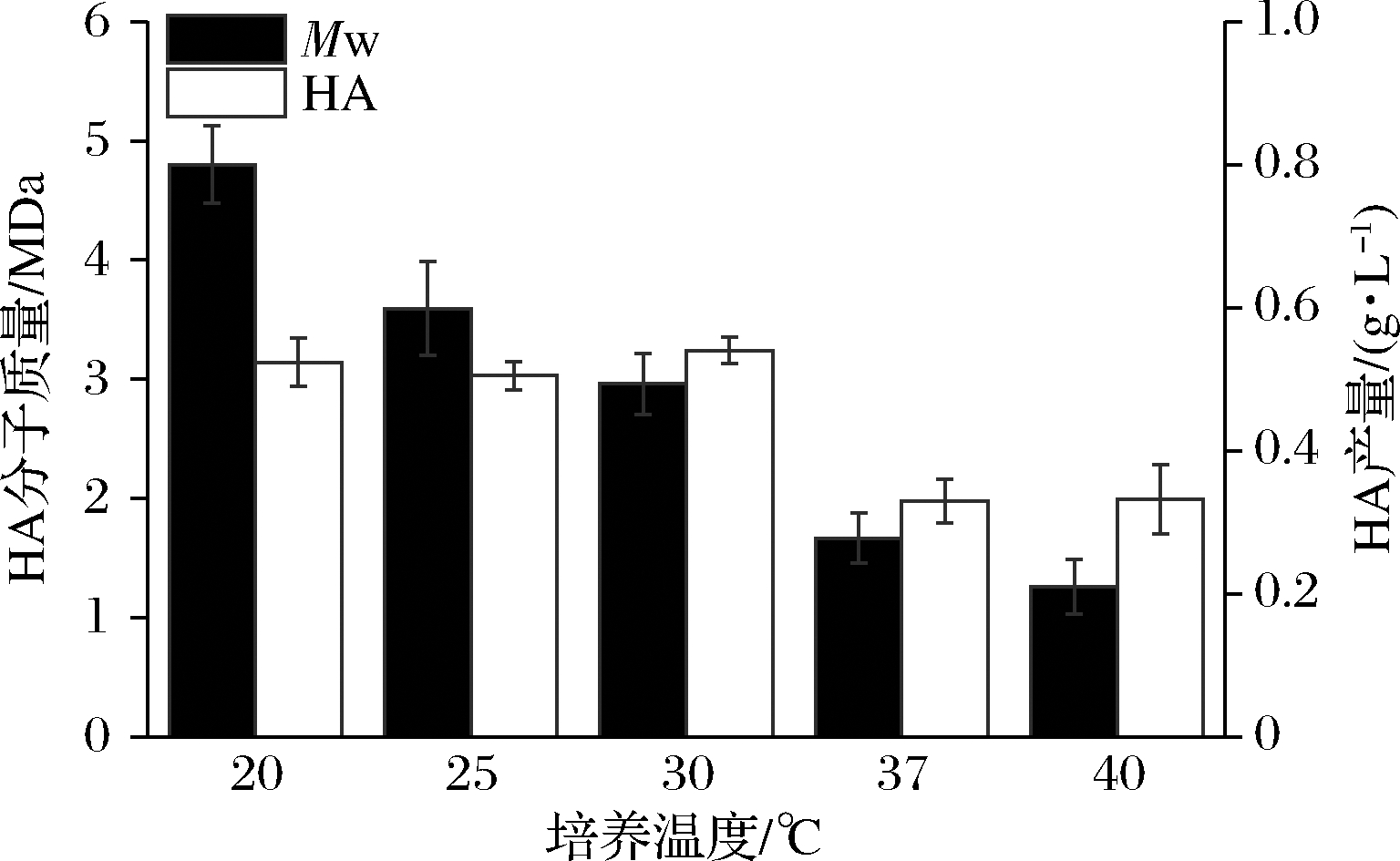
图4 不同培养温度对谷氨酸棒杆菌合成透明质酸分子 质量和产量的影响
Fig.4 Effects of different culture temperatures on the Mw and yield of HA synthesized by C.glutamicum
2.6 重组菌株 3 L 发酵罐分批补料生产高分子质量透明质酸
为验证产高分子质量透明质酸重组菌株的实际生产能力,在3 L发酵罐进行分批补料培养重组菌株ugdA。结果如图5所示,发酵48 h时,OD600最高达到58。发酵72 h时,高分子质量透明质酸产量达到1.5 g/L。在发酵过程中,透明质酸分子质量有波动性变化,其中发酵32和48 h时,分子质量分别为4.6 M和4.7 MDa。

图5 重组菌株ugdA 3 L发酵罐中分批补料时间曲线图
Fig.5 Fermentation curves of the recombinant strain ugdA in 3 L fermentor
3 结论
通过在谷氨酸棒杆菌中表达密码子优化后的P.multocida来源的Ⅱ型透明质酸合酶基因,实现了透明质酸积累,并优化提取胞内透明质酸工艺,透明质酸分子质量达到1.51 MDa。其次,构建了透明质酸合酶突变体pmhasA T104A,分子质量提高至1.8 MDa。在此基础上对合成途径中UDP-葡萄糖醛酸脱氢酶基因ugdA进行表达强化,并确定最优培养温度条件为20 ℃,透明质酸分子质量达到4.8 MDa。最后,将重组菌株在3 L发酵罐中进行放大培养,验证该菌株生产高分子质量透明质酸的能力,高分子质量透明质酸产量最终达到1.5 g/L,分子质量达4.7 MDa。该研究在谷氨酸棒杆菌表达Ⅱ型透明质酸合酶实现高分子质量透明质酸的安全高效合成,对于推动工业发酵生产高分子质量透明质酸有重要意义。
[1] KOGAN G, OLTÉS L,STERN R,et al.Hyaluronic acid:A natural biopolymer with a broad range of biomedical and industrial applications[J].Biotechnology Letters,2007,29(1):17-25.
OLTÉS L,STERN R,et al.Hyaluronic acid:A natural biopolymer with a broad range of biomedical and industrial applications[J].Biotechnology Letters,2007,29(1):17-25.
[2] GUILLAUMIE F,FURRER P,FELT-BAEYENS O,et al.Comparative studies of various hyaluronic acids produced by microbial fermentation for potential topical ophthalmic applications[J].Journal of Biomedical Materials Research Part A,2010,92A (4):1 421-1 430.
[3] AGARWAL S,DUFFY B,CURTIN J,et al.Effect of high-and low-molecular-weight hyaluronic-acid-functionalized-AZ31 Mg and Ti alloys on proliferation and differentiation of osteoblast cells[J].ACS Biomaterials Science & Engineering,2018,4(11):3 874-3 884.
[4] ZHAO Y F,QIAO S P,HOU X L,et al.Bioengineered tumor microenvironments with naked mole rats high-molecular-weight hyaluronan induces apoptosis in breast cancer cells[J].Oncogene,2019,38(22):4 297-4 309.
[5] OHTSUKI T,ASANO K,INAGAKI J,et al.High molecular weight hyaluronan protects cartilage from degradation by inhibiting aggrecanase expression[J].Journal of Orthopaedic Research,2018,36(12):3 247-3 255.
[6] CHITNIS A S,ETTER K,HOLY C E,et al.Real-world impact of the high concentration non-avian high molecular weight hyaluronan on pain medication use among osteoarthritis patients[J].Current Medical Research and Opinion,2019,35(9):1 523-1 527.
[7] LIU L,LIU Y F,LI J H,et al.Microbial production of hyaluronic acid:Current state,challenges,and perspectives[J].Microbial Cell Factories,2011,10:99.
[8] KANG Z,ZHOU Z X,WANG Y,et al.Bio-based strategies for producing glycosaminoglycans and their oligosaccharides[J].Trends in Biotechnology,2018,36(8):806-818.
[9] JEONG E,SHIM W Y,KIM J H.Metabolic engineering of Pichia pastoris for production of hyaluronic acid with high molecular weight[J].Journal of Biotechnology,2014,185:28-36.
[10] POURZARDOSHT N,RASAEE M J.Improved yield of high molecular weight hyaluronic acid production in a stable strain of Streptococcus zooepidemicus via the elimination of the hyaluronidase-encoding gene[J].Molecular Biotechnology,2017,59(6):192-199.
[11] DE OLIVEIRA J D,CARVALHO L S,GOMES A M V,et al.Genetic basis for hyper production of hyaluronic acid in natural and engineered microorganisms[J].Microbial Cell Factories,2016,15(1):1-19.
[12] JIN P,KANG Z,YUAN P H,et al.Production of specific-molecular-weight hyaluronan by metabolically engineered Bacillus subtilis 168[J].Metabolic Engineering,2016,35:21-30.
[13] WIDNER B,BEHR R,VON DOLLEN S,et al.Hyaluronic acid production in Bacillus subtilis[J].Applied and Environmental Microbiology,2005,71(7):3 747-3 752.
[14] HOFFMANN J,ALTENBUCHNER J.Hyaluronic acid production with Corynebacterium glutamicum:Effect of media composition on yield and molecular weight[J].Journal of Applied Microbiology,2014,117(3):663-678.
[15] JEEVA P,SHANMUGA DOSS S,SUNDARAM V,et al.Production of controlled molecular weight hyaluronic acid by glucostat strategy using recombinant Lactococcus lactis cultures[J].Applied Microbiology and Biotechnology,2019,103(11):4 363-4 375.
[16] XIA Y Z,LI K,LI J J,et al.T5 exonuclease-dependent assembly offers a low-cost method for efficient cloning and site-directed mutagenesis[J].Nucleic Acids Research,2019,47(3):e15.
[17] BITTER T,MUIR H M.A modified uronic acid carbazole reaction[J].Analytical Biochemistry,1962,4(4):330-334.
[18] ZHANG L P,HUANG H,WANG H,et al.Rapid evolution of hyaluronan synthase to improve hyaluronan production and molecular mass in Bacillus subtilis[J].Biotechnology Letters,2016,38(12):2 103-2 108.
[19] WILLIS L M,WHITFIELD C.KpsC and KpsS are retaining 3-deoxy-D-manno-oct-2-ulosonic acid (Kdo) transferases involved in synthesis of bacterial capsules[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(51):20 753-20 758.
[20] WILLIS L M,WHITFIELD C.Structure,biosynthesis,and function of bacterial capsular polysaccharides synthesized by ABC transporter-dependent pathways[J].Carbohydrate Research,2013,378:35-44.
[21] JING W,DEANGELIS P L.Dissection of the two transferase activities of the Pasteurella multocida hyaluronan synthase:Two active sites exist in one polypeptide[J].Glycobiology,2000,10(9):883-889.
[22] MANDAWE J,INFANZON B,EISELE A,et al.Directed evolution of hyaluronic acid synthase from Pasteurella multocida towards high-molecular-weight hyaluronic acid[J].ChemBioChem,2018,19(13):1 414-1 423.
[23] WANG Y,HU L T,HUANG H,et al.Eliminating the capsule-like layer to promote glucose uptake for hyaluronan production by engineered Corynebacterium glutamicum[J].Nature Communications,2020,11:3 120.
[24] LI Y Y,LI G Q,ZHAO X,et al.Regulation of hyaluronic acid molecular weight and titer by temperature in engineered Bacillus subtilis[J].3 Biotech,2019,9(6):1-9.