随着全球经济的快速发展,人类对能源的需求急剧增加,传统化石燃料带来的环境污染以及能源短缺问题日益严重,寻找和开发利用可再生的清洁型能源来代替传统的化石燃料已成为关注焦点之一[1]。燃料乙醇是一种理想的清洁生物燃料,可以通过木质纤维素原料水解产物糖化和发酵生产。酿酒酵母因其具有生长周期短、发酵能力强等特性而被认为是生产燃料乙醇的主要微生物。但是,由于木质纤维素结构致密复杂、成分多样,很难被酵母细胞直接利用,因此预处理是必不可少的环节。然而,预处理过程中会产生一系列木质纤维素发酵抑制物,如酚类化合物、弱酸类和呋喃类[2]。它们常常会影响酵母细胞的生长和发酵酶系的正常功能,进而降低发酵产物乙醇的产率[3-4]。酚类化合物来源于木质素的降解[5-6]、糖类物质的分解[7],虽然含量比较低,但对细胞毒害作用比有机酸和呋喃类化合物更大。其中酚醛抑制物直接影响发酵菌株的乙醇发酵,酚酸和酚醇化合物通过影响菌体生长而间接影响乙醇发酵[8]。然而,由于缺乏酚类物质准确定性定量方法,目前对酚类化合物的毒性机制研究还不够深入。有学者推测是因为酚类物质能造成细胞膜损伤,使细胞膜的完整性丧失、选择透过性的能力降低,从而影响酵母细胞的正常生长和发酵性能[9-10]。ZHANG等[11]研究酚类化合物可以抑制细胞内酶的活性,使酵母念珠菌SB18内木糖醇通路中有关酶失活。KEWELOH等[12]研究发现苯酚通过影响细胞膜上蛋白质和脂质含量的比例来改变细胞膜的功能。ISHIDA等[13]研究发现添加高浓度的香草醛能抑制细胞的翻译过程,影响蛋白质的生物合成,从而抑制酿酒酵母的生长和发酵。另外,酚类化合物的毒性强弱与其分子质量大小以及取代基的位置有关。有研究指出,酚类物质对木质纤维素水解液发酵过程存在强烈的抑制[14-15],且分子质量越低的酚类化合物毒性作用越强[16-17]。因此,如何解除酚类抑制剂对水解和发酵的不利影响是木质纤维素产乙醇过程中的一个重要挑战。
儿茶酚是木质纤维素水解液中常见的抑制物,但酿酒酵母应对儿茶酚的响应机制尚未阐明。本研究通过转录组技术分析,从mRNA水平揭示儿茶酚胁迫下酿酒酵母的响应机制,为进一步研究提高酿酒酵母酚类物质耐受性的方法提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
酿酒酵母(Saccharomyces cerevisiae GGSF16),由广西科技大学微生物研究室提供。
1.1.2 培养基
酵母浸出粉胨葡萄糖培养基(yeast extract peptone dextrose medium,YPD)(g/L):葡萄糖20,蛋白胨20,酵母粉10,115 ℃灭菌30 min。
发酵培养基(g/L):葡萄糖180,蛋白胨20,酵母粉10,115 ℃灭菌30 min。
1.1.3 主要试剂
儿茶酚(AR),天津市大茂化学试剂厂;RNA提取试剂盒,大连宝生物;DNA Marker,北京索莱宝科技有限公司;Tris,BioFroxx公司。
1.2 仪器与设备
DY-6D 型DNA电泳仪,北京市六一仪器厂;ZWYD—2402型摇床,上海智诚分析仪器制造有限公司;H2100R离心机,湖南湘仪实验室仪器开发有限公司;LDZH-100KBS立式压力蒸汽灭菌器,上海申安医疗器械厂;456-GC气相色谱仪,天美创科仪器(北京)有限公司;高效液相色谱仪,日本株式会社日立制作所。
1.3 实验方法
1.3.1 儿茶酚对酵母细胞发酵力的测定
挑取一环酵母细胞接种于YPD培养基中,30 ℃,150 r/min,摇床培养14 h;按10%接种量接入新鲜的YPD中培养至对数中期(OD600=1.109),最后按10%接种量转接入发酵培养基中,将儿茶酚加入到发酵培养基中使其质量浓度为1.2 g/L,以未添加儿茶酚的菌液为对照组,30 ℃,150 r/min培养24 h,每隔2 h取上清液适当稀释,用高效液相色谱仪检测葡萄糖质量浓度,用气相色谱仪检测乙醇质量浓度。
1.3.2 转录组测序
将活化的菌种按2%接种量接种于YPD培养基,30 ℃,150 r/min摇床中培养至指数中期,将儿茶酚加入到YPD培养基中,使其质量浓度为1.2 g/L,并随机分为对照组(ck1、ck2、ck3)和儿茶酚组(cat1、cat2、cat3),继续培养2 h后收集细胞用于后续RNA提取。取新鲜培养的菌液200 μL,参照酵母RNA提取试剂盒说明书提取RNA。质量检测合格的总RNA和mRNA用于后续建库测序,转录组测序工作均在深圳华大基因科技服务有限公司完成。
1.3.3 测序数据处理及其生信分析
对测序所得到的原始数据(raw reads)首先去除含有接头的Reads、未知碱基N含量过高、低质量的reads得到clean reads,作为本研究的基本数据。将clean reads与指定的参考基因组进行序列比对以及利用表达定量软件RSEM对基因的表达水平进行定量分析。使用DESeq2算法对样品之间的差异表达基因进行筛选,以P<0.05为差异值,fold change ≥2为差异倍数进行差异表达基因筛选,并进行差异基因的GO功能富集和KEGG代谢通路富集分析。
2 结果与分析
2.1 儿茶酚对酵母细胞发酵的影响
为研究儿茶酚对酵母细胞的影响,本试验在1.2 g/L的儿茶酚培养条件下,测定了葡萄糖消耗及乙醇生成。由图1可知,对照组在6~12 h葡萄糖消耗较快,18 h基本消耗完全,乙醇在6~16 h产量较多,并在18 h趋于稳定;而在添加儿茶酚后,葡萄糖消耗延迟了4 h,即在10 h才开始消耗,乙醇生成含量明显低于对照组,说明儿茶酚影响了酵母细胞乙醇发酵的性能,并延长了发酵周期。
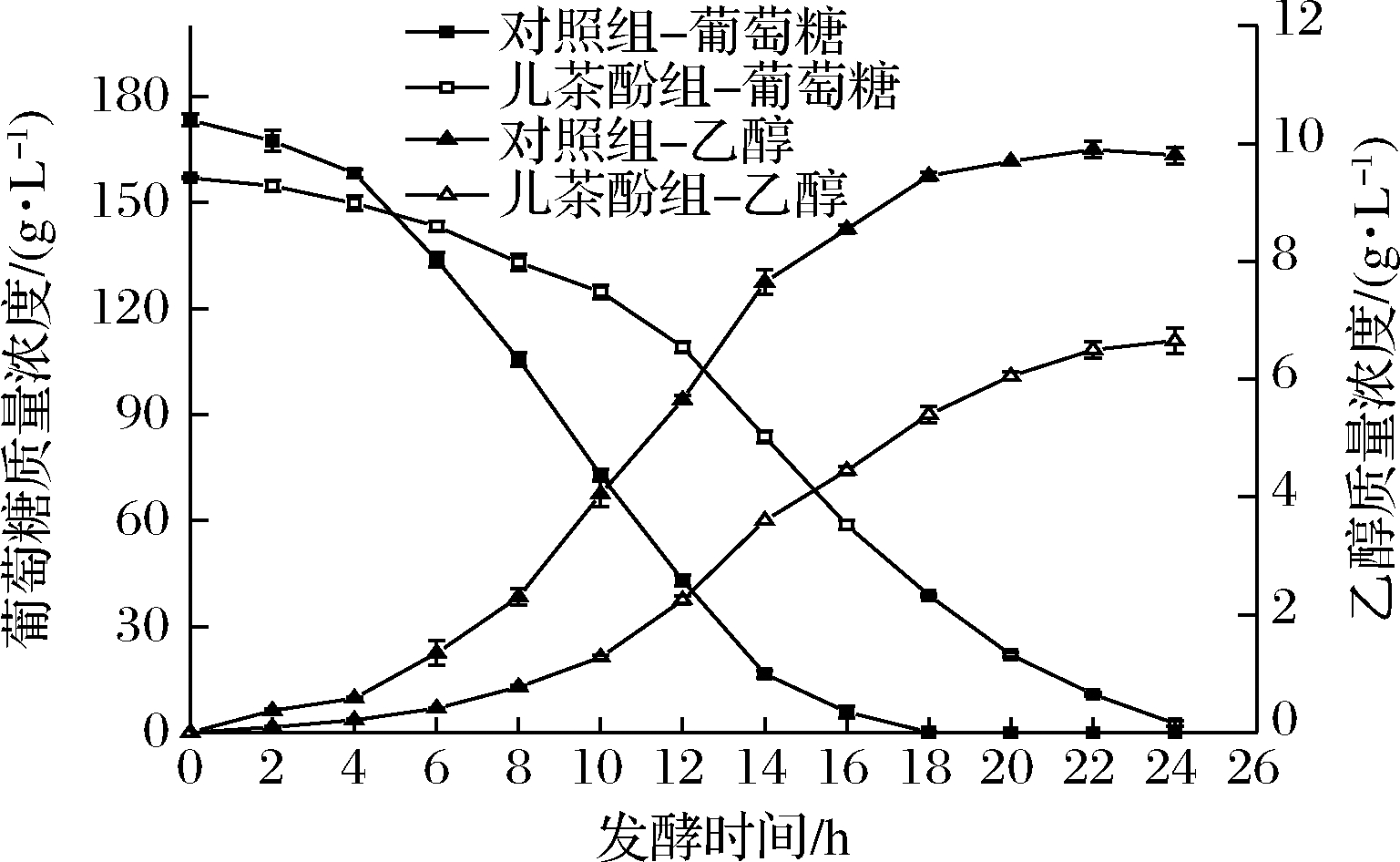
图1 儿茶酚对酵母细胞乙醇发酵的影响
Fig.1 Effect of catechol on ethanol fermentation of yeast cells
2.2 测序数据质控
将原始数据经过一系列数据处理来过滤杂质得到过滤数据(clean data),如表1所示。每个样品平均产出6.85 GB的过滤数据,Q20、Q30均≥89%。用HISAT软件将clean reads比对到参考基因组序列,如表2所示,每个样品平均能比对到基因组中的reads数占总数的90.9%,比对上参考基因组唯一位置的clean reads比例占89.6%,说明测序结果基本达到可接受范围,满足后续实验分析的需求。
表1 测序数据质量统计
Table 1 Quality of sequencing data
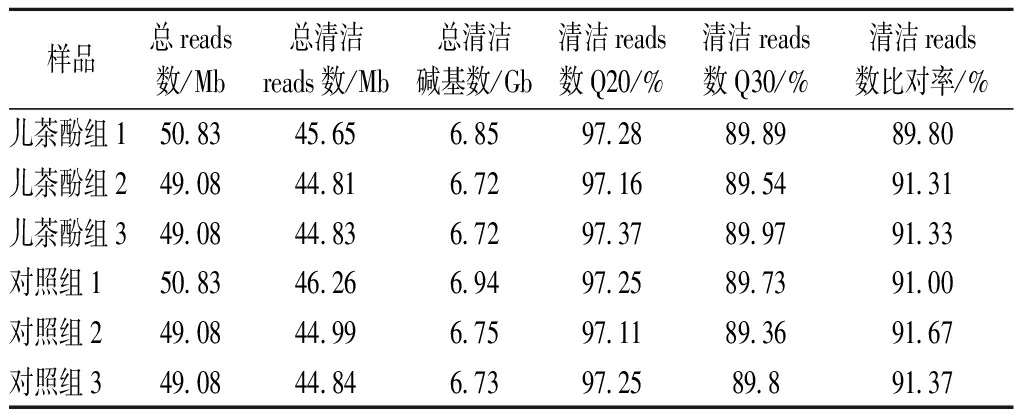
样品总reads数/Mb总清洁 reads数/Mb总清洁碱基数/Gb清洁reads数Q20/%清洁reads数Q30/%清洁reads数比对率/%儿茶酚组150.8345.656.8597.2889.8989.80儿茶酚组249.0844.816.7297.1689.5491.31儿茶酚组349.0844.836.7297.3789.9791.33对照组150.8346.266.9497.2589.7391.00对照组249.0844.996.7597.1189.3691.67对照组349.0844.846.7397.2589.891.37
注:Q20(%),Q30(%):对质控后测序数据进行质量评估,Q20、Q30分别指测序质量在99%和99.9%以上的碱基占总碱基的百分比,一般Q20在85%以上,Q30在80%以上表示测序质量良好
表2 参考基因组比对结果
Table 2 Results of reference genome alignment
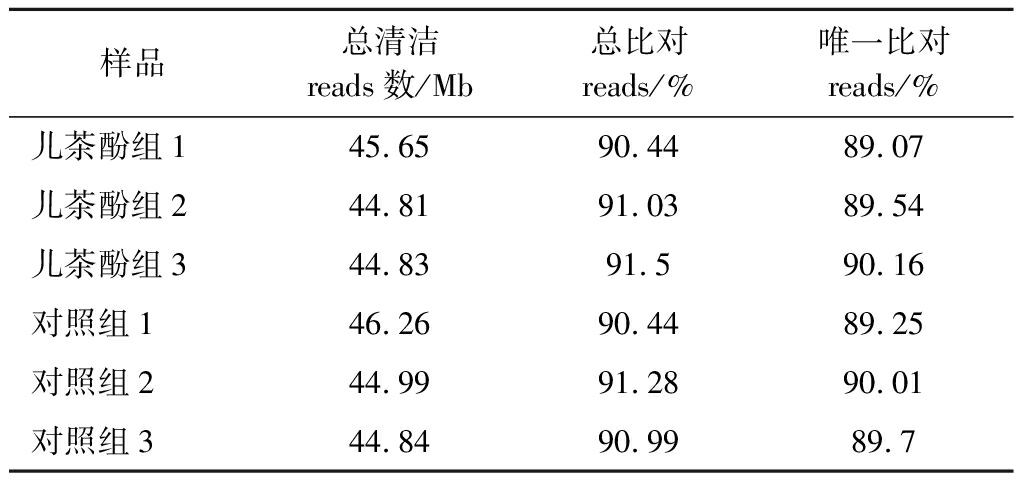
样品总清洁reads数/Mb总比对reads/%唯一比对reads/%儿茶酚组145.6590.4489.07儿茶酚组244.8191.0389.54儿茶酚组344.8391.590.16对照组146.2690.4489.25对照组244.9991.2890.01对照组344.8490.9989.7
2.3 测序样品相关性分析
对照组(ck)和儿茶酚组(cat)均有3个生物学重复,共有6组转录组测序数据。由图2可知处理组与对照组间相关性系数约为0.92,平行样品间相关系数为1或接近于1,说明样品间重复性良好,两组间差异相对较大,结果可信度高。

图2 样品相关性热图
Fig.2 Heat map of sample correlation 注:图中右侧和下侧为样本名称,对照组(ck1、ck2、ck3)和儿茶酚组 (cat1、cat2、cat3);左侧和上侧为样本聚类情况,不同颜色的方块 代表两个样本的相关性高低
2.4 差异表达基因筛选
以q<0.05以及fold change ≥2为筛选条件,对儿茶酚组(cat)和对照组(ck)差异表达基因进行检测,如图3所示,共筛选出223个,其中上调基因172个,下调基因52个。说明样品间存在一定量的差异表达基因,且部分基因在差异对中表达量较大,结果也表明当酿酒酵母受到儿茶酚胁迫时,大部分基因在酵母细胞内出现了差异表达。
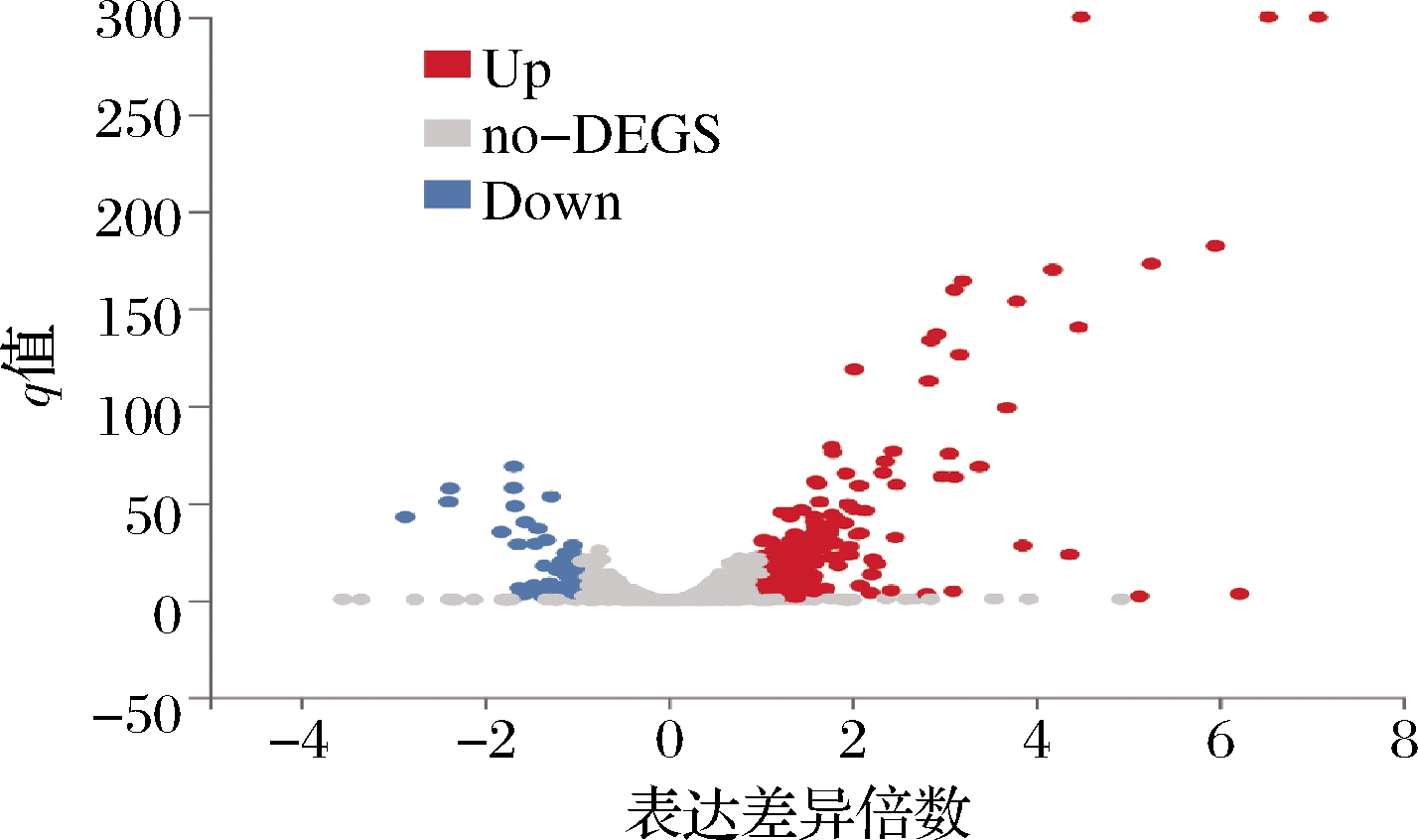
图3 差异表达基因火山图
Fig.3 Volcano map of DEGs 注:Up代表上调差异表达基因,Down代表下调差异表达基因, no-DEGS非显著差异基因
2.5 差异表达基因功能富集和信号通路分析
对筛选出进行差异表达基因进行GO富集分析,如图4所示,横坐标为富集比例,纵坐标为富集的GO term,气泡的大小表示注释到某个GO Term上的差异基因数目,颜色代表富集P值,颜色越深代表P值越小。差异表达基因主要富集在氧化还原过程、氧化还原酶活性、药物跨膜转运、细胞对氧化应激的反应、抗氧化活性、谷胱甘肽代谢过程、半胱氨酸的生物合成过程、葡萄糖跨膜转运以及铁硫团簇结合等通路。在生物体内,不同基因相互协调行使其生物学功能,为了进一步理解这些差异基因在代谢途径里的变化情况,我们采用KOBAS在线分析工具进行了KEGG信号功能注释,结果发现对照组(ck)与儿茶酚组(cat)共富集到67个信号通路,挑选富集最显著的14条通路在图形中进行展示,如图5所示,横坐标为富集比例,纵坐标为KEGG Pathway,气泡的大小表示注释到某个KEGG Pathway上的基因数目,颜色代表富集p值,颜色越深代表P值越小。这些信号通路分析主要涉及到代谢途径、碳代谢、硫代谢、半胱氨酸和蛋氨酸代谢、谷胱甘肽代谢、ABC(ATP-binding cassette) 转运蛋白以及磷酸戊糖途径等多条代谢通路。以上结果表明,儿茶酚胁迫下,酿酒酵母发生了氧化应激,通过改变代谢途径来适应胁迫压力,并加速碳代谢,为酵母抵抗胁迫提供能量以及提高胞内抗氧化活性物质,从而抵抗儿茶酚的胁迫作用。
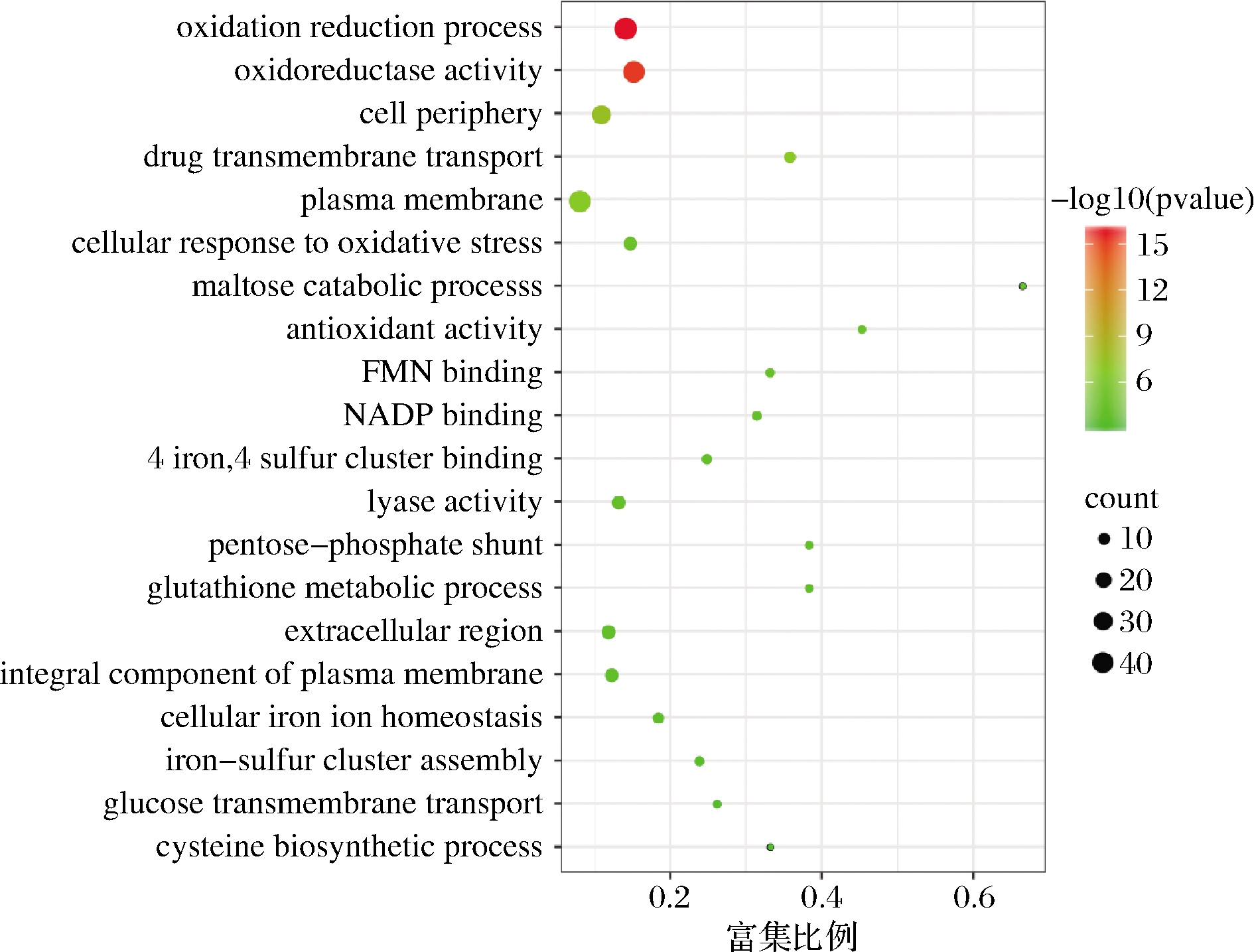
图4 差异表达基因GO富集分析
Fig.4 GO enrichment analysis of DEGs

图5 差异表达基因KEGG富集分析
Fig.5 KEGG pathway analysis of DEGs
3 讨论
3.1 涉及硫代谢相关的差异表达基因分析
在生物体中硫元素主要以半胱氨酸、谷胱甘肽形式存在,半胱氨酸能够调控胞内氧化还原水平,而且还是很多酶的关键氨基酸,参与了众多代谢反应[18-19]。谷胱甘肽是一类重要抗氧化物质,能够将活性氧自由基还原,生成氧化型谷胱甘肽以发挥抗氧化功能,保护细胞内重要细胞器免受损伤[20]。在酿酒酵母中,可以通过硫代谢相关的途径合成半胱氨酸和谷胱甘肽以发挥其生物学功能。本研究中,一些涉及硫代谢相关通路的基因出现了明显的上调,如ssu1、sul2、met16、met14、met5、met17、cys3、gsh1、gsh2、gex1、gex2、glr1等(表3)。ssu1和sul2分别编码亚硫酸盐/硫酸盐转运蛋白,可以将胞外的硫酸盐离子转运入细胞内,增强了硫的吸收。有研究表明,当酵母处于硫饥饿的情况下,sul2会过量表达,如果添加合适的硫源,它们的表达量急剧下降[21]。met16、met14、met5、cys3与半胱氨酸的合成有关,其中腺苷酰硫酸激酶和亚硫酸盐还原酶的编码基因met14和met5分别上调了2.24倍和1.46倍。met16编码3′-磷酸腺苷硫酸还原酶基因的表达量显著提高了3.08倍,该酶催化3′-磷酸腺苷硫酸盐还原为腺苷-3′,5′-二磷酸盐和游离亚硫酸盐,参与硫酸盐同化和甲硫氨酸代谢。半胱氨酸合成酶A和胱硫醚γ-裂解酶的编码基因met17和cys3的表达量分别上调了1.77和1.59倍,这2个酶都参与了半胱氨酸的合成。由此看出,酿酒酵母细胞内半胱氨酸含量的提高不仅与增强的硫吸收有关,还与上调半胱氨酸合成相关的酶有关,从而为谷胱甘肽的合成提供了充足的前体氨基酸。gsh1、gsh2、gex1、gex2这几个基因参与了谷胱甘肽的合成,其表达量也显著的提高。gsh1编码γ-谷氨酰半胱氨酸合成酶,该酶是谷胱甘肽合成代谢过程中的限速酶,催化谷胱甘肽生物合成的第一步,而在本实验中,发现其表达量显著上调2.16倍。有研究表明通过gsh1过表达使得GSH产量提高了约50%,说明gsh1表达量的提高有利于GSH的合成[22]。gsh2编码谷胱甘肽合成酶,其表达量也上调了1.98倍,该酶催化由γ-谷氨酰半胱氨酸和甘氨酸合成谷胱甘肽。谷胱甘肽含量的提高不仅与关键前体氨基酸半胱氨酸的提高有关,还和上调的限速酶编码基因gsh1和gsh2有关。王立梅等[23]发现酿酒酵母突变株在遭受内源性活性氧过氧化氢的胁迫下,通过调节谷胱甘肽合成限速酶活力加强了谷胱甘肽的合成。另外发现上调基因gex1、gex2、glr1,分别编码谷胱甘肽逆转运蛋白和谷胱甘肽还原酶,在谷胱甘肽逆转运蛋白作用下,把谷胱甘肽转运到活性氧较多的细胞器加速活性氧的清除,同时生成氧化型谷胱甘肽,氧化型谷胱甘肽再被谷胱甘肽还原酶(GLR1)还原成还原性谷胱甘肽继续参与抗氧化作用。KIM等[24]研究发现过表达了谷胱甘肽合成途径的基因gsh1和glr1,可以加强酵母细胞对糠醛的耐受能力。
3.2 涉及氧化还原相关的差异表达基因分析
通过转录组学的分析,发现一些参与氧化应激相关的基因表达量显著提高(表3)。如编码超氧化物歧化酶(superoxide dismutase,SOD)的基因sod1、sod2,SOD是酿酒酵母细胞内重要的抗氧化酶,其中sod1编码含铜和锌的超氧化物歧化酶存在细胞质中,sod2编码含锰的超氧化物歧化酶存在线粒体中,该酶能够催化超氧负离子和H+的反应,生成O2和H2O2。上调基因ctt1、ira1编码的过氧化氢酶作用下,将H2O2催化为H2O,从而清除儿茶酚胁迫下氧化应激产生的活性氧。因此,猜测在儿茶酚胁迫下酵母细胞产生大量的活性氧,酿酒酵母可以合成多种酶类来清除活性氧产生的毒性。
酿酒酵母为了降低儿茶酚对细胞的毒性作用,还可以通过上调功能还原酶和大量具有NAD(P)H依赖性还原酶的活性来实现,本研究发现了一些显著上调的基因(见表3),如ddi3、ddi2、oye3、oye2、ecm4,其中ddi3、ddi2、ecm4分别编码氰胺水合酶、谷胱甘肽-氢醌还原酶,都显著高表达,DDI3、DDI2对氰胺具有解毒作用,ecm4能够依赖性地将有毒的GS-三氯对苯二酚还原为三氯对苯二酚,因此猜测这3个基因参与了酿酒酵母对儿茶酚解毒作用。oye3、oye2编码老黄酶,研究报道这2个基因可能在甾醇代谢、氧化应激反应和细胞程序性死亡中具有潜在作用[25]。另外有研究发现老黄酶可介导酿酒酵母对丙烯醛的抗性,而丙烯醛为生物系统中活性氧与生物膜脂质过氧化的产物[26-27]。因此,认为oye2、oye3在本研究中介导了由活性氧引起的脂质过氧化产物的抗性,从而解除此类化合物对细胞的毒性,提高酿酒酵母在儿茶酚胁迫下的耐受性。此外,研究发现一些重要的ABC转运蛋白基因参与了酿酒酵母在儿茶酚胁迫下的调控作用,如pdr10、pdr5、pdr18、yor1、ycf1、snq2。其中pdr10、pdr18和pdr5编码的ABC转运蛋白起药物/毒素转运和多药物外排泵的作用,pdr10主要由Pdr1p和Pdr3p调节,pdr5受Pdr1p积极调控的多药转运蛋白,研究表明pdr10、pdr5在细胞排毒代谢和多效性耐药过程起着重要的作用[28]。基于以上结果,可以推测酿酒酵母通过ABC转运蛋白家族将儿茶酚运送到胞外进行细胞排毒,从而介导酿酒酵母对儿茶酚的耐受性。
3.3 涉及碳代谢相关的差异表达基因分析
酿酒酵母应对儿茶酚的胁迫,消耗辅因子NAD(P)H和ATP导致氧化还原不平衡,从而使细胞代谢损伤,面对这样的问题,酿酒酵母选择了改变代谢途径来适应胁迫压力。一些相关的基因表达水平发生了变化(表3),如zwf1、gnd1、gnd2、tdh1、err1、err3以及编码铁硫簇相关的基因nbp35、dre2、tah18、nar1、nfs1、isu2。其中zwf1编码的6-磷酸葡萄糖脱氢酶,戊糖磷酸途径的第一步反应,催化葡萄糖-6-磷酸生成磷酸葡萄糖酸-δ-内脂,是酵母细胞中NADPH产生的主要途径之一,参与适应氧化应激反应。而gnd1和gnd2编码6-磷酸葡萄糖酸脱氢酶,该酶催化磷酸葡萄糖酸生成核酮糖-5-磷酸,在戊糖磷酸途径中催化NADPH再生反应。这2个限速酶通过磷酸戊糖途径产生大量的NADPH,为细胞的各种合成反应提供还原力,维持了细胞内氧化还原水平。编码3-磷酸甘油醛脱氢酶基因tdh1表达量上调了3.58倍,该酶主要参与糖酵解中催化3-磷酸甘油醛氧化成1,3-二磷酸甘油酸生成NADH。err1、err3编码烯醇化酶,一种磷酸丙酮酸水合酶,催化2-磷酸甘油酸生成磷酸烯醇式丙酮酸,加速了磷酸烯醇式丙酮酸向丙酮酸的转化生成ATP。nbp35和isu2是铁硫蛋白合成所需的线粒体蛋白,参与线粒体和胞质蛋白的Fe-S簇组装。nfs1编码半胱氨酸脱硫酶,在铁硫簇组装中提供硫原子。nar1为胞质铁硫蛋白组装的亚基,ZHAO等[29]报道,nar1缺失导致细胞寿命缩短和对百草枯的敏感性提高,对氧化应激具有保护作用。
表3 儿茶酚胁迫下酿酒酵母应答的相关基因
Table 3 Related genes of S.cerevisiae response to catechol stress

类别基因名基因注释差异倍数对数值显著性q值硫代谢met163′-磷酸腺苷硫酸还原酶1.74 498 8847.87E-35met14腺苷酰硫酸激酶1.287 934 1975.06E-21met17OAH/OAS硫解酶1.177 661 0291.84E-21ssu1质膜亚硫酸盐泵2.924 176 6461.43E-137sul2硫酸盐转运蛋白0.991 245 4176.49E-22cys3胱硫醚γ-裂解酶1.059 852 3187.21E-24gsh1γ-谷氨酰半胱氨酸合成酶1.234 827 8647.42E-46gsh2谷胱甘肽合成酶1.103 533 7091.29E-19gex1谷胱甘肽逆转运蛋白2.426 406 8751.03E-05gex2谷胱甘肽逆转运蛋白3.104 414 5282.40E-05glr1谷胱甘肽还原酶1.925 979 0162.41E-40氧化还原过程sod1超氧化物歧化酶1.452 385 9992.72E-47sod2超氧化物歧化酶1.354 984 4176.34E-17ddi3氰胺水合酶7.077 868 4180oye3老黄酶5.962 940 4255.32E-183oye2老黄酶4.188 357 7668.90E-171ecm4谷胱甘肽-氢醌还原酶4.496 222 5370pdr10ABC转运蛋白2.986 087 6012.23E-64pdr5ABC转运蛋白1.648 441 3422.29E-51pdr18ABC转运蛋白1.256 337 9985.83E-13ycf1ABC转运蛋白1.345 595 5422.40E-31zwf16-磷酸葡萄糖脱氢酶1.304 363 3467.38E-46碳代谢gnd16-磷酸葡萄糖酸脱氢酶1.40 631 7192.10E-14gnd26-磷酸葡萄糖酸脱氢酶2.064 575 9541.20E-34tdh13-磷酸甘油醛脱氢酶1.959 981 0495.10E-50err1烯醇化酶1.919 877 0254.14E-24err3烯醇化酶1.976 464 0195.29E-24nbp35铁硫蛋白1.282 229 9675.54E-27dre2Fe-S簇组装蛋白1.79 572 0387.19E-77tah18二黄素氧化还原酶Ⅰ1.168 374 9261.94E-18nar1铁硫蛋白1.051 821 2038.67E-21nfs1半胱氨酸脱硫酶1.003 160 5313.19E-23isu2铁硫蛋白1.071 233 1223.25E-13
tah18编码NADPH依赖的二黄素氧化还原酶Ⅰ,它们参与了黄素转运蛋白和生物合成,直接参与氧化还原过程,该过程将电子转移到线粒体的电子传输链中并和Fe-S簇组装蛋白(DRE2)之间的相互作用,对于调节氧化应激诱导的细胞死亡和胞质中Fe-S蛋白的合成至关重要,且DRE2-TAH18复合物可以防止H2O2诱导的细胞死亡[30]。由此可知,酿酒酵母在儿茶酚胁迫下,通过改变代谢途径以及上调铁硫蛋白合成相关的基因,在氧化应激反应中维持细胞内氧化还原水平和充足的能量发挥着重要的作用,从而提高了酵母细胞对儿茶酚的抵抗能力。
4 结论
酿酒酵母对木质纤维素水解液抑制剂儿茶酚的适应性和耐受性存在复杂的基因相互作用、调控网络和协同调控。本研究在转录组水平上研究了酿酒酵母潜在的儿茶酚应激反应机制。儿茶酚胁迫下可引起酿酒酵母氧化性损伤,影响细胞内相关酶活性,从而影响细胞的正常生长。酿酒酵母通过提高硫代谢途径来响应儿茶酚的胁迫,加强了半胱氨酸和谷胱甘肽的合成,在抗氧化、清除活性氧方面有着重要的作用;其次,通过上调涉及氧化还原相关功能酶的基因表达来响应儿茶酚胁迫,从而清除酵母细胞内过多的ROS,并在多种ABC转运蛋白作用下对酿酒酵母细胞排毒;最后,通过加速碳代谢,为酵母抵抗胁迫提供能量以及维持细胞内NAD(P)H水平,在酵母的适应和能量再平衡中发挥作用,使酵母能够生存和适应儿茶酚胁迫。通过本研究探讨酿酒酵母儿茶酚胁迫响应机制,为进一步研究提高酿酒酵母酚类物质耐受性的方法提供理论指导意义。
[1] MOSIER N,WYMAN C,DALE B,et al.Features of promising technologies for pretreatment of lignocellulosic biomass[J].Bioresource Technology,2005,96(6):673-686.
[2] 齐本坤,万印华.木质纤维素预处理液膜分离脱毒技术研究进展[J].生物产业技术,2018(4):56-67.
QI B K,WAN Y H.Research progress on application of membrane separation technology in detoxification of lignocellulosic prehydrolysates[J].Biotechnology & Business,2018(4):56-67.
[3] HATAKEYAMA H,HATAKEYAMA T.Lignin structure,properties,and applications[M].Berlin,Heidelberg:Springer Berlin Heidelberg,2009.
[4] 林贝, 李健秀,刘雪凌.木质纤维素水解副产物对乙醇发酵的影响及应对措施[J].生物技术通报,2018,34(3):23-30.
LIN B,LI J X,LIU X L.The effects of by-products of hydrolyzing lignocellulose on ethanol fermentation and relevant countermeasures[J].Biotechnology Bulletin,2018,34(3):23-30.
[5] 谢瑶嬛, 曹淑贞,王风芹,等.木质纤维素水解液中酚类抑制物去除的研究进展[J].纤维素科学与技术,2016,24(2):93-98.
XIE Y H,CAO S Z,WANG F Q,et al.Recent advances in detoxification of phenolic compounds in lignocellulose hydrolysate[J].Journal of Cellulose Science and Technology,2016,24(2):93-98.
[6] SARAVANAKUMAR T,PARK H S,MO A Y,et al.Detoxification of furanic and phenolic lignocellulose derived inhibitors of yeast using laccase immobilized on bacterial cellulosic nanofibers[J].Journal of Molecular Catalysis B:Enzymatic,2016,134:196-205.
[7] POPOFF T,THEANDER O,RØMMING C,et al.Formation of aromatic compounds from carbohydrates.part III.reaction of D-glucose and D-fructose in slightly acidic,aqueous solution[J].Acta Chemica Scandinavica,1976,30b:397-402.
[8] WANG H Y,LI Q,KUANG X L,et al.Functions of aldehyde reductases from Saccharomyces cerevisiae in detoxification of aldehyde inhibitors and their biotechnological applications[J].Applied Microbiology and Biotechnology,2018,102(24):10 439-10 456.
[9] 庄军平, 林鹿,庞春生,等.木质纤维素稀水解液脱毒研究进展[J].现代化工,2009,29(2):19-23.
ZHUANG J P,LIN L,PANG C S,et al.Research advances in detoxification of lignocellulose hydrolysates-making[J].Modern Chemical Industry,2009,29(2):19-23.
[10] PALMQVIST E,HAHN-H GERDAL B.Fermentation of lignocellulosic hydrolysates.II:Inhibitors and mechanisms of inhibition[J].Bioresource Technology,2000,74(1):25-33.
GERDAL B.Fermentation of lignocellulosic hydrolysates.II:Inhibitors and mechanisms of inhibition[J].Bioresource Technology,2000,74(1):25-33.
[11] ZHANG J M,GENG A L,YAO C Y,et al.Effects of lignin-derived phenolic compounds on xylitol production and key enzyme activities by a xylose utilizing yeast Candida athensensis SB18[J].Bioresource Technology,2012,121:369-378.
[12] KEWELOH H,WEYRAUCH G,REHM H J.Phenol-induced membrane changes in free and immobilized Escherichia coli[J].Applied Microbiology and Biotechnology,1990,33(1):66-71.
[13] ISHIDA Y,NGUYEN T T M,IZAWA S.The yeast ADH7 promoter enables gene expression under pronounced translation repression caused by the combined stress of vanillin,furfural,and 5-hydroxymethylfurfural[J].Journal of Biotechnology,2017,252:65-72.
[14] VARGA E,KLINKE H B,RÉCZEY K,et al.High solid simultaneous saccharification and fermentation of wet oxidized corn stover to ethanol[J].Biotechnology and Bioengineering,2004,88(5):567-574.
[15] 李文莉, 缪冶炼,陈介余,等.木质素降解物对酿酒酵母的毒性[J].生物加工过程,2015,13(1):82-88.
LI W L,MIAO Y L,CHEN J Y,et al.Inhibition of lignin degradation products on Saccharomyces cerevisiae[J].Chinese Journal of Bioprocess Engineering,2015,13(1):82-88.
[16] KLINKE H B,THOMSEN A B,AHRING B K.Inhibition of ethanol-producing yeast and bacteria by degradation products produced during pre-treatment of biomass[J].Applied Microbiology and Biotechnology,2004,66(1):10-26.
[17] BüCHERT J,PULS J,POUTANEN K.The use of steamed hemicellulose as substrate in microbial conversions[J].Applied Biochemistry and Biotechnology,1989,20-21(1):309-318.
[18] SPADARO D,YUN B W,SPOEL S H,et al.The redox switch:Dynamic regulation of protein function by cysteine modifications[J].Physiologia Plantarum,2010,138(4):360-371.
[19] 乔新华, 胡明曦,张玉英,等.细胞的氧化还原平衡调控及巯基修饰[J].生命科学,2015,27(3):374-382.
QIAO X H,HU M X,ZHANG Y Y,et al.Cellular redox regulation and thiol modification[J].Chinese Bulletin of Life Sciences,2015,27(3):374-382.
[20] MAILLOUX R J,MCBRIDE S L,HARPER M E.Unearthing the secrets of mitochondrial ROS and glutathione in bioenergetics[J].Trends in Biochemical Sciences,2013,38(12):592-602.
[21] BOER V M,DE WINDE J H,PRONK J T,et al.The genome-wide transcriptional responses of Saccharomyces cerevisiae grown on glucose in aerobic chemostat cultures limited for carbon,nitrogen,phosphorus,or sulfur[J].Journal of Biological Chemistry,2003,278(5):3 265-3 274.
[22] FAN X Y,HE X P,GUO X N,et al.Increasing glutathione formation by functional expression of the γ-glutamylcysteine synthetase gene in Saccharomyces cerevisiae[J].Biotechnology Letters,2004,26(5):415-417.
[23] 王立梅, 任清华,郑丽雪,等.内源性氧化胁迫促进酿酒酵母合成谷胱甘肽的潜在机制分析[J].食品科学,2017,38(4):26-31.
WANG L M,REN Q H,ZHENG L X,et al.Elucidation of the underlying mechanism by which endogenous oxidative stress promotes glutathione synthesis of Saccharomyces cerevisiae[J].Food Science,2017,38(4):26-31.
[24] KIM D,HAHN J S.Roles of the Yap1 transcription factor and antioxidants in Saccharomyces cerevisiaes tolerance to furfural and 5-hydroxymethylfurfural,which function as thiol-reactive electrophiles generating oxidative stress[J].Applied and Environmental Microbiology,2013,79 (16):5 069-5 077.
[25] ODAT O,MATTA S,KHALIL H,et al.Old yellow enzymes,highly homologous FMN oxidoreductases with modulating roles in oxidative stress and programmed cell death in yeast[J].Journal of Biological Chemistry,2007,282(49):36 010-36 023.
[26] TROTTER E W,COLLINSON E J,DAWES I W,et al.Old yellow enzymes protect against acrolein toxicity in the yeast Saccharomyces cerevisiae[J].Applied and Environmental Microbiology,2006,72(7):4 885-4 892.
[27] UCHIDA K,KANEMATSU M,MORIMITSU Y,et al.Acrolein is a product of lipid peroxidation reaction[J].Journal of Biological Chemistry,1998,273(26):16 058-16 066.
[28] MA M G,LIU Z L.Comparative transcriptome profiling analyses during the lag phase uncover YAP1,PDR1,PDR3,RPN4 and HSF1 as key regulatory genes in genomic adaptation to the lignocellulose derived inhibitor HMF for Saccharomyces cerevisiae[J].BMC Genomics,2010,11:660.
[29] ZHAO W,FANG B X,NIU Y J,et al.Nar1 deficiency results in shortened lifespan and sensitivity to paraquat that is rescued by increased expression of mitochondrial superoxide dismutase[J].Mechanisms of Ageing and Development,2014,138:53-58.
[30] VERNIS L,FACCA C,DELAGOUTTE E,et al.A newly identified essential complex,Dre2-Tah18,controls mitochondria integrity and cell death after oxidative stress in yeast[J].PLoS One,2009,4(2):e4 376.