短小芽孢杆菌(Brevibacillus choshinensis)是一种革兰氏阳性菌,具有胞外蛋白酶活性低、蛋白合成与分泌能力强以及非致病性等优点[1]。相较于目前被广泛用于蛋白表达的枯草芽孢杆菌(Bacillus subtilis),B.choshinensis不仅具有更低的胞外蛋白酶活性[2],而且其胞外分泌蛋白质量浓度可高达30 g/L[3],这几乎是B.subtilis最高胞外蛋白浓度的1.5倍[4]。因此,B.choshinensis被逐渐用于重组表达目的蛋白[5-7]。然而,目前常用于B.choshinensis表达系统的强启动子仅有P2启动子[8],高效强启动子的缺乏严重限制了B.choshinensis表达系统的进一步开发及应用[1]。
启动子是一个重要的基因表达调控元件,选择高效强启动子是促进目的基因高效表达的一种有效方法[9]。高效强启动子可通过对已有启动子的关键位点(如-35或-10区)进行修饰以及挖掘新启动子2种方式获得[10-11]。随着转录组测序(RNA-Seq)技术的发展,目前可基于基因的mRNA含量来挖掘新强启动子,且该方法较传统载体陷阱捕捉的方法具有时间短、劳动成本低等优点[12-14]。因此,基于转录组数据挖掘新强启动子的方法逐渐得到推广[15]。例如,LIU等[16] 基于地衣芽孢杆菌的转录组数据及基因的RPKM(reads per kilobase of exon model per million mapped reads)值挖掘了一个新强启动子pBL9。当谷氨酰转肽酶为报告蛋白时,由启动子pBL9介导的重组酶活性是启动子P43介导的重组酶活性的1.23倍[16]。MENG等[17]基于B.subtilis的转录组数据及基因的RPKM值挖掘了一个新强启动子PsodA。当普鲁兰酶为报告蛋白时,由启动子PsodA介导的重组酶活性是启动子P43介导的重组酶活性的1.82倍[17]。由此可推,基于B.choshinensis转录组数据分析将是获得B.choshinensis内源性新强启动子的一种有效手段。
在本研究中,首先制备了RNA-Seq样品并利用RNA-Seq技术获得了B.choshinensis的转录组数据。然后基于转录组数据挖掘了B.choshinensis内源性新强启动子。最后通过其他报告蛋白以及3 L罐发酵进一步考察了筛选到的新内源性强启动子的通用性及有效性。本研究为B.choshinensis表达系统提供了更多的强启动子,促进了B.choshinensis表达系统的开发与应用。
1 材料与方法
1.1 材料
1.1.1 菌种与质粒
菌株B.choshinensis HPD31-SP3、B.choshinensis/pNCMO2-P2-amyS(BCWPS)、B.choshinensis/pNCMO2-P2-SI(BCWPSI)及B.choshinensis/pNCMO2-P2-Cut(BCWPCUT)、重组表达载体pNCMO2-P2-amyS(pNCP2S)、pNCMO2-P2-SI(pNCP2SI)和pNCMO2-P2-Cut(pNCP2Cut)均为作者所在课题组前期保藏,其他菌株及质粒均由本研究所构建。
1.1.2 实验试剂
DNA聚合酶,大连宝生生物工程有限公司;质粒提取试剂盒、DNA胶回收试剂盒、细菌RNA提取试剂盒,北京天根生化科技有限公司;One-step Cloning kit一步克隆试剂盒,南京诺唯赞生物科技有限公司;其他试剂均为国产或进口的分析纯级。
1.1.3 仪器与设备
PCR仪,美国Bio-Rad公司;摇床培养箱,上海知楚生物科技有限公司;3 L发酵罐,美国NBS公司;离心机,德国Eppendorf公司;可见分光光度计,北京优普通用科技有限公司;NanoDrop2000,赛默飞世尔科技(中国)有限公司。
1.1.4 培养基
TM液体培养基、TM固体培养基、SHC电转培养基、微量金属离子液均参考邹亮等[18]的研究报告。3 L 罐发酵基础培养基组分包括:15 g/L牛肉浸膏,15 g/L多聚蛋白胨,10 g/L葡萄糖,10 mL/L微量金属离子液。3 L罐发酵补料培养基为300 g/L葡萄糖。
1.2 实验方法
1.2.1 分子操作方法
重组载体pNCPAS、pNCPBS、pNCPCS、pNCPDS、pNCPES、pNCPFS、pNCPGS、pNCPHS、pNCPIS、pNCPJS、pNCPKS、pNCPLS、pNCPMS、pNCPNS、pNCPOS、pNCPPS、pNCPQS、pNCPRS、pNCPSS分别由启动子PA、PB、PC、PD、PE、PF、PG、PH、PI、PJ、PK、PL、PM、PN、PO、PP、PQ、PR、PS替换重组载体pNCP2S中的P2启动子获得。重组载体pNCESI和pNCECut分别由启动子PE替换重组载体pNCP2SI和pNCP2Cut中的启动子P2获得。不同启动子片段分别用表1中对应的引物,依B.choshinensis HPD31-SP3基因组为模板,通过PCR获得。载体片段pNCMO2-amyS、pNCMO2-SI和pNCMO2-Cut均是通过表1中的FP/RP为引物,分别依重组表达载体pNCP2S、pNCP2SI和pNCP2Cut为模板,通过PCR获得。含有不同启动子的重组表达载体是由对应的启动子片段与载体片段pNCMO2-amyS或pNCMO2-SI或pNCMO2-Cut通过一步克隆试剂盒进行连接获得。将含有不同启动子的重组表达载体通过电转化的方式转到B.choshinensis HPD31-SP3中分别获得对应的重组菌BCWAS、BCWBS、BCWCS、BCWDS、BCWES、BCWFS、BCWGS、BCWHS、BCWIS、BCWJS、BCWKS、BCWLS、BCWMS、BCWNS、BCWOS、BCWSP、BCWQS、BCWRS、BCWSS、BCWESI和BCWECUT。B.choshinensis HPD31-SP3的电转感受态的制备及重组质粒的电转化方法均参考LI等[19]的研究报告。
表1 本研究中所使用的引物
Table 1 Primers used in this study

引物名称碱基序列 (5′-3′)F-PAAAAACTTTAAATTTTTTTCGAACCCTAAAACCGGTACTGTTACTTTR-PAACGACCCTTCTTTTTTTCATTGTTTACACCTCCCGTGCTCTAF-PBAAAACTTTAAATTTTTTTCGTTCAGGCGAAGCATGTCATCTR-PBACGACCCTTCTTTTTTTCATACGACTCACTCCTCTCTTTCTTATAACGF-PCAAAACTTTAAATTTTTTTCGAACGCTCTTCGCAAACTGGAR-PCACGACCCTTCTTTTTTTCATCGCTTCTTCTCCCCCATTTGF-PDAAAACTTTAAATTTTTTTCGGATGAAACGTCGGTGCCAGCR-PDACGACCCTTCTTTTTTTCATCGGTAACGCCTCCTTTAACCTF-PEAAAACTTTAAATTTTTTTCGGGAAGTCGTCCGTTACCTGCTR-PEACGACCCTTCTTTTTTTCATTGAATCTCCGGCCTCCCTTF-PFAAAACTTTAAATTTTTTTCGTGGCCGAATACCAGAGCTTCR-PFACGACCCTTCTTTTTTTCATTAGCTCCTTGAGGTCAACACTCACF-PGAAAACTTTAAATTTTTTTCGTAACGCATTGCCACTGGAAAR-PGACGACCCTTCTTTTTTTCATTAGTGAACCTCCCTTCGCCAF-PHAAAACTTTAAATTTTTTTCGTGTTAAAGCGTATGAAAAACTGTCTACCR-PHACGACCCTTCTTTTTTTCATCTGTTCTTCAATCCATATGATGCAGF-PIAAAACTTTAAATTTTTTTCGTATGGCGGATGGAGTGGGAR-PIACGACCCTTCTTTTTTTCATCCACCGCCATTCTTGTCTCAF-PJAAAACTTTAAATTTTTTTCGCTTCGTCTTCTTCATCTTCTTCGTCR-PJACGACCCTTCTTTTTTTCATATTAGAATCTCTCCTTTTTTATAATTATTATTGAF-PKAAAACTTTAAATTTTTTTCGTGTACGCTGTAAAAACCATGGCR-PKACGACCCTTCTTTTTTTCATTTATTTGTTAACTAGTATTTTAATTTGGTTAACAF-PLAAAACTTTAAATTTTTTTCGTATATTTCATCGTGTCCTGCCGR-PLACGACCCTTCTTTTTTTCATCGGGTTCGATTTTCTGTCATGF-PMAAAACTTTAAATTTTTTTCGCATGAACTGCAACCTGCAGAAGR-PMACGACCCTTCTTTTTTTCATCATGAGCGTAAACAGGATTTTACGF-PNAAAACTTTAAATTTTTTTCGGATGTGAAAGGATAGCTGCGTGR-PNACGACCCTTCTTTTTTTCATGATTGTACGTCCTACACCCGACCF-POAAAACTTTAAATTTTTTTCGAAAAATTGATATTGGCGCTCTTTTR-POACGACCCTTCTTTTTTTCATTTTCTAAACAGACATTTCTCCGTGGF-PPAAAACTTTAAATTTTTTTCGTTGAAGTCGGCGTAGCACACAR-PPACGACCCTTCTTTTTTTCATTAAACAAACCCTCCTTCAAAACGF-PQAAAACTTTAAATTTTTTTCGTAAGGACATGTTCCTCGAAGAGCR-PQACGACCCTTCTTTTTTTCATAACTTCGTATTGACGCATACCTTTTF-PRAAAACTTTAAATTTTTTTCGCCGCTGTTCATCTGGCTGGR-PRACGACCCTTCTTTTTTTCATTCGTTTCACTCCTTCCTGCGF-PSAAAACTTTAAATTTTTTTCGGATATGCTCCGTCGAGAGCAAR-PSACGACCCTTCTTTTTTTCATTGGCGGAACCCAATTGCCFP ATGAAAAAAAGAAGGGTCGTTAACARP CGAAAAAAATTTAAAGTTTTTTTCGT
1.2.2 重组菌培养方法
摇瓶种子液培养、摇瓶发酵培养及3 L罐发酵培养方法均参考邹亮等[18]的研究报告,其中的区别在于摇瓶发酵及3 L罐发酵是在本研究所列的培养基中进行,并且发酵温度均为33 ℃。
1.2.3 菌体浓度测定方法
菌体浓度是通过测定发酵液在600 nm处的光密度值(OD600)获得,同时为保证数据的准确性,需要对发酵液进行适当的稀释使600 nm处的可见分光光度计读数为0.2~0.8。
1.2.4 酶活性测定方法
α-淀粉酶、蔗糖异构酶及角质酶的酶活性测定方法及单位酶活性定义分别参考李祝等[20]、刘军彤等[21]以及SU等[22]的研究报告。
1.2.5 RNA样品的提取及检测、反转录文库构建及测序
通过RNA提取试剂盒提取发酵菌体的总RNA。通过1%的琼脂糖核酸电泳分析RNA样品的完整度和纯度;通过NanoDrop 2000分析RNA样品的浓度及纯度;通过Agilent 2100分析RNA样品的完整度值(RNA integrity number,RIN)。检测合格后的RNA样品在干冰的保护下送往武汉华大医学检验所有限公司进行后续的RNA富集纯化、反转录文库的构建及测序。
1.2.6 数据分析
测序下机后的Raw Reads通过SOAPnuke软件和trimmomatic软件过滤掉包含接头、低质量以及未知碱基含量高的数据从而获得Clean Reads。以B.choshinensis HPD31-SP3基因组(GenBank:CP069127)为参考基因组,通过HISAT软件将获得的Clean Reads与参考基因组进行比对分析。通过RSEM软件计算各基因表达水平,并利用FPKM(fragment per kilobases per millionreads)值对不同基因的表达水平进行标准化处理。任意2个样品间的Pearson系数是利用R软件计算所得。利用GO数据库(http://www.geneontology.org/)和KEGG数据库(https://www.kegg.jp/kegg/pathway.html)分别对鉴定到的基因进行GO功能和KEGG功能的分类分析;利用DOOR数据库(http://161.117.81.224/DOOR3/)、BPROM数据库(http://www.softberry.com/berry.phtml?topic=bprom&group=programs & subgroup = gfindb)以及DBTBS数据库(http://dbtbs.hgc.jp/)分别对目的基因的操纵子结构、启动子的-35和-10区以及启动子的σ因子和转录因子进行预测。
2 结果与分析
2.1 RNA-Seq样品的制备
将重组菌BCWPS进行3 L罐发酵培养,提取处于稳定期前期的菌体总RNA作为RNA-Seq测序样品。另外,为了保证实验数据的科学有效性,将重组菌BCWPS的3 L罐发酵实验进行3次生物学重复。如图1重组菌发酵曲线所示,在3批生物学重复实验中,重组菌BCWPS均在发酵培养48 h时进入稳定期。因此,提取发酵48 h时的菌体总RNA作为RNA-Seq测序样品。为了方便后续研究,将由3批生物学重复实验获得的样品分别命名为B_1_T、B_2_T和B_3_T。

空心图标为OD600,实心图标为α-淀粉酶活性
图1 重组菌BCWPS的3 L罐发酵曲线
Fig.1 The 3 L fermenter fermentation curve of recombinant
strain BCWPS
2.2 RNA样品的质量检测
质量合格的RNA样品是获得有效RNA-Seq数据的重要保障。浓度、纯度及完整度是评判RNA样品质量的3个重要指标。其中评判RNA样品纯度的指标包括OD260/OD280和OD260/OD230,评判RNA样品完整度的指标主要为RIN。当OD260/OD280值为1.8~2.2,且OD260/OD230值>2.0时则认为RNA样品具有良好的纯度;当RIN越接近10时,RNA样品的完整度越高。一般情况下,当RIN>8 时即可认为样品的完整度较好。对于细菌样品,为保证有足量的RNA(≥1 μg)用于cDNA文库的构建,RNA样品的质量浓度应>150 ng/μL。如表2所示,样品B_1_T、B_2_T和B_3_T对应RNA样品的OD260/OD280、OD260/OD230及RIN均达标且质量浓度均>1 000 ng/μL,因此均可以用于后续的cDNA建库分析。
表2 RNA样品的质量检测结果
Table 2 Quality test results of RNA samples
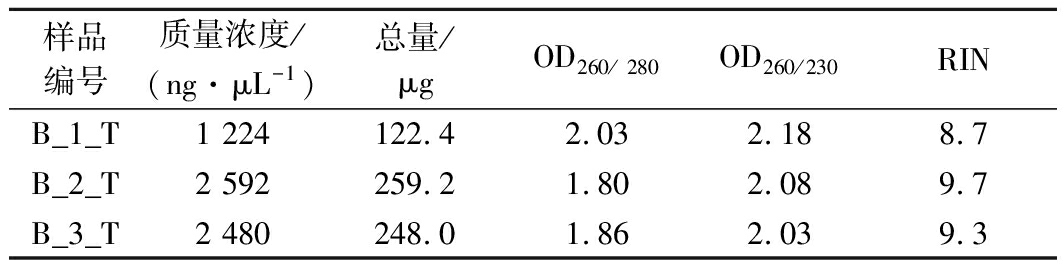
样品编号质量浓度/(ng·μL-1)总量/μgOD260/ 280OD260/230RINB_1_T1 224122.42.032.188.7B_2_T2 592259.21.802.089.7B_3_T2 480248.01.862.039.3
2.3 RNA-Seq结果的统计分析
2.3.1 测序数据的准确度分析
由表3中的RNA-Seq数据可知,各样品的Clean Bases均≥3.40 Gb;Clean Reads Q20均>98%;Clean Reads Q30均>91%;Clean Reads Ratio均>97%。由此可知,获得的转录组数据准确度较高,可用于后续分析。
表3 RNA-Seq数据统计表
Table 3 Statistical table of RNA-Seq data
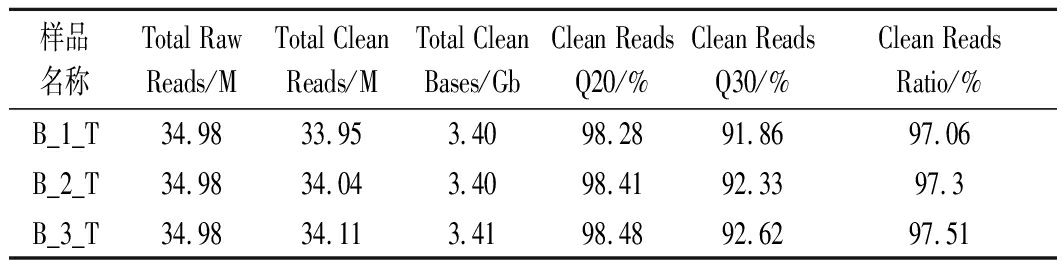
样品名称Total Raw Reads/MTotal Clean Reads/MTotal Clean Bases/GbClean Reads Q20/%Clean Reads Q30/%Clean Reads Ratio/%B_1_T34.9833.953.4098.2891.8697.06B_2_T34.9834.043.4098.4192.3397.3B_3_T34.9834.113.4198.4892.6297.51
注:Q20和Q30分别表示测序质量在99%和99.9%以上的条目占总条目的比例,一般当Q20值>85%或Q30值>80%即可认为具有较好的准确度
2.3.2 测序数据与参考基因组的匹配度分析
由表4中的Clean Reads与B.choshinensis HPD31-SP3基因组(GenBank:CP069127)的比对结果可知,各样品的Total Mapping值均>80%,Uniquely Mapping值均>75%。由此可知,获得的Clean Reads与参考基因组的匹配度良好,可进行后续分析。
表4 Clean Reads与参考基因组的比对结果
Table 4 Comparison results of Clean Reads and
reference genome
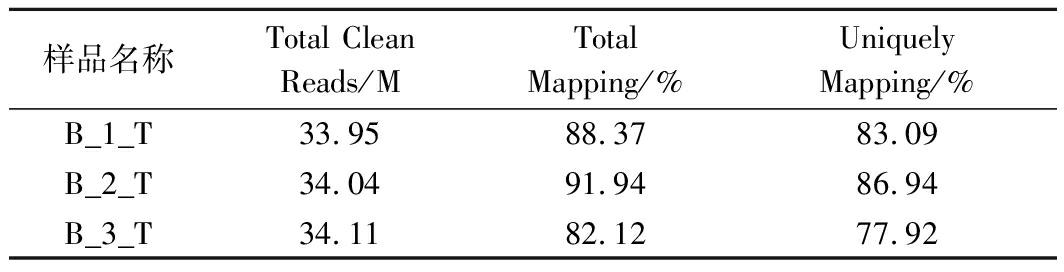
样品名称Total Clean Reads/MTotal Mapping/%Uniquely Mapping/%B_1_T33.9588.3783.09B_2_T34.0491.9486.94B_3_T34.1182.1277.92
2.3.3 测序数据在全基因组水平的转录分析
RNA-Seq数据共检测到5 998个基因,将检测到的基因分别从GO和KEGG功能分类2个角度做全基因组水平的转录分析,分析结果分别如图2和图3所示。由图2可知,RNA-Seq检测到的基因共分布在34个GO分类条目中,其中基因数量最多的GO分类条目为催化活性(catalytic activity),占基因总数的41.5%。由图3可知,RNA-Seq检测到的基因共分布在25个KEGG分类条目中,其中基因数量最多的KEGG分类条目为全局和总览图(global and overview maps),占基因总数的13.8%。

图2 基因GO分类分析
Fig.2 GO classification analysis of genes

图3 基因KEGG分类分析
Fig.3 KEGG classification analysis of genes
2.3.4 测序样品的相关性分析
在本研究中,样品B_1_T、B_2_T和B_3_T间的相关性是通过任意2个样品间相关性Person系数的平方(R2)进行分析。如图4所示,任意2个样品间的R2值均>0.95,因此3个样品间具有很好的相关性,实验数据可靠,满足后续分析的要求。

图4 样品相关性热图
Fig.4 Samples correlation heat map
2.4 高转录水平基因的挖掘
目前基于RNA-Seq数据挖掘强启动子的研究所采用的方法均仅根据FPKM或RPKM值选择高转录水平基因[15-17]。然而,仅基于基因FPKM或RPKM值而不考虑基因功能的高表达基因筛选方法,最终获得的强启动子通常难以显著提高报告蛋白表达水平。因此,在本研究中我们采用了一种高转录水平基因新筛选方法,即在基因筛选过程中同时考虑了基因FPKM 值及基因功能。本研究中采用的高转录水平基因新方法是基于以下3个原则:(1)根据GO及KEGG功能分类结果,筛选每个GO条目和KEGG条目中FPKM值最高的前3个基因组成初级靶基因文库;(2)按照图2和图3中GO和KEGG分类条目的排列顺序,去除初级靶基因文库中重复的基因,获得二级靶基因文库;(3)以胞外培养基中含量最丰富的细胞壁蛋白编码基因(hwp)的FPKM值为基准,去除二级靶基因文库中FPKM值低于该基准的基因。若某个GO或KEGG分类条目中所有基因的FPKM均低于该基准,则仅保留该条目中FPKM值最大的基因,最终获得可用于后续启动子筛选的终极靶基因文库。基于上述3个筛选原则,最终获得了包含19个基因的终极靶基因文库。为了方便后续研究,将筛选到的基因分别命名为Bsa、Bsb、Bsc、Bsd、Bse、Bsf、Bsg、Bsh、Bsi、Bsj、Bsk、Bsl、Bsm、Bsn、Bso、Bsp、Bsq、Bsr、Bss,对应的FPKM平均值如图5所示。

图5 基因的FPKM值
Fig.5 FPKM value of genes
2.5 强启动子的序列分析
基于上述获得的高转录水平基因在基因组中的操纵子结构预测结果,直接选取基因编码框起始密码子5′端500 bp内的序列为基因的启动子序列,然后对启动子的-35、-10区以及对应的σ因子和转录因子进行预测。如表5所示,筛选到的19个启动子中总共预测到了11种σ因子和32种转录因子。
表5 启动子序列特点
Table 5 Promoter sequence characteristics

基因类型启动子-35和-10区的预测(5′-3′)σ因子转录因子BsaOperonPATTGACTTCCTTTTGGGAAGTTGATACGATSigA/SigF/SigW/SigXCcpA/CtsR/RocR/XreBsbGenePBTTGAATAGAATAACAGTATTGATGTAAGATSigE/SigW/SigXAbrB/CcpA/DegU/GlnR/GltC/PerR/PurR/RocR/TnrA/XreBscOperonPCTTTCCATGAACAATTGTAAAATSigBCcpA/CodY/ComK/DegU/Fur/MntR/Spo0A/ZurBsdOperonPDTTAAAAGGAGAAGAAAAAGGGTAAGATSigB/SigHBkdR/CodY/ComABseOperonPETTTTCTTTCTTTTCCCGATTGTGATACCATSigD/SigLComA/CtsR/LevR/Mta/ResD/SinR/TnrABsfOperonPFTTGACGCTTTTTTACGGCCCATGTATAATSigA/ SigF/ SigHAbrB/FNR/Spo0A/SpoIIID/TnrABsgOperonPGTCGCAAGAAGTAAGCTAGGCTGATACTATNDCcpA/IolR/PucR/PurRBshGenePHCTGCCAGGCATTTAGAAAAGCATGGTTAGAAGSigHAbrB/SpoIIIDBsiGenePITTCTTTGGCGATTGCCTTCGGTTATCATSigA/SigE/SigFComK/DegU/HrcA/MntR/ResD/XreBsjGenePJTTGACAAATGCTCGTATTAGTTTATAATSigAAraR/BkdR/Fur/PerR/PurRBskOperonPKTTTACATTTGGCTGAATTATCGGAATATATTSigF/SigLBkdR/CtsR/MntR/ResDBslGenePLTTACTATATAAATTAATTAGAAGTATTATTATSigB/SigG/SigKFur/GlnR/Hpr/IolR/Mta/PerR/SinR/TnrABsmOperonPMATGTTAGCGTCCTTAACTAACATTTTTAAGATSigHCodY/CtsR/LexABsnGenePNTAGGAGTACAGCAATCCTGCTACCCTSigF/SigHCcpA/ComK/DegU/PucR/SpoIIID/ZurBsoGenePOTTGCTATTCTACCGTGTTCTCTGTAATATSigD/SigLAbrB/CtsR/SinRBspOperonPPTTTATATCGTTCAGAACATGTCCATACTAGAATSigF/SigG/SigL/SigXComA/CtsR/GltR/IolR/PucR/SinRBsqOperonPQTTGCTTCGCGAGATTTGCTTTGCTATAATNDBkdR/CcpA/MtaBsrOperonPRTTGGATGAAGCGGTTTATGAATCCGTGAAGATNDBkdR/CcpA/CodY/HrcA/LexA/TnrABssOperonPSTTGACAACACTCTTCCCTCCCTCAAAAATSigA/SigH/SigLAbrB/CcpA/CtsR/IolR/SpoIIID/TnrA
注:下划线分别表示-35和-10区;ND表示没有预测到σ因子
2.6 强启动子的筛选及验证
2.6.1 摇瓶发酵筛选
以α-淀粉酶为报告蛋白构建含有不同启动子的B.choshinensis重组菌筛选文库,以含有P2启动子的重组菌BCWPS为对照,以发酵上清液中的α-淀粉酶活性为筛选指标,进行强启动子筛选文库的摇瓶发酵筛选。如图6所示,不同启动子介导α-淀粉酶在B.choshinensis中的重组表达水平具有明显的差异,其中启动子PE和PF介导的重组菌BCWES和BCWFS的胞外α-淀粉酶活性均高于对照菌BCWPS。重组菌BCWES和BCWFS摇瓶发酵后胞外α-淀粉酶活性分别为7 880.1和4 375.4 U/mL,分别是对照菌BCWPS胞外α-淀粉酶活性(3 367.9 U/mL)的2.3和1.3倍。
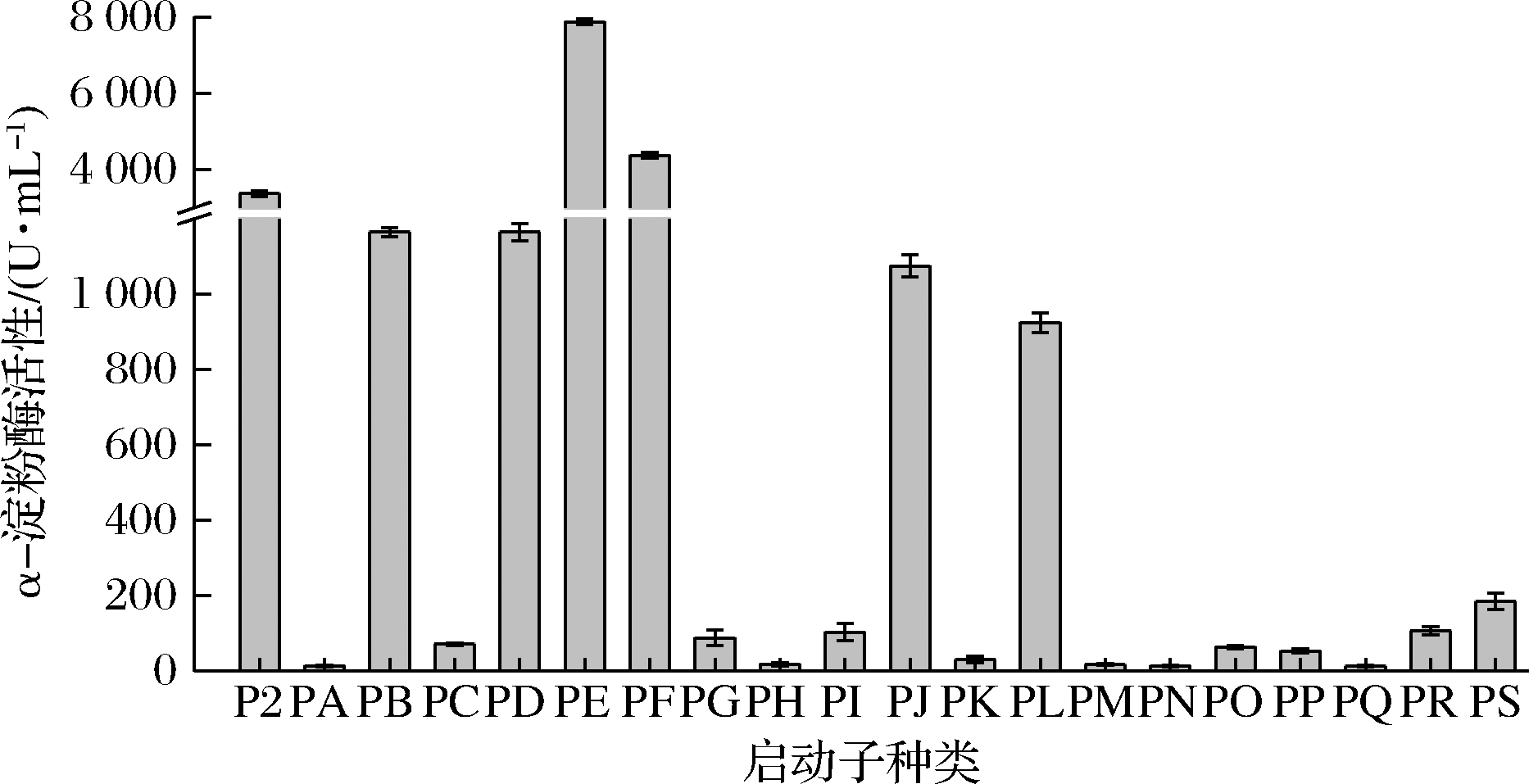
图6 含有不同启动子的短小芽孢杆菌重组菌的摇瓶培养
Fig.6 Shake-flask culture of B.choshinensis recombinant
strains with different promoters
2.6.2 启动子的转录活性分析
为了考察启动子PE、PF和P2的转录活性,在本研究中分别测定了重组菌BCWES、BCWFS和BCWPS 在摇瓶发酵24、36和48 h时α-淀粉酶基因的mRNA含量。如图7所示,启动子PE、PF和P2具有不同的转录活性。其中,启动子PE和PF的转录活性在重组菌摇瓶发酵过程中不断增强,启动子P2的转录活性在重组菌摇瓶发酵过程中却先降低后增加。另外,在24和36 h时启动子PE的转录活性均高于启动子PF和P2,而启动子P2在48 h的转录活性均高于启动子PE和PF。综上,启动子的转录活性与重组菌胞外α-淀粉酶活性无明显的线性关系,这表明除启动子的转录活性外,重组菌胞外α-淀粉酶活性还与其他因素有关,比如基因组中启动子所对应的目的基因功能及mRNA转录后修饰。因启动子PE介导的重组菌BCWES胞外α-淀粉酶活性最高,因此仅选择启动子PE进行后续的研究。

图7 不同启动子对mRNA表达量的影响
Fig.7 Effects of different promoters on mRNA expression
2.6.3 启动子PE的通用性分析
为了考察启动子PE的通用性,在本研究中另外选取蔗糖异构酶和角质酶作为报告蛋白,分别构建由启动子PE介导的蔗糖异构酶和角质酶B.choshinensis重组菌BCWESI和BCWECUT。将分别由P2启动子介导的蔗糖异构酶和角质酶重组菌BCWPSI和BCWPCUT与重组菌BCWESI和BCWECUT进行摇瓶发酵48 h后,重组菌BCWPSI和BCWESI胞外上清液中蔗糖异构酶活性分别为29.0和65.9 U/mL,重组菌BCWPCUT和BCWECUT胞外上清液中角质酶活性分别为21.0和26.5 U/mL。其中,重组菌BCWESI胞外蔗糖异构酶活性是重组菌BCWPSI的2.3倍,重组菌BCWECUT胞外角质酶活性是重组菌BCWPCUT的1.3倍。由此可知,启动子PE较P2所体现的正效果不局限于α-淀粉酶,对提高其他报告蛋白在B.choshinensis HPD31-SP3中的重组表达也具有一定的通用性。
2.6.4 重组菌BCWES的3 L罐发酵验证
为了进一步验证启动子PE的效果,我们将重组菌BCWES进行3 L罐发酵验证。如图8所示,当发酵72 h时,发酵液的菌体浓度及胞外上清液中α-淀粉酶活性均达到最大值,分别为23.2和5 626.2 U/mL。而由P2启动子介导的对照菌BCWPS在3L罐发酵过程中最高的菌体浓度及胞外α-淀粉酶活性分别为13.4和2 358.1 U/mL(图1)。重组菌BCWES最高的菌体浓度及胞外α-淀粉酶活性分别是重组菌BCWPS的1.7和2.4倍。因此,与重组菌BCWPS相比,尽管菌体浓度的增加有利于提高重组菌BCWES胞外α-淀粉酶活性,但PE作为一种较P2更高效的强启动子也是重组菌BCWES具有较高胞外α-淀粉酶活性的一个不可忽视的重要原因。综上,PE启动子与广泛应用于B.choshinensis中的强启动子P2相比是一种更高效的新内源性强启动子。

图8 重组菌BCWES的3 L罐发酵曲线
Fig.8 The 3 L fermenter fermentation curve of recombinant
strain BCWES
3 结论
本研究通过RNA-Seq技术获得了B.choshinensis的转录组数据,并基于基因FPKM值及基因功能,挖掘到了新内源性强启动子PE和PF。其中,以α-淀粉酶为报告蛋白,启动子PE介导的重组菌BCWES经摇瓶和3 L罐发酵后的胞外α-淀粉酶活性分别是P2启动子介导的对照菌BCWPS胞外α-淀粉酶活性的2.3和2.4倍。另外,以蔗糖异构酶和角质酶为新的报告蛋白证明了启动子PE具有一定的通用性。因此,本研究为B.choshinensis表达系统提供了新的强启动子,有助于进一步扩大B.choshinensis表达系统的应用范围。
[1] YAO D B, ZHANG K, WU J.Available strategies for improved expression of recombinant proteins in Brevibacillus expression system:A review[J].Critical Reviews in Biotechnology, 2020, 40(7):1 044-1 058.
[2] SIMONEN M, PALVA I.Protein secretion in Bacillus species[J].Microbiological Reviews,1993, 57(1):109-137.
[3] MIZUKAMI M,ONISHI H,HANAGATA H, et al.Efficient production of trastuzumab fab antibody fragments in Brevibacillus choshinensis expression system[J].Protein Expression and Purification, 2018, 150:109-118.
[4] POHL S, HARWOOD C R.Heterologous protein secretion by Bacillus species[J].Advances in Applied Microbiology, 2010, 73(10):1-25.
[5] 胡艳红, 龚雪梅, 丁柳柳, 等.利用短短芽孢杆菌进行酮还原酶CgKR2的高效表达与纯化[J].中国生物工程杂志, 2019, 39(8):59-65.
HU Y H, GONG X M, DING L L, et al.Highly efficient expression and purification of ketoreductase CgKR2 using Brevibacillus choshinensis SP3[J].China Biotechnology, 2019, 39(8):59-65.
[6] LAKSMI F A, IMAMURA H, TSURUMARU H, et al.Expression, folding, and activation of halophilic alkaline phosphatase in non-halophilic Brevibacillus choshinensis[J].The Protein Journal, 2020, 39(1):46-53.
[7] ICHIKAWA S, ICHIHARA M, ITO T, et al.Glucose production from cellulose through biological simultaneous enzyme production and saccharification using recombinant bacteria expressing the β-glucosidase gene[J].Journal of Bioscience and Bioengineering, 2019, 127(3):340-344.
[8] HANAGATA H, MIZUKAMI M, MIYAUCHI A.Efficient expression of antibody fragments with the Brevibacillus expression system[J].Antibodies, 2014, 3(2):242-252.
[9] LIAO Y L, HUANG L G, WANG B, et al.The global transcriptional landscape of Bacillus amyloliquefaciens XH7 and high-throughput screening of strong promoters based on RNA-seq data[J].Gene, 2015, 571(2):252-262.
[10] JIAO S, LI X, YU H M, et al.In situ enhancement of surfactin biosynthesis in Bacillus subtilis using novel artificial inducible promoters[J].Biotechnology and Bioengineering, 2017, 114(4):832-842.
[11] LI Y, DONG C H, HU M, et al. Identification of flower-specific promoters through comparative transcriptome analysis in Brassica napus[J]. International Journal of Molecular Sciences, 2019, 20(23): 5 949.
[12] CROUCHER N J, THOMSON N R.Studying bacterial transcriptomes using RNA-seq[J].Current Opinion in Microbiology, 2010, 13(5):619-624.
[13] MCCLEARY W R.Application of promoter swapping techniques to control expression of chromosomal genes[J].Applied Microbiology and Biotechnology, 2009, 84(4):641-648.
[14] YANG H L, LIAO Y L, WANG B, et al.Complete genome sequence of Bacillus amyloliquefaciens XH7, which exhibits production of purine nucleosides[J].Journal of Bacteriology, 2011, 193(19):5 593-5 594.
[15] LIU X, YANG H Y, ZHENG J W, et al.Identification of strong promoters based on the transcriptome of Bacillus licheniformis[J].Biotechnology Letters, 2017, 39(6):873-881.
[16] LIU X, WANG H, WANG B, et al.High-level extracellular protein expression in Bacillus subtilis by optimizing strong promoters based on the transcriptome of Bacillus subtilis and Bacillus megaterium[J].Protein Expression and Purification, 2018, 151:72-77.
[17] MENG F Q, ZHU X Y, NIE T, et al.Enhanced expression of pullulanase in Bacillus subtilis by new strong promoters mined from transcriptome data, both alone and in combination[J].Frontiers in Microbiology, 2018.DOI:10.3389/fmicb.2018.02635.
[18] 邹亮, 吴敬, 陈晟.重组蔗糖异构酶在短短芽孢杆菌中的表达及发酵优化[J].食品与生物技术学报, 2019, 38(1):22-28.
ZOU L, WU J, CHEN S.Expression and fermentation optimization of recombinant sucrose isomerase in Brevibacillus choshinensis[J].Journal of Food Science and Biotechnology, 2019, 38(1):22-28.
[19] LI Z, SU L Q, DUAN X G, et al.Efficient expression of maltohexaose-forming α-amylase from Bacillus stearothermophilus in Brevibacillus choshinensis SP3 and its use in maltose production[J].BioMed Research International, 2017.DOI:10.1155/2017/5479762.
[20] 李祝, 段绪果, 宿玲恰, 等.基于角质酶共表达策略的大肠杆菌胞外生产耐高温 α-淀粉酶及其发酵条件优化[J].食品与生物技术学报, 2019, 38 (11):9-17.
LI Z, DUAN X G, SU L Q, et al.Enhancing extracellular production of recombinant α-amylase in Escherichia coli through co-expression with cutinase and optimizing fermentation conditions[J].Journal of Food Science and Biotechnology, 2019, 38 (11):9-17.
[21] 刘军彤, 吴敬, 陈晟.分散泛菌蔗糖异构酶在大肠杆菌中的表达及发酵优化[J].生物工程学报, 2016, 32(8):1 070-1 080.
LIU J T, WU J, CHEN S.Expression and production optimization of sucrose isomerase from Pantoea dispersa in Escherichia coli[J].Chinese Journal of Biotechnology, 2016, 32(8):1 070-1 080.
[22] SU L Q, HONG R Y, KONG D M, et al.Enhanced activity towards polyacrylates and poly(vinyl acetate) by site-directed mutagenesis of Humicola insolens cutinase[J].International Journal of Biological Macromolecules, 2020, 162:1 752-1 759.