角蛋白是一种结构稳定、不溶于水的蛋白质,主要存在于羽毛、羊毛、毛发、蹄和指甲中[1]。全世界每年会产生大量的角蛋白废弃物,比如羽毛废弃物和劣质羊毛[2]。这些富含角蛋白的废弃物能够转化成为其他增值产品,如饲料和肥料[3]。目前物理法和化学法处理是提取的主要策略,这些策略常常涉及到高温或强酸强碱的使用,不仅会造成空气、土壤和水的污染,也会降低角蛋白分解产物的价值[4]。由真菌、放线菌等多种微生物产生的角蛋白酶能有效地水解角蛋白废弃物,是一种有效的替代方法[5]。角蛋白酶因其宽泛的底物专一性以及较强的水解能力,广泛地运用于饲料[6]、制革[7]、化妆品[8]、医药[9]等领域。
虽然角蛋白酶能够有效降解角蛋白废弃物,但是在具体应用场景中的耐受性和稳定性仍然受到挑战[10]。在前期的研究中我们发现,角蛋白酶KerZ1的最适反应温度为60 ℃,并在此温度下对羽毛角蛋白废物有着高效的水解作用。但是,KerZ1在60 ℃下的半衰期仅为18 min,这对于整个催化过程显然是不利的[11]。由于较高的反应温度不仅能够提高催化反应速率并且能够降低微生物污染的风险[12],因此,提高角蛋白酶的热稳定性将可以有效提升其在实际应用中的价值。
目前,蛋白质工程是提高生物催化剂热稳定性的有效手段,主要分为定向进化与理性设计两类[13]。定向进化是一种较为普遍的策略,因为其对蛋白质的相关信息需求较小,但是该策略需要消耗大量的时间和资源进行突变文库的筛选和数据分析[14]。而理性设计以蛋白质的结构信息为基础,通过计算机分析出可能会提高热稳定性的氨基酸相互作用,然后利用定点突变手段引入突变位点并验证效果[15]。与定向进化相比,理性设计可以有效减少突变库的大小,增加定向进化的效率,是一种可行性较高的策略。
蛋白质结构中的柔性环区域loop是提高其稳定性的潜在目标。Loop是一类多样化的二级结构,包括转角、无规则卷曲以及其他连接二级结构的氨基酸链[16]。大量的研究结果表明,loop在调节酶的催化作用[17],稳定性[18]以及底物特异性[19]方面发挥着重要作用。因此,预测出蛋白质结构中的柔性区域,刚化该柔性区域可以进一步提高蛋白质的稳定性。B-factor分析和分子动力学模拟是两种常用的研究蛋白质柔性区域的方法。B-factor是从X光衍射晶体结构中获得的,可以有效反映氨基酸残基的灵活性,但是它依赖于晶体结构的分辨率,不同分辨率的蛋白质数据会影响对氨基酸灵活性的预测[20]。分子动力学模拟主要是在原子水平上模拟蛋白质在某个时间段的运动轨迹,并可以准确提供蛋白质在其生理环境中的灵活性。与B-factor分析相比,分子动力学模拟需要消耗大量的计算资源,对计算机的运算能力也有较高的要求。目前,已经有各种方法改造柔性区域从而提高蛋白质的热稳定性,比如添加盐桥[21],引入二硫键或脯氨酸[22]以及迭代饱和突变[23]等。
基于同源性分析的方法已经被广泛应用于提高蛋白质的热稳定性。一种相对典型的方法就是将目标蛋白酶的序列或结构与嗜热蛋白酶进行比较,确定突变的关键位点。β-转角是最小的二级结构,由4个残基(位置i,i+1,i+2,i+3)组成,通常分布在柔性区域中[24]。对构成426条蛋白质链的7 153个β-转角残基的统计分析表明,在特定的β-转角上,某些氨基酸的出现频率会高于其他氨基酸[24],所以将不同位置上的氨基酸替换为频率更高的氨基酸就有可能提高目标蛋白质的热稳定性。基于这些假设,我们通过在柔性区域中的β-转角引入氨基酸突变以提高角蛋白酶的热稳定性,获得了热稳定提高的角蛋白酶,拓宽了其在工业生产中的应用。
1 材料与方法
1.1 实验材料
1.1.1 菌株和质粒
本研究所用大肠杆菌(Escherichia coli) JM109、枯草芽孢杆菌(Bacillus subtilis) WB600及质粒pP43 NMK均由本实验室保藏。
1.1.2 主要实验试剂
PrimeSTAR® Max DNA Polymerase,大连宝生物工程有限公司;限制性内切酶Dpn I,Thermo-Fisher Scientific公司;Bradford试剂盒,生工生物工程(上海)股份有限公司;HisTrapTM HP预装柱,GE Healthcare公司;其他试剂均为国产分析纯。
1.1.3 PCR引物设计
本研究中PCR扩增所用引物通过上海生工生物工程股份有限公司来合成,引物见表1。
1.1.4 培养基及培养条件
LB培养基(g/L):酵母粉 5、蛋白胨 10、NaCl 10,固体培养基则加20 g/L的琼脂粉,121 ℃灭菌15 min。根据实际需要添加相应的抗生素。
表1 本研究中所用引物
Table 1 Primers used for PCR in this study

引物名称引物DNA序列(5'-3')A128K-FCTTGGGGGAAAATCAGGCTCGACAGCGATGAAA128K-RGAGCCTGATTTTCCCCCAAGGCTCATATTGATAACATA128D-FCTTGGGGGAGACTCAGGCTCGACAGCGATGAAACAA128D-RGAGCCTGAGTCTCCCCCAAGGCTCATATTGATAACATA128P-FCTTGGGGGACCATCAGGCTCGACAGCGATGAAA128P-RGAGCCTGATGGTCCCCCAAGGCTCATATTGATAACATS129D-FGGGGGAGCAGACGGCTCGACAGCGATGAAACAGS129D-RGTCGAGCCGTCTGCTCCCCCAAGGCTCATATTGAS129H-FGGGGGAGCACACGGCTCGACAGCGATGAAACAGS129H-RGTCGAGCCGTGTGCTCCCCCAAGGCTCATATTGAS129 N-FGGGGGAGCAAACGGCTCGACAGCGATGAAACAGS129 N-RGTCGAGCCGTTTGCTCCCCCAAGGCTCATATTGAS129V-FGGGGGAGCAGTCGGCTCGACAGCGATGAAACAGS129V-RGTCGAGCCGACTGCTCCCCCAAGGCTCATATTGAS129Y-FGGGGGAGCATACGGCTCGACAGCGATGAAACAGS129Y-RGTCGAGCCGTATGCTCCCCCAAGGCTCATATG130P-FGGAGCATCACCATCGACAGCGATGAAACAGGCG130P-RGCTGTCGATGGTGATGCTCCCCCAAGGCTCAG130V-FGGAGCATCAGTCTCGACAGCGATGAAACAGGCG130V-RGCTGTCGAGACTGATGCTCCCCCAAGGCTCATAS158Y-FAGCGGATCTTACGGAAACACGAATACAATTGGCTATC-CTGS158Y-RGTGTTTCCGTAAGATCCGCTGTTCCCTGCN160D-FTCTTCAGGAGACACGAATACAATTGGCTATCCTGCGN160D-RGTATTCGTGTCTCCTGAAGATCCGCTGTTCCCN160S-FTCTTCAGGATCTACGAATACAATTGGCTATCCTGCGN160S-RGTATTCGTAGATCCTGAAGATCCGCTGTTCCC
注:加下划线的碱基为替换的密码子
发酵培养基(g/L):蔗糖 20、酵母粉 10、蛋白胨 20、Na2HPO4·12H2O 6、KH2PO4 3、MgSO4·7H2O 0.3,121 ℃灭菌15 min。枯草芽孢杆菌发酵使用。
大肠杆菌、枯草芽孢杆菌培养温度37 ℃,旋转式摇床220 r/min。
1.2 突变体酶的构建
利用Quick ChangeTM定点突变的方法构建突变体角蛋白酶[25]。PCR反应产物经过Dpn I酶切处理后,转化E.coli JM109感受态细胞,转化产物涂布于LB平板上(100 μg/mL氨苄青霉素),于37 ℃过夜培养。从LB平板上挑取阳性转化子,提取质粒之后送上海生工进行测序验证。将测序正确的重组质粒转化表达载体B.subtilis WB600,转化产物涂布于LB平板上(50 μg/mL卡那霉素),于37 ℃过夜培养。得到的转化子即为目标角蛋白酶重组菌。
1.3 突变体酶的表达及纯化
挑取B.subtilis WB600转化子转接至2 mL LB培养基中(50 μg/mL卡那霉素),于37 ℃、220 r/min摇床过夜培养。过夜培养的种子液按体积分数2%转接至25 mL发酵培养基中,于37 ℃、220 r/min摇床培养24 h。将发酵获得的菌液于4 ℃、7 000 r/min离心10 min,得到粗酶液。利用镍离子亲和色谱柱纯化蛋白质。用20 mmol/L pH 7.4 Tris-HCl缓冲液平衡预装柱,将处理后的粗酶液上样,然后用含有50 mmol/L咪唑的20 mmol/L pH 7.4的Tris-HCl缓冲液进行梯度洗脱得到纯酶。利用SDS-PAGE和Bradford试剂盒检测蛋白的纯度和浓度。
1.4 角蛋白酶酶活力的测定
取50 μL适当稀释的酶液,加入150 μL 50 mmol/L的Gly-NaOH溶液(pH 10)作为缓冲液和100 μL 25 g/L的水溶性角蛋白作为底物,混匀后于60 ℃下反应20 min;加入200 μL 0.5 mol/L的三氯乙酸终止反应,室温12 000 r/min离心2 min。取上清液200 μL,加入1 mL 50 g/L的Na2CO3溶液和200 μL的福林酚试剂,混匀后于50 ℃下显色10 min,使用分光光度计测定波长660 nm下的吸光度;空白对照是在加入底物之前先加入反应终止剂三氯乙酸,其余操作同上。
酶活力单位定义:一个活力单位(U)为吸光度在波长660 nm下每分钟升高0.001[11]。
1.5 酶的热稳定性研究
半衰期(t1/2)测定:将纯化后的KerZ1及其突变体酶置于60 ℃水浴中,每隔10 min取样并测定残余酶活力。以水浴处理前酶活力为100%,以后每次取样酶活力为相对酶活力。作以残余酶活力的ln值为纵坐标,时间t为横坐标的散点图,通过对数据点的线性拟合获得角蛋白酶失活速率常数k,然后带入公式:t1/2=ln2/k计算半衰期[26]。
半失活温度(T50)测定:将纯化后的KerZ1及其突变体酶置于不同的温度下(30~80 ℃)水浴处理20 min,测定其残余酶活力。以各自最高酶活力为100%,其余温度下酶活力为相对酶活力。以相对酶活力为纵坐标,温度为横坐标作图,计算半失活温度。
1.6 动力学参数研究
将角蛋白底物按照体积分数稀释(0%、0.2%、0.4%、0.6%、0.8%、1.0%、1.2%),参照酶活力测定方法测定不同底物浓度下的反应初速,计算米氏常数(Km)和最大反应速率(Vmax),并进一步计算催化常数kcat以及催化效率kcat/Km。
1.7 分子动力学模拟
角蛋白酶KerZ1与模板greglin以及subtilisin的复合蛋白体KerA(PDB 4 gi3,1.75 Å resolution)的成熟酶氨基酸序列具有99%的同源性,通过在线模拟服务器SWISS MODEL(https://swissmodel.expasy.org/interactive),以PDB数据库中KerA的晶体结构为模板,同源模拟角蛋白酶KerZ1及其突变体酶的晶体结构。使用AMBER16进行分子动力学模拟,力场选定ff14SB[27],以水分子盒子包裹同源建模模型,并设定盒子边界与蛋白质表面的最小距离为12 Å,在溶剂中添加150 mmol/L的NaCl以平衡系统的正负电荷,整个分子动力学模拟包括水平衡、侧链平衡、能量最小化、50 ps的0~300 K的升温过程和20 ns的动力学模拟。利用AMBER16自带的分析工具计算每个氨基酸残基的均方根浮动(root mean square fluctuation,RMSF)。
2 结果与分析
2.1 角蛋白酶柔性环区域的改造
利用PyMOL软件对KerZ1(PDB 4 gi3)中的二级结构进行标注,一共确定了15个loop环区域。通过将KerZ1的PDB文件提交至在线服务器HotSpot Wizard 3.0(http://loschmidt.chemi.muni.cz/hotspot-wizard)中计算每个氨基酸残基的B-factor值[28]。B-factor主要反映原子构象状态的一种“模糊度”,其值越高,模糊度越大,对应结构构象越不稳定。每个loop环区域的B-factor通过环路内每个氨基酸残基的平均B-factor计算得到,KerZ1的15个loop环区域及其B-factor值计算结果见表2。
表2 基于B-factor值的Loop环灵活性等级表
Table 2 Flexibility rank of loops based on B-factors
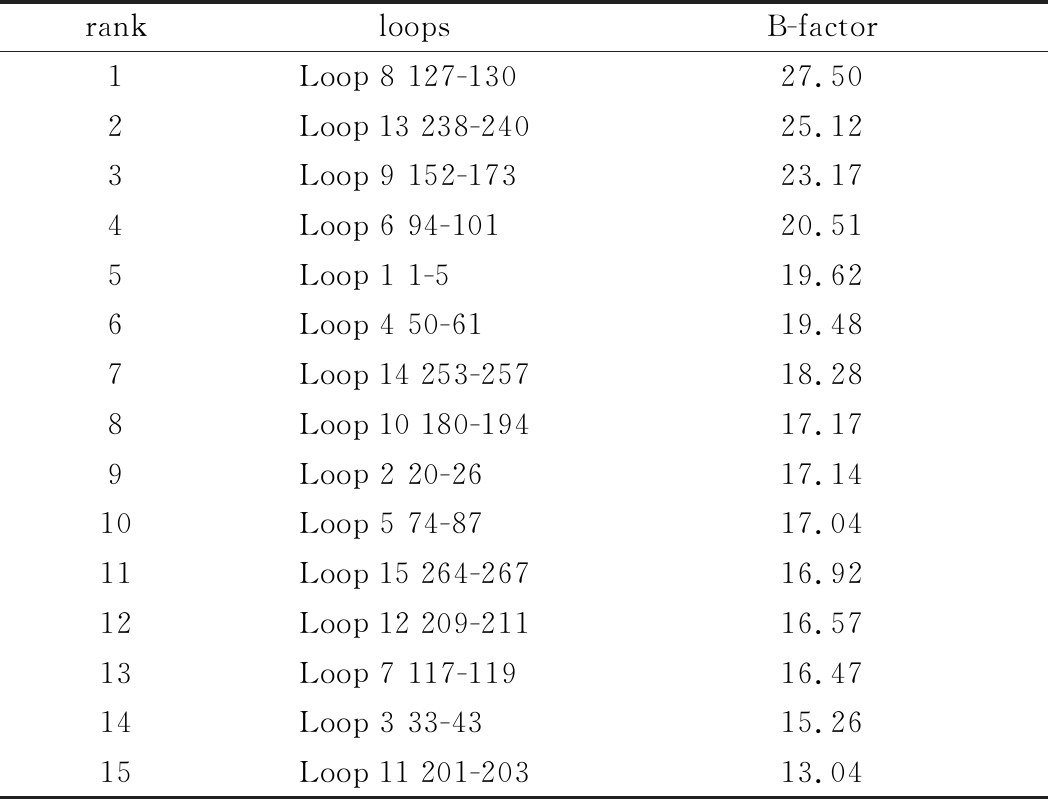
rankloopsB-factor1Loop 8 127-13027.502Loop 13 238-24025.123Loop 9 152-17323.174Loop 6 94-10120.515Loop 1 1-519.626Loop 4 50-6119.487Loop 14 253-25718.288Loop 10 180-19417.179Loop 2 20-2617.1410Loop 5 74-8717.0411Loop 15 264-26716.9212Loop 12 209-21116.5713Loop 7 117-11916.4714Loop 3 33-4315.2615Loop 11 201-20313.04
各loop环区域的B-factor值比较结果如图1所示。Loop8 127~130和Loop13 238~240表现出最高的灵活性。Loop9 152~173由22个氨基酸残基组成,是角蛋白酶结构中最长的环区域,也表现出较高的灵活性。
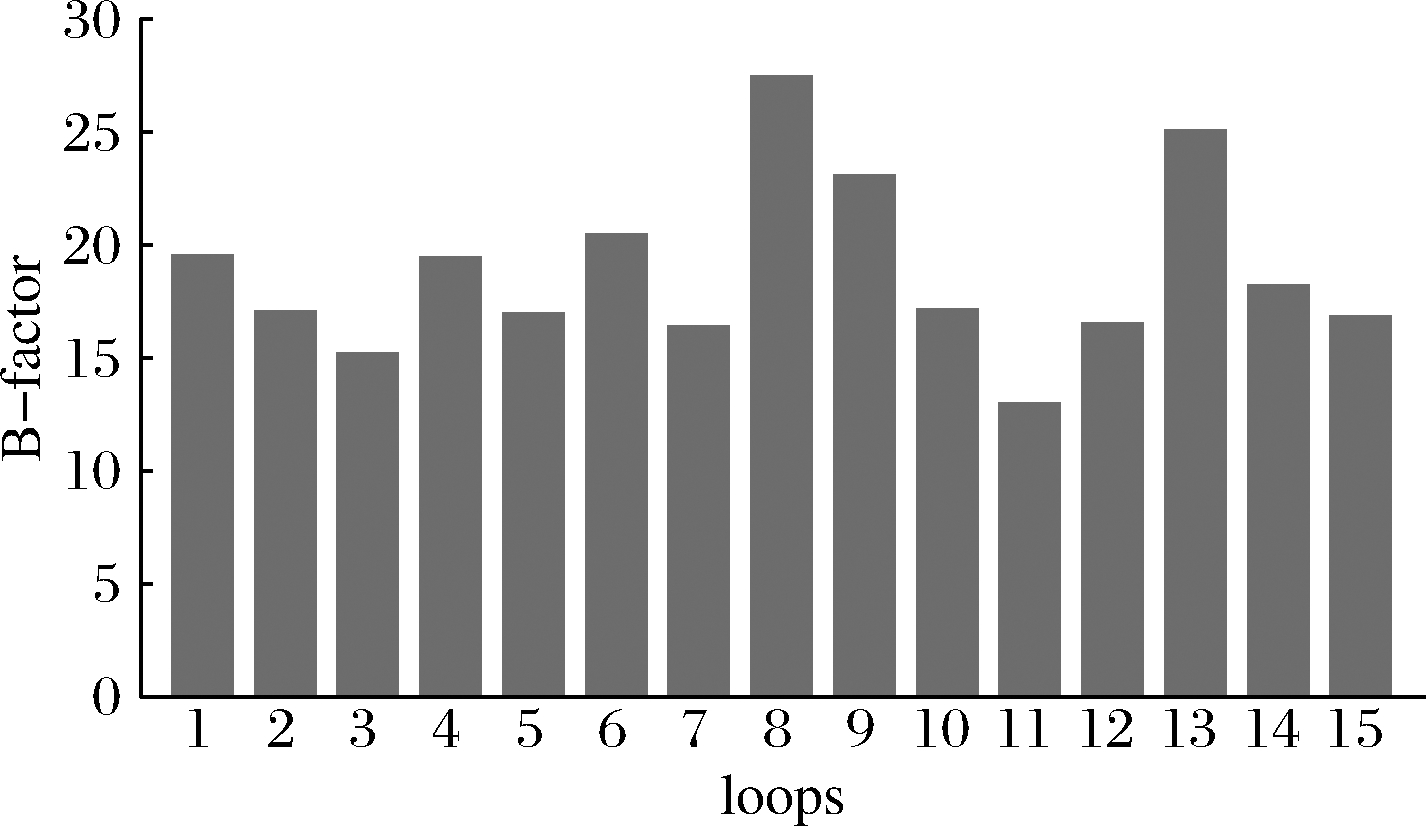
图1 角蛋白酶KerZ1中loop环区域的B-factor值比较
Fig.1 Comparison of B-factor of loops in KerZ1
分子动力学模拟也被用于分析KerZ1结构的灵活性。在300 K下,对KerZ1的结构进行了20 ns的分子动力学模拟,并计算每个氨基酸残基的RMSF值,结果如图2所示。RMSF值反映了构象的灵活度,其值越高,对应的结构柔性越大。结果表明,RMSF值虽然从不同的角度反映构象的灵活度,但是总体的结果与B-factor分析的结果一致,说明两者在预测蛋白质柔性区域方面具有相似性。尽管相关性很强,但是结果中也存在一定差异。例如,根据在300 K下分子动力学模拟计算的RMSF值,KerZ1最灵活的3个环区域是loop8、loop9以及loop5,而B-factor分析下最灵活的3个区域是loop8、loop13以及loop9。因此,结合B-factor分析与分子动力学模拟,最终确定KerZ1的2个柔性环区域作为改造对象,分别是loop8 127~130和loop9 152~173。
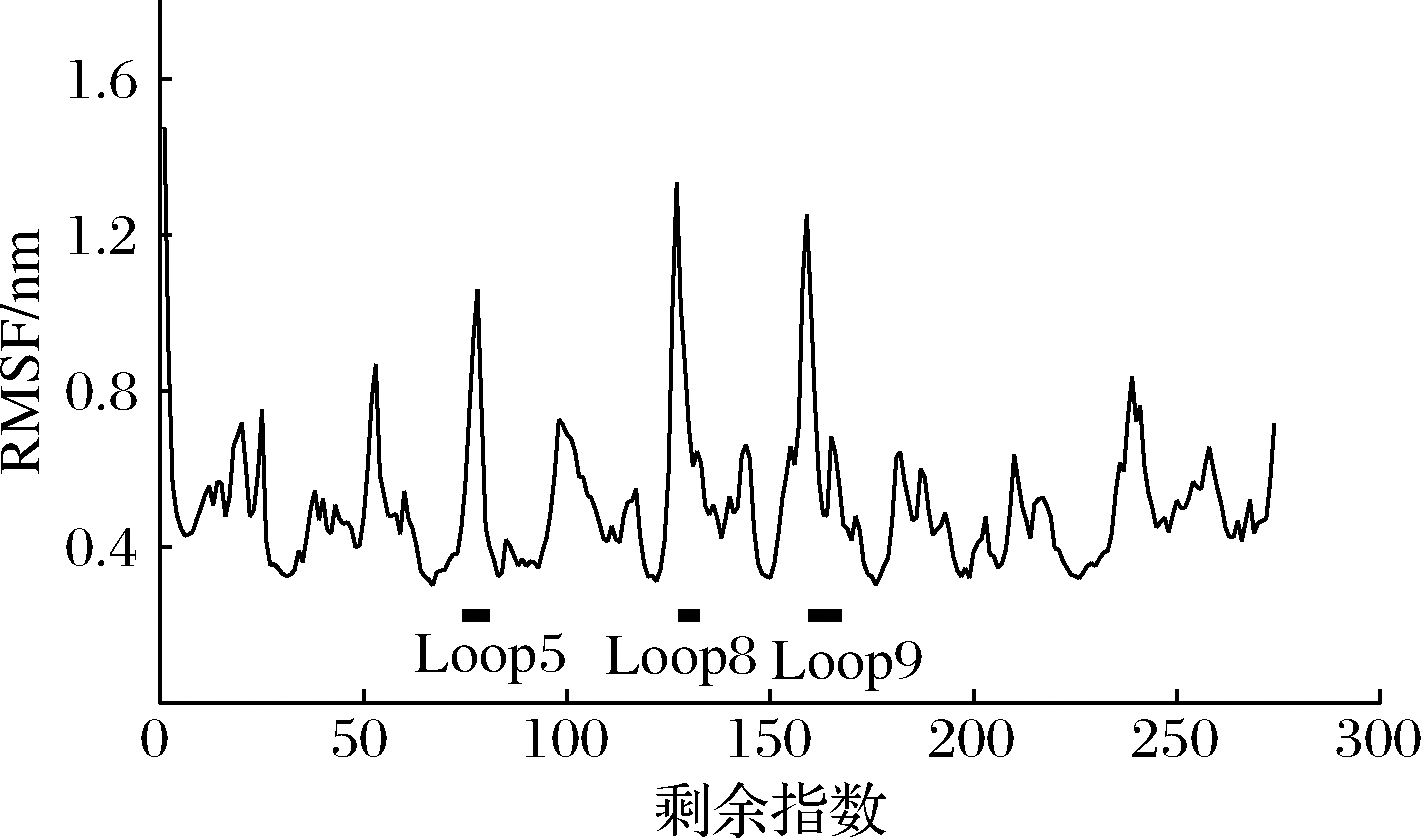
图2 在300 K下角蛋白酶KerZ1氨基酸残基的RMSF值
Fig.2 Normalized RMSF values of KerZ1 amino acid residues at 300 K
同源性分析最典型的例子是将目标蛋白酶的序列或结构与嗜热蛋白酶进行比对,确定突变的关键位点。YU等[29]基于同源性分析对大肠杆菌转酮酶柔性区域的β-转角进行了改造,提高了热稳定性。通过对β-转角残基的统计分析,不同类型β-转角上的部分氨基酸频率会高于其它氨基酸。例如,在Ⅱ型β-转角的i位置上,半胱氨酸与脯氨酸的出现频率最高,如果Ⅱ型β-转角的i位置的氨基酸不是半胱氨酸或脯氨酸,将其突变为其中一种就有可能提高目标蛋白质的热稳定性。因此,基于这些假设,我们从PDBsum数据库中获得了角蛋白酶3个柔性环区域内的β-转角信息,设计引入了13个单点突变体,设计结果如表3所示。
表3 基于β-转角氨基酸位置偏好的突变体设计
Table 3 Design of variants based on β-turn amino acid positional preference.

柔性环区域β-转角转角类型引入的氨基酸突变loop8 127~130127-GASG-130ⅧA128D,A128K,A128PS129D,S129H,S129 N,S129V,S129YG130P,G130Vloop9 152~173158-SGNT-161II’S158YN160D,N160S
2.2 角蛋白酶及其突变体酶的表达与纯化
利用Quick ChangeTM将表3确定的氨基酸残基引入KerZ1,构建的重组质粒转化B.subtilis WB600中进行突变体酶的表达。为了初步确定突变体酶的热稳定性,测定了KerZ1及突变体酶粗酶液在60 ℃水浴处理1 h条件下的残余酶活力,结果如图3所示,突变体酶A128D表现出较好的热稳定性,其残余酶活力高于KerZ1,而其他突变体酶的残余酶活力均低于KerZ1。通过镍亲和色谱柱进一步纯化KerZ1与A128D,并进行SDS-PAGE分析,KerZ1与A128D的分子质量约为30 kDa(图4)。
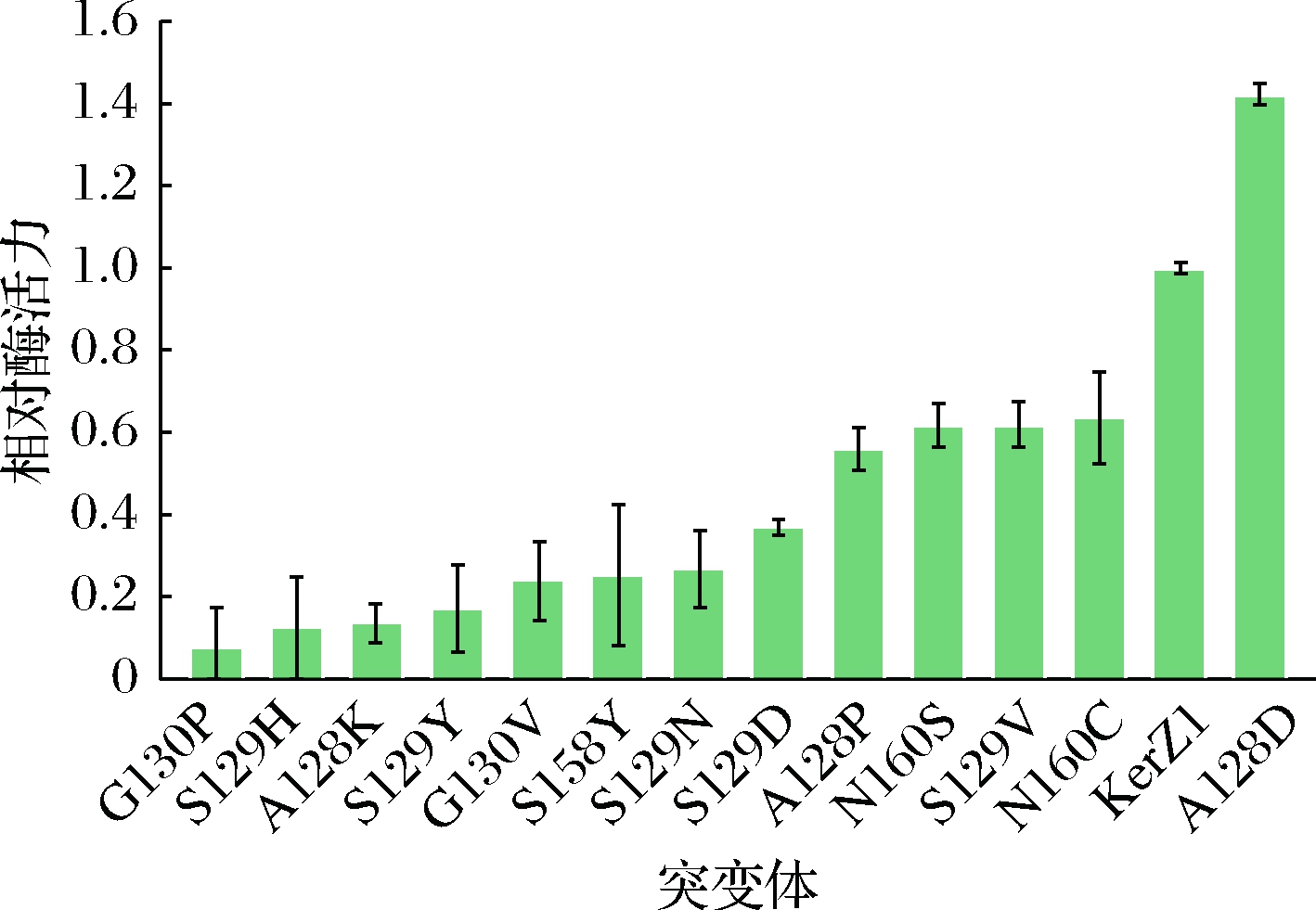
图3 KerZ1及其突变体酶在60 ℃下处理1 h的残余酶活力
Fig.3 Residual enzyme activity of KerZ1 and its mutants at 60 ℃ for 1 h

M-蛋白marker;泳道1-KerZ1;泳道2-A128D
图4 KerZ1和A128D的SDS-PAGE分析
Fig.4 SDS-PAGE analysis of KerZ1 and A128D
2.3 角蛋白酶热稳定性测定
将纯化后的KerZ1和A128D置于不同温度下(30~80 ℃)水浴处理20 min研究其温度适应性。如图5-a所示,KerZ1和A128D在50~80 ℃明显下降。KerZ1在60和80 ℃水浴处理时分别损失了48%和100%的酶活性,A128D表现出比KerZ1更好的热稳定性,A128D的热失活温度(T50)为67.4 ℃,与KerZ1的热失活温度61.6 ℃相比有所提高。考虑到60 ℃是KerZ1的最佳酶活性温度,且在该温度下酶活力下降明显,因此选择60 ℃条件进一步验证其热稳定性的提高。
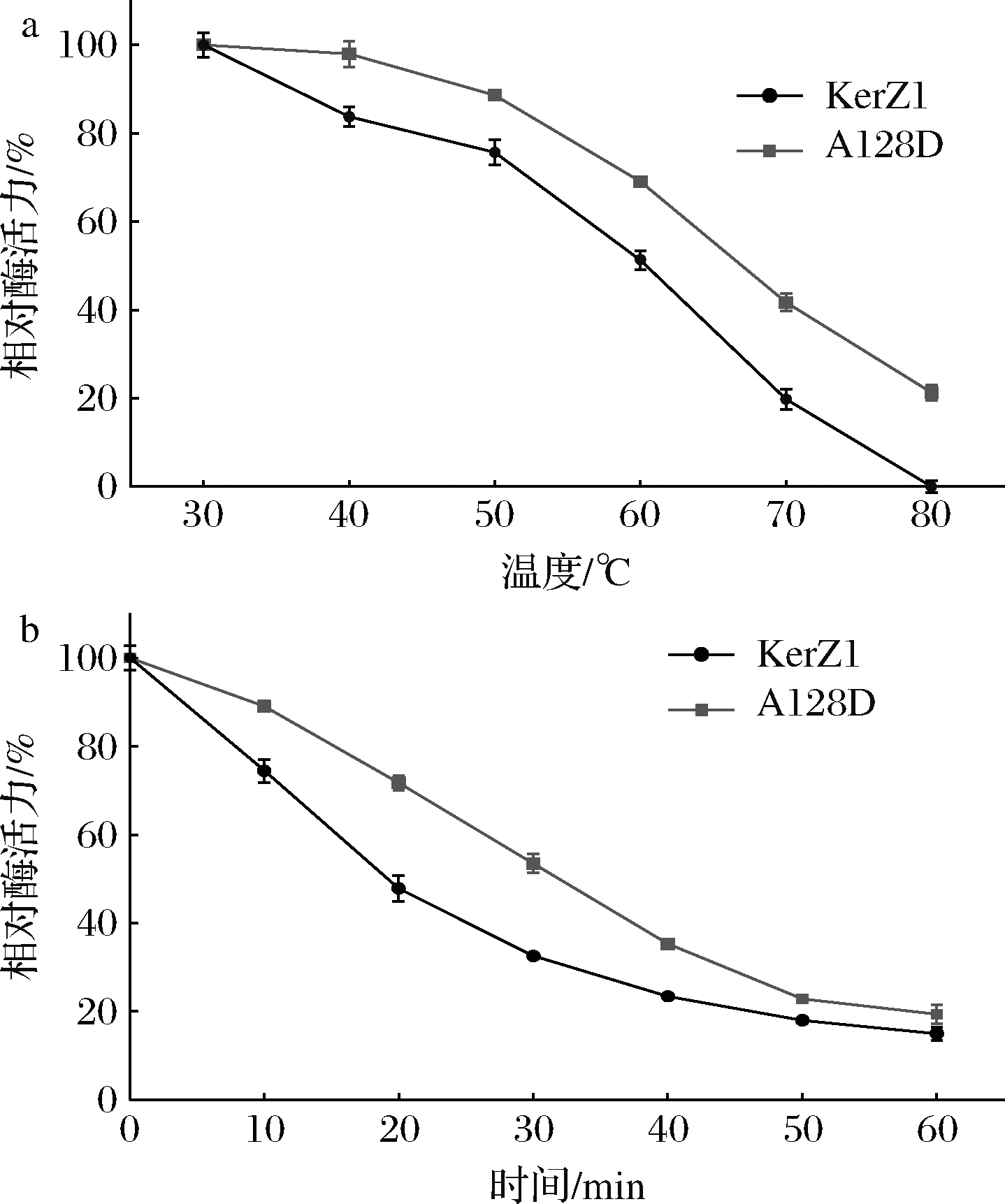
a-不同温度下的热稳定性;b-60 ℃下的半衰期
图5 KerZ1和A128D的热稳定性及半衰期测定
Fig.5 Determination of thermostability and half-life of KerZ1 and A128D
将KerZ1和A128D置于60 ℃水浴中处理1 h,每隔10 min取样测其残余酶活力并计算60 ℃条件下的半衰期(t1/2),结果如图5-b所示,A128D的半衰期(t1/2)为30.44 min,是KerZ1半衰期(18.12 min)的1.68倍。综上所述,在KerZ1的β-转角中引入的突变D128有效提高了角蛋白酶的热稳定性。
2.4 角蛋白酶的动力学参数
以不同浓度的角蛋白为底物测量KerZ1和A128D在60 ℃下的酶活性和动力学参数,结果如表4所示,A128D的Km值低于KerZ1,表明A128D对底物的亲和力有所提升。而在催化效率方面,A128D的kcat/Km与KerZ1相比提高了6.29%,表明催化效率有所提高。因此,A128D在热稳定性提升的同时在动力学方面的表现也优于KerZ1。
表4 KerZ1和A128D的酶动力学参数
Table 4 Kinetic parameters of KerZ1 and A128D

酶Km/(mol·L-1)kcat/(s-1)kcat/Km(L·mol-1·s-1)比酶活力/(U·mg-1)KerZ10.63±0.0531.26±0.5449.62193 192.57±21.88A128D0.61±0.0832.17±0.3252.74204 725.42±28.94
2.5 突变体酶的稳定性机制分析
分子动力学模拟计算得到的RMSF值揭示了其热稳定性提高的机制。如图6所示,引入的突变D128与A128相比有更低的RMSF值,整个loop8区域的氨基酸残基的RMSF都明显降低,这说明A128D的loop8的结构变得更加稳定,从而提高了KerZ1的热稳定性。通过突变体A128D的三维结构进一步分析了KerZ1热稳定性提高的机制。

图6 KerZ1和A128D残基灵活性比较
Fig.6 The residual flexibility of KerZ1 and A128D
如图7-a所示,A128所在的loop8位于蛋白质结构表面,但是其周围的氨基酸G126、G127、S129、G130以及S131都是亲水性氨基酸。因此,作为一种疏水性氨基酸,A128可能破坏了loop8结构的稳定性,从而导致KerZ1稳定性的下降。通过降低蛋白质表面的疏水性提高其热稳定性已经在黄素氧化还原蛋白的研究中得到了证实[30]。在本研究中,当A128突变为D后,作为一种亲水性氨基酸,D128与周围亲水性氨基酸提高了蛋白质表面的亲水性,这可能使loop8变得更加稳定(图7-b)。因此,A128D表面亲水性的增加可能是其热稳定性提高的主要原因之一。
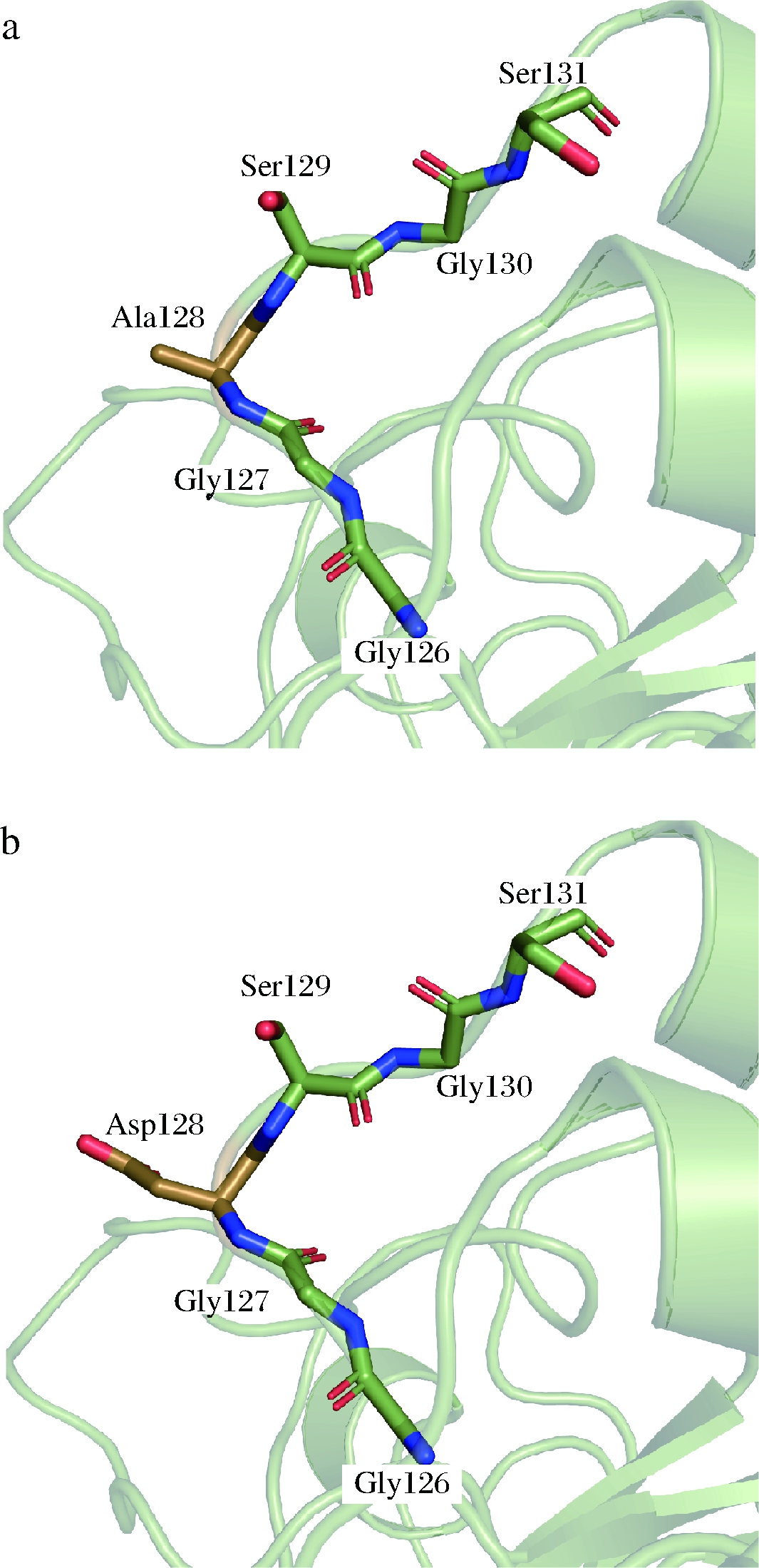
图7 KerZ1 (a)和A128D (b)三维结构以及突变残基位置的示意图
Fig.7 Schematic diagram of the three-dimensional structure and mutant residue positions of KerZ1 (a) and A128D (b)
3 结论
角蛋白酶KerZ1在其最适反应温度60 ℃下的热稳定性较差,这限制其实际应用的潜力。通过B-factor分析和分子动力学模拟对角蛋白酶KerZ1中的柔性环区域进行了不稳定性预测,并基于β-转角的统计学分析结果在柔性环区域的β-转角中引入了氨基酸突变,获得一个热稳定性显著提高的突变体酶A128D。进一步,基于分子动力学模拟分析了突变体热稳定性提高的机制,结果表明,与KerZ1相比,A128D在柔性环loop8这一区域中表现出了更低的RMSF值。通过三维结构分析突变位点残基以及周围氨基酸残基的分布,发现引入的D128为亲水性更好的氨基酸,与周围的亲水性氨基酸一起增加了蛋白质表面的亲水性,从而提高了热稳定性。总之,本研究首次对角蛋白酶中的β-转角进行了改造,有效地提高了角蛋白酶的热稳定性,也为其他工业用酶的热稳定性改造提供了思路。
[1] SHAVANDI A,SILVA T H,BEKHIT A A,et al.Keratin:dissolution,extraction and biomedical application[J].Biomaterials Science,2017,5(9):1 699-1 735.
[2] HASSAN M A,ABOL-FOTOUH D,OMER A M,et al.Comprehensive insights into microbial keratinases and their implication in various biotechnological and industrial sectors:A review[J].International Journal of Biological Macromolecules,2020,154:567-583.
[3] GUPTA R,RAMNANI P.Microbial keratinases and their prospective applications:An overview[J].Applied Microbiology and Biolechnology,2006,70(1):21-33.
[4] MARTINEZ J P D,CAI G Q,NACHTSCHATT M,et al.Challenges and opportunities in identifying and characterising keratinases for value-added peptide production[J].Catalysts,2020,10(2):184.
[5] BRANDELLI A.Bacterial keratinases:useful enzymes for bioprocessing agroindustrial wastes and beyond[J].Food and Bioprocess Technology,2008,1(2):105-116.
[6] PENG Z,MAO X,ZHANG J,et al.Effective biodegradation of chicken feather waste by co-cultivation of keratinase producing strains[J].Microbial Cell Factories,2019,18(1):84.
[7] CAO S,LI D,MA X,et al.A novel unhairing enzyme produced by heterologous expression of keratinase gene (kerT) in Bacillus subtilis[J].World Journal of Microbiology and Biotechnology,2019,35(8):122.
[8] SANGHVI G,PATEL H,VAISHNAV D,et al.A novel alkaline keratinase from Bacillus subtilis DP1 with potential utility in cosmetic formulation[J].International Journal of Biological Macromolecules,2016,87:256-262.
[9] MARTINEZ Y N,CAVELLO I,HOURS R,et al.Immobilized keratinase and enrofloxacin loaded on pectin PVA cryogel patches for antimicrobial treatment[J].Bioresource Technology,2013,145:280-4.
[10] 唐雪明, 王正祥,诸葛健.具有工业应用价值的高热稳定性极端酶[J].食品与发酵工业,2001,27(5):65-70.
TANG X M,WANG Z X,ZHUGE J.High termostable extremozymes for industrial application[J].Food and Fermentation Industries,2001,27(5):65-70.
[11] PENG Z,MAO X,ZHANG J,et al.Biotransformation of keratin waste to amino acids and active peptides based on cell-free catalysis[J].Biotechnology for Biofuels,2020,13:61
[12] EIJSINK V G,BJØRK A,GÅSEIDNES S,et al.Rational engineering of enzyme stability[J].Journal of Biotechnology,2004,113(1-3):105-120.
[13] LIU Q,XUN G H,FENG Y.The state-of-the-art strategies of protein engineering for enzyme stabilization[J].Biotechnology Advances,2019,37(4):530-537.
[14] YANG H Q,LIU L,LI J H,et al.Rational design to improve protein thermostability:Recent advances and prospects[J].ChemBioEng Reviews,2015,2(2):87-94.
[15] ACEVEDO-ROCHA C G,HOEBENREICH S,REETZ M T.Iterative saturation mutagenesis:A powerful approach to engineer proteins by systematically simulating Darwinian evolution[J].Methods Mol Biol,2014,1179:103-128.
[16] NESTL B M,HAUER B.Engineering of flexible loops in enzymes[J].ACS Catalysis,2014,4(9):3 201-3 211.
[17] MALABANAN M M,AMYES T L,RICHARD J P.A role for flexible loops in enzyme catalysis[J].Current Opinion in Structural Biology,2010,20(6):702-710.
[18] TANG H,SHI K,SHI C,et al.Enhancing subtilisin thermostability through a modified normalized B-factor analysis and loop-grafting strategy[J].Journal of Biological Chemistry,2019,294(48):18 398-18 407.
[19] GUNASEKARAN K,MA B Y,NUSSINOV R.Triggering loops and enzyme function:Identification of loops that trigger and modulate movements[J].Journal of Molecular Biology,2003,332(1):143-159.
[20] YUAN Z,BAILEY T L,TEASDALE R D.Prediction of protein B-factor profiles[J].Proteins,2005,58(4):905-12.
[21] FEI B,XU H,CAO Y,et al.A multi-factors rational design strategy for enhancing the thermostability of Escherichia coli AppA phytase[J].Journal of Industrial Microbiology & Biotechnology,2013,40(5):457-464.
[22] 王睿, 喻晓蔚,徐岩.理性设计二硫键提高华根霉脂肪酶热稳定性[J].微生物学通报,2018,45(11):2 311-2 319.
WANG R,YU X W,XU Y.Rationale design of disulfide bond in Rhizopus chinensis lipase to improve thermostability[J].Microbiology China,2018,45(11):2 311-2 319.
[23] BOEHLEIN S K,SHAW J R,STEWART J D,et al.Enhancing the heat stability and kinetic parameters of the maize endosperm ADP-glucose pyrophosphorylase using iterative saturation mutagenesis[J].Archives of Biochemistry and Biophysics,2015,568:28-37.
[24] GURUPRASAD K,RAJKUMAR S.Beta-and gamma-turns in proteins revisited:A new set of amino acid turn-type dependent positional preferences and potentials[J].Journal of Biosciences,2000,25(2):143-56.
[25] ZHENG L,BAUMANN U,REYMOND J L.An efficient one-step site-directed and site-saturation mutagenesis protocol[J].Nucleic Acids Research,2004,32(14):e115.
[26] O’FAGAIN C.Enzyme stabilization-recent experimental progress[J].Enzyme and Microbial Technology,2003,33(2-3):137-149.
[27] MAIER J A,MARTINEZ C,KASAVAJHALA K,et al.ff14SB:Improving the accuracy of protein side chain and backbone parameters from ff99SB[J].Journal of Chemical Theory and Computation,2015,11(8):3 696-3 713.
[28] SUMBALOVA L,STOURAC J,MARTINEK T,et al.HotSpot Wizard 3.0:web server for automated design of mutations and smart libraries based on sequence input information[J].Nucleic Acids Research,2018,46(W1):W356-W362.
[29] YU H,YAN Y,ZHANG C,et al.Two strategies to engineer flexible loops for improved enzyme thermostability[J].Scientific Reports,2017,7:41212.
[30] AYUSO-TEJEDOR S,ABIAN O,SANCHO J.Underexposed polar residues and protein stabilization[J].Protein Engineering,Design and Selection,2011,24(1-2):171-177.