低聚果糖是指果糖经β-2,1糖苷键连接而成的,聚合度为2~9的功能性低聚糖[1]。作为一种强效益生元,它可以促进肠道内益生菌的增殖[2],降低血脂、预防高胆固醇[3],促进矿物质的吸收[4],有助减肥和预防糖尿病,并可以增强机体免疫力[5]。因此,低聚果糖常被添加于牛奶制品、调味品、婴幼儿食品及饲料中,在食品、保健品等领域应用广泛。
低聚果糖在洋葱、蒜、甜菜和一些谷物中广泛存在,但含量并不高,且提取难度较大。目前市场上售卖的低聚果糖主要是利用生物酶技术——果糖基转移酶(EC 2.4.1.9)或内切菊粉酶进行生产。果糖基转移酶在反应过程中形成的副产物葡萄糖不仅增加了下游纯化工艺的成本,同时也抑制了果糖基转移酶的活性,阻碍蔗糖的进一步转化,产物含量低(<55%)且生产成本高[6]。内切菊粉酶(endo-inulinase,EC 3.2.1.7)能随机催化水解菊粉内部的β-2,1糖苷键,产生三糖、四糖、五糖等低聚果糖[7]。使用内切菊粉酶生产低聚果糖操作简单,副产物少,产率高达90%,具有很高的商业应用价值[8]。
目前,我国内切菊粉酶的催化活力难以达到工业化生产的要求,且相关研究主要集中于野生菌的筛选、基因的克隆表达及酶学性质研究,关于内切菊粉酶催化机制及三维结构改造的研究则相对较少[7]。随着生物信息学和分子生物学的高速发展,如何提高内切菊粉酶的催化活力也有了新的研究思路。如同源建模可以简单准确地根据已知的蛋白质结构模型预测目标蛋白的三维结构[9],分子对接则可以帮助理解蛋白与底物的相互作用方式,指导人们理性选取氨基酸改造靶点,有效地对蛋白质进行改造[10]。
2012年,内切菊粉酶晶体结构首次被解析[11],该蛋白来自Aspergillus ficuum,至今仍是唯一被解析的内切菊粉酶晶体。为了进一步提高内切菊粉酶的催化活力,本研究以该晶体作为模板,对来源于Lipomyces starkeyi NRRL Y-11557的内切菊粉酶INU3B[12]进行同源建模,并以蔗果五糖(GF4,G代表葡萄糖,F代表果糖)作为底物进行分子对接,确定INU3B结合口袋处的关键氨基酸。在此基础上,将关键氨基酸突变为丙氨酸来扩大底物结合口袋,以期提高酶催化活力。
1 材料与方法
1.1 材料与试剂
重组质粒pET22b(+)-inu3B为本实验室前期构建,并克隆至大肠杆菌E.coli BL21(DE3)中保存[12]。重组大肠杆菌的培养采用Luria-Bertani (LB)培养基:NaCl 5 g/L,蛋白胨 10 g/L,酵母提取物 5 g/L。
E.coli感受态细胞制备试剂盒,大连TaKaRa生物工程有限公司;质粒提取剂盒、Cycle Pure试剂盒等,OMEGA Bio-tek公司;氨苄青霉素、异丙基硫代半乳糖苷(IPTG),生工生物工程(上海)股份有限公司;SDS-PAGE凝胶快速配制试剂盒、Bradford蛋白浓度测定试剂盒,上海碧云天生物技术有限公司;用作底物的菊粉(I2255),Sigma公司;KH2PO4、K2HPO4·3H2O、NaCl、甘油、胰蛋白胨等其他试剂,国药集团有限公司。
1.2 仪器与设备
T100 Thermal Cycle PCR仪,美国Bio-Rad公司;GelDoc-ItTM 凝胶成像系统,美国UVP公司;Nano Drop 2000微量分光光度计,美国Thermo Fisher Scientific公司;HYL-C3组合式摇床,太仓市强乐实验设备有限公司;Sonics Vibra-Celltm超声破碎仪,美国Sonics公司;MR-96T型智能酶标仪,骋克仪器(上海)有限公司;XMTD-204数显式恒温水浴,上海博讯医疗设备厂;Powerpac Basic蛋白电泳仪,美国Bio-Rad公司;冷冻离心机,德国Eppendorf公司; KTA蛋白纯化系统,美国GE 医疗公司。
KTA蛋白纯化系统,美国GE 医疗公司。
1.3 实验方法
1.3.1 内切菊粉酶的同源建模
采用SWISS-MODEL在线程序 (https://swissmodel.expasy.org/)建模,相似性>30%的蛋白质为同源模型的候选参考蛋白质。利用评估程序ProCheck (https://servicesn.mbi.ucla.edu/PROCHECK/)对模型可靠性进行评估。用软件Pymol对目标蛋白的三维模型进行结构分析。
1.3.2 GF4与内切菊粉酶的分子对接
受体结构:用AutoDockTools 1.5.6对同源建模得到的蛋白质结构进行去水、加氢等处理,保存蛋白原有电荷,采用刚性对接,生成pdbqt文件。
配体结构:通过Chem3D绘制并生成小分子配体蔗果五糖GF4的三维结构。用AutoDock Tools 1.5.6 处理后,生成pdbqt文件。
选定对接区域:通过结合口袋对接,确保选定的蛋白区域完全包裹催化三联体。
分子对接:调动相应的代码,采用AutoDock Vina[13]进行对接。
1.3.3 定点突变
以质粒pET22b(+)-inu3B为模板,加入相应的突变引物(表1),用高保真聚合酶Phanta Max Super-Fidelity DNA Polymerase进行PCR扩增。从E.coli中提取的质粒被甲基化,用Dpn I对PCR产物进行处理排除模板的干扰后通过cycle pure纯化回收,并转入E.coli BL21(DE3)感受态细胞中。选取阳性转化子提取质粒送测序公司测序。
表1 突变引物序列
Table 1 List of primers used for site-directed mutagenesis

突变位点引物序列(5'-3')Met46M46A-FCCAGAAGAGAACTGGgcGAATGAGCCCAATGGACaM46A-RGGGCTCATTCgcCCAGTTCTCTTCTGGCACGPHE104F104A-FGATGGGATATGGGCTgccACAGGAACCTCTTACTTTGF104A-RGAGGTTCCTGTggcAGCCCATATCCCATCTGCGThr105T105A-FGGATATGGGCTTTCgCAGGAACCTCTTACTTTGT105A-RGAGGTTCCTGcGAAAGCCCATATCCCATCTGCGVal239V239A-FCAGGTTGGGAGgcgCCAGACTTTTTCGAACTTCAGATTGAAGV239A-RGTTCGAAAAAGTCTGGcgcCTCCCAACCTGAAATTCCACCThr260T260A-FGTGTTGATTGTGgCTCCTGCCGCTGGATCT260A-RGCGGCAGGAGcCACAATCAACACCCATTTCSER326S326A-FCTTGCTGCAGTTATGAACgcTTATGGTGTTAACCCCS326A-RGGGGTTAACACCATAAgcGTTCATAACTGCAGCAAGTYR327Y327A-FGCTGCAGTTATGAACAGTgcTGGTGTTAACCCCCY327A-RGGGTTAACACCAgcACTGTTCATAACTGCAGCAAG
注:a,小写字母为进行突变的碱基
1.3.4 重组菌的诱导表达
将突变后的重组E.coli接种至50 mL含氨苄青霉素的LB液体培养基中,37 ℃、180 r/min培养12 h后以2%的接种量转移至50 mL新鲜的TB培养基中。37 ℃、180 r/min培养约2 h (OD600约为0.6~1.0)后加入诱导剂IPTG至终浓度为0.1 mmol/L,转移至20 ℃、180 r/min诱导20 h。
1.3.5 重组菌粗酶液的提取
取诱导培养后的发酵液30 mL,3 000×g 离心5 min后收集菌液,加入15 mL 50 mmol/L磷酸盐缓冲液(pH 6.5)重悬清洗,5 000×g离心5 min,弃上清液后再加入15 mL相同的缓冲液。冰浴条件下超声波细胞破碎,300 W,超声3 s、间隔7 s,120次。破壁后,4 ℃,5 000×g离心10 min,上清液即为粗酶液,用于下一步纯化。
1.3.6 重组内切菊粉酶的纯化
重组蛋白C端带有6×His标签,选择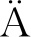 KTA蛋白质纯化系统,采用His Trap HP亲和层析柱纯化重组蛋白。用磷酸盐缓冲液(500 mmol/L NaCl,20 mmol/L NaH2PO4,pH 7.4)分别配制浓度为20和500 mmol/L的咪唑溶液,上样后通过20~500 mmol/L线性梯度的咪唑溶液洗脱蛋白质。目的蛋白在350 mmol/L咪唑时被洗脱并经过PD-10脱盐柱进行脱盐。经SDS-PAGE鉴定后得到的蛋白样品置于4 ℃保存。
KTA蛋白质纯化系统,采用His Trap HP亲和层析柱纯化重组蛋白。用磷酸盐缓冲液(500 mmol/L NaCl,20 mmol/L NaH2PO4,pH 7.4)分别配制浓度为20和500 mmol/L的咪唑溶液,上样后通过20~500 mmol/L线性梯度的咪唑溶液洗脱蛋白质。目的蛋白在350 mmol/L咪唑时被洗脱并经过PD-10脱盐柱进行脱盐。经SDS-PAGE鉴定后得到的蛋白样品置于4 ℃保存。
1.3.7 酶活力测定
采用DNS法测定内切菊粉酶酶活力[14]。取200 μL纯化后的酶液与800 μL 20 g/L的菊粉混合,70 ℃水浴反应10 min 后加入1 mL DNS试剂停止反应。沸水浴10 min,取出冷却至室温,取1 mL反应液加蒸馏水稀释一定的倍数并在540 nm波长下测量吸光度。根据果糖标准曲线计算生成的还原糖的量。通过Bradford方法测量蛋白质浓度,牛血清白蛋白用作标准蛋白质[15]。
内切菊粉酶活力的定义为:在pH为5.5、温度为70 ℃的条件下,每分钟水解菊粉生成1 μmol还原糖所需的酶量(mg)为1个酶活力单位(U)。
2 结果与讨论
2.1 内切菊粉酶INU3B的同源建模
将来自L.starkeyi NRRL Y-11557的内切菊粉酶INU3B的氨基酸序列上传到Swiss-Model 蛋白模拟在线服务器。INU3B与来自A.ficuum的内切菊粉酶(3 sc7.1.A)同源性达68.1%,以它为模板进行同源建模,图1为INU3B获得的三维结构。内切菊粉酶属于糖苷水解酶第32家族,它的三维结构具有该家族的共同特点,N端由5个叶片形成的封闭催化活性口袋,每个叶片包含4个反向平行的β-折叠片,形成经典的“W”拓扑结构;C端由2个反向平行的β折叠片组成,每个β折叠片包括6个β折叠股,组装成类似三明治的结构[16]。其中,Glu48(WMNDEPNG)是亲核试剂,Asp181(RDP)是过渡态稳定剂,Glu238(EVP)是酸/碱催化剂,这3个氨基酸构成了内切菊粉酶的催化三联体,负责底物的结合和催化[16],以蓝色棍棒模型进行标识。催化三联体对蛋白活性起着至关重要的作用,对这3个位点的突变常常会导致酶的完全失活[17]。
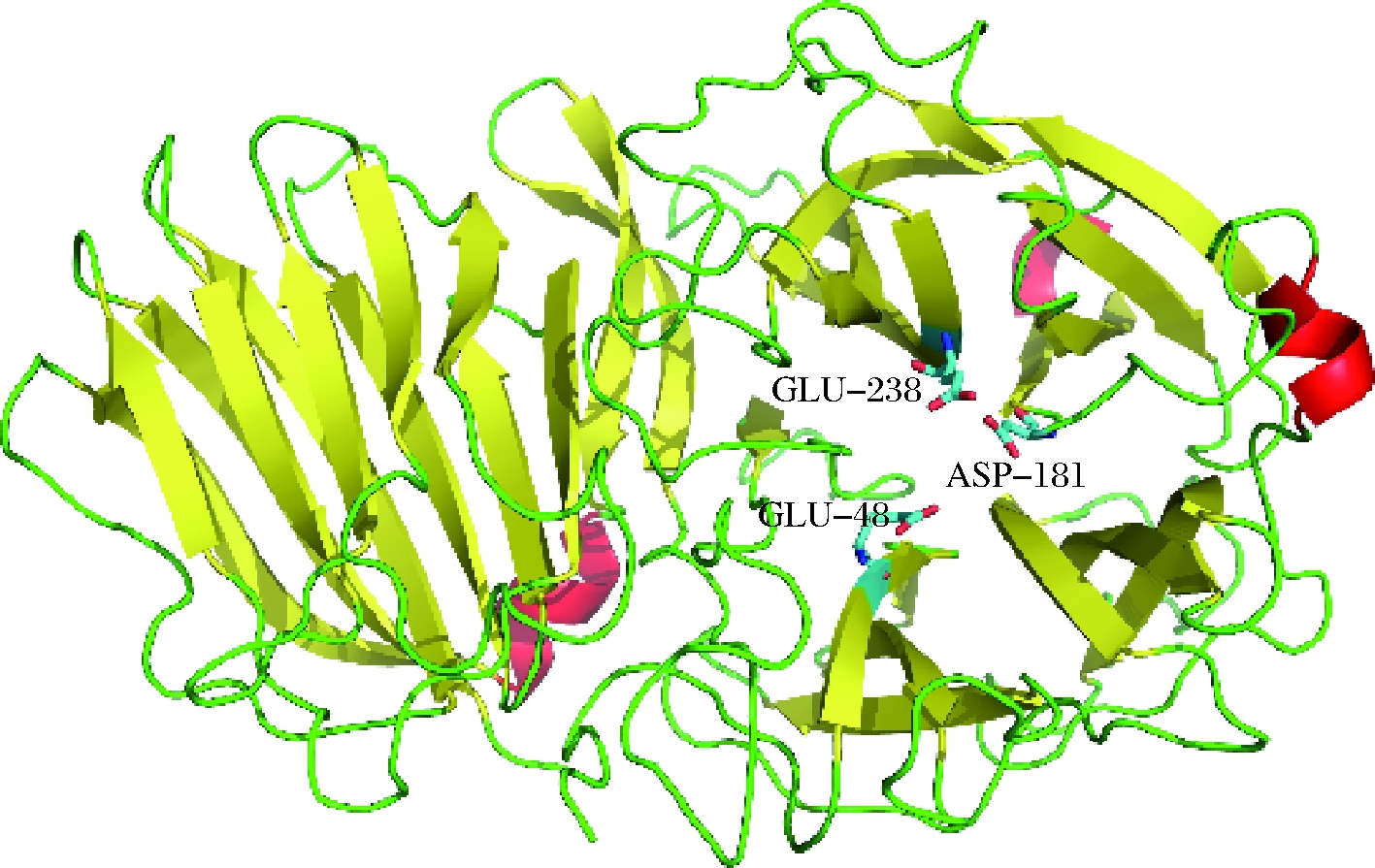
图1 INU3B的同源建模结构
Fig.1 Homology model for 3D structure of INU3B
利用评估程序ProCheck对INU3B蛋白模型的可靠性进行评估,结果如图2-a所示:88.1%位于最适区域,10.2%位于其他允许区域,1.5%位于一般允许区域,1个残基(0.2%)位于不允许区域,说明该模型基本合理。对INU3B进行表面电势分析,如图2-b所示,INU3B活性口袋的表面具有较强电负性,有利于底物稳定结合在酶的活性空腔中。
a-Ramachandran图;b-表面电势分析
图2 INU3B的Ramachandran图和表面电势分析
Fig.2 Ramachandran plot and potential diagram of INU3B
2.2 INU3B与GF4的分子对接
以INU3B催化三联体所在区域为对接盒子,通过Autodock Vina对INU3B和底物分子GF4进行对接,选出最合理的构象。结果如图3-a所示,GF4深入到INU3B结合口袋内部,与口袋良好匹配。利用Pymol进行分析,INU3B底物结合口袋中距GF4分子4 Å以内的氨基酸残基共25个。其中,N47、Y133、Q140、E238、N270、N325能与GF4形成氢键,如图3-b所示,这些氨基酸可能在酶与底物的结合和催化过程中起着重要作用。
2.3 突变位点的选择
丙氨酸(Ala)是一种不带电荷的疏水性氨基酸,侧链为甲基,具有较小的空间位阻,对蛋白质的结构影响较小[18]。内切菊粉酶的底物为菊粉,菊粉是由果糖构成的线性直链多糖,末端带有1个葡萄糖,聚合度高、分子量大[19-20],因此猜测将相关位点突变为丙氨酸可以增加底物结合口袋,从而有效地提高内切菊粉酶的活力[21]。
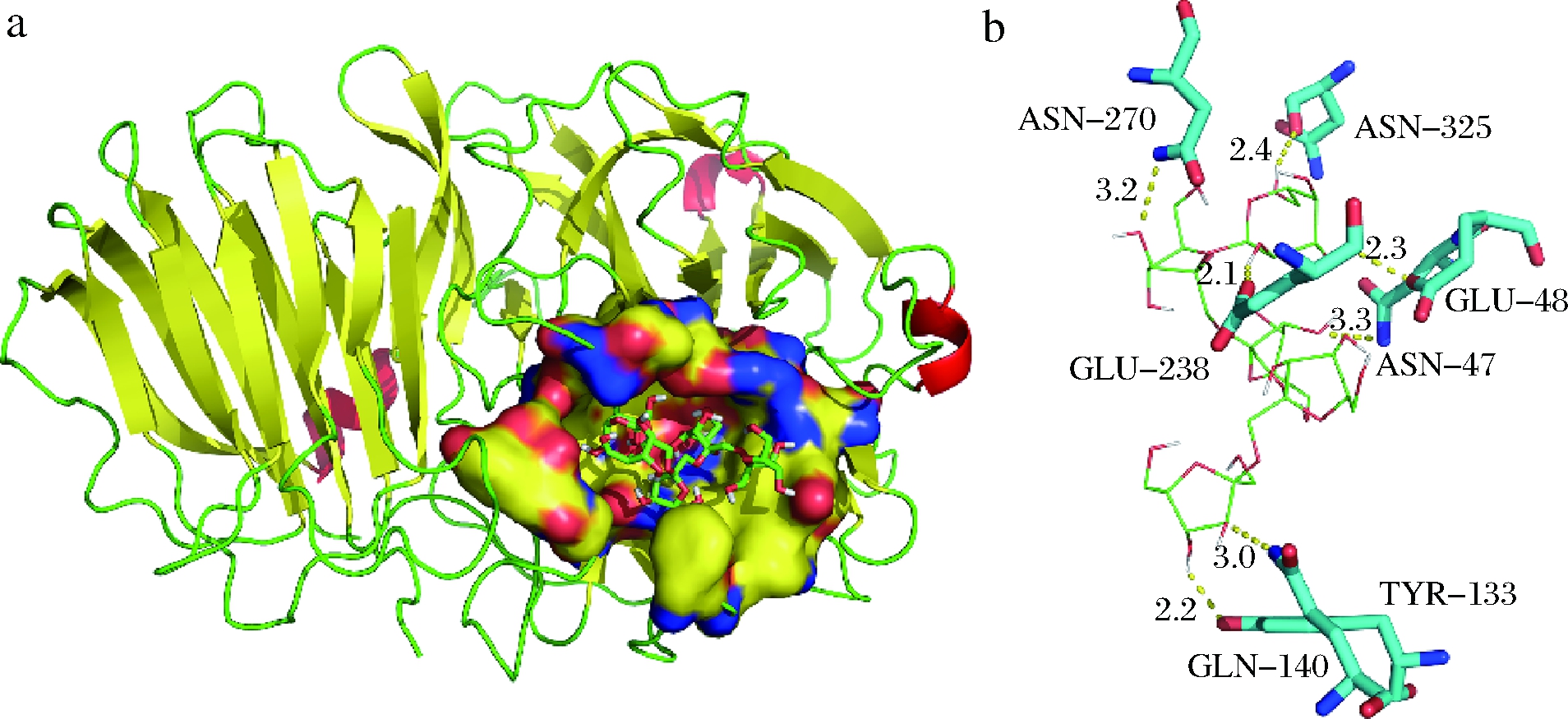
a-INU3B-GF4复合体的对接模型;b-INU3B与GF4的氢键作用
图3 GF4与INU3B的相互作用
Fig.3 The interaction between INU3B and GF4
通过序列比对(图4),对结合口袋处的25个氨基酸进行保守性分析。结果如表2所示,大多数氨基酸都具有一定的保守性。在进化过程中绝对保守的氨基酸一般有着重要作用,因此不突变。而保守性极低的氨基酸,在催化过程中发挥的作用相对较小,因此不突变。对能与底物分子形成氢键的6个氨基酸的位点的突变往往会造成酶活力的降低,因此不突变。由于G73、A262、G328原来的侧链就比较小,因此也不进行丙氨酸突变。因此,最终选择了7个氨基酸位点(M46、F104、T105、V239、T260、S326、Y327)分别进行丙氨酸突变。

图4 内切菊粉酶的氨基酸序列比对
Fig.4 A snapshot of alignment of endo-inulinase amino acid sequences
注:组成结合口袋的氨基酸用*标出
表2 底物结合口袋氨基酸的保守性分析
Table 2 Conservative analysis of amino acids in substrate-binding pocket

组别保守性/%氨基酸位点绝对保守100N47a E48 Q64 R180 D181E238 G304相对保守50~100M46 W72 G73 F104 T105Y133 Q140 V239T260 A262N270 N325 S326 Y327 G328低保守<50F71 L75 W102
注:a-下划线氨基酸能够与底物分子形成氢键
2.4 突变体的构建与分析
以pET22b(+)-inu3B为模板进行全质粒扩增,PCR产物经Dpn I处理后转化至感受态中,挑选阳性转化子测序后获得相应的突变体。正确点突变的重组E.coli诱导表达后,进行纯化、脱盐,并进行SDS-PAGE验证。从图5可以看出所有重组蛋白均为单一条带,可用于进一步分析。
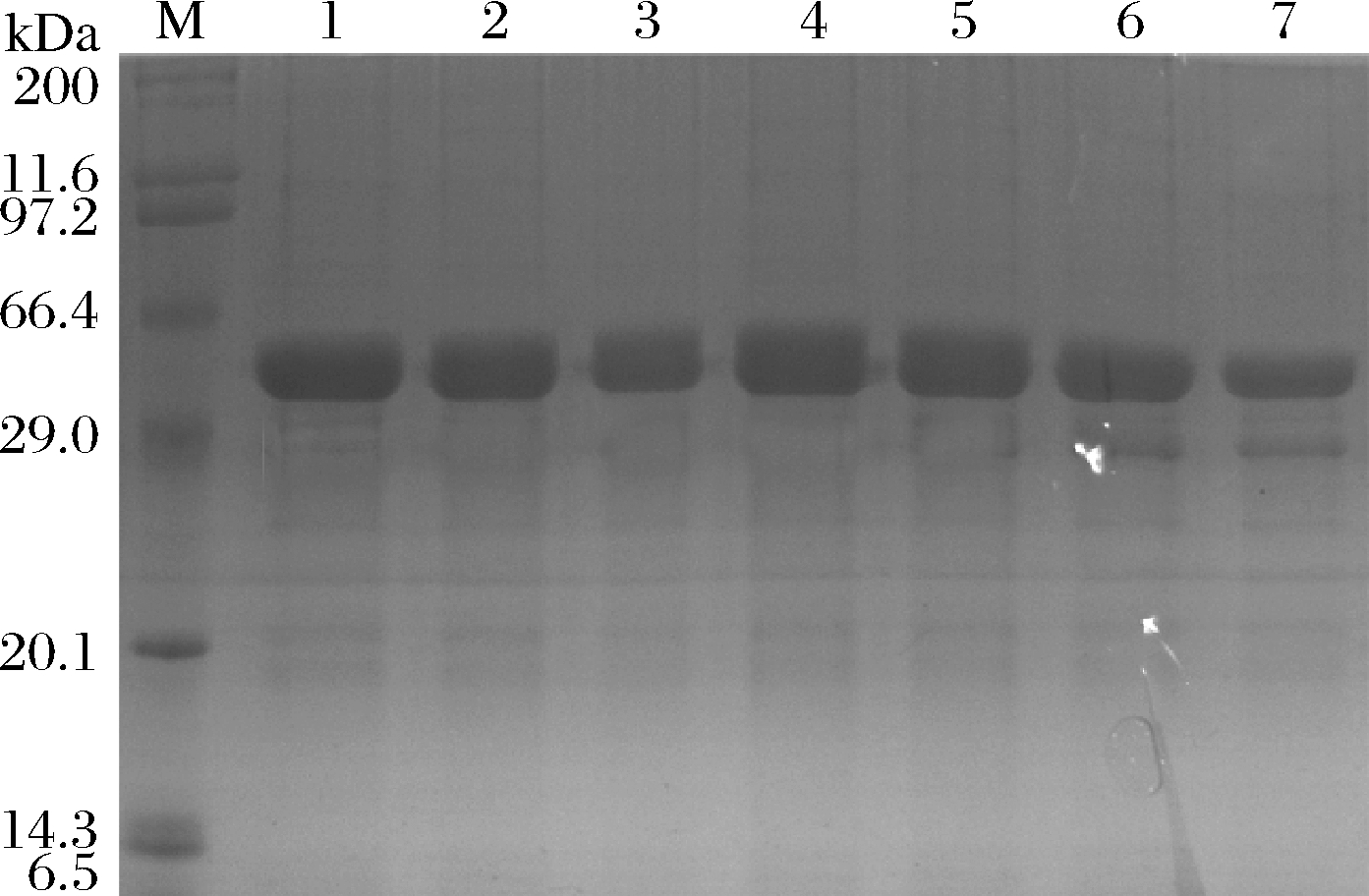
M-蛋白质marker;1-7:INU3B、M46A、F104A、T105A、V239A、T260A、Y327A
图5 INU3B与突变酶的蛋白电泳分析
Fig.5 SDS-PAGE analysis of INU3B and mutants
对纯化后的突变蛋白进行酶活力测定,实验结果如图6所示。大部分突变体的催化活性大幅下降,突变体F104A、T260A、S326、Y327A几乎检测不到酶活性,突变体T105A、V239A对底物的活力远小于INU3B。7个突变体中仅M46A酶活力提高了22%,比酶活达到2 611.40 U/mg。将内切菊粉酶INU3B的晶体结构提交至Eris (https://dokhlab.med.psu.edu/eris/login.php)[22]在线服务器预测M46A突变对热稳定性的影响,该突变使得氨基酸的去折叠自由能下降,ΔΔG为-2.72 kcal/mol,因此该突变很可能增加酶的热稳定性,这在工业应用上十分有利。
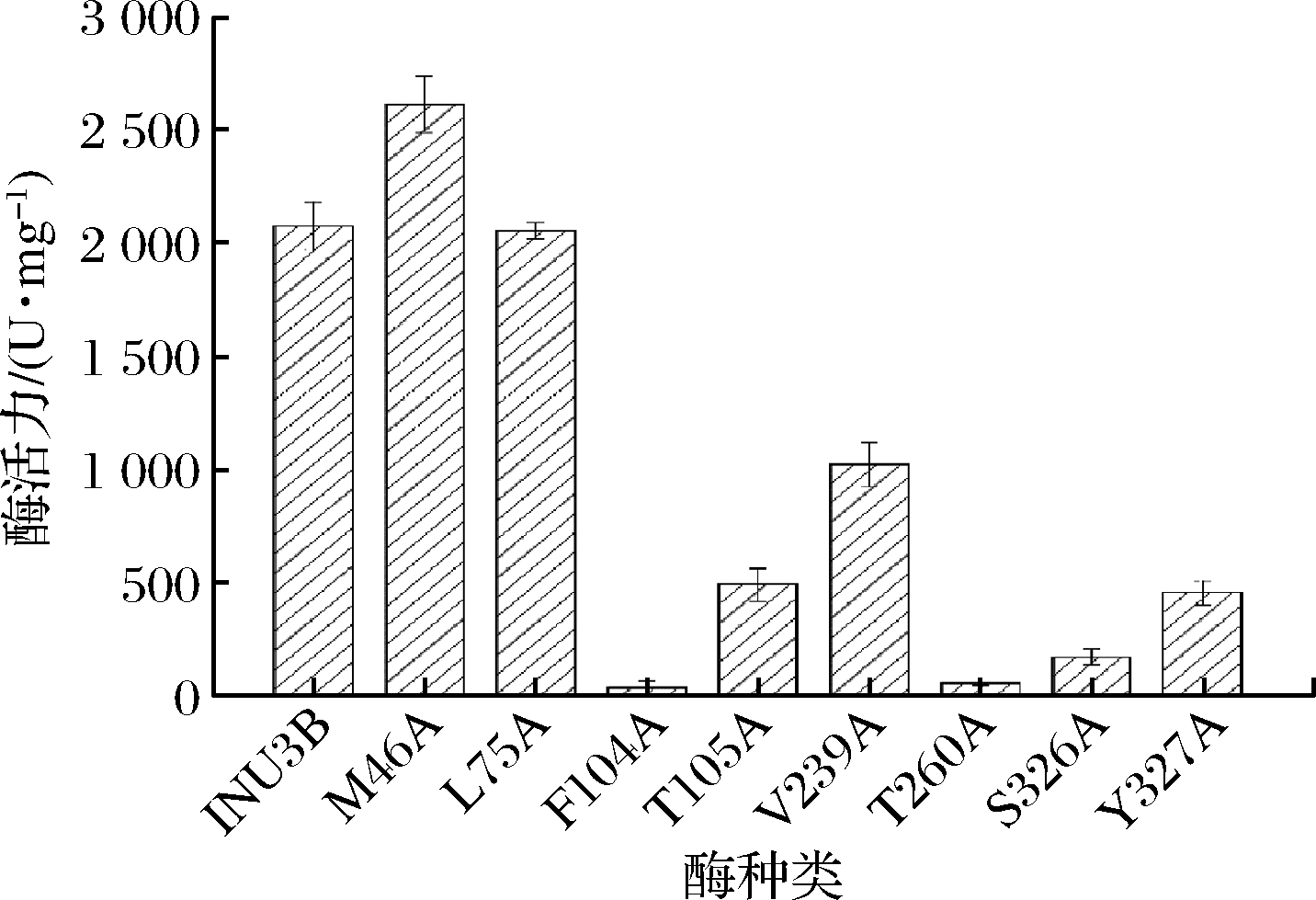
图6 INU3B及突变体内切菊粉酶活力测定
Fig.6 The specific enzyme activity of INU3B and mutant endo-inulinases generated by site-directed mutagenesis
M46位于结合口袋的底部且指向外部 (图7)。野生酶中M46与K337形成一个氢键,突变后A46仍然能与K337形成氢键,氢键的位置不变,突变未对在46 位的氨基酸的氢键相互作用产生明显的影响。有文献指出,Ala主要通过空间效应而非形成氢键或疏水键来调节酶活性[18]。因此推测,该位点突变为丙氨酸后侧链变短,减小了结合口袋的外表面积、体积和深度(图7),一定程度上使INU3B-GF4复合体结构更为紧凑,增强了底物与酶结合,从而增强了催化活力。
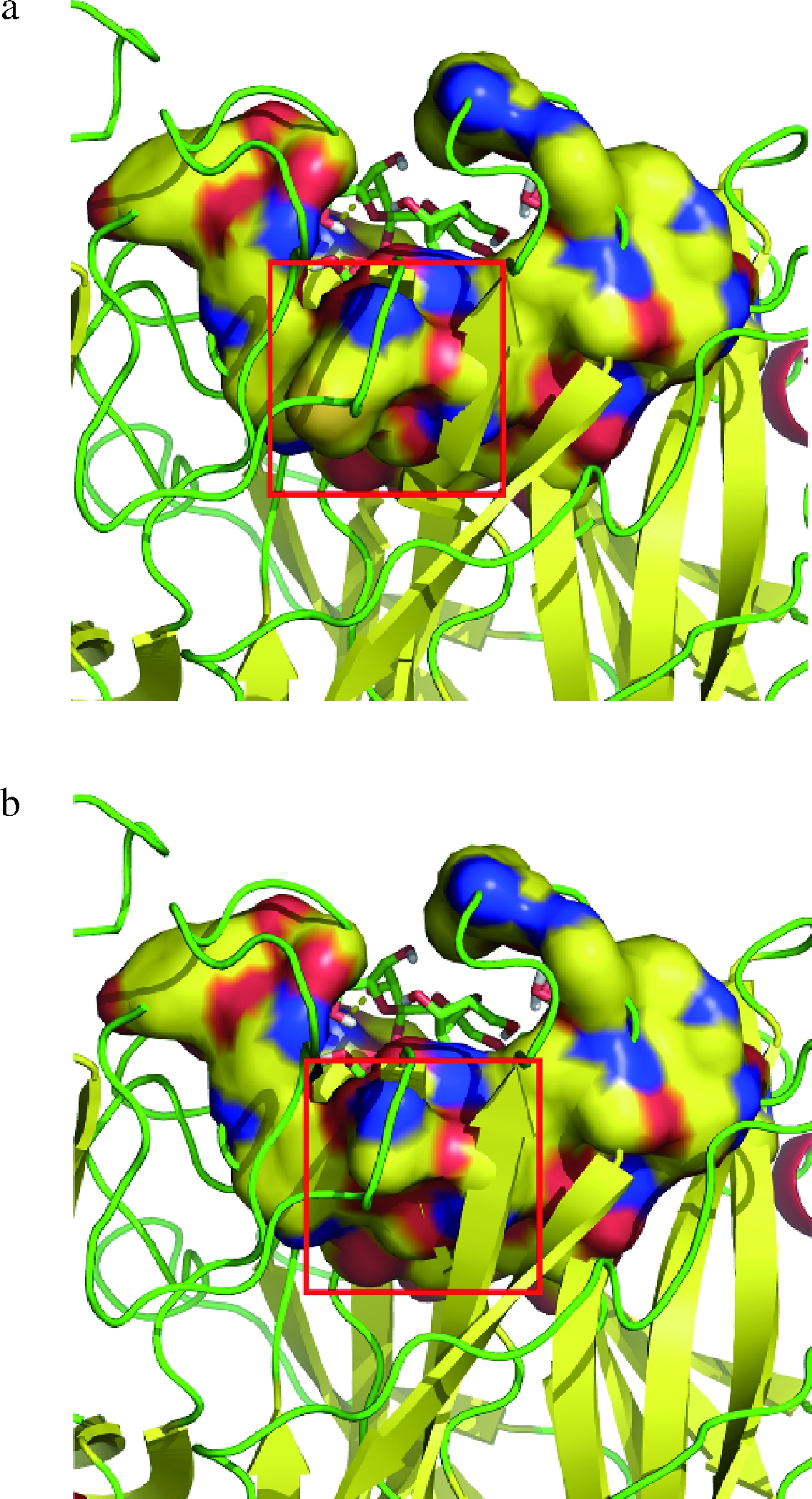
a-INU3B;b-M46A
图7 INU3B和突变体M46A结合口袋的形态
Fig.7 Shape of the binding pocket for INU3B and mutant M46A
F104、T260、S326、Y327位于结合口袋的侧面(图8),与底物分子较近。这些位点通过自身或者相互作用来形成稳定的结构,对结合口袋的构象有着关键的作用。F104的侧链较长且指向口袋内部,这缩短了与底物分子的距离,同时侧链末端的苯环为口袋的结构提供了支撑。因此突变之后,结合口袋的结构遭到破坏,酶活急剧下降。S326、Y327位于loop环上,共同组成了口袋末端,把底物限制在里面,突变成丙氨酸后,降低了酶-底物复合体的紧密度和稳定性从而降低了酶活性。
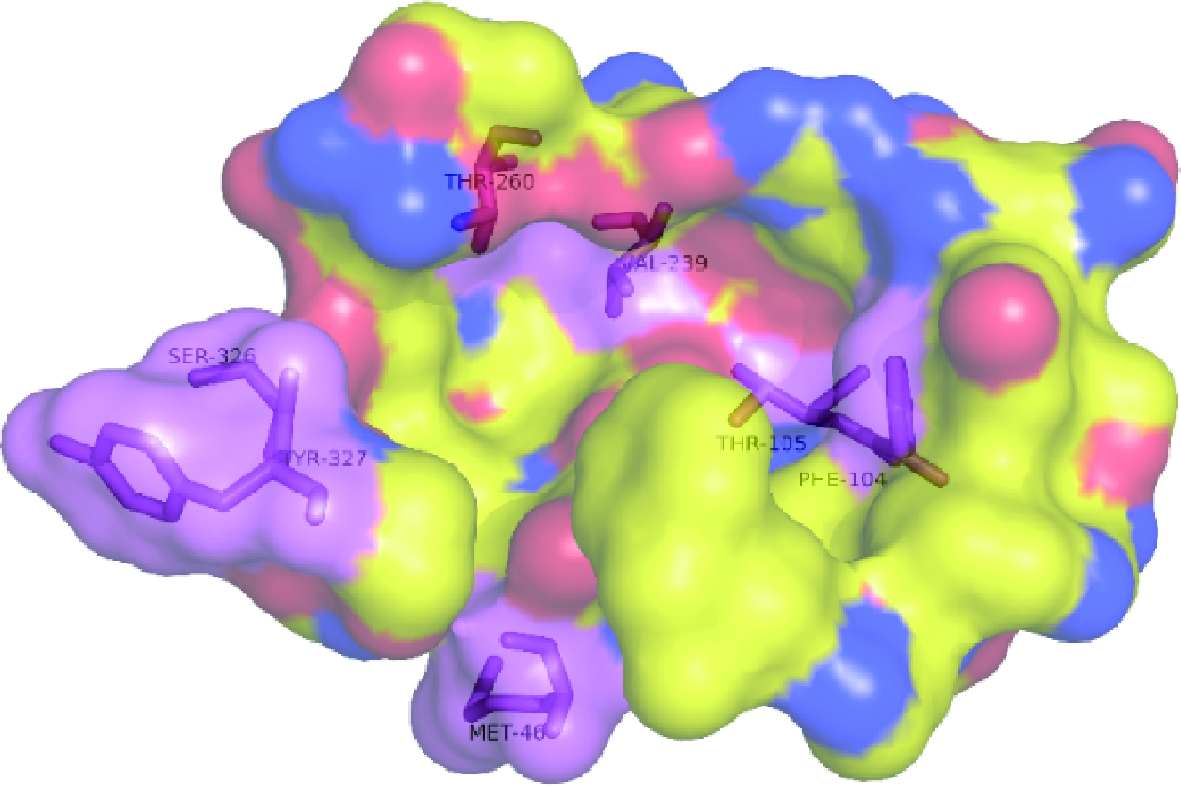
图8 突变位点在结合口袋中的位置 (突变氨基酸用紫色标出)
Fig.8 All mutants in the binding pocket
T105、V239位于结合口袋的凹槽底部(图8),与底物分子相对较远。所带的侧链基团也相对较小,突变成丙氨酸后对空间位阻影响不大。此外,比较发现突变前后氢键数量几乎没有减少,这也可能是其突变后仍保留了部分酶活性的原因。无论如何,这些负突变的形成表明了结合口袋附近的氨基酸对内切菊粉酶催化活性具有重大影响。
3 结论
本研究通过同源建模构建INU3B的三维蛋白结构模型,并与GF4进行分子对接。分析底物结合口袋的氨基酸,并进行丙氨酸突变以增大结合口袋,筛选获得了唯一的正向突变体M46A,比酶活力达2 611.40 U/mg,是原始酶INU3B的1.26倍。此外,其余6株突变体的酶活力均呈现不同程度的下降,说明结合口袋附近的氨基酸对内切菊粉酶催化活性影响重大。深入了解酶的结构与功能有助于指导酶分子进行定向改造,将是提高内切菊粉酶催化活力和低聚果糖的产量的有效途径。后续可以深入探究M46突变成丙氨酸后对酶学性质及功能的影响,同时可以在此基础上对结合口袋处的关键氨基酸进行饱和突变以深入解析相关功能。
[1] SANGEETHA P T,RAMESH M N,PRAPULLA S G.Recent trends in the microbial production,analysis and application of fructooligosaccharides[J].Trends Food Sci Technol,2005,16(10):442-457.
[2] O’CALLAGHAN A,VAN SINDEREN D.Bifidobacteria and their role as members of the human gut microbiota[J].Front Microbiol,2016,7:925.
[3] AL-SHERAJI S H,ISMAIL A,MANAP M Y,et al.Prebiotics as functional foods:A review[J].J Funct Food,2013,5(4):1 542-1 553.
[4] SCHMIDT K,COWEN P J,HARMER C J,et al.Prebiotic intake reduces the waking cortisol response and alters emotional bias in healthy volunteers[J].Psychopharmacology (Berlin),2015,232(10):1 793-1 801.
[5] KANGO N,JAIN SC.Production and properties of microbial inulinases:Recent advances[J].Food Biotechnol,2011,25(3):165-212.
[6] GANAIE M A,LATEEF A,GUPTA U S.Enzymatic trends of fructooligosaccharides production by microorganisms[J].Appl Biochem Biotechnol,2014,172(4):2 143-2 159.
[7] LIU G L,CHI Z,CHI Z M.Molecular characterization and expression of microbial inulinase genes[J].Crit Rev Microbiol,2013,39(2):152-165.
[8] CHEN M,LEI X,CHEN C Z,et al.Cloning,overexpression,and characterization of a highly active endoinulinase gene from Aspergillus fumigatus CL1 for production of inulo-oligosaccharides[J].Appl Biochem Biotechnol,2015,175(2):1 153-1 167.
[9] WATERHOUSE A,BERTONI M,BIENERT S,et al.SWISS-MODEL:Homology modelling of protein structures and complexes[J].Nucleic Acids Res,2018,46(W1):W296-W303.
[10] 曲戈,赵晶,郑平,等.定向进化技术的最新进展[J].生物工程学报,2018,34(1):1-11.
[11] POUYEZ J,MAYARD A,VANDAMME A M,et al.First crystal structure of an endo-inulinase,INU2,from Aspergillus ficuum:Discovery of an extra-pocket in the catalytic domain responsible for its endo-activity[J].Biochimie,2012,94(11):2 423-2 430.
[12] BAO M,NIU C T,XU X,et al.Identification,soluble expression,and characterization of a novel endo-inulinase from Lipomyces starkeyi NRRL Y-11557[J].Int J Biol Macromol,2019,137:537-544.
[13] TROTT O,OLSON A J.AutoDock Vina:Improving the speed and accuracy of docking with a new scoring function,efficient optimization,and multithreading[J].Journal of Computational Chemistry,2010,31(2):455-461.
[14] MILLER G L.Use of dinitrosalicylic acid reagent for determination of reducing sugar[J].Anal Biochem,1959,31(3):426-428.
[15] BRADFORD M M.A rapid method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].Anal Biochem,1976,72(s 1-2):248-254.
[16] LAMMENS W,LE ROY K,SCHROEVEN L,et al.Structural insights into glycoside hydrolase family 32 and 68 enzymes:Functional implications[J].Journal of Experimental Botany,2009,60(3):727-740.
[17] KIM K Y,NASCIMENTO A S,GOLUBEV A M,et al.Catalytic mechanism of inulinase from Arthrobacter sp.S37[J].Biochemical and Biophysical Research Communications,2008,371(4):600-605.
[18] NI H,ZENG S Q,QIN X,et al.Molecular docking and site-directed mutagenesis of a Bacillus thuringiensis chitinase to improve chitinolytic,synergistic lepidopteran-larvicidal and nematicidal activities[J].Int J Biol Sci,2015,11(3):304-315.
[19] GLIBOWSKI P,PIKUS S.Amorphous and crystal inulin behavior in a water environment[J].Carbohydr Polym,2011,83(2):635-639.
[20] 马俊,安启坤,唐文竹.菊粉外切酶的异源表达、纯化及酶学性质[J].食品与发酵工业,2019,45(4):25-30.
[21] MIAO R,XIE H,HO F M,et al.Protein engineering of α-ketoisovalerate decarboxylase for improved isobutanol production in Synechocystis PCC 6803[J].Metab Eng,2018,47:42-48.
[22] YIN S Y,DING F,DOKHOLYAN N V.Eris:An automated estimator of protein stability[J].Nat Methods,2007,4(6):466-467.