酸肉是采用自然发酵的方式制得的一种特色发酵食品,因其味道独特、香气宜人和口感较佳,在湖南省、贵州省和江西省等地广泛流传[1]。酸肉的制作流程为将盐均匀涂抹在生鲜肉上,而后将处理好的鲜肉放入密封性能良好的土陶中,同时加入辣椒、米粉、胡椒和糯米粉等辅料进行密封发酵,发酵完成至少需要2周[2]。近年来,国内学者围绕酸肉制作工艺进行了诸多优化实验,其中姜亚[3]得出制作酸肉最优的发酵工艺参数为糯米粉添加量25%、食盐添加6%、35 ℃发酵60 d;韦诚等[4]通过研究发现发酵周期为50~80 d的酸肉品质最佳;罗珍岑等[5]用乳酸链球菌素(Nisin)对酸肉贮藏时间进行了研究,结果显示Nisin的添加可以显著延长产品的贮藏时间。微生物类群对发酵食品的品质形成和食用安全性均具有较大的影响[6],但是目前有关酸肉微生物类群的研究仍相对较少。
经过10多年的发展,高通量测序技术逐渐的被改进并优化,相较于上一代测序技术,其通量更高、测序成本更低且方便易操作[7],目前该技术被广泛应用于风干发酵肉[8]、发酵蔬菜[6]以及发酵饮料[9]微生物多样性解析中。本研究采用MiSeq高通量测序技术对湖南省张家界市慈利县和湖南省湘西土家族苗族自治州古丈县酸肉微生物多样性进行揭示,在对其门属含量、α多样性和β多样性进行比较分析的基础上,使用BugBase以及PICRUSt软件对酸肉中细菌表型和所行使的功能进行预测。通过本研究的实施以期为后续酸肉产品品质的改善提供数据支撑。
1 材料与方法
1.1 材料与试剂
酸肉:纳入本研究的酸肉样品共有12 个,依次编号为CL1~CL5和GZ1~GZ7,其中CL1~CL5采集自慈利县城西市场和柳林农贸市场,GZ1~GZ7采集自古丈县农贸市场和默戎镇农贸市场。所有酸肉样品均以猪肉为原料、切片大小一致且发酵时间为1个月左右。
rTaq酶、脱氧核糖核苷三磷酸、10×聚合酶链式反应缓冲液,北京全式金生物技术有限公司;微生物基因组提取试剂盒,德国QIAGEN公司;338F/806R正反向引物,武汉天一辉远生物科技有限公司。
1.2 仪器与设备
R930机架式服务器,美国DELL公司;FluorChem FC3化学发光凝胶成像系统,美国ProteinSimple公司;CR21N型高速离心机,日本日立金属株式会社;Vetiri梯度基因扩增仪,美国AB公司;Illumina MiSeq高通量测序平台,美国Illumina公司;ND-2 000C微量紫外分光光度计,美国Nano Drop公司。
1.3 实验方法
1.3.1 酸肉微生物宏基因组提取、16S rRNA序列扩增及高通量测序
使用试剂盒对酸肉中微生物宏基因组DNA进行提取,参照郭壮等[10]的扩增体系及条件对细菌进行PCR扩增,而后将检测合格且清洁的扩增产物邮寄至上海美吉生物医药科技有限公司,通过MiSeq PE300平台进行高通量测序。
1.3.2 序列质控及生物信息学分析
使用QIIME平台对质控后的下机序列进行分析[11],首次使用PyNAST工具将序列对齐[12],继而使用UCLUST算法构建分类操作单元(operational taxonomic unit,OTU)[13],然后使用ChimeraSlayer工具删除含有嵌合体的OTU[14],并从余下的OTU中挑选代表性序列在RDP[15]、Greengenes[16]和SILVA[17]数据库中进行综合比对,进而明确酸肉细菌类群的构成和相对含量,紧接着使用FastTree软件构建系统发育树[18],最后对细菌类群的α多样性和β多样性进行分析,其中α多样性指标包含Chao1指数和Shannon指数,β多样性分析为非加权主坐标分析(principal coordinate analysis,PCoA)和加权非加权组平均法(unweighted pair-group method with arithmetic mean,UPGMA)聚类分析。本研究将平均相对含量>1.0%的门和属定义为优势门和优势属。
1.3.3 BugBase表型及PICRUSt基因功能预测
将OTU数据文件和样品分类信息文件上传至BugBase网站(https://bugbase.cs.umn.edu/)进行样品表型预测[19];选取序列文件使用PICRUSt工具进行酸肉微生物基因功能预测,同时使用蛋白质直系同源簇数据库(clusters of orthologous groups of proteins,COGs)对预测的功能进行注释[20]。
1.3.4 数据分析
使用Past软件进行Mann-Whitney分析,使用R软件进行气泡图、瀑布图、相关性图和热图的绘制,使用Origin 2017软件绘制主坐标分析图。
2 结果与分析
2.1 酸肉细菌类群测序的基本情况
本研究使用Illumina MiSeq高通量测序技术对采集自慈利和古丈地区的酸肉细菌多样性进行了解析,其测序产出序列信息如表1所示。
表1 测序深度、序列比对情况和α多样性指标
Table 1 Sequencing depth, sequence alignment and α diversity index
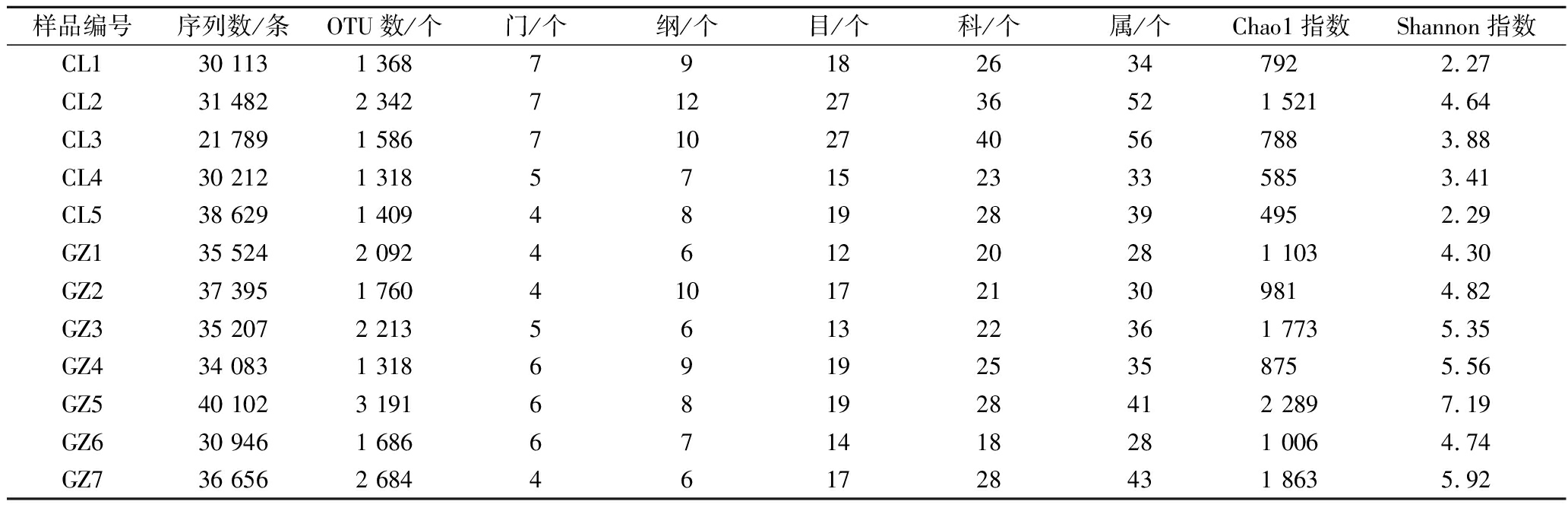
样品编号序列数/条OTU数/个门/个纲/个目/个科/个属/个Chao1指数Shannon指数CL130 1131 368791826347922.27CL231 4822 3427122736521 5214.64CL321 7891 5867102740567883.88CL430 2121 318571523335853.41CL538 6291 409481928394952.29GZ135 5242 092461220281 1034.30GZ237 3951 7604101721309814.82GZ335 2072 213561322361 7735.35GZ434 0831 318691925358755.56GZ540 1023 191681928412 2897.19GZ630 9461 686671418281 0064.74GZ736 6562 684461728431 8635.92
注:计算Chao1和Shannon指数时的测序深度均为20 510 条序列
由表1可知,12 个酸肉经测序共得到402 138 条高质量序列,其中有23 条序列比对失败,因此共有402 115 条高质量序列参与OTU的构建,按照97%相似度对序列进行划分,去除嵌合体序列后,得到17 088个OTU,并将其划分为9 个门、16 个纲、38 个目、59 个科和98 个属。由表1亦可知,GZ5的Chao1和Shannon指数均为最高,其数值分别为2 289和7.19,说明GZ5的群落丰度及多样性较其他酸肉均为最高。经Mann-Whitney检验发现,慈利地区酸肉细菌类群的Chao1和Shannon指数均显著偏低(P<0.05),这说明其菌群丰度和多样性均低于古丈地区酸肉。
2.2 酸肉细菌类群各分类学地位的含量及差异分析
由图1-a可知,慈利地区酸肉中平均相对含量>1.0%的细菌门有厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria),平均相对含量分别为93.86%、2.92%和2.63%;古丈地区酸肉中平均相对含量>1.0%的细菌门仅有Firmicutes,平均相对含量为99.07%。经Mann-Whitney检验发现,慈利地区酸肉中Firmicutes的含量显著偏低(P<0.05),而其他细菌门的含量在不同地区酸肉中差异不显著(P>0.05)。
由图1-b可知,慈利地区酸肉中平均相对含量>1.0%的细菌属为乳酸杆菌属(Lactobacillus)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、棒状杆菌属(Corynebacterium)和色盐杆菌属(Chromohalobacter),平均相对含量分别为40.23%、44.35%、7.94%、2.40%和1.64%;古丈地区酸肉中平均相对含量>1.0%的细菌属为Lactobacillus、Staphylococcus、Weissella、Lactococcus(乳球菌属)和Macrococcus(巨型球菌属),平均相对含量分别为62.61%、5.18%、22.79%、3.65%和1.33%。经Mann-Whitney检验发现,慈利地区酸肉中Lactococcus的含量显著低于古丈地区(P<0.05),分别为0.05%和3.65%,而其他细菌属的含量差异则不显著(P>0.05)。
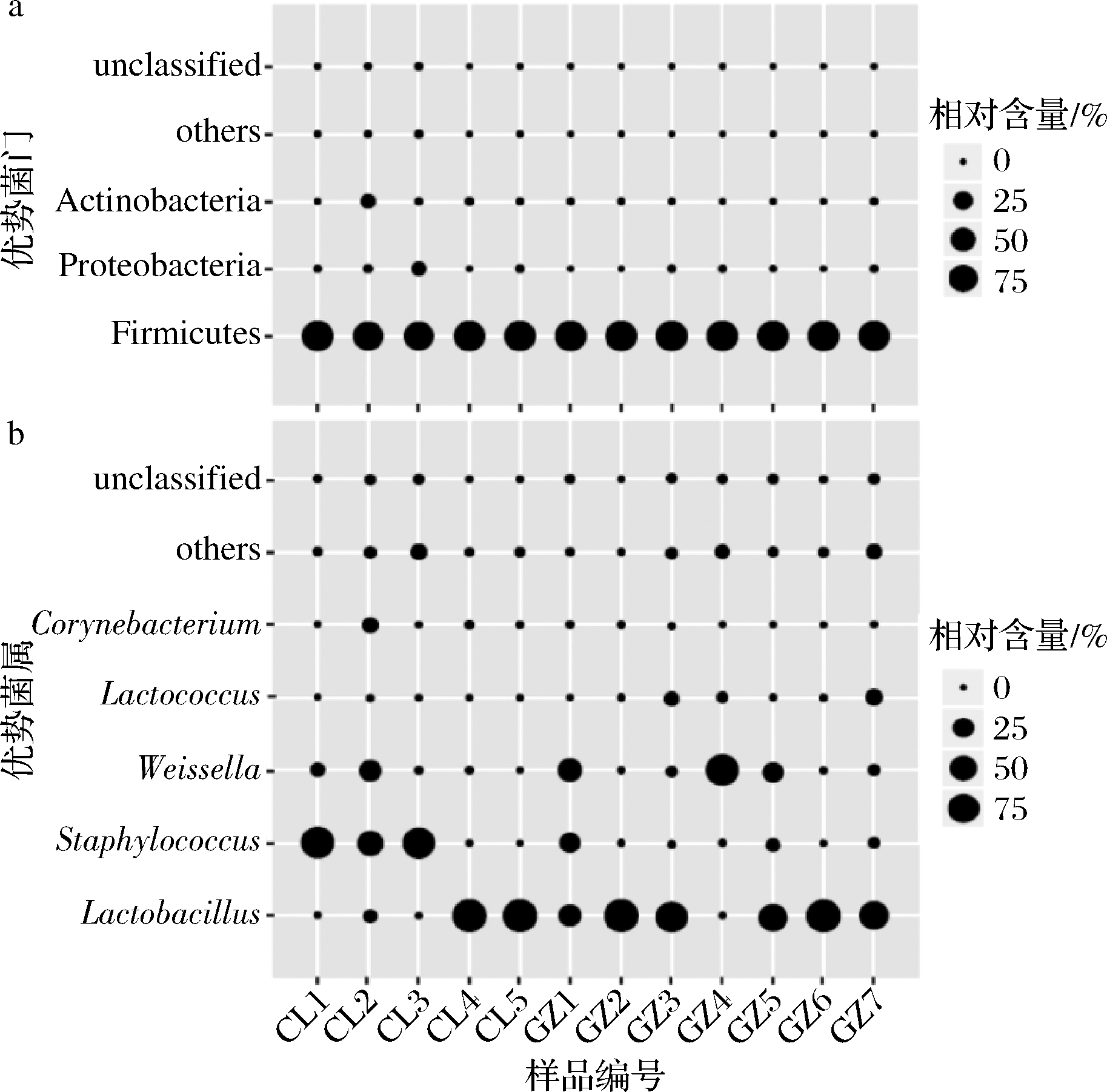
a-门水平;b-属水平
图1 基于门和属水平的酸肉细菌类群构成分析
Fig.1 Bacterial taxa composition analysis of sour meat based on phylum and genus levels
此外,Lactobacillus和Weissella在慈利和古丈地区酸肉中的累计平均相对含量分别为48.17%和85.40%,且古丈地区酸肉中亦含有3.65%的Lactococcus。通过研究云南地区酸肉细菌类群构成,米瑞芳等[21]发现Lactobacillus、Weissell和Lactococcus为其优势菌属,平均相对含量分别为53.68%、22.31%和8.63%。由此可见,由于不同地区的酸肉均以猪肉为原料,并采用厌氧发酵制作而成,因此为乳酸菌的生长创造了有利的环境,进而促使其成为了优势细菌类群。乳酸菌可使酯类物质含量明显增加,进而提升了酸肉的风味品质[22]。除酸肉外,腊肉亦是湖北和湖南地区较为常见的发酵肉制品,采用高通量测序技术,董蕴等[23]对恩施地区腊肉的菌群多样性进行了解析,结果发现其蕴含的细菌类群主要为Staphylococcus、嗜冷杆菌属(Psychrobacter)和假交替单胞菌属(Pseudoalteromonas)。酸肉和腊肉细菌类群存在明显差异的原因可能在于两者的制作工艺不同,腊肉为开放式发酵,且增加了烟熏等操作工艺[24]。
若某一OTU存在于所有12 个酸肉样品中,则将其定义为核心OTU,本研究共发现了6 个核心OTU,其平均相对含量及注释结果如图2所示。

图2 核心OTU平均相对含量的瀑布图
Fig.2 Waterfall plot representing the average relative content of core OTUs
由图2可知,6 个核心OTU分别为OTU1407(Staphylococcus)、OTU3714(Lactobacillus)、OTU13870(Weissella)、OTU6884(Lactococcus)、OTU366(Corynebacterium)和OTU9607(Macrococcus),其平均相对含量分别为14.89%、5.01%、1.79%、1.62%、0.99%和0.63%,累计平均相对含量达24.9%。由此可见,2个地区制作的酸肉共有部分细菌类群,这与属水平上的研究结果一致。Staphylococcus和Macrococcus均隶属于葡萄球菌科(Staphylococcaceae)[25],有研究发现Staphylococcus具有降低广式腊肠中亚硝酸盐含量的作用[26],而隶属于Macrococcus的M. caseolyticus(溶酪大球菌)则可以加快广式腊肠中脂肪和蛋白质的氧化与风味的降解[27],因而划分到OTU1407和OTU9607的细菌类群对酸肉品质的形成可能具有积极的意义。值得一提的是,部分隶属于Staphylococcus的细菌类群还具有潜在的致病性,如AN等[28]研究发现,S.epidermidis(表面葡萄球菌)可导致脂溢性皮炎的发生,BUBECK WARDENBURG等[29]研究发现S.aureus(金黄色葡萄球菌)可导致肺炎,严重情况下甚至还能导致死亡,因此在后续研究中积极开展隶属于Staphylococcus菌株的筛选和安全性评价,对酸肉制品的产业化推动具有积极的意义。
进一步分析发现,OTU10585(芽孢杆菌属)仅存在于5 个慈利地区酸肉样品中,但其相对含量均低于0.1%,何江红等[30]研究发现Bacillus在川藏牦牛酸肉中具有较强的产蛋白酶和淀粉酶能力,由于酸肉制作过程中常加入糯米粉或米粉为原料,因而推测划分到OTU10585的细菌可能会通过分解蛋白质和淀粉进而对酸肉的品质产生影响。此外,本研究没有发现有OTU仅存在于7 个古丈地区酸肉样品中。为进一步解析核心细菌类群间的互作关系,本研究对核心OTU的相关性进行了分析,其结果如图3所示。OTU13870(Weissella)和OTU366(Corynebacterium)之间呈现显著正相关(P<0.05),且其相关系数为0.92,由此推测隶属于Weissella与Corynebacterium的细菌类群在酸肉中可能存在一定的共生关系。
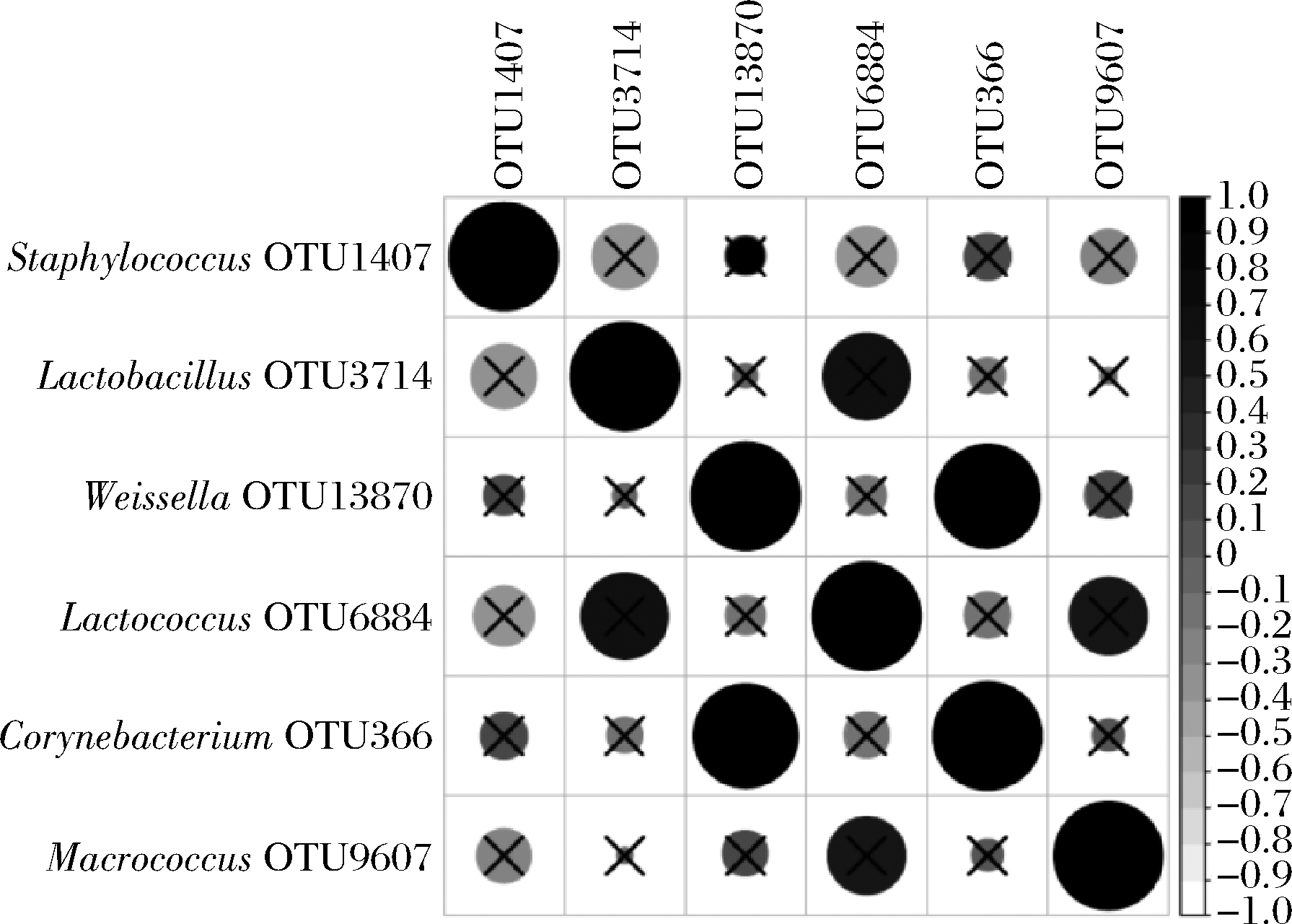
图3 核心OTU的相关性分析
Fig.3 Correlation analysis of core OTUs
注:圆圈颜色越深且直径越大,表明其相关系数越大
2.3 两个地区酸肉细菌类群的β多样性分析
本研究进一步采用非加权PCoA和加权UPGMA聚类分析对2个地区酸肉细菌类群的β多样性进行解析,其结果如图4所示。由图4-a可知,2个地区制作的酸肉样品在空间排布上呈现出分离趋势,其中慈利地区样品主要分布在2、3象限,而古丈地区样品分布在1、2、4象限。图4-b的结果亦呈现出相似的现象,这说明2个地区的酸肉细菌群落结构存在一定差异。
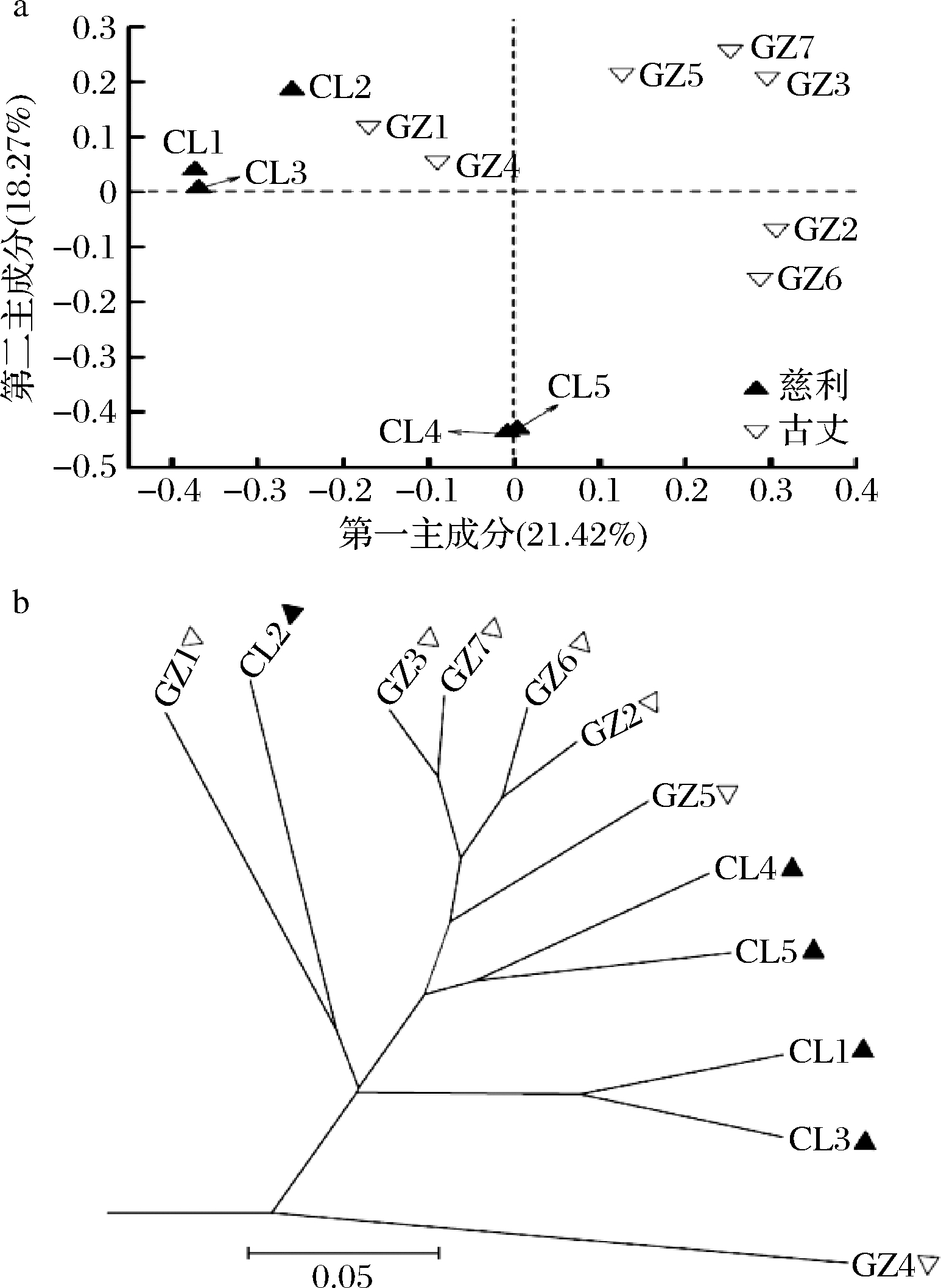
a-非加权PCoA;b-加权UPGMA聚类分析
图4 非加权PCoA和加权UPGMA聚类分析
Fig.4 Unweighted PCoA and weighted UPGMA clustering analysis
2.4 酸肉细菌类群的表型预测及功能预测分析
本研究使用BugBase对2个地区酸肉细菌类群的表型进行了预测,结果如图5所示。
由图5可知,2个地区酸肉细菌类群在好氧、移动原件含量、生物膜形成、氧化胁迫耐受、革兰氏阳性、革兰氏阴性和致病潜力等表型上无显著差异(P>0.05),而在厌氧和兼性厌氧表型上分别有显著(P<0.05)和非常显著差异(P<0.01),这说明慈利地区酸肉细菌类群中厌氧细菌的相对含量显著高于古丈地区(P<0.05),而兼性厌氧型细菌的相对含量则呈现相反的趋势(P<0.01)。本研究进一步对酸肉细菌类群的基因功能进行了预测,其结果如图6所示。

a-厌氧;b-好氧;c-移动原件含量;d-兼性厌氧;e-生物膜形成;f-氧化胁迫耐受;g-革兰氏阳性;h-革兰氏阴性;i-致病潜力
图5 慈利和古丈地区酸肉细菌类群的表型预测
Fig.5 Phenotypic prediction of sour meat samples from Cili and Guzhang regions
注:*代表差异显著(P<0.05),**代表差异非常显著(P<0.01)
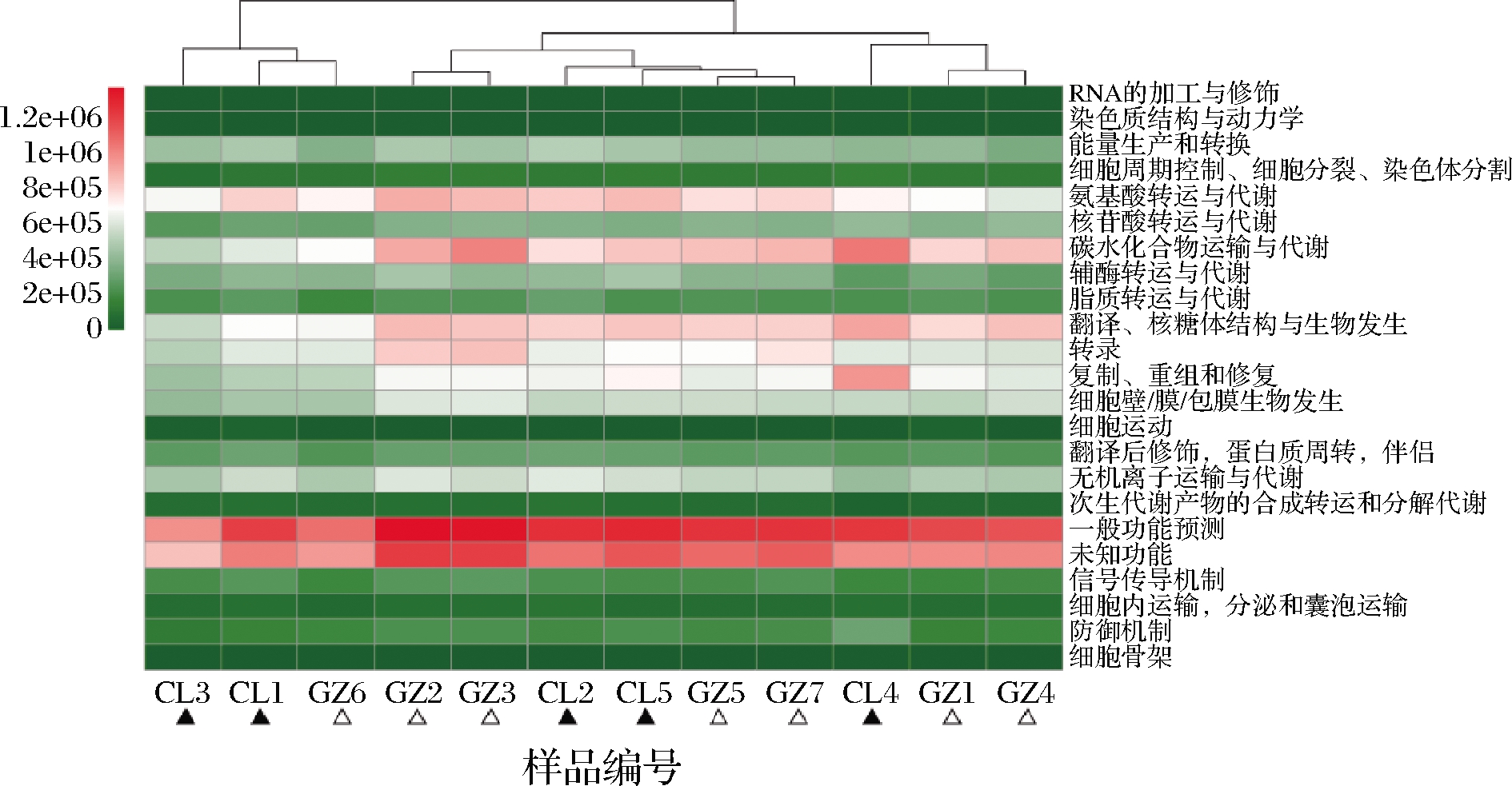
图6 慈利和古丈地区酸肉细菌类群功能预测的热图
Fig.6 Functional prediction heat map of sour meat samples from Cili and Guzhang regions
由图6可知,慈利和古丈地区酸肉细菌类群在“氨基酸转运与代谢”、“碳水化合物运输与代谢”和“翻译、核糖体结构与生物发生”功能上的相对含量较高,这说明2个地区酸肉细菌类群可能在自身代谢相关功能上表达较高。经Mann-Whitney分析发现,2个地区酸肉细菌类群在各功能上的表达差异均不显著(P>0.05)。
3 结论
使用高通量测序技术解析发现,慈利和古丈地区酸肉中的细菌类群主要由Lactobacillus、Staphylococcus和Weissella构成,主要为厌氧菌或兼性厌氧菌,且细菌类群在“氨基酸转运与代谢”、“碳水化合物运输与代谢”以及“翻译、核糖体结构与生物发生”等自身代谢相关功能上表达较高。同时,通过研究2个地区的酸肉发现,不同地区制作的酸肉细菌类群多样性存在一定的差异。
[1] 冉春霞, 谭小蓉, 王静, 等.我国传统酸鲊肉制品的研究现状及展望[J].中国酿造, 2015, 34(11):14-19.
RAN C X, TAN X R, WANG J, et al.Present status and prospects of research on traditional sour Zha meat products in China[J].China Brewing, 2015, 34(11):14-19.
[2] LYU J, YANG Z X, XU W H, et al.Relationships between bacterial community and metabolites of sour meat at different temperature during the fermentation[J].International Journal of Food Microbiology, 2019, 307:108286.
[3] 姜亚. 酸肉生产工艺优化及其细菌多样性的研究[D].南宁:广西大学, 2014.
JIANG Y.Study on the acid meat production optimization and bacterial diversity[D].Nanning:Guangxi University, 2014.
[4] 韦诚, 常荣, 段珍珍, 等.发酵时长对酸肉物理特性及食用安全性的影响[J].食品与发酵工业, 2019, 45(8):86-92.
WEI C, CHANG R, DUAN Z Z, et al.Effects of fermentation time on physical properties and food safety of sour meat[J].Food and Fermentation Industries, 2019, 45(8):86-92.
[5] 罗珍岑, 段珍珍, 谭小琴, 等.Nisin对酸肉常温贮藏中营养卫生及感官品质的影响[J].食品与发酵工业, 2019, 45(23):176-182.
LUO Z C, DUAN Z Z, TAN X Q, et al.Effects of Nisin on nutritional hygiene and sensory quality of sour pork stored at room temperature[J].Food and Fermentation Industries, 2019, 45(23):176-182.
[6] LIANG H P, YIN L G, ZHANG Y H, et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai, a traditional Chinese fermented vegetable food, as assessed by Illumina MiSeq sequencing, DGGE and qPCR assay[J].Annals of Microbiology, 2018, 68(2):111-122.
[7] MAO Y, WEI B Y, TENG J W, et al.Analyses of fungal community by Illumina MiSeq platforms and characterization of Eurotium species on Liupao tea, a distinctive post-fermented tea from China[J].Food Research International, 2017, 99:641-649.
[8] JUSTYNA P, ANNALISA R, VINCENZA P, et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J].Food Microbiology, 2015, 46(8):342-356.
[9] WURIHAN, BAO L S, HASIGAOWA, et al.Bacterial community succession and metabolite changes during the fermentation of koumiss, a traditional Mongolian fermented beverage[J].International Dairy Journal, 2019, 98 (7):1-8.
[10] 郭壮, 葛东颖, 尚雪娇, 等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技, 2018, 39(22):93-98;106.
GUO Z, GE D Y, SHANG X J, et al.Comparative analysis on the diversity of bacterial microflora in degenerated and normal pit mud[J].Science and Technology of Food Industry, 2018, 39(22):93-98;106.
[11] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[12] XIAO Y, ZHENG Y, WU S, et al.Bacterial community structure of autotrophic denitrification biocathode by 454 pyrosequencing of the 16S rRNA gene[J].Microbial Ecology, 2015, 69(3):492-499.
[13] GLASSMAN S I, MARTINY J B H.Broadscale ecological patterns are robust to use of exact sequence variants versus operational taxonomic units[J].mSphere, 2018.DOI:10.1128/mSphere.00148-18.
[14] ZIMMERMAN N, IZARD J, KLATT C G, et al.The unseen world:Environmental microbial sequencing and identification methods for ecologists[J].Frontiers in Ecology and the Environment, 2014, 12(4):224-231.
[15] BACCI G, BANI A, BAZZICALUPO M, et al.Evaluation of the performances of ribosomal database project (RDP) classifier for taxonomic assignment of 16S rRNA Metabarcoding sequences generated from Illumina-Solexa NGS[J].Journal of Genomics, 2015, 3(4):36-39.
[16] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al.Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Applied and Environmental Microbiology, 2006, 72(7):5 069-5 072.
[17] YILMAZ P, PARFREY L W,YARZA P, et al.The SILVA and "All-species Living Tree Project (LTP)" taxonomic frameworks[J].Nucleic Acids Research, 2014, 42:643-648.
[18] PRICE M N, DEHAL P S, ARKIN A P.Fast Tree:Computing large minimum evolution trees with profiles instead of a distance matrix[J].Molecular Biology and Evolution, 2009, 26(7):1 641-1 650.
[19] WARD T, LARSON J, MEULEMANS J, et al.BugBase predicts organism-level microbiome phenotypes[J].bioRxiv, 2017.DOI:10.1101/133462.
[20] LANGILLE M G I,ZANEVELD J, CAPORASO J G, et al.Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J].Nature Biotechnology, 2013, 31(9):814-821.
[21] 米瑞芳, 陈曦, 熊苏玥, 等.传统自然发酵酸肉中细菌群落多样性与风味品质分析[J].食品科学, 2019, 40(2):85-92.
MI R F, CHEN X, XIONG S Y, et al.Bacterial community diversity and flavor characteristics of traditional naturally fermented sour meat[J].Food Science, 2019, 40(2):85-92.
[22] 米瑞芳, 陈曦, 戚彪, 等.乳杆菌发酵剂对酸肉挥发性风味成分的影响[J].肉类研究, 2018, 32(4):48-55.
MI R F, CHEN X, QI B, et al.Effect of Lactobacillus starter cultures on the volatile flavor components of sour meat[J].Meat Research, 2018, 32(4):48-55.
[23] 董蕴, 王玉荣, 王尧, 等.基于变性梯度凝胶电泳和MiSeq高通量测序技术分析恩施地区腊肉的细菌多样性[J].肉类研究, 2018, 32(10):37-42.
DONG Y, WANG Y R, WANG Y, et al.Evaluation of bacterial diversity in Chinese bacon from Enshi by denatured gradient gel electrophoresis and MiSeq high-throughput sequencing[J].Meat Research, 2018, 32(10):37-42.
[24] 文开勇, 汪月, 文鹏程, 等.四川传统腊肉中微生物群落结构研究[J].食品与发酵工业, 2020, 46(3):36-42.
WEN K Y, WANG Y, WEN P C, et al.Study on microbial community structure in Sichuan traditional bacon[J].Food and Fermentation Industries, 2020, 46(3):36-42.
[25] MAZHAR S, HILL C, MCAULIFFE O.The genus Macrococcus:An insight into its biology, evolution, and relationship with Staphylococcus[J].Advances in Applied Microbiology,2018, 105:1-50.
[26] 田甜, 詹锐琪, 张雅琳, 等.微生物发酵技术对广味香肠安全性提升的研究[J].中国调味品, 2019, 44(12):26-30.
TIAN T, ZHAN R Q, ZHANG Y L,et al.Study on safety improvement of cantonese sausage by microbial fermentation technology[J].China Condiment, 2019, 44(12):26-30.
[27] 吴燕涛, 赵谋明, 孙为正, 等.内源性发酵剂Macrococcus caseolyticus发酵广式腊肠的风味物质成分分析[J].食品工业科技, 2011, 32(7):207-209;213.
WU Y T, ZHAO M M, SUN W Z, et al.Analysis of volatile compounds of cantonese sausage fermented by Macrococcus caseolyticus separated from cantonese sausage[J].Science and Technology of Food Industry, 2011, 32(7):207-209;213.
[28] AN Q, SUN M, QI R Q, et al.High Staphylococcus epidermidis colonization and impaired permeability barrier in facial seborrheic dermatitis[J].Chinese Medical Journal, 2017, 130(14):1 662-1 669.
[29] BUBECK WARDENBURG J, BAE T, OTTO M, et al.Poring over pores:α-hemolysin and panton-valentine leukocidin in Staphylococcus aureus pneumonia[J].Nature Medicine, 2007, 13(12):1 405-1 406.
[30] 何江红, 王荣钰, 舒小芳, 等.川西藏区牦牛酸醡肉中产蛋白酶和淀粉酶菌株的分离鉴定及其生理特性分析[J].中国调味品, 2018, 43(12):60-65.
HE J H, WANG R Y, SHU X F, et al.Isolation and identification of protease and amylase producing strains in sour yak meat from western Sichuan Tibetan areas and analysis of their physiological characteristics[J].China Condiment, 2018, 43(12):60-65.