中国白酒是世界上著名蒸馏酒之一,具有千年历史。白酒的生产过程是在一个开放和不受控制的环境中,来自大曲、高粱、空气、土壤和未消毒的生产工具中的微生物可能为白酒的生产提供丰富的微生物来源。其中,大曲是一个非常重要的角色。酱香型高温大曲是以小麦为主要原料、高温发酵而成,是白酒酿造所需的糖化发酵剂和生香剂,提供功能微生物及其代谢物,有助于风味物质的产生,形成独特的酱香型白酒风味[1-4]。
近年来多项研究利用高通量测序对大曲的微生物群落进行了广泛的研究[5-6]。JIN等[6]研究酱香型大曲微生物群落,表明芽孢杆菌属(Bacillus)、乳杆菌属(Lactobacillus)、Thermoactinomyces、Saccharomyces、Aspergillus和Thermomyces在大曲中是优势微生物。大曲中微生物的相互作用产生了具有多种香气成分和复杂风味的白酒。地衣芽孢杆菌是优势菌群之一,高产吡嗪类、挥发性酸类、芳香族和酚类等化合物,增强酱香风味[7]。Bacillus velezensis和Bacillus subtilis进行生物强化改善大曲风味,增加酯类、吡嗪类和醇类等挥发性化合物的含量[8]。大曲的生产是在开放且复杂的工作环境中进行,DU等[9]对大曲微生物来源进行了分析,大曲的细菌群落主要来源于小麦原料,而真菌群落大多来自制作环境。由此可见,小麦对大曲微生物组成和白酒独特风味的形成有着重要的影响。然而,基于这方面的研究却很少。之前的研究集中于小麦原料的质量上[10],而忽略了小麦原料微生物对大曲微生物区系的影响。许多微生物会栖息在原辅料表面,与原辅料一起进入到发酵体系中。例如,在葡萄酒发酵中,葡萄皮表面的微生物可以在发酵中生长和存活,参与葡萄酒发酵并增强葡萄酒的感官复杂性[11-12]。同样地,由于地理因素和环境生长条件的差异,形成了独特的小麦原料微生物区系。这些微生物为大曲微生物群落的演化带来更多的可能性,进而影响功能微生物的代谢,影响白酒的品质和风味。
本次研究采用了高通量测序技术,揭示了来自中国5个省份不同产地的21个小麦样品的微生物群落结构。模拟固态发酵制备大曲,采用顶空固相微萃取(headspace solid-phase microextraction,HS-SPME)结合GC-MS分析模拟大曲的风味物质。这项研究有助于阐明小麦原料微生物对大曲风味物质的影响,为制曲原料的选择提供参考依据,对保持大曲风味组成的一致性具有重要意义。
1 材料与方法
1.1 样品采集
21个小麦样品分别来自贵州,安徽、四川、湖北和河南5个省区不同产地,高温大曲产自茅台镇(贵州省,中国),所有样品于-20 ℃下保存备用。
1.2 主要试剂
DNA提取试剂盒,德国QIAGEN公司;2×Taq PCR Master Mix,宝日医生物技术(北京)有限公司。
1.3 仪器与设备
Nano-Drop2000 微量核酸测定仪,无锡莱弗思生物实验器材有限公司;可伸缩的试验箱,上海博讯实业有限公司医疗设备厂;C1000 PCR仪,赛默飞世尔有限公司;Leco Pegasus GC-HRT 4D 气相色谱-质谱仪,美国力可公司;7890B 气质色谱仪、TR-FFAP毛细管气相色谱柱(30 m×0.25 mm×0.25 μm),美国安捷伦公司。
1.4 实验方法
1.4.1 样品微生物总DNA的提取
取200 mg样品加入适量液氮研磨后使用DNeasy PowerSoil Kit试剂盒提取样品总DNA,步骤参考说明书;使用Nano-Drop2000微量核酸测定仪检测DNA的含量,并用1%的琼脂糖凝胶电泳检验DNA的完整性。
1.4.2 PCR扩增及测序
细菌16S rDNA全长扩增引物为27F(5′-AGRGTTYGATYMTGGCTCAG-3′)和1492R(5′-RGYTACCTTGTTACGACTT-3′);真菌ITS区扩增引物为ITS1(5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2(5′-GCTGCGTTCTTCATCGATGC-3′)。PCR扩增反应体系(50 μL):1 μL模板DNA,上下游引物各1 μL,2×Taq PCR Master Mix 25 μL,22 μL 无菌双蒸水。反应条件:94 ℃预变性3 min,94 ℃变性30 s,55 ℃退火45 s,72 ℃延伸45 s,循环30次,72 ℃延伸10 min[13]。扩增序列由武汉华大基因科技有限公司进行测序。16S rDNA序列使用Pacbio测序平台,ITS扩增序列使用Hiseq2500测序平台。
1.4.3 高通量测序数据分析
对原始数据进行过滤、去除接头以及低质量序列。使用FLASH软件(v1.2.11)[14],利用重叠关系将双末端测序列连接配对,获得高质量标签。利用UPARSE软件(v7.0.1090)[15]在97%相似度下进行聚类得到可操作分类单元(operational taxonomic unit, OTU),并用UCHIME软件(v4.2.40)[16]去除嵌合体得到OTU代表序列。利用RDP classife软件(v2.2)[16]将OTU代表序列与数据库比对进行物种注释。细菌OTU使用Greengenes(v201305)数据库、真菌OTU使用UNITE(v201407.03)数据库比对。
Alpha多样性使用MOTHUE软件(v1.31.2)计算[17],Beta多样性和样品聚类分析使用QIIME软件(v1.8.0)计算[18]。聚类树和物种丰度柱状图使用phytools和R语言(v3.5.1)绘制。
1.4.4 固态发酵实验
取750 g小麦原料,加入52.5 g母曲和285 mL无菌水混合均匀后,捏制成型。放入可伸缩的试验箱, 在30 ℃下发酵30 d。以高温大曲生产工艺为基础进行固态发酵[19]。
1.4.5 大曲挥发性成分的测定
取2 g粉碎大曲样品放入15 mL顶空瓶中,在50 ℃自动SPME装置中平衡5 min,以500 r/min萃取30 min后进入GC-MS系统。气相色谱条件:TR-FFAP毛细管气相色谱柱(30 m×0.25 mm×0.25 μm);进样口温度250 ℃;氦气流速1 mL/min;升温程序:初始温度45 ℃,保持3 min,以4 ℃/min升温速率升温至230 ℃,保持6 min。质谱条件:电子轰击电离;电子能量为70 eV;四级杆温度为150 ℃,离子源温度为230 ℃,采集模式为全扫描,扫描范围35~550 m/z[20]。
2 结果与分析
2.1 小麦样品微生物多样性分析
采用高通量测序技术,分析21个小麦样品的微生物组成。样品详细信息见表1。
细菌16S rDNA全长测序,共获得171 147个高质量序列,并按97%的相似度聚类得到23 572个OTU。对于真菌,共获得1 002 154个高质量序列,共聚类得到1 492个OTU。Shannon指数稀释曲线分析表明小麦样品细菌16S rDNA和真菌ITS测序到达平台期,说明本次测序水平下覆盖了样品中绝大多数的微生物群落信息,充分的展现群落多样性(图1)。
表1 小麦样品信息
Table 1 Wheat sample information
序号样品名称品种产地地区省份1LHKM-1软质小麦老河口湖北省2LHKM-2软质小麦老河口湖北省3NZM软质小麦南漳湖北省5XYM-2软质小麦襄阳湖北省6YJM-1软质小麦延津河南省7YJM-2软质小麦延津河南省8THM-1软质小麦唐河河南省9THM-2软质小麦唐河河南省10GSM软质小麦固始河南省11HXM-2软质小麦滑县河南省12XCM软质小麦西昌四川省13BDM软质小麦西昌四川省14NCM软质小麦南充四川省15FYM-1软质小麦阜阳安徽省16FYM-2软质小麦阜阳安徽省17PTM软质小麦合肥安徽省18M1903软质小麦仁怀贵州省19M1904软质小麦仁怀贵州省20M1905软质小麦仁怀贵州省21M1906软质小麦仁怀贵州省
本研究通过Shannon指数和物种数目(observed species)对样品微生物的Alpha多样性进行表征。Alpha多样性结果显示了不同产地小麦样品多样性的差异。对于细菌群落,湖北省小麦样品的群落丰富度(species richness)和多样性(species diversity)均高于其他4个省份,四川省小麦样品Alpha多样性最低(图2-a)。对于真菌群落,四川省小麦样品的群落丰富度和多样性均高于其他4个省份,河南省小麦样品的Alpha多样性最低(图2-b)。
Beta多样性由主坐标分析(principal co-ordinates analysis,PCoA)表征,显示不同产地小麦样品OTU存在差异。对于细菌群落,PC1解释了总变异的41.75%,PC2解释了总变异的22.83%(图2-c),表明同一省份不同产地小麦样品的细菌群落相似性很低。对于真菌群落,PC1解释了总变异的41.9%,PC2解释了总变异的30.05%,揭示了小麦样品真菌群落相似性较高(图2-d)。

a-细菌测序稀释曲线;b-真菌测序稀释曲线
图1 小麦样品中细菌和真菌测序稀释曲线
Fig.1 Rarefaction curves of bacterial and fungal community of wheat samples
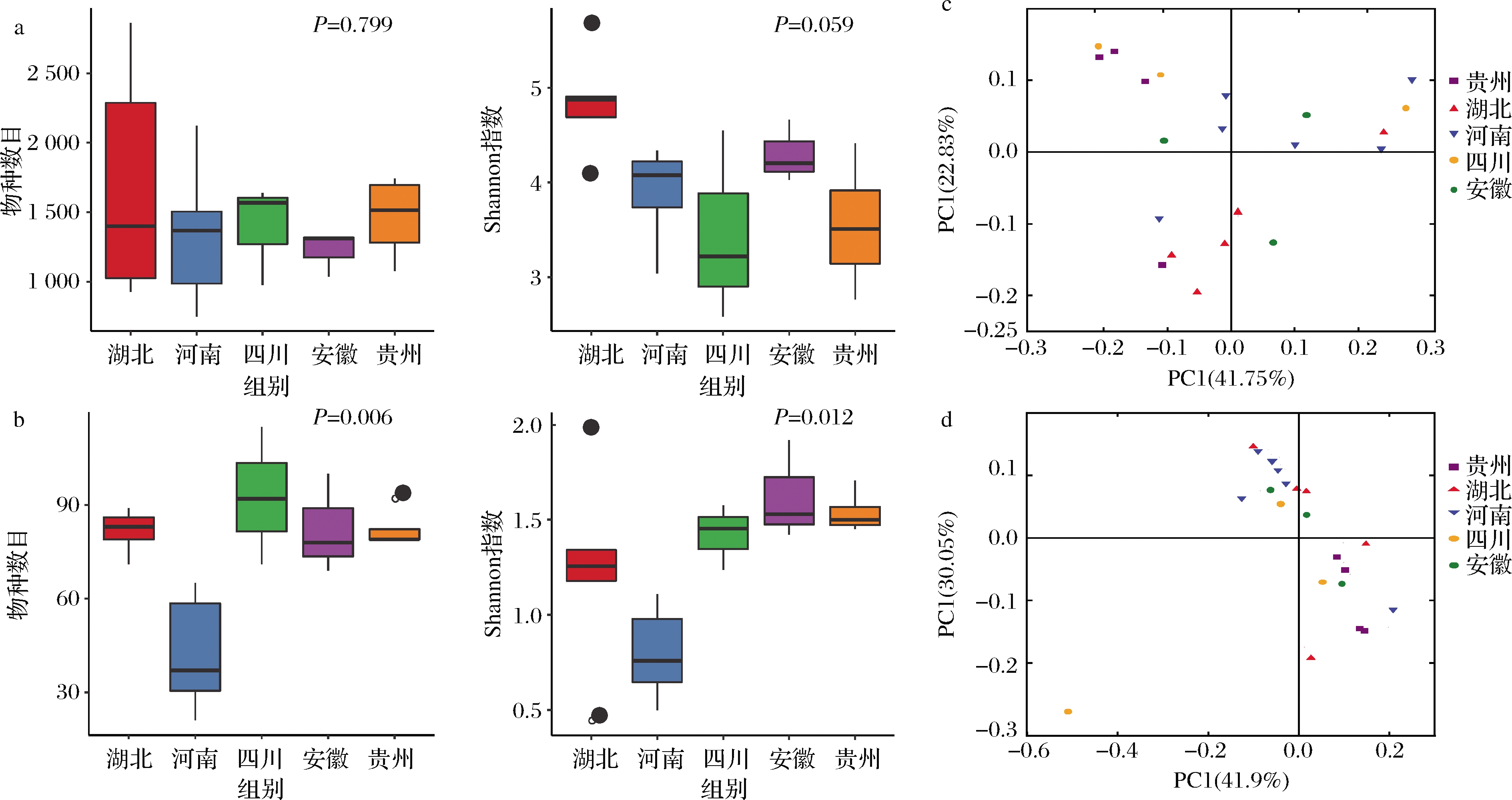
a-细菌Alpha多样性分析;b-真菌Alpha多样性分析;c-细菌PCoA分析;d-真菌PCoA分析
图2 小麦样品细菌和真菌多样性分析
Fig.2 Alpha diversity estimates of bacterial and fungal communities
PCoA分析揭示了不同产地小麦样品的细菌群落结构有较大差异,而真菌群落结构则差异相对较小。小麦原料微生物包括细菌和真菌微生物[21]。小麦细菌微生物大部分寄附在种子表面,内生细菌在植物体内生物量相对较少[22-23]。小麦表面细菌受生长环境中纬度、温度、湿度等自然条件的影响[21,24],因此,不同产区小麦样品由于生长环境不同,细菌微生物组成有所差异。小麦真菌微生物主要受田间和贮藏环境的影响[21,24],在贮存期间,贮藏真菌逐渐代替田间真菌,不同贮藏环境下,优势真菌差异较小[25]。小麦内生真菌在田间生长期和贮存期间优势真菌没有明显差异,种子内部的真菌物种差异不明显[26-28],因此不同产地小麦真菌微生物群落结构差异相对较小,本文分析结果与此前报道结果相符。此外,细菌是大曲风味物质的主要贡献者。因此,根据细菌微生物群落的相似性,我们将21个小麦样品分为4组,具体样品信息见表2。
表2 重分组样品信息
Table 2 Regroup wheat sample information
组别样品名称品种产地地区省份BDM软质小麦西昌四川省M1903软质小麦仁怀贵州省AM1905软质小麦仁怀贵州省M1906软质小麦仁怀贵州省NCM软质小麦南充四川省FYM.1软质小麦阜阳安徽省FYM.2软质小麦阜阳安徽省BGSM软质小麦固始河南省HXM.2软质小麦滑县河南省THM.2软质小麦唐河河南省LHKM.1软质小麦老河口湖北省THM.1软质小麦唐河河南省CXCM软质小麦西昌四川省YJM.1软质小麦延津河南省LHKM.2软质小麦老河口湖北省NZM软质小麦南漳湖北省PTM软质小麦合肥安徽省DXYM.1软质小麦襄阳湖北省XYM.2软质小麦襄阳湖北省YJM.2软质小麦延津河南省M1904软质小麦仁怀贵州省
2.2 小麦样品细菌群落组成差异分析
21个小麦样品检测出的细菌门主要包括厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)和蓝藻门(Cyanobacteria)。属水平共检测出466个属。不同产地的小麦样品中细菌群落组成存在差异(图3-a)。A组样品中泛菌属(Pantoea)、假单胞菌属(Pseudomonas)及Massilia是相对丰度较高的细菌属。样品BDM以泛菌属(57.28%)、鞘氨醇单胞菌属(Sphingomonas)(5.72%)和假单胞菌属(4.01%)为优势细菌属。B组样品中泛菌属是主要的细菌属。短杆菌属(Curtobacterium)(38.98%)、假单胞菌属(2.28%)和鞘氨醇单胞菌属(0.80%)在样品FYM.1中占主导地位。与A、B两组样品不同,Massilia在C组样品中占优势地位。样品YJM.1中Massilia相对丰度最高(10.88%),其次为Frigoribacterium(2.93%)和鞘氨醇单胞菌(0.85%)。与C组样品相似,Massilia也是D组样品中最丰富的细菌属。样品PTM中Massiilia、假单胞菌属和鞘氨醇单胞菌属占比较高,分别为20.28%、20.08%和6.88%。
种水平上共检测出917个细菌种群。如图3-b所示,Pantoea agglomerans在A组样品中占绝对优势。样品BDM的优势细菌种群为P.agglomerans(54.16%)、Pseudomonas brenneri(3.16%)和Curtobacterium flaccumfaciens(1.17%)。B组样品中,样品FYM.1中Curtobacterium citreum相对丰度最高(33.12%),其次为C.flaccumfaciens(4.5%)和Sphingomonas aerolata(0.48%)。C组样品中Massiilia aurea是主要的细菌种群。样品YJM.1中M.aurea、Frigoribacterium faeni和Massilia brevitalea占主要地位,相对丰度分别为6.30%,1.51%和1.39%。D组样品,除样品PTM和XYM.1外,M.aurea是其他样品中最丰富的细菌种群。样品PTM中Sphingomonas faeni(17.28%)为优势细菌,样品XYM.1中C.flaccumfaciens(5.81%)相对丰度较高。总体而言,P.agglomerans 和 M.aurea是小麦样品主要的细菌种群。

a-属水平细菌群落结构;b-种水平细菌群落结构
图3 小麦样品细菌微生物群落结构(相对丰度>0.5%)
Fig.3 Composition of dominant bacterial genera/ species of wheat samples (relative abundance>0.05%)
2.3 小麦样品真菌群落组成差异分析
真菌门主要包括子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota)。21个样品共检测出200个属,其微生物群落组成差异较小。如图4-a所示,A组样品中链格孢属(Alternaria)、附球菌属(Epicoccum)和枝孢属(Cladosporium)是主要的真菌属。样品BDM中链格孢菌属相对丰度最高(61.84%)。B组样品中,附球菌属是样品FYM.1(37.67%)和样品GSM(67.99%)的优势真菌。在C组样品LHKM.1中,链格孢属(91.38%)是最丰富的真菌属。对于D组样品,样品XYM.1中曲霉属占比较高(40.00%),其次是附球菌属(18.57%)和裸孢壳属(Emericella)(16.71%)。
种水平共检测出172个真菌种群。如图4-b所示,A组样品以黑附球菌(Epicoccum nigrum)和黄曲霉菌(Aspergillus cibarius)为优势真菌。样品BDM、M1906和NCM以黄曲霉菌为主要的真菌,相对丰度分别为61.82%、36.11%和29.39%。B组样品中,Alternaria alternata是样品FYM.2(58.98%)、HXM.1(84.26%)和THM.2(84.12%)的优势真菌。C组样品中,Tilletia bromi是样品XCM的重要真菌种群,但在其他样品中相对丰度非常低(<0.5%)。D组样品中,样品XYM.1中的优势真菌群落为黄曲霉菌(38.95%),而其他小麦样品中A. alternata相对丰度较高。总体而言,A.alternate和黑附球菌是小麦样品中的主要真菌群落。
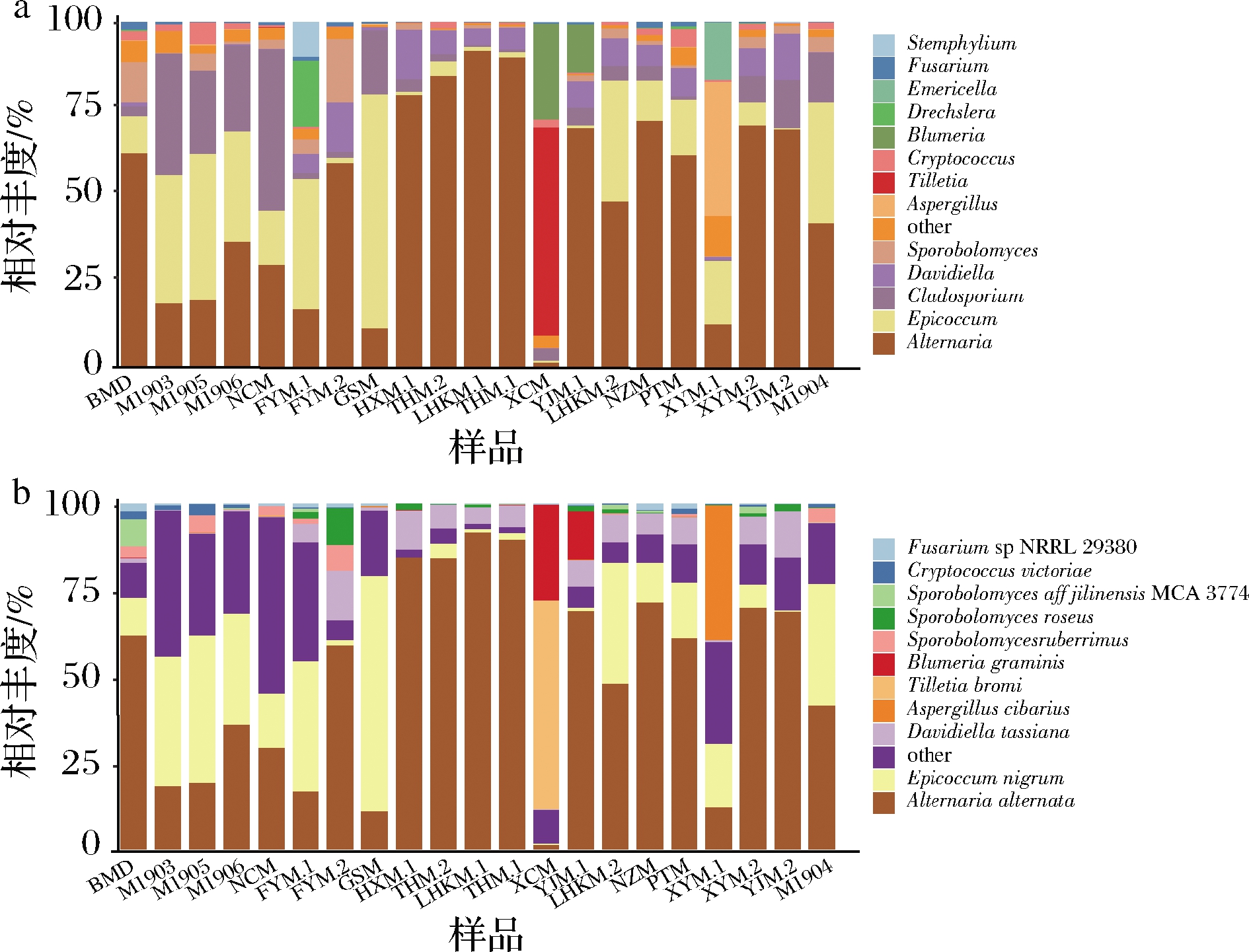
a-属水平真菌群落结构;b-种水平真菌群落结构
图4 小麦样品真菌微生物群落结构(相对丰度>0.5%)
Fig.4 Composition of dominant fungal genera/species of wheat samples (relative abundance>0.05%)
聚类分析表明,不同产地小麦样品微生物组成存在差异(图5)。结果表明,细菌种水平上,样品BDM与M1906细菌群落结构相似性最高。样品FYM与XYM.1、YJM.1与XCM、PTM与THM.2的细菌群落组成最为相似,而真菌群落组成有所不同。大曲的细菌主要来源于小麦原料,而真菌则来自大曲的生产环境和工具。基于PCoA分析(图2-c),每组分别选择一种代表性样品(A组:BDM;B组:FYM.1;C组:YJM.1;D组:PTM)进行固态发酵实验。为了探究小麦微生物组成与大曲风味物质间的相互关系,使用与代表性样品细菌群落结构相似性较高的小麦样品进行模拟制曲,比较其风味物质的差异。

a-细菌属水平聚类分析;b-真菌属水平聚类分析
图5 小麦样品微生物聚类树与丰度组合图 (相对丰度>0.5%)
Fig.5 Unweighted pair group method with arithmetic mean analysis(relative abundance>0.05%)
2.4 小麦微生物组成与大曲风味物质关系分析
通过HS-SPME-GC-MS测定挥发性物质。如图6-a所示,4个代表性样品(A组:BDM;B组:FYM.1;C组:YJM.1;D组:PTM)共鉴定出212种挥发性成分,包括醇类(16)、酮类(20)、酯类(19)、吡嗪类(20)、烷烃类(27)、酸类(5)、醛类(5)、芳烃类(10)和其他类(90)。模拟的大曲中醇类和吡嗪类物质是高丰度的挥发性物质。为了更好地评估模拟大曲的风味,选用11种特征风味物质[29],比较其在模拟大曲中的差异,计算其相对含量,结果如表3所示。
结果表明,模拟大曲的特征风味物质有所不同。样品BDM-D中鉴定出5种特征风味物质、YJ-D、PTM-D和FY-D中鉴定出7种(图6-b)。模拟大曲以四甲基吡嗪为主体风味物质,其中样品BDM-D和PTM-D中四甲基吡嗪相对含量为突出(>70%),四甲基吡嗪是酱香香气的重要风味物质之一。此外,2,3-丁二醇在模拟大曲的相对含量高于其他5种风味物质,模拟大曲中丁酸、苯乙醇和苯甲醛相对含量较低(<1%)。
主成分分析(principal components analysis,PCA)显示了模拟大曲中风味物质组成的相似性和差异性。如图7所示,8个大曲样品形成独立的5簇。样品M1906-D与BDM-D距离较近,独立形成一簇;样品TH-D与PTM-D远离其他样品,单独形成一簇;样品FY-D与XY-D、YJ-D与XC-D距离较近,分别形成一簇。样品M1906-D与BDM-D特征风味物质的种类和相对含量较为相似,以四甲基吡嗪为主(相对含量>70%)。样品XY-D检测出7种特征风味物质,种类与样品FY-D相同,且风味物质的组成较为相近。样品XC-D与YJ-D风味物质的组成也较为相近,四甲基吡嗪是主要的风味物质,2,3-丁二醇的含量相对较高。同样地,样品TH-D和PTM-D的大曲风味物质组成较为相似。微生物区系聚类中(图5-a),BDM与M1906在相同分支,细菌群落结构相似性最高;样品FYM与XYM.1在相近分支、YJM.1与XCM、PTM与THM.2分支距离较短,细菌微生物群落组成最为相似。PCA分析与微生物区系聚类分析结果相似,总的来说,小麦样品细菌群落结构相似性越高,模拟大曲的特征风味物质组成有着更高的相似度。
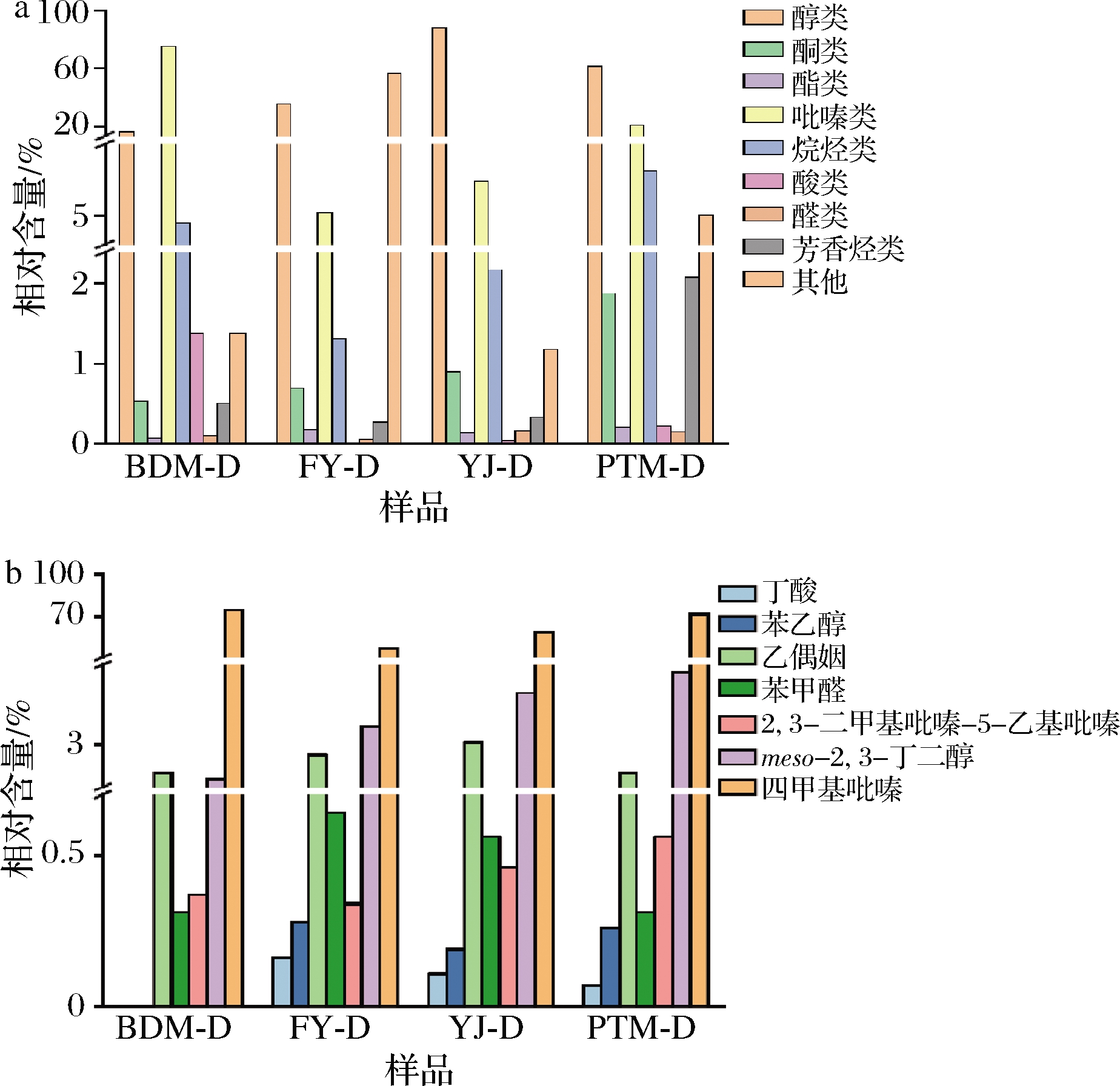
a-大曲挥发性物质种类分布;b-大曲中特征风味物质的相对含量
图6 大曲挥发性物质
Fig.6 Bar graph of the concentrations of the volatile compounds in Daqu
表3 大曲中11种特征风味物质的相对含量 单位:%
Table 3 Distribution of the 11 characteristic flavor compounds in simulated Daqu samples
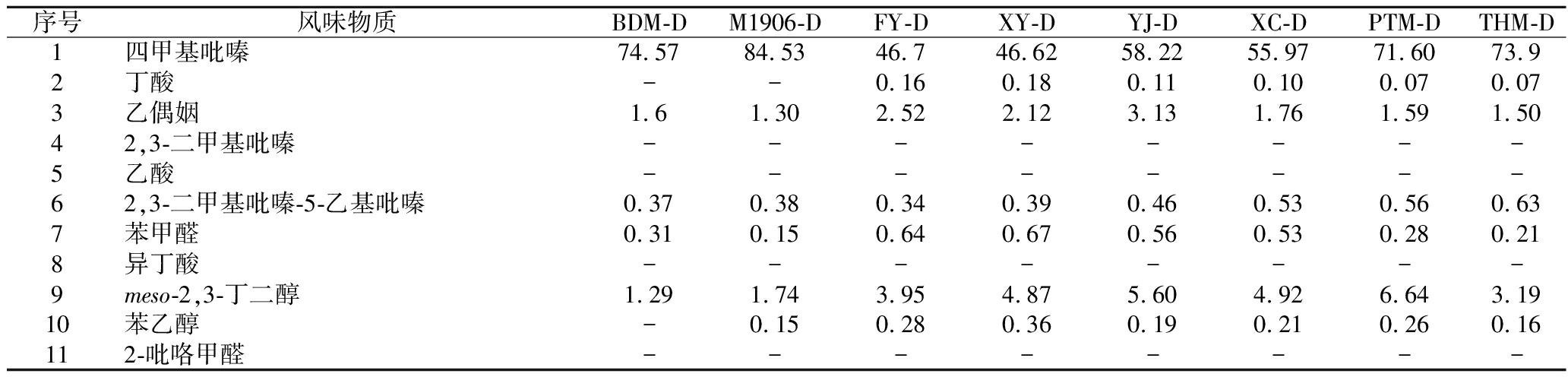
序号风味物质BDM-DM1906-DFY-DXY-DYJ-DXC-DPTM-DTHM-D1四甲基吡嗪74.5784.5346.746.6258.2255.9771.6073.92丁酸--0.160.180.110.100.070.073乙偶姻1.61.302.522.123.131.761.591.5042,3-二甲基吡嗪--------5乙酸--------62,3-二甲基吡嗪-5-乙基吡嗪0.370.380.340.390.460.530.560.637苯甲醛0.310.150.640.670.560.530.280.218异丁酸--------9meso-2,3-丁二醇1.291.743.954.875.604.926.643.1910苯乙醇-0.150.280.360.190.210.260.16112-吡咯甲醛--------
注:“-”代表未检出该特征风味物质

图7 大曲风味物质组成PCA分析
Fig.7 Principal components analysis of flavor compounds in Daqu samples
3 结论与讨论
白酒是中国传统发酵食品的典型代表,而大曲是白酒酿造过程中微生物富集的重要步骤。本研究中,通过16S rDNA全长测序和ITS rDNA测序分析了来自中国5个省份不同地区的21个小麦原料的微生物群落。通过固态发酵实验,比较了模拟大曲中挥发性物质的差异。结果表明,细菌组成中,样品BDM,FYM.1,YJM.1和PTM分别以P.agglomerans(54.16%)、Curtobacterium citreum(33.12%)、M.aurea(6.30%)和S.faeni(17.28%)为主要的细菌。就真菌而言,样品BDM(61.84%)、YJM.1(69.11%)和PTM(61.41%)以链格孢菌属为主;而FYM.1样品以附球菌属(37.67%)为优势真菌。使用不同产地小麦制曲时,大曲的风味物质有所不同,模拟大曲以四甲基吡嗪为主体风味物质。样品BDM和M905产区小麦原料制曲时,可能会增强酱香风味,增加吡嗪类物质的含量。此外,选择样品PTM和YJM产区小麦制备大曲,2,3-丁二醇含量可能会有所提高。通过微生物聚类分析和大曲风味物质组成PCA分析,小麦样品微生物与风味物质组成有密切的相关性,小麦原料微生物相似度较高,大曲风味物质组成更加相似,对了解不同产区小麦原料生产的大曲风味成分组成具有指导意义。
高温大曲是由未经灭菌的原料经过固态发酵制成的,而小麦是主要原料。这些小麦原料的表面和内部组织是各种微生物的天然栖息地。自然和内在因素都会影响微生物群落的生长和分布,形成独特的微生物群落。制曲原料会影响大曲的微生物群落结构,进而影响风味微生物的代谢,直接或间接影响白酒品质和风味。本研究有助于阐明制曲原料微生物组成对大曲风味及白酒品质的影响,为制曲原料的选择提供参考依据,对保持大曲风味物质的一致性和白酒的感官复杂性具有重要意义。
[1] 任聪, 杜海, 徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报, 2017, 57(6):885-898.
REN C, DU H, XU Y.Advances in microbiome study of traditional Chinese fermented foods[J].Acta Microbiologica Sinica, 2017, 57(6):885-898.
[2] 孙利林, 李立郎, 胡萍, 等.酱香型白酒大曲的微生物菌群结构及风味成分分析[J].现代食品科技, 2020,36(8):299-306;193.
SUN L L, LI L L, HU P, et al.Analysis of microbial community structure and flavor composition of Maotai-flavor Daqu[J].Modern Food Science and Technology, 2020,36(8):299-306;193.
[3] WANG M Y, YANG J G, ZHAO Q S, et al.Research progress on flavor compounds and microorganisms of Maotai flavor Baijiu[J].Journal of Food Science, 2019, 84(1):6-18.
[4] 王雪山, 杜海, 徐岩.清香型白酒发酵过程中微生物种群空间分布[J].食品与发酵工业, 2018, 44(9):1-8.
WANG X S, DU H, XU Y.Spatial distribution of microbial community during Chinese light-flavor liquor fermentation[J].Food and Fermentation Industries, 2018, 44(9):1-8.
[5] ZHENG X W, YAN Z, ROBERT NOUT M J, et al.Characterization of the microbial community in different types of Daqu samples as revealed by 16S rRNA and 26S rRNA gene clone libraries[J].World Journal of Microbiology and Biotechnology, 2015, 31(1):199-208.
[6] JIN Y, LI D Y, AI M, et al.Correlation between volatile profiles and microbial communities:A metabonomic approach to study Jiang-flavor liquor Daqu[J].Food Research International, 2019, 121:422-432.
[7] ZHANG R, WU Q, XU Y.Aroma characteristics of Moutai-flavour liquor produced with Bacillus licheniformis by solid-state fermentation[J].Letters in Applied Microbiology, 2013, 57(1):11-18.
[8] HE G Q, DONG Y, HUANG J, et al.Alteration of microbial community for improving flavor character of Daqu by inoculation with Bacillus velezensis and Bacillus subtilis [J].LWT, 2019, 111:1-8.
[9] DU H, WANG X S, ZHANG Y H, et al.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].International Journal of Food Microbiology, 2019, 297:32-40.
[10] 刘淼, 丁丽, 赵梦梦, 等.基于主成分分析的不同小麦品种大曲发酵动态品质评价[J].食品与发酵工业, 2020, 46(6):19-24.
LIU M, DING L, ZHAO M M, et al.Comprehensive quality evaluation of Daqu of different wheat varieties based on principal components analysis[J].Food and Fermentation Industries, 2020, 46(6):19-24.
[11] BOKULICH N A, JOSEPH C M L, ALLEN G, et al.Next-generation sequencing reveals significant bacterial diversity of botrytized wine[J].PloS One, 2012, 7(5):e36357.
[12] CIANI M, COMITINI F, MANNAZZU I, et al.Controlled mixed culture fermentation:A new perspective on the use of non-Saccharomyces yeasts in winemaking[J].FEMS Yeast Research, 2010, 10(2):123-133.
[13] AI M, QIU X, HUANG J, et al.Characterizing the microbial diversity and major metabolites of Sichuan bran vinegar augmented by Monascus purpureus [J].International Journal of Food Microbiology, 2019, 292:83-90.
[14] ![]() , SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2 957-2 963.
[15] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods, 2013, 10(10):996-998.
[16] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2 194-2 200.
[17] SCHLOSS P D, WESTCOTT S L, RYABIN T, et al.Introducing mothur:Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J].Applied and Environmental Microbiology, 2009, 75(23):7 537-7 541.
[18] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5):335-336.
[19] FAN G S, TENG C, XU D, et al.Improving ethyl acetate production in Baijiu manufacture by Wickerhamomyces anomalus and Saccharomyces cerevisiae mixed culture fermentations[J].BioMed Research International, 2019.DOI:10.1155/2019/1470543.
[20] 沈海月. 酱香型白酒香气物质研究[D].无锡:江南大学,2010.
SHEN H Y.Study on aroma substances of sauce-flavored liquor[D].Wuxi:Jiangnan University, 2010.
[21] 李贺勤, 江绪文.种子微生物的研究进展[J].中国农学通报, 2014, 30(21):47-51.
LI H Q, JIANG X W.Progression of seed-associated microorganism[J].Chinese Agricultural Science Bulletin, 2014, 30(21):47-51.
[22] 王鸣岐. 种子微生物及种子生理的现状与展望[J].复旦学报(自然科学), 1959(2):197-205.
WANG M Q.Seed microarganisms in relation to seed physiology, present situation and pespective[J].Journal of Fudan University(Natural Science), 1959(2):197-205.
[23] 彭雪霁, 周建新, 鞠兴荣, 等.小麦及小麦粉中微生物污染与控制研究进展[J].粮食储藏, 2009,38(3):37-42.
PENG X J, ZHOU J X, JU X R,et al.Research and advance on microbe contamination and its control in wheat and wheat flour[J].Grain Storage, 2009,38(3):37-42.
[24] 王志伟, 纪燕玲, 陈永敢.植物内生菌研究及其科学意义[J].微生物学通报, 2015, 42(2):349-363.
WANG Z W, JI Y L, CHEN Y G.Studies and biological significances of plant endophytes[J].Microbiology China, 2015, 42(2):349-363.
[25] 李彪, 李国长, 柳琴.微生物对小麦及其加工品污染的调查分析[J].粮食储藏, 2003,32(5):36-38.
LI B, LI G C, LIU Q.Investigation of microorganism contamination on wheat and its by-products[J].Grain Storage, 2003,32(5):36-38.
[26] 凌长有. 小麦内生菌和根际土微生物多样性的研究[D].武汉:华中农业大学,2018.
LIN C Y.Microbial diversity on endophyte and rhizosphere soil in wheat [D].Wuhan:Huazhong Agricultural University, 2018.
[27] 杨文兰, 马桂珍.冀东地区小麦种子内寄生真菌的种类及致病性的研究[J].河北职业技术师范学院学报, 2001, 15(3):25-29.
YANG W L, MA G Z.Study of parasitic fungi in wheat seed in east Hebei province and its pathogenicity[J].Journal of Hebei Vocation-Technical Teachers College, 2001, 15(3):25-29.
[28] 岳红宾, 王守正, 袁红霞, 等.小麦种子内生真菌区系分析[J].河南农业大学学报, 1996, 30(4):354-357.
YUE H B, WANG S Z, YUAN H X, et al.Analysis of endophytic fungi flora in wheat seeds[J].Acta Agriculturae Universitatis Henanensis, 1996, 30(4):354-357.
[29] 陈宗校, 林琳, 韩莹, 等.基于风味物质组成的高温功能大曲应用方法[J].中国酿造, 2017, 36(1):98-101.
CHEN Z X, LIN L, HAN Y, et al.Application of functional high-temperature Daqu based on the composition of flavor compounds[J].China Brewing, 2017, 36(1):98-101.