食品掺假是人们一直以来比较关注的问题,乳及乳制品作为一种富含营养物质的日常消费品,成为了非常容易受到掺假影响的食品[1],在世界各国都存在掺假现象[2]。随着人们消费观念的提高,高品质、高营养的水牛乳和羊乳产品逐渐获得消费者的认可[3-4]。为谋求商业利益,不法商家用牛奶代替山羊奶或者水牛奶[5-7],进而引发经济利益、宗教习俗、过敏等问题[8-10]。因此,亟需鉴定乳制品的乳源种类,以保护消费者免受掺假产品的侵害。
目前鉴别乳源成分的方法主要为蛋白质和DNA序列分析。相比于蛋白质,DNA由于稳定性高更适合作为各类乳制品检测分析的目标物[11]。DNA的分析方法中,PCR因其特异性好、灵敏度高而被广泛应用[12]。普通PCR产物分离通常需要结合RFPL[13]、电泳[14]、测序等开管检测方法,容易产生污染。随着科学技术的发展,实时PCR被引入鉴定乳制品的真伪。实时PCR包括探针法和染料法[15],其中探针法价格贵且设计较为复杂,染料法虽然成本低、通用性好,但相对于探针法特异性不强。高分辨熔解曲线是一种在实时PCR扩增完成后监测扩增子熔解行为的分析方法,可以区分目标基因序列的微小差异,具有灵敏、快速、高通量的优点,常被应用在基因分型、突变扫描和甲基化检测领域[16-19]。近年来,PCR高分辨熔解曲线(high-resolution melting, HRM)广泛用于肉制品、海鲜、乳制品等食品的真实性鉴定和溯源分析[19-20]。在检测乳及乳制品掺假方面,特异性引物结合PCR-HRM方法被用于区分希腊菲达奶酪中牛、山羊、绵羊3种动物源性成分[6]、鉴别水牛乳制品中的牛乳成分[21]。基于特异性引物的PCR-HRM方法在鉴别乳源成分时,需要进行复杂的引物设计,在进行多重检测时需要添加多对引物,使得反应体系复杂、优化困难且浪费试剂。通用引物克服了特异性引物的不足,不需要复杂的引物设计及优化就能实现在同一反应体系中检测多个目标物。本文将通用引物与HRM相结合实现对山羊、绵羊、牛、水牛4个物种的鉴别检测,达到分析乳及乳制品中多个物种的目的,为维护消费者合法权益以及乳产品市场安全提供技术支撑。
1 材料与试剂
1.1 实验材料
生牛乳、生羊乳,西安市未央区奶牛场和市场;生水牛乳和绵羊乳,淘宝网,鲜奶于-20 ℃保存备用。用于验证特异性的非目标DNA(驴、马、兔子、鸡、鸭、鸽子、鱼、猪),四川华汉三创有限公司。不同品牌的牛、山羊、绵羊、水牛乳产品(液态乳、全脂奶粉、脱脂奶粉及配方奶粉)等样品分别购自西安市以及网上不同的超市。
1.2 试剂与仪器
磁珠法血液基因组DNA提取试剂盒、2×Taq PCR Master Mix、DNA marker,天根生化科技有限公司;通用引物,生工生物工程股份有限公司;EvaGreen,Biotium公司。MyGo Pro荧光定量PCR仪,IT-IS Life Science Ltd;MYSPIN 12微型离心机,Thermo Fisher Scientific;5424R高速冷冻离心机,Eppendorf 有限公司;Q6000超微量分光光度计,美国Quawell。
2 实验方法
2.1 提取DNA
将购买的液态乳和固体乳粉的脂肪去除后,按照磁珠法血液基因组DNA提取试剂盒操作步骤,提取所有样品的DNA。采用超微量核酸定量仪测定DNA的浓度和纯度,将DNA稀释至20 ng/μL,放于-4 ℃冰箱备用。
2.2 设计通用引物
根据美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)所公布的GU295658(山羊)、AF010406(绵羊)、AF492351(奶牛)和NC_006295(水牛)的基因序列,通过BioEdit对比其核苷酸全序列,依据物种的保守性和特异性,按照引物设计原则进行设计,保证通用引物可同时特异性扩增4个物种,且扩增产物间的序列具有差异性,能够通过高分辨率熔解曲线分析鉴别。
2.3 PCR-HRM反应体系建立
PCR反应体系组成为:5 μL 2×Tap PCR Master Mix,1 μL(20 ng/μL)DNA模板,0.5 μL的上游引物和下游引物,0.5 μL 20×EvaGreen染料,最后加水补充至10 μL。PCR反应程序为:94 ℃预变性5 min,94 ℃变性15 s,60 ℃退火20 s,72 ℃延伸30 s,进行30个循环;72 ℃再延伸5 min,扩增完成后立即对扩增产物进行高分辨熔解程序:65~95 ℃升温熔解,熔解梯度为0.05 ℃/s。在该温度范围熔解,既可以去除引物二聚体的干扰,也可以保证得到足够有效的熔解数据进行HRM分析。扩增产物采用琼脂糖凝胶电泳技术验证片段大小。
2.4 PCR-HRM可行性和特异性验证
将通用引物扩增的目标序列导入BioEdit对比4个物种的基因序列差异性,确认DNA序列具有可区分性。进一步将4条目标序列导入u-melt线上软件拟合高分辨熔解曲线,分析是否可以相互区分。将从牛、水牛、绵羊、山羊乳及其制品中提取的DNA作为模板,进行PCR-高分辨熔解实验,对比拟合曲线与实验所得曲线,确认实验的可行性。将购买的鸡、猪、马、兔和鱼等动物的DNA稀释至20 ng/μL分别加入PCR-HRM反应体系,观察有无扩增曲线,取扩增产物做琼脂糖凝胶电泳实验,确认通用引物的通用性及特异性。
2.5 PCR-HRM体系检测多元混合物
为了进一步验证所建立的方法的区分能力,将山羊、绵羊、牛、水牛4个物种的DNA按照排列组合方式预先混合,其中单组分4种、双组分6种、3组分4种和4组分1种共计15种混合物,对所有DNA混合物执行相同的PCR-HRM实验程序,考察该检测方法对混合物的区分能力。
2.6 PCR-HRM体系检测不同掺假比例样品
取20 ng/μL的山羊、水牛乳DNA于离心管中,分别将不同比例的牛乳DNA与其混合,比例依次为5%、10%、50%、80%、90%,取1.0 μL上述混合DNA作为模板进行PCR扩增,以5%的掺假为方法的可应用性的限度,并以单个物种DNA来源样本熔解曲线为参考,对不同掺假比例的样品进行检测,考察所建立的方法的实际应用性,以及对掺假成分的相对定量。
2.7 PCR-HRM体系检测实际样品
为了验证本方法的实用性及准确性,对市售的14种乳制品分别采用PCR-HRM和sanger测序进行鉴定,确认实际应用价值。
3 结果与分析
3.1 通用引物的选取设计
经筛选得到了一对通用引物,正向引物序列为5′-TTTGGTCCCAGCCTTCCTGTT-3′,反向引物序列为5′-CTTAGTCAAACTTTCGTTT-3′。利用BioEdit软件比对该通用引物对应的4个物种的完整扩增序列(以山羊的扩增序列为参照)。比对结果如图1所示,图中黑框范围内的是通用引物的碱基序列,圆点代表4个物种之间的相同碱基,可以很明显地看到,4个物种的碱基之间有差异,说明使用该通用引物,理论上可以区分出这4个不同的物种,其中序列差异最小的为水牛和牛,存在5%的序列差异,绵羊和奶牛的序列差异最大为13%,4个物种碱基序列平均差异约为10%。

图1 通用引物对应的4个物种的核苷酸序列差异对比
Fig.1 Sequence alignment of four species corresponding to universal primers
3.2 PCR-HRM方法可行性分析
利用u-Melt软件(https://dna-utah.org/umelt/quartz/)拟合通用引物对应的牛、水牛、山羊、绵羊基因序列的高分辨熔解曲线,拟合结果如图2-a所示。拟合得到4条独立特异的熔解曲线,曲线之间可以相互区分,证明理论上可以通过高分辨熔解曲线区分4个物种(图2-a)。进一步用提取的DNA做PCR-高分辨熔解验证实验,结果见图2-b,实验获得的熔解曲线与理论拟合的曲线类似,其中水牛熔解曲线出现在最右端,因为其靶序列含有较高的GC,相反的,牛的靶序列GC比例最小,因此牛的熔解曲线位于最左侧,通用引物对应的牛和水牛基因序列差异最小,但采用熔解曲线分析可以明显区分,且区分效果较好。实验结果表明4个物种对应的熔解曲线可以相互区分,因此验证了该鉴定方法用于山羊、绵羊、牛、水牛4个物种鉴别分析的可行性。图2-c中将该通用引物用于扩增其他物种以验证特异性,4个目标物种均可以有效扩增,此外扩增曲线显示鸽子、鱼、猪的Ct在27,而BLAST结果显示该对引物是无法扩增这3个物种,因此该扩增曲线可能由于样本提取时受污染所致。琼脂糖凝胶电泳图中只有牛、水牛、山羊、绵羊有明显条带,且符合预期长度,证明该通用引物对牛、水牛、山羊、绵羊具有通用性和对其他物种具有特异性。
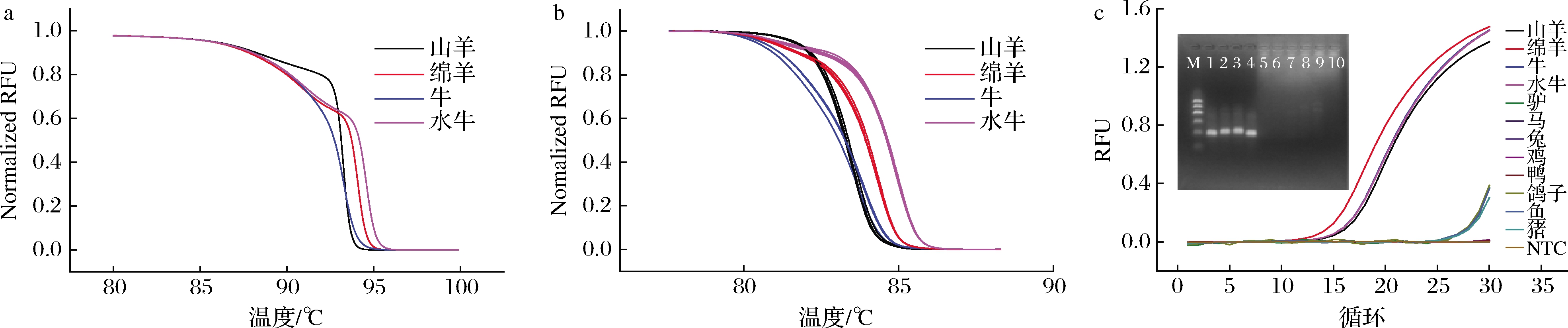
a-拟合熔解曲线;b-实验熔解曲线;c-扩增曲线及琼脂糖凝胶电泳图
图2 PCR-HRM可行性验证
Fig.2 Verification of PCR-HRM feasibility
注:图c中M是marker,1~10泳道分别对应于山羊、绵羊、牛、水牛、驴、马、兔子、鸡、鸭DNA
3.3 PCR-HRM体系检测多元混合物结果
采用通用引物扩增单组分(山羊、绵羊、牛、水牛)、二元混合物(山羊+牛、绵羊+牛、水牛+牛、山羊+水牛、山羊+绵羊、绵羊+水牛)、三元混合物(山羊+绵羊+牛、山羊+绵羊+水牛、绵羊+牛+水牛、山羊+牛+水牛)、四元混合物(山羊+绵羊+牛+水牛),并对以上15种样本进行高分辨熔解,熔解结果见图3。归一化曲线图中大部分样本的曲线形状独特,可以相互区分,有几个样本的曲线发生了重叠,差异性很小。根据WITTWER等[18]的研究显示,差异化曲线的区分效果更好,分析15种不同样本对应的差异化曲线,区分能力优于归一化熔解曲线,与文献报道一致。在图3-c、图3-d的二维和三维主成分分析图中,同一样本的3个平行被划为1个聚类,含有不同物种的样本分布在不同位置,混合物可以依据聚类位置相互区分。结果证明该方法可以对15种目标物进行鉴别,区分能力强大,适合多元混合物的掺假检测。
3.4 PCR-HRM体系检测不同掺假比例样品
将山羊和牛的DNA按照不同比例混合,每个混合物做3个平行,进行PCR-HRM检测,分析由熔解曲线得到的主成分分析(principal component analysis,PCA),结果如图4-a所示,每个样本的3个平行由于主成分相同被划为1个聚类,最左和最右边分别是100%牛和100%山羊,随着牛DNA在样本中占比增加,聚类逐渐靠近100%牛,说明掺假比例和聚类在PCA图中的PC1有一定的关系,因此以PCA图中各聚类PC1数值的平均值为纵坐标,混合比例为横坐标做标准曲线,线性回归模型相关系数R2=0.995。用相同的方法做牛和水牛DNA的掺假实验,得到相似结果,线性回归模型相关系数R2=0.998。根据文献报道,掺入5%的外源物即可通过掺假获得经济利益[22],因此以5%为最低检测标准,本实验证明可以检测到占比为5%的牛DNA,确认PCR-HRM可以实现对山羊乳DNA、水牛乳DNA中掺假牛乳DNA的相对定量。

a-归一化熔解曲线;b-差异化熔解曲线;c-二维PCA分析图;d-三维PCA分析图
图3 PCR-HRM多元混合物检测结果
Fig.3 Multivariate mixtures detection results by PCR-HRM

a-山羊和牛掺比PCA图;b-山羊和牛掺比标准曲线图;c-水牛和牛掺比PCA图;d-水牛和牛掺比标准曲线图
图4 山羊、水牛和牛不同掺假比例样品的检测结果
Fig.4 Results of pure goat and buffalo milk DNA mixed with bovine in progression proportions
3.5 市售样品的应用性检测
因为牛奶价格较低且含有过敏蛋白,所以采用建立的检测方法测定了山羊和水牛乳及其制品中牛乳的掺假情况。对市售的14种乳制品进行PCR-HRM实验,分别设置牛、山羊、水牛标准品对照,依据不同品牌乳制品在PCA图中的聚类分布位置确认其主成分。检测结果如表1所示,本文建立的方法一共检测了12个主成分为牛和山羊乳的产品,其中有2个标签标示为纯山羊乳,却检测到牛的DNA,其余产品成分均与标签标识成分一致。此外还对2种主成分为牛和水牛乳的产品进行检测,结果显示样本13、14都含有牛和水牛2种成分,样本13符合标签,样本14不符合。通过对以上14种市售乳制品的检测鉴定,确认有3种乳制品的实测成分与标签不符,在纯山羊乳、纯水牛乳制品中掺入了牛乳成分。对样品进行Sanger测序,其中3组样品的测序结果如图5所示。样品1、4、7分别为牛羊混合、纯牛、纯山羊,测序结果(扩增序列77~102 bp)显示,纯山羊、纯牛的测序峰都是单峰,没有杂峰出现,而牛、山羊混合的样品测序峰比较杂乱,并且出现了重叠峰,与本文建立方法的检测结果一致。实验结果证明基于通用引物的PCR-HRM具有实际应用能力,可用于乳及乳制品的真实性鉴定。
表1 通用引物PCR-HRM反应体系对市售牛、 山羊、水牛乳制品的检测应用
Table 1 Application of PCR-HRM reaction system in the detection of bovine, goat, water buffalo milk products
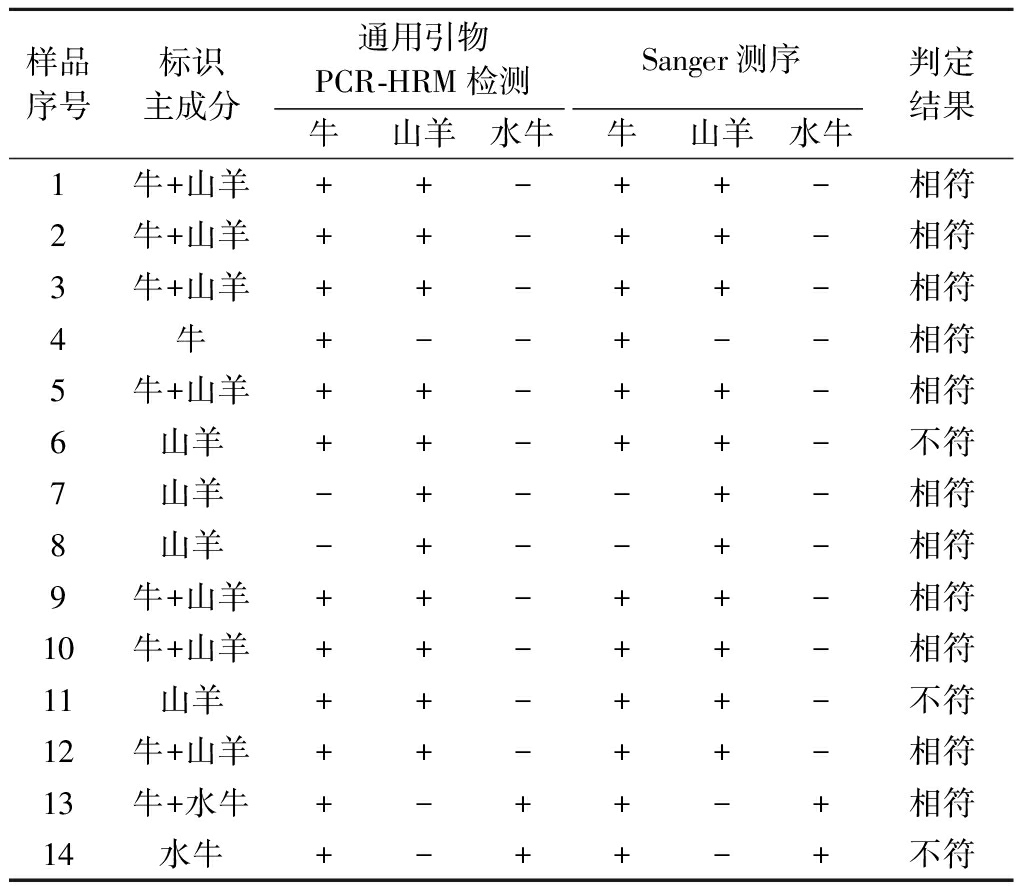
样品序号标识主成分通用引物PCR-HRM检测Sanger测序牛山羊水牛牛山羊水牛判定结果1牛+山羊++-++-相符2牛+山羊++-++-相符3牛+山羊++-++-相符4牛+--+--相符5牛+山羊++-++-相符6山羊++-++-不符7山羊-+--+-相符8山羊-+--+-相符9牛+山羊++-++-相符10牛+山羊++-++-相符11山羊++-++-不符12牛+山羊++-++-相符13牛+水牛+-++-+相符14水牛+-++-+不符
注:+代表检测出该目标DNA,-代表未检测出目标DNA
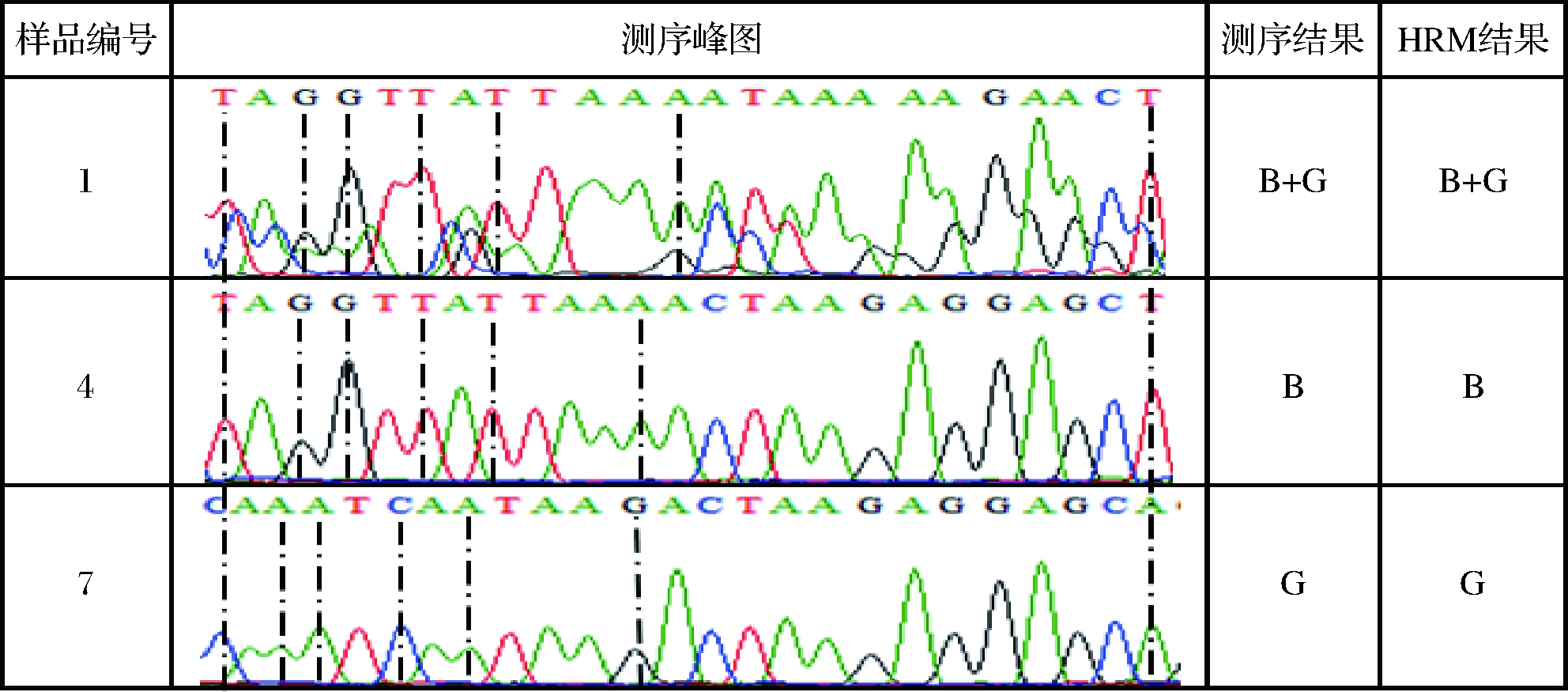
图5 部分市售乳制品测序结果
Fig.5 Sanger sequencing results of some commercial dairy products
4 结论
本研究针对牛、水牛、山羊、绵羊的基因序列设计了一对通用引物,建立了基于通用引物的PCR-HRM检测方法。该通用引物的PCR产物借助HRM分析可以成功区分以上4个物种,且不能扩增其他物种,具特异性。同时该方法可鉴别多元混合物,具备分析复杂、多目标样本的能力,可以检出掺假比例为5%的牛DNA,符合市场检测需求。将该方法用于市售乳及其制品的鉴别,检测结果与测序结果一致,检测结果可靠。与普通熔解曲线相比,高分辨熔解曲线因采用饱和染料具备更高的分辨率和区分能力,在分析同源性高的牛、水牛亲缘物种时,也能显示良好的区分效果。此外,PCR-HRM方法不用开管操作,减小了检测环境及待测样本受PCR产物污染的可能性,并且操作简单,经短时培训后即可进行实验操作和数据分析,更重要的是基于通用引物进行PCR,可在1次反应中同步检测4个物种、检测通量高、检测成本低,适用于各类企业、厂家、研究所等。采用相同的方法优化筛选相应引物,所建立的方法也可用于牦牛奶、骆驼奶等高附加值乳制品的掺假分析,从而为食品中多种动物源性成分的掺假检测提供技术支撑。
[1] 王文强,文豪,张文众,等.基于美国药典委EMA数据库的全球经济利益驱动型掺假和食品欺诈的分析[J].食品安全质量检测学报,2019,10(3):804-810.
WANG W Q,WEN H,ZHANG W Z,et al.Analysis of global economically motivated adulteration and food fraud based on the EMA database of the United States Pharmacopoeia[J].Journal of Food Safety & Quality,2019,10(3):804-810.
[2] GONÇALVES J,PEREIRA F,AMORIM A,et al.New method for the simultaneous identification of cow,sheep,goat,and water buffalo in dairy products by analysis of short species-specific mitochondrial DNA targets[J].Journal of Agricultural and Food Chemistry,2012,60(42):10 480-10 485.
[3] 林波, 李玲,唐艳,等.水牛乳及其乳制品中掺入牛属动物乳源的检测方法研究进展[J].中国乳品工业,2012,40(6):47-49;57.
LIN B,LI L,TANG Y,et al.Research progress in detecting methods of adulteration of bovine milk into buffalo milk and its products[J].China Dairy Industry,2012,40(6):47-49;57.
[4] 张昊阳, 严林,党高平,等.羊乳制品掺假检测技术研究进展[J].中国乳业,2019(8):132-136.
ZHANG H Y,YAN L,DANG G P,et al.Research progress on detection technology of adulteration of goat milk products[J].China Dairy,2019(8):132-136.
[5] AGRIMONTI C,PIRONDINI A,MARMIROLI M,et al.A quadruplex PCR (qxPCR) assay for adulteration in dairy products[J].Food Chemistry,2015,187:58-64.
[6] GANOPOULOS I,SAKARIDIS I,ARGIRIOU A,et al.A novel closed-tube method based on high resolution melting (HRM) analysis for authenticity testing and quantitative detection in Greek PDO Feta cheese[J].Food Chemistry,2013,141(2):835-840.
[7] KIM M J,KIM H Y.Direct duplex real-time loop mediated isothermal amplification assay for the simultaneous detection of cow and goat species origin of milk and yogurt products for field use[J].Food Chemistry,2018,246:26-31.
[8] 程晋霞, 曾静,马丹,等.食品中牛奶过敏原成分LAMP检测方法的建立[J].食品安全质量检测学报,2014,5(4):1 039-1 044.
CHENG J X,ZENG J,MA D,et al.Establishment of loop-mediated isothermal amplification method for detection of milk allergen in foods[J].Journal of Food Safety & Quality,2014,5(4):1 039-1 044.
[9] 党慧杰, 刘振民,郑远荣.牛乳主要过敏原及其检测技术研究进展[J].食品安全质量检测学报,2020,11(3):765-770.
DANG H J,LIU Z M,ZHENG Y R.Research progress of cow milk allergens and their detection techniques[J].Journal of Food Safety & Quality,2020,11(3):765-770.
[10] ABBAS O,ZADRAVEC M,BAETEN V,et al.Analytical methods used for the authentication of food of animal origin[J].Food Chemistry,2018,246:6-17.
[11] 宋宏新, 刘建兰,徐秦峰.羊乳及其制品中掺入牛乳成分的双重PCR检测[J].陕西科技大学学报,2018,36(1):34-38;44.
SONG H X,LIU J L,XU Q F.A duplex real-time PCR for the detection of bovine milk in goat's milk[J].Journal of Shaanxi University of Science & Technology,2018,36(1):34-38;44.
[12] LO Y T,SHAW P C.DNA-based techniques for authentication of processed food and food supplements[J].Food Chemistry,2018,240:767-774.
[13] ABDEL-RAHMAN S M,AHMED M M M.Rapid and sensitive identification of buffalo's,cattle's and sheep’s milk using species-specific PCR and PCR-RFLP techniques[J].Food Control,2007,18(10):1 246-1 249.
[14] 刘鸣畅, 侯艳梅,杨艳歌,等.毛细管电泳技术检测羊乳婴幼儿配方粉中的牛乳成分[J].中国食品学报,2019,19(5):270-275.
LIU M C,HOU Y M,YANG Y G,et al.Detection of cow milk components in goat milk from infant formula using capillary gel electrophoresis method[J].Journal of Chinese Institute of Food Science and Technology,2019,19(5):270-275.
[15] SAFDAR M,JUNEJO Y.Development and validation of fast duplex real-time PCR assays based on SYBER Green florescence for detection of bovine and poultry origins in feedstuffs[J].Food Chemistry,2015,173:660-664.
[16] GUNDRY C N,VANDERSTEEN J G,REED G H,et al.Amplicon melting analysis with labeled primers:A closed-tube method for differentiating homozygotes and heterozygotes[J].Clinical Chemistry,2003,49(3):396-406.
[17] LIEW M,PRYOR R,PALAIS R,et al.Genotyping of single-nucleotide polymorphisms by high-resolution melting of small amplicons[J].Clinical Chemistry,2004,50(7):1 156-1 164.
[18] WITTWER C T,REED G H,GUNDRY C N,et al.High-resolution genotyping by amplicon melting analysis using LCGreen[J].Clinical Chemistry,2003,49(6):853-860.
[19] 徐秦峰, 马西亚,闵红卫,等.高分辨熔解技术在食品真实溯源检测中的应用[J].陕西科技大学学报,2018,36(6):42-50.
XU Q F,MA X Y,MIN H W,et al.Applications of high resolution melting technique in food authentication and food traceability[J].Journal of Shaanxi University of Science & Technology,2018,36(6):42-50.
[20] DRUML B,CICHNA-MARKL M.High resolution melting (HRM) analysis of DNA-its role and potential in food analysis[J].Food Chemistry,2014,158:245-254.
[21] SAKARIDIS I,GANOPOULOS I,ARGIRIOU A,et al.High resolution melting analysis for quantitative detection of bovine milk in pure water buffalo mozzarella and other buffalo dairy products[J].International Dairy Journal,2013,28(1):32-35.
[22] RUSSO R,REGA C,CHAMBERY A.Rapid detection of water buffalo ricotta adulteration or contamination by matrix-assisted laser desorption/ionisation time-of-flight mass spectrometry[J].Rapid Communications in Mass Spectrometry,2016,30(4):497-503.