大米,是稻谷经过清理、砻谷、碾米、成品整理等工序后制成的成品[1]。当今世界的大米加工工艺已经比较成熟[2],制品的品质是关系消费者需求的主要因素,目前已将研究重点转移至稻米的深加工和综合利用上。为满足消费者对食品提出的安全、营养、保健的等方面的要求,需不断开发新的大米产品[3]。大米经过清洗、浸泡、磨浆、发酵等工序后,可加工成大米乳酸饮料、米发糕、米酒等制品。米浆发酵作为中国传统米制品加工的重要工序,经过此加工过程,微生物群落组成复杂多样,且易滋生杂菌,导致发酵不易控制,从而出现质量问题及安全隐患[4]。现有研究多集中在发酵米制品的加工工艺及食味品质,对米浆发酵过程中微生物群落变化的研究较少。研究米浆发酵体系的微生物群落结构对改善发酵米制品品质及风味具有重要的作用[5]。
高通量测序是20世纪70年代由FREDERICK发明的双脱氧链终止法核酸测序技术。近年来被广泛应用于食醋、白酒、泡菜、豆豉等发酵过程中微生物群落的测定[6-7],该技术能快速、准确地对发酵食品的微生物组成及其丰度变化进行多方位的研究,在食品微生物快速检测中有着重要的应用前景[8-9]。本研究基于高通量测序技术,探究传统自然发酵过程中不同发酵阶段的微生物生长变化规律,为后续复合菌剂的制备和米制品质量控制提供理论基础。
1 材料与方法
1.1 材料与试剂
新米浆、老浆,乐山阙记食品有限公司;Qubit3.0 DNA检测试剂盒(Q10212),Life;E.A.N.ATM Mag-Bind Soil DNA Kit(M5635-02),美国OMEGA公司;2×Hieff® Robust PCR Master Mix(10105ES03)、Hieff NGSTM DNA Selection Beads(2601ES56),翌圣(Yeasen)生物科技(上海)股份有限公司。
1.2 仪器与设备
Pico-21台式离心机,Thermo Fisher;GL-88B旋涡混合器,海门市其林贝尔仪器制造有限公司;TND03-H-H混匀型干式恒温器,深圳拓能达科技有限公司;DYY-6C电泳仪电源、DYCZ-21电泳槽,北京市六一仪器厂;FR-1000凝胶成像系统,上海复日科技有限公司;Q32866 Qubit®3.0荧光计,Invitrogen;ETC 811 PCR仪,北京东胜创新生物科技有限公司;Research plus 0.5~10 μL移液器,Eppendorf。
1.3 实验方法
1.3.1 样品预处理
参照传统自然发酵法,将100 g新米浆中接入10 g老浆[m(老浆)∶m(新米浆)=1∶10],密封后在16~20 ℃发酵40 h,分别在0、8、16、24、32、40 h取样10 g,并将细菌分别标号为(A0-bac)~(A5-bac),真菌分别标号为(A0-fun)~(A5-fun)。
参照文献[10-11]的方法,将每个时间点的米浆样品各取5 g,加入20 mL磷酸缓冲盐溶液(PBS)(0.137 mol/L NaCl,0.002 7 mol/L KCl,0.01 mol/L Na2HPO4,0.002 mol/L KH2PO4,pH 7.0)悬浮,并加入0.5 g玻璃珠后充分涡旋5 min。在4 ℃离心机中2 000 r/min离心5 min,收集上清液。将沉淀用PBS洗涤2次并收集上清液。将所有上清液于4 ℃,10 000 r/min离心10 min弃去上清液,收集沉淀。收集的沉淀用1 mL的PBS重悬,转入1.5 mL的EP管中,-20 ℃冻存备用。
1.3.2 总DNA提取
参考李荣源等[10]的方法,对样品进行处理后,使用DNA抽提试剂盒(OMEGA,美国)进行DNA提取,步骤严格参照DNA抽提试剂盒说明书,并用1%琼脂糖凝胶电泳检测提取质量。
1.3.3 PCR扩增及高通量测序
参考高乾坤等[12]的方法,以检验合格的细菌基因组为模板,引物针对16S rRNA基因V3/V4区合成特异引物,Nobar_341F(CCTACGGGNGGCWGCAG),Nobar_805R(GACTACHVGGGTATCTAATCC)。以提取的总真菌的基因组为模板,引物针对16S rRNA基因ITS1-ITS2区合成特异引物,ITS1:(CTTGGTCATTTAGAGGAAGTAA),ITS2:(GCTGCGTTCTTCATCGATGC)。根据PCR扩增产物浓度进行等浓度混样,使用2%琼脂糖凝胶电泳检测效果。将合格的样品送至生工生物工程(上海)股份有限公司进行Illumina MiSeq高通量测序。
1.4 数据处理
利用Usearch软件将最终优化数据在97%相似度水平下进行运算分类单位(operational taxonomic unit,OTU)聚类分析;基于OTU聚类分析结果,利用Mothur软件对α多样性指数进行分析;基于分类学信息,利用统计学方法对各分类水平进行群落结构的统计分析;利用方差分解将数据差异反映在二维坐标上绘制主成分分析图。作图软件使用R软件和origin 8.0。实验重复3次。
2 结果与分析
2.1 细菌多样性分析
2.1.1 细菌OTU聚类分析
为研究各样本间的微生物组成,以97%的相似度水平对所有样本的有效序列进行OTUs聚类分析并进行物种注释。16S rRNA的V4区间经过滤和双端拼接后共得到383 814条有效序列,聚类得到447个OTU。Venn图可以显示所有样本共有OTU和特有OTU的数目,为直观显示样品OTU组成数目的相似性及重叠性,对米浆发酵过程各时间点的细菌的OTU进行统计,结果如图1所示。米浆整个发酵过程中细菌共有的OTU数为16个。发酵初期样品中特有OTU数为15个,随着发酵时间的延长,在32 h时特有OTU数目为1个,其他时间点均为0个,证明在环境中获得的细菌数量少。将每个OTU与细菌分类学数据库对比后得到其物种注释,共2个门,8个属水平的细菌。结果表明,自然发酵过程与老浆细菌种类相似,证明发酵过程感染的细菌量少,与孟燕华[13]的研究结果一致。
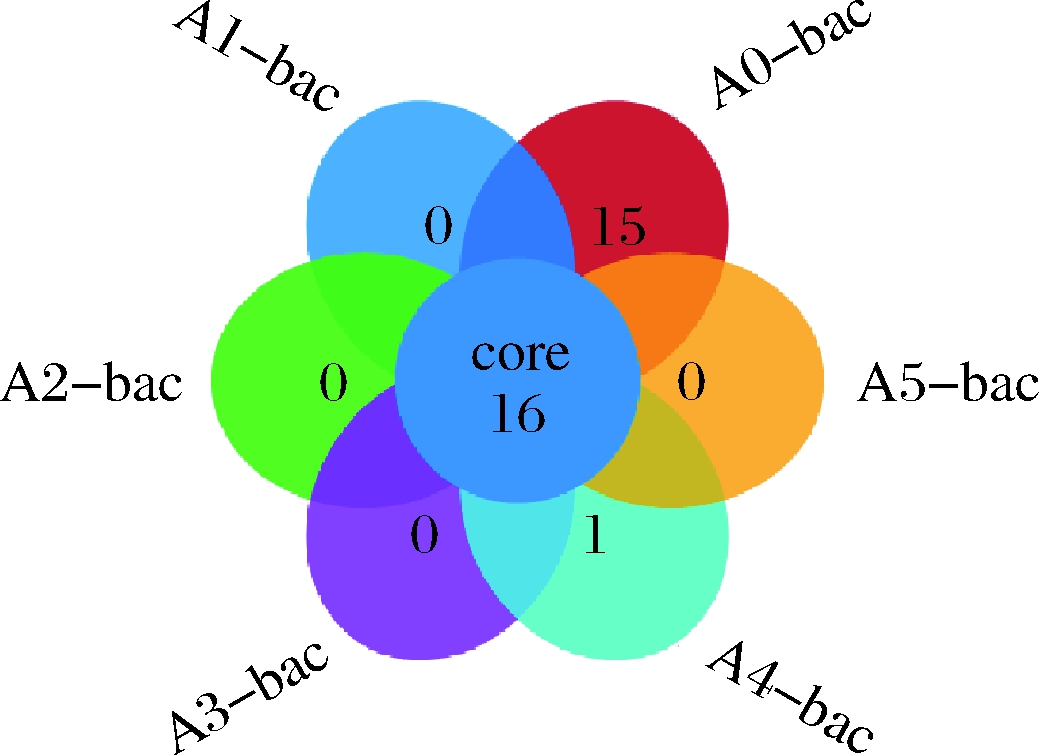
图1 不同发酵阶段细菌OTU数
Fig.1 Number of bacterial OTU in different stages of fermentation
2.1.2 细菌α多样性分析
α多样性分析能反映微生物群落的丰度和多样性,利用统计学分析指数(P<0.05)估计群落的物种丰度和多样性。Chao指数体现群落丰度,其值越大表明群落丰富度越大。OTUs体现物种的丰富度,ACE指数用于估计群落中OTU数目的指数。Shannon和Simpson指数都能体现群落多样性,Shannon值越大,Simpson值越小,群落多样性越高。Shannoneven是反映物种个体数目在群落中分配的均匀程度的指数。由表1可知,在整个发酵过程中,OTUs、Chao和Ace指数在0~16 h时增加,然后减少至32 h后再增加,物种丰富度和多样性在16 h最高,32 h最低。覆盖率均在99.94%~99.99%,证明样本中的细菌几乎都被检测到,该数据可靠。
表1 样品中细菌α多样性指数测定结果
Table 1 α-Diversity of bacteria in samples
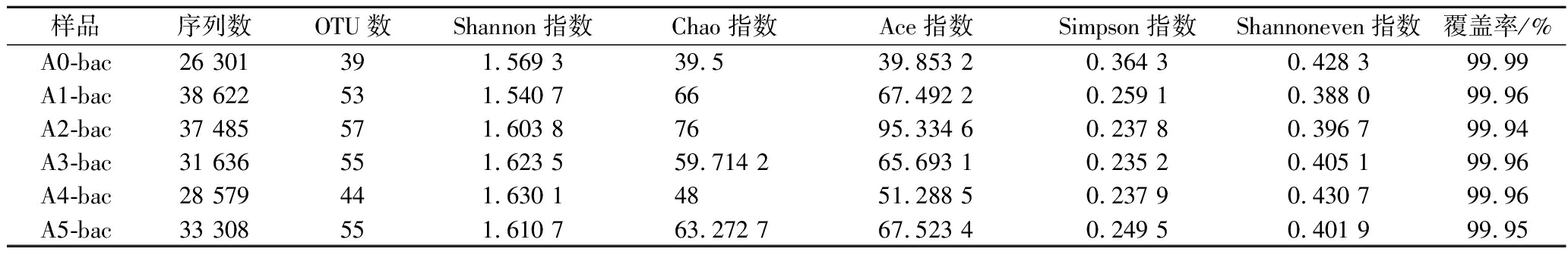
样品序列数OTU数Shannon指数Chao指数Ace指数Simpson指数Shannoneven指数覆盖率/%A0-bac26 301391.569 339.5 39.853 20.364 30.428 399.99A1-bac38 622531.540 76667.492 20.259 10.388 099.96A2-bac37 485571.603 87695.334 60.237 80.396 799.94A3-bac31 636551.623 559.714 265.693 10.235 20.405 199.96A4-bac28 579441.630 14851.288 50.237 90.430 799.96A5-bac33 308551.610 763.272 767.523 40.249 50.401 999.95
2.1.3 细菌物种组成分析
2.1.3.1 门水平米浆发酵过程群落结构分析
米浆发酵体系中共检测出2个门的细菌(图2),在发酵初期厚壁菌门(Firmicutes)占全部细菌群落的89.22%,螺旋菌门(Spirochaetota)占10.33%,其他物种占0.46%。随着发酵时间的延长,厚壁菌门的丰度增加至99.40%,螺旋菌门的丰度降低至0.05%。5组样品中厚壁菌门的丰度均最高,可见厚壁菌门是米浆发酵体系的优势微生物。厚壁菌门是半固态发酵食品中的重要微生物,如乳制品、酒类、酱油等[14],这与本研究结果一致。

图2 门水平细菌相对丰度
Fig.2 Relative abundance of bacteria at phylum level
2.1.3.2 属水平米浆发酵过程群落结构分析
米浆发酵体系共检测出8个属的细菌,其他物种合并为Other(图3)。由图3可知,属水平上发酵体系的细菌主要有乳杆菌属,短螺旋体属(Brachyspira)、芽孢杆菌属(Bacillus)和需盐芽孢杆菌属(Virgibacillus)。发酵初期,乳杆菌属的相对丰度为84.46%,短螺旋体属的丰度为10.33%,芽孢杆菌属的丰度为5.05%。随着发酵时间的延长,乳杆菌属的丰度逐渐增加至97.09%,短螺旋体属的丰度逐渐降低至0.02%,芽孢杆菌属的丰度逐渐降低至0.59%。芽孢杆菌是广泛存在于环境中的腐生菌,多属于需氧或兼性厌氧菌。杨磊等[15]在浓香型酒醅中分离得到1株地衣芽孢杆菌。邓岳等[16]发现在传统工艺酿造的酱油中分离得到的芽孢杆菌是酸类、酮类、含硫类、吡嗪类等低阈值化合物合成的重要微生物。大多数乳酸菌能在低氧发酵过程中利用糖类产生乳酸结合乙醇后形成乳酸乙酯,可为发酵体系提供特殊的风味物质并降低发酵体系的pH值,所产生的乳酸菌素能抑制杂菌生长。故乳酸菌在保证产品风味和品质方面起着极其重要的作用,这与李美伦等[17]的研究结果一致。
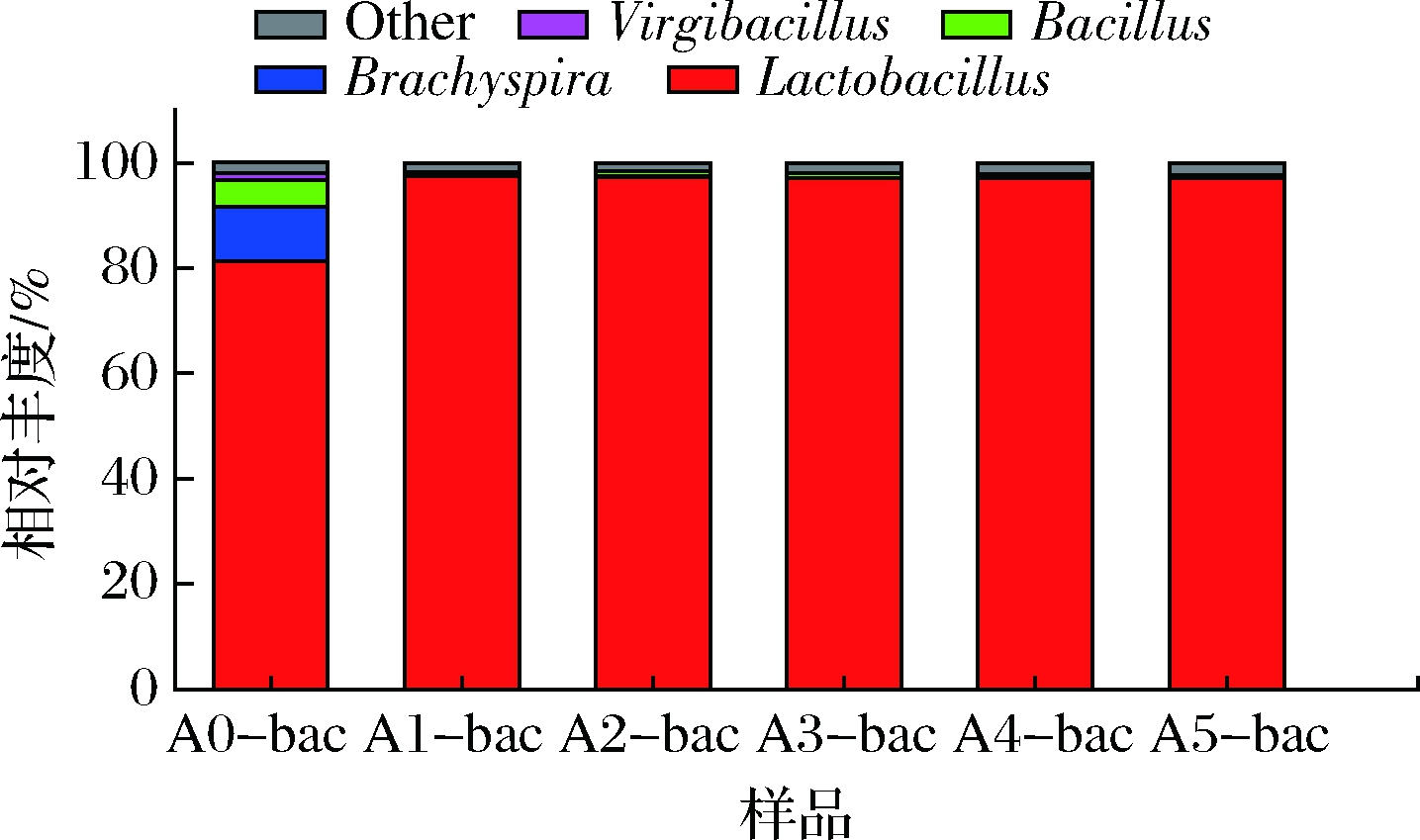
图3 属水平细菌相对丰度
Fig.3 Relative abundance of bacteria at the genus level
2.1.3.3 属水平米浆发酵过程微生物群落主成分分析
主成分分析可找出数据中最主要的元素和结构。X轴和Y轴代表选定的主成分轴,图中不同颜色的点代表不同样本来源,各点间的距离代表相似程度,越近相似度越高。由图4可知,各样本间有明显差异。A0-bac在坐标轴左侧上方且与其他5个样本的距离较远,表明刚接入老浆时细菌多样性与后续发酵过程差异较大。A1-bac至A5-bac这5个样品均在坐标轴右侧且距离相对较近,表明环境中的细菌变化较小,这与高赟[18]的研究结果相似。
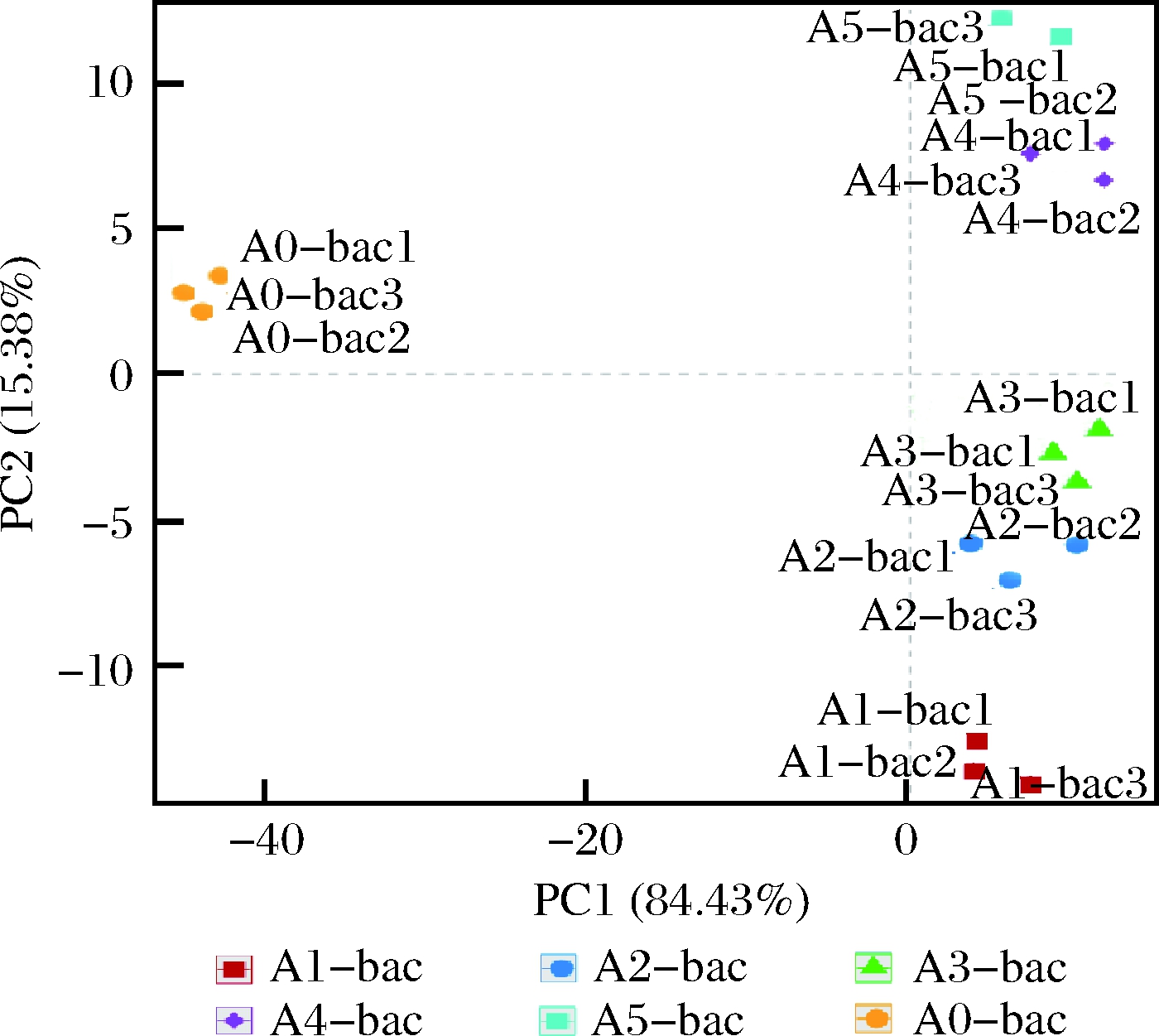
图4 属水平细菌主成分分析
Fig.4 PCA of bacteria at genus level
2.2 真菌多样性分析
2.2.1 真菌OTU聚类分析
真菌的ITS1经过滤和双端拼接后共得到433 198条有效序列,聚类得到870个OTU。对米浆发酵过程各时间点的细菌OTU进行统计,结果如图5所示。真菌的共有OTU数目37个。发酵初期特有OTU数为27个,在第8 h减少至8个,在第16 h又增加至13个,发酵终点时特有OTU数为8个。证明从环境中获得一定数量的真菌。将每个OTU与细菌分类学数据库对比后得到其物种注释,共2个门,9个属水平的真菌。结果表明,在整个发酵过程中,由于乳酸菌产酸导致部分真菌不适应低pH环境,故生长被抑制,这与文献[19-20]的研究结果一致。
2.2.2 真菌α多样性分
真菌α多样性如表2所示。在米浆发酵全过程40 h中,OTUs、Chao和Ace指数在发酵初期开始增加至第8 h,然后降低并在发酵后期略有升高。Shannon和Simpson指数分别在40 h时达到最大和最小,说明在发酵终点真菌多样性最高,即此时真菌丰度最高,这与古明亮等[21]的研究结果一致。
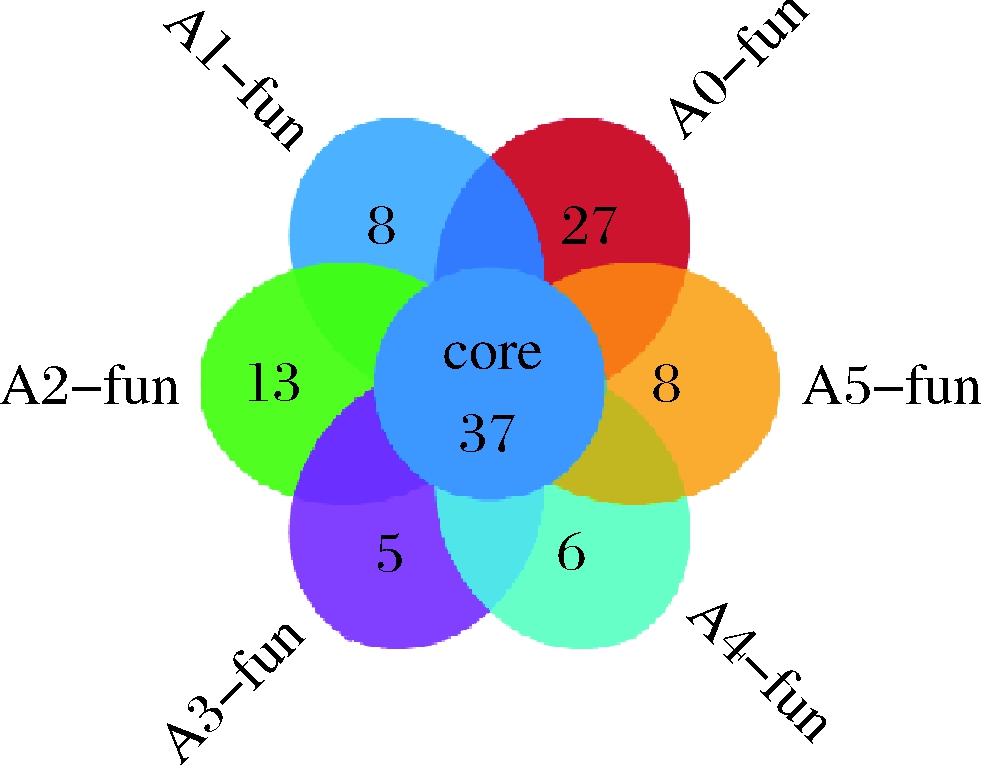
图5 不同发酵阶段真菌OTU数
Fig.5 Number of fungal OTU in different stages of fermentation
表2 样品真菌α多样性指数测定结果
Table 2 α-Diversity of fungal in samples
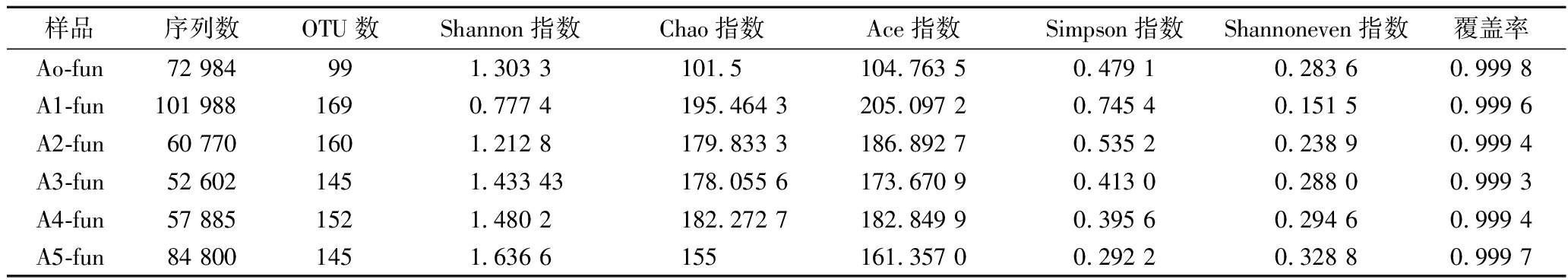
样品序列数OTU数Shannon指数Chao指数Ace指数Simpson指数Shannoneven指数覆盖率Ao-fun72 984991.303 3101.5104.763 50.479 10.283 60.999 8A1-fun101 9881690.777 4195.464 3205.097 20.745 40.151 50.999 6A2-fun60 7701601.212 8179.833 3186.892 70.535 20.238 90.999 4A3-fun52 6021451.433 43178.055 6173.670 90.413 00.288 00.999 3A4-fun57 8851521.480 2182.272 7182.849 90.395 60.294 60.999 4A5-fun84 8001451.636 6 155161.357 00.292 20.328 80.999 7
2.2.3 真菌物种组成分析
2.2.3.1 门水平米浆发酵过程群落结构分析
米浆发酵体系中共检测出2个门的真菌,分别为子囊菌门(Ascomycota)和担子菌门(Basidiomycota)。子囊菌门在发酵体系占主导地位,在发酵初期其相对丰度占真菌群落的98.48%,随着发酵时间的延长其含量几乎无变化,证明其在发酵过程中起重要的作用,为体系的主要优势菌。担子菌门的丰度在整个发酵过程中都低于1.5%,作为真菌中最高等的门类,多数为大型真菌,故在米浆发酵体系的作用不大且不易生长,此结果与张文齐等[22]的研究结果相似。
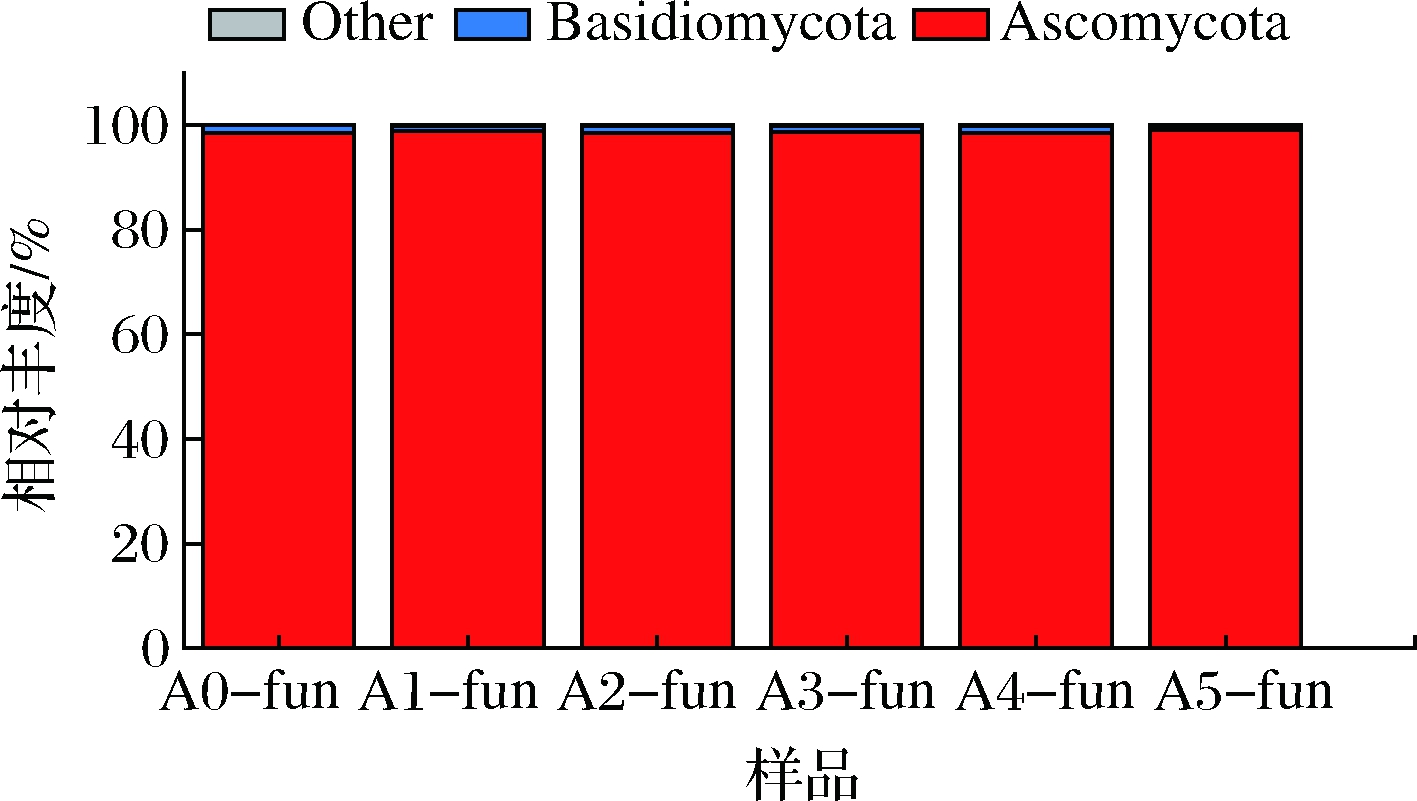
图6 门水平真菌相对丰度
Fig.6 Relative abundance of fungi at the phylum level
2.2.3.2 属水平米浆发酵过程群落结构分析
米浆发酵体系中共检测出9个属的真菌。由图7可知,米浆发酵体系主要有哈萨克斯坦酵母属(Kazachstania)、双足囊菌属(Dipodascus)、酿酒酵母菌属(Saccharomyces)、假丝酵母属(Candida)、伊萨酵母属(Issatchenkia)、木克拉酵母属(Mrakia)、曲霉属(Aspergillus)、丝孢酵母属(Trichosporon)。哈萨克斯坦酵母属在发酵初期丰度为73.49%,8 h丰度达到86.86%然后开始减小,至发酵终点丰度为46.87%,为发酵体系的主要优势菌。哈萨克斯坦酵母属参与发酵体系乙酸乙酯、β-苯乙醇、异戊醇等香气物质的产生,并能利用糖类产生乙醇和CO2,使米浆浆体变得多孔蓬松并获得特殊风味,提升了产品的口感和风味[23],这与王雪山[24]的研究结果一致。
2.3.3.3 属水平米浆发酵过程群落主成分分析
由图8可知,各样本间有明显差异。A0-fun在坐标轴左上侧且与其他5个样本距离较远,表明刚接入老浆时真菌多样性较大,A3-fun和A4-fun的距离较近,表明在发酵24~32 h时真菌的多样性达到相对稳定,其多样性差异不明显。A1-fun至A5-fun这5个样品的差异较明显,表明环境中的真菌变化较大,这与方冠宇[25]的研究结果相似。

图7 属水平真菌相对丰度
Fig.7 Relative abundance of fungi at genus level
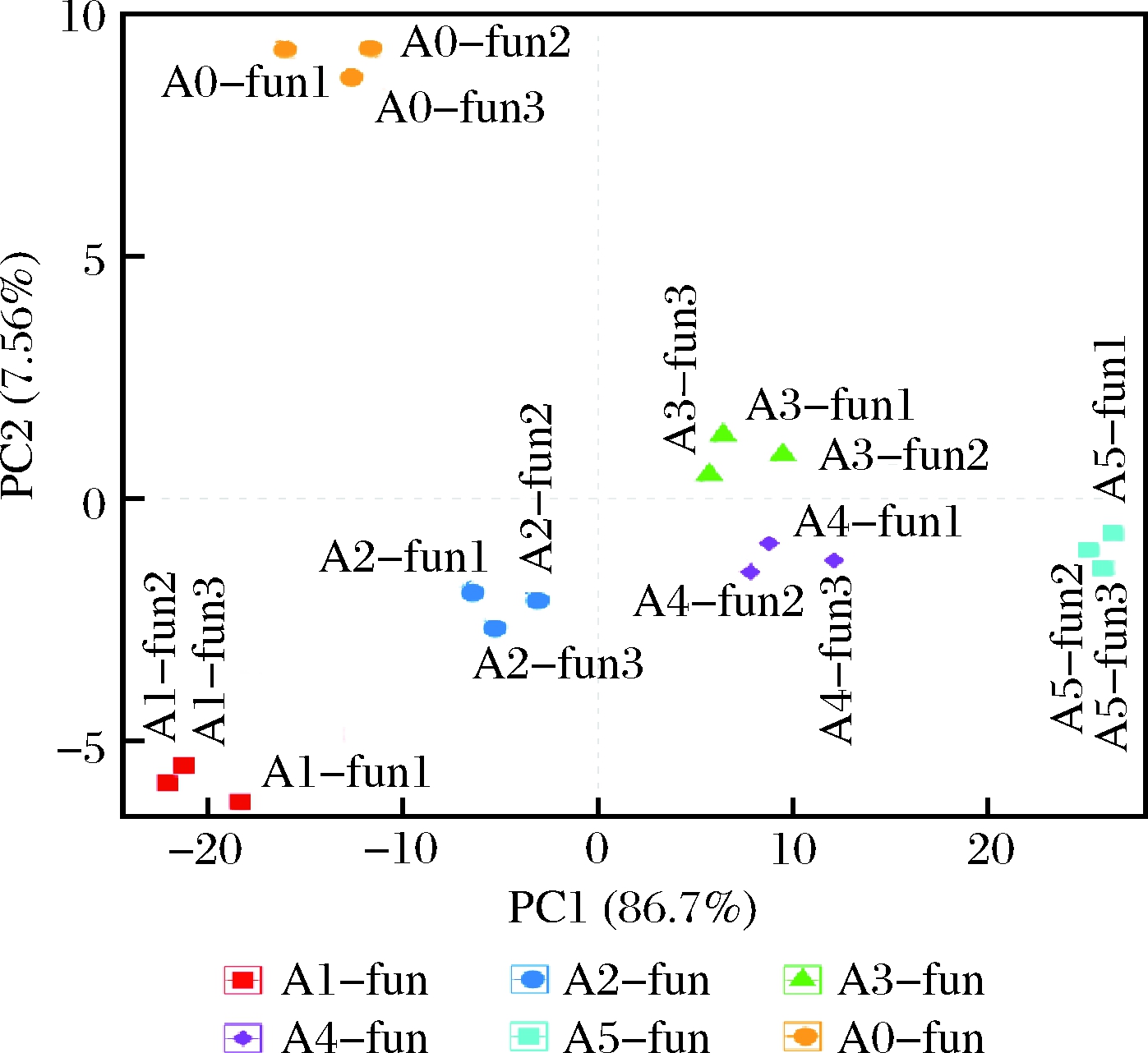
图8 属水平真菌主成分分析
Fig.8 PCA of fungi at genus level
3 结论
本研究采用高通量测序技术对米浆不同发酵时间微生物多样性进行分析。研究发现,在门水平上,厚壁菌门和子囊菌门为发酵体系主要优势菌门。在属水平上,乳杆菌属、哈萨克斯坦酵母属、双足囊菌属和酿酒酵母属为发酵体系主要优势菌属,这些优势菌主要影响着发酵体系特殊风味物质的生成和发酵米制品的品质。高通量测序技术能分析各时间点的微生物组成及丰度变化规律,为发酵米制品的生产提供了一定的理论依据。
[1] 安红周,陈会会,尹文婷,等.加工精度对大米食用品质及风味的影响[J].中国粮油学报,2021,36(1):1-7.
AN H Z,CHEN H H,YIN W T,et al.Effect of milling degree on the edible quality and flavor of rice[J].Journal of the Chinese Cereals and Oils Association,,2021,36(1):1-7.
[2] 刘厚清, 河野元信.优良食味大米的生产、加工技术[J].中国粮油学报,2017,32(9):182-187.
LIU H Q,KAWANO M.The production & processing technology on good eating taste of rice[J].Journal of the Chinese Cereals and Oils Association,2017,32(9):182-187.
[3] 刘厚清, 景梦瑶,周涛.白米的加工精度对食味及经济性的影响[J].粮食加工,2018,43(5):36-41.
LIU H Q,JING M Y,ZHOU T.The effect of the processing precision of white rice on the taste and economy[J].Grain Processing,2018,43(5):36-41.
[4] 阳盈盈. 自然发酵米发糕微生物分析及其优势菌的应用[D].长沙:湖南农业大学,2014.
YANG Y Y.Analysis of microbe in naturally fermented rice cake and application of its dominant strains[D].Changsha:Hunan Agricultural University,2014.
[5] 席慧婷, 江平屿,陈星光,等.复合菌发酵米粉的制作工艺优化及其香气成分[J].食品工业科技,2019,40(11):204-210;217.
XI H T,JIANG P Y,CHEN X G.Optimization of production process of fermented rice flour with compound bacteria and its aroma compound bacteria and its aroma components[J].Science and Technology of Food Industry,2019,40(11):204-210;217.
[6] 向凡舒, 张婷,董蕴,等.糯米酸细菌多样性解析及其表型和基因功能预测[J].中国粮油学报,2021,36(1):14-20.
XIANG F S,ZHANG T,DONG Y,et al.Bacterial divercity analysis and prediction of phenotype and gene function of fermented glutinous rice[J].Journal of the Chinese Cereals and Oils Association,2021,36(1):14-20.
[7] 倪慧, 葛东颖,杨甜甜,等.基于MiSeq高通量测序技术奶疙瘩细菌群落结构研究[J].中国乳品工业,2020,48(12):16-19.
NI H,GE D Y,YANG T T,et al.Study on bacterial diversity of kurut by MiSeq high-throughput sequencing technology[J].China Dairy Industry,2020,48(12):16-19.
[8] 张明珠, 吴学凤,穆冬冬,等.基于高通量测序的窖泥原核微生物群落结构及其理化因子相关性分析[J/OL].食品科学,2021.https://kns.cnki.net/kcms/detail/11.2206.TS.20201213.1312.002.html.
ZHANG M Z,WU X F,MU D D,et al.Correlation analysis of pit mud prokaryotic microbial community structure and its physicochemical indexes based on high throughput sequencing[J/OL].Food Science,2021.https://kns.cnki.net/kcms/detail/11.2206.TS.20201213.1312.002.html.
[9] 文鹤, 李浩,付薇,等.基于高通量测序分析快速发酵曲霉型豆豉可培养细菌多样性[J/OL].基因组学与应用生物学,2021.https://kns.cnki.net/kcms/detail/45.1369.Q.20201207.1517.006.html.
WEN H,LI H,FU W,et al.High-throughput sequencing analysis of rapid fermentation of aspergillus-type douchi cultivable Bacterial diversity[J/OL].Genomics and Applied Biology,2021.https://kns.cnki.net/kcms/detail/45.1369.Q.20201207.1517.006.html.
[10] 李荣源, 卢红梅,秦兴,等.高通量测序分析赤水晒醋各生产阶段微生物群落结构变化[J].食品科学,2020,41(24):78-86.
LI R,LU H M,QIN X,et al.High-throughput sequencing study on changes in microbial community structure in different production stages of Chishui sun-dried vinegar[J].Food Science,2020,41(24):78-86.
[11] LI S,LI P,LIU X,et al.Bacterial dynamics and metabolite changes in solid-state acetic acid fermentation of Shanxi aged vinegar[J].Applied Microbiology and Biotechnology,2016,100(10):4 395-4 411.
[12] 高乾坤, 焦琳舒,杜贺超,等.高通量测序分析不同产地带鱼冷藏时微生物群落多样性[J].食品科学,2018,39(18):127-132.
GAO Q K,JIAO L S,DU H C,et al.High-throughput sequencing analysis of microbial community diversity of hairtail from different waters during chilled storage[J].Food Science,2018,39(18):127-132
[13] 孟燕华. 利用高通量测序技术分析传统食醋酿造过程中的细菌多样性[D].太原:山西大学,2019.
MENG Y H.Bactreial diversity during brewing processes of traditional viengar by high-throughput sequencing[D].Taiyuan:Shanxi University,2019.
[14] 杜瑞, 王柏辉,罗玉龙,等.应用Illumina MiSeq测序技术比较传统发酵乳、肉食品中细菌多样性[J].中国食品学报,2021,21(2):269-277.
DU R,WANG B H,LUO Y L,et.al.Comparison of bacterial diversity in traditional fermented dairy and meat products by Illumina MiSeq sequencing[J].Journal of Chinese Institute of Food Science and Technology,2021,21(2):269-277.
[15] 杨磊, 陈良.浓香型酒醅中一株耐热地衣芽孢杆菌产淀粉酶和蛋白酶培养条件的优化[J].酿酒,2021,48(2):49-53.
YANG L,CHEN L.Optimization of conditions for amylase and protease production of a thermotolerant bacillus licheniformis strain[J].Liquor Making,2021,48(2):49-53.
[16] 邓岳, 杨阳,梁丽静,等.传统工艺酿造酱油微生物多样性与风味研究[J].中国调味品,2021,46(3):104-108.
DENG Y,YANG Y,LIANG L J,et al.Study on microbial diversity and flavor of soy sauce brewed by traditional technology[J].China Condiment,2021,46(3):104-108.
[17] 李美伦, 姜萌艺,龚川杰,等.乳酸菌、酵母菌的筛选鉴定及其在米发糕中的应用[J].食品与机械,2019,35(5):14-20.
LI M L,JIANG M Y,GONG C J,et al.Screening and identification of lactic acid bacteria and yeasts and its application in fermented rice cake[J].Food & Machinery,2019,35(5):14-20.
[18] 高赟. 维生素C二步发酵营养组学的研究[D].天津:天津大学,2012.
GAO Y.Nutriomics study in the second step of vintamin C fermentation[D].Tianjin:Tianjin University,2012.
[19] 刘贞, 刘小翠,赵思明,等.发酵米浆中高发酵性能酵母菌和乳酸菌的筛选和鉴定[J].食品科学,2010,31(7):232-235.
LIU Z,LIU X C,ZHAO S M,et al.Screening and identification of strains of yeast and Lactobacillus with high fermentation performance from fermented rice slurry[J].Food Science,2010,31(7):232-235.
[20] 王丹丹. 小米发酵物菌种分离与在小米饮料中的应用[D].天津:天津科技大学,2018.
WANG D D.Isolation of Millet fermentation strains and its application in millet beverage[D].Tianjin:Tianjin University of Science and Technology,2018.
[21] 古明亮, 刘群,祝建梅.丹棱冻粑发酵米浆中乳酸菌和酵母菌分析[J].粮食与食品工业,2017,24(5):57-59.
GU M L,LIU Q,ZHU J M.Analysis of lactic bacteria and yeasts in the fermenting rice slurry of Danling Dongba[J].Cereal & Food Industry,2017,24(5):57-59.
[22] 张文齐,麻和平,刘彩云,等.高通量测序分析不同采集保存温度下甘南牧区牦牛酸奶真菌多样性[J].食品工业科技,2021, 42(13):163-169.
ZHANG W Q,MA H P,LIU C Y,et al.Analysis of fungal diversity of yak yogurt collected and preserved at different temperatures in Gannan pastoral area based on high-throughput sequencing[J].Science and Technology of Food Industry, 2021, 42(13):163-169.
[23] CHIKOWSKI R,LARSSON K H,GIBERTONI T B.Taxonomic novelties in Trechispora (Trechisporales,Basidiomycota) from Brazil[J].Mycological Progress,2020,19(12):1 403-1 414.
[24] 王雪山. 不同环境清香类型白酒发酵微生物种群结构比较及溯源解析[D].无锡:江南大学,2018.
WANG X S.Microbial community structure and microbial source tracking of Chinese light-flavor liquor fermentation in different environments[D].Wuxi:Jiangnan University,2018.
[25] 方冠宇. 浙江玫瑰醋搅拌工艺及发酵过程中微生物与风味物质相关性研究[D].杭州:浙江工商大学,2019.
FANG G Y.Research on the stirring technology and the correlation between microorganism and flavor substance in Zhejiang rosy viengar fermentation process[D].Hangzhou:Zhejiang Gongshang University,2019.