熊果苷是一类天然的糖苷类化合物,由葡萄糖和对苯二酚(hydroquinone,HQ)通过糖苷键连接而成,自然界中主要存在于植物果皮及树叶中。熊果苷具有抗菌[1-2]、抗氧化[3-4]等作用,因而在医药领域具有较好的应用。此外,熊果苷作为亮肤剂被广泛用于各类化妆品中[5]。由于葡萄糖和HQ之间可形成α-和 β-两种构型的糖苷键,因此熊果苷也被分为α-熊 果苷和β-熊果苷两种。天然存在的熊果苷均是β-型, α-熊果苷可通过有机合成和微生物转化[6]的方法获得。相较于β-熊果苷,α-熊果苷在美白功效及安全性方面都更具优势[7-8]。约60年前,日本学者通过化学法成功合成了α-熊果苷[9],但该方法存在产物立体选择性差、反应条件剧烈、产生大量副产物等的不足[10],因此,更符合绿色环保发展理念的生物转化法合成α-熊果苷成了目前研究的热点。α-熊果苷的生物转化是在一类葡萄糖基转移酶的作用下,将葡萄糖基转移至HQ的酚羟基上,并特异性形成α-糖 苷键[11-12]。目前研究发现的7种微生物来源的葡萄糖基转移酶均以HQ作为受体底物,但不同种类的葡萄糖基转移酶需要不同的供体底物,如蔗糖、麦芽糖、淀粉等[12-13]。
蔗糖磷酸化酶(sucrose phosphorlase,SPase,EC 2.4.1.7)是第一个被研究用于生物转化合成α-熊果苷的酶,该酶以蔗糖和HQ作为供体和受体底物,特异性合成α-熊果苷。在催化过程中,蔗糖并不需要使用二磷酸腺苷(adenosine diphosphate,ADP)、二磷酸尿苷(uridine diphosphate,UDP)这类昂贵的物质作为前体[14],由此推测该酶可利用蔗糖中糖苷键断裂时释放的能量来形成α-熊果苷中的糖苷键;此外,该酶的催化活力也不依赖于辅因子[15-16]。KITAO等[17]的研究表明SPase能够将α-葡萄糖基转移到23种酚类及其相关化合物中,且该酶对于α-熊果苷的合成表现出很高的转糖基化效率,摩尔转移率达到了65%。变异链球菌(Streptococcus mutans)来源的蔗糖磷酸化酶(SmSP)稳定性较好,对蔗糖具有最高的底物亲和力。
本文在课题组前期研究的基础上,以枯草芽孢杆菌(Bacillus subtilis) WB600和B.subtilis WB800为出发菌株,对S.mutans UA159来源的蔗糖磷酸化酶(SmSPI336L,在本文中表示为Smut)进行异源表达,通过采用整合表达、优化核糖体结合位点(ribosome binding site, RBS)序列和优化全细胞催化条件的策略,获得了具有高效催化潜力的重组B.subtilis BS8-P43-RBSopt-3Smut,α-熊果苷的产量可达到 119.44 g/L, 底物HQ的摩尔转化率为96.56%。
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
研究使用的菌株、质粒见表1。大肠杆菌(Escherichia coli) JM109用于重组DNA实验,B.subtilis WB600、B.subtilis WB800为本实验的出发菌株。菌株、质粒全部保藏在本实验室。
表1 本研究所用菌株和质粒
Table 1 Strains and plasmids used in this study
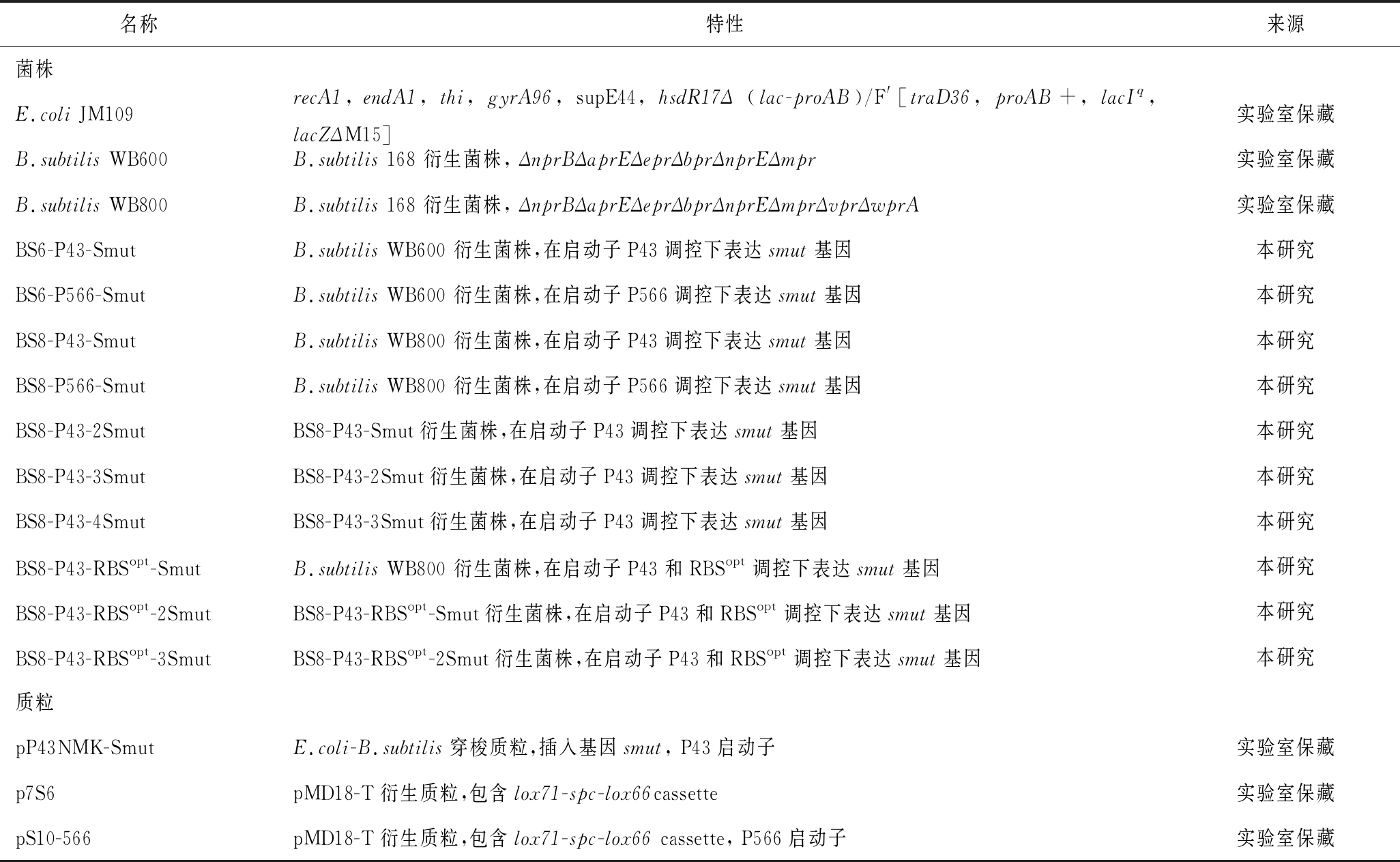
名称特性来源菌株E.coli JM109recA1, endA1, thi, gyrA96, supE44, hsdR17Δ (lac-proAB)/F'[traD36, proAB+, lacIq, lacZΔM15]实验室保藏B.subtilis WB600B.subtilis 168 衍生菌株, ΔnprBΔaprEΔeprΔbprΔnprEΔmpr实验室保藏B.subtilis WB800B.subtilis 168 衍生菌株, ΔnprBΔaprEΔeprΔbprΔnprEΔmprΔvprΔwprA实验室保藏BS6-P43-SmutB.subtilis WB600 衍生菌株,在启动子P43调控下表达smut基因本研究BS6-P566-SmutB.subtilis WB600 衍生菌株,在启动子P566调控下表达smut基因本研究BS8-P43-SmutB.subtilis WB800 衍生菌株,在启动子P43调控下表达smut基因本研究BS8-P566-SmutB.subtilis WB800 衍生菌株,在启动子P566调控下表达smut基因本研究BS8-P43-2SmutBS8-P43-Smut衍生菌株,在启动子P43调控下表达smut基因本研究BS8-P43-3SmutBS8-P43-2Smut衍生菌株,在启动子P43调控下表达smut基因本研究BS8-P43-4SmutBS8-P43-3Smut衍生菌株,在启动子P43调控下表达smut基因本研究BS8-P43-RBSopt-SmutB.subtilis WB800 衍生菌株,在启动子P43和RBSopt调控下表达smut基因本研究BS8-P43-RBSopt-2SmutBS8-P43-RBSopt-Smut衍生菌株,在启动子P43和RBSopt调控下表达smut基因本研究BS8-P43-RBSopt-3SmutBS8-P43-RBSopt-2Smut衍生菌株,在启动子P43和RBSopt调控下表达smut基因本研究质粒pP43NMK-SmutE.coli-B.subtilis穿梭质粒,插入基因smut, P43启动子实验室保藏p7S6pMD18-T衍生质粒,包含lox71-spc-lox66cassette实验室保藏pS10-566pMD18-T衍生质粒,包含lox71-spc-lox66 cassette, P566启动子实验室保藏
1.1.2 培养基与试剂
种子培养基(g/L):蛋白胨10,酵母粉5,NaCl 10。121 ℃灭菌20 min。
发酵培养基(g/L):蛋白胨12,酵母粉24,KH2PO4 2.31,K2HPO4 12.54,甘油 4 mL/L。115 ℃,20 min。
培养基中添加相应的抗生素(μg/mL):氨苄青霉素100、卡那霉素50、壮观霉素100。
PB缓冲液(20 mmol/L,pH 7.0):分别配制0.2 mol/L 的NaHPO4和0.2 mol/L的Na2HPO4,从中各取39 mL和61 mL,加入蒸馏水定容至1 L,即获得20 mmol/L,pH 7.0的PB缓冲液。
HPLC流动相:10 mmol/L稀磷酸与甲醇混合,体积比为8∶2。
Prime STAR(max)DNA聚合酶、DNA marker,TaKaRa公司;Taq DNA聚合酶,南京诺唯赞生物科技有限公司;分子操作相关试剂盒,上海生工生物工程有限公司;α-熊果苷的标准品,Sigma-Aldrich;其他常规试剂均为分析纯。
1.2 实验方法
1.2.1 Smut表达框的构建及整合
使用引物yqhB-rh-1F/R,yqhB-rh-4F/R,从B.subtilis WB600基因组上分别扩增待整合位点的上下游同源臂各1 000 bp;使用引物yqhB-rh-2F/R,以质粒p7S6为模板,扩增lox71-spc-lox66序列;使用引物yqhB-rh-3F/R,以质粒pP43NMK-Smut为模板,扩增带有P43启动子的目的基因片段P43-smut。
使用引物yqhB-rh-zong-F/R,通过融合PCR技术将上下游同源臂、lox71-spc-lox66序列和P43-smut序列进行融合,获得待整合表达框yqhB-P43-smut。所用引物见表2。
将表达框yqhB-P43-smut片段分别转化B.subtilis WB600和B.subtilis WB800的感受态细胞,通过壮观霉素抗性标记来筛选阳性转化子BS6-P43-Smut和BS8-P43-Smut。
1.2.2 Smut表达框中启动子的替换及表达框整合
使用引物yqhB-rh-2F和pS10-2R,以质粒pS10-566为模板,扩增带有P566启动子序列的lox71-spc-lox66-P566片段;使用引物smut-3F和yqhB-rh-3R,以质粒pP43NMK-Smut为模板,扩增目的基因smut。
使用引物yqhB-rh-zong-F/R,通过融合PCR技术将1.2.1中的上下游同源臂、lox71-spc-lox66-P566片段和smut片段进行融合,获得待整合表达框yqhB-P566-smut。所用引物见表2。
将表达框yqhB-P566-smut片段分别转化B.subtilis WB600和B.subtilis WB800的感受态细胞,通过壮观霉素抗性标记来筛选阳性转化子BS6-P566-Smut和BS8-P566-Smut。
1.2.3 P43-Smut的多位点整合
使用引物yitR-rh-1F/R和yitR-rh-4F/R, amyE-rh-1F/R和amyE-rh-4F/R, ymzB-rh-1F/R和ymzB-rh-4F/R分别扩增得到待整合位点yitR, amyE, ymzB的上下游同源臂。使用引物yitR-rh-2F/3R, amyE-rh-2F/3R, ymzB-rh-2F/3R,以1.2.1中构建完成的表达框yqhB-P43-smut为模板,分别扩增相应的片段。使用引物yitR-rh-zong-F/R, amyE-rh-zong-F/R, ymzB-rh-zong-F/R,通过融合PCR技术得到待整合表达框yitR-P43-smut, amyE-P43-smut, ymzB-P43-smut。所用引物见表2。
将3个表达框片段依次转化BS8-P43-Smut的感受态细胞,通过壮观霉素抗性标记来筛选阳性转化子BS8-P43-2Smut, BS8-P43-3Smut, BS8-P43-4Smut。由于多重抗性标记会影响筛选效率,因此在每一轮基因整合之前,都使用cre-lox系统将壮观霉素抗性标记消除。
1.2.4 smut基因的RBS序列优化及多位点整合
使用RBS预测网站“RBS calculator”(https://salislab.net/software/predict_rbs_calculator)设计smut基因的最优RBS序列RBSopt。
使用引物yqhB-rh-1F和smut-RBS-opt-R,smut-RBS-opt-F和yqhB-rh-4R,以1.2.1中构建完成的表达框yqhB-P43-smut为模板,分别扩增相应的片段。使用引物yqhB-rh-zong-F/R,通过融合PCR技术将这2个片段进行融合,得到将原始RBS序列替换为RBSopt序列的待整合表达框yqhB-P43-RBSopt-smut。所用引物见表2。将表达框yqhB-P43-RBSopt-smut转化B.subtilis WB800的感受态细胞,筛选得到重组菌株BS8-P43-RBSopt-Smut。
表2 本研究所用引物
Table 2 Primers used in this study

引物名称碱基序列(5'-3')yqhB-rh-1FGATCAATTTTGGATCGCCGC yqhB-rh-1RGTACCGAGCTCGAATTCCATCCTATAGAGTCCGCCTTTTTCyqhB-rh-2FGCGGACTCTATAGGATGGAATTCGAGCTCGGTACCCyqhB-rh-2RGTATTTTTTGAGAAGATCGTCGACGATTCTACCGTTCGyqhB-rh-3FGGTAGAATCGTCGACGATCTTCTCAAAAAATACTACCTGTCCyqhB-rh-3RGAAAAAGCGGGGCACGTTAT-TCGAAAGATATGGTCTGATCATTCTCyqhB-rh-4FGACCATATCTTTCGAATAACGTGCCCCGCTTTTTCyqhB-rh-4RCGTCTCTCGTATGAAGGCTCyqhB-rh-zong-FCGATTGAAAATTCTACGGCAACGyqhB-rh-zong-RCCGCAATTTCAGCTGTTTCCsmut-RBS-opt-FGCTGCTAGATACCAAAAGGAGGTTTTTTTTATGCC-AATCATTAACAAGACGATGCsmut-RBS-opt-RAAAAACCTCCTTTTGGTATCTAGCAGCTAC-ATCGTCTATAATGGTACCGC
续表2

引物名称碱基序列(5'-3')pS10-2FTGTGAGGAGGATTTGTTTGCGAATTCGAGCTCGGTACCpS10-2RCTTGTTAATGATTGGCATTTTTATCAC-CTCCTTTCTGATAATATAACATAsmut-3FAGAAAGGAGGTGATAAAAATGCCAATCATTAACAAGACGsmut-3RCCAGAAAGTCGTTACTCGGTTTAT-TCGAAAGATATGGTCTGATCATTCyitR-rh-1FGCTGACGTCATCCTTAAAAACCyitR-rh-1RTACCGAGCTCGAATTCCTACTCTACTAATTTTTT-ATTGACATAAATAATAACCCCyitR-rh-4FCCATATCTTTCGAATAAAAAAAGAAGGGCAGAGCGyitR-rh-4RCCATTGTTTGCCGACATGCamyE-rh-1FCTCATCTCTTCAGGTTCTGGC amyE-rh-1RGTACCGAGCTCGAATTCGGTCCTGCCAGAACCAAATGamyE-rh-4FCCATATCTTTCGAATAAGGGCAAGGCTAGACGGamyE-rh-4RAATGTTTTTAAGGACGCCGGymzB-rh-1FGATGGACAAACTTGGACAACCCymzB-rh-1RTACCGAGCTCGAATTCGGCTGTCAGCTGTGCTG ymzB-rh-4FCCATATCTTTCGAATAAGCGTAGCGCCTTTGAGCymzB-rh-4RGGCAGATCCGATGCCTATAAAAATCyitR-rh-2FAAAATTAGTAGAGTAGGAATTCGAGCTCGGTACCCyitR-rh-3RCTCTGCCCTTCTTTTTTTAT-TCGAAAGATATGGTCTGATCATTCTC amyE-rh-2FTTGGTTCTGGCAGGACCGAATTCGAGCTCGGTACCCamyE-rh-3RCCGTCTAGCCTTGCCCTTAT-TCGAAAGATATGGTCTGATCATTCTCymzB-rh-2FCAGCACAGCTGACAGCCGAATTCGAGCTCGGTACCCGymzB-rh-3RCTCAAAGGCGCTACGCTTAT-TCGAAAGATATGGTCTGATCATTCTCyitR-rh-zong-FTGGCTATTTCAGAGGGAGCGyitR-rh-zong-RAATCACGGCCAAATGTCTTGCamyE-rh-zong-FTTTATTCCAATCCTGGCGCCamyE-rh-zong-RAAGTTCAGCTCAGTGATACCTGymzB-rh-zong-FGAGTTTGCGATTGGACAAACAGymzB-rh-zong-RTGCCATCATAACGACAGGTG
使用1.2.3中的方法构建整合表达框yitR-P43-RBSopt-smut和ymzB-P43-RBSopt-smut,然后依次转化BS8-P43-RBSopt-Smut的感受态细胞,筛选得到重组菌株BS8-P43-RBSopt-2Smut和BS8-P43-RBSopt-3Smut。
1.2.5 α-熊果苷的全细胞催化
取重组菌株的单菌落接种于20 mL种子培养基中,37 ℃,220 r/min振荡培养10 h。然后以1% (体积分数)的转接比例将种子液转接至30 mL的发酵培养基中,37 ℃,220 r/min振荡培养11 h。将发酵液低温低速离心15 min以收集细胞,用pH 7.0,20 mmol/L 的PB缓冲液将细胞洗涤2次。将细胞加入含有50 g/L HQ、310.9 g/L蔗糖的催化反应体系中,该催化反应是在pH 7.0,20 mmol/L的PB缓冲液中进行的。催化容器为250 mL摇瓶,反应体积为20 mL。催化条件为30 ℃,220 r/min振荡反应22~34 h。
1.2.6 α-熊果苷的HPLC检测
使用HPLC的方法检测α-熊果苷的浓度。将Agilent 1200 HPLC配备的紫外检测器设置波长281 nm,使用Agilent SB-Aq色谱柱(4.6 mm×250 mm,5 μm)。流动相为10 mmol/L的稀磷酸和甲醇,体积比为 8∶2, 流速为0.6 mL/min。控制柱温为35 ℃,进样量为10 μL。 样品处理方法:将全细胞催化液以最高转速在4 ℃离心10 min后,取上清液用超纯水进行稀释,再使用0.22 μm滤膜除去稀释液中的杂质。
2 结果与分析
2.1 蔗糖磷酸化酶Smut的整合表达及启动子替换
为使Smut在B.subtilis中稳定表达,我们选择B.subtilis WB600[18]和B.subtilis WB800[19]作为出发菌株,将P43-smut表达框分别整合至上述出发菌株基因组的yqhB位点。同时,为使Smut高效表达,我们使用在B.subtilis中能够高效启动淀粉酶基因bla转录的P566启动子[19]替换P43启动子。分别得到了BS6-P43-Smut、BS6-P566-Smut、BS8-P43-Smut和BS8-P566-Smut这4株重组菌株,如图1-a、图1-b所示。然后将这4株重组菌株进行全细胞催化合成α-熊果苷,以验证Smut在不同启动子的调控下及在不同的宿主中表达时α-熊果苷的合成情况,如图1-c所示,在B.subtilis WB800中使用P43启动子时,α-熊 果苷的产量最高,为29.14 g/L,底物HQ的摩尔转化率为23.56%;而在B.subtilis WB800中使用强启动子P566时,α-熊果苷的产量最低,为10.10 g/L,表明在B.subtilis WB800中,使用P43启动子调控Smut的表达对于全细胞催化合成α-熊果苷的效果更好。而在B.subtilis WB600中使用P43和P566启动子对于α-熊果苷的产量没有明显差别,分别为11.71、12.55 g/L。上述结果表明,以B.subtilis WB800作为表达宿主,使用P43启动子调控Smut的表达有利于获得较高产量的α-熊果苷。
在重组菌株BS8-P43-Smut的基础上,我们进一步增加Smut的拷贝数:将构建的2拷贝P43-smut整合表达框转化BS8-P43-Smut,获得菌株BS8-P43-2Smut;将3拷贝P43-smut整合表达框转化BS8-P43-2Smut,获得菌株BS8-P43-3Smut;将4拷贝P43-smut整合表达框转化BS8-P43-3Smut,获得菌株BS8-P43-4Smut。然后将上述4株重组菌株进行全细胞催化,α-熊果苷的产量如图1-d所示,α-熊果苷产量与smut的拷贝数的增加呈正相关;当整合4个拷贝smut时,α-熊果苷的产量为54.05 g/L,底物HQ的摩尔转化率为43.70%,表明增加smut基因的拷贝数能在一定程度上提高α-熊果苷的产量。
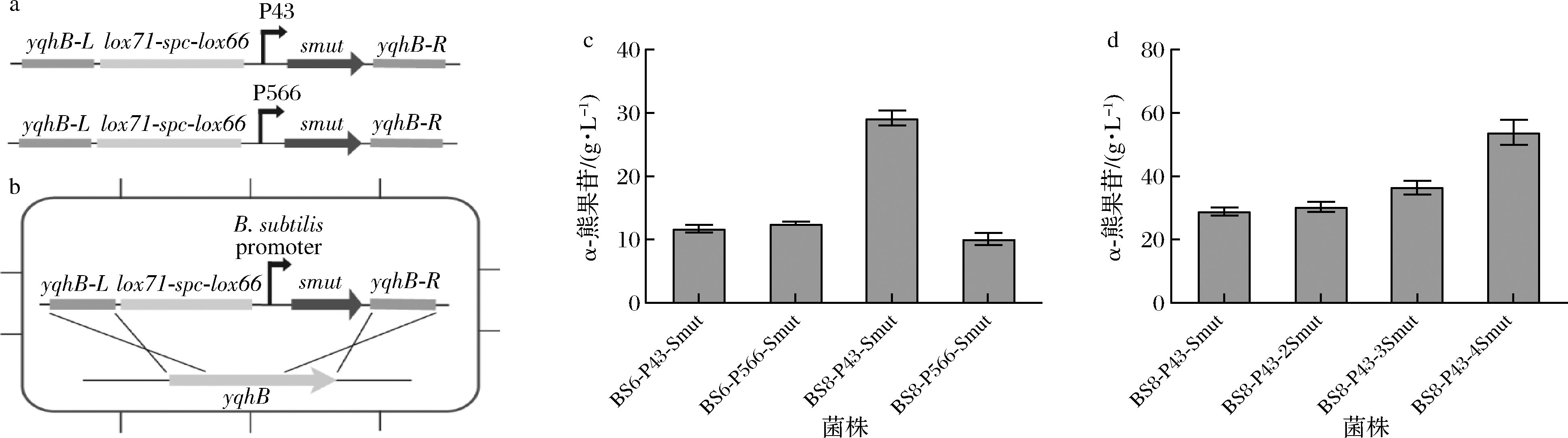
a-蔗糖磷酸化酶基因smut的整合表达框;b-蔗糖磷酸化酶基因smut在B.subtilis中同源重组的示意图;c-4株重组菌株全细胞催化22 h时合成α-熊果苷的能力;d-分别整合1、2、3、4拷贝的蔗糖磷酸化酶基因smut的重组菌株在全细胞催化22 h时合成α-熊果苷的能力
图1 蔗糖磷酸化酶Smut整合表达合成α-熊果苷的情况
Fig.1 Integrated expression of Smut to produce α-arbutin
2.2 蔗糖磷酸化酶的RBS序列优化
为进一步提高α-熊果苷的产量,我们对P43-smut表达框中RBS序列进行优化[18-19]。使用RBS预测网站“RBS calculator”设计蔗糖磷酸化酶基因smut的最优RBS序列,获得了理论最优RBS序列RBSopt:5′-ACGATGTAGCTGCTAGATACCAAAAGGA-GGTTTTTTTT-3′[20-21]。预测结果如图2-a所示。
将原始的RBS序列替换成RBSopt并构建1拷贝P43-RBSopt-smut整合表达框转化B.subtilis WB800菌株,获得重组菌株BS8-P43-RBSopt-Smut;将2拷贝P43-RBSopt-smut整合表达框转化BS8-P43-RBSopt-Smut,获得重组菌株BS8-P43-RBSopt-2Smut;将3拷贝P43-RBSopt-smut整合表达框转化BS8-P43-RBSopt-2Smut,获得重组菌株BS8-P43-RBSopt-3Smut。然后将这3株重组菌株进行α-熊果苷的全细胞催化,结果如图2-b所示。当将原始RBS序列替换为RBSopt后,BS8-P43-RBSopt-Smut菌株的α-熊果苷合成能力就已接近于BS8-P43-4Smut菌株,为46.33 g/L,底物HQ的摩尔转化率为37.46%;BS8-P43-RBSopt-2Smut和BS8-P43-RBSopt-3Smut菌株合成α-熊果苷的能力不断提高,其中BS8-P43-RBSopt-3Smut菌株全细胞催化合成α-熊果苷的产量为99.14 g/L,底物HQ的摩尔转化率为80.15%。上述结果表明,通过优化RBS序列和增加拷贝数能够实现目标蛋白酶Smut的高效表达,最终提高α-熊果苷的产量。
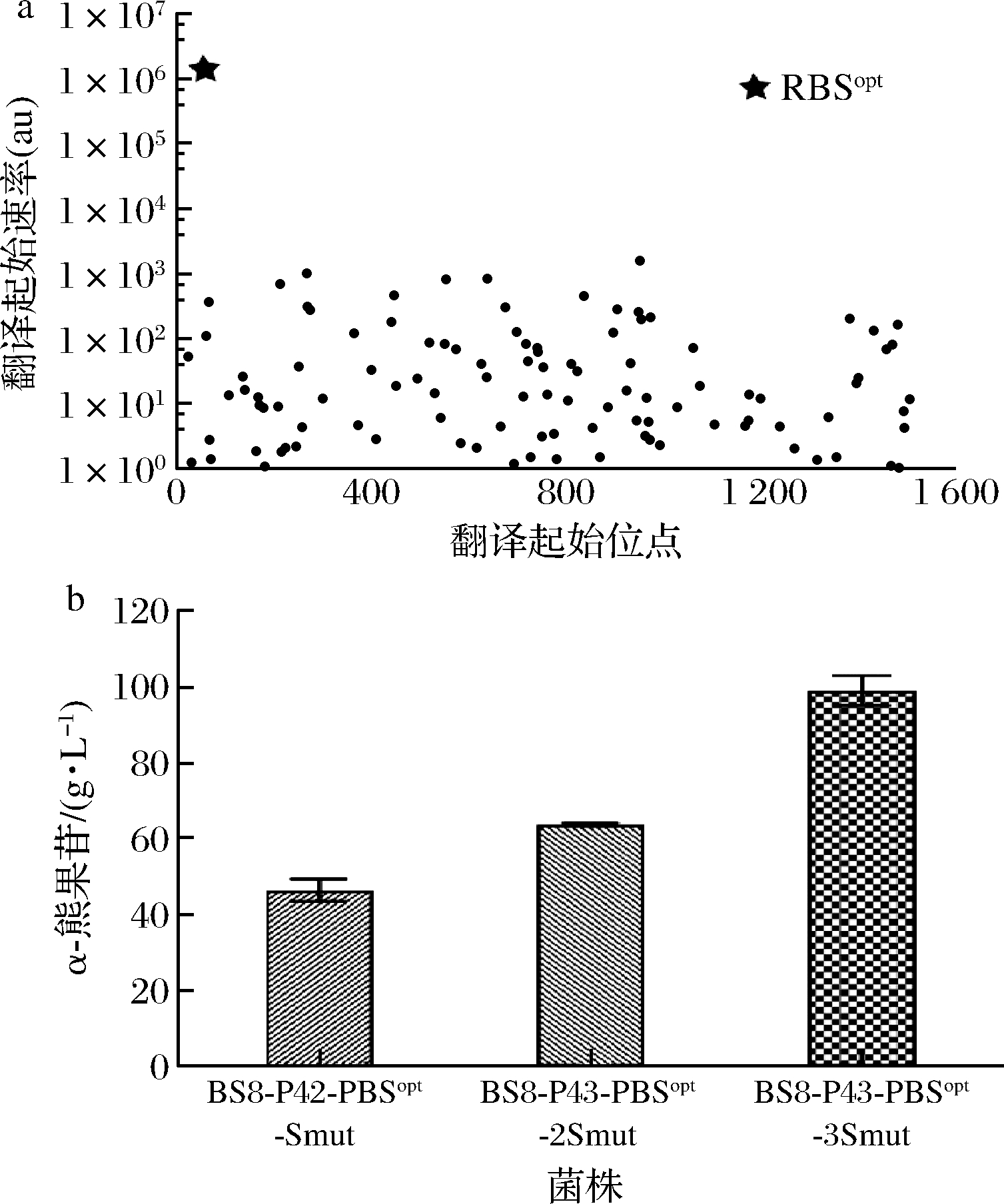
a-“RBS caculator”预测的不同RBS序列对应的翻译速率;b-重组菌株BS8-P43-RBSopt-Smut, BS8-P43-RBSopt-2Smut, BS8-P43-RBSopt-3Smut全细胞催化22 h时合成α-熊果苷的情况
图2 蔗糖磷酸化酶基因smut的RBS序列优化后菌株合成α-熊果苷的情况
Fig.2 Production of α-arbutin after optimization of the RBS sequence of smut gene
2.3 全细胞催化合成α-熊果苷的条件优化
在前面的研究中,我们使用5 mL的反应体系进行全细胞催化,但是发现多个批次之间的α-熊果苷产量不够稳定。此外,HPLC检测时发现催化22 h的反应液在产物峰之后始终伴随着一个面积较大的杂峰(图3-a)。通过与HQ标准品的出峰时间(图3-b)比对,确定该杂峰对应的物质就是HQ,这说明在全细胞催化反应进行到22 h时还有一部分HQ未被转化为产物。因此,我们尝试将反应体系扩大至 20 mL 并延长催化时间,发现不同批次之间α-熊果苷的产量保持稳定,表明扩大反应体系有利于α-熊果苷的稳定合成。结果如图3-d所示,当催化时间延长至34 h时,BS8-P43-RBSopt-Smut、BS8-P43-RBSopt-2Smut和BS8-P43-RBSopt-3Smut菌株催化生成的α-熊 果苷含量均有明显提高,其中BS8-P43-RBSopt-3Smut菌株累积合成了119.44 g/L的α-熊果苷,底物HQ的摩尔转化率为96.56%。催化时间超过34 h 后,α-熊果苷的产量趋于稳定,在46 h时检测到α-熊果苷质量浓度为121.60 g/L。表明α-熊果苷的催化合成在前34 h就基本完成。同时,我们对催化液中的底物HQ浓度进行检测,如图3-e所示,发现随着催化时间的延长,HQ的浓度不断降低,这也间接证明了适当地延长催化时间可以提高α-熊果苷的产量。
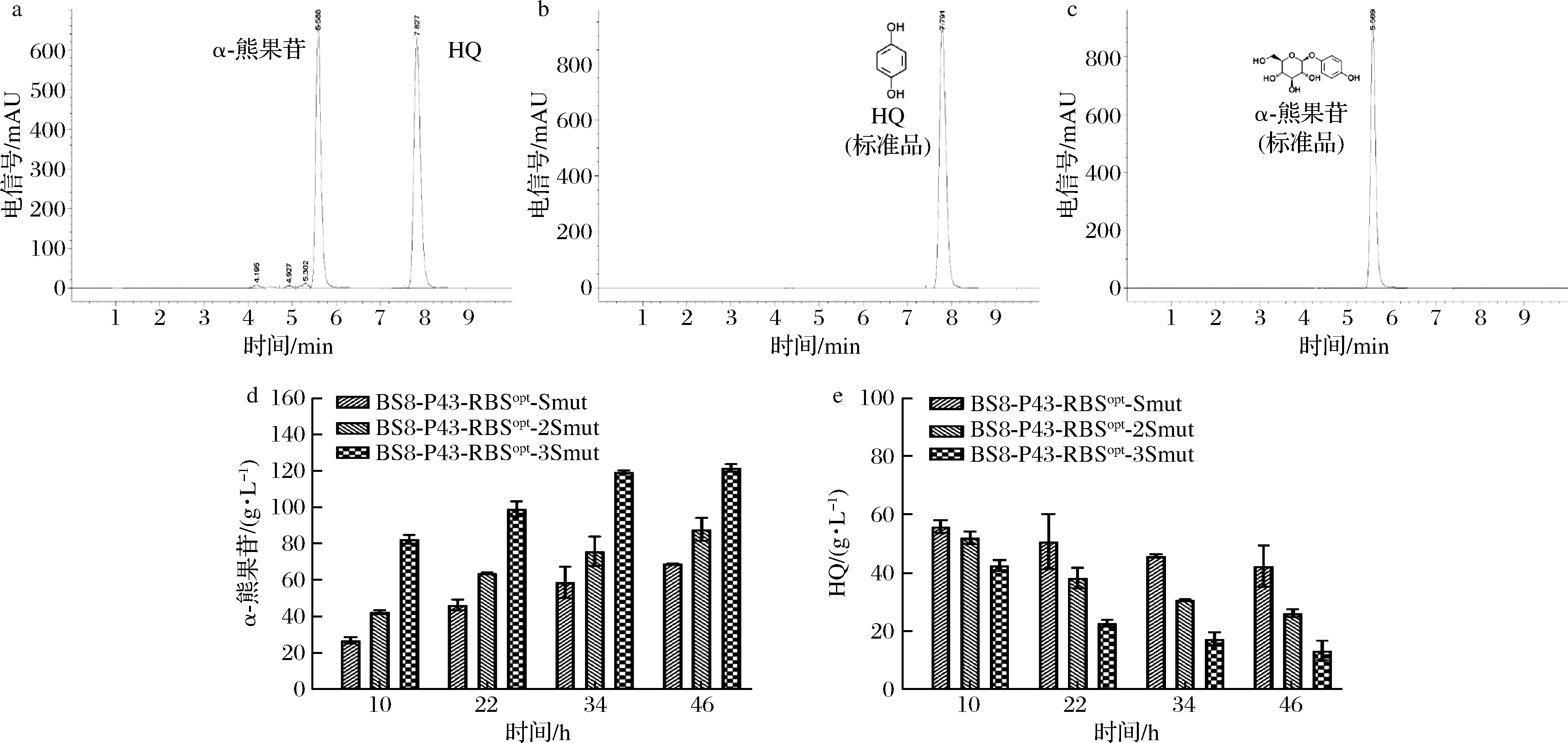
a-重组菌株全细胞催化22 h时催化液中α-熊果苷和对HQ对应的出峰时间和峰面积;b-HQ标准品高效液相色谱出峰时间;c- α-熊果苷标准品高效液相色谱出峰时间;d-重组菌株在不同催化时间时α-熊果苷的积累量;e-重组菌株在不同催化时间时底物HQ的残留量
图3 延长全细胞催化时间对α-熊果苷合成情况的影响
Fig.3 Production of α-arbutin when extending the whole cell catalysis time
3 结论与讨论
前期的研究对变异链球菌来源的蔗糖磷酸化酶进行分子改造,获得了具有高效催化合成α-熊果苷潜力的SmSPI336L。本文在此基础上,将SmSPI336L在B.subtilis WB800菌株中异源整合表达,获得的重组菌株BS8-P43-4Smut在全细胞催化22 h时α-熊 果苷的产量54.05 g/L,底物HQ的摩尔转化率为43.70%。通过RBS序列优化策略,并对催化条件进行优化后,重组菌株BS8-P43-RBSopt-3Smut在全细胞催化34 h时α-熊果苷的产量为119.44 g/L,底物HQ的摩尔转化率为96.56%。与目前已报道的α-熊果苷合成方法相比,利用该方法具有操作简便、产物转化率高且产量稳定的优点,为实现α-熊果苷的工业化生产提供了一个新的策略。但是,本研究中全细胞催化合成α-熊果苷是在摇瓶中完成的,为满足工业化生产的需要,接下来需要放大催化体系,对发酵条件、全细胞催化的策略进行系统地优化。
[1] JURICA K, GOBIN I, KREMER D, et al.Arbutin and its metabolite hydroquinone as the main factors in the antimicrobial effect of strawberry tree (Arbutus unedo L.) leaves[J].Journal of Herbal Medicine, 2017, 8:17-23.
[2] TABATA M, TSUKADA M, FUKUI H.Antimicrobial activity of quinone derivatives from Echium lycopsis callus cultures[J].Planta Medica, 1982, 44(4):234-236.
[3] IOKU K, TERAO J J, NAKATANI N.Antioxidative activity of arbutin in a solution and liposomal suspension[J].Bioscience, Biotechnology, and Biochemistry, 1992, 56(10):1 658-1 659.
[4] BANG S H, HAN S J, KIM D H.Hydrolysis of arbutin to hydroquinone by human skin bacteria and its effect on antioxidant activity[J].Journal of Cosmetic Dermatology, 2008, 7(3):189-193.
[5] HAN R Z, LI J H, SHIN H D, et al.Recent advances in discovery, heterologous expression, and molecular engineering of cyclodextrin glycosyltransferase for versatile applications[J].Biotechnology Advances, 2014, 32(2):415-428.
[6] DINMUKHAMED T, HUANG Z Y, LIU Y F, et al.Current advances in design and engineering strategies of industrial enzymes[J].Systems Microbiology and Biomanufacturing, 2021, 1(1):15-23.
[7] SUGIMOTO K, NISHIMURA T, KURIKI T.Development of α-arbutin:Production at industrial scale and application for a skin-lightening cosmetic ingredient[J].Trends in Glycoscience and Glycotechnology, 2007, 19(110):235-246.
[8] SUGIMOTO K, NISHIMURA T, NOMURA K, et al.Inhibitory effects of α-arbutin on melanin synthesis in cultured human melanoma cells and a three-dimensional human skin model[J].Biological & Pharmaceutical Bulletin, 2004, 27(4):510-514.
[9] ZHU X T, TIAN Y Q, ZHANG W L, et al.Recent progress on biological production of α-arbutin[J].Applied Microbiology and Biotechnology, 2018, 102(19):8 145-8 152.
[10] SEO D H, JUNG J H, LEE J E, et al.Biotechnological production of arbutins (α- and β-arbutins), skin-lightening agents, and their derivatives[J].Applied Microbiology & Biotechnology, 2012, 95(6):1 417-1 425.
[11] KITAO S, SEKINE H.α-D-glucosyl transfer to phenolic compounds by sucrose phosphorylase from Leuconostoc mesenteroides and production of α-arbutin[J].Bioscience, Biotechnology, and Biochemistry, 1994, 58(1):38-42.
[12] NISHIMURA T, KOMETANI T, TAKII H, et al.Purification and some properties of X-amylase from Bacillus subtilis X-23 that glucosylates phenolic compounds such as hydroquinone[J].Journal of Fermentation and Bioengineering, 1994, 78(1):31-36.
[13] NISHIMURA T, KOMETANI T, TAKII H, et al.Acceptor specificity in the glucosylation reaction of Bacillus subtilis X-23 α-amylase towards various phenolic compounds and the structure of kojic acid glucoside[J].Journal of Fermentation and Bioengineering, 1994, 78(1):37-41.
[14] ZHANG W, LIU Z M, GONG M Y, et al.Metabolic engineering of Escherichia coli for the production of Lacto-N-neotetraose (LNnT)[J].Systems Microbiology and Biomanufacturing, 2021,1(3):291-301.
[15] GOEDL C, SAWANGWAN T, WILDBERGER P, et al.Sucrose phosphorylase:A powerful transglucosylation catalyst for synthesis of α-D-glucosides as industrial fine chemicals[J].Biocatalysis and Biotransformation, 2010, 28(1):10-21.
[16] DENG J Y, LYU X, LI J H, et al.Recent advances and challenges in microbial production of human milk oligosaccharides[J].Systems Microbiology and Biomanufacturing, 2021, 1(1):1-14.
[17] KITAO S, SEKINE H.Transglucosylation catalyzed by sucrose phosphorylase from Leuconostoc mesenteroides and production of glucosyl-xylitol[J].Bioscience, Biotechnology, and Biochemistry, 1992, 56(12):2 011-2 014.
[18] AERTS D, VERHAEGHE T F, ROMAN B I, et al.Transglucosylation potential of six sucrose phosphorylases toward different classes of acceptors[J].Carbohydrate Research, 2011, 346(13):1 860-1 867.
[19] ABOLBAGHAEI A, SILKE J R, XIA X H.How changes in anti-SD sequences would affect SD sequences in Escherichia coli and Bacillus subtilis[J].G3-Genes Genomes Genetics, 2017, 7(5):1 607-1 615.
[20] REIS A C, SALIS H M.An automated model test system for systematic development and improvement of gene expression models[J].ACS Synthetic Biology, 2020, 9(11):3 145-3 156.
[21] ESPAH BORUJENI A, CETNAR D, FARASAT I, et al.Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences[J].Nucleic Acids Research, 2017, 45(9):5 437-5 448.