核黄素又名维生素B2,与动植物的生长密切相关,在生物体内常以黄素单核苷酸(flavin mononucleotide,FMN)和黄素腺嘌呤二核苷酸(flavin adenine dinucleotide,FAD)两种形式存在,发挥辅酶的作用。核黄素常被用作饲料添加剂、营养添加剂、辅助药物等,广泛应用于饲料、食品、医疗等领域[1]。目前核黄素工业大规模生产的主要方法为微生物发酵法,枯草芽孢杆菌(Bacillus subtilis)、棉囊阿舒酵母(Assulta gossypii)等都是工业上常用的核黄素生产菌株[2-3]。
在核黄素的合成途径中,鸟苷三磷酸(guanosine triphosphate,GTP)是重要前体之一。它既可以利用糖类及氨基酸为原料进行头合成,也可以利用嘌呤碱为前体进行补救合成[4]。在工业生产中,培养基成分复杂,尤其是酵母粉或酵母抽提物中含有一些嘌呤类物质,理论上既可提供GTP合成的原料,又会对从头合成产生抑制作用[5]。但在以酵母提取物、玉米浆为氮源的工业培养基中,补救合成途径并不是核黄素合成的主要途径[6]。因此通过嘌呤转运蛋白的敲除调节胞内嘌呤类物质的含量,进而解除培养基中嘌呤类物质对从头合成的抑制是一种潜在的提高核黄素产量的手段。枯草芽孢杆菌基因组中存在7个被注释为嘌呤转运蛋白的基因(pbuG、nupG、pbuE、ywdJ、nupNOPQ、pbuX、pbuO)。根据文献报道,PbuE为次黄嘌呤和腺嘌呤的外排蛋白[7-8],PbuG为鸟嘌呤和次黄嘌呤的内运蛋白[9],PbuX为黄嘌呤的内运蛋白[10],PbuO为鸟嘌呤和次黄嘌呤的内运蛋白[9]。然而7种转运蛋白对全部4种嘌呤碱的转运能力,以及敲除转运蛋白对核黄素产量的影响还没有被系统的研究过。
本研究发现培养基中添加一定浓度的嘌呤碱对核黄素的合成有抑制作用。转运蛋白缺失后,这种抑制的程度会发生改变。根据抑制程度的改变和转运蛋白缺失突变体生长表型的变化鉴定了7种嘌呤转运蛋白对4种嘌呤碱的转运功能,并且本文构建了一系列转运蛋白缺失菌株并测定了这些菌株的核黄素产量。
1 材料与方法
1.1 实验材料
1.1.1 实验菌株
实验涉及的菌株如表1所示。
表1 本实验所用菌株
Table 1 Strains used in this study

菌株特征来源RB.subtilis 168, araR:Para-neo, ribO*, rib+(gsiB), rpe*本实验室保藏RFR, ΔpurF::cat本实验构建RPR, ΔpbuG本实验构建RGR, ΔnupG本实验构建RER, ΔpbuE本实验构建RYR, ΔywdJ本实验构建RNR, ΔnupNOPQ本实验构建RXR, ΔpbuX本实验构建ROR, ΔpbuO本实验构建RFPRF, ΔpbuG本实验构建RFGRF, ΔnupG本实验构建RFERF, ΔpbuE本实验构建RFYRF, ΔywdJ本实验构建RFNRF, ΔnupNOPQ本实验构建RFXRF, ΔpbuX本实验构建RFORF, ΔpbuO本实验构建RNXRN, ΔpbuX本实验构建RNXORNX, ΔpbuO本实验构建RNXPRNX, ΔpbuG本实验构建RNXOPRNXO, ΔpbuG本实验构建
1.1.2 主要培养基
LB培养基(g/L):酵母抽提物5,胰蛋白胨10,氯化钠10。
SPI(g/L):硫酸铵1.96,磷酸氢二钾13.72,磷酸二氢钾5.88,柠檬酸钠0.98,七水硫酸镁0.196,葡萄糖5,酸水解酪素0.2,酵母抽提物1。
SPⅡ(g/L):硫酸铵1.92,磷酸氢二钾13.45,磷酸二氢钾5.76,柠檬酸钠0.96,七水硫酸镁0.192,葡萄糖4.9,酸水解酪素0.196,酵母抽提物0.98,氯化钙0.006,氯化镁0.238。
发酵培养基(g/L):白玉米浆干粉15,玉米浆干粉1.5,蔗糖40,硫酸镁0.5,硫酸铵5,酵母抽提物5,磷酸氢二钾3,磷酸二氢钾1,灭菌前用饱和NaOH调节pH至7.2,121 ℃,灭菌30 min后,根据需要添加不同浓度的嘌呤碱。
SP基本盐培养基(g/L):SPI(不添加酸水解酪素和酵母抽提物),色氨酸0.05,根据需要添加不同浓度的嘌呤碱。
1.1.3 主要试剂
腺嘌呤碱、次黄嘌呤碱、黄嘌呤碱、鸟嘌呤碱,北京索莱宝科技有限公司。
1.1.4 仪器与设备
EDC-810基因扩增仪,东胜国际贸易有限公司;水平电泳仪,北京市六一仪器厂;94-Z水平摇床,宁波新芝生物科技股份有限公司;V-1600可见分光光度仪,上海美谱达仪器有限公司。
1.2 实验方法
1.2.1 嘌呤转运蛋白缺失突变菌株构建方法
本实验所涉及的基因序列均来自NCBI(https://www.ncbi.nlm.nih.gov/),使用SnapGene设计所用引物,由擎科生物公司合成(表2)。
表2 本实验所用引物
Table 2 Primers used in this study

引物序列(5'-3')UPpurF-FGAAGATGCGAATGAAATGCTGAAGGUPpurF-RGGCTTAAATGAAGAATGCGGCGTCTTCAACTAAAGCACCCATTAGTTCAACAAACGAAACM-FTCTTCAACTAAAGCACCCATTAGTTCAACAAACGCMpurF-RATTGTCAGATAGGCCTAATGACTGGCTTTTATAACAGGATACAGTGCTTCCTCACGTAAAAGAAGCAGDNpurF-FCAGGATACAGTGCTTCCTCACGDNpurF-RGCCTATTTAATCGGGCGTGTGUPpbuG-FCGTGAAACGGTCGGAGACAUPpbuG-RTGAACTAATGGGTGCTTTAGTTGAAGATGAAATACCCATACCAAGGACACRpbuG-RGTAGACGTACCAAGCACGGCTGAAATAC-CCATACCAAGGACAACAGAAAACGCAAAAAACTTATTCATTCAGTTTTCGTGCGDNpbuG-FGCCGTGCTTGGTACGTCTACDNpbuG-RGCCATGTCCATCATCCTCCUPnupG-FGAATCGCAGAGCGGGCATUPnupG-RTGAACTAATGGGTGCTTTAGTTGAAGAGTCTACTGTTTCATTCGCCATCACCRnupG-RGGACGTCAGGAATGTCGTCGGTCTACT-GTTTCATTCGCCATCACTGACGGAAAGGCAAACTTATTCATTCAGTTTTCGTGCGDNnupG-FCGACGACATTCCTGACGTCCDNnupG-RGGTGTTTGCCCAGTGGTGCUPnupN-FCCAGCGAGCTTGACGTGATCUPnupN-RTGAACTAATGGGTGCTTTAGTTGAAGACCTACGTCAGTAACCATCGCCCRnupN-RAAATGCCGACTCCGTCTTGATCAAGC-CCCTACGTCAGTAACCATCGCCACACTGAATTTGTTTTTGCTTATTCATTCAGTTTTCGTGCGDNnupN-FGGCTTGATCAAGACGGAGTCGDNnupN-RCTGTTTTGCGGAGTCCTGTCAC
续表2

引物序列(5'-3')UPpbuX-FGTTATAATCAAAAGCCTGGCGGCUPpbuX-RTGAACTAATGGGTGCTTTAGTTGAAGAAATCAGAGGAACGACAATGGCGCRpbuX-RAATATTTAAGACGACTGCAGTAAAGCTG-CAATCAGAGGAACGACAATGGCGCCGGCATACATGGCAAGATTATTCATTCAGTTTTCGTGCGDNpbuX-FGCAGCTTTACTGCAGTCGTCTTADNpbuX-RATCGATCACTTTTTTTCCGCCTGUPpbuO-FATCTGTCACAACTTGCACAAACCUPpbuO-RTGAACTAATGGGTGCTTTAGTTGAAGACCATTGTGAAAAAGGTCGTCAGCCRpbuO-RGATGACGAGCGGATGAATCTCTCTCCAT-TCCATTGTGAAAAAGGTCGTCAGCCCTGCTATGATTTCTTGTTATTCATTCAGTTTTCGTGCGDNpbuO-FAATGGAGAGAGATTCATCCGCTCDNpbuO-RTCTATATCGTTTTGCCGGCGATT
根据文献报道的方法进行无痕敲除[11],以B.subtilis 168基因组为模板,分别扩增基因的上游同源臂(UP)、下游同源臂(DN)部分,以带有cat+araR(CR)的菌为模板扩增含有筛选标记CR以及与同向重复序列DR(direct repeats)的部分,进行3片段融合PCR,将产物用SPI-II转化法转入R菌中[12],利用DR在LB培养基中振荡培养进行同源重组,将筛选标记去除,得到敲除菌株。具体敲除过程如图1所示。

图1 同源重组敲除原理图
Fig.1 Schematic diagram of the method of construction of the deletion mutants with homologous recombination
注:目标基因Gene;同源重复序列DR: direct repeats;筛选标记CR:cat+araR;cat为氯霉素抗性基因,araR为阿拉伯糖阻遏蛋白基因
1.2.2 嘌呤从头合成缺陷菌株的构建
通过同源重组将嘌呤合成途径中关键酶PRPP转酰胺酶编码基因purF使用氯霉素抗性基因cat替换,阻断嘌呤的从头合成,构建嘌呤从头合成缺陷菌株RF、RFP、RFG、RFE、RFY、RFN、RFX、RFO。
1.2.3 转运蛋白缺失对生长影响的测定
挑取新鲜的单菌落至5 mL LB液体培养基中,37 ℃, 200 r/min振荡培养12 h,取适量菌液至1.5 mL EP管中,离心弃上清液,用同体积的无菌水洗涤1次,再用同体积的无菌水重悬并微调至相同OD600值,取50 μL转接至添加不同嘌呤碱的SP基本盐培养基中,37 ℃,200 r/min,培养30~40 h,每隔10~12 h测定其OD600。
1.2.4 核黄素浓度的测定
采用分光光度法测定核黄素浓度,取适量体积的发酵液,稀释合适倍数,溶于0.1 mol/L NaOH 避光放置20 min,12 000 r/min,离心1 min,取上清液测定其在444 nm处的吸光度,确定核黄素浓度[13]。
1.2.5 摇瓶发酵验证
从-80 ℃冻存管中取20 μL菌液,涂布于LB固体平板上,37 ℃倒置培养24 h,刮取全部菌苔至1 mL无菌水中,混匀后取适量菌液,以初始OD600=0.1接至含80 mL发酵培养基的500 mL带挡板的三角瓶中,37 ℃,180 r/min振荡培养42 h。
2 结果与分析
2.1 嘌呤转运蛋白缺失菌株的构建
根据文献报道以及在KEGG(https://www.kegg.jp/)及subtiwiki(http://subtiwiki.uni-goettingen.de/)上查找到7种嘌呤转运蛋白基因:pbuG、nupG、pbuE、ywdJ、nupNOPQ、pbuX、pbuO。以实验室保藏的1株有一定核黄素产量的枯草芽孢杆菌R为出发菌株,根据1.2.1中的方法分别敲除了以上7种嘌呤转运蛋白基因,构建了一系列嘌呤转运蛋白缺失的菌株。
以敲除pbuG基因为例,以B.subtilis 168基因组为模板,用引物UPpbuG-F、UPpbuG-R(含DR),和引物DNpbuG-F、DNpbuG-R分别扩增pbuG基因上下游的UP和DN片段,以实验室保藏菌株中带有CR片段的基因组为模板,用引物CR-F、CRpbuG-R(含DR)扩增出CR片段。将所扩增出的UP、CR、DN进行片段融合,将融合后的产物转入菌株R中,通过氯霉素抗性(8 μg/mL)筛选到CR替换pbuG的阳性克隆,用引物UPpbuG-F、DNpbuG-R进行菌落PCR验证,正确的条带大小应为5 248 bp。正确的克隆转接至无抗性的LB液体培养基中培养,直接重复序列DR重组之后,弹出筛选标记CR,涂布在含40 μg/mL新霉素的LB固体培养上,即可得到筛选到目的基因的敲除的阳性克隆,用引物UPpbuG-F、DNpbuG-R对克隆进行菌落PCR验证,大小为3 179 bp即为敲除成功,未敲除成功的条带大小为5 248 bp。
2.2 不同嘌呤物质添加浓度的确定及其对核黄素产量的影响
根据文献报道,腺苷一磷酸(adenosine monophosphate, AMP)、腺苷二磷酸(adenosine diphosphate,ADP)、腺苷三磷酸(adenosine triphosphate,ATP)、鸟苷一磷酸(guanosine monophosphate,GMP)、鸟苷二磷酸(guanosine diphosphate,GDP)、GTP等均会抑制嘌呤的从头合成途径[14-15]。本实验基于如下假设:一旦发酵培养基中嘌呤碱浓度超过阈值,虽然通过补救合成途径可以生成少量嘌呤核苷酸,但这些嘌呤核苷酸会对从头合成途径产生抑制,并且补救合成的嘌呤核苷酸不能弥补这种抑制导致的从头合成GTP的损失,最终导致核黄素产量的降低。为了证明我们的假设,首先尝试在发酵培养基中添加不同浓度的4种嘌呤碱并检测出发菌株R的核黄素产量变化。
发酵培养基中的腺嘌呤、次黄嘌呤、黄嘌呤、鸟嘌呤设置7个终质量浓度梯度,分别为0、1、20、50、100、500、1 000 mg/L,添加1 000 mg/L的腺嘌呤、次黄嘌呤、黄嘌呤及100 mg/L的鸟嘌呤时核黄素产量降低最明显,分别为29.22%、10.15%、17%、10.27%(图2),说明该浓度的嘌呤碱对菌株R的核黄素合成具有较强的抑制效果。因此确定后续实验中,4种嘌呤碱的添加终质量浓度分别为腺嘌呤1 000 mg/L、次黄嘌呤1 000 mg/L、黄嘌呤1 000 mg/L、鸟嘌呤100 mg/L。
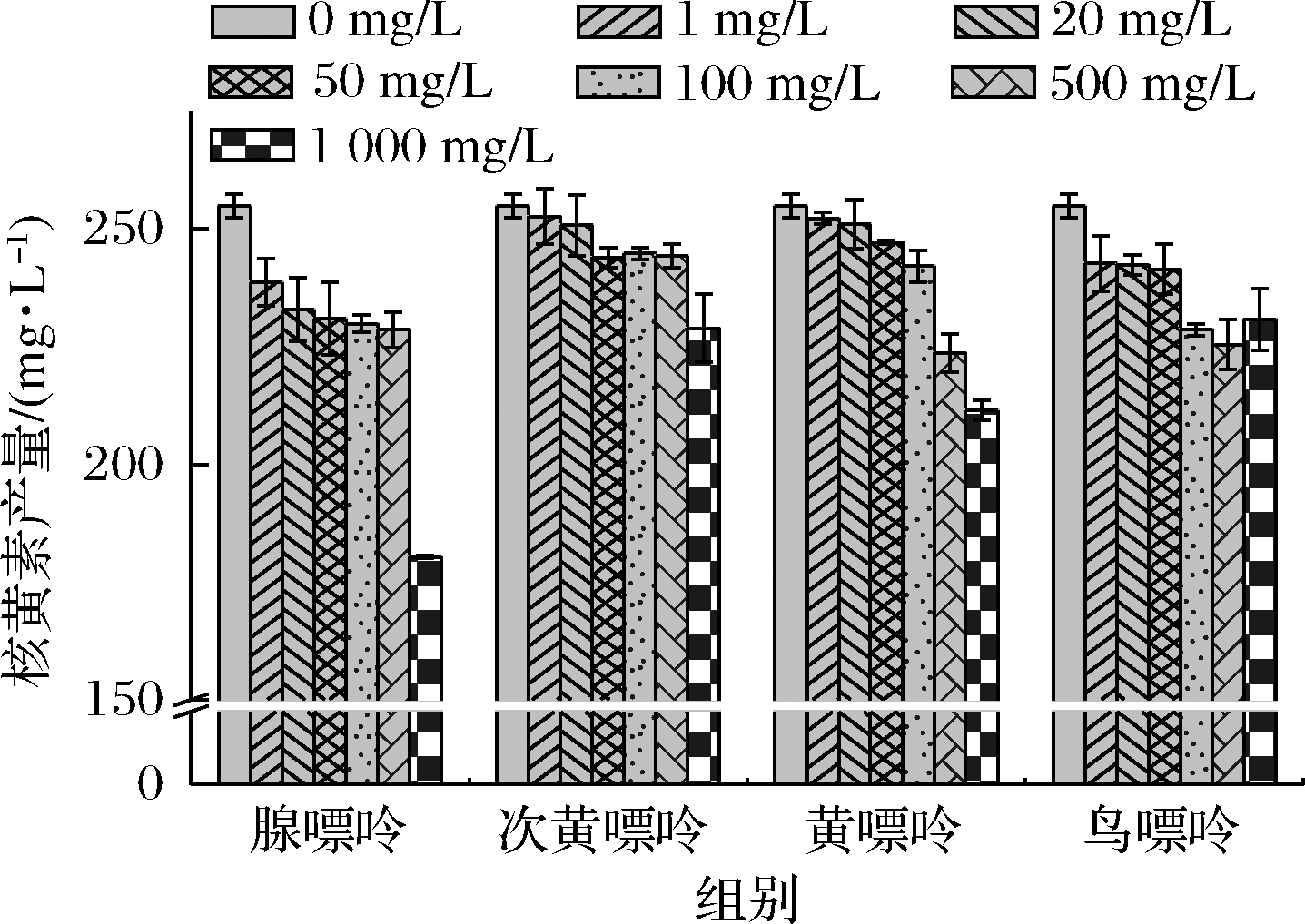
图2 添加不同浓度的嘌呤碱对核黄素产量的影响
Fig.2 The effect of different concentration of purine bases on riboflavin production
敲除嘌呤内运蛋白和外排蛋白,可以阻碍发酵培养基中的嘌呤碱进入细胞,或细胞中嘌呤碱的排出,导致嘌呤从头合成途径抑制程度的改变,而影响核黄素的产量。敲除nupNOPQ、pbuX、pbuO后,腺嘌呤对菌株核黄素产量的抑制程度分别减弱了65.30%、75.80%、58.50%,敲除pbuG、pbuE、ywdJ后,腺嘌呤对菌株核黄素产量的抑制程度分别增强了10.15%、159.90%、106.53%(图3-a);敲除nupG、pbuE、ywdJ、nupNOPQ、pbuX后,次黄嘌呤对菌株核黄素产量的抑制程度分别增强66.10%、45.60%、448.60%、197.20%、 245.80%(图3-b);敲除pbuG、pbuE、ywdJ、nupNOPQ、pbuO后,黄嘌呤对菌株核黄素产量的抑制程度分别减弱了66.10%、55.50%、82.60%、44.70%、34.40%,敲除nupG后,黄嘌呤对菌株核黄素产量的抑制程度增强了61.30%(图3-c);敲除pbuG、pbuE、ywdJ、nupNOPQ、pbuX、pbuO后,鸟嘌呤对菌株核黄素产量的抑制程度分别减弱了66.80%、217.90%、 133.60%、66.50%、82.20%、179.20%(图3-d)。 说明嘌呤转运蛋白的缺失确实影响了嘌呤碱对核黄素合成的抑制作用。
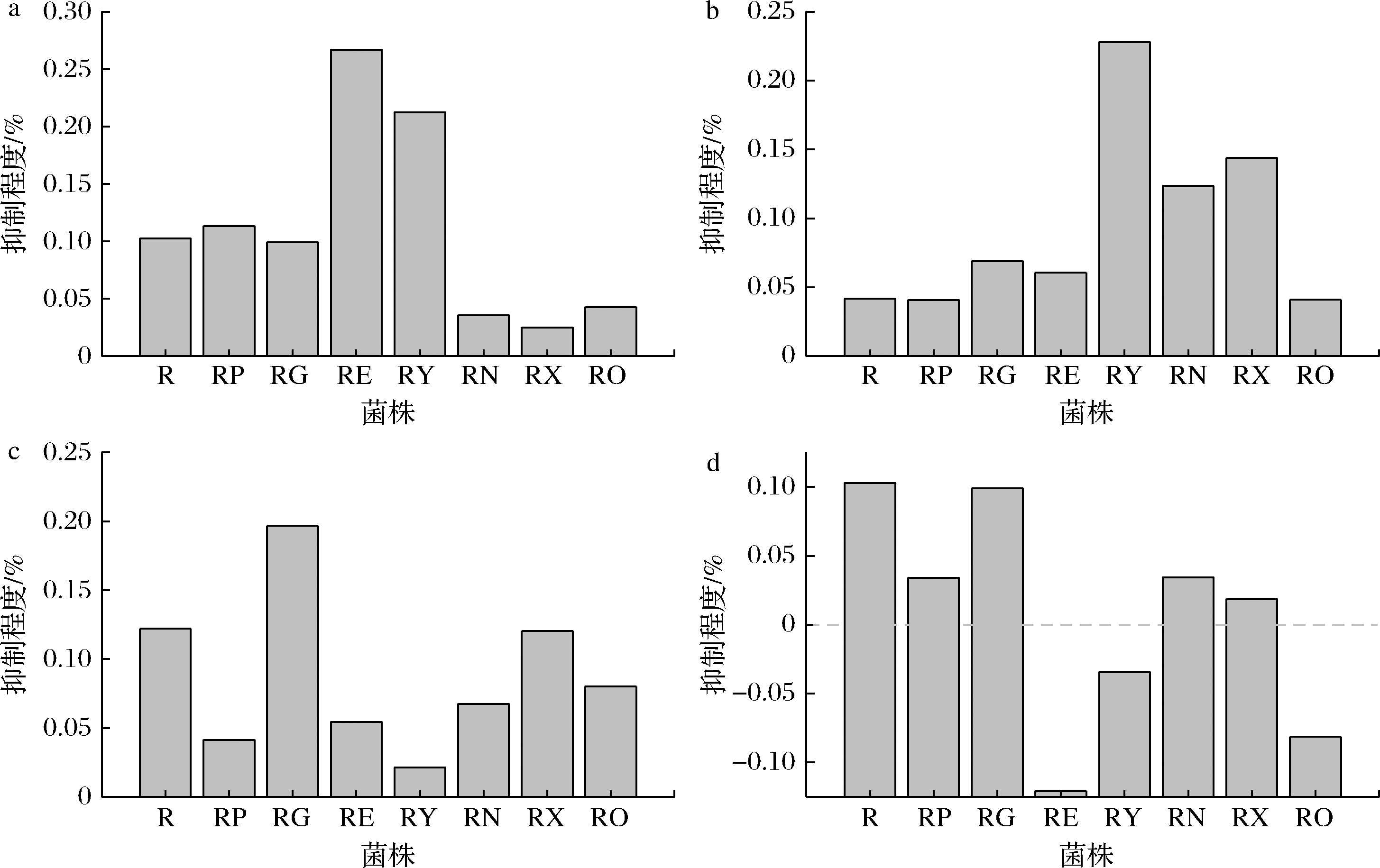
a-1 000 mg/L腺嘌呤;b-1 000 mg/L次黄嘌呤;c-1 000 mg/L黄嘌呤;d-100 mg/L鸟嘌呤
图3 嘌呤转运蛋白缺失对抑制程度的影响
Fig.3 The effect of purine transporters deletion on inhibition
2.3 嘌呤转运蛋白缺失对菌株生长的影响
在阻断嘌呤从头合成途径后,细胞只能利用胞外的嘌呤碱进行补救合成以维持细胞生长,嘌呤转运蛋白的敲除会影响细胞利用胞外嘌呤碱的能力,从而使细胞的生长速度发生改变。首先利用同源重组将嘌呤操纵子中的purF用氯霉素抗性基因cat替换,构建了嘌呤从头合成缺陷菌株RF。在SP基本盐培养基中,若不添加任何嘌呤碱,在培养47 h后通过吸光度法检测不到菌株RF生长,而出发菌株R的OD600可长至0.7左右,因此确定其成功阻断了嘌呤从头合成途径。经过测定RF菌株在SP基本盐培养基中分别添加终质量浓度为10 mg/L腺嘌呤(图4-a),20 mg/L次黄嘌呤(图4-b),20 mg/L 黄嘌呤(图4-c),20 mg/L鸟嘌呤(图4-d)后培养至47 h时可恢复到与出发菌株R生长相近的水平。
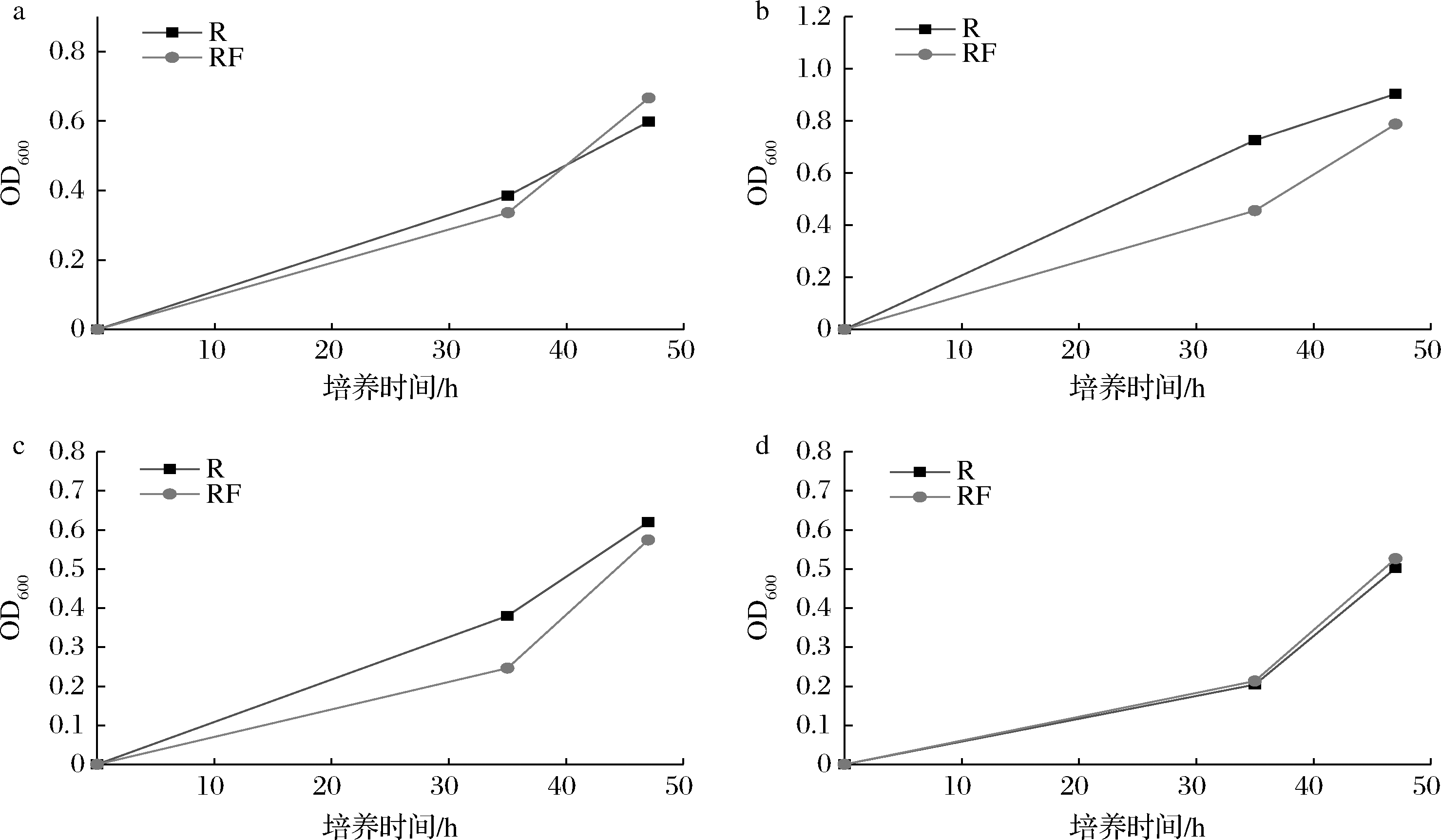
a-10 mg/L腺嘌呤;b-20 mg/L次黄嘌呤;c-20 mg/L黄嘌呤;d-20 mg/L鸟嘌呤
图4 嘌呤从头合成缺陷菌株RF与出发菌株R在添加嘌呤碱后生长情况
Fig.4 The growth of RF and R supplemented with purine bases
由图5可知,敲除nupG、pbuE、ywdJ后,菌株在添加腺嘌呤的SP基本盐培养基中生物量分别提高了10.43%、57.55%、20.50%,敲除nupNOPQ、pbuX、pbuO后,菌株在添加腺嘌呤的培养基中生物量分别降低了6.83%、8.27%、6.65%;敲除nupG、pbuE、nupNOPQ、pbuX后,菌株在添加次黄嘌呤的培养基中生物量分别提高了11.65%、9.61%、16.69%、24.57%;敲除nupG后菌株在添加黄嘌呤的培养基中生物量分别提高了36.38%,敲除pbuG、pbuE、ywdJ、pbuX、pbuO后,菌株在添加黄嘌呤的培养基中生物量分别降低了5.87%、31.65%、19.25%、18.92%、29.04%;敲除nupG、pbuE、ywdJ后,菌株在添加鸟嘌呤的培养基中生物量分别提高了8.12%、50.84%、30.63%,敲除pbuG、pbuX、pbuO后,菌株在添加鸟嘌呤的培养基中生物量分别降低了38.44%、8.12%、15.47%。

图5 嘌呤转运蛋白缺失对生物量的影响
Fig.5 The effect of purine transporters deletion on biomass
2.4 嘌呤转运蛋白的功能分析
在本实验中,我们通过嘌呤转运蛋白缺失对嘌呤碱对核黄素合成的抑制及缺失菌株在添加嘌呤碱的基本培养基中细胞生物量的变化两方面实验判断了7种嘌呤转运蛋白对4种嘌呤碱的转运功能(表3)。一方面,我们通过核黄素产量的变化初步判断PbuG可以外排腺嘌呤,内运黄嘌呤和鸟嘌呤;NupG可外排次黄嘌呤和黄嘌呤;PbuE、YwdJ可能是主要的腺嘌呤外排蛋白,同时可以外排次黄嘌呤,内运黄嘌呤和鸟嘌呤;NupNOPQ、PbuX、PbuO可内运腺嘌呤及鸟嘌呤,NupNOPQ、PbuX可以外排次黄嘌呤, NupNOPQ、PbuO还可内运黄嘌呤。另一方面,通过生长实验可以初步判定PbuG可以内运黄嘌呤和鸟嘌呤;NupG可以外排4种嘌呤碱;PbuE可以外排腺嘌呤、次黄嘌呤和鸟嘌呤,内运黄嘌呤;YwdJ可外排腺嘌呤、鸟嘌呤;NupNOPQ可外排次黄嘌呤,内运腺嘌呤和黄嘌呤;PbuX可外排次黄嘌呤,内运腺嘌呤、黄嘌呤和鸟嘌呤;PbuO可内运腺嘌呤、黄嘌呤和鸟嘌呤。
表3 由产量和生长实验的推测结果以及文献报道的嘌呤转运蛋白功能结果
Table 3 The function of transporters deduced from yield and growth experiments and the functions reported in previous studies

注:“/”左侧代表由核黄素产量变化推测的嘌呤转运蛋白功能;“/”右侧代表由生物量变化推测的嘌呤转运蛋白功能;“内”代表内运;“外”代表外排;“-”代表无影响
组别PbuGNupGPbuEYwdJNupNOPQPbuXPbuO腺嘌呤外/--/外外/外外/外内/内内/内内/内次黄嘌呤-/-外/外外/外外/-外/外外/外-/-黄嘌呤内/内外/外内/内内/-内/内-/内内/内鸟嘌呤内/内-/外内/外内/外内/-内/内内/内文献所报道的功能内运鸟嘌呤与次黄嘌呤外排次黄嘌呤与腺嘌呤-内运黄嘌呤内运鸟嘌呤与次黄嘌呤鉴定方法嘌呤类似物生长抑制[9]嘌呤类似物生长抑制[7-8]-同位素标记[10]嘌呤类似物生长抑制[9]
综合上述两方面实验结果,发现只有其中1个实验证明的结果有7个,2个实验产生相反的结果有2个,两个实验得到的一致结果有17个。其中一致的结果如下:PbuG可以内运黄嘌呤、鸟嘌呤;NupG可外排次黄嘌呤和黄嘌呤;PbuE可以外排腺嘌呤和次黄嘌呤,内运黄嘌呤;YwdJ可外排腺嘌呤;NupNOPQ可外排次黄嘌呤,内运腺嘌呤和黄嘌呤;PbuX可外排次黄嘌呤,内运腺嘌呤、鸟嘌呤;PbuO可内运腺嘌呤、黄嘌呤和鸟嘌呤。
通过比较文献中利用其他实验方法验证的嘌呤转运蛋白的功能,发现其与本文中的结果具有一定的一致性(表3)。
2.5 通过改造嘌呤转运蛋白提高核黄素的产量
通过摇瓶发酵测定了嘌呤转运蛋白缺失菌株的核黄素产量。如图6所示,发现不同嘌呤转运蛋白缺失对核黄素的合成有一定的促进或抑制作用。pbuG、nupNOPQ、pbuX的缺失菌株与出发菌株R相比,核黄素产量分别提高了7.53%、10.74%、5.84%;而pbuE、ywdJ的缺失菌株与出发菌株R相比,核黄素产量分别降低了7.72%、6.48%。
在测定添加不同浓度的嘌呤碱对核黄素产量影响的实验中发现添加较低浓度的腺嘌呤和鸟嘌呤就会对核黄素产量产生抑制作用。因此推测在本实验所用培养基中,转运蛋白对腺嘌呤和鸟嘌呤的转运能力是对核黄素产量的关键影响因素。而单独敲除腺嘌呤/鸟嘌呤内运蛋白基因(除pbuO)和腺嘌呤外排蛋白基因分别提高和降低了核黄素产量。因此对本文中确定的3个编码腺嘌呤内运蛋白基因nupNOPQ、pbuX、pbuO进行了敲除构建了菌株RNXO,同时构建了敲除nupNOPQ、pbuX的菌株RNX,经摇瓶验证发现RNXO和RNX相较于出发菌株R的核黄素产量分别提高了11.39%、12.17%。在RNXO和RNX基础上又分别敲除了鸟嘌呤内运蛋白基因pbuG,得到了RNXOP和RNXP,二者的核黄素产量较出发菌株R分别提高了17.81%、19.14% (图6)。
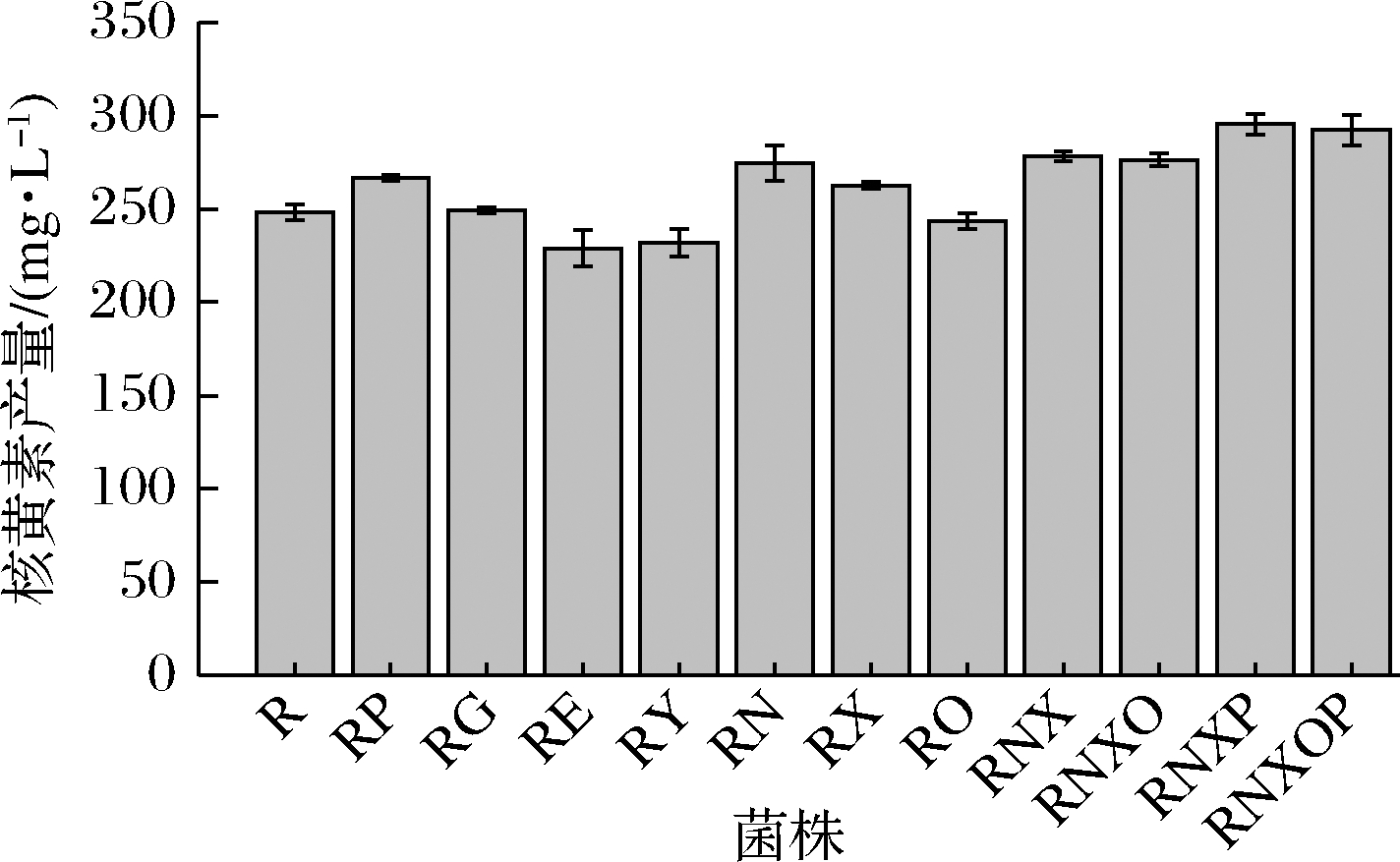
图6 嘌呤转运蛋白缺失菌株的核黄素产量
Fig.6 Riboflavin production of purine transporters deficient strains
3 结论与讨论
经本实验鉴定PbuE、YwdJ具有外排腺嘌呤的功能,NupNOPQ、PbuX、PbuO具有内运腺嘌呤的功能;NupG、PbuE、NupNOPQ、PbuX具有外排次黄嘌呤的功能;NupG具有外排黄嘌呤的功能;PbuG、PbuE、NupNOPQ、PbuO具有内运黄嘌呤的功能;PbuG、PbuX、PbuO具有内运鸟嘌呤的功能。基于其转运功能,在实验室已有菌株R基础上对腺嘌呤及鸟嘌呤的内运蛋白基因nupNOPQ、pbuX、pbuG进行了敲除,将核黄素产量提高了19.14%。
文献报道,嘌呤转运蛋白的敲除会影响嘌呤核苷的产量,嘌呤核苷的生产菌株中也会存在一些嘌呤转运蛋白的突变[16-17]。这些敲除和突变可能影响了菌株嘌呤核苷酸的从头合成能力。本文中鉴定的一些腺嘌呤和鸟嘌呤的转运蛋白与核黄素产量有一定的关系。除pbuO以外,敲除其他腺嘌呤/鸟嘌呤的内运蛋白基因后核黄素产量均有所提高,而敲除外排蛋白的菌株核黄素产量均降低。文献报道PbuO的鸟嘌呤的转运能力在大于100 μmol/L时才能体现[8],因此在我们的培养基中鸟嘌呤的含量没有达到该浓度,而当培养基中额外添加100 mg/L 的鸟嘌呤后,敲除pbuO提高了核黄素的产量(图3)。因此由于发酵培养基成分的差异,对嘌呤转运蛋白敲除的效果也会出现差异。本文对嘌呤转运蛋白的敲除结果对核黄素生产菌株的改造具有一定的参考意义。
前人对于枯草芽孢杆菌生产核黄素的方法已经有了详细的研究,在最新的综述中,这些研究方法已经被归纳汇总[3,18]。WANG等[19]通过整合基因组和转录组数据解析了1株高产核黄素枯草芽孢杆菌的高产机制,并通过逆向代谢工程从头构建了核黄素生产菌株,其摇瓶产量达到4.2 g/L。BOUMEZBEUR等[20]通过点突变核糖开关,在保持细胞正常生长的前提下解除了嘌呤操纵子和核黄素操纵子的转录水平调控,提高了核黄素的产量。而本文首次报道了通过改造嘌呤转运蛋白提高核黄素产量的方法,为核黄素高产菌株的构建提供了新的思路。
[1] SCHWECHHEIMER S K, PARK E Y, REVVELTA J L, et al.Biotechnology of riboflavin[J].Applied Microbiology and Biotechnology, 2016, 100(5):2 107-2 119.
[2] SU Y, LIU C, FANG H, et al.Bacillus subtilis:A universal cell factory for industry, agriculture, biomaterials and medicine[J].Microbial Cell Factories, 2020, 19(1):1-12.
[3] AVERIANOVA L A, BALABANOVA L A, SON O M, et al.Production of vitamin B2 (Riboflavin) by microorganisms:An overview[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:570828.
[4] 沈同, 王镜岩, 赵邦悌, 等, 生物化学 [M].第二版.北京:高等教育出版社, 1991.
SHEN T, WANG J Y, ZHAO B D, Biochemistry[M].2nd ed.Beijing:Higher Education Press, 1991.
[5] SCHWECHHEIMER S K, BECKER J, PEYRIGA L, et al.Metabolic flux analysis in Ashbya gossypii using 13C-labeled yeast extract:Industrial riboflavin production under complex nutrient conditions[J].Microbial Cell Factories, 2018, 17(1):162.
[6] SUN Y W, LIU C, TANG W Z, et al.Manipulation of purine metabolic networks for riboflavin production in Bacillus subtilis[J].ACS Omega,2020, 5(45):29 140-29 146.
[7] JOHANSEN L E, NYGAARD P, LASSEN C, et al.Definition of a second Bacillus subtilis pur regulon comprising the pur and xpt-pbuX operons plus pbuG, nupG (yxjA), and pbuE (ydhL)[J].Journal of Bacteriology, 2003, 185(17):5 200-5 209.
[8] NYGAARD P, SAXILD H H.The purine efflux pump PbuE in Bacillus subtilis modulates expression of the PurR and G-Box (XptR) regulons by adjusting the purine base pool size[J].Journal of Bacteriology, 2005, 187(2):791-794.
[9] SAXILD H H, BRUNSTEDT K, NIELSEN K I, et al.Definition of the Bacillus subtilis PurR operator using genetic and bioinformatic tools and expansion of the PurR regulon with glyA, guaC, pbuG, xpt-pbuX, yqhZ-folD, and pbuO[J].Journal of Bacteriology, 2001, 183(21):6 175-6 183.
[10] CHRISTIANSEN L C, SCHOU S, NYGAARD P, et al.Xanthine metabolism in Bacillus subtilis:Characterization of the xpt-pbuX operon and evidence for purine- and nitrogen-controlled expression of genes involved in xanthine salvage and catabolism[J].Journal of Bacteriology, 1997, 179(8):2 540-2 550.
[11] LIU S H, ENDO K, ARA K, et al.Introduction of marker-free deletions in Bacillus subtilis using the AraR repressor and the ara promoter[J].Microbiology, 2008, 154(9):2 562-2 570.
[12] WU Y K, CHEN T C, LIU Y F, et al.Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis[J].Nucleic Acids Research, 2020, 48(2):996-1 009.
[13] SHAH H, PATEL S, PATEL B, SOLANKI N, et al.Development and validation of UV-visible spectrometric method for estimation of water soluble vitamin riboflavin[J].International Journal of Pharmaceutical Sciences and Research, 2012, 3(9):3 462-3 466.
[14] 王永成, 陈涛, 石婷, 等.嘌呤核苷及其衍生物的代谢工程[J].中国生物工程杂志, 2015, 35(5):87-95.
WANG Y C, CHEN T, SHI T, et al.Progress in biosynthesis of purine nucleosides and their derivatives by metabolic engineering[J].China Biotechnology, 2015, 35(5):87-95.
[15] SHI T, WANG Y C, WANG Z W, et al.Deregulation of purine pathway in Bacillus subtilis and its use in riboflavin biosynthesis[J].Microbial Cell Factories, 2014, 13(1):1-16.
[16] SHEREMET A S, GRONSKIY S V, AKHMADYSHIN R A, et al.Enhancement of extracellular purine nucleoside accumulation by Bacillus strains through genetic modifications of genes involved in nucleoside export[J].Journal of Industrial Microbiology & Biotechnology, 2011, 38(1):65-70.
[17] ZAKATAEVA N P, GRONSKIY S V, SHEREMET A S, et al.A new function for the Bacillus PbuE purine base efflux pump:Efflux of purine nucleosides[J].Research in Microbiology, 2007, 158(8-9):659-665.
[18] LIU S, HU W Y, WANG Z W, et al.Production of riboflavin and related cofactors by biotechnological processes[J].Microbial Cell Factories, 2020, 19(1):31.
[19] WANG G L, SHI T, CHEN T, et al.Integrated whole-genome and transcriptome sequence analysis reveals the genetic characteristics of a riboflavin-overproducing Bacillus subtilis[J].Metabolic Engineering, 2018, 48:138-149.
[20] BOUMEZBEUR A H, BRUER M, STOECKLIN G, et al.Rational engineering of transcriptional riboswitches leads to enhanced metabolite levels in Bacillus subtilis[J].Metabolic Engineering, 2020, 61:58-68.