酿酒酵母(Saccharomyces cerevisiae)在营养缺陷或极端情况下,双倍体的酵母会进入减数分裂过程,最终在二倍体细胞中产生4个单倍体孢子,从而对抗外部环境[1]。单个酿酒酵母孢子的直径在2 μm左右,可用作大分子生物材料[2-4]。然而现有报道中,尚未成功解析酿酒酵母孢子壁的结构[5-8]。本实验室主要对酿酒酵母孢子进行研究,并且通过高浓度盐洗脱酿酒酵母孢子表面,在洗脱液中发现了RNA[9]。
HEK293T细胞来源于人胚胎肾细胞HEK293细胞的优化,是生物分子研究中常用到的细胞之一,由于HEK293T细胞的转染效率高,已成为广大研究者研究基因功能的一个首选细胞株[10]。HEK293T细胞不同于巨噬细胞,没有明显的吞噬作用[11-13]。实验室发现HEK293T可以吞噬酿酒酵母孢子,且内化效率较高[9]。本研究使用购自Sigma公司的聚苯乙烯乳胶颗粒(latex beads amine-modified polystyrene, beads),其直径为2 μm。HEK293T细胞不能内化beads,但是beads通过静电结合酿酒酵母孢子表面分子后,可以被HEK293T细胞内化。
因此,本文着力研究酿酒酵母孢子表面分子与beads交联后,能否出现吞噬作用,以及影响吞噬效率的因素[14-15]。本研究为诱导非巨噬细胞内化大颗粒机制研究、酿酒酵母孢子表面的特性研究奠定了基础[16-17],也为未来酿酒酵母孢子的医用提供了理论基础。
1 材料与方法
1.1 实验材料
1.1.1 菌株
酿酒酵母菌株是以SK-1背景下的二倍体菌株AN120作为野生型(wild type,WT)菌株;大肠杆菌DH5α。本研究所用菌株均为本实验室保存。
1.1.2 酶及试剂
Lyso-Tracker Red,碧云天生物技术公司;RNase、Protease、Loading buffer,宝生物工程(大连)有限公司(TaKaRa);PBS缓冲液、Agrose M、酵母提取物、胰蛋白胨、腺嘌呤、乙酸钾、葡萄糖、氯化钠、山梨醇,生工生物工程有限公司;Liticase溶菌酶、山梨醇、RNAiso Plus、beads,Sigma;DNA marker 1kb plus,北京全式金生物技术有限公司;氯仿、异丙醇、戊二醛,中国医药集团有限公司。
1.1.3 培养基及其他溶液配制
LB培养基(g/L):酵母提取物5,胰蛋白胨10, NaCl 5,琼脂粉20(固体培养基)。
YPAD液体培养基(g/L):酵母提取物10,胰蛋白胨20,腺嘌呤3,葡萄糖20。
YPACe培养基(g/L):酵母提取物10,胰蛋白胨20,腺嘌呤3,乙酸钾20。
KACe培养基(g/L):乙酸钾20。
1.4 mol/L 山梨醇:255 g山梨醇溶解于1 L 0.5% PBS缓冲液。
1.1.4 仪器与设备
DS-Ri2共聚焦荧光显微镜,Nikon公司;TY7622振荡混合器,易扩中国有限公司;QB-128旋转培养器,其林贝尔仪器制造有限公司;HYL-A电热恒温摇床,上海三发科学仪器有限公司;MX 150高速冷冻离心机,日本日立 HITACHI公司;DYY-6C凝胶电泳仪,北京六一仪器厂;Universal Hood II凝胶成像仪,Bio-Rad(美国)公司;JY92-IIN超声波细胞粉碎机,宁波新芝生物科技有限公司;NanoDrop2000,赛默飞世尔科技有限公司公司。
1.2 实验方法
1.2.1 酵母培养与产孢
本文用到的酵母为AN120(野生型,二倍体)。酵母营养细胞使用YPAD培养基,酵母产孢使用YPACe培养基和KACe培养基。
先将酿酒酵母接入5 mL的YPAD培养基于试管中,放入30 ℃,220 r/min的摇床上培养24 h;将5 mL培养物转入100 mL的YPACe培养基于摇瓶中,放入30 ℃,220 r/min的摇床上培养24 h;离心收集菌体,再将菌体转入100 mL的KACe培养基于摇瓶中,放入30 ℃,220 r/min的摇床上培养24 h;取5 μL在显微镜下观察产孢率,统计酿酒酵母和孢子的数量,再用孢子数量除以总数即为产孢率,实验中的产孢率要达到90%;最后离心收集孢子。
1.2.2 孢子的纯化
将收集的细胞重悬于5 mL 1.4 mol/L山梨醇中,向其中加入20 μL的1 mg/mL溶菌酶充分混匀,37 ℃孵育2 h后弃上清液。向孢子中加入5 mL水,超声破碎20 min,将子囊膜破碎,在显微镜下观察,90%以上的孢子处于游离状态。以3 000×g,2 min离心收集菌体。用0.5% TritonX-100洗涤3~6次,每次均以3 000×g,2 min离心收集菌体,随后用水洗涤3次,每次均以3 000×g,2 min,最后离心收集孢子。
1.2.3 酿酒酵母孢子表面RNA的提取
将纯化好的孢子进行称重,加入适量水,使孢子终质量浓度为0.1 g/mL,取500 μL,9 000×g离心1 min, 去除水;加入500 μL 0.6 mol/L NaCl溶液,放入振荡器振荡3 min;15 000 r/min 离心,取上清液500 μL, 加入RNAiso Plus(TaKaRa)500 μL[18]。充分混匀,冰上放置5 min;加入200 μL氯仿(国药),迅速混匀,冰上静置15 min;12 000×g 4 ℃ 离心15 min; 取600 μL上清液转入新的1.5 mL 离心管,加入 600 μL 异丙醇,冰上静置30 min,随后12 000×g 4 ℃ 离心10 min,弃上清液;加入500 μL 75%乙醇清洗沉淀,7 500×g 4 ℃ 离心5 min;去除上清液,冰上静置10 min, 加入50 μL DEPC水。最后用Nano Drop检测 RNA的浓度。
1.2.4 酿酒酵母营养细胞与大肠杆菌RNA提取
酵母细胞于5 mL YPAD过夜培养,取1 mL于1.5 mL 离心管中,9 000×g离心1 min,加入适量玻璃珠,加入RNAiso Plus(TaKaRa)1 mL 于振荡器上振荡1 min,破碎细胞,放置冰上5 min;加入200 μL氯仿,迅速混匀,冰上静置15 min;12 000×g 4 ℃ 离心15 min;取600 μL上清液转入新的1.5 mL 离心管,加入 600 μL异丙醇,冰上静置30 min,12 000×g 4 ℃ 离心10 min,倒去上清液;加入500 μL 75%乙醇清洗沉淀,7 500×g 4 ℃ 离心5 min;去除上清液,冰上静置10 min,加入50 μL DEPC水。最后在Nano Drop检测 RNA的浓度。
大肠杆菌于5 mL LB培养基过夜培养,操作步骤同上。
1.2.5 酿酒酵母孢子表面分子的提取
将纯化好的孢子进行称重,加入适量水,控制孢子终质量浓度为0.1 g/mL,取500 μL,9 000×g离心1 min, 弃上清液;加入500 μL 0.6 mol/L NaCl溶液,放入振荡器振荡3 min;15 000 r/min 离心,取上清液500 μL。 考虑到NaCl的浓度对于吞噬效率以及表面分子的影响,对0.6 mol/L NaCl进行10倍稀释,具体稀释比例可根据后期需要的RNA的量,计算得出对应的 0.6 mol/L NaCl的量,再进行10倍稀释。
1.2.6 beads的交联
不加入戊二醛交联:将beads置于2 mL离心管中,RNA组(加入定量的RNA),酿酒酵母表面分子组(按照RNA的量,计算出加入酿酒酵母表面分子的量),加入DEPC水,定容至2 mL。最后置于旋转仪上(4 ℃,24 h),充分交联[19]。
戊二醛交联:将beads置于2 mL离心管中,向离心管中加入200 μL 2%戊二醛预处理beads,置于30 ℃ 培养箱1 h;离心(5 000×g, 1 min),并用去离子水洗涤,再离心,重复3次;RNA组(加入定量的RNA),酿酒酵母表面分子组(按照RNA的量,计算出加入酿酒酵母表面分子的量),加入DEPC水,定容至2 mL。最后置于旋转仪上(4 ℃,24 h),充分交联。
1.2.7 RNA的检测
对于酿酒酵母孢子表面分子,分别用RNase(0.5 U) 进行处理,37 ℃,1 h,再进行1.2.6中的交联。
1.2.8 HEK293T细胞的培养
在长满HEK293T细胞的培养皿中,去除培养液,加入10 mL新的培养液充分悬浮,并取适量转入新的24孔培养板中,计数,数量约为2.5×106个,37 ℃培养。
1.2.9 beads的吞噬与显微镜观察计数
将1.2.6中的终产物,3 000×g 2 min 离心,去除上清液,加入10 μL 水,混匀,取10 μL加入1.2.8中的HEK293T细胞培养液中,加入30 μL lysosome tracker,37 ℃ 放置1 h。取出培养板,除去培养液,加入100 μL PBS。在显微镜下观察吞噬现象,并计数,计算吞噬的数量,从而得出吞噬效率。
2 结果与分析
2.1 HEK293T细胞对beads的吞噬
首先,在本研究室前期的研究工作中,发现酿酒酵母孢子能够被HEK293T细胞吞噬,孢子直径2 μm左右,所以HEK293T细胞对于微米级的颗粒也有着吞噬的作用。为此,研究中购买来自Sigma公司的聚苯乙烯乳胶珠可用于创建乳胶凝集系统,称为beads,beads表面带有氨基基团,呈正电荷,直径2 μm。用beads来进行吞噬研究。研究中将beads和HEK293T细胞共同培养1 h,然后共聚焦显微镜下观察beads内化现象。如图1所示, beads本身具有蓝色荧光,在显微镜DAPI下能明显观察到;lysosome tracker显示的红色即为被吞噬的beads,所以在TRAIC下能观察到红色的圆圈即为被吞噬;BF为拍到的beads和HEK293T细胞;MERGE为前3张图的重叠图。结果显示beads能被HEK293T细胞吞噬。考虑到研究中加入的beads的数量是否会改变吞噬效率,因此研究了不同的beads量对于吞噬效率的影响,如图2所示,100个HEK293T细胞吞噬的beads的数目即为吞噬百分比,进而来表示吞噬的效率,吞噬百分比越高即为100个HEK293T细胞吞噬的beads数目越多。在加入不同量的beads条件下,100个HEK293T细胞吞噬的beads数目在1~2个左右,且差距不大,在后续的研究中使用8×107个beads做吞噬研究。
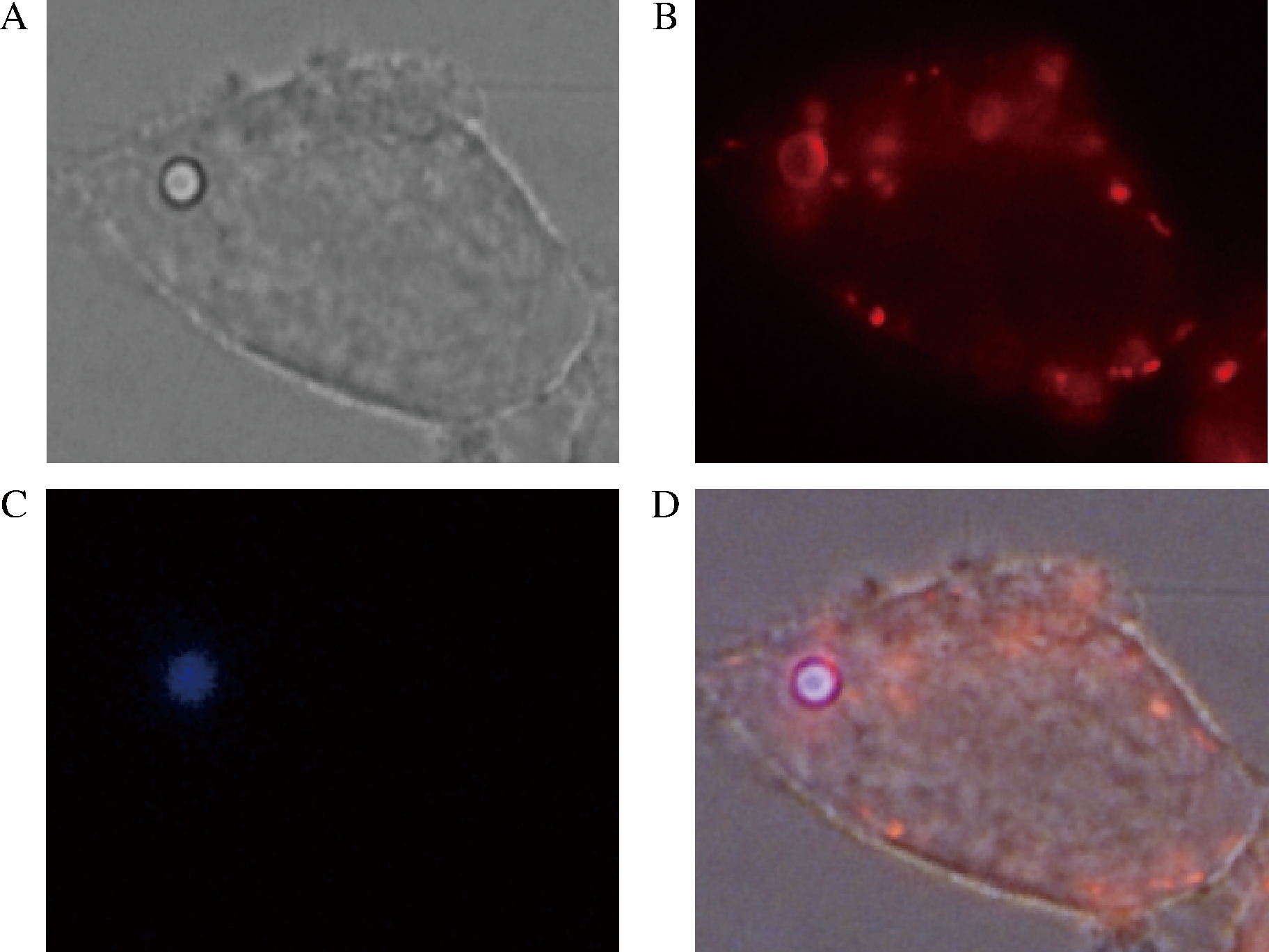
A-BF; B-TRAIC; C-DAPI; D-MERGE
图1 HEK293T细胞吞噬beads
Fig.1 HEK293T cell phagocytizes beads

图2 不同beads量的吞噬效率
Fig.2 The phagocytosis efficiency of different amount of beads
2.2 RNA对于吞噬的影响
本研究用0.6 mol/L NaCl溶液洗脱酿酒酵母孢子表面,从NaCl洗脱液中提取RNA再进行纯化,然后将RNA和beads一起孵育绑定,RNA带负电荷,氨基修饰的beads带正电荷,通过正负电荷作用力进行绑定,形成RNA-beads后再进行吞噬实验。研究猜测不同的RNA量对于吞噬效率有影响,所以用不同浓度的RNA绑定beads。为了探究不同来源的RNA是否对beads的吞噬有影响,分别从大肠杆菌、酿酒酵母营养细胞中提取了RNA,并与酿酒酵母孢子壁RNA以及购买的tRNA作了比较,不同RNA的凝胶电泳如图3所示。如图4-A所示,50 000 ng的RNA效果最优,因此后续的研究都选用50 000 ng的RNA进行反应;吞噬效率的结果如图4-B所示,结果表明所有RNA对于吞噬都有促进作用,但是来自酿酒酵母孢子表面的RNA和tRNA有更明显的促进作用,同时由图3看出,酿酒酵母孢子表面RNA和tRNA 都是长度非常短的RNA,猜测短链RNA更能影响beads的吞噬效率。
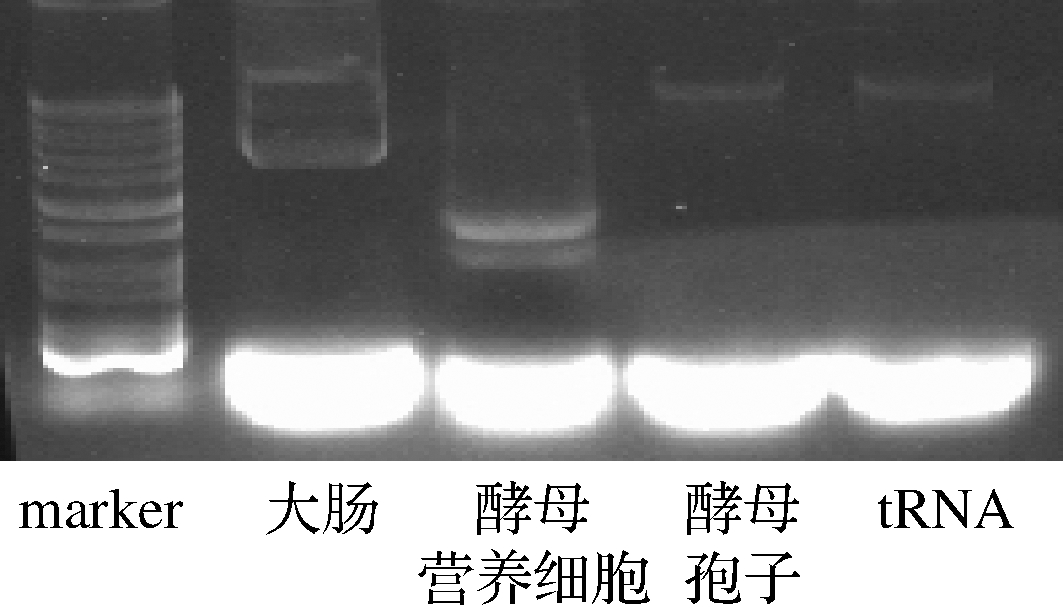
图3 不同RNA纯化后的凝胶电泳图
Fig.3 Gel electrophoresis of different purified RNA
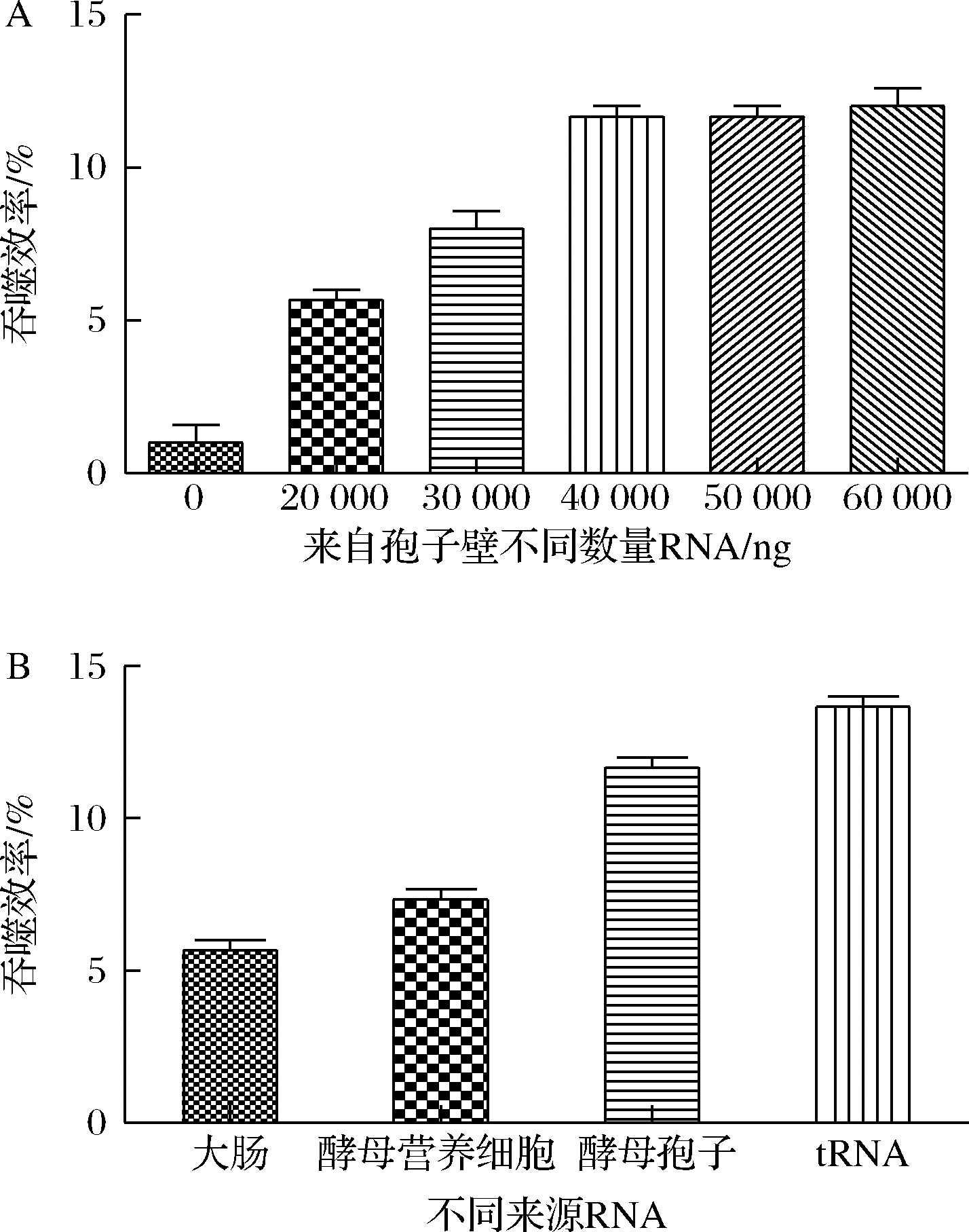
A-不同RNA的量;B-不同来源RNA
图4 不同RNA的量、不同来源RNA对于吞噬效率的影响
Fig.4 The phagocytosis efficiency of different amount of RNA and different sources of RNA
2.3 RNase对beads和RNA-beads吞噬作用影响
为了进一步确认RNA对于beads的吞噬有着促进作用,因此有必要在体系中分别用RNase处理beads和RNA-beads,再和HEK293T细胞共同孵育,进行吞噬实验。如图5所示,结果显示RNase不会对beads的吞噬产生影响。对于从酿酒孢子表面的RNA进行纯化,并对样品分别加入RNase(0.5 U),37 ℃, 1 h,再进行绑定然后观察吞噬结果。研究发现RNase可以明显的降低吞噬效率。因此研究中纯化的RNA确实对吞噬有促进作用。

图5 RNase对只有beads吞噬效率的影响和对加入RNA的beads吞噬效率的影响
Fig.5 Effects of RNase on the phagocytosis efficiency of beads and phagocytosis efficiency of beads with RNA
2.4 酿酒酵母孢子表面分子和戊二醛交联作用促进吞噬作用
本研究选择用0.6 mol/L NaCl进行洗脱酿酒酵母孢子表面的分子,由于RNA也是从NaCl洗脱液中提取纯化获得,研究猜想是否洗脱液也会影响beads的内化,所以用beads直接绑定孢子表面洗脱液,对洗脱液进行了10倍稀释以防高浓度的NaCl影响beads对分子的绑定。如图6所示,NaCl的洗脱液相对于对照组、纯化后的RNA都有明显提升。考虑到酿酒酵母表面分子的洗脱液中可能还有其他物质,所以使用交联剂戊二醛去研究是否其他分子也影响了beads的吞噬,戊二醛能够作为交联剂,对于氨基、羧基等有着交联作用。结果见图6,戊二醛的加入,使吞噬作用有了显著提升,这说明孢子洗脱液中除RNA外还有其他分子也可以影响beads的吞噬。研究中对戊二醛的影响做了猜测,戊二醛可能交联了酿酒孢子表面除RNA外的其他分子,对于该分子需要进一步研究。
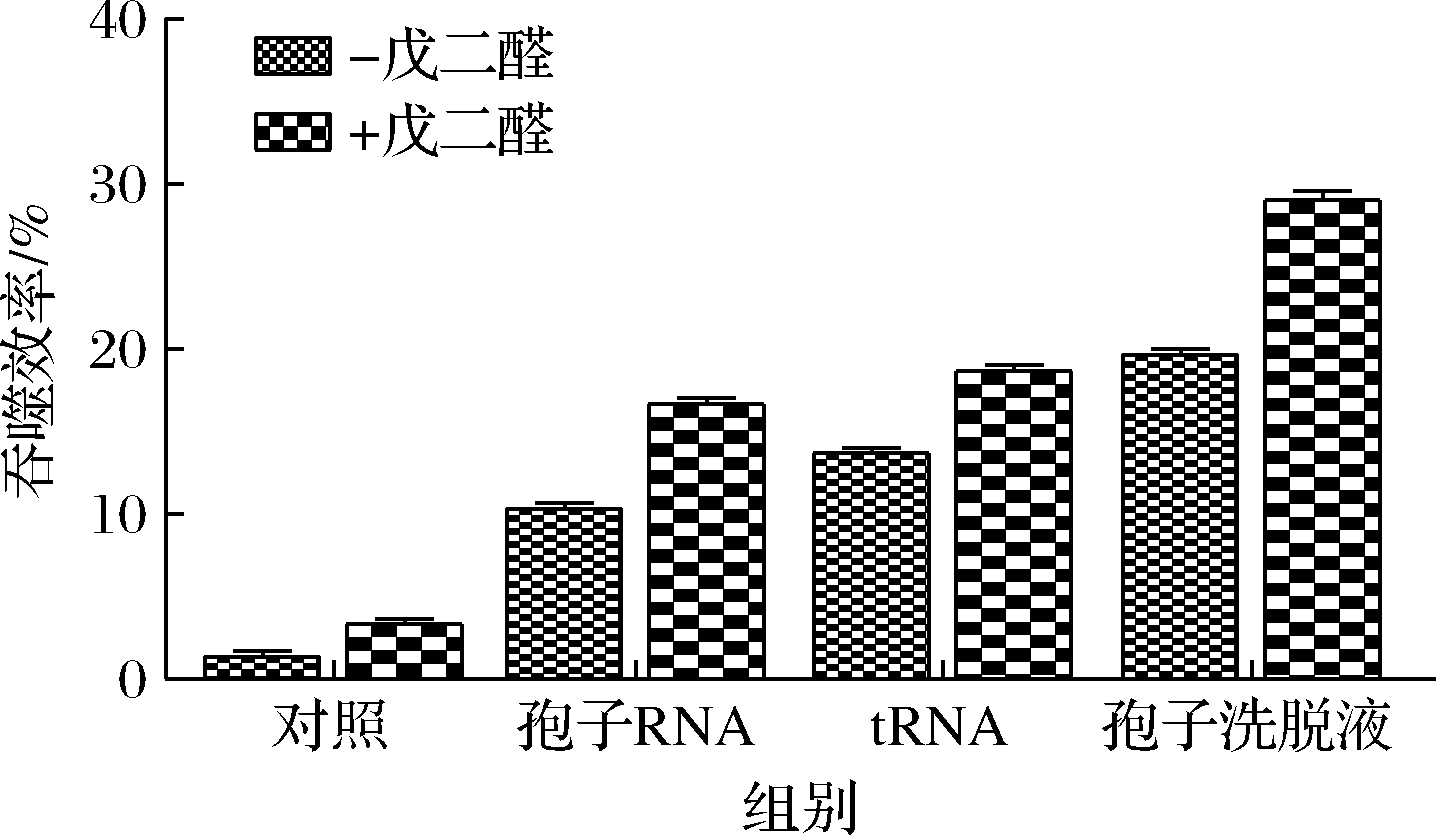
图6 戊二醛的加入对不同分子对于吞噬效率的影响
Fig.6 Effect of glutaraldehyde on phagocytosis efficiency of different molecules
2.5 酿酒酵母孢子表面分子洗脱液中NaCl浓度对吞噬作用影响
本研究选择用0.6 mol/L NaCl进行洗脱酿酒酵母孢子表面的分子,考虑到NaCl浓度影响,先前实验在洗脱后进行了稀释。在此实验中,加入了不稀释组,并在稀释组与不稀释组2组的最后体系中测量RNA和蛋白质的量,如表1所示,没有明显差异。在吞噬作用实验中,如图7所示,2组也不存在差异,因此NaCl浓度对于beads绑定分子吞噬没有影响。
表1 不同浓度NaCl洗脱液中蛋白浓度和RNA总量
Table 1 Protein concentration and total RNA in different concentrations of NaCl of spore eluent
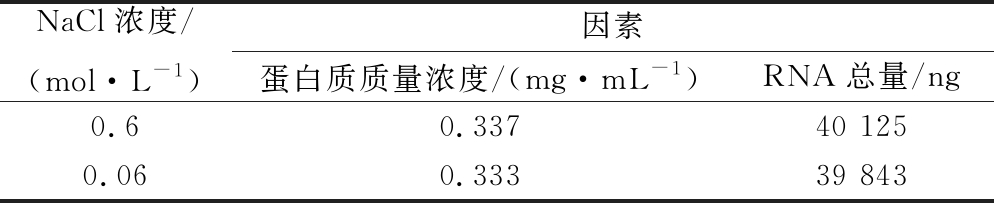
NaCl浓度/(mol·L-1)因素蛋白质质量浓度/(mg·mL-1)RNA总量/ng0.60.33740 1250.060.33339 843

图7 不同浓度NaCl对于吞噬效率的影响
Fig.7 Effects of different concentrations of NaCl on phagocytosis efficiency
2.6 RNase对酿酒酵母孢子表面分子促吞噬作用的影响
研究发现高盐洗脱液也可以诱导beads内化,是否高盐洗脱液中诱导内化的关键分子是RNA,为此用RNase处理了高盐洗脱液。研究中加入了RNase(0.5 U),在37 ℃放置1 h,再进行绑定交联然后吞噬观察结果,如图8所示,研究发现RNase的处理可以使吞噬数量大幅降低。因此酿酒酵母孢子表面洗脱液中RNA是调节内化的关键分子。

图8 RNase对酿酒酵母表面分子和beads吞噬效率的影响
Fig.8 Effects of RNase on the phagocytosis efficiency of beads with molecules from spore wall
3 讨论
首先,本研究发现酿酒酵母孢子表面的分子可以诱导2 μm颗粒被HEK293T细胞内吞,并对HEK293T细胞内化beads数量做了评估。进一步分析显示酿酒酵母孢子表面的RNA可显著影响beads的吞噬,并发现其他物种的RNA也具有诱导beads内化的功能,这说明RNA诱导beads内化具有普遍性。同时研究发现高盐洗脱液诱导beads内化的效率要比纯化后的RNA更为显著,这说明孢子表面的其他分子也具有调节beads内化的能力,对于该分子有待进一步研究和探讨。
酿酒酵母孢子表面结构复杂,由内到外依次是甘露糖层,葡聚糖层,壳聚糖层和二酪氨酸层,最新研究发现孢子表面具有脂滴等结构。0.6 mol/L NaCl 洗脱液中包含了除RNA以外的其他分子促进beads的吞噬,到底是什么分子促进了这一过程需要进一步的探索。在研究中,使用洗脱液孵育戊二醛处理过的beads的吞噬效率最高。对酿酒酵母孢子表面分子和非巨噬细胞的吞噬研究,将为未来药物运输的材料和传递提供新思路。
[1] NEIMAN A M.Ascospore formation in the yeast Saccharomyces cerevisiae[J].Microbiology and Molecular Biology Reviews, 2005, 69(4):565-584.
[2] 王晓文, 殷政, 高晓冬, 等.表达人源 HPD 基因对酿酒酵母孢子壁二酪氨酸层正确组装的干扰[J].食品与发酵工业, 2019, 45(15):17-23.
WANG X W, YIN Z, GAO X D, et al.Expression of human HPD gene interferes correct assembly of the dityrosine layer on the spore wall of Saccharomyces cerevisiae[J].Food and Fermentation Industries, 2019, 45(15):17-23.
[3] SHI L B, LI Z J, TACHIKAWA H, et al.Use of yeast spores for microencapsulation of enzymes[J].Applied and Environmental Microbiology, 2014, 80(15):4 502-4 510.
[4] BRIZA P, BREITENBACH M, ELLINGER A, et al.Isolation of two developmentally regulated genes involved in spore wall maturation in Saccharomyces cerevisiae[J].Genes & Development, 1990, 4(10):1 775-1 789.
[5] LONGTINE M S, RD M A K, DEMARINI D J, et al.Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae[J].Yeast, 1998, 14(10):953-961.
[6] NEIMAN A M. Sporulation in the budding yeast Saccharomyces cerevisiae[J].Genetics, 2011, 189(3):737-765.
[7] BASIONY M, YANG Y, LIU G Y, et al.Studies on the properties of the sporulation specific protein dit1 and its product formyl tyrosine[J].Journal of Fungi, 2020, 6(2):77.
[8] JIN Y F, OKAMOTO M, CHIBANA H, et al.Functional characteristics of Svl3 and Pam1 that are required for proper cell wall formation in yeast cells[J].Yeast, 2020, 37(7-8):359-371.
[9] YANG Y, WANG Q, LIU G Y, et al.Studies on the mechanism of non-phagocytic uptake of budding yeast spores in mammalian cells[C].The Joint Annual Meeting of 71st JSDB and 19th PSSJ.KOBE:Office of JSDB and PSSJ, 2019.
[10] HU J W, HAN J Z, LI H R, et al.Human embryonic kidney 293 cells:A vehicle for biopharmaceutical manufacturing, structural biology, and electrophysiology[J].Cells Tissues Organs, 2018, 205(1):1-8.
[11] SAKAGUCHI M, MURATA H, YAMAMOTO K, et al.TIRAP, an adaptor protein for TLR2/4, transduces a signal from RAGE phosphorylated upon ligand binding[J].PLoS One, 2011, 6(8):e23132.
[12] MEDRA O-FERNANDEZ I, REYES R, OLAZABAL I, et al.RIAM (Rap1-interacting adaptor molecule) regulates complement-dependent phagocytosis[J].Cellular and Molecular Life Sciences:CMLS, 2013, 70(13):2 395-2 410.
O-FERNANDEZ I, REYES R, OLAZABAL I, et al.RIAM (Rap1-interacting adaptor molecule) regulates complement-dependent phagocytosis[J].Cellular and Molecular Life Sciences:CMLS, 2013, 70(13):2 395-2 410.
[13] TANG C, ZHANG Y X, TANG D, et al.An easy NaV1.8 channel expression system based on HEK293T cell line[J].Toxicon, 2019, 158(20):518-524.
[14] ALIPANAHI B, DELONG A, WEIRAUCH M T, et al.Predicting the sequence specificities of DNA- and RNA-binding proteins by deep learning[J].Nature Biotechnology, 2015, 33(8):831-838.
[15] GAO C, HANES M S, BYRD-LEOTIS L A, et al.Unique binding specificities of proteins toward isomeric asparagine-linked glycans[J].Cell Chemical Biology, 2019, 26 (4):535-547.
[16] OKADA H, NEIMAN A M, OHYA Y.Assay for spore wall integrity using a yeast predator[J].Cold Spring Harbor protocols, 2016(8):138-142.
[17] LIU G Y, GAO X D, NAKANISHI H.Unique properties of the S.cerevisiae spore wall and its applications[J].Trends in Glycoscience and Glycotechnology, 2020, 32(190):E189-E193.
[18] RAHA S, MERANTE F, PROTEAU G, et al.Simultaneous isolation of total cellular RNA and DNA from tissue culture cells using phenol and lithium chloride[J].Gene Analysis Techniques, 1990, 7(7):173-177.
[19] SATO N, FUJIMURA T, MASUZAWA M, et al.Recombinant Mycobacterium leprae protein associated with entry into mammalian cells of respiratory and skin components[J].Journal of Dermatological Science, 2007, 46(2):101-110.