蒎烯是一种来自松柏类植物的天然单萜化合物,在调味剂、香料和农药中具有重要作用[1]。蒎烯二聚体因为其高体积能值的特性,可作为再生燃料替代航空燃料JP-10[2]。目前工业获得蒎烯的方法是从松柏类植物中提取,这种方法不仅浪费原料,还会产生大量的污染[1]。
蒎烯的生物合成是由焦磷酸香叶酯(geranyl pyrophosphate, GPP)经过蒎烯合酶催化而来[3],在酿酒酵母中GPP通过甲羟戊酸(mevalonate, MVA)通路合成[4],其中前体乙酰辅酶A的供应[5]、甲羟戊酸[6]、异戊烯焦磷酸(isopentenyl pyrophosphate, IPP)和二甲基烯丙焦磷酸酯(dimethylallyl pyrophosphate, DMAPP)的转化[7]是关键节点。由于GPP在酿酒酵母中会被转化为法尼基焦磷酸(farnesyl pyrophosphate, FPP),不利于蒎烯的合成,所以焦磷酸香叶酯合成酶突变体ERG20F96W-N127W被引入酿酒酵母提高GPP的含量[8]。陈天华等[9]通过上述方法在酿酒酵母中引入蒎烯合酶Pt30后发酵测得了蒎烯的产量,但是产量水平仍然很低。
蒎烯合酶作为蒎烯合成通路关键酶对蒎烯的最终产量产生直接的影响[10]。蒎烯合酶的研究目前主要集中在大肠杆菌中异源表达,来自火炬松、大冷杉等松柏类植物的蒎烯合酶在大肠杆菌中的表达已见报道[10-11]。但是野生型蒎烯合酶在异源表达中不能收到良好的效果,有研究发现蒎烯合酶Pt1的Q457位点的突变可以影响到其对Mn2+的偏好性,从而提高酶活性和蒎烯产量。虽然酿酒酵母是合成萜类物质的优良宿主[12-17],但蒎烯合酶Pt30在酿酒酵母中的表达也没有很好的效果[9]。基于此,本研究对蒎烯合酶Pt30的保守性Q456位点进行饱和突变以获得更好的突变体,为高效生产蒎烯提供借鉴。
1 材料与方法
1.1 实验材料
本实验使用的菌株和质粒见表1。PrimeSTAR®HS DNA高保真聚合酶、限制性核酸内切酸(BamH I、EcoR I、Not I、Nhe I、Kpn I等),大连宝生物有限公司;ABclonal MultiF Seamless Assembly,武汉爱博泰克生物科技有限公司;氨苄青霉素(ampicillin, Amp)、酵母无氨基氮源(yeast nitrogen base W/O amino acids, YNB)、氨基酸(亮氨酸、色氨酸、组氨酸、尿嘧啶),上海生工生物工程股份有限公司;蒎烯标准品,Sigma-aldrich公司;Tryptone、Yeast Extract,Oxiod公司;质粒提取试剂盒、DNA片段回收和凝胶回收试剂盒,上海捷瑞生物工程有限公司;TSQ8000气相质谱联用仪,美国赛默飞世尔科技公司;GC-2014岛津气相色谱仪,日本岛津实验器材有限公司;HP-5MS气相色谱柱,安捷伦科技有限公司;UV-2000可见分光光度计,尤尼柯仪器有限公司。
表1 本研究使用的菌株和质粒
Table 1 Strains and plasmids used in this study

菌株(质粒)相关特性来源E.coli JM109基因克隆宿主TaKaRaS.cerevisiae PK2-1CMATa ura3-52 trp1-289 leu2-3_112 his3Δ1 MAL2-8c SUC2实验室保藏Sc.MPSS.cerevisiae PK2-1C YPL062 WΔ::KanMX δ::PTEF1-tHMG1,PGAP-IDI1, LEU2, NTS::PTDH3-ERG20WW, PPGK1-IDI1, HIS实验室保藏p424-PTEF12 μ, PTEF1, TCYC1, TRP1, Amp实验室保藏p424-tPt30p424-PTEF1- tPt30(48 amino acid truncation) 本研究p424-tPt30Gp424-PTEF1- tPt30(Q456G)本研究p424-tPt30Ap424-PTEF1- tPt30(Q456A)本研究p424-tPt30Vp424-PTEF1- tPt30(Q456V)本研究p424-tPt30Lp424-PTEF1- tPt30(Q456L)本研究p424-tPt30Ip424-PTEF1- tPt30(Q456I)本研究p424-tPt30Pp424-PTEF1- tPt30(Q456P)本研究p424-tPt30Fp424-PTEF1- tPt30(Q456F)本研究p424-tPt30Yp424-PTEF1- tPt30(Q456Y)本研究p424-tPt30 Wp424-PTEF1- tPt30(Q456 W)本研究p424-tPt30Sp424-PTEF1- tPt30(Q456S)本研究p424-tPt30Tp424-PTEF1- tPt30(Q456T)本研究p424-tPt30Cp424-PTEF1- tPt30(Q456C)本研究p424-tPt30Mp424-PTEF1- tPt30(Q456M)本研究p424-tPt30 Np424-PTEF1- tPt30(Q456 N)本研究p424-tPt30Dp424-PTEF1- tPt30(Q456D)本研究p424-tPt30Ep424-PTEF1- tPt30(Q456E)本研究p424-tPt30Kp424-PTEF1- tPt30(Q456K)本研究p424-tPt30Rp424-PTEF1- tPt30(Q456R)本研究p424-tPt30Hp424-PTEF1- tPt30(Q456H)本研究
1.2 引物
本研究使用的引物见表2。
表2 本研究使用的引物
Table 2 Primers used in this study

注:F表示上游引物;R表示下游引物;下划线部分为突变位点核苷酸的序列
引物名称引物序列(5'-3')tPt30-FCTAAGTTTTCTAGAACTAGTGGATCCATGATG-TCTATGTCTAGTACCACCAGTGtPt30-RCGAATTCCTGCAGCCCGGGGCGGCCGCT-TAAAGTGGGACAGTTTCAACGACGQ456G-FGTGCCTACATCGGAGAAGCCAAGTGGATCAGTAGTGGQ456G-RCACTTGGCTTCTCCGATGTAGGCACCGATAAAGGCCQ456A-FGTGCCTACATCGCCGAAGCCAAGTGGATCAGTAGTGGQ456A-RCACTTGGCTTCGGCGATGTAGGCACCGATAAAGGCCQ456V-FGTGCCTACATCGTCGAAGCCAAGTGGATCAGTAGTGGQ456V-RCACTTGGCTTCGACGATGTAGGCACCGATAAAGGCCQ456I-FGTGCCTACATCATCGAAGCCAAGTGGATCAGTAGTGGQ456I-RCACTTGGCTTCGATGATGTAGGCACCGATAAAGGCCQ456P-FGTGCCTACATCCCTGAAGCCAAGTGGATCAGTAGTGGQ456P-RCACTTGGCTTCAGGGATGTAGGCACCGATAAAGGCCQ456F-FGTGCCTACATCTTCGAAGCCAAGTGGATCAGTAGTGGQ456F-RCACTTGGCTTCGAAGATGTAGGCACCGATAAAGGCCQ456Y-FGTGCCTACATCTATGAAGCCAAGTGGATCAGTAGTGGQ456Y-RCACTTGGCTTCATAGATGTAGGCACCGATAAAGGCCQ456W-FGTGCCTACATCTGGGAAGCCAAGTGGATCAGTAGTGGQ456W-RCACTTGGCTTCCCAGATGTAGGCACCGATAAAGGCCQ456S-FGTGCCTACATCAGTGAAGCCAAGTGGATCAGTAGTGGQ456S-RCACTTGGCTTCACTGATGTAGGCACCGATAAAGGCCQ456T-FGTGCCTACATCACCGAAGCCAAGTGGATCAGTAGTGGQ456T-RCACTTGGCTTCGGTGATGTAGGCACCGATAAAGGCCQ456C-FGTGCCTACATCTGTGAAGCCAAGTGGATCAGTAGTGGQ456C-RCACTTGGCTTCACAGATGTAGGCACCGATAAAGGCCQ456M-FGTGCCTACATCATGGAAGCCAAGTGGATCAGTAGTGGQ456M-RCACTTGGCTTCCATGATGTAGGCACCGATAAAGGCCQ456N-FGTGCCTACATCAACGAAGCCAAGTGGATCAGTAGTGGQ456N-RCACTTGGCTTCGTTGATGTAGGCACCGATAAAGGCCQ456D-FGTGCCTACATCGACGAAGCCAAGTGGATCAGTAGTGGQ456D-RCACTTGGCTTCGTCGATGTAGGCACCGATAAAGGCCQ456E-FGTGCCTACATCGAAGAAGCCAAGTGGATCAGTAGTGGQ456E-RCACTTGGCTTCTTCGATGTAGGCACCGATAAAGGCCQ456K-FGTGCCTACATCAAGGAAGCCAAGTGGATCAGTAGTGGQ456K-RCACTTGGCTTCCTTGATGTAGGCACCGATAAAGGCCQ456R-FGTGCCTACATCAGAGAAGCCAAGTGGATCAGTAGTGGQ456R-RCACTTGGCTTCTCTGATGTAGGCACCGATAAAGGCCQ456H-FGTGCCTACATCCATGAAGCCAAGTGGATCAGTAGTGGQ456H-RCACTTGGCTTCATGGATGTAGGCACCGATAAAGGCC
1.3 培养基与培养方法
LB培养基(g/L):酵母提取物5,蛋白胨10,氯化钠10。
YPD培养基(g/L):酵母浸膏10,蛋白胨20,葡萄糖20。
发酵培养基:YNB 6.7 g/L,葡萄糖20 g/L,尿嘧啶200 mg/L。
培养方法:挑取单菌落,接种于10 mL发酵培养基进行活化,30 ℃,200 r/min培养24 h;以2%的接种量再转接到10 mL发酵培养基培养24 h作为种子液;将得到的种子液转接到装有50 mL发酵培养基的250 mL锥形瓶中,控制起始生物量OD600为0.1。待菌株生长12 h后,添加10%的十二烷进行两相发酵[12];发酵周期为120 h。期间每24 h取样1 mL测定菌株的生长情况,待发酵结束后测定上层十二烷中的蒎烯产量。
1.4 表达质粒的构建
蒎烯和酶基因Pt30通过苏州金唯智公司合成,以p424-PTEF1为载体,经BamH I单酶切后,利用同源重组酶进行连接,构建成p424-tPt30。Q456位点的点突变通过反向重叠PCR实现,以p424-tPt30为模板,PCR获得点突变的片段,经Dpn I酶消化后转化大肠杆菌,提取转化子质粒并测序验证。
1.5 测定方法
本研究生物量以OD600的值进行表达,通过分光光度计测定。
蒎烯的含量通过气相测定:蒎烯标品建立蒎烯标准曲线,对发酵蒎烯进行定量;气相色谱程序如下:进样口温度与FID检测器温度均为260 ℃;柱温升温程序为:起始温度70 ℃,保持3 min;然后以5 ℃/min升至100 ℃,保持3 min;最后以40 ℃/min升至250 ℃, 保持5 min;进样量为1 μL;载体为氮气;分流比为1∶20。
2 结果与分析
2.1 蒎烯合酶赋予酿酒酵母蒎烯合成能力
酿酒酵母中天然存在MVA通路,是合成萜类物质的优良的宿主;蒎烯合成通路在酿酒酵母中缺少蒎烯合酶催化GPP合成蒎烯。首先我们按照酿酒酵母的密码子偏好性对蒎烯合酶Pt30进行密码子优化经苏州金唯智公司合成,并通过信号肽预测将得到的蒎烯合酶Pt30的N端48个氨基酸截断后构建了蒎烯合酶表达质粒;将其直接转化酿酒酵母,构建具有完整的蒎烯合成通路的工程酵母,将得到的工程菌进行两相发酵,气相测定的结果中并没有检测到蒎烯的产生。已知GPP作为单萜类物质的直接前体物质,在酿酒酵母中会转化成FPP,造成胞内的含量很微弱[8],可能造成蒎烯的产量太少,达不到气相的检测限。因此,气相质谱分析被用于发酵结果的检测,质谱分析结果见图1,与标准品对比确定检测到了蒎烯的存在。
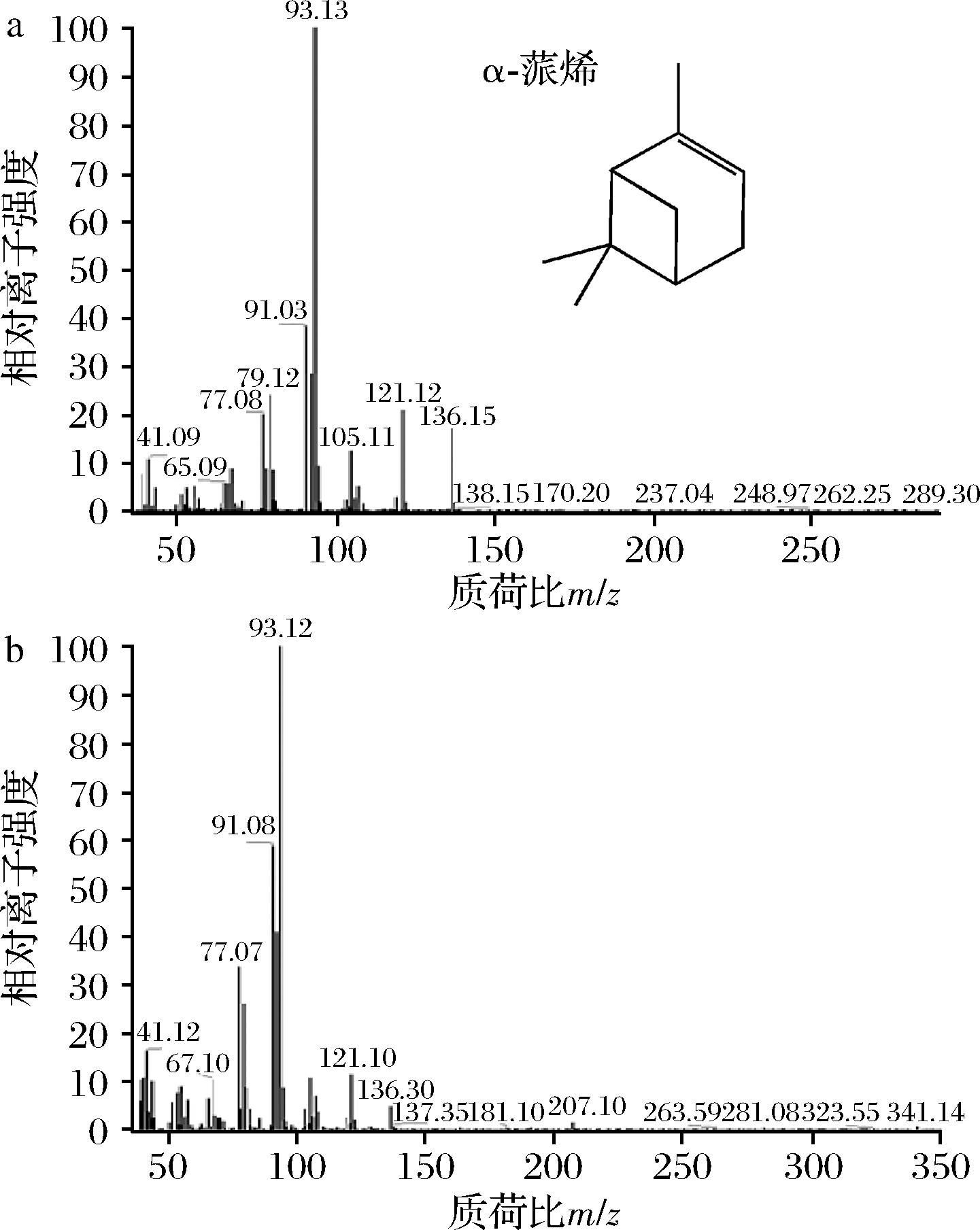
a-蒎烯标准品的质谱图;b-样品的质谱图
图1 两相发酵十二烷有机相气相质谱结果
Fig.1 Analysis of pinene yield in dodecane phase by GC-MC
2.2 蒎烯合酶突变促进蒎烯合成
为了提高蒎烯的合成,将蒎烯合酶表达质粒转化到构建的强化MVA通路的菌株Sc.MPS中,经过两相发酵后,在蒎烯标准品的保留时间还是没有检测到蒎烯的出峰,可能是蒎烯合酶的活性在酿酒酵母中太低。为了提高蒎烯合酶在酿酒酵母中的活性,考虑对蒎烯合酶进行突变。有文献报道,与Pt30同样来自于火炬松的蒎烯合酶Pt1,在Q457位点的突变可以改变Pt1的离子偏好性[11],从而提高其在大肠杆菌中的酶活性。野生型的Pt30酶需要Mn2+较高的胞内环境保持高酶活性[18],在酿酒酵母中胞内Mn2+浓度在2~10 μmol/L,不足以维持蒎烯合酶的高活性[19]。Pt30在酿酒酵母中表达时,在共表达ERG20WW的情况下,蒎烯的产量只有0.329 mg/L[9];与ERG20WW融合表达之后产量也只有6.55 mg/L[9];这是关于蒎烯合酶表达在酿酒酵母中唯一的报道。所以我们将包括这2个蒎烯合酶的9种蒎烯合酶进行氨基酸对比,结果如图2所示,发现在Q457位点附近具有保守性,所以将Pt30的Q456位点被饱和突变并在蒎烯合成通路强化的菌株中过表达,希望改变蒎烯合酶Pt30在酿酒酵母中的表现。经过两相发酵后,大多数的突变体还是不能检测到样品在蒎烯保留时间的出峰,但是Pt30(Q456I)和Pt30(Q456K)检测到了明显蒎烯出峰,并通过蒎烯标准品获得的蒎烯标准曲线确定了蒎烯的产量,结果见图3,蒎烯的产量分别为0.041、0.017 mg/L。这2个突变体明显提高了蒎烯合酶在酿酒酵母中生产蒎烯的能力,实现了气相检测的从无到有的质的变化。如图4所示,我们还发现在2株突变体发酵的过程中,蒎烯产量较高的Pt30(Q456I)突变体发酵时的生长弱于Pt30(Q456K)突变体,可能是由于合成了更多的蒎烯,造成生长曲线的显著延后,这也合理解释了蒎烯对酵母菌株的毒性。我们利用COACH-D[20-21]对Pt30建立蛋白模型,如图3-a所示,发现Q456位点位于蛋白外围的一个α螺旋上,离活性中心较远,但是与Mn2+的结合位点距离很近,从突变后侧链的结果来看,原来的Q456存在一个碳氧双键,2个突变体的侧链结构更加简单,但是对于结构更加简单的蛋白的突变没有获得更优的结果,可能归因于侧链的长度对Mn2+的结合位点的影响。推测可能突变后的蛋白对离子的结合强度发生了变化,一定程度上改变了蒎烯合酶对Mn2+的依赖,从而通过对这一位点的突变确实改变了蒎烯合酶在酿酒酵母中的表现,这项成果也为未来理性设计蒎烯合酶提供了一定的借鉴。
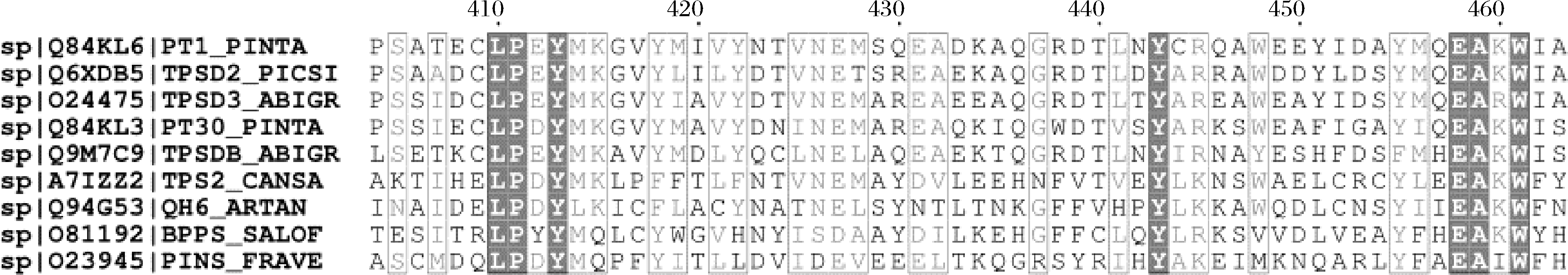
图2 蒎烯合酶的氨基酸序列对比
Fig.2 Multiple sequence comparison of pinene synthase
注:sp | UniProt ID | 蛋白命名_来源

a-Pt30(Q456I)和Pt30(Q456K)蛋白模型;b-Pt30(Q456I)和Pt30(Q456K)蒎烯产量
图3 突变体Pt30(Q456I)和Pt30(Q456K)提高酿酒酵母蒎烯产量
Fig.3 Mutants of Pt30 (Q456I) and Pt30 (Q456K) increase pinene titer in S.cerevisiae
注:图3-a中圆球为Mn2+
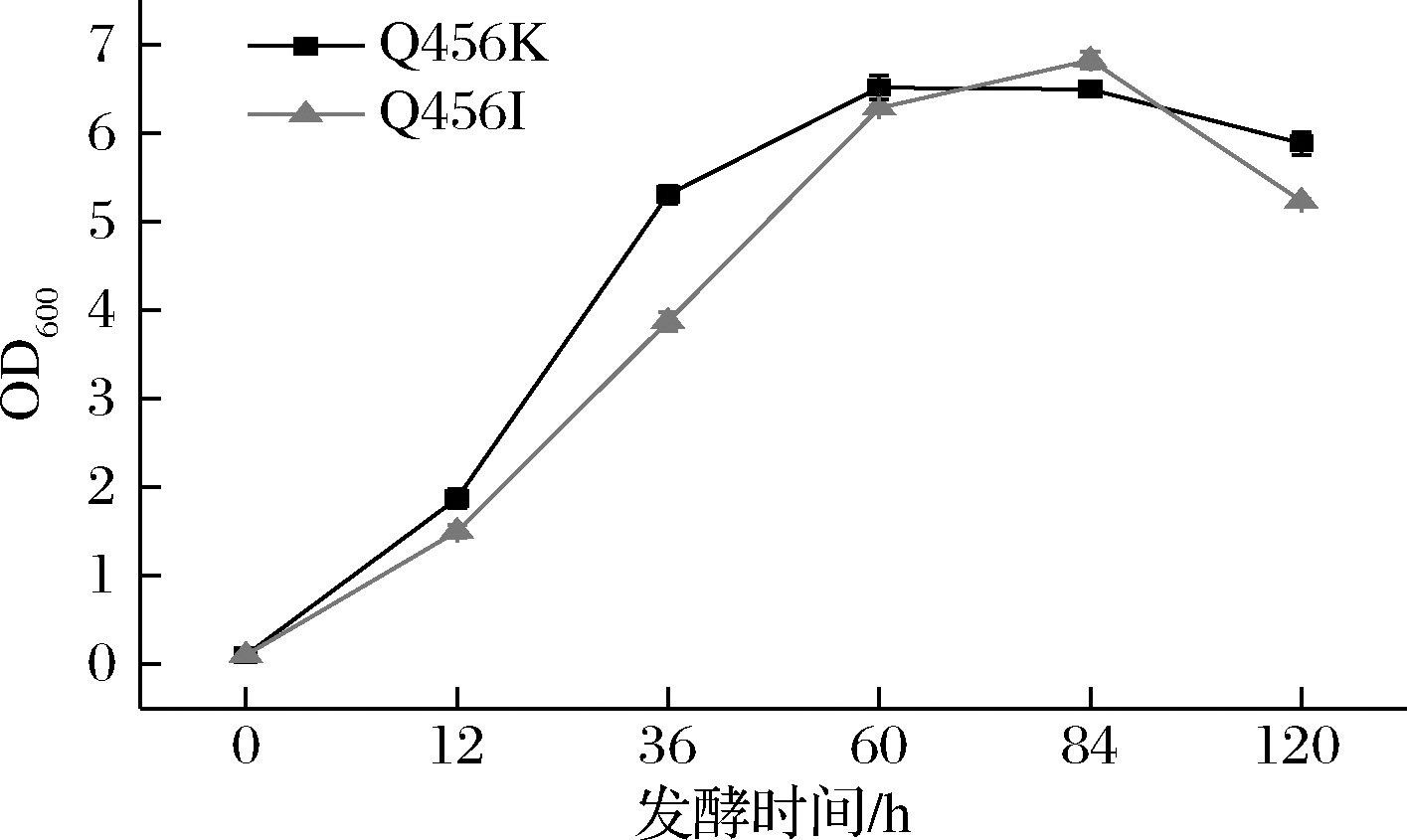
图4 突变体Pt30(Q456I)和Pt30(Q456K)两相发酵的生长曲线
Fig.4 Growth curve of Pt30 (Q456I) and Pt30 (Q456K) during two-phase fermentation
3 结论
蒎烯是一个应用前景极大的单萜类化合物,对于其绿色生物合成的研究还亟待挖掘,本研究首先将蒎烯合酶引入酿酒酵母中,通过质谱分析检测到蒎烯的存在,确定酿酒酵母合成蒎烯的能力。通过进一步将蒎烯合酶表达质粒在MVA通路强化的菌株中表达,还是没有通过气相检测到蒎烯的出峰。为了提高蒎烯合酶在酿酒酵母中的活性,通过比较包括Pt1和Pt30在内的9种蒎烯合酶的氨基酸序列,发现蒎烯合酶在Q457位点附近具有保守性。为了提高蒎烯合酶的在酿酒酵母中的表现,在构建的蒎烯合成通路强化的酿酒酵母菌株中,过表达构建的蒎烯合酶Pt30的Q456位点饱和突变体,希望获得在酿酒酵母胞内表现更好的蒎烯合酶,经过两相发酵后,发现Pt30(Q456I)和Pt30(Q456K)的点突变,显著提高了酿酒酵母合成蒎烯的产量,这证明了Q456位点对Pt30的重要性。虽然目前获得的蒎烯产量无法与共表达ERG20WW及融合表达ERG20WW相比,但是解决蒎烯合酶的活性问题对于蒎烯高效合成更加重要。对蒎烯合酶的建模发现Q456这一位点在蛋白中的Mn2+的结合位点附近,对酿酒酵母蒎烯合成的影响很大,本研究获得的蒎烯合酶的突变体也可以为酿酒酵母高效合成蒎烯提供了借鉴。
[1] BEHR A, JOHNEN L.Myrcene as a natural base chemical in sustainable chemistry:A critical review[J].ChemSusChem, 2009, 2(12):1 072-1 095.
[2] HARVEY B G, WRIGHT M E, QUINTANA R L.High-density renewable fuels based on the selective dimerization of pinenes[J].Energy & Fuels, 2010, 24(1):267-273.
[3] YANG J M, NIE Q J, REN M, et al.Metabolic engineering of Escherichia coli for the biosynthesis of alpha-pinene[J].Biotechnology for Biofuels, 2013, 6(1):60.
[4] VICKERS C E, WILLIAMS T C, PENG B Y, et al.Recent advances in synthetic biology for engineering isoprenoid production in yeast[J].Current Opinion in Chemical Biology, 2017, 40:47-56.
[5] CHEN Y, WANG Y, LIU M, et al.Primary and secondary metabolic effects of a key gene deletion (ΔYPL062W) in metabolically engineered terpenoid-producing Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2019, 85(7).DOI:10.1128/aem.01990-18.
[6] IGNEA C, CVETKOVIC I, LOUPASSAKI S, et al.Improving yeast strains using recyclable integration cassettes, for the production of plant terpenoids[J].Microbial Cell Factories, 2011, 10(1):1-18.
[7] LIU J D, ZHANG W P, DU G C, et al.Overproduction of geraniol by enhanced precursor supply in Saccharomyces cerevisiae[J].Journal of Biotechnology, 2013, 168(4):446-451.
[8] IGNEA C, PONTINI M, MAFFEI M E, et al.Engineering monoterpene production in yeast using a synthetic dominant negative geranyl diphosphate synthase[J].ACS Synthetic Biology, 2014, 3(5):298-306.
[9] 陈天华, 张若思, 姜国珍, 等.产蒎烯人工酵母细胞的构建[J].化工学报, 2019, 70(1):179-188.
CHEN T H, ZHANG R S, JIANG G Z, et al.Metabolic engineering of Saccharomyces cerevisiae for pinene production[J].CIESC Journal, 2019, 70(1):179-188.
[10] SARRIA S, WONG B, MART N H G, et al.Microbial synthesis of pinene[J].ACS Synthetic Biology, 2014, 3(7):466-475.
N H G, et al.Microbial synthesis of pinene[J].ACS Synthetic Biology, 2014, 3(7):466-475.
[11] TASHIRO M, KIYOTA H, KAWAI-NOMA S, et al.Bacterial production of pinene by a laboratory evolved pinene synthase[J].ACS Synthetic Biology, 2016, 5(9):1 011-1 020.
[12] YEE D A, DENICOLA A B, BILLINGSLEY J M, et al.Engineered mitochondrial production of monoterpenes in Saccharomyces cerevisiae[J].Metabolic Engineering, 2019, 55:76-84.
[13] LIU T, ZHANG C B, LU W.Heterologous production of levopimaric acid in Saccharomyces cerevisiae[J].Microbial Cell Factories, 2018, 17(1):1-10.
[14] CATALDO V F, ARENAS N, SALGADO V, et al.Heterologous production of the epoxycarotenoid violaxanthin in Saccharomyces cerevisiae[J].Metabolic Engineering, 2020, 59:53-63.
[15] JIANG G Z, YAO M D, WANG Y, et al.Manipulation of GES and ERG20 for geraniol overproduction in Saccharomyces cerevisiae[J].Metabolic Engineering, 2017, 41:57-66.
[16] CHENG S, LIU X, JIANG G Z, et al.Orthogonal engineering of biosynthetic pathway for efficient production of limonene in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2019, 8(5):968-975.
[17] LINDAHL A L, OLSSON M E, MERCKE P, et al.Production of the artemisinin precursor amorpha-4,11-diene by engineered Saccharomyces cerevisiae[J].Biotechnoligy Letter, 2006, 28(8):571-580.
[18] PHILLIPS M A, WILDUNG M R, WILLIAMS D C, et al.cDNA isolation, functional expression, and characterization of (+)-α-pinene synthase and (-)-α-pinene synthase from loblolly pine (Pinus taeda):Stereocontrol in pinene biosynthesis[J].Archives of Biochemistry and Biophysics, 2003, 411(2):267-276.
[19] JONES R P, GADD G M.Ionic nutrition of yeast-physiological mechanisms involved and implications for biotechnology[J].Enzyme and Microbial Technology, 1990, 12(6):402-418.
[20] WU Q, PENG Z L, ZHANG Y, et al.COACH-D:Improved protein-ligand binding sites prediction with refined ligand binding poses through molecular docking[J].Nucleic Acids Research, 2018, 46(W1):W438-W442.
[21] YANG J Y, ROY A, ZHANG Y.Protein-ligand binding site recognition using complementary binding-specific substructure comparison and sequence profile alignment[J].Bioinformatics, 2013, 29(20):2 588-2 595.