酪醇[2-(4-羟基苯基)乙醇,tyrosol]是一种天然的多酚类物质,广泛存在于红景天、女贞、橄榄等植物中,具有抗氧化、消炎等多种药理活性[1]。目前酪醇的合成主要有植物提取、化学合成及微生物代谢生产3种途径。由于植物提取率低、化学合成反应复杂等问题的存在,微生物细胞工厂的优势逐渐凸显。在微生物代谢中,酪醇是酪氨酸代谢途径的下游产物,是橄榄苦苷、羟基酪醇、红景天苷等重要天然产物的关键前体。微生物中酪醇的合成途径主要有3种:(1) SATOH等[2]在大肠杆菌中表达植物罂粟(Papaver somniferum)来源的酪氨酸脱羧酶(tyrosine decarboxylase,TDC)和藤黄微球菌(Micrococcus luteus)来源的酪胺氧化酶(tyramine oxidase,TYO),并结合大肠杆菌内源的乙醇脱氢酶(alcohol dehydrogenase,ADH),实现了酪醇的从头合成,产量达到69 mg/L;(2) YANG等[3]在大肠杆菌中过量表达来源于酿酒酵母的苯丙酮酸脱羧酶(phenylpyruvate decarboxylase,ARO10),得到了800.40 mg/L 酪醇;(3) CHUNG等[4]对拟南芥(Arabidopsis thaliana)、矮牵牛(Petunia hybrid)和荷兰芹(Petroselinum crispum)3种来源的芳香醛合成酶(aromatic aldehyde synthase,AAS)进行了检测,发现表达PcAAS菌株的产量最高,达12.9 mg/L。在这3条途径中,仅AAS可以将酪氨酸一步转化为生产酪醇所需的直接前体物质。因此,本研究将从此途径入手开展酪醇的生物合成研究。
ZHOU等[5-6]在大肠杆菌基因组中挖掘得到一系列梯度强度的组成型启动子,并用于强化己二酸的生产。为了提升酪醇的产量,本研究以E.coli BL21 (DE3)为出发菌株,使用组成型启动子PcspA[5]取代PT7启动子,并运用该质粒过量表达PcAAS来构建酪醇合成的生物途径。将调控分支代谢途径基因敲除阻断竞争途径和过量表达乙醇脱氢酶来强化代谢流的方法结合起来,通过使用高、中、低3种不同强度的组成型启动子对途径中的关键酶AAS和ADH进行组合调控,实现了大肠杆菌合成酪醇的代谢途径优化。研究结果为后续采用发酵法生产酪醇和相关的下游产物奠定了良好的基础。
1 材料与方法
1.1 材料
1.1.1 菌株及质粒
本研究使用及构建的菌株和质粒分别见表1和表2。
表1 本研究使用的菌株
Table 1 Strains used in this study
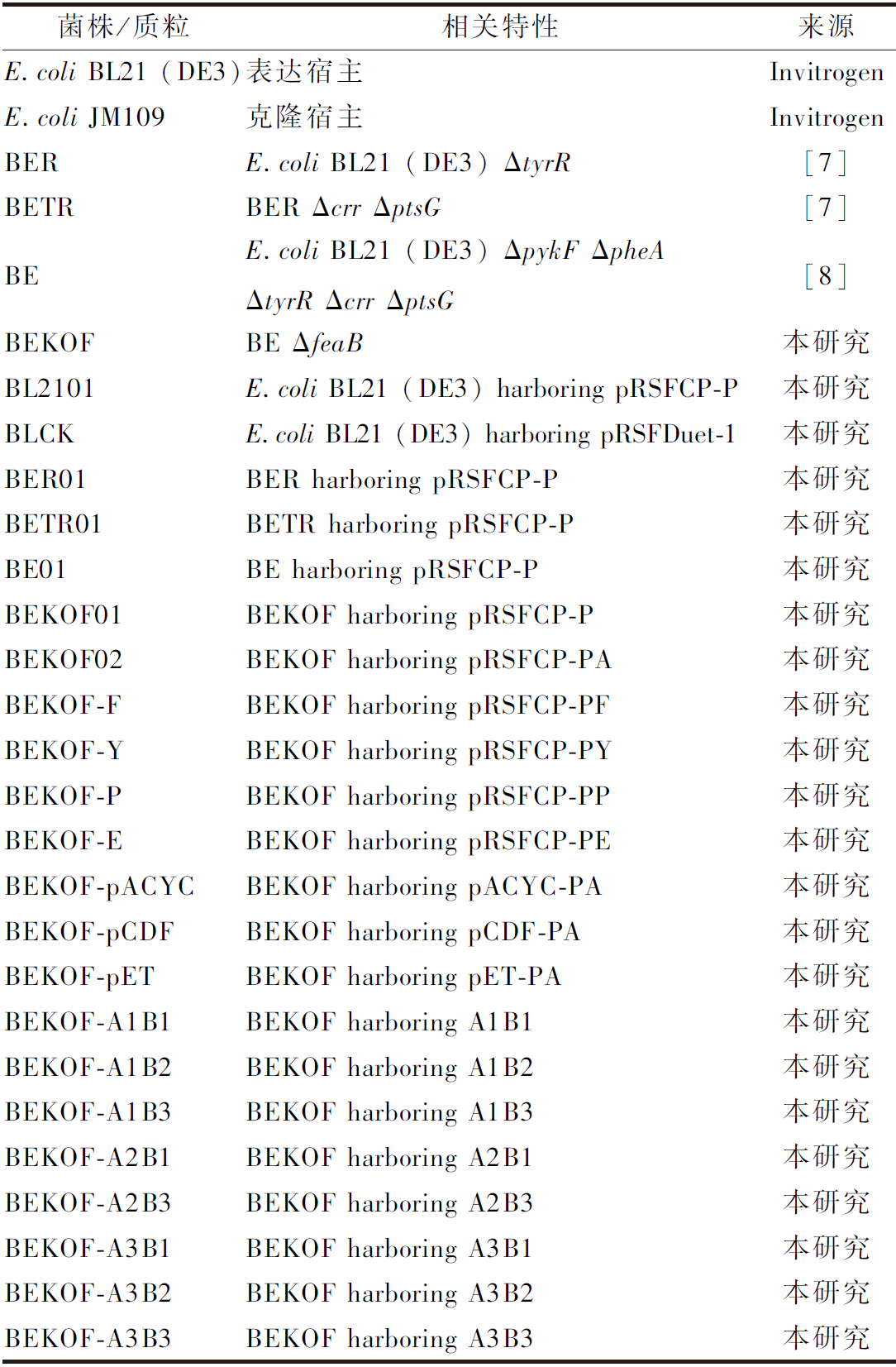
菌株/质粒相关特性来源E.coli BL21 (DE3)表达宿主InvitrogenE.coli JM109克隆宿主InvitrogenBERE.coli BL21 (DE3) ΔtyrR[7]BETRBER Δcrr ΔptsG[7]BEE.coli BL21 (DE3) ΔpykF ΔpheAΔtyrR Δcrr ΔptsG[8]BEKOFBE ΔfeaB本研究BL2101E.coli BL21 (DE3) harboring pRSFCP-P本研究BLCKE.coli BL21 (DE3) harboring pRSFDuet-1本研究BER01BER harboring pRSFCP-P本研究BETR01BETR harboring pRSFCP-P本研究BE01BE harboring pRSFCP-P本研究BEKOF01BEKOF harboring pRSFCP-P本研究BEKOF02BEKOF harboring pRSFCP-PA本研究BEKOF-FBEKOF harboring pRSFCP-PF本研究BEKOF-YBEKOF harboring pRSFCP-PY本研究BEKOF-PBEKOF harboring pRSFCP-PP本研究BEKOF-EBEKOF harboring pRSFCP-PE本研究BEKOF-pACYCBEKOF harboring pACYC-PA本研究BEKOF-pCDFBEKOF harboring pCDF-PA本研究BEKOF-pETBEKOF harboring pET-PA本研究BEKOF-A1B1BEKOF harboring A1B1本研究BEKOF-A1B2BEKOF harboring A1B2本研究BEKOF-A1B3BEKOF harboring A1B3本研究BEKOF-A2B1BEKOF harboring A2B1本研究BEKOF-A2B3BEKOF harboring A2B3本研究BEKOF-A3B1BEKOF harboring A3B1本研究BEKOF-A3B2BEKOF harboring A3B2本研究BEKOF-A3B3BEKOF harboring A3B3本研究
表2 本研究使用的质粒
Table 2 Plasmids used in this study

质粒相关特性来源pRSFDuet-1Vector, PT7 promoter, KanRInvitrogenpETDuet-1Vector, PT7 promoter, AmpRInvitrogenpACYCDuet-1Vector, PT7 promoter, CmRInvitrogenpCDFDuet-1Vector, PT7 promoter, StrRInvitrogenpRSFCPPT7 promoter replaced by constitutive promoter cspA本研究pRSFCP-PpRSFCP derivative with aas gene from P. crispum, KanR本研究pRSFCP-PApRSFCP-P derivative withadh6 gene from Saccharomyces cerevisiae, KanR本研究pRSFCP-PPpRSFCP-P derivative with adhP gene from E. coli, KanR本研究pRSFCP-PYpRSFCP-P derivative with yahK gene from E. coli, KanR本研究pRSFCP-PFpRSFCP-P derivative with frmA gene from E. coli, KanR本研究pRSFCP-PEpRSFCP-P derivative with adhE gene from E. coli, KanR本研究pACYC-PAthe origin of pRSFCP-PA replaced by p15A ori本研究pCDF-PAthe origin of pRSFCP-PA replaced by CDF ori本研究pET-PAthe origin of pRSFCP-PA replaced by pBR322 ori本研究A1B1aas promoted by PyjeFE and adh6 promoted by PyjeFE本研究A1B2aas promoted by PyjeFE and adh6 promoted by PcspA本研究A1B3aas promoted by PyjeFE and adh6 promoted by Pinfc-rplT本研究A2B1aas promoted by PcspA and adh6 promoted by PyjeFE本研究A2B3aas promoted by PcspA and adh6 promoted by Pinfc-rplT本研究A3B1aas promoted by Pinfc-rplT and adh6 promoted by PyjeFE本研究A3B2aas promoted by Pinfc-rplT and adh6 promoted by PcspA本研究A3B3aas promoted by Pinfc-rplT and adh6 promoted by Pinfc-rplT本研究
1.1.2 培养基及试剂
种子培养基(g/L):胰蛋白胨10.0,酵母粉5.0,NaCl 10.0。
发酵培养基F1(g/L):葡萄糖 25.0,甘油 10.0,(NH4)2SO4 7.5,K2HPO4·3H2O 3.0,KH2PO4 2.0,MgSO4·7H2O 1.0,柠檬酸钠 1.0,维生素B1 0.1,酵母粉 7.0,微量元素营养液1 mL/L,依据需求加入适量的抗生素。
微量元素营养液(g/L):Al2(SO4)3·18H2O 2.0,CoSO4·7H2O 0.75,CuSO4·5H2O 2.5,H3BO3 0.5,MnSO4·H2O 24.0,NiSO4·6H2O 2.5,ZnSO4·7H2O 15.0。
酵母粉,Oxoid公司;胰蛋白胨及所有使用的抗生素粉末,上海生工有限公司;酪醇标准品,阿拉丁;其余试剂购自国药集团。
1.2 实验方法
1.2.1 基因组编辑方法
基因组编辑方法参考JIANG等[9]的研究。敲除feaB基因(phenylacetaldehyde dehydrogenase gene)所用的sgRNA序列为5′-CGATCTTTGATCCGGCCACC-3′,其PAM序列为GGG,所用相关引物如表3所示。
表3 基因敲除使用的引物
Table 3 Primers for gene knockout

引物名称序列(5'-3')FEAB-UP-FCGGTGTAAAACCAGGCATCGFEAB-UP-RCCGCCTGCTCAAATCCACTAACCGTGTTGACGATCGAGAAACFEAB-DOWN-FTTCTCGATCGTCAACACGGTTAGTGGATTTGAGCAGGCGGFEAB-DOWN-RACACACACCGACTTAGTTTCACPTARGET-FEAB-FCGATCTTTGATCCGGCCACCGTTTTAGAGCTAGAAATAGCAPTARGET-FEAB-RGGTGGCCGGATCAAAGATCGACTAGTATTATACCTAGGACTGAGCT
1.2.2 重组质粒的构建方法
质粒构建相关引物如表4所示。质粒构建采用Gibson组装的方法进行。首先通过简单PCR获得目的片段,对得到的PCR产物纯化后使用Gibson组装酶进行连接。将连接产物采用热激法转化E.coli JM109,次日挑取转化子进行菌落PCR并将得到的阳性克隆进行Sanger测序。
表4 质粒构建使用的引物
Table 4 Primers for plasmid constructions

引物名称序列 (5'-3')pACYCDuet-FGGTACCCTCGAGTCTGGTAAAGAAApACYCDuet-RTCACTCATTAGGCACCGGGAAAS/ADH-F (pACYC)TCCCGGTGCCTAATGAGTGAGCTAACTTACATTAATTGCGTTGCAAS/ADH-R (pACYC)TTTCTTTACCAGACTCGAGGGTACCCTAGTCTGAAAATTCTTTGTCGTAGCA2-cspA-F (pRSF)CTTACATTAATTGCGTTGCGCAATCATAAATATGAAAAATAATTGTTGCATCA2-cspA-R (aas)TCGATACTGCCCATGAATTCAGTGTATTACCTTTAATAATTAAGTGTGCA1-yjeFE-F (pRSF)CTTACATTAATTGCGTTGCGCTTTCAGCTAACGGGCGCTCA1-yjeFE-R (aas)TCGATACTGCCCATGAATTCGTCAGAGCCCCTCGATCTCAA3-infc-rplT-F (pRSF)CTTACATTAATTGCGTTGCGCGCGGGCATTCGTGTTAAAGCA3-infc-rplT-R (aas)TCGATACTGCCCATGAATTCACCTTATTCCTCCAATTGTTTAAGACB2-cspA-F (aas)GAAGTGCTCAGTTAAAGATCTCAATTGGA-TATCGGAATCATAAATATGAAAAATAATTGTTGCATB2-cspA-R (adh)TCAGGATAAGACATGGATCCAGTGTATTACCTTTAATAATTAAGTGTGCCB1-yjeFE-F (aas)TCGTGGAAGTGCTCAGTTAAAGATCTCAATTGGATATCGGTTTCAGCTAACGGGCGCTB1-yjeFE-R (adh)TCAGGATAAGACATGGATCCGTCAGAGCCCCTCGATCTCAB3-infc-rplT-F (aas)TCGTGGAAGTGCTCAGTTAAAGATCTCAATTGGATATCGGGCGGGCATTCGTGTTAAAGCB3-infc-rplT-R (adh)TCAGGATAAGACATGGATCCACCTTATTCCTCCAATTGTTTAAGACcspA-ADH6-FCCACGCGATCGCTGACGTCGGTACCAATCATAAATATGAAAAATAATTGTTGCATCACcspA-ADH6-RAGGATAAGACATGGATCCAGTGTATTACCTTTAATAATTAAGTGTGCADH6-cspA-FATTATTAAAGGTAATACACTGGATCCATGTCTTATCCTGAGAAATTTGAAGGADH6-cspA-RTTTCTTTACCAGACTCGAGGGTACCCTAGTCTGAAAATTCTTTGTCGTAGCcspA-PcAAs-FTTACATTAATTGCGTTGCGCAATCATAAATATGAAAAATAATTGTTGCATCACcspA-PcAAs-RTCGATACTGCCCATGAATTCAGTGTATTACCTTTAATAATTAAGTGTGCCPcAAs-cspA-FTTATTAAAGGTAATACACTGAATTCATGGGCAGTATCGACAPcAAs-cspA-RGGCCGATATCCAATTGAGATCTTTAACTGAGCACTTCCACGAGCpRSF-FAGATCTCAATTGGATATCGGCCpRSF-RGCGCAACGCAATTAATGTAAGTadhE-F (cspA)ACTTAATTATTAAAGGTAATACACTATGGCTGTTACTAATGTCGCTGAfrmA-F (cspA)ACTTAATTATTAAAGGTAATACACTATGAAATCACGTGCTGCCGTadhP-F (cspA)ACTTAATTATTAAAGGTAATACACTATGAAGGCTGCAGTTGTTACGAadhE-R (cspA)TTTCTTTACCAGACTCGAGGGTACCTTAAGCGGATTTTTTCGCTTTTTTCTfrmA-R (cspA)TTTCTTTACCAGACTCGAGGGTACCTCAGTAACGAATTACGGTTCGAATGadhP-F (cspA)TTTCTTTACCAGACTCGAGGGTACCTTAGTGACGGAAATCAATCACCATcspA-RAGTGTATTACCTTTAATAATTAAGTGTGCcspA-F (pRSFMC)CCACGCGATCGCTGACGTCGGTACCAATCATAAATATGAAAAATAATTGTTGCATCAC
1.2.3 摇瓶发酵方法
在LB平板上划线分离得到单菌落,将其接种于LB培养基中并加入适量抗生素,在37 ℃,220 r/min的摇床中过夜培养(10~14 h)。以3%的接种量转接至发酵培养基F1中,将装有20 mL培养基的250 mL 锥形瓶置于30 ℃,220 r/min的摇床中振荡培养,适时取样。
1.2.4 样品处理及HPLC检测方法
由于酪氨酸微溶于水,利用酪氨酸的官能团—NH2 和H+可进行化学反应这一特性,用2 mol/L的HCl将发酵液样品按体积比2∶1稀释,以确保酪氨酸完全溶解。用去离子水将发酵液样品进一步稀释,使最终产物浓度适宜分析方法的检测范围。稀释完成后,12 000 r/min离心10 min。收集上清液并过滤处理得到用于HPLC测定的样品。
HPLC测定的色谱柱为C18柱ODS-2 Hypersil(250 mm×4.6 mm,5 μm),紫外检测器的检测波长为280 nm,A相为含有0.1%的三氟乙酸的去离子水,B相为含有0.1%三氟乙酸的乙腈,使用1.0 mL/min的流速进行梯度洗脱,流速为1.0 mL/min,柱温40 ℃, 进样量10 μL。酪氨酸和酪醇的出峰时间分别约为10.0、12.5 min。
2 结果与分析
2.1 酪醇合成途径的构建
本研究采用的代谢调控策略如图1所示,选择E.coli BL21 (DE3)作为出发菌株,利用化学转化的方法将构建的重组质粒pRSFCP-P(携带有芳香醛合成酶PcAAS的基因)导入其中,得到菌株BL2101,以转入空质粒pRSFDuet-1的菌株作为对照菌株(菌株BLCK)。结果表明,24 h时菌株BL2101发酵液中有酪醇产生,但随着发酵时间的延长,产量并未有明显增加,菌株BL2101的最终产量为105.97 mg/L。液相-质谱分析结果表明该产物为酪醇(图2)。
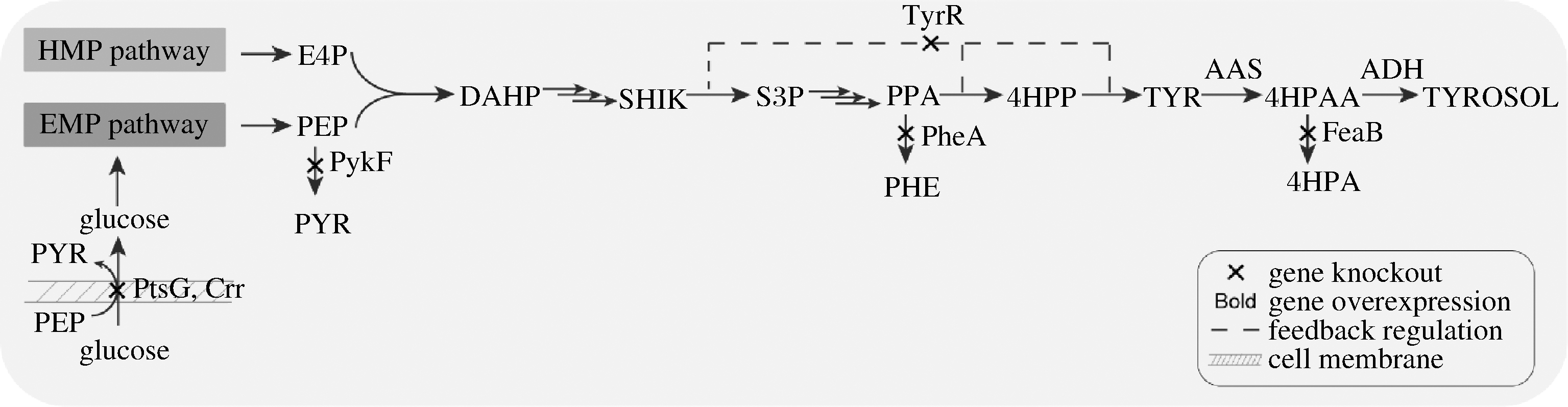
图1 本研究相关代谢工程策略示意图
Fig.1 Metabolic strategies used in this study
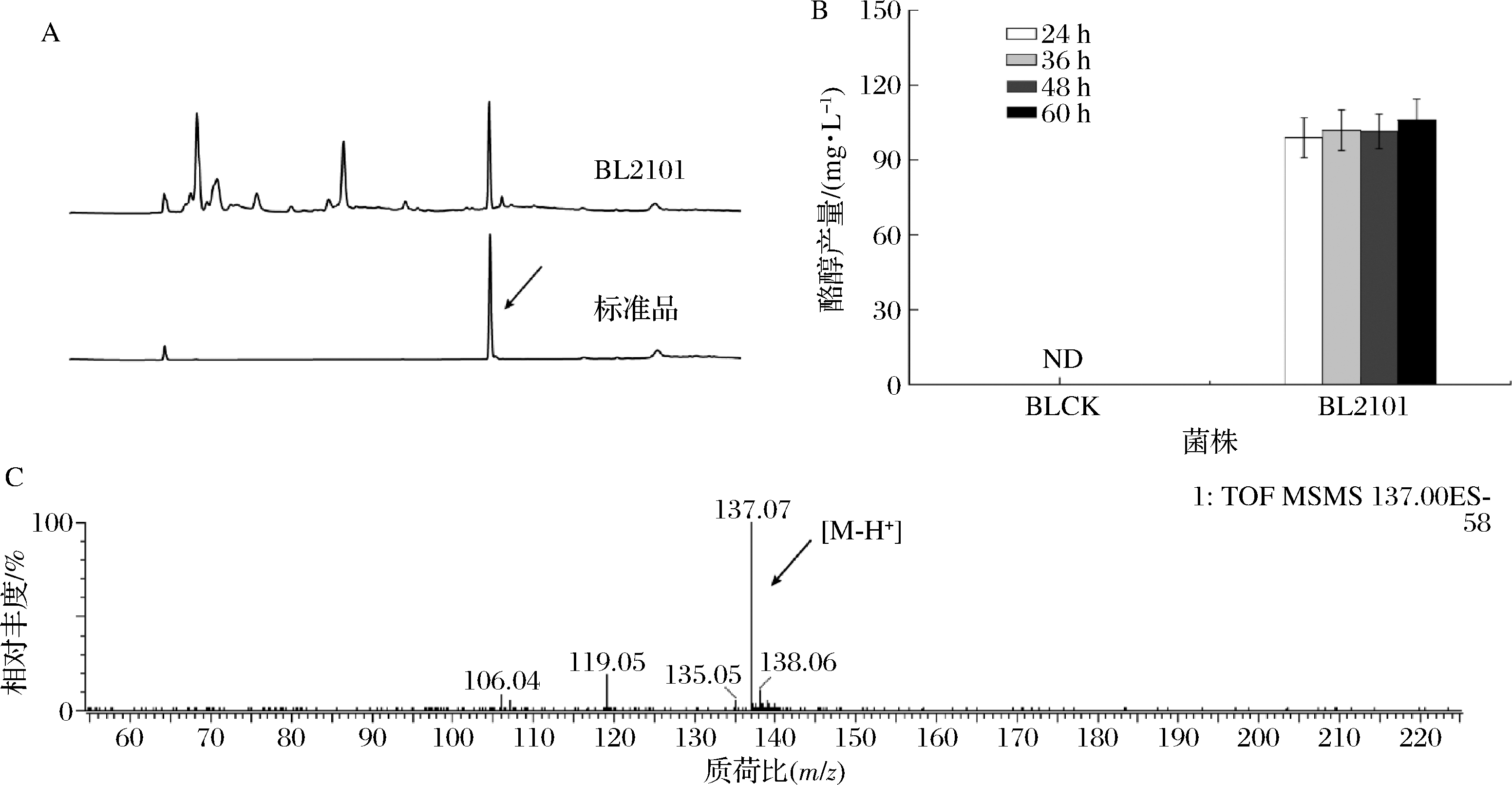
A-发酵24 h样品及分析标准品液相检测图;B-从头合成发酵产生的酪醇产量;C-发酵样品质谱图
图2 样品的高效液相色谱及质谱检测图
Fig.2 HPLC and MS analysises of fermentation samples
注:“ND”表示未检出(下同)
2.2 分支代谢途径的阻遏
酪醇合成依赖的莽草酸途径是苯丙氨酸、色氨酸、酪氨酸以及相关衍生物合成的重要代谢途径。下调支路表达可以有效增强流向产物的代谢流。首先,在缺失反馈抑制基因(transcriptional regulatory protein gene,tyrR)的菌株BER中转入质粒pRSFCP-P获得重组菌株BER01,酪醇产量为60.97 mg/L。在菌株BER的基础上,继续敲除在葡萄糖转运过程中会使磷酸烯醇式丙酮酸转变为丙酮酸的基因ptsG和crr(均为葡萄糖转运过程相关基因),并通过质粒过量表达PcAAS获得的菌株BETR01,酪醇产量为76.24 mg/L。在此基础上,敲除苯丙氨酸竞争途径的基因pheA(prephenate dehydratase gene)以及会使磷酸烯醇式丙酮酸流向丙酮酸的基因pykF (pyruvate kinase),并表达重组质粒pRSFCP-P获得菌株BE01,其酪醇产量达131.22 mg/L,相比其他2株重组菌株,酪醇产量分别提升了115.22%及72.11%。另外,通过敲除会使代谢流流向副产物对羟基苯乙酸的基因feaB,并在该菌株中过量表达PcAAS获得菌株BEKOF01,酪醇产量达到1 055.36 mg/L(图3)。相比菌株BE01,酪醇产量提升了704.2%;相比在E.coli BL21 (DE3)中过量表达PcAAS,酪醇产量提升了896.23%。
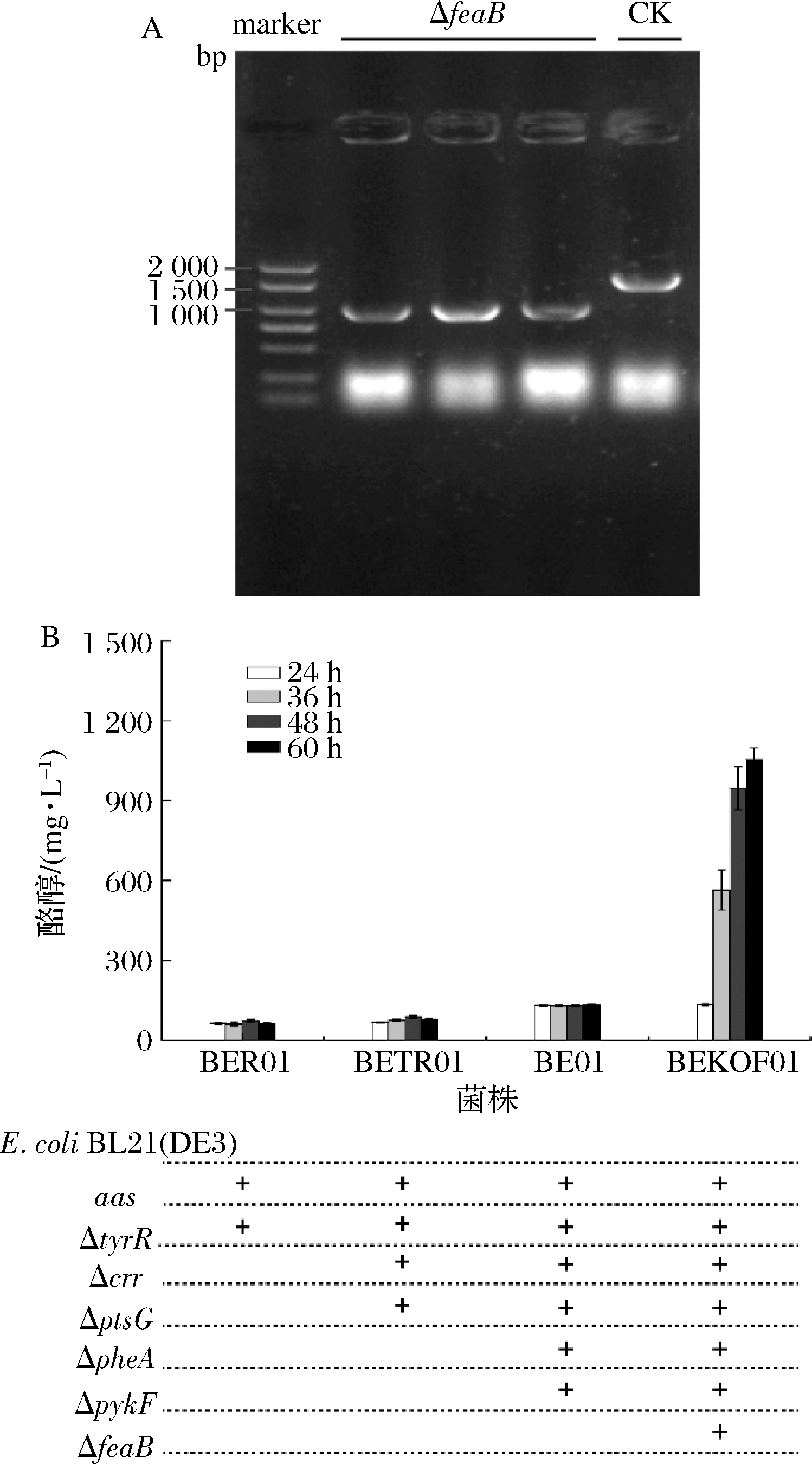
A-feaB敲除电泳图;B-基因缺失对酪醇产量的影响
图3 敲除调控分支代谢途径基因对酪醇产量的影响
Fig.3 Effects of relieving bottlenecks to tyrosol prodution
注:“+”表示基因的过量表达或缺失
2.3 乙醇脱氢酶的筛选
对羟基苯乙醛的还原反应是酪醇合成代谢途径的最后一步,对酪醇生产具有重要影响。本研究利用大肠杆菌内源的乙醇脱氢酶实现该步反应,酪醇产量达到1 055.36 mg/L。为了深入研究乙醇脱氢酶活性对酪醇积累的影响,本研究考察了4种来源于大肠杆菌的乙醇脱氢酶(AdhE[10]、AdhP[11]、YahK[12]及FrmA[13])及1种来源于酿酒酵母的乙醇脱氢酶(Adh6)[14-15] (图4)。结果表明,过量表达大肠杆菌来源的乙醇脱氢酶效果不佳;过量表达FrmA仅获得516.92 mg/L的酪醇;过量表达YahK仅产生了96.33 mg/L的酪醇;过量表达AdhE的菌株发酵液及过量表达AdhP的菌株发酵液并未检测到酪醇;过量表达酿酒酵母来源的Adh6效果显著(菌株BEKOF02),酪醇产量达1 516.86 mg/L。 相比于未表达Adh6的菌株(菌株BEKOF01),酪醇产量提升了43.73%。
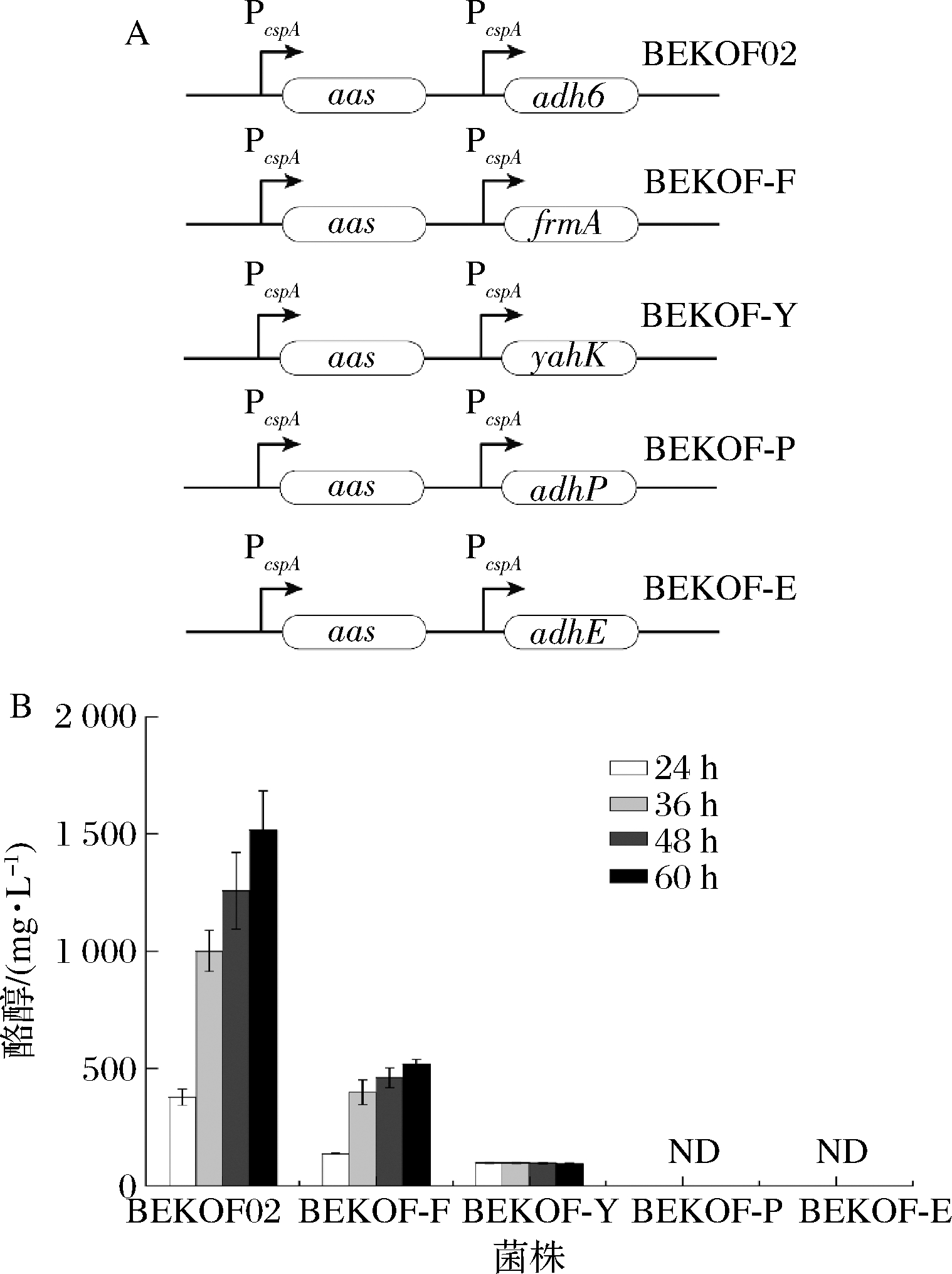
A-乙醇脱氢酶筛选;B-酪醇产量
图4 敲除调控分支代谢途径基因对酪醇产量的影响
Fig.4 Relieving bottlenecks in tyrosol biosynthesis pathway
2.4 关键酶PcAAS和ScADH6的组合调控
酶表达量的调节是代谢工程改造过程中的重要方法。在本研究中,通过调整质粒拷贝数的方法,尝试平衡酪醇生产的代谢流。质粒pRSFDuet-1、pETDuet-1、pACYCDuet-1和pCDFDuet-1含有的复制子分别是RSF、pBR322、p15A和CDF,其拷贝数分别为100、40、20和10[16]。由于本研究已使用pRSFDuet-1作为载体,因此将PcAAS及ScADH6的表达框克隆至其余3个质粒中,获得菌株BEKOF-pET、BEKOF-pACYC及BEKOF-pCDF。3株菌株产量均低于50.00 mg/L (图5、表5)。
因此,为了进一步优化代谢流,选择具有不同表达强度的启动子PyjeFE(低强度)、PcspA(中强度)、Pinfc-rplT(高强度)[5]分别用于调节PcAAS及ScADH6的表达强度。结果表明,A2B1(使用启动子PcspA表达PcAAS,启动子PyjeFE表达ADH6)的组合产量较BEKOF02(使用启动子PcspA表达PcAAS,启动子PcspA表达ADH6)产量提升了293.60 mg/L,酪醇产量达到1 810.46 mg/L(图5、表5)。
表5 复制子或启动子对酪醇产量的影响
Table 5 Effect of replications and promoters on tyrosol production

菌株名称产量/(mg·L-1)BEKOF-pACYC47.03BEKOF-pCDF11.13BEKOF-pET22.21BEKOF-A1B128.62BEKOF-A1B222.36BEKOF-A1B313.31BEKOF-A2B11 810.46BEKOF02(CK)1 516.86BEKOF-A2B3899.62BEKOF-A3B133.23BEKOF-A3B212.15BEKOF-A3B327.39
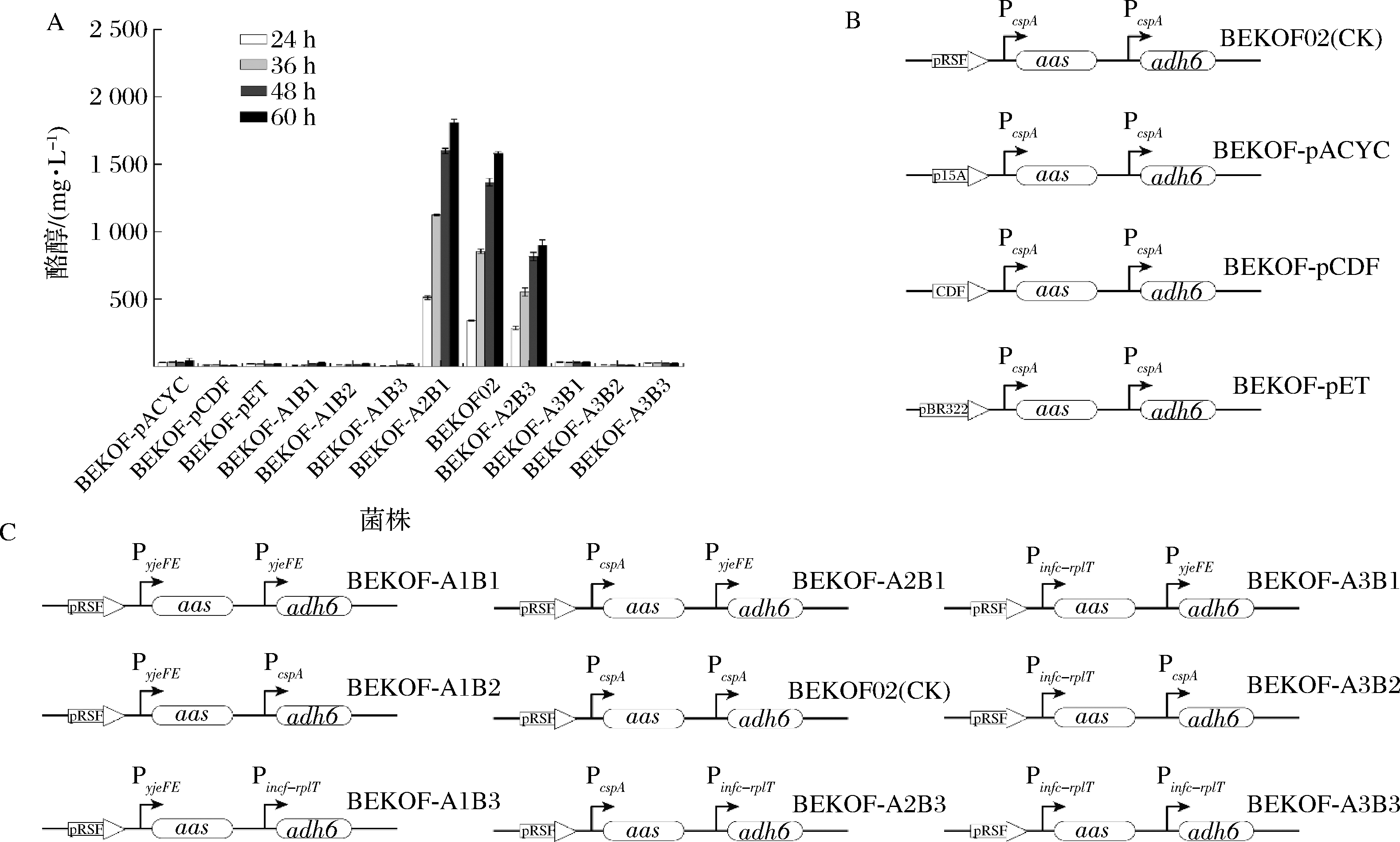
A-酪醇产量;B-复制子筛选;C-启动子组合表达筛选
图5 关键酶表达量的调控
Fig.5 Regulations of expression levels
2.5 最佳菌株BEKOF-A2B1的发酵优化
葡萄糖和甘油是大肠杆菌培养过程中常用的碳源,在细胞生长过程中发挥着重要作用。此外,葡萄糖和甘油也是细胞糖酵解途径的底物,其含量的高低直接决定了代谢所产生的产物含量。本研究中的出发培养基中含有葡萄糖25 g/L及甘油10 g/L,以此为基础,进行了葡萄糖和甘油浓度的调整。图6表明了不同配比下发酵60 h时酪醇的产量、菌体OD600以及发酵液pH。25 g/L葡萄糖和20 g/L甘油的组合具有优势(图6-A),但发酵液pH较低,导致大肠杆菌难以进行正常的代谢活动。因此,通过提高培养基中氮源浓度的方法或外源添加来调节发酵过程中的pH,结果表明外源添加5 g/L碳酸钙效果最佳,酪醇产量达到2 120.58 mg/L。
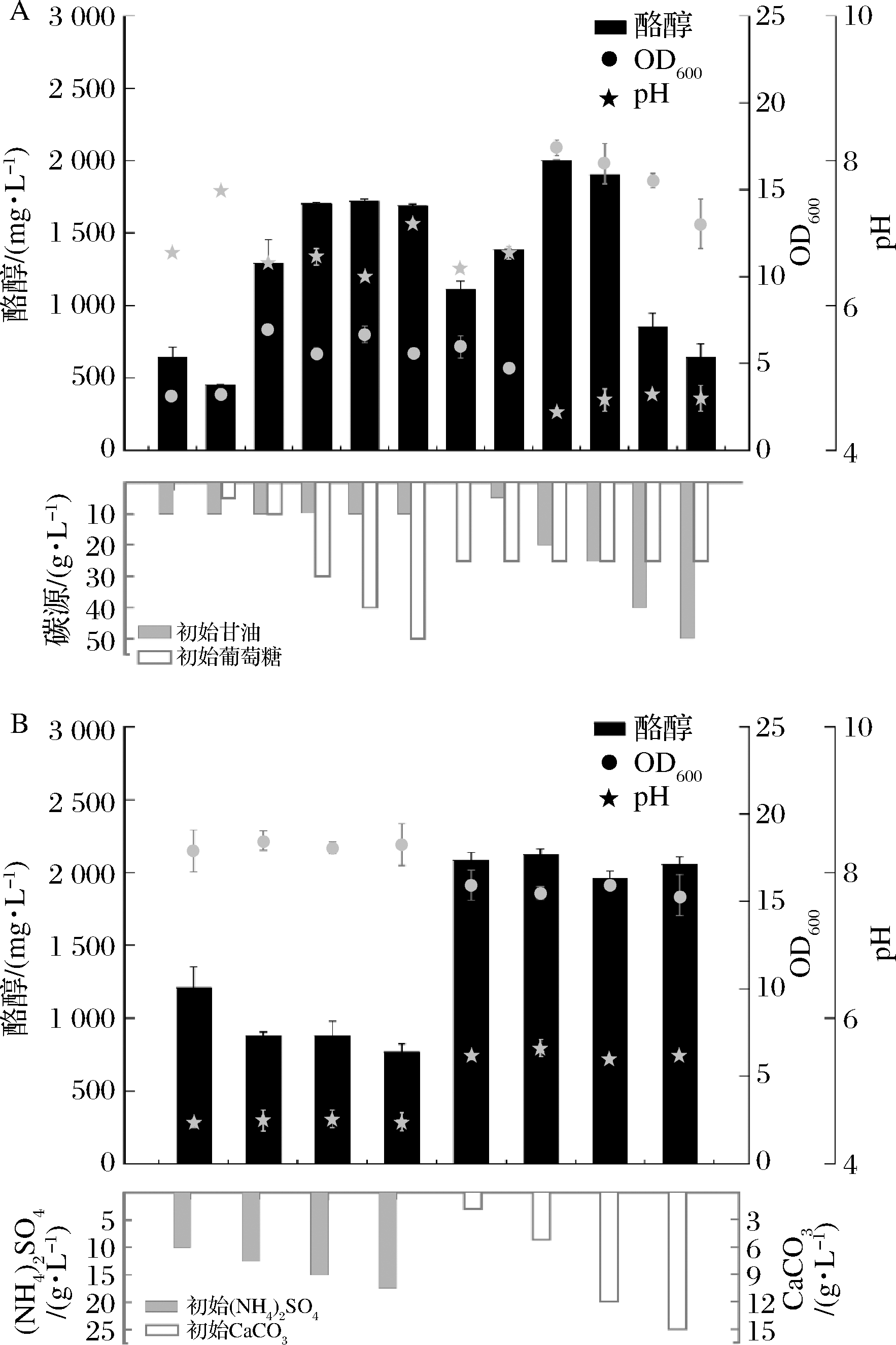
A-碳源浓度优化;B-氮源浓度优化及CaCO3浓度优化
图6 摇瓶水平发酵优化
Fig.6 Fermentation optimization on shake flask
3 讨论
酪醇因具有显著的抗氧化、抗肿瘤等功能药效成为近年来的研究热点[17]。酿酒酵母由于具有完整的Ehrlich途径,成为研究者们较为青睐的表达宿主。但是,由于较长的发酵周期使得酿酒酵母作为生产菌株具有了一定的局限性,如LIU等[18]改造的对香豆酸生产菌株1个发酵周期需要96 h,GUO等[19]改造获得的具有酪醇生产能力的酿酒酵母的发酵周期为196 h。大肠杆菌因发酵周期较短且易于基因工程改造等优点成为了另一种选择。研究表明,在宿主细胞中表达来源荷兰芹的芳香醛合成酶可以构建酪醇生物合成途径[4,19-20]。本研究将经密码子优化的PcAAS在E.coli BL21 (DE3)中过量表达,酪醇产量达到105.97 mg/L。此外,在缺失了不同分支代谢途径调控基因的菌株中过量表达PcAAS,以及敲除对酪醇积累具有显著抑制作用的feaB基因,酪醇积累量达到1 055.36 mg/L,而未缺失feaB基因的菌株产量仅为131.22 mg/L,研究结果与此前的报道类似[3, 13, 21]。
在多数研究中,醛基到羟基的还原反应依赖于大肠杆菌中天然存在的醇脱氢酶[4, 13]。过量表达乙醇脱氢酶可以强化酪醇合成途径的代谢流。GUO等[20]在酿酒酵母中表达来自大肠杆菌的乙醇脱氢酶,提高了酪醇的产量。本研究过量表达ScADH6使得酪醇的产量由1 055.36 mg/L提高至1 516.86 mg/L。另外,LIU等[18]通过同时优化生物合成途径中关键酶的启动子,显著提高了对香豆酸的产量。在本研究使用中等强度启动子启动PcAAS与低强度启动子启动ScADH6的组合表现出较大潜力,酪醇产量达1 810.46 mg/L。 在此基础上,通过优化碳氮源初始浓度以及加入CaCO3改善发酵过程pH稳定性,酪醇产量提高至2120.58 mg/L。在摇瓶发酵水平上,较XU等[13]研究中大肠杆菌YMG5A*R的产量1 506.96 mg/L及LIU等[22]研究中酿酒酵母LYTY16的产量702.30 mg/L,酪醇分别提升了40.72%和201.95%。但是,在发酵罐水平上,对比XU等[13]研究中的3.90 g/L和LIU等[22]研究中的9.90 g/L,仍存在一定差距。因此,在后续研究中,可以结合对前体供给途径的系统改造和发酵过程优化,使其成为羟基酪醇等高价值化合物合成的良好平台菌株。
[1] CHANDRAMOHAN R, PARI L, RATHINAM A, et al.Tyrosol, a phenolic compound, ameliorates hyperglycemia by regulating key enzymes of carbohydrate metabolism in streptozotocin induced diabetic rats[J].Chemico-Biological Interactions, 2015, 229:44-54.
[2] SATOH Y, TAJIMA K, MUNEKATA M, et al.Engineering of a tyrosol-producing pathway, utilizing simple sugar and the central metabolic tyrosine, in Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2012, 60:979-984.
[3] YANG H Q, XUE Y X, YANG C, et al.Modular engineering of tyrosol production in Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2019, 67:3 900-3 908.
[4] CHUNG D, KIM S Y, AHN J H.Production of three phenylethanoids, tyrosol, hydroxytyrosol, and salidroside, using plant genes expressing in Escherichia coli[J].Scientific Reports, 2017, 7:2 578.
[5] ZHOU S H, DING R P, CHEN J, et al.Obtaining a panel of cascade promoter-5′-UTR complexes in Escherichia coli[J].Acs Synthetic Biology, 2017, 6(6):1 065-1 075.
[6] ZHOU Y, ZHAO M, ZHOU S H, et al.Biosynthesis of adipic acid by a highly efficient induction-free system in Escherichia coli[J].Journal of Biotechnology, 2020, 314:8-13.
[7] ZHOU S H, HAO T T, ZHOU J W.Fermentation and metabolic pathway optimization to de novo synthesize (2S)-naringenin in Escherichia coli[J].Journal of Microbiology and Biotechnology, 2020, 30(10):1 574-1 582.
[8] FORDJOUR E, ADIPAH F K, ZHOU S H, et al.Metabolic engineering of Escherichia coli BL21 (DE3) for de novo production of L-DOPA from D-glucose[J].Microbial Cell Factories, 2019, 18(1):74.
[9] JIANG Y, CHEN B, DUAN C L, et al.Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J].Applied and Environmental Microbiology, 2015, 81(7):2 506-2 514.
[10] WU J J, ZHANG X, ZHU Y J, et al.Rational modular design of metabolic network for efficient production of plant polyphenol pinosylvin[J].Scientific Reports, 2017, 7(1):1-15.
[11] CAO Y X, XIAO W H, ZHANG J L, et al.Heterologous biosynthesis and manipulation of alkanes in Escherichia coli[J].Metabolic Engineering, 2016, 38:19-28.
[12] LI C Z, JIA P, BAI Y J, et al.Efficient synthesis of hydroxytyrosol from L-3,4-Dihydroxyphenylalanine using engineered Escherichia coli whole cells[J].Journal of Agricultural and Food Chemistry, 2019, 67(24):6 867-6 873.
[13] XU W, YANG C, XIA Y Y, et al.High-level production of tyrosol with noninduced recombinant Escherichia coli by metabolic engineering[J].Journal of Agricultural and Food Chemistry, 2020, 68(16):4 616-4 623.
[14] MU IZ-CALVO S, BISQUERT R, PUIG S, et al.Overproduction of hydroxytyrosol in Saccharomyces cerevisiae by heterologous overexpression of the Escherichia coli 4-hydroxyphenylacetate 3-monooxygenase[J].Food Chemistry, 2020, 308:125646.
IZ-CALVO S, BISQUERT R, PUIG S, et al.Overproduction of hydroxytyrosol in Saccharomyces cerevisiae by heterologous overexpression of the Escherichia coli 4-hydroxyphenylacetate 3-monooxygenase[J].Food Chemistry, 2020, 308:125646.
[15] SHEN X L, WANG J, GALL B K, et al.Establishment of novel biosynthetic pathways for the production of salicyl alcohol and gentisyl alcohol in Engineered Escherichia coli[J].ACS Synthetic Biology, 2018, 7(4):1 012-1 017.
[16] WU J J, DU G C, ZHOU J W, et al.Metabolic engineering of Escherichia coli for (2S)-pinocembrin production from glucose by a modular metabolic strategy[J].Metabolic Engineering, 2013, 16:48-55.
[17] CASADEY R, CHALLIER C, ALTAMIRANO M, et al.Antioxidant and antimicrobial properties of tyrosol and derivative-compounds in the presence of vitamin B2.Assays of synergistic antioxidant effect with commercial food additives[J].Food Chemistry, 2021, 335:127576.
[18] LIU Q L, YU T, LI X W, et al.Rewiring carbon metabolism in yeast for high level production of aromatic chemicals[J].Nature Communications, 2019, 10(1):4 976.
[19] GUO W, HUANG Q L, FENG Y H, et al.Rewiring central carbon metabolism for tyrosol and salidroside production in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2020, 117(8):2 410-2 419.
[20] GUO W, HUANG Q L, LIU H, et al.Rational engineering of chorismate-related pathways in Saccharomyces cerevisiae for improving tyrosol production[J].Frontiers in Bioengineering and Biotechnology, 2019, 7:152.
[21] XUE Y X, CHEN X Z, YANG C, et al.Engineering Eschericha coli for enhanced tyrosol production[J].Journal of Agricultural and Food Chemistry, 2017, 65(23):4 708-4 714.
[22] LIU H Y, TIAN Y J, ZHOU Y, et al.Multi-modular engineering of Saccharomyces cerevisiae for high-titre production of tyrosol and salidroside[J].Microbial Biotechnology, 2020.DOI:10.1111/1751-7915.13667.