传统大豆酱是中国的四大发酵豆制品之一[1],以大豆为原料,经过蒸煮、制醅、晾晒、前发酵、盐封固态发酵和后发酵等工序加工而成,采用自然发酵,保留了传统大豆酱独特的风味。其中前发酵阶段是将大豆浸泡、蒸煮和挤压后制成长方体的酱醅,置于发酵室中进行为期约2个月的自然发酵,在此阶段主要是霉菌分泌多种酶类发挥主要作用;低盐固态发酵阶段,是将发酵室中成熟的酱醅刷洗粉碎后放入发酵桶中,加入盐度为(20±1)°B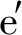 的盐水混合均匀再继续发酵3个月左右,此阶段中霉菌基本停止生长,耐盐乳酸菌和酵母菌开始大量繁殖,可发酵糖被代谢生成乙醇、乳酸乙酯和乙酸乙酯等香气物质[2],赋予传统大豆酱独特的风味。本研究采用Illumina MiSeq测序技术对传统大豆酱前发酵阶段和低盐固态发酵阶段进行细菌和真菌群落结构的分析,明确在不同发酵阶段中发挥主要作用的微生物,有利于挖掘功能性菌株,缩短发酵时间,改善传统大豆酱的风味。
的盐水混合均匀再继续发酵3个月左右,此阶段中霉菌基本停止生长,耐盐乳酸菌和酵母菌开始大量繁殖,可发酵糖被代谢生成乙醇、乳酸乙酯和乙酸乙酯等香气物质[2],赋予传统大豆酱独特的风味。本研究采用Illumina MiSeq测序技术对传统大豆酱前发酵阶段和低盐固态发酵阶段进行细菌和真菌群落结构的分析,明确在不同发酵阶段中发挥主要作用的微生物,有利于挖掘功能性菌株,缩短发酵时间,改善传统大豆酱的风味。
1 材料与方法
1.1 材料与试剂
传统大豆酱由吉林省田野泉酿造有限公司提供,对同一发酵室2个不同发酵阶段的5个时间节点的传统大豆酱进行取样,第1个阶段为酱醅放入发酵室的第1天(A),酱醅发酵的中间时间节点30 d(B)和在发酵室的最后一天50 d(C),称之为前发酵阶段;第2个阶段为酱醅刷酱粉碎后加入盐水进入低盐固态发酵阶段的起始点75 d(D)和结束点165 d(E),在每个时间节点取5个平行样品共25个样品,分别标记为A1~A5、B1~B5、C1~C5、D1~D5、E1~E5,样品无菌采集后立即置于-20 ℃冰箱中保藏待用。
表1 材料信息
Table 1 Material information

分组发酵时间/d编号 A1A1、A2、A3、A4、A5B30B1、B2、B3、B4、B5C50C1、C2、C3、C4、C5D75D1、D2、D3、D4、D5E165E1、E2、E3、E4、E5
M0491L-Q5®High-Fidelity DNA Polymerase扩增聚合酶,美国纽英伦生物技术有限公司;P7589-Quant-iT PicoGreen dsDNA Assay Kit荧光定量染料、75510-019-Agarose琼脂糖凝胶试剂、AM9870-TAE琼脂糖凝胶电泳缓冲液,美国英杰公司;DL2000-DNA Marker,日本宝生物股份有限公司。
1.2 仪器与设备
2720 PCR仪,美国应用生物系统公司;FLX800T酶标仪,美国伯腾仪器(BioTek)有限公司;DYY-6C电泳仪,北京六一生物科技有限公司;BG-gdsAUTO 130型凝胶成像系统,北京百晶生物技术有限公司;高通量测序平台,美国Illumina公司。
1.3 实验方法
1.3.1 传统大豆酱不同发酵阶段微生物多样性测定
将采集的5个不同发酵时间段的大豆酱样本,每个样本5个平行,送至上海派森诺生物科技股份有限公司进行测定。测定流程主要包括:微生物组总DNA的提取,对细菌使用引物338F(5′-ACTCCTACGGGAGGCGCAGCA-3′)和806R(5′-GG-ACTACHVGGGTWTCTAAT-3′)扩增16S rRNA基因的V3~V4结构域。真菌使用内部转录间隔区ITS区用引物ITS5(5′-GGAAGTAAAAGTCGTAACAAGG-3′)和ITS1(5′-GCTGCGTTCTTCATCGATGC-3′)扩增。将扩增产物进行回收纯化,荧光定量处理,使用Illumina公司的TruSeq Nano DNA LT Library prep Kit进行测序文库的制备,上机进行高通量测序。
1.3.2 生物信息分析
采用Illumina平台对群落DNA片段进行双端(Paired-end)测序,使用QIIME2(2019.4)对测序原始数据进行分析处理,得到有效序列;通过UCLUST序列比对工具,对获得的序列按97%的序列相似度进行归并和操作分类单元数(operational taxonomic unit,OTU)划分,并选取每个OTU中丰度最高的序列注释;利用R语言工具绘制各分类水平下的微生物多样性群落结构图,对各样本在不同分类水平上的群落结构进行分析。
2 结果与分析
2.1 传统大豆酱不同发酵阶段微生物多样性分析
2.1.1 传统大豆酱α多样性分析
α多样性是以Chao1指数表征丰富度,以Shannon和Simpson指数表征多样性,以coverage指数表征覆盖度[3]。本研究得到的传统大豆酱不同发酵阶段微生物群落α多样性指数详情见表2。
通过测序,去除嵌合体后序列量,得到的高质量序列量,16S rRNA 测序得到 1 795 797条高质量的序列,ITS 结果共得到2 615 938条高质量的序列。Chao1指数越大,说明群落丰富度越高[4],本研究结果中细菌的 Chao 1指数均比真菌高很多,说明传统大豆酱发酵过程中主要优势菌群是细菌,所有豆酱样品coverage指数都在99.71%以上,这表明样品中菌群型基本被鉴定出来。
表2 传统大豆酱α多样性分析
Table 2 α-diversity index of bacteria and fungi in traditional soybean pastes

组别样品细菌真菌Chao1ShannonSimpsoncoverageChao1ShannonSimpsoncoverageAA1200.553.030.75125.121.100.421A2170.613.130.80132.221.250.451A3212.773.630.86126.001.130.421A4197.772.910.75123.911.110.431A5191.662.850.75126.311.110.431BB1423.214.280.901139.301.960.441B2594.754.770.911108.201.630.371B3447.644.780.921117.671.580.361B4467.404.440.88185.492.000.531B5563.434.980.931110.011.810.401CC1533.023.160.511118.312.060.461C2589.613.760.591102.202.910.721C3442.842.950.49198.992.410.611C4529.184.040.65197.191.920.441C5338.911.610.26176.681.170.271DD1385.384.480.89198.451.940.451D2460.824.510.91191.371.760.551D3514.764.600.911120.032.800.641D4500.974.380.901214.523.520.811D5493.694.140.881181.553.000.671EE1516.734.760.931176.903.750.811E2511.384.630.931145.133.740.821E3547.234.940.941160.813.960.851E4566.804.780.931152.663.800.841E5532.884.920.941160.083.720.811
2.1.2 传统大豆酱不同发酵阶段微生物多样性Venn图分析
Venn图中1个圆圈代表1组样本,不同的颜色代表不同的分组。由图1-a可知,不同分组间细菌群落共有OTU数为20,独有的OTU数量分别为320、805、599、554和1 072。由图1-b可知,不同分组间真菌群落共有OTU数为27,独有的OTU数量分别为20、86、62、143和131。其中,细菌在A时独有的OTU数最少,在E时最多。真菌OTU数在A时最少,在D时最多。
在前发酵阶段,细菌和真菌OTU数呈现的趋势相同,在B(30 d)时最多;但在低盐固态发酵阶段D(75 d)和E(165 d)中,E时的细菌OTU数高于D,但真菌OTU数不及E时,可能由于在低盐固态发酵阶段,高的食盐含量使得豆酱的水分活度降低,微生物生长受到抑制,一些厌氧耐高渗透压的细菌适应了环境细菌迅速增长[5-6],使得好氧性真菌数量下降。
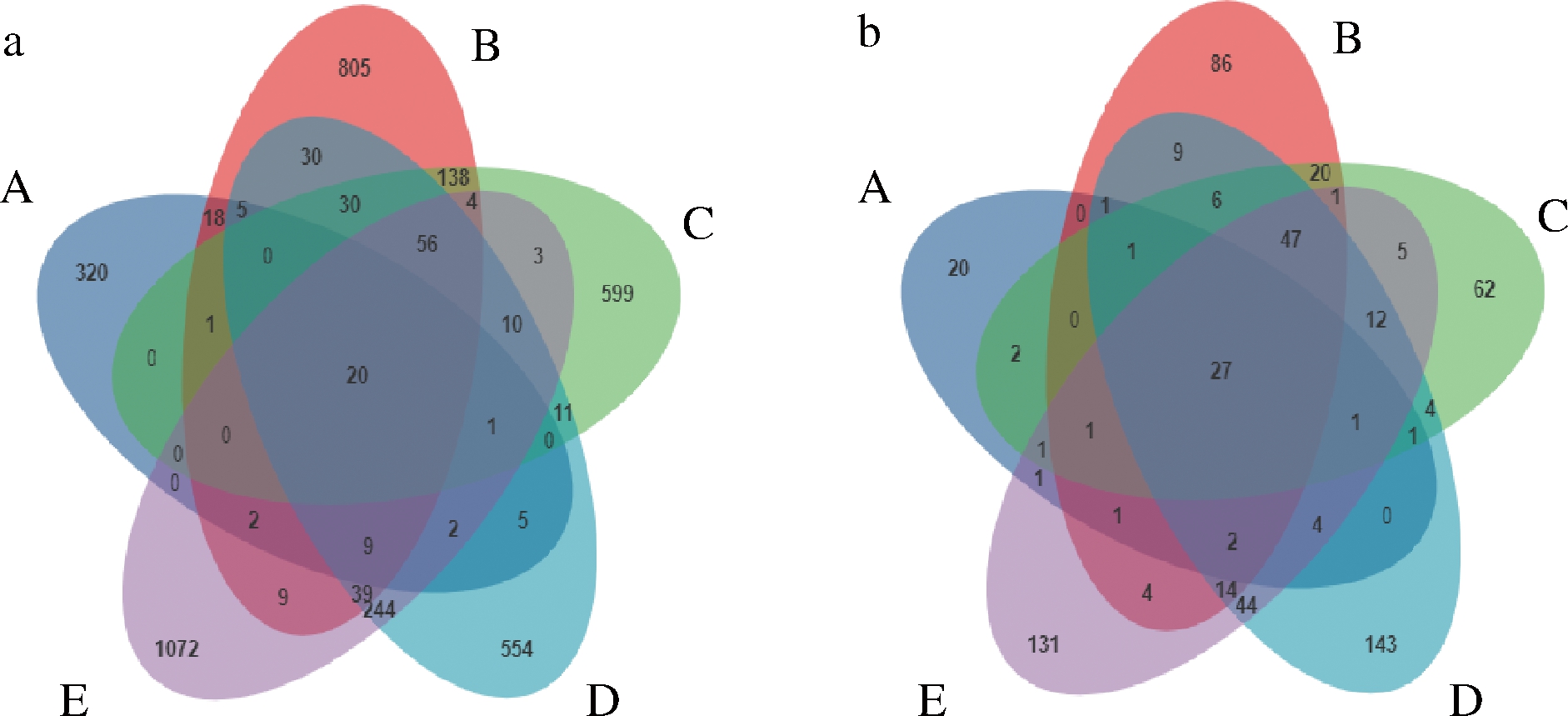
a-细菌;b-真菌
图1 传统大豆酱细菌和真菌Venn图分析
Fig.1 Venn diagram analysis of traditional soybean paste
由图2可知,细菌第一主成分(PC1)贡献率为35.3%,第二主成分(PC2)的贡献率为25.9%;真菌的第一主成分(PC1)贡献率和第二主成分(PC2)的贡献率分别为46.7%和36.4%。传统东北大豆酱5个不同发酵时间的样品细菌丰富度整体相对分散,说明不同发酵阶段细菌群落结构具有差异性,但是D、E相对较近,有重叠现象出现,说明盐封入缸半固态发酵阶段细菌群落结构具有相似性;而真菌在 B和C、D与E相对较近,即发酵30和50 d(发酵室酱醅阶段)、75 和165 d(盐封入缸半固态发酵阶段)可说明在同一环境下真菌群落非常相似。

a-细菌;b-真菌
图2 传统大豆酱细菌和真菌距离矩阵与主成分分析
Fig.2 Distance matrix and PCA of bacteria and fungi in traditional soybean paste
2.2 传统大豆酱不同发酵阶段微生物群落组成分析
2.2.1 细菌群落结构分析
根据物种注释结果,从微生物分类门的水平上来看,细菌门主要包括厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria)等。其中厚壁菌门在不同发酵阶段中占比均较高。
从微生物分类属水平上来看,不同的发酵阶段有不同的优势细菌属,但在所有样品中芽孢杆菌属(Bacillus)均存在。在传统大豆酱发酵1 d时,主要细菌属有芽胞杆菌属占比55.19%、狭义梭菌(Clostridium sensu stricto 12)占比19.07%、魏斯氏菌属(Weissella)和明串珠菌属(leuconostoc)均为乳酸菌属,占比分别为11.23%和9.28%。这一结果与陈浩等[7]的研究结果相似。乳酸菌是人体肠道系统中最主要的有益菌,能够产生良好的感官风味和丰富的营养物质,在各种发酵食品中发挥关键作用[8-10]。芽胞杆菌属能分泌多种酶类和抗生素;魏斯氏菌属可以增加在食品发酵过程中的有机酸、酯等含量[11],促进风味物质的合成,是参与食品发酵的微生物;明串珠菌可以作为食品发酵中的起始剂、调味剂和膨松剂,改善食品的安全性和感官特性[12]。李璐等[13]用肠膜明串珠菌发酵的菠萝果汁能够延缓维生素C的流失,感官评价较优。
在酱醅发酵中后期30和50 d(B和C)时,随着发酵时间增长,芽胞杆菌属在逐渐降低,枝芽胞菌属(Virgibacillus)在30 d时出现且增长迅猛,在50 d达到最大占比为85.47%。在发酵30 d时还有厌氧菌(Anaerosalibacter)、片球菌属(Pediococcus)和克罗彭斯特菌属(Kroppenstedtia)等,发酵50 d时优势细菌属与30 d基本一致,说明酱醅在发酵室中相同环境下细菌群落结构基本相似。左丽丽等[14]对传统发酵豆酱中乳酸菌进行分离和鉴定,初步推断出戊糖片球菌是吉林市农家酱发酵过程中的优势乳酸菌群,对传统豆酱的风味起着至关重要的作用。
在低盐固态发酵起始阶段75 d(D)主要优势细菌属为四联球菌属(Tetragenococcus)占总数52.71%,还有少量狭义梭菌(Clostridium sensu stricto 12);在终止阶段165 d(E)四联球菌和芽孢杆菌属有降低趋势,但同时出现了乳杆菌属(Lactobacillus)占比32.94%,主要优势细菌属同75 d时一致。嗜盐四联球菌是发酵食品常用的微生物之一[15],可以用于酿造酱油、发酵食品和腌制品中,改善产品的风味。

a-门水平;b-属水平
图3 不同分类水平下的传统大豆酱主要细菌群落结构
Fig.3 Bacterial community structure of traditional soybean paste under different classification levels
2.2.2 真菌群落结构分析
由图4-a可知,在门水平下,最丰富真菌菌门是子囊菌门(Ascomycota)、比较丰富的毛霉菌门(Mucoromycota)、担子菌门(Basidiomycota) 以及被孢霉门(Mortierellomycota)、壶菌门(Chytridiomycota)、球囊菌门(Glomeromycota)和芽枝霉门(Blastocladiomycota)等少量丰度菌门。其中,在发酵1 d时毛霉菌门为优势菌门,相对丰度高达 96.77%,随着发酵时间的延长,毛霉菌门锐减,子囊菌门增加,在发酵30 d时达到顶峰;在发酵后期出现少量担子菌门,相对丰度仅为 0.32%。
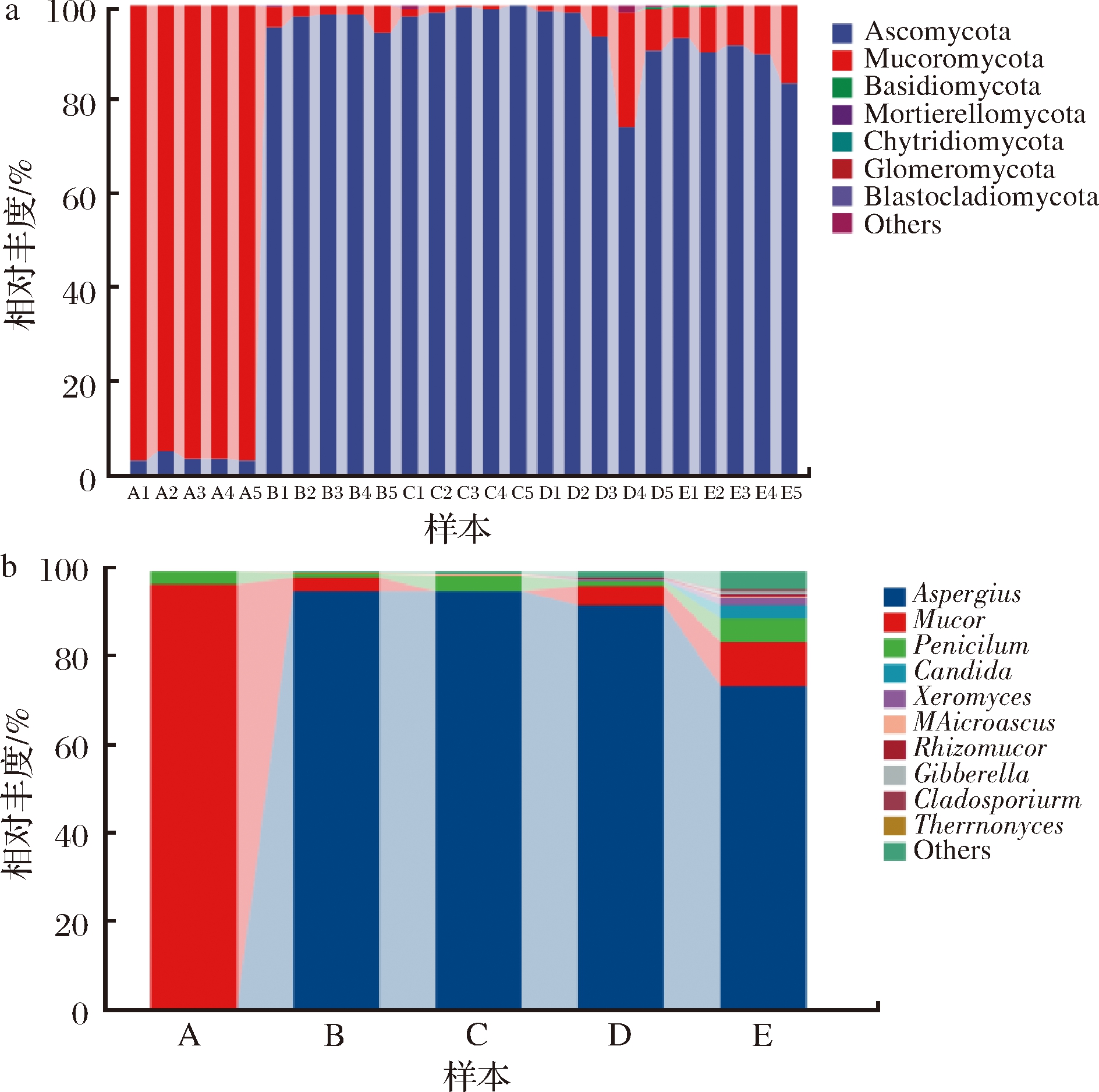
a-门水平;b-属水平
图4 不同分类水平下的传统大豆酱主要真菌群落结构
Fig.4 Community structure of main fungi in traditional soybean paste under different classification levels
由图4-b可见,属水平下,样品在真菌中检测出最丰富菌属是曲霉属(Aspergillus),次之是毛霉属(Mucor)、青霉属(Penicillum)。在发酵初期1 d时毛霉属为主要优势菌,30 d以后曲霉属为主要优势菌;在发酵的最后阶段165 d时出现假丝酵母属(Candida)、耐干霉菌属(Xeromyces),其余还有小囊菌属(Microascus)、根毛霉属(Rhizomucor)、赤霉菌(Gibberrella)、狭义梭菌属。其中,曲霉属[16]具有较强的酶活性,广泛应用于食品发酵;毛霉属能产生果胶酶、凝乳酶和脂肪酶等[17],可用于制曲和酿酒。因此,有必要对豆酱的微生物多样性进行研究,了解豆酱的微生物群落结构,充分利用优势菌株。
2.3 传统大豆酱不同发酵阶段微生物群落差异分析
采用线性判别分析(linear discriminant analysis effect size,LEfSe)法分析传统大豆酱前发酵酱醅微生物群落的差异显著性,寻找生物标识物[18]。由图5-a可知,不同发酵时期豆酱细菌群落结构存在显著丰度差异的。在发酵1 d时,芽胞杆菌属、魏斯氏菌属、明串珠菌属等为显著差异菌属;在30 d时,有Anaerosalibacter、Pediococcus、Kroppenstedtia、Oceanobacillus等为显著差异菌属;在50 d时只有Virgibacillus差异显著;在75 d时,Oligella、Vibrio等为差异显著菌属;在165 d时,只有Lactobacillus为差异显著菌属。由图5-b可知,不同发酵时期豆酱存在真菌有4组是有显著差异的,但是在165 d时差异不显著。发酵1 d时Mucor为显著差异菌属,在30 d时Licanicillum为差异显著菌属;在50 d时Lichtheimia差异显著菌属;在75 d时没有显著差异菌属,但165 d差异显著菌属相对来说比较多,包括Aspergillus、Curvularia、Tausonia、Fusarium和Pichia等。
3 讨论
目前采用高通量测序技术对于大豆酱发酵过程中微生物群落结构的分析已有报道。张颖等[19]利用PCR-DGGE结合高通量测序技术分析了自然发酵豆酱过程中细菌的多样性和动态变化,并得出细菌多样性也随着发酵时间的变化而变化的结论。向凡舒等[20]对鹤峰地区渣豆酱中的微生物研究结果表明芽孢杆菌属和葡萄球菌属是主要的细菌属,青霉属、耐碱酵母属和接合酵母属是主要的真菌属。JUNG等[21]利用焦磷酸测序法研究了韩国豆酱发酵过程中微生物,发现芽孢杆菌和毛霉是主要的细菌和真菌。马岩石等[22]对东北5个不同产地的豆酱微生物群落进行分析,表明细菌属主要为葡萄球菌属、肠杆菌属、明串珠菌属和芽胞杆菌属。本研究采用 Illumina MiSeq高通量测序技术对传统大豆酱2个不同发酵阶段5个时间节点微生物多样性差异分析。其中在传统大豆酱前发酵阶段的细菌群落结构上,主要以厚壁菌门和芽胞菌属为主,在低盐固态发酵阶段中75和165 d时,四联球菌属为主要优势细菌属,还有部分乳杆菌属。在真菌的群落结构上,发酵1 d时毛霉菌门优势菌门,毛霉属为主要优势菌属,其他发酵阶段均以子囊菌门为优势菌门,曲霉属为主要优势菌属。此结果与韩俊燕等[23]对4个不同地区的发酵辣椒制品的研究结果具有相似性。但本研究在细菌属水平和真菌属水平不同发酵阶段优势菌有很大差异,可能与地域及加工环境有密切关系,微生物群落多样性在细菌门上差异原因与部分报道相一致,如上述马岩石等[22]的结论。

a-传统大豆酱细菌LEfSe分析;b-传统大豆酱真菌LEfSe分析
图5 传统大豆酱不同发酵阶段LEfSe分析
Fig.5 Analysis of LEfSe in different fermentation stages of traditional soybean paste
在细菌群落结构上,主要优势菌属也包括明串珠菌属和芽胞杆菌属,与其不同的是四联球菌属和乳杆菌属也均为优势菌属,有研究表明二者会产生优良风味且对人体有益,比如CUI等[24]通过在酱醪中先接种嗜盐四联球菌,后接种耐盐酵母菌的方式,明显提高了醇类、酮类和吡嗪类物质等风味物质的含量。乳杆菌属可以通过发酵碳水化合物产生ATP,广泛用于生产商业酸奶,含乳杆菌的酸奶对肠道有益[25]。
本研究从前发酵酱醅阶段到盐封半固态发酵实验研究,该结果对了解传统大豆酱不同发酵微生物多样性变化能够提供一定的理论数据。为进一步明确优势菌株和风味物质的相关性研究提供依据,可以通过不同发酵天数的传统大豆酱中优势菌属进行筛选,定植发酵,以缩短生产周期,控制和改善风味。
[1] 马艳莉,李里特.发酵豆制品酿造过程中组分和营养功能因子的变化及调控[J].食品科学,2012,33(3):292-299.
MA Y L,LI L T.Changes and regulations of ingredients and nutritional factors in fermented soybean products during fermentation[J].Food Science,2012,33(3):292-299.
[2] 白乐宜,颜振敏,冯梦茹,等.四种芝麻香型白酒中香气活性成分分析[J].食品与发酵工业,2020,46(2):272-276.
BAI L Y,YAN Z M,FENG M R,et al.Analysis of odor-active compounds in four sesame-flavor Chinese Baijiu[J].Food and Fermentation Industries,2020,46(2):272-276.
[3] 张琪,朱丹,牛广财,等.高通量测序分析沙棘酵素自然发酵过程中细菌多样性[J/OL].食品科学,2021.http://kns.cnki.net/kcms/detail/11.2206.TS.20210406.1403.024.html.
ZHANG Q,ZHU D,NIU G C,et al.Bacterial diversity analysis during natural fermentation of sea buckthorn Jiaosu by high-throughput sequencing[J/OL].Food Science,2021.http://kns.cnki.net/kcms/detail/11.2206.TS.20210406.1403.024.html.
[4] YANG B,XIAO L,LIU S,et al.Exploration of the effect of probiotics supplementation on intestinal microbiota of food allergic mice[J].American Journal of Translational Research,2017,9(2):376-385.
[5] 索化夷,赵欣,骞宇,等.永川毛霉型豆豉在发酵过程中微生物总量与区系变化规律[J].食品科学,2015,36(19):124-131.
SUO H Y,ZHAO X,QIAN Y,et al.Changes in microbial load and flora during fermentation Yongchuan Mucor-type Douchi[J].Food Science,2015,36(19):124-131.
[6] 汪孟娟,陈廷涛,姜淑英,等.豆豉发酵中的微生物和功能性组分研究动态[J].中国微生态学杂志,2010,22(1):81-84.
WANG M J,CHEN T T,JIANG S Y,et al.The progress on the microflora and functional components in Douchi[J].Chinese Journal of Microecology,2010,22(1):81-84.
[7] 陈浩,樊游,陈源源,等.传统发酵豆制品中原核微生物多样性的研究[J].食品工业科技,2011,32(9):230-232.
CHEN H,FAN Y,CHEN Y Y,et al.Study on the prokaryotic microbial diversity of Chinese traditional fermented soybean foods[J].Science and Technology of Food Industry,2011,32(9):230-232.
[8] WASSIE M,WASSIE T.solation and identification of lactic acid bacteria from Iranian camel milk[J].International Journal of Advanced Biological and Biomedical Research,2020,8(1):67-74.
[9] TESHOME G.Review on lactic acid bacteria function in milk fermentation and preservation[J].African Journal of Food Science,2015,9(4):170-175.
[10] CAPPELLO M S,ZAPPAROLI G,LOGRIECO A,et al.Linking wine lactic acid bacteria diversity with wine aroma and flavour[J].International Journal of Food Microbiology,2017,243:16-27.
[11] 李巧玉,方芳,堵国成,等.魏斯氏菌在发酵食品中的应用[J].食品与发酵工业,2017,43(10):241-247.
LI Q Y,FANG F,DU G C,et al.The application of Weissella strains in fermented food[J].Food and Fermentation Industries,2017,43(10):241-247.
[12] 张平,张鹏飞,刘斯琪,等.自然发酵豆酱中明串珠菌的分离鉴定[J].食品科学,2018,39(22):110-115.
ZHANG P,ZHANG P F,LIU S Q,et al.Isolation and identification of Leuconostoc from traditional fermented soybean paste[J].Food Science,2018,39(22):110-115.
[13] 李璐,徐玉娟,温靖,等.益生菌对发酵菠萝果汁综合品质的影响[J].中国酿造,2021,40(1):105-110.
LI L,XU Y J,WEN J,et al.Effect of probiotics on the comprehensive quality of fermented pineapple juice[J].China Brewing,2021,40(1):105-110.
[14] 左丽丽,高永欣,王钰涓,等.传统发酵豆酱中乳酸菌的分离及鉴定[J].中国调味品,2020,45(11):117-120.
ZUO L L,GAO Y X,WANG Y J,et al.Separation and identification of lactic acid bacteria in traditionally fermented soybean paste[J].China Condiment,2020,45(11):117-120.
[15] 王博,周朝晖,李铁桥,等.嗜盐四联球菌及其在发酵食品中的应用[J].食品与发酵工业,2017,43(8):267-272.
WANG B,ZHOU Z H,LI T Q,et al.Tetragenococcus halophilus and its application in fermented foods[J].Food and Fermentation Industries,2017,43(8):267-272.
[16] 潘力,王斌,郭勇.曲霉菌的RAPD分析及其在酿造工业中的应用[J].微生物学报,2007,47(3):533-536.
PAN L,WANG B,GUO Y.RAPD analysis of Aspergilli and its application in brewing industry[J].Acta Microbiologica Sinica,2007,47(3):533-536.
[17] 张中义,畅晓霞,钟其顶.酒曲酶系,菌系特征及酿造过程中微生物动态变化[J].酿酒,2008,35(5):6.
ZHANG Z Y,SHEN X X,ZHONG Q D. Some rules and research methods of mold growth and enzyme formation in Monascus[J].Liquor Making,2008,35(5):6.
[18] 朱宏斌,沈伟,王竞,等.宏基因组研究高脂饮食诱导小鼠的肥胖易感性与肠道菌群的关系[J].第三军医大学学报,2017,39(8):773-780.
ZHU H B,SHEN W,WANG J,et al.Correlation of gut microbiota with susceptibility to high-fat diet-induced obesity in mice:A metagenomic study[J].Journal of Third Military Medical University,2017,39(8):773-780.
[19] 张颖,乌日娜,孙慧君,等.豆酱不同发酵阶段细菌群落多样性及动态变化分析[J].食品科学,2017,38(14):30-35.
ZHANG Y,WU R N,SUN H J,et al.Diversity and dynamic changes of the bacterial community during fermentation of soybean paste[J].Food Science,2017,38(14):30-35.
[20] 向凡舒,张振东,望诗琪,等.基于MiSeq高通量测序技术渣豆酱微生物类群研究[J].中国酿造,2020,39(9):48-52.
XIANG F S,ZHANG Z D,WANG S Q,et al.Microbiome of Zha-Doujiang by MiSeq high-throughput sequencing technology[J].China Brewing,2020,39(9):48-52.
[21] JUNG J Y,LEE S H,JEON C O.Microbial community dynamics during fermentation of Doenjang-meju,traditional Korean fermented soybean[J].International Journal of Food Microbiology,2014,185:112-120.
[22] 马岩石,姜明,李慧,等.基于高通量测序技术分析东北豆酱的微生物多样性[J].食品工业科技,2020,41(12):100-105.
MA Y S,JIANG M,LI H,et al.Analysis of microbial diversity of northeast soy sauce based on high-throughput sequencing technology[J].Science and Technology of Food Industry,2020,41(12):100-105.
[23] 韩俊燕,赵国忠,赵建新,等.发酵辣椒细菌多样性的16S rDNA 测序分析[J].中国食品学报,2018,18(5):246-251.
HAN J Y,ZHAO G Z,ZHAO J X,et al.16S rDNA sequencing analysis of bacterial diversity of fermented pepper[J].Journal of Chinese Institute of Food Science and Technology,2018,18(5):246-251.
[24] CUI R Y,ZHENG J,WU C D,et al.Effect of different halophilic microbial fermentation patterns on the volatile compound profiles and sensory properties of soy sauce moromi[J].European Food Research and Technology,2014,239(2):321-331.
[25] RYU J K,LEE H S,KOO B K,et al.Comparison of the biochemical activities of commercial yogurts and Lactobacillus acidophilus-containing yogurt[J].Korean Journal of Clinical Laboratory Science,2015,47(2):59-64.