甲壳素(C8H13O5 N)n,又称几丁质,是存在于各类甲壳动物的外壳及真菌细胞壁中的一种难溶的N-乙酰-D-葡萄糖胺聚合物。甲壳素脱乙酰率达到55%及以上即为壳聚糖,壳聚糖广泛应用于工业、农业、渔业、医疗产业等领域,尤其在生物医学领域,由于其良好的生物相容性,在组织工程、创伤愈合、生物传感器上有很好的应用前景[1-3]。
甲壳素脱乙酰酶(chitin deacetylase,CDA)是专一性的用于甲壳素脱去乙酰基的酶,它可代替现有的浓碱热解法,生产出化学法无法得到的某些高质量的壳聚糖[4-5]。红球菌属(Rhodococcus)是一类呈短杆状、不产孢子、革兰氏染色呈阳性的细菌,可由甲壳素诱导产CDA[6-7]。但研究发现,由于天然大分子甲壳素水溶性低,暴露的乙酰基团过少,导致红球菌无法高效利用天然大分子甲壳素,从而造成酶活力低,应用价值不高[1-2,7-9]。本文作者发现,将蜡状芽胞杆菌与红球菌混合培养时,培养基中的胶体甲壳素降解速度明显增快,对发酵液的脱乙酰酶活力测定结果显示,酶活力得到了极大的提高。本文将2株菌进行混合发酵,以单菌发酵为对比,同时监测甲壳素酶和CDA的酶活力变化,并对两菌的混合时间、接种量配比及发酵条件等单因素进行优化,并进一步设计Plackett-Burman(PB)实验、中心组合设计(central composite design,CCD)实验和响应面优化实验确定适宜两菌株协同发酵的培养基中各成分的水平,为工业化生产CDA提供参考。
1 材料与方法
1.1 实验材料
1.1.1 实验菌种及培养基
实验菌株:Rhodococcus HQcdag从潜江市虾池土壤中筛选得到,并经16S rRNA鉴定;Bacillus cereus CJPE209为本实验室保存菌株。
活化培养基(g/L):蛋白胨10,牛肉膏3,NaCl 5,琼脂 20,pH 6.6。
种子培养基(g/L):蛋白胨10,牛肉膏3,NaCl 5,琼脂 20,pH 6.6。
富集培养基(g/L):胶体甲壳素5,NaCl 8,K2HPO4 0.5,KH2PO4 1,MgSO4 0.5,pH 6.8。
筛选培养基(g/L):胶体甲壳素5,NaCl 8,K2HPO4 0.5,KH2PO4 1,MgSO4 0.5,对硝基乙酰苯胺0.2,琼脂20,pH 7。
基础发酵培养基(g/L):胶体甲壳素5,酪蛋白2,酵母浸粉2,NaCl 8,K2HPO4 0.5,KH2PO4 1,MgSO4 0.5,pH 6.8。
优化发酵培养基(g/L):蛋白胨2.5,K2HPO4 0.6,MgSO4 1.5,酵母浸粉3.0,NaCl 8.5,KH2PO4 0.8,胶体甲壳素8.0。
1.1.2 实验材料
甲壳素,上海源叶生物有限公司;对硝基乙酰苯胺,上海麦克林生化科技有限公司;胶体甲壳素,自制。
1.1.3 仪器与设备
FW100高速万能粉碎机,天津市泰斯特仪器有限公司;YXQ-LS—75211型立式压力蒸汽灭菌器,上海博迅实业有限公司;HH-S型水浴锅,巩义市矛华仪器有限公司;ZSD-1270型全自动生化培养箱,上海智诚分析仪器制造有限公司;TG16-II,高速离心机长沙平凡仪器仪表有限公司;SYNERGY2酶标仪,Gene Company Limited;ZHWY系列双层恒温培养振荡器,中国科学院武汉科学仪器厂。
1.2 实验方法
1.2.1 双菌株协同作用验证
将蜡状芽胞杆菌和红球菌37 ℃分别培养24 h制备种子液,将其单独和按活菌数1∶1接种于基础发酵培养基中,37 ℃、180 r/min发酵36 h,同时监测CDA酶和甲壳素酶的酶活力变化,采用变色圈法[9-10]测定CDA的酶活力,甲壳素酶活力用3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法测定[11]。
1.2.2 双菌株协同发酵条件优化
1.2.2.1 菌种活化
分别测定两菌株的生长曲线,确定其最佳的混合接种时间。
1.2.2.2 种子液的配比优化
根据测定的两菌株的生长曲线,取两菌株的生长对数期用血球计数法测定活菌数,然后按一定数量比例(10∶0、9∶1、8∶2、7∶3、6∶4、5∶5、4∶6、3∶7、2∶8、1∶9、0∶10)换算成体积比同时接入基础发酵培养基中发酵。以对硝基乙酰苯胺为底物,用分光光度法测定酶活力。
1.2.2.3 PB实验设计
发酵条件优化:由上述实验所得种子液最佳配比,在其他条件不变的前提下,依次改变发酵周期、发酵温度、发酵转速、装液量、接种量5因素的水平,用分光光度法测定酶活力,确定促进产酶的最适条件。
发酵培养基成分优化:由上述实验所得最佳发酵条件,在其他条件不变的前提下,依次改变基础发酵培养基的碳源、氮源、无机盐等,确定发酵培养基的最佳组分。在单因素实验的基础上,将上述所得发酵培养基组分根据PB设计正交实验,筛选出影响酶活力的显著性因子[7,12-14],实验的因素水平如表1所示,CDA的酶活力(Y)作为响应值。
表1 Plackett-Burman实验设计因素与水平 单位:g/L
Table 1 Factors and levels of Plackett-Burman design

代码因素水平-1+1X1蛋白胨2.503.75X2酵母浸粉2.503.75X3NaCl5.07.5X4K2HPO41.01.5X5KH2PO41.01.5X6MgSO40.500.75X7胶体甲壳素5.07.5
1.2.2.4 响应面实验设计
基于PB实验的结果,根据CCD中心组合设计原理,以酵母浸粉(X2),NaCl(X3),KH2PO4(X5),胶体甲壳素(X7)4个因素为自变量,以酶活力为响应值,设计4因素5水平响应面实验,因素水平如表2所示[7,12-14]。
表2 CCD设计试验的因素与水平 单位:g/L
Table 2 Factors and levels of variables of CCD
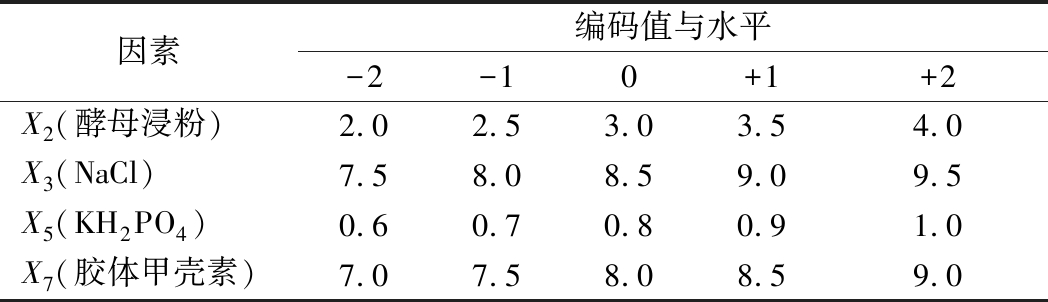
因素编码值与水平-2-10+1+2X2(酵母浸粉)2.02.53.03.54.0X3(NaCl)7.58.08.59.09.5X5(KH2PO4)0.60.70.80.91.0X7(胶体甲壳素)7.07.58.08.59.0
1.2.2.5 数据处理
每个指标重复测定3次,采用Design Expert 8.0软件进行PB实验、CCD实验、响应面实验设计及其数据分析。
2 结果与分析
2.1 双菌株协同发酵效果验证
由图1可知,蜡状芽胞杆菌单独发酵时只产甲壳素酶,红球菌单独发酵只产CDA,但酶活力并不太高。两菌混合发酵时CDA的活力在24 h时最高,比红球菌单独发酵提前了约12 h,且最高点约为其单独发酵的10倍。此外,混合发酵时甲壳素酶的活力与杆菌单独发酵相比也有所提高。可见,甲壳素酶和CDA共同作用,不仅提高了底物胶体甲壳素的降解速率和效果,还对各自酶的产生有促进作用。

图1 菌株B.cereus、Rhodococcus和两菌混合发酵过程中甲壳素酶和甲壳素脱乙酰酶酶活力的变化
Fig.1 Effects of culture time on activity of chitin deacetylase and chitinase during B.cereus,Rhodococcus and co-fermentation
2.2 双菌株协同发酵条件优化
2.2.1 菌种活化
两菌株活化后所测生长曲线如图2所示,由于生长对数期的细菌生长旺盛,产酶活力强。因此选取皆处于两菌株生长对数期的第20 h的种子液,将两菌按比例接入发酵培养基中。
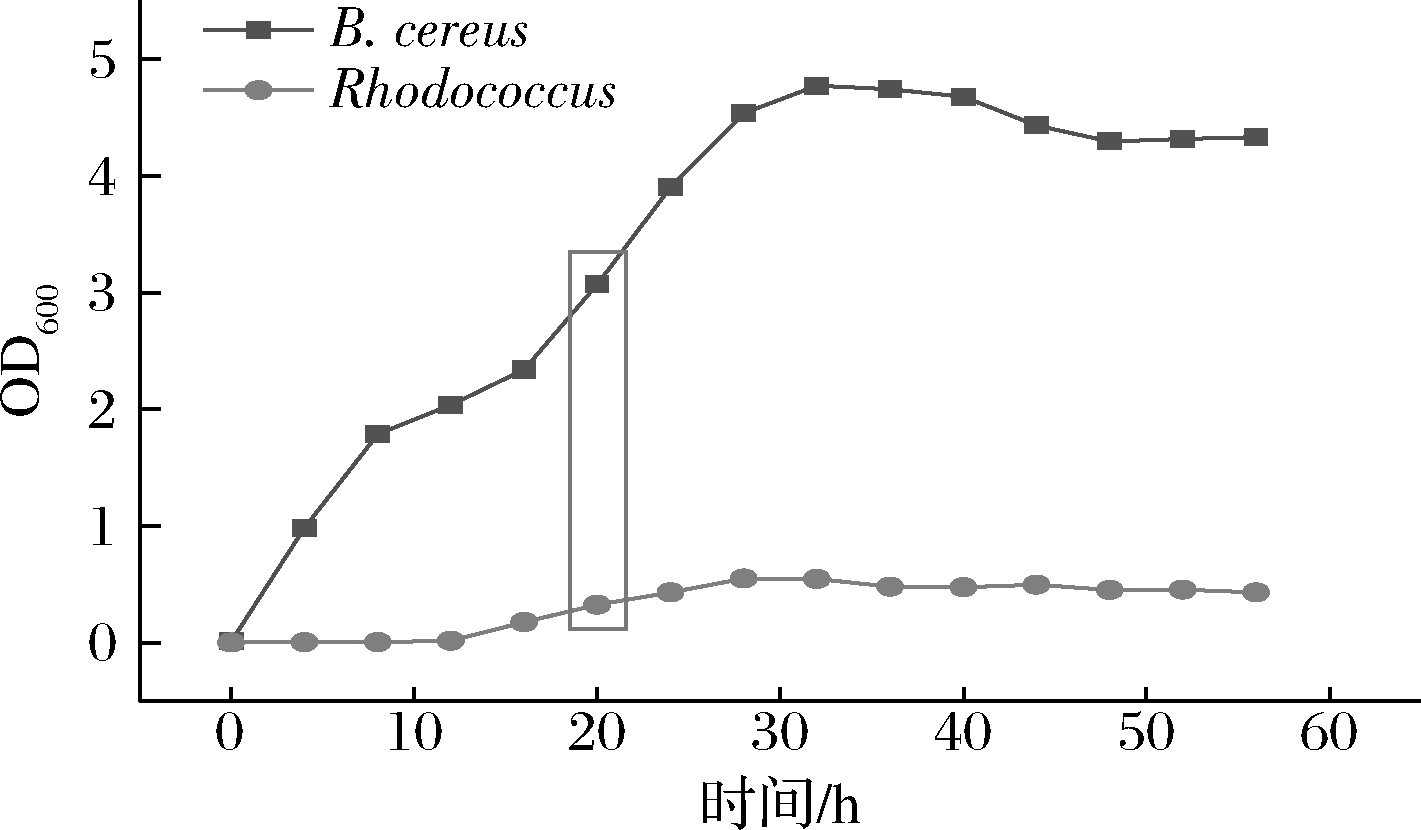
图2 菌株B.cereus和Rhodococcus的生长曲线
Fig.2 Growth curve of strain B.cereus and Rhodococcus
2.2.2 种子液优化
由图3可知,蜡状芽胞杆菌单独发酵时CDA的酶活力为1.43 U,可忽略不计。红球菌单独发酵时,CDA的酶活力为19.47 U,经过混合发酵后,发酵液中的酶活力大幅提升。而当配比为蜡状芽胞杆菌∶红球菌=2∶8时,发酵液中的CDA的酶活力达到最高值,为226.27 U,然后随着蜡状芽孢杆菌比例的增加而逐渐降低。这意味着协同发酵产酶的过程是以红球菌为主导,在此过程中借助蜡状芽胞杆菌分泌的糖苷酶对底物甲壳素的糖链进行水解,使其更易被红球菌利用或其他方式使得酶活力大幅提升。而杆菌比重逐渐增大后,培养液的酶活力呈现下降趋势,很可能是由于杆菌挤压了球菌的生存空间导致。
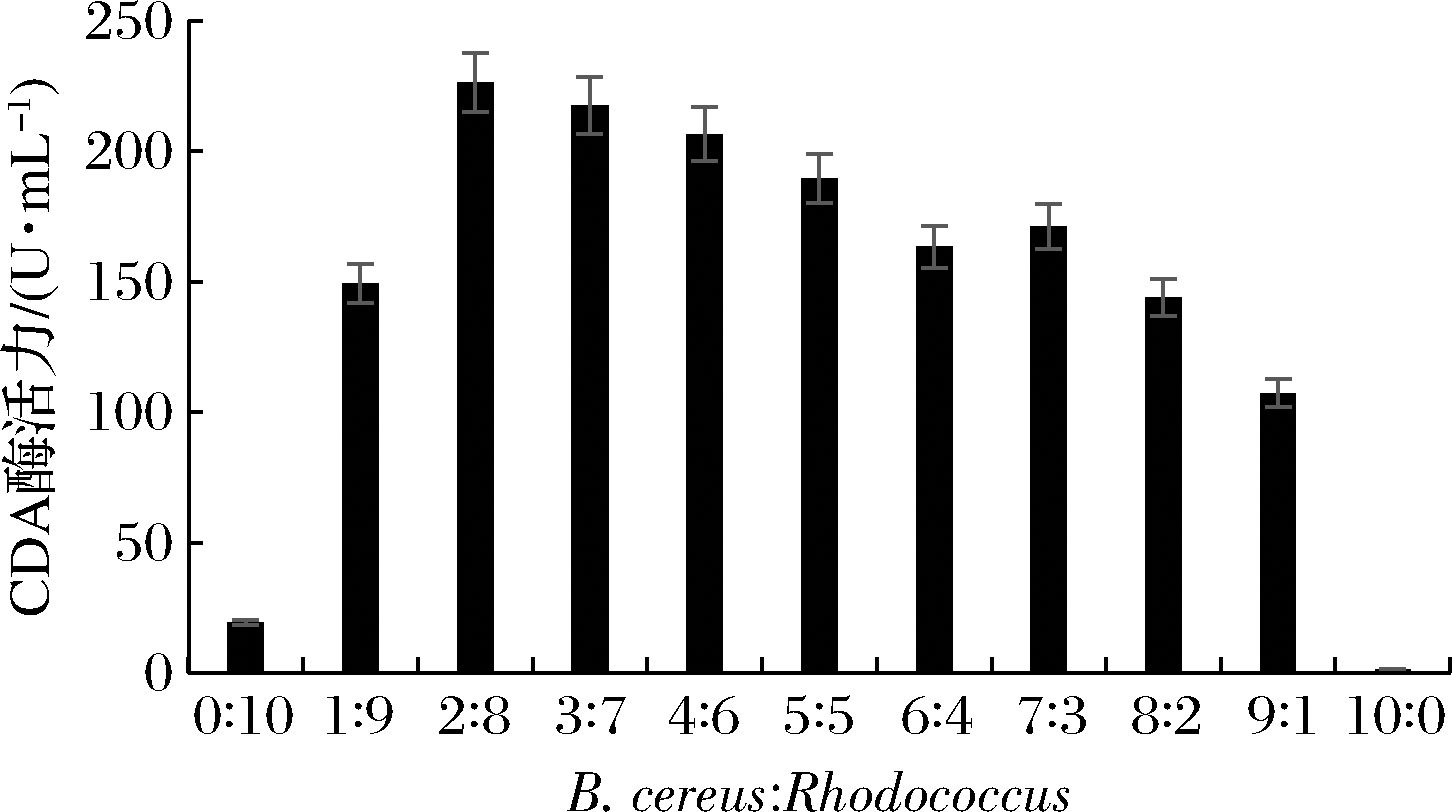
图3 菌株B.cereus和Rhodococcus的接种比例对酶活力的影响
Fig.3 Effects of ratio of B/R on activity of chitin deacetylase
2.3 单因素实验结果
2.3.1 发酵条件的优化
发酵条件优化结果表明,促进产酶的最佳单因素条件分别为发酵周期24 h,发酵温度35 ℃,转速160 r/min,装液量50 mL/250 mL,接种量3%。发酵条件的交互影响较小,因此,在确定发酵条件的基础上,再对发酵培养基的成分进一步优化。
2.3.2 PB实验设计结果
PB实验的分析结果如表3所示,模型具有高度的显著性,失拟项不显著。其中酵母浸粉(X2)、NaCl(X3)、KH2PO4(X5)和胶体甲壳素(X7)的P值在5%水平上是显著的,这说明酵母浸粉、NaCl、KH2PO4和胶体甲壳素是促进CDA产生的显著影响因素。
表3 PB设计试验的回归分析
Table 3 Regression analysis for the PB design
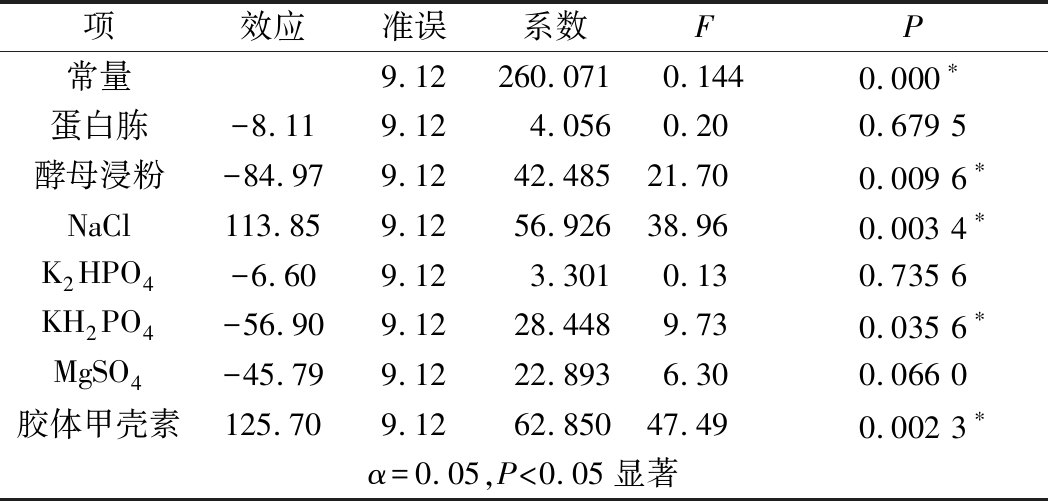
项效应准误系数FP常量9.12260.0710.1440.000∗蛋白胨-8.119.124.0560.200.679 5酵母浸粉-84.979.1242.48521.700.009 6∗NaCl113.859.1256.92638.960.003 4∗K2HPO4-6.609.123.3010.130.735 6KH2PO4-56.909.1228.4489.730.035 6∗MgSO4-45.799.1222.8936.300.066 0胶体甲壳素125.70 9.1262.85047.490.002 3∗α=0.05,P<0.05显著
注:*代表显著因素
2.3.3 响应面实验设计结果
响应面运行结果如表4所示,Yα为检测值,![]() 为预测值。由表4可知,第7组中培养基配比为酵母浸粉3.0 g/L、NaCl 8.5 g/L、KH2PO4 0.8 g/L、胶体甲壳素8.0 g/L时检测到的CDA的酶活力最高,为443.37 U/mL。用Design Expert 8.0软件对表4实验数据进行二次多项式回归拟合,得到二次多元回归方程:
为预测值。由表4可知,第7组中培养基配比为酵母浸粉3.0 g/L、NaCl 8.5 g/L、KH2PO4 0.8 g/L、胶体甲壳素8.0 g/L时检测到的CDA的酶活力最高,为443.37 U/mL。用Design Expert 8.0软件对表4实验数据进行二次多项式回归拟合,得到二次多元回归方程:![]()
表4 CCD设计试验与结果
Table 4 Experiment design and results of CCD
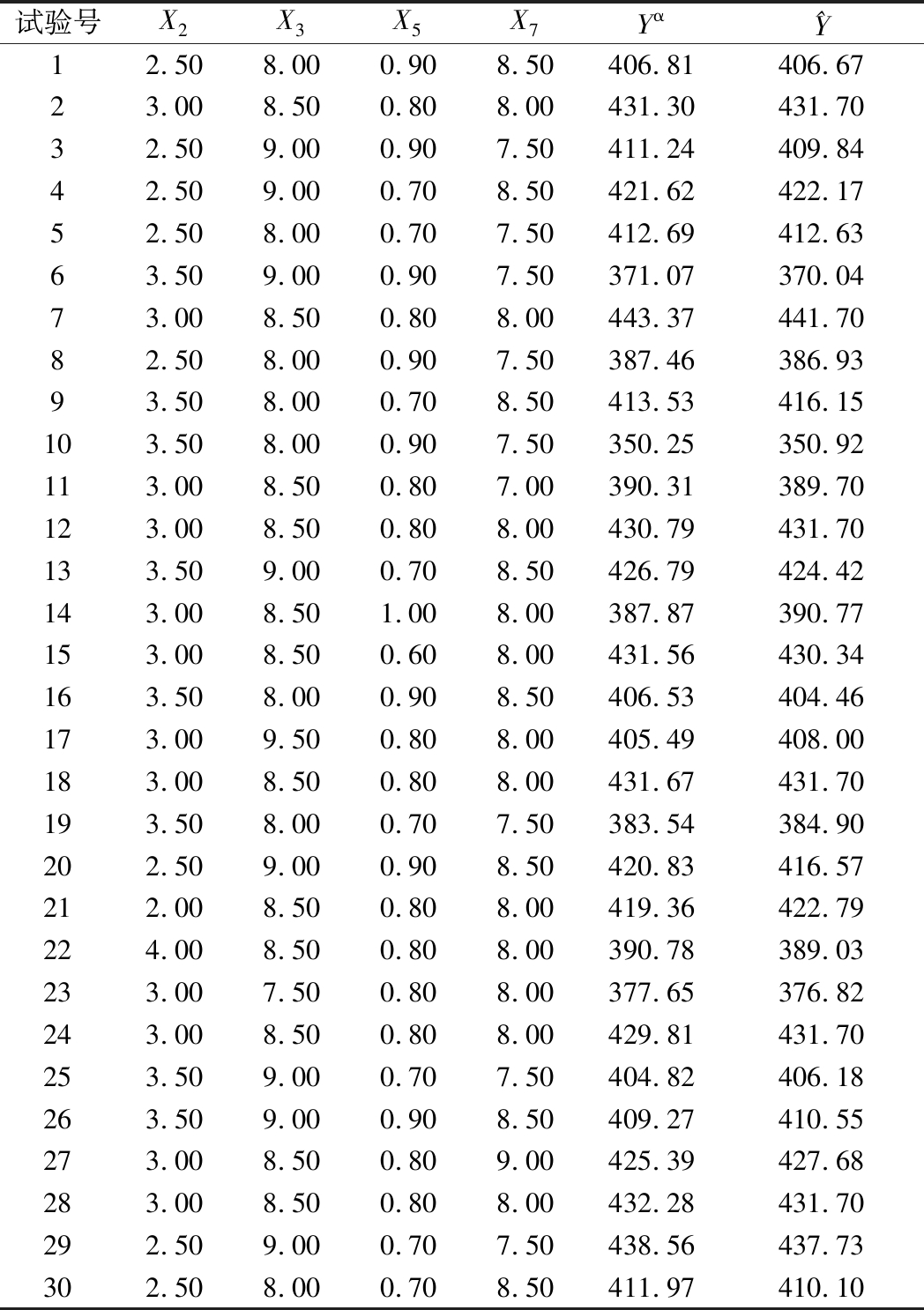
试验号X2X3X5X7Yα^Y12.508.000.908.50406.81406.6723.008.500.808.00431.30431.7032.509.000.907.50411.24409.8442.509.000.708.50421.62422.1752.508.000.707.50412.69412.6363.509.000.907.50371.07370.0473.008.500.808.00443.37441.7082.508.000.907.50387.46386.9393.508.000.708.50413.53416.15103.508.000.907.50350.25350.92113.008.500.807.00390.31389.70123.008.500.808.00430.79431.70133.509.000.708.50426.79424.42143.008.501.008.00387.87390.77153.008.500.608.00431.56430.34163.508.000.908.50406.53404.46173.009.500.808.00405.49408.00183.008.500.808.00431.67431.70193.508.000.707.50383.54384.90202.509.000.908.50420.83416.57212.008.500.808.00419.36422.79224.008.500.808.00390.78389.03233.007.500.808.00377.65376.82243.008.500.808.00429.81431.70253.509.000.707.50404.82406.18263.509.000.908.50409.27410.55273.008.500.809.00425.39427.68283.008.500.808.00432.28431.70292.509.000.707.50438.56437.73302.508.000.708.50411.97410.10
采用Design Expert 8.0软件对表4的实验结果进行分析。由表5可知,模型具有高度的显著性,失拟项不显著,R2=99.3%,表明回归方程对实验拟合情况较好,检验结果与模型计算结果无显著差异,约有99.3%实验结果可以用此模型来解释。其中,![]() 和交互因素X2X5、X2X7、X3X7、X5X7对产CDA有显著作用。
和交互因素X2X5、X2X7、X3X7、X5X7对产CDA有显著作用。
表5 响应面二次式模型的方差分析表
Table 5 ANOVA table of response surface quadratic model
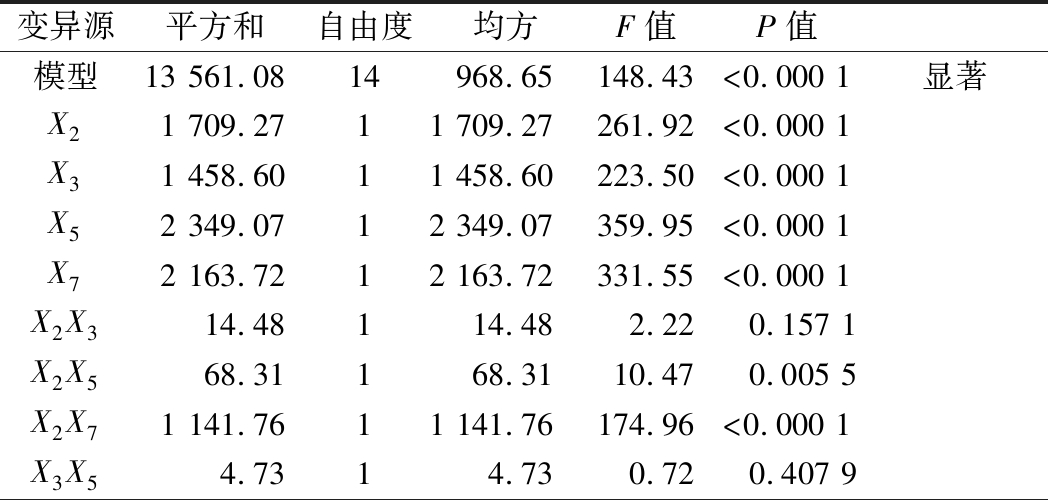
变异源平方和自由度均方F值P值模型13 561.0814968.65148.43<0.000 1显著X21 709.2711 709.27261.92<0.000 1X31 458.6011 458.60223.50<0.000 1X52 349.0712 349.07359.95<0.000 1X72 163.7212 163.72331.55<0.000 1X2X314.48114.482.22 0.157 1X2X568.31168.3110.47 0.005 5X2X71 141.7611 141.76174.96<0.000 1X3X54.7314.730.72 0.407 9
续表5

变异源平方和自由度均方F值P值X3X7169.521169.5225.980.000 1X5X7496.401496.4076.06<0.000 1X221 140.5111 140.51174.76<0.000 1X232 646.8012 646.80405.58<0.000 1X25766.721766.72117.49<0.000 1X27907.911907.91139.12<0.000 1残差97.89156.53失拟项85.86108.593.57 0.086 4不显著纯误差12.0352.41总离差13 658.9729R2=0.992 8 R2(调整)=0.986 1 R2(预测)=0.962 5
通过软件对回归方程中的X2X5、X2X7、X3X7、X5X7交互项绘制响应面分析图,结果如图4所示。用方程预测的结果表明,酵母浸粉3.0 g/L、NaCl 8.5 g/L、KH2PO4 0.8 g/L、甲壳素8.0 g/L是CDA生产的最佳发酵培养基组成,与表4中的运行结果一致。
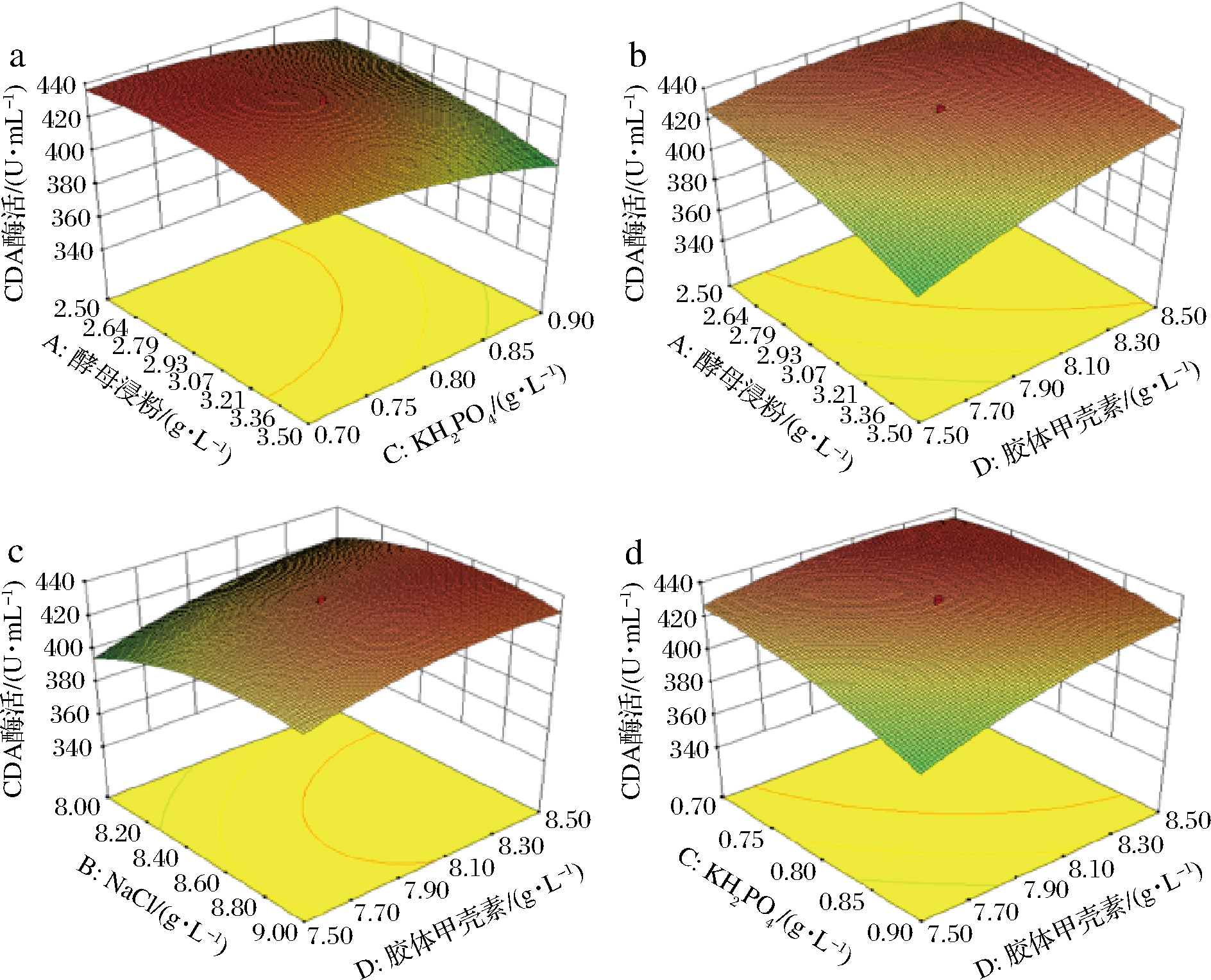
a-酵母浸粉和KH2PO4;b-酵母浸粉和胶体甲壳素;c-NaCl和胶体甲壳素;d-KH2PO4和胶体甲壳素
图4 不同因素的交互作用对混合菌株发酵产CDA的影响响应面图
Fig.4 Response surface contour plots of CDA production by complex bacteria optimization of variables
3 结论
本文发现了蜡状芽孢杆菌和红球菌混合发酵,可大幅提高CDA产量的现象,并测得两菌株接种量最佳配比为2∶8,相比红球菌单独发酵酶活力提升了约12倍。对混合发酵产CDA的培养基条件进行响应面优化。响应面实验结果表明,两菌协同发酵培养基的最优组分为:蛋白胨0.25 g/L、K2HPO4 0.6 g/L、MgSO4 1.5 g/L、酵母浸粉3.0 g/L、NaCl 8.5 g/L、KH2PO4 0.8 g/L、胶体甲壳素8.0 g/L。优化后菌株产CDA的活力约为优化前的1.96倍。
天然甲壳素由于其高度延伸的氢键半晶结构使其难以溶于一般的稀酸、稀碱及有机溶剂,在没有经过处理的情况下难以被CDA作用[2,15-18]。有文献表明,在预先使用类似糖苷酶的多糖裂解单加氧酶氧化裂解甲壳素原纤维表面上的糖链后,其晶体内部的乙酰基暴露出来,大大加快了CDA后续的脱乙酰进程[16,19-20]。根据文献提示,蜡状芽胞杆菌可产多种糖苷酶(如甲壳素酶),因此产生此现象的原因很可能是由于蜡状芽胞杆菌产生的甲壳素酶通过作用于糖链的糖苷键,使甲壳素分子链长度减小并破坏其晶态结构,内部乙酰基暴露从而更易被CDA作用的同时诱导红球菌产生更多的CDA[1,15-17,21]。
作者已验证了蜡状芽胞杆菌中甲壳素酶基因(CP028009.1)和红球菌中甲壳素脱乙酰酶基因(MH244430.1)的存在。下一阶段将通过对其及壳聚糖酶(CP029454.1)、甲壳二糖酶(CP001176.1)等甲壳素酶系基因的转录水平分析,来进一步揭示双菌株协同降解甲壳素,并促进红球菌产脱乙酰酶的机理。同时作者也在进一步研究诱导红球菌产脱乙酰酶的物质,实验发现,蜡状芽胞杆菌单独发酵灭菌后的培养液仍能诱导红球菌产脱乙酰酶。因此下一阶段将对该菌株发酵甲壳素的产物进行检测和分离,并对红球菌做产酶诱导实验。但发酵液中成分复杂,要准确的分析出发酵液中产酶诱导物的组分,对于其纯化制备的精度和产量有着很高的要求。
[1] SILVA D S,DELEZUK J A M,LA PORTA F A,et al.Comparison of experimental and theoretical data on the structural and electronic characterization of chitin and chitosan[J].Current Physical Chemistry,2016,5(3):206-213.
[2] CRINI G.Historical review on chitin and chitosan biopolymers[J].Environmental Chemistry Letters,2019,17(4):1 623-1 643.
[3] LIN S,QIN Z,CHEN Q M,et al.Efficient immobilization of bacterial GH family 46 chitosanase by carbohydrate-binding module fusion for the controllable preparation of chitooligosaccharides[J].Journal of Agricultural and Food Chemistry,2019,67(24):6 847-6 855.
[4] ARAGUNDE H,BIARNÉS X,PLANAS A.Substrate recognition and specificity of chitin deacetylases and related family 4 carbohydrate esterases[J].International Journal of Molecular Sciences,2018,19(2):412.
[5] BLAIR D E,HEKMAT O,SCHüTTELKOPF A W,et al.Structure and mechanism of chitin deacetylase from the fungal pathogen Colletotrichum lindemuthianum[J].Biochemistry,2006,45(31):9 416-9 426.
[6] 华苟根,郭坚华.红球菌属的分类及应用研究进展[J].微生物学通报,2003,30(4):107-111.
HUA G G,GUO J H.The taxonomy and application of Rhodococcus[J].Microbiology,2003,30(4):107-111.
[7] SUN Y Y,ZHANG J Q,WU S J,et al.Statistical optimization for production of chitin deacetylase from Rhodococcus erythropolis HG05[J].Carbohydrate Polymers,2014,102:649-652.
[8] MA Q Y,GAO X Z,BI X Y,et al.Isolation,characterisation,and genome sequencing of Rhodococcus equi:A novel strain producing chitin deacetylase[J].Scientific Reports,2020,10:4329.
[9] 柴金龙,王敏卜,杭加豪,等.产几丁质脱乙酰酶海洋细菌的筛选鉴定及产酶条件优化[J].中国酿造,2019,38(10):89-95.
CHAI J L,WANG M B,HANG J H,et al.Screening,identification and enzyme production conditions optimization of chitin deacetylase-producing marine bacteria[J].China Brewing,2019,38(10):89-95.
[10] LIU J L,JIA Z J,LI S,et al.Identification and characterization of a chitin deacetylase from a metagenomic library of deep-sea sediments of the Arctic Ocean[J].Gene,2016,590(1):79-84.
[11] 陈立功,吴家葳,张庆芳,等.产低温几丁质酶菌株的筛选、鉴定与产酶条件优化[J].食品工业科技,2020,41(23):85-90;98.
CHEN L G,WU J W,ZHANG Q F,et al.Screening and identification of a cold-adapted chitinolytic bacterium strain and optimization of its chitinase fermentation conditions[J].Science and Technology of Food Industry,2020,41(23):85-90;98.
[12] HE Y H,XU J P,WANG S J,et al.Optimization of medium components for production of chitin deacetylase by Bacillus amyloliquefaciens Z7,using response surface methodology[J].Biotechnology & Biotechnological Equipment,2014,28(2):242-247.
[13] LIAQAT F,SÖZER BAHADI R P,ELIBOL M,et al.Optimization of chitosanase production by Bacillus mojavensis EGE-B-5.2i[J].Journal of Basic Microbiology,2018,58(10):836-847.
[14] REN J,LIN W T,SHEN Y J,et al.Optimization of fermentation media for nitrite oxidizing bacteria using sequential statistical design[J].Bioresource Technology,2008,99(17):7 923-7 927.
[15] WANG Z W.Advance in study and application on chitinase produced by microbes[J].Letters in Biotechnology,2006,17(3):439-442.
[16] VIENS P,DUBEAU M P,KIMURA A,et al.Uptake of chitosan-derived D-glucosamine oligosaccharides in Streptomyces coelicolor A3(2)[J].FEMS Microbiology Letters,2015,362(9):fnv048.
[17] WIN N,STEVENS W.Shrimp chitin as substrate for fungal chitin deacetylase[J].Applied Microbiology and Biotechnology,2001,57(3):334-341.
[18] NAQVI S,CORD-LANDWEHR S,SINGH R,et al.A recombinant fungal chitin deacetylase produces fully defined chitosan oligomers with novel patterns of acetylation[J].Applied and Environmental Microbiology,2016,82(22):6 645-6 655.
[19] LIU Z L,GAY L M,TUVENG T R,et al.Structure and function of a broad-specificity chitin deacetylase from Aspergillus nidulans FGSC A4[J].Scientific Reports,2017,7(1):1746.
[20] CHAMBON R,PRADEAU S,FORT S,et al.High yield production of Rhizobium NodB chitin deacetylase and its use for in vitro synthesis of lipo-chitinoligosaccharide precursors[J].Carbohydrate Research,2017,442:25-30.
[21] 刘力睿,潘杰,李猛.微生物几丁质酶的研究进展、应用及展望[J].生物资源,2020,42(5):494-504.
LIU L R,PAN J,LI M.Recent research progress,application and future perspectives on microbial chitinases[J].Biotic Resources,2020,42(5):494-504.