大曲是中国传统白酒酿造中特有的关键糖化发酵剂,主要包括菌系、酶系和物系,菌系和酶系是生成乙醇和复杂风味化合物产生的必要条件,物系为酿酒提供部分发酵原料和风味形成的前体物质[1],它们质量的好坏直接决定着酒的产率和品质,素有“曲为酒之骨”之说[2]。青稞酒以青稞为原料,中低温青稞大曲为糖化发酵剂,采用固态发酵和固态蒸馏取酒,是中国白酒酿造中唯一采用曲粮合一的传统工艺,具有清蒸清烧四次清、花岗岩窖池发酵的工艺特点。
大曲的制作是开放式的混菌发酵,在自然发酵过程中富集了环境中的大量微生物,微生物群落结构及其演变对于大曲制作的过程条件把控和最终曲的质量具有重要作用。王鹏等[3]根据风味贡献程度、相互作用关系强度和相对丰度确定 Lactobacillus、Saccharomyces、Candida、Rhizopus、Saccharomycopsis、Pichia、Dipodascus、Bacillus、Thermoascus 和 Lactococcus 等 10 个属为浓香型白酒发酵过程的核心微生物群。WANG 等[4]借助高通量扩增子测序解析浓香型白酒酒醅、大曲和窖泥原核微生物群落结构差异,判定发酵酒醅微生物主要来源。同时,大曲原料的配比、质量的优劣以及不同的工艺技术方法,对大曲的类别和质量有很大的影响。目前对于青稞酒的研究主要集中在酿造微生物及原酒风味组分[5],对青稞大曲制作过程中微生物菌群结构及其群落演变并不太清楚,解析青稞大曲制做过程中微生物群落演替规律,判定制曲过程中核心功能微生物及其驱动因子对于今后提升青稞大曲的质量至关重要。王鹏[6]研究了浓香型白酒酒醅发酵过程中的核心微生物,通过核心功能微生物的强化应用,提升了白酒酿造品质的控制。本文通过高通量扩增子测序解析3种不同配料大曲发酵过程中的微生物群落结构,采用气质联用测定大曲发酵过程中不同时间点样品中的重要风味化合物,并使用多元统计学分析发酵过程中微生物菌群结构及群落演替与主要代谢物质之间的网络共线性关系,找出大曲发酵过程中的优势微生物种群,为后期青稞大曲功能微生物的筛选及强化大曲品质提供理论依据。
1 材料与方法
1.1 试验样品
实验样品取自某青稞酒厂两种不同配料大曲,分别在制曲过程第 0、2、3、4、5、8、12、15和30 d(出房)取样,于-20 ℃冰箱保藏备用。
1.2 试验试剂和仪器
1.2.1 主要试剂
NaH2PO4、Na2HPO4、NaOH、NaCl,国药(集团)上海化学试剂有限公司;异戊醇氯仿、乙酸钠、十二烷基硫酸钠(sodium dodecyl snlfate,SPS)、饱和苯酚溶液,生工生物工程(上海)股份有限公司。
0.1 mm玻璃珠、0.5 cm玻璃珠,Sigma公司。
1.2.2 溶液配制
0.1 mol/L PBS缓冲液:0.05 mol/L Na2HPO4、0.042 3 mol/L NaH2PO4(精确称取8.19 g Na2HPO4和5.075 g NaH2PO4,加入去离子水定容至1 L,121 ℃高压灭菌15 min,保存备用)。
Buffer Z溶液:10 mmol/L Tris-HCl、150 mmol/L NaCl(母液1 mol/L的Tris-HCl,精确称取12.1 g Tris加入到80 mL去离子水中,再加入4.2 mL浓盐酸,定容至100 mL,pH 8.0,121 ℃高压灭菌15 min保存备用)。
100 g/L SDS溶液:精确称取10 g SDS溶于去离子水中,定容至100 mL,121 ℃高压灭菌15 min备用。
1.2.3 主要仪器
电子天平,Mettler-Toledo 公司;Centrifuge 5804R高速冷冻离心机,德国Eppendorf公司,Milli-Q 超纯水系统,美国Millipore 公司;607EUR珠磨仪,美国Biospec Products公司;Nano Drop—1000紫外可见分光光度计,核酸浓度检测仪,美国赛默飞科技公司;GC6890 N-MSD5975气相色谱质谱联用仪,美国安捷伦科技有限公司。
1.3 试验方法
1.3.1 大曲和酒醅理化指标的测定
大曲和酒醅的水分、酸度、淀粉含量、糖化酶、淀粉酶和酯化酶等理化指标均依据QB/T 4257—2011 方法测定,所有分析均以干重计。大曲温度采用温度计实时测定。酒醅温度使用温度计插入窖池1.2 m深处测定。
1.3.2 样品中挥发性化合物含量的检测
1.3.2.1 样品的预处理
取5.000 g待测样品,置于50 mL离心管中,加入去离子水20 mL,振荡1 min混匀,冰浴超声波30 min,再振荡1 min,8 000 r/min离心5 min,取8 mL上清液置于装有3 g NaCl的顶空瓶中。
1.3.2.2 GC-MS检测
挥发性化合物的测定采用气相色谱-质谱联用仪;Agilent 6890 N气相色谱仪和Agilent 5975质谱检测器,色谱柱为DB-Wax(30 m×0.25 mm×0.25 μm;J&W Scientific),内标为分析级薄荷醇(106.25 mg/L),检测条件参考文献[7]。
1.3.3 样本DNA 提取、PCR扩增和高通量测序
称取7 g酒醅样品,参照李小龙[8]等的方法提取样品中的DNA。细菌用16S的V3-V4区引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GACTACHVGGGTWTCTAAT-3′)进行扩增;对于真菌,用ITS2区引物ITS3(5′-GCATCGATGAAGAACGCAGC-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC-3′)扩增[8]。PCR 细菌扩增程序为:95 ℃预热5 min,95 ℃变性30 s,55 ℃退火30 s;72 ℃延伸40 s,共25个循环,最后72 ℃ 保持10 min。真菌扩增程序为:95 ℃预热2 min,95 ℃变性30 s,61 ℃退火30 s,72 ℃延伸45 s,共32个循环,最后72 ℃保持10 min。PCR产物根据已有的报道方法进行纯化[9]。纯化后的产物用Nanodrop ND-1000 UV-Vis 定量,然后将PCR 产物等分子量混合,并根据Low Sample Protocol进行文库的制备,最后在 2×300 Illumina MiSeq平台进行双端测序。
1.3.4 序列处理分析
对测序下机的原始序列进行去除标签、引物和接头序列及去除嵌合体等处理。然后把高质量的序列根据97%的序列相似度聚成不同的操作性分类单元(operational taxonomic unit,OTU)[8,10],并计算Chao1、shannon多样性指数[11]。
1.4 数据分析
1.4.1 主成分分析
根据高通量扩增子测序数据,统计不同取样点微生物属的相对丰度,借助SPSS 19.0进行主成分分析,揭示大曲制作和酒醅发酵过程中微生物群落的演替轨迹。
1.4.2 冗余分析
通过R语言的Vegan包计算制曲过程中环境因子和微生物群落之间的关系,分析环境因子对群落结构的影响,并进行蒙特卡洛置换检验[12]。
1.4.3 网络图绘制
用SPSS软件计算微生物与代谢物之间的Pearson相关系数,找出相关系数显著并且大于0.5的作为可视化对象[13]。使用Gephi软件对大曲发酵过程中微生物和代谢物之间的相关关系进行可视化,判定与制曲过程中的核心微生物[14]。
2 结果与分析
2.1 制曲过程微生物多样性及菌群结构
采用MiSeq 测序技术确定2种配料大曲制作过程中的微生物群落组成。通过质控后,细菌得到1 064 488条高质量的序列,平均每个样本有(53 339±6 000)条;获得6 735个OTUs,每个样品得到(374±105)个OTUs。真菌质控后共得到1 277 271条高质量的序列,平均每个样本具有(70 959±29 131)条;获得4 665个OTUs,每个样品得到(259±45)个OTUs。各样品的覆盖率均在99%以上,说明样本测序深度和数据质量足够可靠[15]。
比较了2种不同配料方案大曲发酵过程中微生物Chao1和Shannon指数,如图1所示,方案一[m(青稞)∶m(豌豆)=7∶3]大曲中原核微生物群落的Chao1和Shannon指数要低于方案二[m(青稞)∶m(豌豆) ∶m(小麦)= 5∶3∶2],2种方案大曲中真菌Chao1和Shannon指数没有明显的差异,表明2种不同方案细菌微生物多样性差异显著,真菌物种多样性没有明显的差异。
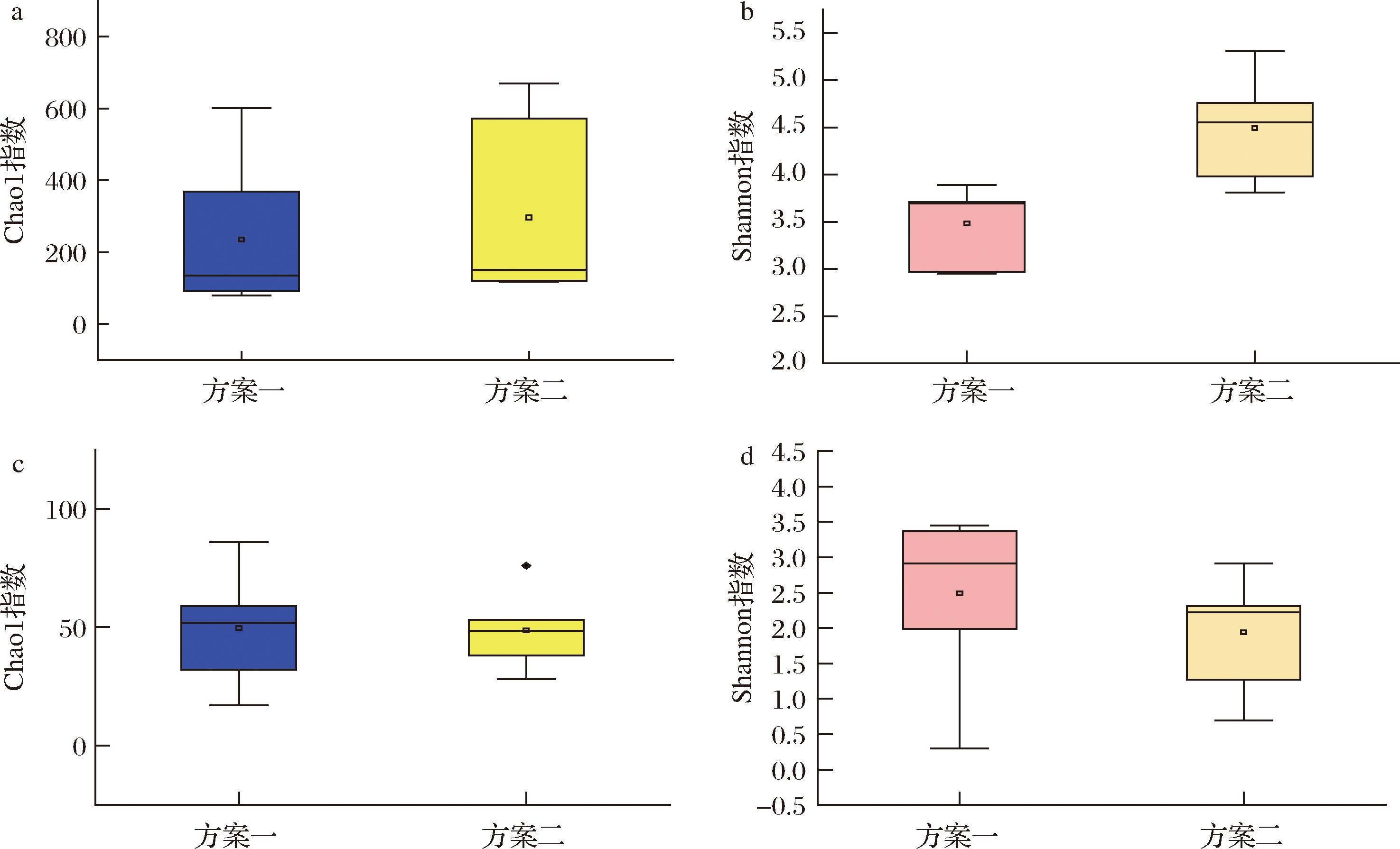
a-细菌Chao1指数;b-细菌Shannon指数;c-真菌Chaol指数;d-真菌Shannon 指数
图1 不同配料大曲微生物组成多样性分析
Fig.1 Diversity analysis of microbial composition of Daqu with different ingredients
注:方案一-m(青稞)∶m(豌豆)=7∶3;方案二-m(青稞)∶m(豌豆)∶m(小麦)=5∶3∶2 (下同)
基于OTU相对丰度的主成分分析表明(图2),方案一和方案二在前期(0和5)细菌和真菌群落都较为相似,而从发酵第5天开始,2种方案细菌群落演替轨迹比较相似,真菌群落演替出现明显的分化。

a-细菌;b-真菌
图2 不同配料大曲制作过程中基于OTU水平的微生物群落的主成分分析
Fig.2 Principal component analysis of microbial community based on OTU level during the production of Daqu with different ingredients
利用高通量测序解析了不同配料大曲中微生物群落结构和组分,图3为2种不同配料方案大曲中微生物群落结构和组分图(OTU≥0.1%)。统计发现,真菌群落中0~4 d主要由Saccharomycopsis、Alternaria、Cladosporium主导,从第4天开始一直到出房,Saccharomycopsis逐渐占据优势,同时Pichia和Wickerhamomyces具有一定的优势。对于细菌,在整个过程中Pantoea占到绝对的优势,Staphylococcus也具有很高的优势,在2个方案中占比没有明显差异。总的来说,青稞大曲制作过程中真菌Pichia、Saccharomyces具有较高的优势,Lactobacillus在发酵前期所占比例上升平稳,在发酵后期在酒醅中占主要地位。在整个发酵过程中,方案二的真菌结构在后期发生了明显变化,Issatchenkia比方案一又说增加;在细菌组成上,方案二在发酵后期Lactobacillus的丰度比方案一高,说明小麦的加入引起了方案二菌群结构的变化。生产实际表明方案二大曲质量总体评价好于方案一大曲,说明一定量小麦的加入影响了大曲微生物群落结构和演替,微生物组成变化是造成大曲质量差异的主要原因。
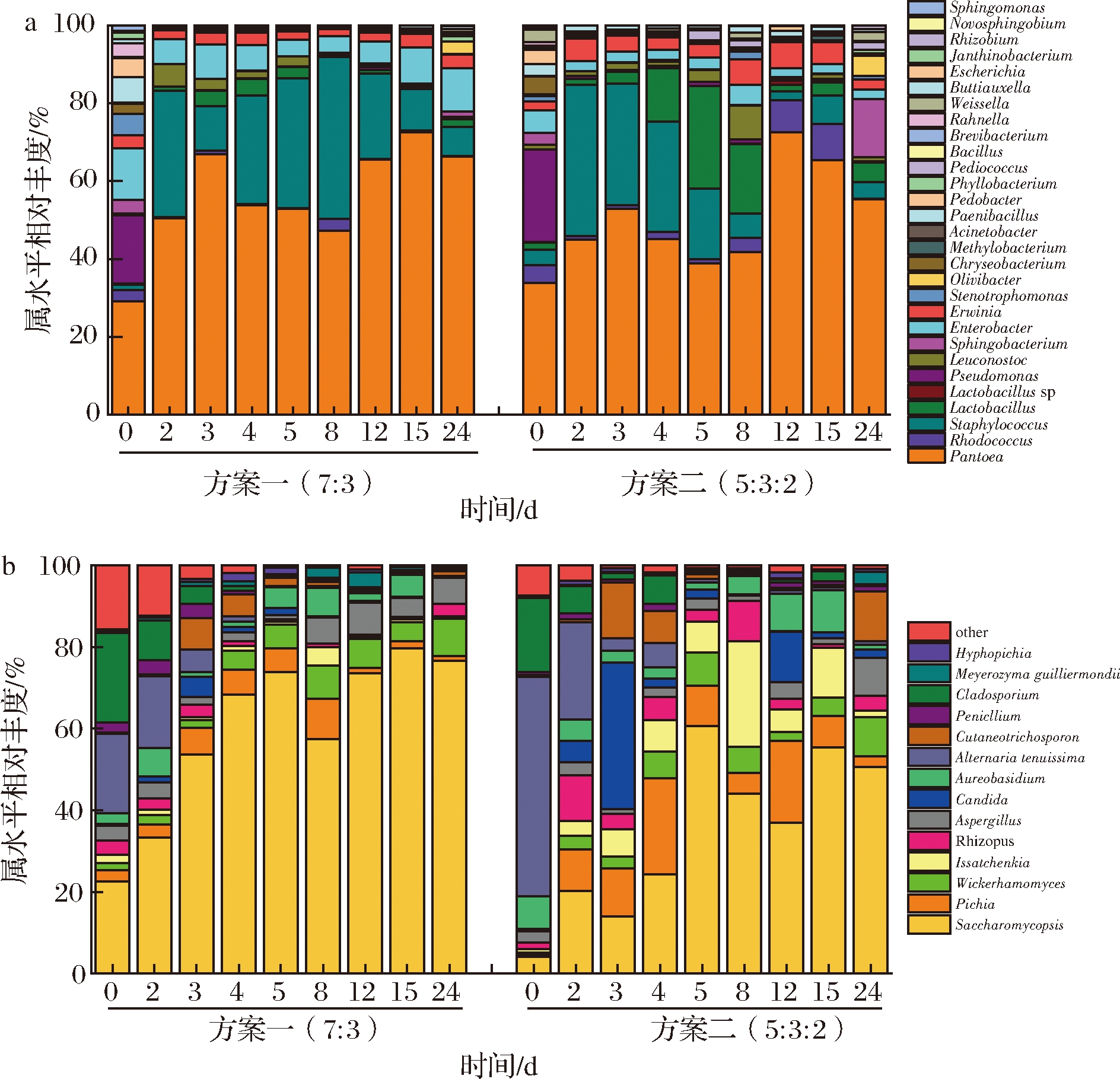
a-细菌;b-真菌
图3 大曲在属水平上的微生物群落结构
Fig.4 Distribution of microbial community at the genus level in Daqu during fermentation
2.2 制曲过程代谢物质变化
在制曲过程中,方案一分析出风味物质有104种,包括35种醇类、6 种酸类、14种酯类、4种吡嗪类化合物;方案二130种,包括38种醇类、11种酸类、25种酯类、7种吡嗪类化合物(图4)。整体上方案一风味物质总种类低于方案二,2种方案酯类物质的种类较丰富。通过化合物的相对含量的聚类分析可看出(图5),大曲中醇类、醛类、酚类在出房时含量较高,醇类占60%~80%,酸类在培养至第4天时含量最高,后期降低明显。酯类含量也较高,培养前期占10%左右,4~15 d时降低,第21 天时又增加明显,达到最高值。

a-方案一;b-方案二
图4 大曲培养风味物质种类
Fig.4 Types of flavor substances in Daqu culture
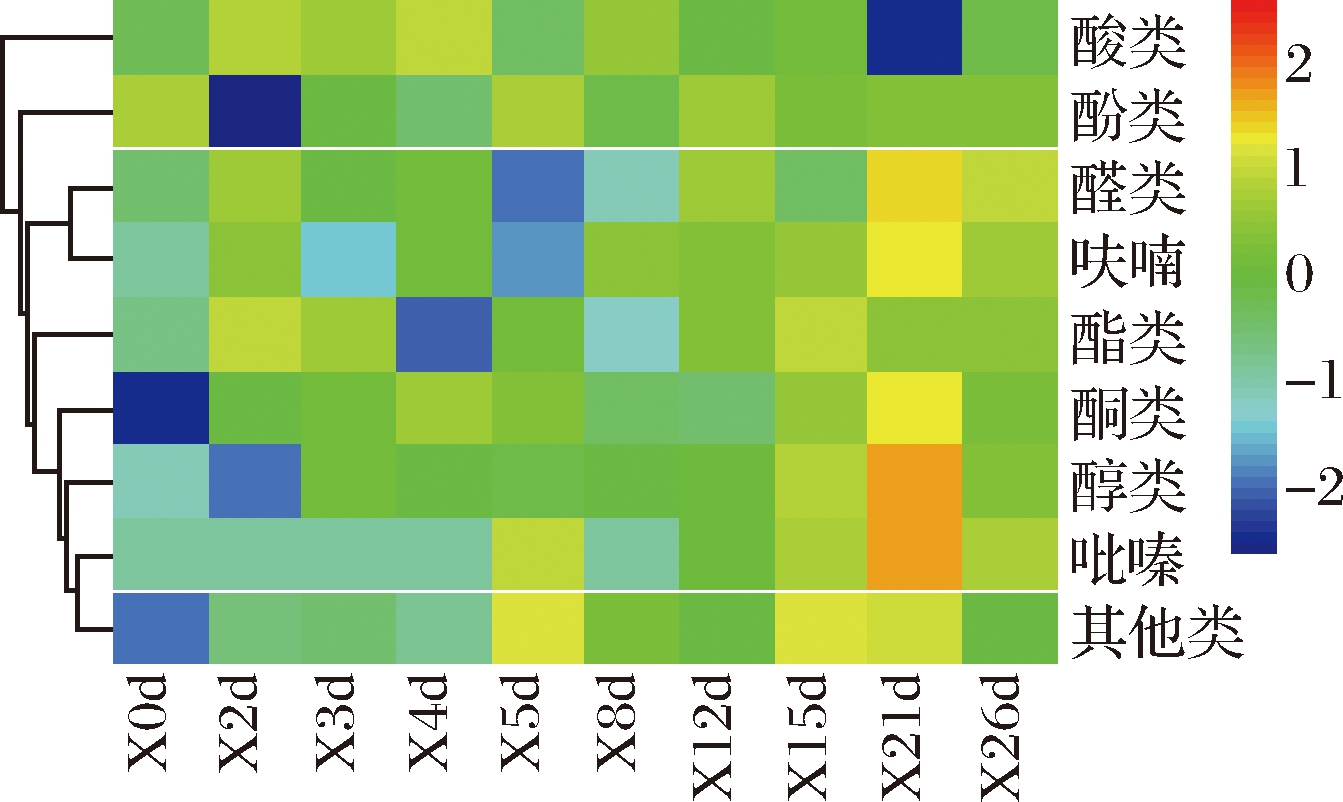
图5 大曲培养过程中风味化合物的变化热图
Fig.5 Heat map of flavor compounds during Daqu culture
2.3 制曲过程核心微生物判定
选取在制曲过程中丰度在前15的细菌属和前 15的真菌属,计算这30个属与61种代谢物之间的Pearson 相关系数(R),选取 R>0.5 作为有效的网络连接并绘图(图6)。可以发现,细菌与代谢产物的连接数要远远大于真菌,与细菌相关的主要化合物是乙醛(V18、V19、V20、V21、V22);与真菌相关的化合物主要有醇、酸、酯和吡嗪类,细菌属Acinetobacter、Olivibacter只和吡嗪类化合作的有显著的相关性;另外,真菌与酮类化合物的相关性较强,其中绝大多数细菌与1-辛烯-3-酮(V27)和伞花烃(V17)有显著的相关性;Lactobacillus、Pediococcus、Saccharomycopsis与醇、酸、酯都有显著的相关性,Wickerhamomyces、Aureobasidium与醇、酯、吡嗪有一定的相关性。以上结果表明,在大曲发酵过程中与重要的代谢产物相关的细菌有Lactobacillus、Leuconostoc、Pediococcus,真菌有Saccharomycopsis、Pediococcus、Wickerhamomyces和Aureobasidium,其中Acinetobacter、Olivibacter和Meyerozyma对吡嗪的贡献较大。
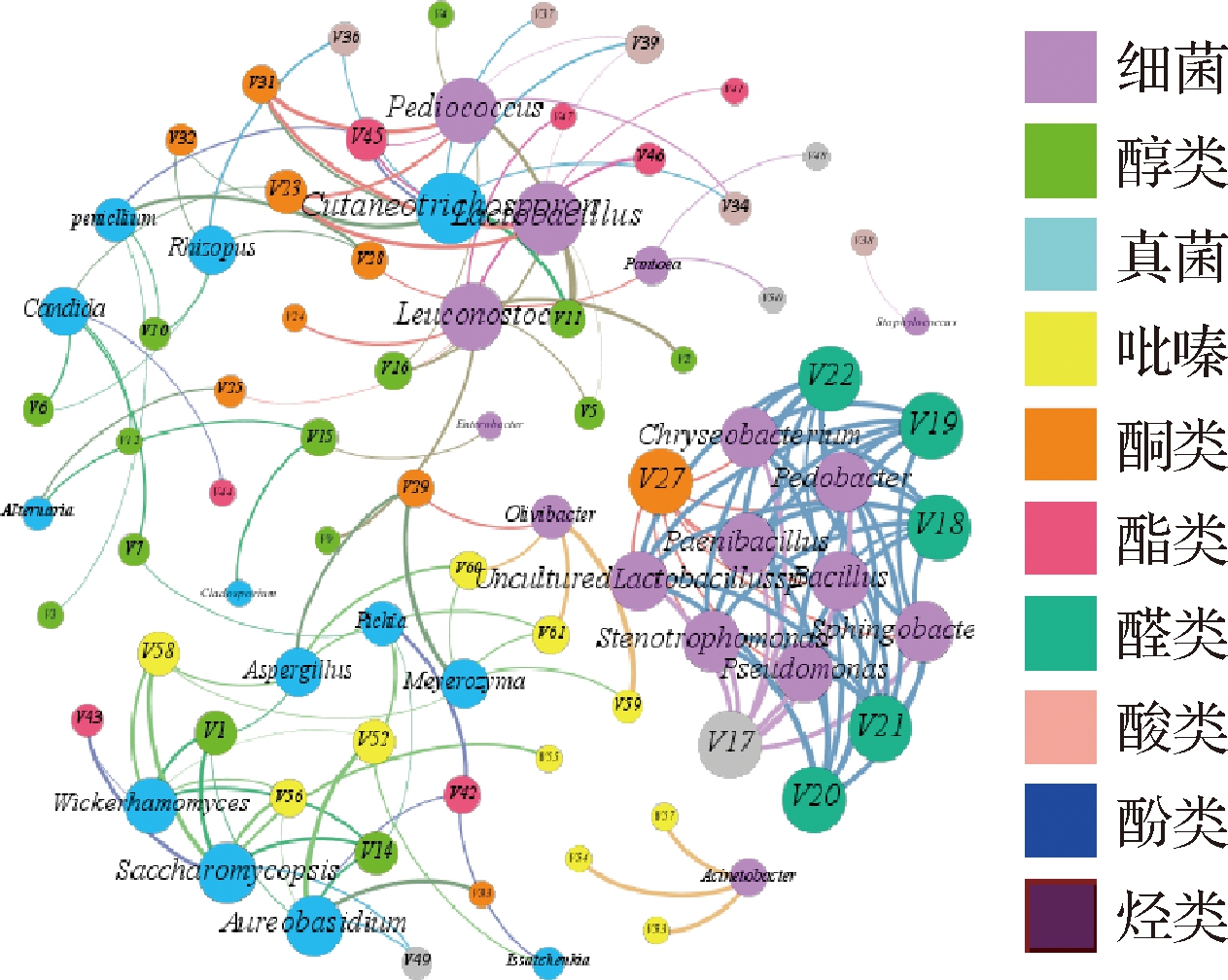
图6 微生物与代谢产物网络图
Fig.6 Network of microorganisms and metabolites
2.4 环境因子对制曲微生物的影响
环境因素会影响微生物的组成和分布,通过冗余分析解析了微生物与青稞大曲制作过程关键环境因子之间的关系(图7),在制曲过程中与糖化和液化相关的微生物有Wickerhamomyces、Rhizopus、Aspergillus、Hypocreales、Ralstonia、Bacteria、Burkholderia、Bacillus;与产酸有关的微生物主要有Lactobacillales、Leuconostoc、Pediococcus、Lactobacillaceae、Lactobacillus、Enterobacteriaceae、Saccharomycetales;与氨态氮相关的微生物主要有Penicillium、Aspergillaceae、Microbotryomycetes、Fusarium、Acinetobacter、Bacillus。蒙特卡洛置换检验的结果表明(表1),酿造过程中温度、酸度和乙醇是影响不同季节中微生物群落组成差异的重要原因。
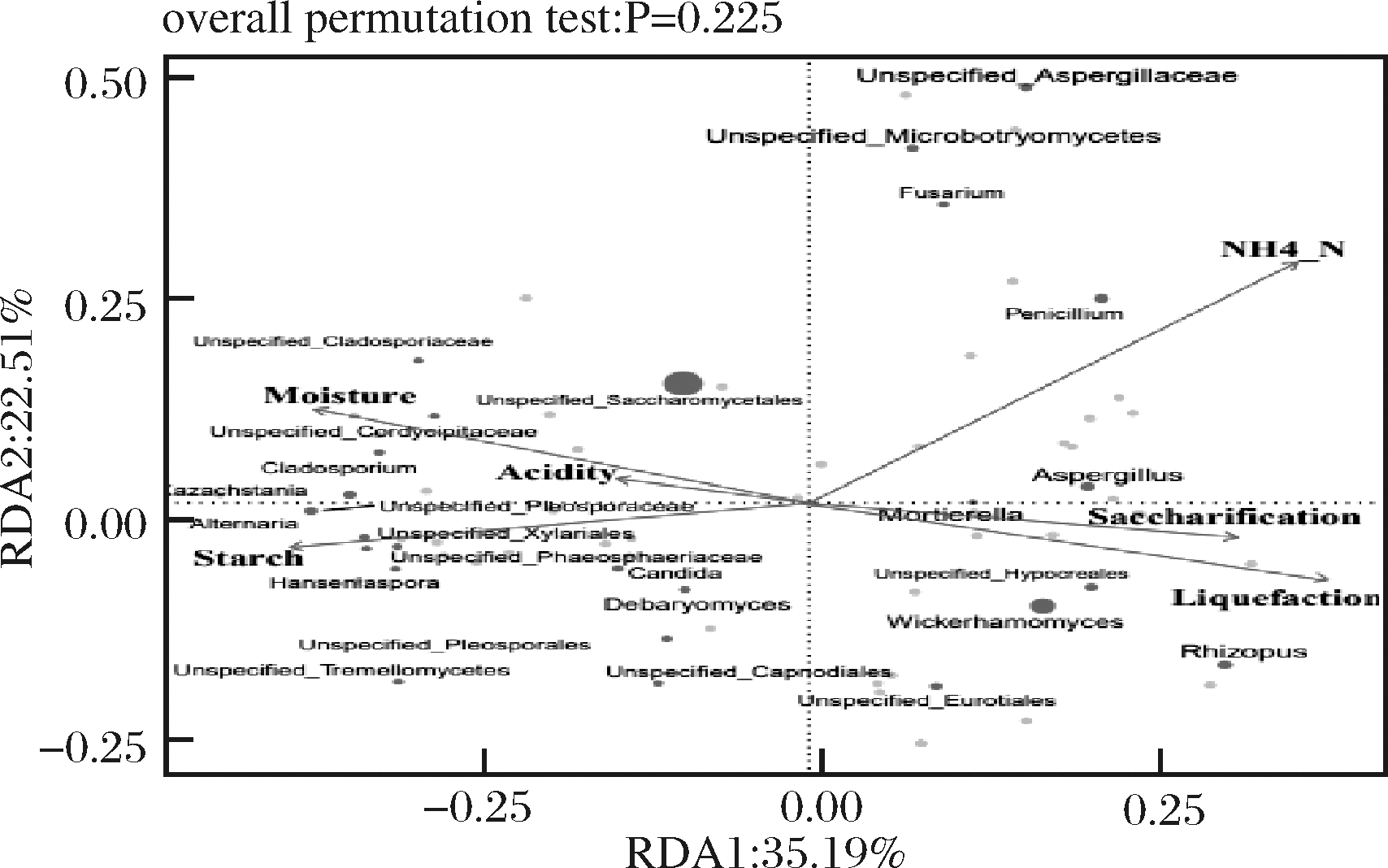
图7 环境因子与大曲微生物之间的相关性
Fig.7 Correlation between environmental factors and Daqu microorganisms
理化因子对制曲过程群落分布的解释率为37.86%,游离氨态氮对微生物群的分布具有极显著相关性(r2>0.5,P<0.001),说明环境中的氮对微生物群落结构演替有重要的推动作用。环境因子变化与大曲微生物群落衍变有显著地相关性,随着原料中小麦含量增加,氨态氮含量变化会驱动大曲中微生物群落衍变。
表1 微生物群与环境因子之间的蒙特卡洛置换检验结果
Table 1 Monte-Carlo permutation test of the core microbiota with environmental factors.
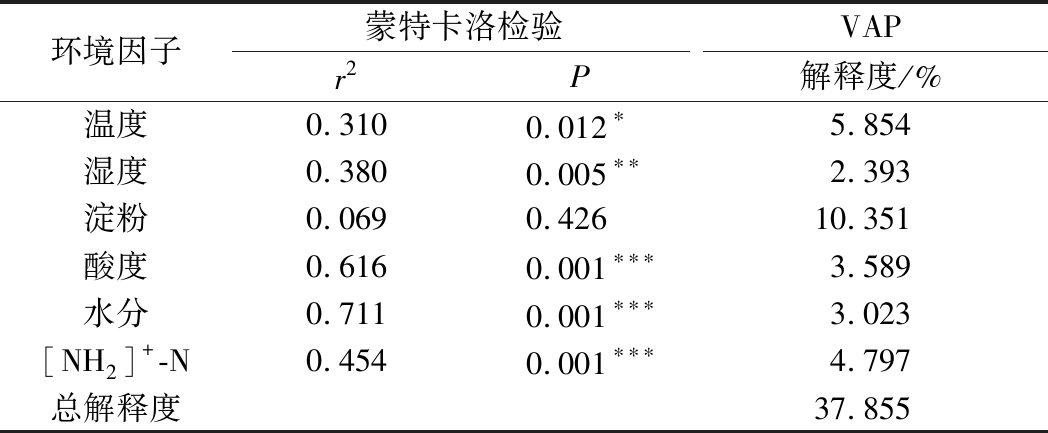
环境因子蒙特卡洛检验VAPr2P解释度/%温度0.3100.012∗5.854湿度0.3800.005∗∗2.393淀粉0.0690.42610.351酸度0.6160.001∗∗∗3.589水分0.7110.001∗∗∗3.023[NH2]+-N0.4540.001∗∗∗4.797总解释度37.855
3 结论
2种不同配料大曲在制曲过程中真菌微生物菌群结构存在差异性,方案二大曲中Lactobacillus和Issatchenkia的含量要高于方案一。初步确定青稞大曲的优势微生物属细菌为Pantoea、Staphylococcus,真菌包括Pichia、Saccharomycopsis。通过微生物与代谢产物网络可视化分析,初步判定在大曲发酵过程中与重要的代谢产物相关的微生物有Lactobacillus、Leuconostoc、Pediococcus、Saccharomycopsis、Pediococcus、Wickerhamomyces、Aureobasidium。制曲过程中与糖化和液化相关的微生物有Wickerhamomyces、Rhizopus、Aspergillus、Hypocreales、Ralstonia、Bacteria、Burkholderia、Bacillus;与产酸有关的微生物主要有Lactobacillales、Leuconostoc、Pediococcus、Lactobacillaceae、Lactobacillus、Enterobacteriaceae和Saccharomycetales;与氨态氮相关的微生物主要有Penicillium、Aspergillaceae、Microbotryomycetes、Fusarium和Acinetobacter、Bacillus。这些菌很可能是青稞大曲制作过程中的核心微生物。在大曲制备过程中游离氨态氮对微生物群的分布具有极显著相关性,随着原料中小麦含量增加,氨态氮含量变化会驱动大曲中微生物群落衍变。
本研究借助高通量测序方法解析了青稞大曲制作过程中微生物群落结构和演替规律,初步判定了制曲过程中的核心微生物属。同时分析了青稞大曲配料中适量加入小麦对大曲微生物菌群结构和大曲质量的影响。但本研究只通过高通量测序解析了微生物群落结构,初步判定了制曲过程中的优势微生物属,后续还需要结合酿酒过程中酒醅微生物变化,明晰大曲微生物对酒醅酿造微生物的贡献,为今后功能菌的筛选及强化大曲提供科学依据。
[1] 申孟林,张超,王玉霞.白酒大曲微生物研究进展[J].中国酿造,2016,35(5):1-5.
SHEN M L,ZHANG C,WANG Y X.Research progress on microorganism in Daqu liquor[J].China Brewing,2016,35(5):1-5.
[2] 吴生文,张志刚,李旭晖.大曲微生物在大曲酒生产中的研究开发现状及发展前景[J].中国酿造,2011,30(5):8-12.
WU S W,ZHANG Z G,LI X H.Current research and development prospects of Daqu microbes in the production of Daqu liquor[J].China Brewing,2011,30(5):8-12.
[3] 王鹏,吴群,徐岩.中国白酒发酵过程中的核心微生物群及其与环境因子的关系[J].微生物学报,2018,58(1):142-153.
WANG P,WU Q,XU Y.Core microbiota in Chinese liquor fermentation and associations with environmental factors[J].Acta Microbiologica Sinica,2018,58(1):142-153.
[4] WANG X,DU H,XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor[J].International Journal of Food Microbiology,2017(244):27-35.
[5] 黄和强,张芬军,李善文,等.夏冬两季青稞酒发酵过程中酒醅微生物菌群多样性分析[J].酿酒,2021,48(2):71-76.
HUANG H Q,ZHANG F J,LI S W,et al.Analysis of the microbial diversity during highland barley Baijiu brewing[J].Liquor Making,2021,48(2):71-76.
[6] 王鹏.地衣芽孢杆菌强化对浓香型白酒酿造微生物群落结构和代谢的影响[D].无锡:江南大学,2017.
WANG P.Effect of Bacillus licheniformis inoculation on the microbial community and metabolism during strong flavored liquor brewing process[D].Wuxi:Jiangnan University,2017.
[7] KONG Y,WU Q,ZHANG Y,et al.In situ analysis of metabolic characteristics reveals the key yeast in the spontaneous and solid-state fermentation process of Chinese light-style liquor[J].Applied and Environmental Microbiology,2014,80(12):3 667-3 676.
[8] 李小龙,吴群,徐岩.乙醇与酸度协同作用推动芝麻香型白酒固态发酵过程的微生物群落演替[J].微生物学通报,2019,46(1):1-10.
LI X L,WU Q,WU Y.Synergism between ethanol and acidity drives microbial community succession during roasted sesame-flavor liquor solid-state fermentations[J].Acta Microbiologica Sinica,2019,46(1):1-10.
[9] LI X,MA E,YAN L,et al.Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J].International Journal of Food Microbiology,2011,146(1):31-37.
[10] 王宇乔,黄和强,徐岩,等.氨基酸对青稞酒酿造微生物群落演替及风味代谢的驱动[J].微生物学通报,2021,48(2):414-425.
WANG Y Q,HUANG H Q,XU Y,et al.Effects of amino acids on microbial community succession and flavor metabolism in highland barley Baijiu fermentation[J].Microbiology,2021,48(2):414-425.
[11] LI S,LI P,LIU X,et al.Bacterial dynamics and metabolite changes in solid-state acetic acid fermentation of Shanxi aged vinegar[J].Applied Microbiology and Biotechnology,2016,100(10):4 395-4 411.
[12] OKSANEN J,KINDT R,LEGENDRE P,et al.The vegan package[J].Community Ecology Package,2007,10:631-637.
[13] PONOMAROVA O,GABRIELLI N,SÉVIN D C,et al.Yeast creates a niche for symbiotic lactic acid bacteria through nitrogen overflow[J].Cell Systems,2017,5(4):345-357.e6.
[14] SHANNON P,MARKIEL A,OZIER O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J].Genome Research,2003,13(11):2 498-2 504.
[15] LEMOS L N,FULTHORPE R R,TRIPLETT E W,et al.Rethinking microbial diversity analysis in the high throughput sequencing era[J].Journal of Microbiological Methods,2011,86(1):42-51.