谷氨酰胺转胺酶(transglutaminase,TG)能够催化蛋白质肽链中谷氨酰胺残基的γ-羧酰胺基与各种酰基受体发生酰基转移反应,是一种有效的蛋白质交联剂[1-3]。微生物谷氨酰胺转胺酶(microbial transglutaminase,MTG)具有低蛋白分子质量、非Ca2+依赖性和较广的底物范围等优点,在食品工业[4-6]、生物医药[7]、组织工程[7]、定点蛋白交联[8]和同源抗体偶联药物的构建等行业均具有广泛的应用价值[8-10]。近年来,通过分子进化获得更高活性和热稳定性的突变酶,并扩大其应用范围的研究一直受到国内外学者的密切关注。
我们在前期研究中,成功构建了一种5位点组合突变的毕赤酵母菌株pro/rDNA-mtg-mut2,其表达的重组酶MTG-MUT2为5个氨基酸组合突变体:S2P-S23V-Y24N-R215A-H289Y。MTG-MUT2比酶活力为(39.3±1.6) U/mg,是野生酶的2.4倍[野生酶(16.7±0.7) U/mg];最佳酶反应温度是55 ℃;50和60 ℃的半衰期分别为446.9和 27.6 min,分别是野生酶的11.7和10.2倍(野生酶在50和60 ℃的半衰期分别为38.5和2.7 min)。本研究首先通过高密度发酵获得粗酶液,分离纯化得到纯酶;并对该纯酶的3个组分进行分离,分析糖基化对酶活性的影响及其影响机制。然后,研究该纯酶催化氨基酸交联反应,旨在拓展重组酶MTG-MUT2的应用。
1 材料与方法
1.1 材料
1.1.1 菌株
毕赤酵母重组菌株pro/rDNA-mtg-mut2为本实验室构建并保藏。
1.1.2 仪器
AKTA Pure蛋白质层析纯化系统、Superdex 75 prep grade,GE公司;BWS465-785S拉曼光谱仪,美国必答泰克公司。实验用水为超纯水。
1.1.3 试剂与培养基
N-carboxybenzoyl-L-glutaminyl-glycine(N-CBZ-Gln-Gly)、L-谷氨酸-γ-单羟胺酸、牛血清蛋白,Sigma-Aldrich公司;还原型谷胱甘肽,华美生物工程有限公司;anti-MTG,Eurogentec公司;L-谷氨酰胺(纯度>99%),国药集团试剂有限公司;L-赖氨酸(纯度>99%),麦克林试剂公司。其他试剂均为国产分析纯。
毕赤酵母培养基:YPD、BMGY、BMMY,参照Invitrogen公司的操作手册配制,毕赤酵母培养温度28 ℃,诱导温度25 ℃[9-10]。
1.2 实验方法
1.2.1 pro/rDNA-mtg-mut2 的高密度发酵及纯化
高密度发酵在14 L发酵罐中进行。采用起始发酵体积为3 L的发酵体系,初始培养基组成:2.4 L BMGY(4%甘油)+300 mL YNB+300 mL PPB,发酵参数设定为:pH 6.0,生长温度28 ℃,搅拌转速800 r/min,通气量10 L/min。当罐内基础甘油用完后,溶氧会在短时间内上升,此时开始补加甘油,50%甘油(含12 ml/L PTM1)恒速补料25 mL/(L·h)。当菌体湿重达到(195±2)g/L时停止补甘油,开始补加100%甲醇诱导(甲醇含12 mL/L PTM1)。甲醇诱导采用3步补料(根据甲醇电极判断补料时间点),诱导温度调整为25 ℃。整个发酵过程中控制溶氧水平为25%~30%[9-11],诱导40 h。每8 h取样测定菌体湿重和酶活力,采用非还原SDS-PAGE检测,胶浓度10%,Western Blot鉴定目标蛋白,鉴定用抗体为anti-MTG。发酵产物的分离纯化采用His Trap HP组氨酸标记亲和层析法,操作步骤见参考文献[9-10]。纯化样品检测方法同上。
1.2.2 MTG-MUT2酶活力测定
异羟肟酸比色法测定MTG酶活力,按照参考文献[12-14]的方法进行。MTG酶活力定义为:在37 ℃下,MTG每分钟催化底物(N-CBZ-Gln-Gly)生成1 μmoL谷氨酸-单羟胺酸(氧肟酸)为1个酶活单位。蛋白浓度测定采用Bradford方法,标准蛋白为牛血清蛋白[13]。
1.2.3 糖基化对MTG-MUT2酶活性的影响
将1.2.2 所得纯化样品过分子筛(Superdex 75 prep grade),并在同一条件下测定这3个组分的酶活力,以探索糖基化对酶活性的影响,酶活力测定方法见1.2.2。流程为:平衡→上样→洗脱。PBS(pH 7.4)平衡分子筛,上样流速为2 mL/min,PBS洗脱,手动收集各个紫外峰组分,SDS-PAGE检测,胶浓度10%,每孔上样量2.5 μg,考马斯亮蓝R-250染色。
1.2.4 MTG-MUT2催化条件优化
为了进一步探讨MTG-MUT2对蛋白质中氨基酸的交联作用,以L-谷氨酰胺和L-赖氨酸作为底物,首先分析反应底物的配比对产物生成的影响。固定L-谷氨酰胺的浓度为300 mmol/L,L-赖氨酸与其的比例为A∶0.9、B∶1.0、C∶1.1、D∶1.2,在55 ℃、pH 6.5下反应2 h,比较谷赖二肽产量,确定2种底物最适合的添加比例。接着,研究L-谷氨酰胺的浓度分别为100、200、300、400、500、600 mmol/L时,L-赖氨酸和L-谷氨酰胺添加比例的最适比例,比较谷赖二肽产量。
1.2.5 MTG-MUT2催化产物的检测
采取柠檬酸钠还原法制备银纳米颗粒(Ag nanoparticles,AgNPs),Ag+被还原成直径为纳米级的AgNPs。具体制备依据文献[14-19]的方法进行制备。
将待测样品用PBS缓冲液(pH 7.2)稀释为100 μg/mL。取50 μL待测样本与50 μL修饰后的AgNPs混合后放入微量比色皿中进行表面增强拉曼散射光谱(surface enhanced Raman scattering,SERS)测试。激发波长为785 nm,积分时间5 ms,激光功率20 mW。数据采集及光谱处理均采用光谱仪自带软件。
2 实验结果
2.1 pro/rDNA-mtg-mut2在14 L发酵罐中的表达和纯化
典型的发酵曲线如图1-a所示,包括间歇培养、甘油进料和甲醇诱导阶段。在间歇培养阶段,酵母种子在含4%甘油(pH 6.0)的BMGY培养基中培养,28 ℃控制,持续17~18 h,当细胞湿重(wet cell weight,WCW)达到140 g/L,开始补甘油。在甘油补料阶段,补50%的甘油(含有12 mL/L PTM1溶液),将DO控制在30%,温度维持在28 ℃,至WCW达到约(195±2)g/L时,补料结束。当溶氧(dissolved oxygen,DO)出现急剧上升,开始甲醇诱导(100%甲醇,含12 mL/L PTM1),甲醇采取3步诱导:3.6、7.2和10.8 mL/(L·h),诱导过程DO保持在大约30%(图1-a)。甲醇诱导持续40 h,当酵母WCW达到约340~350 g/L时结束。在甲醇诱导的前32 h,MTG-MUT2的酶活力和表达逐渐增加(图1-b~图1-d);然而,诱导32 h后,MTG-MUT2酶活力和表达量增加不明显,发酵结束时,发酵上清液的最高酶活力为7.45 U/mL(图1-b)。
2.2 MTG-MUT2的纯化和SDS-PAGE分析
对pro/rDNA-mtg-mut2高密度发酵的70 h培养上清液进行1步镍柱亲和层析纯化,收集洗脱液215 mL。如表1所示,酶活力为12.3 U/mL,蛋白质量浓度274.3 μg/mL,蛋白纯化2.7倍,回收率为87.4%。SDS-PAGE结果如图2所示,重组MTG-MUT2有3条特异性蛋白带,分别为1条非糖基化条带和2条糖基化条带,大小为38 k~42 kDa,这与前期实验结果及文献报道的1~3条带的结果相一致[10-12]。
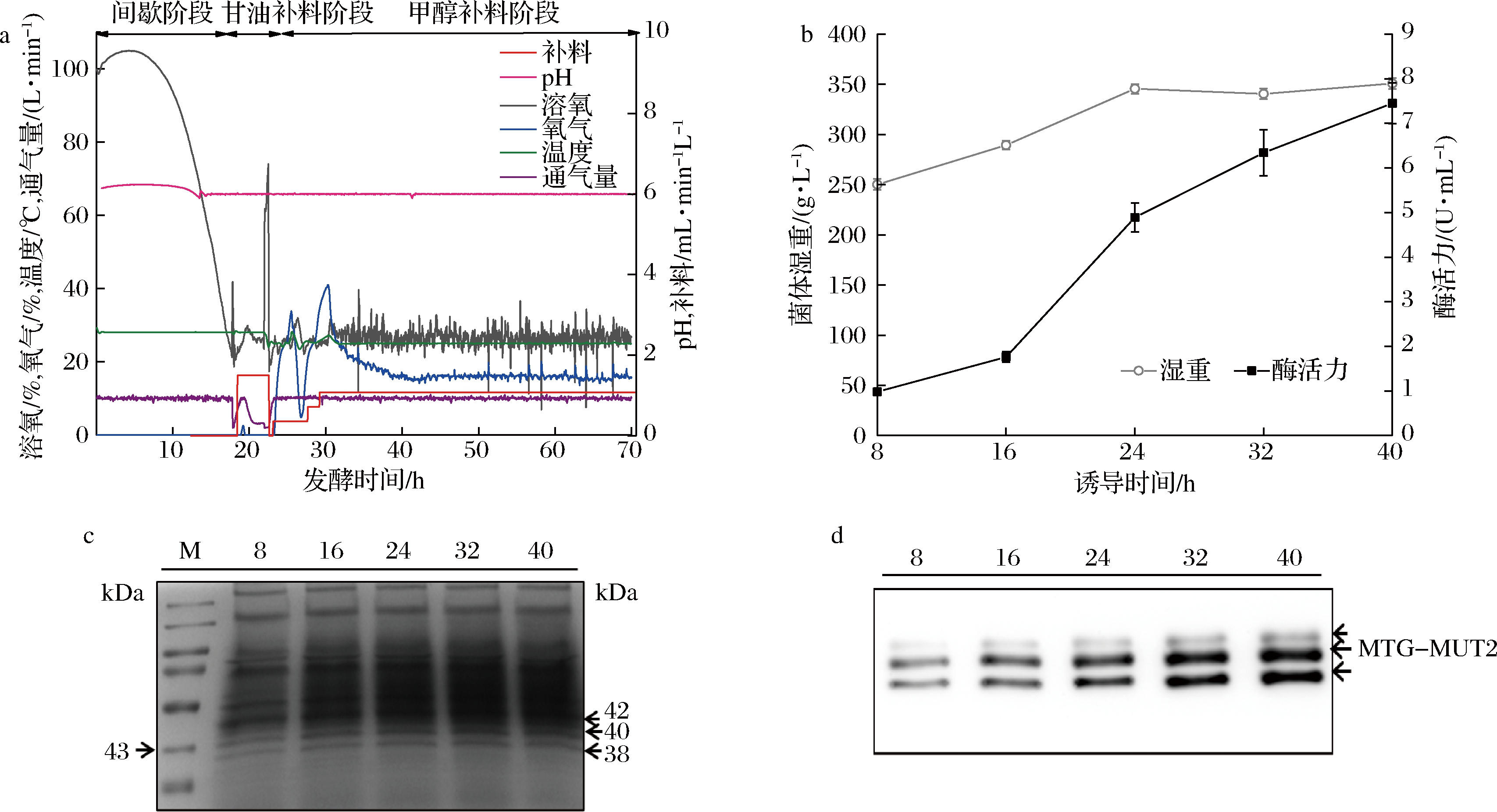
a-发酵曲线;b-不同诱导时间的生物量和酶活力;c-SDS-PAGE不同诱导时间发酵上清液;d-WB检测不同诱导时间发酵上清液
图1 pro/rDNA-mtg-mut2高密度发酵过程的检测
Fig.1 Parameters detected at different induction time of high-density pro/rDNA-mtg-mut2 fermentation
表1 重组MTG的纯化方案
Table 1 Purification scheme of recombinant MTG-MUT2

纯化步骤总蛋白/mg总酶活力/U比酶活力/(U·mg-1)纯化倍数 回收率%/粗酶液182.42 646.414.51100Ni2+柱纯化59.72 313.638.72.787.4
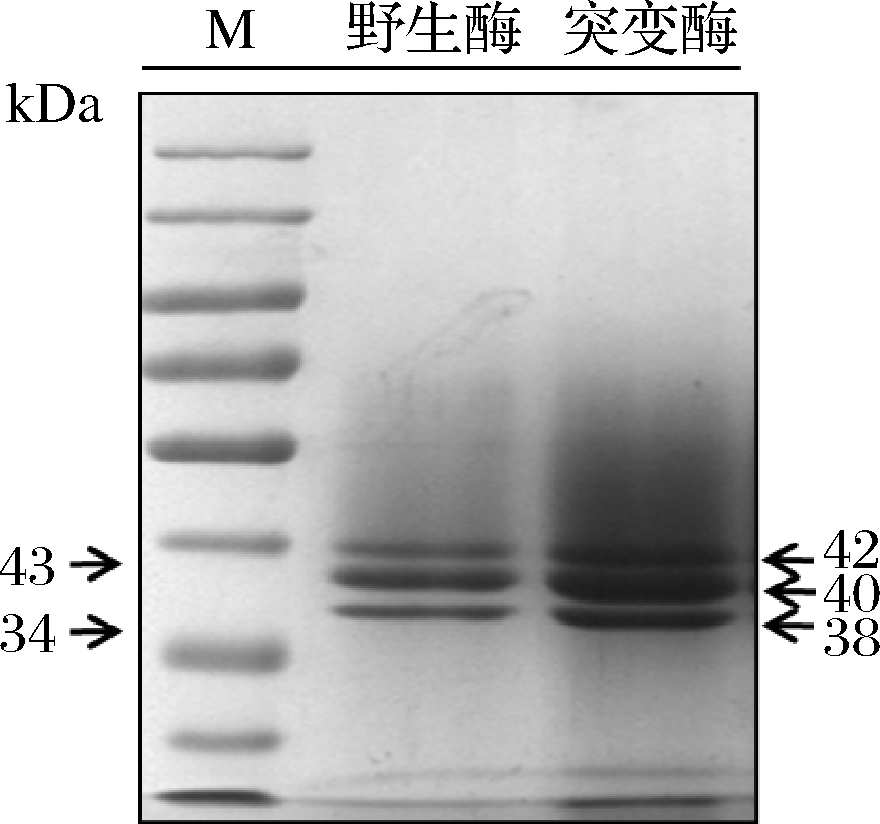
图2 SDS-PAGE检测纯化MTG-MUT2
Fig.2 Detection of purified MTG-MUT2
2.3 糖基化对MTG-MUT2酶活性的影响
MTG-MUT2的3个组分分离结果如图3所示。与未分离蛋白条带比较,3条蛋白带的主带已分离,根据分子质量从大到小依次为42、40、38 kDa,但由于2条带表观分子质量相差只有约2 kDa,所以条带没有完全分开,但不影响后续酶活力的分析。以未分离酶作为对照,在同一条件下检测这3个组分(42、40、38 kDa)的酶活力,结果分别为:10.6、14.7和8.7 U/mL,40 kDa糖蛋(14.7 U/mL)是非糖基化酶(8.7 U/mL)的1.7倍;2个位点均发生糖基化的酶活力次之(10.6 U/mL),是非糖基化酶的1.2倍。
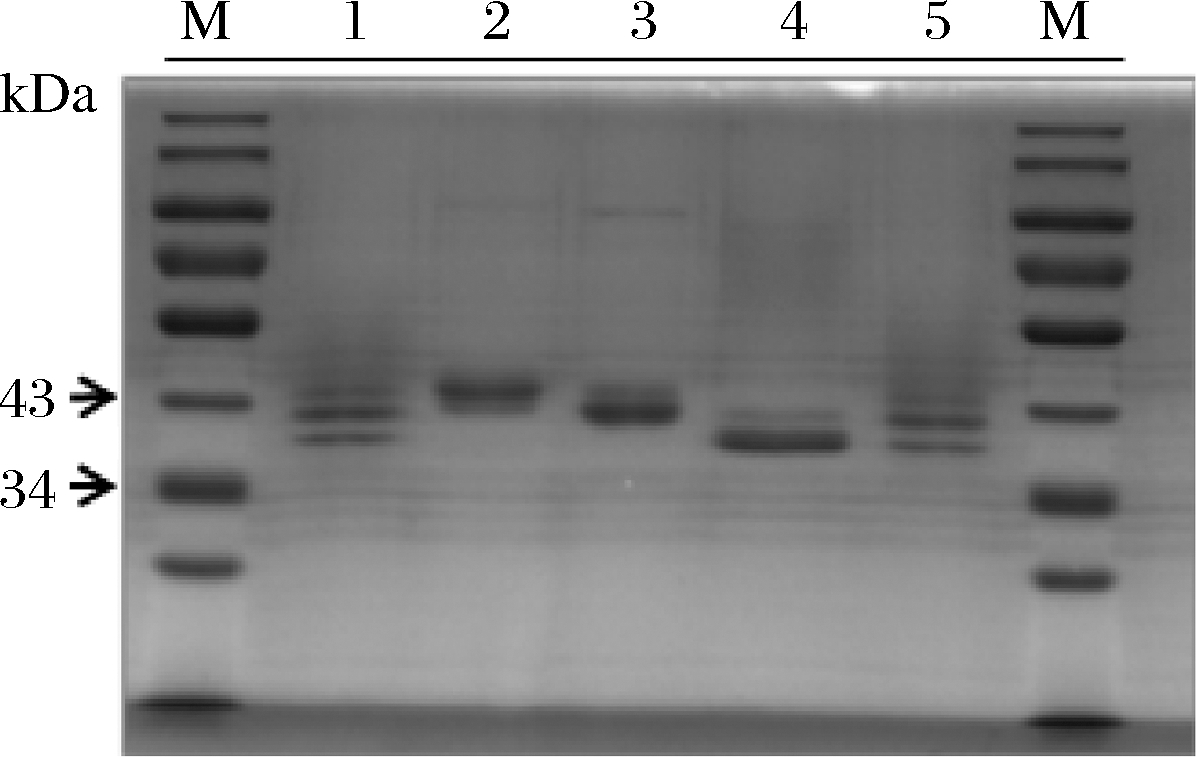
1,5-未过分子筛样品(不同批次);2~4-分离后的蛋白样品
图3 MTG-MUT2分离后组分的SDS-PAGE检测
Fig.3 Components seperated by MTG-MUT2 detected by SDS-PAGE
2.4 MUT2催化条件优化
固定谷氨酰胺的浓度为300 mmol/L,L-赖氨酸与其的比例为A∶0.9、B∶1.0、C∶1.1、D∶1.2,在55 ℃、pH 6.5下反应2 h,结果如图4所示(图中数据均为扣除基线和溶剂的峰)。拉曼光谱特征峰在298~2 000 cm-1,与Gln和Lys比较,Lys与Gln连接产物可能的峰在719、755、1 180、1 288、1 476、1 602 cm-1(图4)。其中719、755 cm-1主要是酰胺Ⅳ的CO面内弯曲所致,1 180 cm-1是NCαC骨架伸缩振动的特征峰,1 288 cm-1主要是酰胺Ⅲ中NH 面内弯曲引起的峰,因此1 288 cm-1可作为Gln的γ-羧酰胺基与Lys的酰基形成酰胺键(肽键)的重要指征,是2个氨基酸交联的重要依据,其峰强可反映谷赖二肽的生成量。
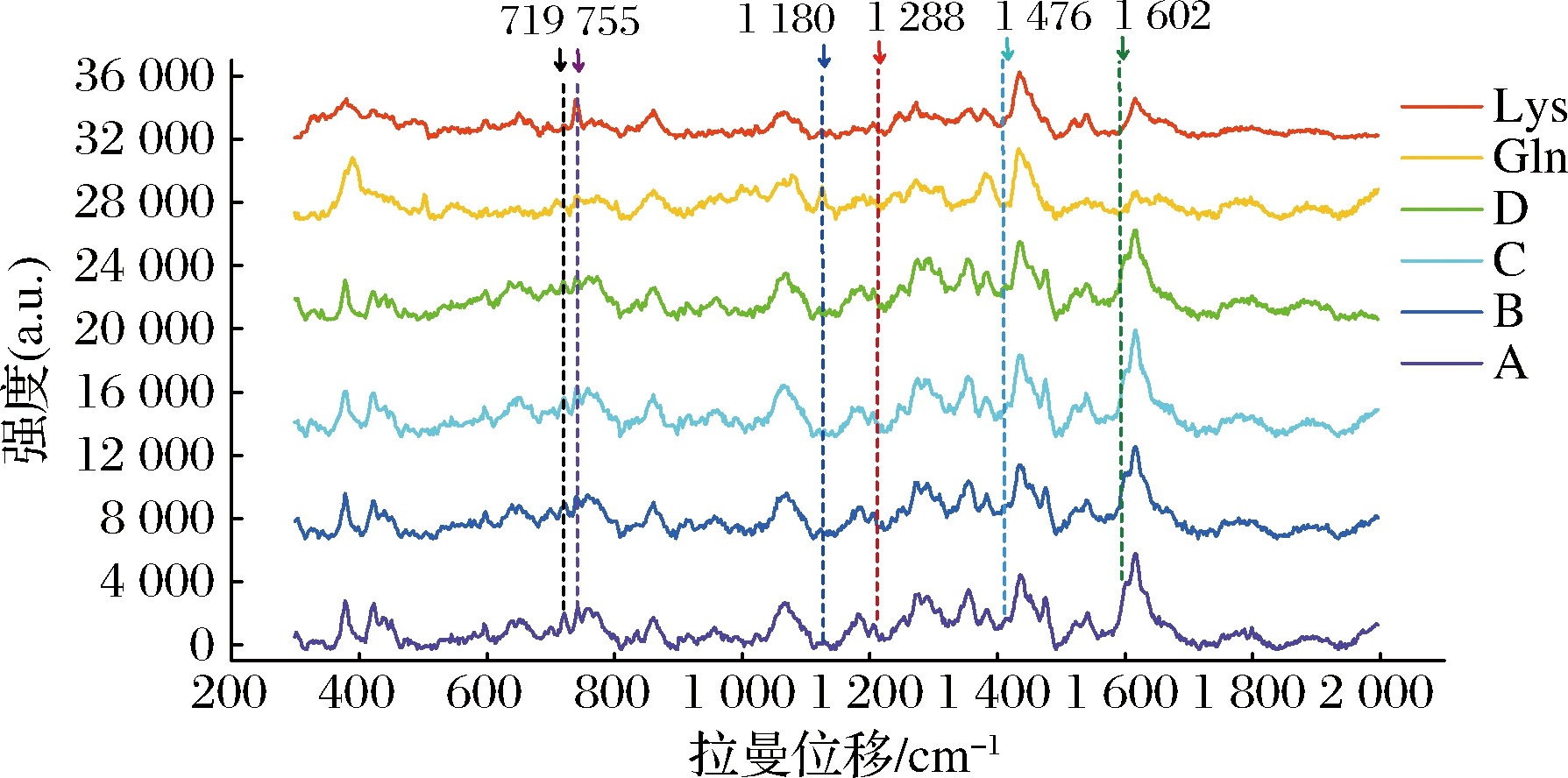
图4 Lys和Gln交联反应产物的拉曼光谱图
Fig.4 Raman spectra of Lys and Gln cross-linked reaction products
谷赖二肽在1 288 cm-1的拉曼光谱峰强度随着底物赖氨酸浓度的增加而增大,但当比例大于1.1时,峰强增加有限(峰强度3 550.7 a.u.),所以当赖氨酸浓度为330 mmol/L,谷氨酰胺浓度为300 mmol/L,即比例1.1为底物的最佳浓度比(图5-a)。L-赖氨酸和L-谷氨酰胺的添加比例为1.1,谷赖二肽的产量如图5-b所示。随着L-谷氨酰胺的浓度从100 mmol/L 增加到600 mmol/L,谷赖二肽在1 288 cm-1的拉曼光谱峰强度随着底物谷氨酰胺浓度的增加而增大,分别为3 191.8、3 250.1、3 550.7、3 764.7、3 994.6和4 274.7 a.u.。
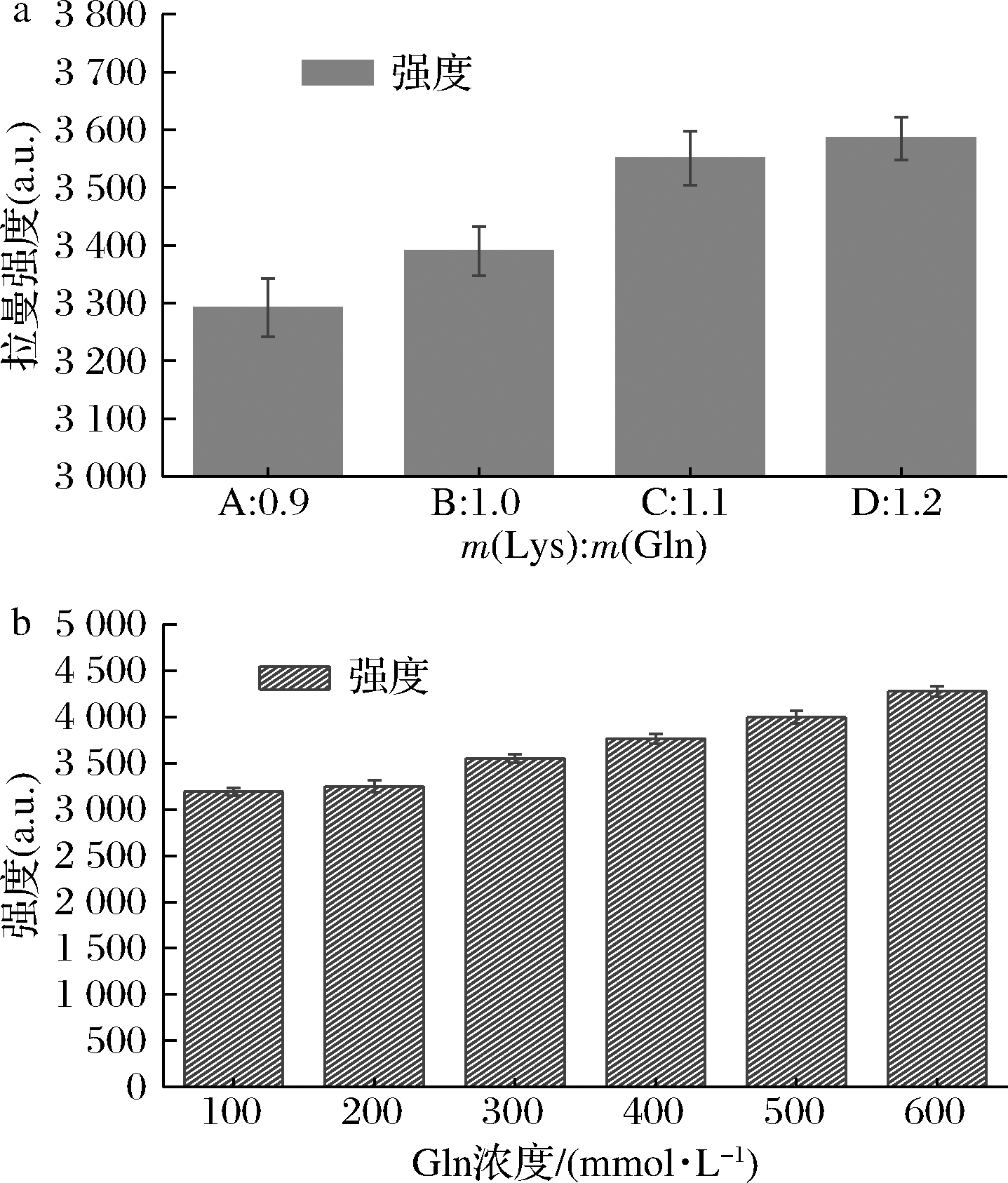
a-Lys/Gln对催化反应的影响;b-Gln浓度对催化反应的影响
图5 底物的配比和底物浓度对MTG-MUT2催化反应的影响
Fig.5 Effect of substrate ratio and substrate concentration on MTG-MUT2 catalytic reaction
3 结论与讨论
本研究对前期构建的毕赤酵母pro/rDNA-mtg-mut2,在14 L发酵罐高密度发酵表达获得MTG-MUT2,对其纯化酶的催化交联反应进行了研究,并探索了糖基化对酶活力的影响。
研究表明,在pH 为6.0时,诱导WCW为(195±2) g/L,采取甲醇3步补料工艺3.6 mL/(L·h) 5~6 h、7.2 mL/(L·h) 2~3 h和10.8 mL/(L·h)持续到下罐,产酶速率最快,为8.89 U/g WCW;酶活性最高,达到3.12 U/mL。一般诱导时间越长,目的蛋白的表达量越高,但诱导时间过长,可能会影响共表达2种蛋白(前导肽PRO和MTG-MUT2)的平衡[3,10-11],反而会抑制MTG-MUT2的活性表达,故本研究中诱导时间不超过45 h。在优化的最适合培养条件下,对该菌株在14 L发酵罐中高密度发酵表达,表达产物通过1步镍柱亲和层析纯化,蛋白纯化2.7倍,回收率为87.4%。
表达的重组蛋白在内质网中正确折叠是其正常分泌的重要条件,这是影响毕赤酵母高效表达蛋白的重要因素,分子伴侣和N-糖基化对有些蛋白的正确折叠是必不可少的[20-23]。本研究中Western Blot鉴定结果显示MTG-MUT2在34~43 kDa处有3条特异性蛋白带,表明有2个位点均参与了MTG-MUT2的糖基化修饰。分析其晶体结构发现MTG-MUT2有2个N-糖基化位点:N282和N297,N282环绕在活性位点C64附近,推测通过N-糖基化修饰增加肽链的亲水性,从而使肽链能够避免修饰位点与附近其他蛋白发生疏水作用,从而提高蛋白折叠的可能性,并增大了与底物的接触面积,赋予其较高的酶活。另一个糖基化位点N297,与N282相距较近,因位阻作用影响其糖基化以及与底物结合,从而影响酶活,因而N297只是一个潜在的糖基化位点,这就解释了2个位点均发生糖基化的蛋白酶活(10.6 U/mL)要低于1个位点糖基化(N282)的酶活(14.7 U/mL),但是均高于非糖基化蛋白酶活(8.7 U/mL)。以上研究结果表明,糖基化有助于MTG-MUT2的正确折叠,增大了与底物的接触面积,从而赋予其较高的酶活。
本研究还对该纯化酶催化L-谷氨酰胺的酰基转移给L-赖氨酸的交联反应条件进行了研究,并采用SERS快速、灵敏分析交联反应产物[24-26]。实验结果显示底物赖氨酸的拉曼特征峰位于651、1 203、1 354、1 518 cm-1,谷氨酰胺的拉曼特征峰位于384、501 cm-1,赖氨酸与谷氨酰胺连接产物可能的峰位于719、755、1 180、1 288、1 476、1 602 cm-1(图4)。其中1 288 cm-1主要是酰胺Ⅲ中NH 面内弯曲引起的峰,因此1 288 cm-1可作为Gln的γ-羧酰胺基与Lys的酰基形成酰胺键(肽键)的重要指征,是2个氨基酸交联的重要依据,其峰强可反映谷赖二肽的生成量。确定了赖氨酸和谷氨酰胺的最适添加比例为1.1;在最适反应条件下(反应温度55 ℃,最适pH 6.5),当谷氨酰胺浓度为600 mmol/L、赖氨酸浓度为660 mmol/L时,反应2 h,谷赖二肽在1 288 cm-1的特征峰强度为4 274.7,高于野生酶在同等反应条件下峰强的20%(野生酶:2 992.5),显示出该酶在较高反应温度下较好的氨基酸交联反应。双缩脲反应适用于分子中含两个或两个以上肽键(—CO—NH—)等基团的物质,不能用于二肽的检测。研究结果表明,采用银溶胶作为SERS基底,能够快速简便、高效灵敏检测谷氨酰胺转氨酶催化的反应产物。
综上所述,MTG-MUT2具有良好的热稳定性和催化效率高的优良特性,在工业生产中具有良好的应用潜力。MTG-MUT2催化蛋白质内或蛋白质间交联反应的性质,使其在组织工程上有潜在的应用,有待后续研究。
[1] SHLEIKIN A G,DANILOV N P.Evolutionary-biological peculiarities of transglutaminase:Structure,physiological functions,application[J].Journal of Evolutionary Biochemistry and Physiology,2011,47(1):1-14.
[2] LI H B,ZHANG L W,CUI Y H,et al.Expression of soluble recombinant transglutaminase from Zea mays in Pichia pastoris[J].World Journal of Microbiology and Biotechnology,2013,29(5):939-947.
[3] MARTINS I M,MATOS M,COSTA R,et al.Transglutaminases:Recent achievements and new sources[J].Applied Microbiology and Biotechnology,2014,98(16):6 957-6 964.
[4] MIWA N.Innovation in the food industry using microbial transglutaminase:Keys to success and future prospects[J].Analytical Biochemistry,2020,597(15):113 638.
[5] MORENO H M,TOVAR C A,DOM NGUEZ-TIM
NGUEZ-TIM N F,et al.Gelation of commercial pea protein isolate:Effect of microbial transglutaminase and thermal processing[J].Food Science and Technology,2020,40(4):800-809.
N F,et al.Gelation of commercial pea protein isolate:Effect of microbial transglutaminase and thermal processing[J].Food Science and Technology,2020,40(4):800-809.
[6] 李明奇,贺稚非,李洪军.微生物源谷氨酰胺转胺酶修饰蛋白质机理及其在食品方面的应用进展[J].食品与发酵工业,2018,44(12):274-280.
LI M Q,HE Z F,LI H J.Catalytic mechanisms of microbial transglutaminase in modification of protein and its applications in food processing[J].Food and Fermentation Industries,2018,44(12):274-280.
[7] BAI J,HUI J Y,LU Q L,et al.Effect of transglutaminase cross-linking on the allergenicity of tofu based on a BALB/c mouse model[J].Food & Function,2020,11(1):404-413.
[8] DENNLER P,CHIOTELLIS A,FISCHER E,et al.Transglutaminase-based chemo-enzymatic conjugation approach yields homogeneous antibody-drug conjugates[J].Bioconjugate Chemistry,2014,25(3):569-578.
[9] GHARIBZAHEDI S M T,CHRONAKIS I S.Crosslinking of milk proteins by microbial transglutaminase:Utilization in functional yogurt products[J].Food Chemistry,2018,245:620-632.
[10] SONG X P,SHAO C S,GUO Y G,et al.Improved the expression level of active transglutaminase by directional increasing copy of mtg gene in Pichia pastoris[J].BMC Biotechnology,2019,19(1):54.
[11] 宋小平,王雅洁,蔡晶晶,等.基因拷贝数对重组毕赤酵母产谷氨酰胺转胺酶的影响[J].生物工程学报,2020,36(8):1 679-1 688.
SONG X P,WANG Y J,CAI J J,et al.Impact of gene dosage on recombinant transglutaminase production of Pichia pastoris[J].Chinese Journal of Biotechnology,2020,36(8):1 679-1 688.
[12] 蒋秋琪,吕雪芹,崔世修,等.代谢工程改造毕赤酵母发酵生产谷胱甘肽[J].食品与发酵工业,2020,46(17):9-14.
JIANG Q Q,LYU X Q,CUI S X,et al.Metabolic engineered Pichia pastoris for synthesis of glutathione[J].Food and Fermentation Industries,2020,46(17):9-14.
[13] MU D D,LU J J,SHU C,et al.Improvement of the activity and thermostability of microbial transglutaminase by multiple-site mutagenesis[J].Bioscience,Biotechnology,and Biochemistry,2018,82(1):106-109.
[14] TIAN K Z,CAO C C,NIE X M,et al.Sensitive and label-free detection of protein secondary structure by amide III spectral signals using surface-enhanced Raman spectroscopy[J].Chinese Journal of Chemical Physics,2019,32(5):603-610.
[15] TAN J J,ZHANG J H.,LUO Y,et al.Misfolding of human islet amyloid polypeptide at lipid membrane populates through β-sheet conformers without involving α-helical intermediates[J].Journal of the American Chemical Society,2019,141(5):1 941-1 948.
[16] SONNENDECKER C,ZIMMERMANN W.Change of the product specificity of a cyclodextrin glucanotransferase by semi-rational mutagenesis to synthesize large-ring cyclodextrins[J].Catalysts,2019,9(3):242.
[17] GUMULYA Y,HUANG W L,D’CUNHA S A,et al.Engineering thermostable CYP2D enzymes for biocatalysis using combinatorial libraries of ancestors for directed evolution(CLADE)[J].ChemCatChem,2019,11(2):841-850.
[18] POTLURI P R,RAJENDRAN V K,SUNNA A,et al.Rapid and specific duplex detection of methicillin-resistant Staphylococcus aureus genes by surface-enhanced Raman spectroscopy[J].Analyst,2020,145(7):2 789-2 794.
[19] ZHANG Y M,LV Y,CHEN L,et al.Inhibition of epigallocatechin-3-gallate/protein interaction by methyl-β-cyclodextrin in myofibrillar protein emulsion gels under oxidative stress[J].Journal of Agricultural and Food Chemistry,2018,66(30):8 094-8 103.
[20] 张小倩,张腾腾,闫攀,等.蛋白N-糖基化分析方法研究进展[J].生物技术进展,2019,9(3):246-252.
ZHANG X Q,ZHANG T T,YAN P,et al.Advances on the analysis methods of N-glycosylation[J].Current Biotechnology,2019,9(3):246-252.
[21] WILD R,KOWAL J,EYRING J,et al.Structure of the yeast oligosaccharyl transferase complex gives insight into eukaryotic N-glycosylation[J].Science,2018,359:545-550.
[22] RILEY N M,HEBERT A S,WESTPHALL M S,et al.Capturing site-specific heterogeneity with large-scale N-glycoproteome analysis[J].Nature Communications,2019,10:1 311.
[23] CHANG M M,GAIDUKOV L,JUNG G,et al.Small-molecule control of antibody N-glycosylation in engineered mammalian cells[J].Nature Chemical Biology,2019,15(7):730-736.
[24] FAZIO B,D’ANDREA C,FOTI A,et al.SERS detection of biomolecules at physiological pH via aggregation of gold nanorods mediated by optical forces and plasmonic heating[J].Scientific Reports,2016,6:26 952.
[25] 左奇,陈瑶,石彩霞,等.表面增强拉曼散射光谱定量分析技术的研究进展[J].分析化学,2015,43(11):1 656-1 663.
ZUO Q,CHEN Y,SHI C X,et al.Recent developments on quantitative surface-enhanced Raman spectroscopic techniques[J].Chinese Journal of Analytical Chemistry,2015,43(11):1 656-1 663.
[26] ZHAO Z H,HUANG Y Q,FAN Y X,et al.Rapid detection of flusilazole in pears with Au@Ag nanoparticles for surface-enhanced Raman scattering[J].Nanomaterials,2018,8(2):94.