谷氨酸棒杆菌(Corynebacterium glutamicum)是食品安全级菌株,被广泛用于生产许多有价值的氨基酸和副产物[1]。由于其具有完整的分泌途径,无内毒素且可高密度发酵等特性,近年来已发展成为能够生成多种外源蛋白的卓越表达系统。然而,由于原始菌株的自然局限性,仍然存在一些缺点,如产量低,可用遗传操作工具匮乏等[2]。随着基因工程技术以及合成生物学技术的广泛应用,人们有了更多改造宿主的方法,从而提高目标产物的产量,此系统已成为实验室研究及商业生产重组蛋白的热点[3-4]。
目前,随着基因组学和蛋白质组学的深入研究,为挖掘相关目标信息提供了分析手段。目前大多数外源蛋白都依赖微生物表达系统进行生产[5]。然而,由于底盘细胞代谢机制的复杂性,使理性改造存在一定的困难。因此从表型差异入手,结合全基因组重测序,可以进一步分析差异表型相关的候选基因[6],为高效宿主的开发提供方向。目前,分析基因组区域和区域内变化的基因已成为一种挖掘表型差异相关候选基因的有效方法[7]。例如,ZHANG等[8]在全基因组水平筛选并鉴定了15种梨火疫菌的Sec依赖分泌蛋白酶。外源蛋白表达是一个复杂的耗能过程,涉及转录、翻译、运输、分泌等多个过程,受多基因调控,路径复杂。因此基于表型差异,采用组学的方法挖掘不同菌株之间的差异基因,有望为底盘细胞的优化改造找到潜在的靶点,最终达到提升宿主蛋白表达能力的目的。
该研究基于全基因组重测序策略,对谷氨酸棒杆菌外源蛋白高产突变菌株进行单核苷酸多态性(single-nucleotide polymorphisms, SNP)挖掘,并结合生物信息学分析SNP基因的结构极其功能。进一步通过构建SNP基因过表达和敲除菌株,评估这些基因表达变化对菌株生长以及外源蛋白增强型绿色荧光蛋白(enhanced green fluorescence protein, EGFP)和人重组特立帕肽(recombinant human teriparatide, rtPTH)表达的影响。最后利用SWISS-MODEL软件对基因GL002370编码的蛋白进行建模,并分析了蛋白的功能。
1 材料与方法
1.1 菌株与培养条件
在该研究所用的菌株见表1。大肠杆菌JM109,谷氨酸棒杆菌原始菌株和谷氨酸棒杆菌高产蛋白突变菌株(经常温室压等离子体诱变所得)均由本实验室保存。大肠杆菌JM109用于质粒构建,谷氨酸棒杆菌是宿主改造和表达重组蛋白的底盘细胞。在LB培养基中于37 ℃、200 r/min条件下培养大肠杆菌,谷氨酸棒杆菌菌株在LBB培养基(色氨酸10 g/L,酵母提取物5 g/L,NaCl 10 g/L,脑心浸出液10 g/L)中于30 ℃、220 r/min培养。大肠杆菌和谷氨酸棒杆菌培养物中使用的卡那霉素和氯霉素质量浓度分别为30、15 μg/mL。
表1 本研究所用菌株
Table 1 Strains used in this study

菌株描述来源E.coli JM109实验室贮存C.glutamicum CGMCC1.15647野生型实验室贮存over-353过表达菌株;含有con-pEC-353质粒;Kanr本研究over-477过表达菌株;含有con-pEC-477质粒;Kanr本研究over-2063过表达菌株;含有con-pEC-2063质粒;Kanr本研究over-2370过表达菌株;含有con-pEC-2370质粒;Kanr本研究over-2761过表达菌株;含有con-pEC-2761质粒;Kanr该研究ko-353敲除菌株;敲除基因GL000353本研究ko-2370敲除菌株;敲除基因GL002370本研究ko-349-350定点突变菌株,突变区域GL000349和GL000350基因间区本研究ko-476-477定点突变菌株,突变区域GL000476和GL000477基因间区本研究ko-973-974定点突变菌株,突变区域GL000973和GL000974基因间区本研究over-353-EGFP含有con-pEC-353质粒,Kanr;pbtac-HT-11-EGFP质粒,Cmr本研究over-477-EGFP含有con-pEC-477质粒;Kanr;pbtac-HT-11-EGFP质粒,Cmr本研究over-2063-EGFP含有con-pEC-2063质粒;Kanr;pbtac-HT-11-EGFP质粒,Cmr本研究over-2370-EGFP含有con-pEC-2370质粒;Kanr;pbtac-HT-11-EGFP质粒,Cmr本研究over-2761-EGFP含有con-pEC-2761质粒;Kanr;pbtac-HT-11-EGFP质粒,Cmr本研究ko-353-EGFP敲除菌株;含有pbtac-HT-11-EGFP质粒,Cmr本研究ko-2370-EGFP敲除菌株;含有pbtac-HT-11-EGFP质粒,Cmr本研究ko-349-350-EGFP定点突变菌株,含有pbtac-HT-11-EGFP质粒,Cmr本研究ko-476-477-EGFP定点突变菌株,含有pbtac-HT-11-EGFP质粒,Cmr本研究ko-973-974-EGFP定点突变菌株,含有pbtac-HT-11-EGFP质粒,Cmr本研究over-2370-rtPTH含有con-pEC-2370质粒;Kanr;pXMJ19-rtPTH质粒,Cmr本研究ko-973-974-rtPTH定点突变菌株,含有pXMJ19-rtPTH质粒,Cmr本研究
1.2 全基因组重测序分析
将诱变筛选得到的菌株进行全基因组重测序。分别准备50 mL 菌液离心样品(OD600 = 10)送至华大基因公司测序。其在北京基因组研究所分别使用PacBio RS Ⅱ平台和Illumina HiSeq 4000平台对突变菌株进行了重测序,以检测潜在的SNP位点。
1.3 蛋白结构分析
利用NCBI数据库,进行Protein BLAST分析,查找相似性最高的同源序列,获得FASTA文件。利用CD-search查找蛋白保守结构域。
1.4 质粒
在该研究所用的质粒见表2。组成型质粒con-pEC-XK99E,用作谷氨酸棒杆菌中基因过表达载体;质粒pK18 mobsacB为谷氨酸棒杆菌基因敲除载体;质粒pXMJ19为谷氨酸棒杆菌中外源蛋白表达载体。上述3个质粒均由实验室保存。
表2 本研究所用质粒
Table 2 Plasmids used in this study

质粒描述来源con-p19-EGFP由pXMJ19质粒改造,组成型tac启动子;插入EGFP基因;Cmr实验室贮存con-pEC-XK99E由pEC-XK-99E质粒改造,组成型trc启动子;Kanr实验室贮存pK18 mobsacB谷氨酸棒杆菌敲除质粒;蔗糖致死型;Kanr实验室贮存pbtac-HT-11-EGFP诱导型型tac启动子;Cmr实验室贮存con-pEC-353con-pEC-XK99E质粒;插入基因GL000353;Kanr本研究con-pEC-477con-pEC-XK99E质粒;插入基因GL000477;Kanr本研究con-pEC-2063con-pEC-XK99E质粒;插入基因GL002063;Kanr本研究con-pEC-2370con-pEC-XK99E质粒;插入基因GL002370;Kanr本研究con-pEC-2761con-pEC-XK99E质粒;插入基因GL002761;Kanr本研究pK18-353pK18 mobsacB质粒;插入基因GL000353上下游片段;Kanr本研究pK18-2370pK18 mobsacB质粒;插入基因GL002370上下游片段;Kanr本研究pK18-349-350pK18 mobsacB质粒;插入基因GL000349和GL000350突变片段;Kanr本研究pK18-476-477pK18 mobsacB质粒;插入基因GL000476和GL000477突变片段;Kanr本研究pK18-973-974pK18 mobsacB质粒;插入基因GL000973和GL000974突变片段;Kanr本研究pXMJ19-rtPTHpXMJ19质粒;诱导型tac启动子;插入Spy-rtPTH基因;Cmr实验室贮存
1.4.1 过表达质粒构建
根据全基因组重测序数据,分析突变位点。根据谷氨酸棒杆菌基因组序列设计扩增突变基因片段的引物。分别扩增基因片段,同时用EcoR Ⅰ和Pst Ⅰ对质粒con-pEC-XK99E进行酶切。用ABclonal同源同组连接基因片段和酶切载体,连接产物转化大肠杆菌感受态。培养出的转化子采用PCR方法验证。正确的转化子于LB液体培养中培养以提取阳性克隆质粒并测序,测序正确的过表达质粒命名为con-pEC-gene。
1.4.2 敲除质粒构建
以谷氨酸棒杆菌CGMCC1.15647基因组中待敲除的突变基因上下游750 bp为模板设计引物。用引物Ko-gene-LF和Ko-gene-LR扩增上游同源臂,Ko-gene-RF和Ko-gene-RR扩增下游同源臂,引物Ko-gene-LF和Ko-gene-RR分别引入同源序列,便于和pK18 mobsacB载体进行同源重组。用EcoRⅠ和Hind Ⅲ对质粒pK18 mobsacB进行酶切。后续操作同1.3.1,构建的敲除质粒命名为pK18-gene。
1.5 过表达和敲除菌株构建
1.5.1 过表达菌株构建
测序正确的过表达质粒con-pEC-gene分别通过电转化的方法导入谷氨酸棒杆菌感受态,30 ℃培养24 h。培养的单菌落进行PCR验证,构建SNP基因过表达重组菌株。
1.5.2 敲除菌株构建
敲除载体pK18-gene分别电转化导入谷氨酸棒杆菌中。阴性对照为野生型菌株。30 ℃培养24 h后进行蔗糖敏感性筛选,长出的单菌落同时点在LBB+K和LBB+K+蔗糖的固体平板上。12 h后挑选在LBB+K上生长而LBB+K+蔗糖不长的单菌落,用LBB液体培养基过夜培养后转接到LBB+蔗糖液体培养基中培养12 h。取1 μL菌液在LBB+蔗糖固体平板上划线,长出的单菌落分别点在LBB和LBB+K固体平板上,挑选LBB板上长而LBB+K不长的单菌落进行PCR验证或测序,构建SNP基因敲除菌株。
1.6 生长曲线测定
分别将SNP重组菌株接种于10 mL的LBB培养基中于30 ℃、220 r/min培养,12 h后转接至100 mL的LBB培养基中,使初始OD600值为0.3。在30 ℃、220 r/min条件下培养36 h,一定时间取1次样,测定OD600值。
1.7 荧光强度测定
将诱导型质粒pbtac-HT-11-EGFP转化至构建的SNP重组菌株中,挑选单菌落于24孔板中(每孔含2 mL LBB培养基)。在30 ℃、220 r/min条件下培养12 h后,将20 μL菌液转接至新的24孔板中并在每孔加入2 μL IPTG(24 mg/mL),培养 24 h后测量荧光强度和OD600吸光值,计算单位荧光强度(FI/OD600)。
1.8 rtPTH外源蛋白表达和分析
选择有价值的外源蛋白rtPTH进行SNP过表达和敲除重组菌株的进一步分析。将质粒pXMJ19-rtPTH电转至over-2370和ko-973-974,后续操作同1.6。将rtPTH的发酵产物在12 000 r/min下离心1 min,并将上清液制成蛋白样品,进行SDS-PAGE分析。
1.9 SWISS-MODEL预测分析
利用SWISS-MODEL软件,输入蛋白序列,搜索以匹配相关模板,选择匹配性最高的模板,建立蛋白模型,并利用NCBI数据库分析蛋白功能。
2 结果与分析
2.1 突变菌株的全基因组重测序分析
将诱变菌株的全基因组重测序结果进行整理分析后,结果显示突变菌株中共有33个SNP和7个InDel。SNP包括位于编码序列(CDS)中的22个非同义突变和位于基因间区域中的11个其他突变。其中编码序列主要涉及5个基因(表3),除GL002370编码1种预测蛋白以外,GL000353,GL000477和GL002063编码的蛋白质分别具有氧化还原酶,核酸内切酶和酪氨酸重组酶活性,GL002761编码ATP依赖性Clp蛋白酶[9]。在位于GL000349~GL000350,GL000476~GL000477,GL000973~GL000974,GL002062~GL002063,GL002369~GL002370,GL002760~GL002761的基因间区域检测到突变。因此,这些突变可能是增加外源蛋白质产量的相关靶基因,但其具体功能仍不清楚,需要进一步研究以探索这些基因与其表型变化之间的关系。
表3 全基因组重测序突变位点分析
Table 3 The analysis for genome resequencing mutation site
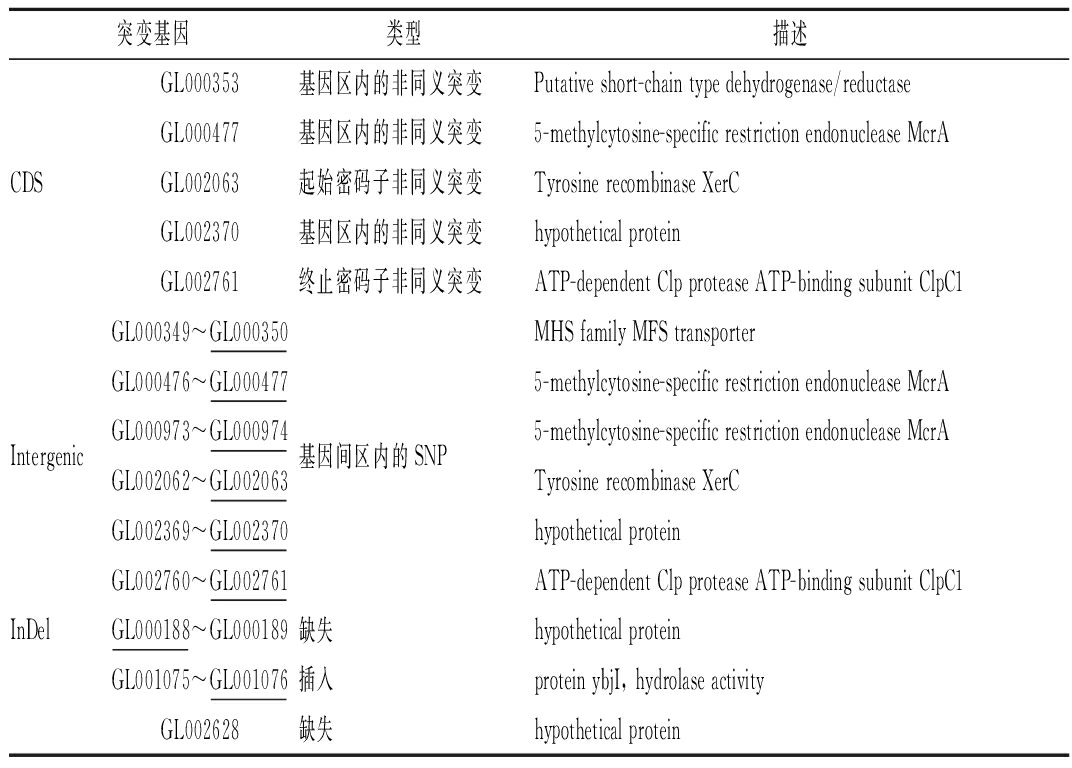
突变基因类型描述GL000353基因区内的非同义突变Putative short-chain type dehydrogenase/reductaseGL000477基因区内的非同义突变5-methylcytosine-specific restriction endonuclease McrACDSGL002063起始密码子非同义突变Tyrosine recombinase XerCGL002370基因区内的非同义突变hypothetical proteinGL002761终止密码子非同义突变ATP-dependent Clp protease ATP-binding subunit ClpC1GL000349~GL000350MHS family MFS transporterGL000476~GL0004775-methylcytosine-specific restriction endonuclease McrAIntergenicGL000973~GL000974基因间区内的SNP5-methylcytosine-specific restriction endonuclease McrAGL002062~GL002063Tyrosine recombinase XerCGL002369~GL002370hypothetical proteinGL002760~GL002761ATP-dependent Clp protease ATP-binding subunit ClpC1InDelGL000188~GL000189缺失hypothetical proteinGL001075~GL001076插入protein ybjI, hydrolase activityGL002628缺失hypothetical protein
注:带下划线的基因是经分析指向的突变靶基因
2.2 SNP基因编码蛋白的结构分析
从分析结果可知,SNP主要集中在GL000353,GL000477,GL002063,GL002370和GL002761这5个基因中,因此,该研究主要分析验证这5个基因对于外源蛋白表达量的影响。首先通过NCBI数据库分析了5个基因编码的蛋白的结构,结果如图1所示。GL000353编码的蛋白共311个氨基酸,是短链型脱氢酶/还原酶。它包含1个NADB结构域,该结构域存在于许多代谢途径的脱氢酶中,例如糖酵解和许多其他氧化还原酶[10]。GL000477编码5-甲基胞嘧啶特异性限制性核酸内切酶McrA,共340个氨基酸,它是典型的限制性修饰(RM)系统中的一种酶,该系统可保护宿主免受外源DNA的侵害[11]。McrA包含1个His-Asn-His (HNH)基序,这个基序的第1个氨基酸在HNH核酸酶中的重要催化功能[12]。GL002063编码特异性酪氨酸重组酶XerC,共315个氨基酸。Xer位点特异性重组系统可以把通过同源重组形成的二聚体染色体转化为单体,它的C末端结构域在核心位点结合,切割和重新连接DNA链,而N末端结构域在很大程度上负责与同源臂结合[13]。GL002370编码1个含有93个氨基酸的蛋白。GL002761编码具有分子伴侣活性和ATP结合亚基的ClpC蛋白酶,由925个氨基酸构成。此蛋白酶有2个Clp-N结构域,1个ClpB-D2-small结构域以及1个UVR结构域[14]。
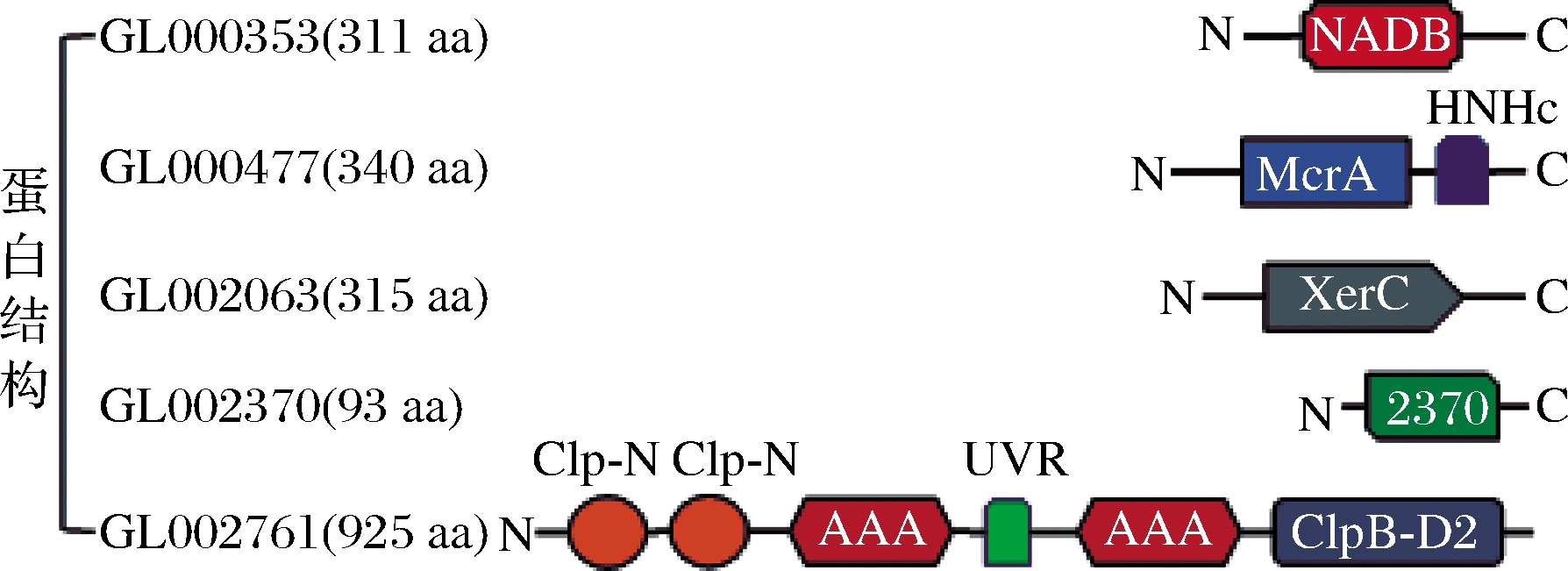
图1 SNP基因编码的蛋白结构示意图
Fig.1 Schematic diagram of the protein structures encoded by the SNP genes
2.3 SNP基因过表达和敲除重组菌株的构建
为了验证SNP基因对于外源蛋白表达的影响,首先构建了SNP基因的过表达和敲除重组菌株。将SNP基因过表达质粒con-pEC-gene电转入谷氨酸棒杆菌中,培养出的转化子进行PCR验证,结果如图2-a所示,成功构建SNP基因过表达菌株:over-353,over-477,over-2063,over-2370,over-2761。将最后培养的敲除菌株进行PCR验证,定点突变菌株进行测序。结果如图2-b和图2-c所示,成功敲除GL000353和GL002370,并且定点突变菌株测序正确。因此,成功构建了SNP基因敲除菌株:ko-353,ko-2370,ko-349-350,ko-476-477,ko-974-973。
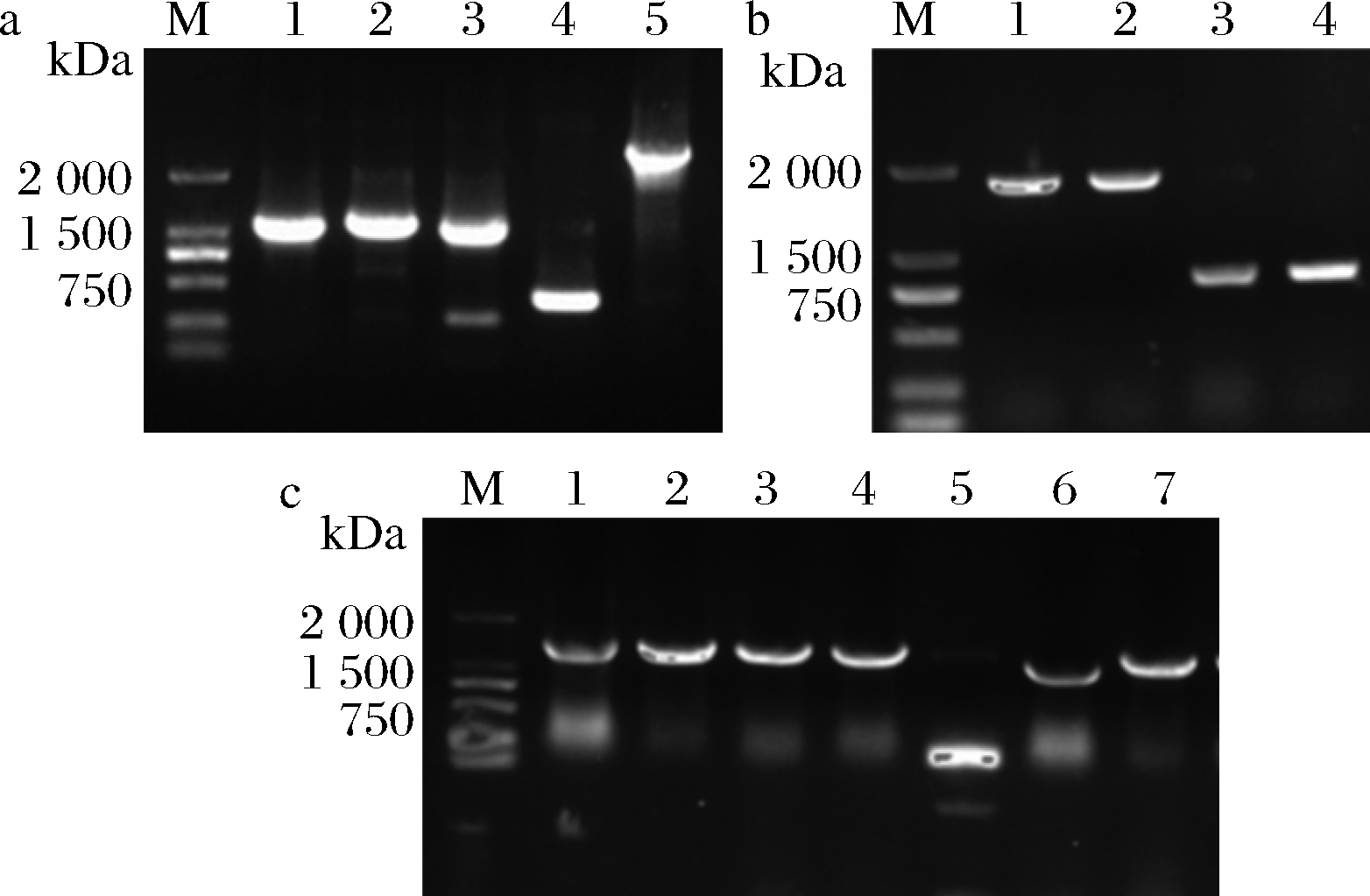
a-SNP过表达菌株PCR验证(M:2 000 marker,1~5:over-353,over-477,over-2063,over-2370,over-2761菌株PCR验证);b-敲除GL000353菌株PCR验证(M:2 000 marker,1:阴性对照,2~4:GL000353敲除菌株);c-敲除GL002370菌株PCR验证(M:2 000 marker,1:阴性对照,2~7:GL002370敲除菌株)
图2 SNP基因过表达和敲除菌株PCR验证
Fig.2 The PCR verification of SNP genes overexpression and knockout strains
2.4 SNP基因过表达和敲除重组菌株生长曲线的测定
为了观察SNP基因的过表达和敲除是否对菌株的生长有影响,进行了生长曲线的测定。结果如图3所示,除了over-353,over-2761和ko-2370前期生长较为缓慢外,其余SNP基因的过表达和敲除对菌株的生长没有太大影响。

a-SNP基因过表达菌株生长曲线;b-SNP基因敲除菌株生长曲线
图3 SNP基因过表达菌株和敲除菌株生长曲线
Fig.3 Growth curve of SNP genes overexpression and knockout strains
注:WT表示野生型(wild type)(下同)
2.5 SNP重组菌株对EGFP外源蛋白表达的影响
为了探究SNP基因重组菌株对于外源蛋白表达的影响,将EGFP作为报告蛋白以观察荧光蛋白表达量的变化。将质粒pbtac-HT-11-EGFP分别电转入SNP基因重组菌株,在多孔板中培养24 h后检测荧光值。结果如图4所示,在SNP过表达菌株中,除over-2761-EGFP的荧光值明显降低以外,其余菌株SNP过表达菌株的荧光值都增加,且over-2370-EGFP的荧光值最高。在SNP敲除菌株中,ko-2370-EGFP和ko-349-350-EGFP荧光值明显降低,而ko-973-974-EGFP的荧光值最高,其次是ko-353-EGFP,ko-476-477-EGFP。其中,over-2761-EGFP荧光值降低可能是GL002761编码的Clp蛋白酶将EGFP外源蛋白降解所致。ko-353-EGFP和over-353-EGFP荧光强度均比野生型(wild type,WT)的高,且前者比后者的提升幅度更明显,可能是由于过表达GL000353编码的脱氢酶/还原酶加快了整个菌株的能量代谢流,使荧光蛋白的表达增加,而敲除该脱氢酶/还原酶可能改变能量代谢的流向,使能量更多的用于外源蛋白的合成。over-477-EGFP,ko-476-477-EGFP和ko-973-974-EGFP均使荧光值增强,而GL000477和GL000974都编码5-甲基胞嘧啶特异性限制性核酸内切酶McrA,该蛋白与外源DNA的导入有关,对增强外源蛋白产量的机制还需要进一步研究。同样over-2370-EGFP显著增强了EGFP的表达,但其编码的蛋白未知,还需要进一步探究。因此,基因GL002370以及GL000974确实与提高蛋白表达产量密切相关。
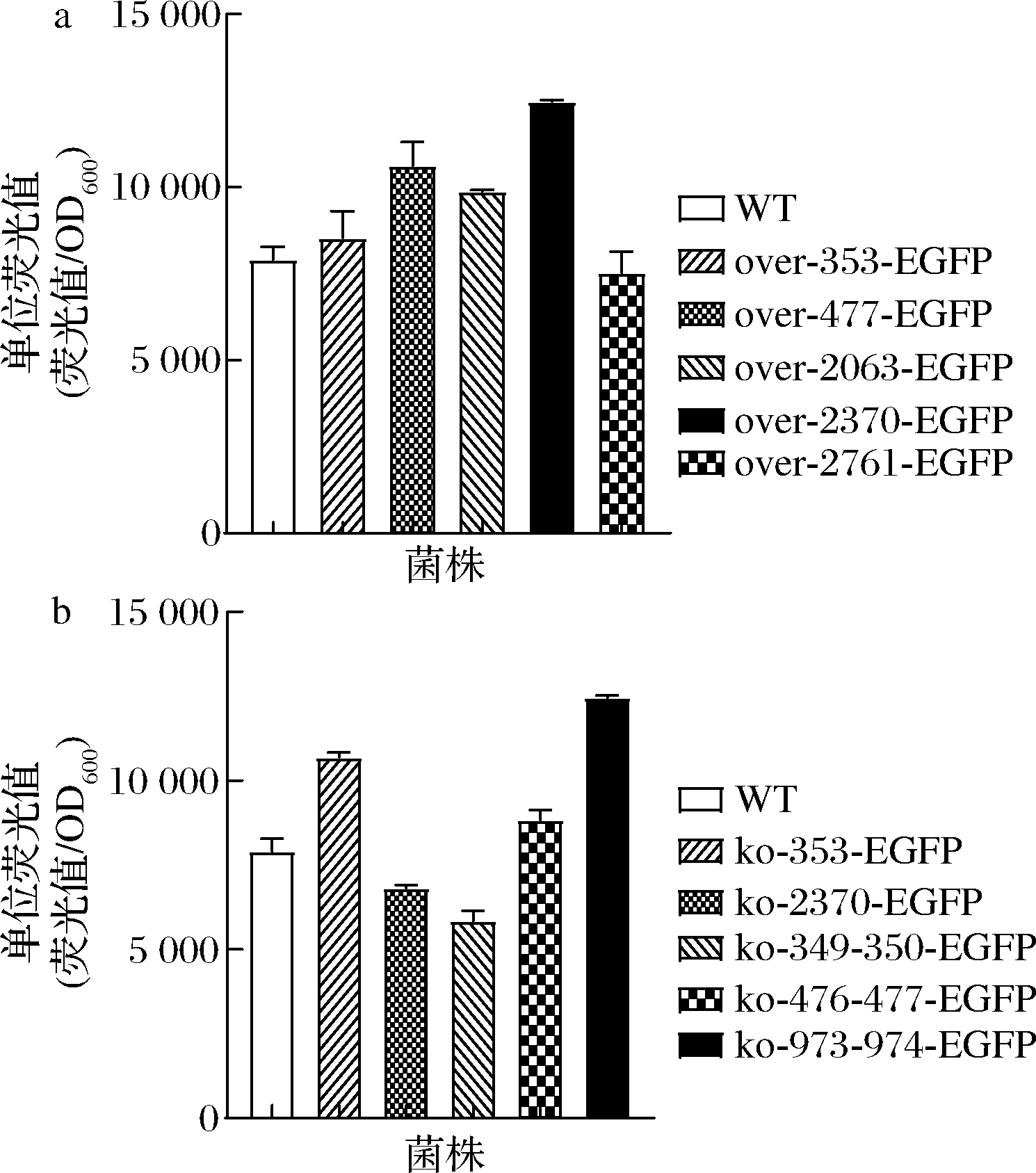
a-SNP过表达菌株荧光值;b-SNP敲除菌株荧光值
图4 SNP基因过表达和敲除菌株单位荧光强度
Fig.4 Unit fluorescence intensity of SNP genes overexpression and knockout strains
2.6 SNP基因重组菌株对其他外源蛋白表达的影响
为了观察SNP基因重组菌株对其他外源蛋白是否具有同样的效果。分别选择上述研究中对EGFP表达量最具影响的2株重组菌,over-2370和ko-973-974。在这2株SNP重组菌株中表达有价值的外源蛋白rtPTH,以验证它们对其他外源蛋白表达量的影响。rhPTH是一种肽药物,包含1~34个N末端氨基酸,已用于治疗骨质疏松症[15]。该研究使用了新的融合标签Spy来促进rtPTH的稳定表达[16]。将质粒pXMJ19-rtPTH分别电导入over-2370和ko-973-974,构建over-2370-rtPTH和ko-973-974-rtPTH重组菌株。重组菌株分别诱导表达24 h后,结果如图5所示。Spy-rtPTH蛋白分子质量为20.8 kDa,over-2370-rtPTH和ko-973-974-rtPTH均具有比WT有更高的产量。因此,在分析的SNP中,GL002370以及GL000974编码的蛋白确实能够使外源蛋白表达量明显增加。
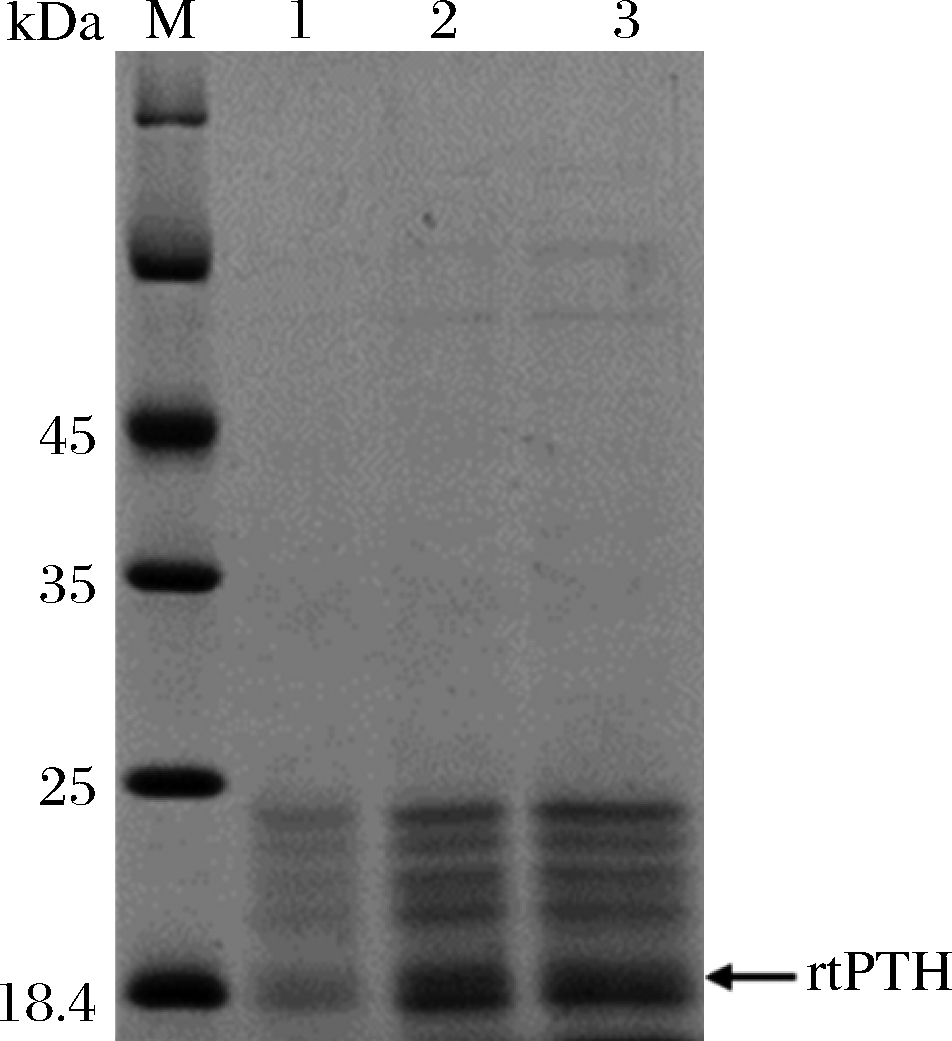
M-26610 marker,1-WT-rtPTH,2-ko-973-974-rtPTH,3-over-2370-rtPTH
图5 rtPTH外源蛋白表达验证
Fig.5 The verification for rtPTH heterologous protein expression
2.7 基因GL002370编码的蛋白预测分析
基于以上蛋白表达结果的分析,基因GL000974和GL002370编码的蛋白对提高外源蛋白产量影响较大,其中基因GL000974编码的蛋白功能已知,而GL002370编码的蛋白功能未知。因此分析了GL002370编码蛋白的结构和功能。结果如图6所示,经SWISS-MODEL分析,预测基因GL002370编码的蛋白与谷氨酸棒杆菌的硫氧还蛋白依赖性砷酸还原酶最为相似,为α-螺旋的同源二聚体结构。硫氧还蛋白依赖性砷酸还原酶(ArsC)属于砷酸盐还原酶的第二家族,该家族使用硫氧还蛋白作为电子供体,在生物砷解毒途径中起重要作用[17]。该家族在结构和功能上与低分子量蛋白质酪氨酸磷酸酶接近,并且具有磷酸酶活性。磷酸酶和激酶共同构成去磷酸化和磷酸化的调控系统,处于能量代谢的关键位置。而外源蛋白表达本身就是一个消耗大量能量的过程,因此推测这个基因可能影响蛋白生成的能量代谢,从而导致了外源蛋白产量的增加,但要明确over-2370影响蛋白产量的作用机理还需要进一步探究。
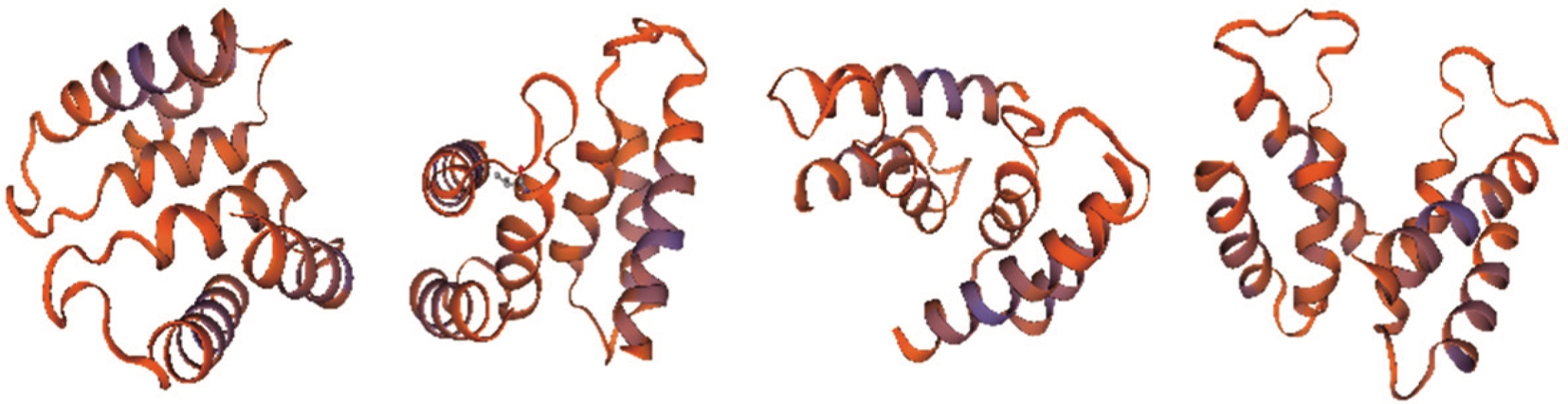
图6 SWISS-MODEL预测基因GL002370编码的蛋白结构图
Fig.6 The GL002370 protein structure predicted by SWISS-MODEL
3 结论与讨论
在这项研究中,为了挖掘与蛋白表达相关的具体基因并获得高产外源蛋白的谷氨酸棒杆菌菌株。首先基于全基因组重测序策略,分析出与蛋白表达相关的靶基因,并利用NCBI数据库分析了蛋白的结构。其次构建了SNP基因过表达和敲除菌株,评价生长曲线以及SNP重组菌株中EGFP的表达水平。其中,SNP过表达菌株over-2370和ko-973-974使蛋白表达水平显著增强。此外,我们选择了有价值的外源蛋白rtPTH以验证SNP基因重组菌株对其他外源蛋白的适用性。最后利用SWISS-MODEL软件进行了基因GL002370编码的蛋白的建模,并分析了蛋白的功能。
但是,在研究过程中存在一些需要进一步研究的问题。首先,目前只研究了单基因过表达和一些基因敲除对于蛋白表达的影响,其他基因的敲除和多基因的组合情况有待进一步研究。其次,基因GL002370编码的具体蛋白以及导致蛋白产量增加的具体机制尚不明确,需要进一步深入研究。最后,提高外源蛋白产量除了宿主改造还有多个方面,例如优良的表达载体,表达元件等,可以探究不同组合之间的相互关系,以及对蛋白产量的影响[18-19]。这些研究都具有重要意义,要求研究人员进一步探索和发现新的目标。
[1] KIM K, CHOE D, LEE D H, et al.Engineering biology to construct microbial chassis for the production of difficult-to-express proteins[J].International Journal of Molecular Sciences, 2020, 21(3):990.
[2] LIU Y F, LIU L, LI J H, et al.Synthetic biology toolbox and chassis development in Bacillus subtilis [J].Trends in Biotechnology, 2019, 37(5):548-562.
[3] LEE J Y, NA Y A, KIM E, et al.The actinobacterium Corynebacterium glutamicum, an industrial workhorse [J].Journal of Microbiology and Biotechnology, 2016, 26(5):807-822.
[4] ZHANG X, ZHANG X M, XU G Q, et al.Integration of ARTP mutagenesis with biosensor-mediated high-throughput screening to improve L-serine yield in Corynebacterium glutamicum [J].Applied Microbiology and Biotechnology, 2018, 102(14):5 939-5 951.
[5] LIU Y F, SU A Q, LI J H, et al.Towards next-generation model microorganism chassis for biomanufacturing[J].Applied Microbiology and Biotechnology, 2020, 104(21):9 095-9 108.
[6] 郭鹤宝, 王星, 何山文, 等.表型特征结合基因组分析鉴定不同菌落形态Bacillus velezensis ACCC 19742 [J].生物技术通报, 2020, 36(2):142-148.
GUO H B, WANG X, HE S W, et al.Phenotypic characteristics combined with genomic analysis to identify different colony morphology Bacillus Velezensis ACCC 19742 [J].Biotechnology Bulletin, 2020, 36(2):142-148.
[7] 戴利铭, 李岚岚, 刘一贤, 等.解淀粉芽孢杆菌生防菌BS-3全基因组测序及生物信息分析 [J].微生物学通报, 2021,48(6):2 073-2 088.
DAI L M, LI L L, LIU Y X, et al.Whole genome sequencing and genomics analysis of Bacillus amyloliquefaciens BS-3 with biocontrol activity[J].Microbiology Bulletin,2021,48(6):2 073-2 088.
[8] ZHANG W B, YAN H L, ZHU Z C, et al.Genome-wide identification of the Sec-dependent secretory protease genes in Erwinia amylovora and analysis of their expression during infection of immature pear fruit [J].Journal of Zhejiang University-Science B(Biomedicine & Biotechnology), 2020, 21(9):716-726.
[9] TRENTINI D B, SUSKIEWICZ M J, HEUCK A, et al.Arginine phosphorylation marks proteins for degradation by a Clp protease [J].Nature, 2016, 539(7 627):48-53.
[10] CROSS E M, ARAG O D, SMITH K M, et al.Structural characterization of a short-chain dehydrogenase/reductase from multi-drug resistant Acinetobacter baumannii[J].Biochemical and biophysical Research Communications, 2019, 518(3):465-471.
O D, SMITH K M, et al.Structural characterization of a short-chain dehydrogenase/reductase from multi-drug resistant Acinetobacter baumannii[J].Biochemical and biophysical Research Communications, 2019, 518(3):465-471.
[11] ![]() M, QIAN L, OKURA R, et al.Bacterial autoimmunity due to a restriction-modification system [J].Current Biology, 2016, 26(3):404-409.
M, QIAN L, OKURA R, et al.Bacterial autoimmunity due to a restriction-modification system [J].Current Biology, 2016, 26(3):404-409.
[12] XU S Y, GUPTA Y K.Natural zinc ribbon HNH endonucleases and engineered zinc finger nicking endonuclease [J].Nucleic Acids Research, 2013, 41(1):378-390.
[13] FERREIRA H, BUTLER-COLE B, BURGIN A, et al.Functional analysis of the C-terminal domains of the site-specific recombinases XerC and XerD [J].Journal of Molecular Biology, 2003, 330(1):15-27.
[14] WANG F, MEI Z Q, QI Y T, et al.Structure and mechanism of the hexameric MecA-ClpC molecular machine [J].Nature, 2011, 471(7 338):331-335.
[15] BAKHTIARI N, AMINI BAYAT Z, SAGHARIDOUZ S, et al.Overexpression of recombinant human teriparatide, rhPTH (1-34) in Escherichia coli:An innovative gene fusion approach [J].Avicenna Journal of Medical Biotechnology, 2017, 9(1):19-22.
[16] RUAN A, REN C, QUAN S.Conversion of the molecular chaperone Spy into a novel fusion tag to enhance recombinant protein expression [J].Journal of Biotechnology, 2020, 307:131-138.
[17] 康振辉. NADPH依赖性硫氧还蛋白还原酶C在植物质体中的作用 [J].中国生物化学与分子生物学报, 2019, 35(2):121-130.
KANG Z H.Roles of NADPH-dependent thioredoxin reductase C in chloroplast[J].Chinese Journal of Biochemistry and Molecular Biology, 2019, 35(2):121-130.
[18] STELLA R G, WIECHERT J, NOACK S, et al.Evolutionary engineering of Corynebacterium glutamicum [J].Biotechnology Journal 2019, 14(9):1800444.
[19] LIU X X, ZHAO Z H, ZHANG W, et al.Bicistronic expression strategy for high-level expression of recombinant proteins in Corynebacterium glutamicum [J].Engineering in Life Sciences, 2017, 17(10):1 118-1 125.