乳酸,也叫2-羟基丙酸,是一种无色无味的单羧基酸[1],被广泛地应用于食品、药品、生物材料等领域[2]。乳酸是聚乳酸的合成单体,聚乳酸是生物可降解塑料的合成原料,被认为是基于化石燃料合成塑料的良好替代品[3]。因羟基的结合位置不同,乳酸可分为D-乳酸(D-lactic acid,D-LA)和L-乳酸,聚D-乳酸和聚L-乳酸按一定比例地混合能够提升聚乳酸材料的热稳定性和结晶度[4]。由于人们绿色环保意识的不断提高,对于生物可降解塑料的需求也不断增加,乳酸的产量仍需不断提高。
截至2008年,全球乳酸年产量约为37万t,有近90%的生产厂家采用发酵法进行生产[5]。2018年,全世界D-乳酸产量是1.6万t,而D-乳酸的需求量约为2.6万t/年[6]。利用发酵法生产乳酸的微生物来源广泛,主要有乳酸杆菌[7],酿酒酵母[8],索氏假丝酵母[9],马氏克鲁维酵母[10],产甘油假丝酵母[11]等。
由于目前以细菌发酵生产乳酸需要控制环境为中性pH,获得的是乳酸盐,需重新酸化后变成乳酸且过程中对营养条件有较多要求,而酵母菌能够利用价格低廉的底物且对pH有更好的耐受性,所以对于酵母菌生产乳酸的研究也越来越多[12]。热带假丝酵母是一种非常规的二倍体酵母[13],结合其自身的特点当前主要应用于长链二元酸的生物生产[14-15]。
本研究以热带假丝酵母CU-206为出发菌株,敲除了丙酮酸脱羧酶(pyruvate decarboxylase,PDC)基因(pdc),整合表达了异源D-乳酸脱氢酶(D-lactate dehydrogenase,D-LDH)基因(d-ldh)和内源2-羟基酸脱氢酶(2-hydroxyacid dehydrogenase,2-HAD)(2-had)基因,探究了热带假丝酵母菌进行乳酸生产的潜力。其中来自大肠杆菌的D-LDH[16]和来自其本身的2-HAD[17]能够催化丙酮酸向D-乳酸的转化。热带假丝酵母生产D-乳酸的参考代谢路径见文献[18]。
1 材料与方法
1.1 试验材料
1.1.1 质粒、菌株和引物
本实验中所使用的载体pMD19-T Simple购自TaKaRa公司,质粒均在此载体的基础上构建获得;质粒Ts-gda324-URA3、Ts-PGAPDH-GFP-TGAPDH由本实验室保藏;菌株CU-206是热带假丝酵母ATCC20336尿嘧啶(URA3)缺陷型菌株;构建菌株见表1。本实验所用的PCR引物见表2,DNA测序等由苏州金唯智生物科技有限公司提供。
表1 本研究所用菌株
Table 1 Strains used in this study
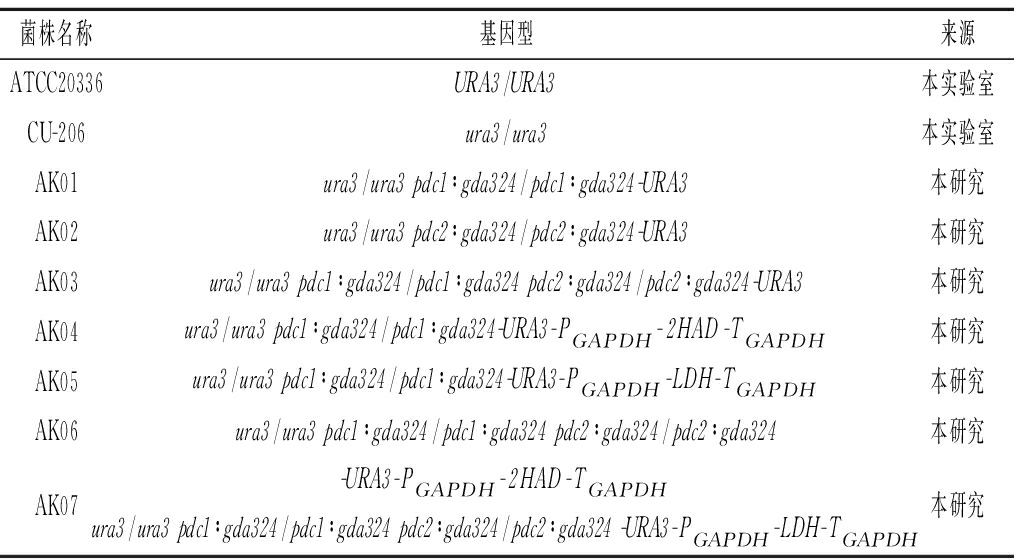
菌株名称基因型来源ATCC20336URA3/URA3本实验室CU-206ura3/ura3本实验室AK01ura3/ura3 pdc1∶gda324/pdc1∶gda324-URA3本研究AK02ura3/ura3 pdc2∶gda324/pdc2∶gda324-URA3本研究AK03ura3/ura3 pdc1∶gda324/pdc1∶gda324 pdc2∶gda324/pdc2∶gda324-URA3本研究AK04ura3/ura3 pdc1∶gda324/pdc1∶gda324-URA3-PGAPDH-2HAD-TGAPDH本研究AK05ura3/ura3 pdc1∶gda324/pdc1∶gda324-URA3-PGAPDH-LDH-TGAPDH本研究AK06ura3/ura3 pdc1∶gda324/pdc1∶gda324 pdc2∶gda324/pdc2∶gda324本研究AK07-URA3-PGAPDH-2HAD-TGAPDH本研究ura3/ura3 pdc1∶gda324/pdc1∶gda324 pdc2∶gda324/pdc2∶gda324 -URA3-PGAPDH-LDH-TGAPDH
表2 本研究所用引物
Table 2 Primers used in this study

引物名称序列(5'-3')酶切位点PDC1-F1CGACGCGTAGATTGCACCAATTGAAAGTCGMlu ⅠPDC1-R1CGACGCGTATTGTTTGGCTTGAGCAACCMlu ⅠPDC1wcF1AACCTCAATAGTCCCCGAA-PDC1wcR1AGACCCGTGTGATAATAGCAA-URA3-FTACTCTAACGACGGGTACAAC-URA3-RACCCGATTTCAAAAGTGCAGA-rPDC1-F1AACTGCAGGTGGAAGACGGTAAAGTCACCGPst ⅠrPDC1-R1GCTCTAGAACGGTGCAAGAGATCTCCACTAXba ⅠPDC2-F1CGACGCGTAACAAAAGATCGGAATCTGCTMlu ⅠPDC2-R1CGACGCGTACCATTTCCAAATCCGTCGTGMlu ⅠPDC2wcF1ATACTGTACTTCCAACGCAAA-PDC2wcR1AAGGAACCCCATGAAATCCAG-rPDC2-F1AACTGCAGGTTGCCGTCCTTGCCTTCTTTGPst ⅠrPDC2-R1GCTCTAGACACCATCAACAGTCGCAACAACXba ⅠPDC1n-F1CGACGCGTCTGAAGTTGTTGAAACCGTCCAMlu ⅠPDC1n-R1CGACGCGTCAGTGGCATAACCAATCGAACMlu ⅠrPDC1n-F1AACTGCAGTTTCTGGTCTTGTAGTTGTAGGPst ⅠrPDC1n-R1GCTCTAGACGTGGTTGAGTTCCACTCCGACXba ⅠPDC2n-F1CGACGCGTTATTCATTGCCCTTGGACCAGMlu ⅠPDC2n-R1CGACGCGTCTTCCAGTAGTCACCGAGTMlu ⅠrPDC2n-F1AACTGCAGTTGTGATCCAACATAATCAAGPst ⅠrPDC2n-R1GCTCTAGAATGCGTCGCTTGACGAAATCATXba Ⅰ2HAD-F1CCCAAGCTTATGACCTCCGCTGTTAGACCHind Ⅲ2HAD-R1GCGTCGACTTAAAAAACGCCTTGCTGTTSa ⅡLDH-F1CCCAAGCTTGCCACCATGAAATTGGCTGTHind ⅢLDH-R1GCGTCGACCTAAACCAATTCATTTGGACSa ⅡP-2HAD-T-f1(1n)TACAACTACAAGACCAGAAAAACGTGGTATGGTTGTAAGA-P-2HAD-T-f1(2n)TTGATTATGTTGGATCACAAAACGTGGTATGGTTGTAAGA-P-2HAD-T-r1(1-2n)TGACGTCCTAGAAGCTTAGTTCTGGTTTAGAAGTAGGGAC-q2had-FGACCTACTGCTGTTACTG-q2had-RGCCACCATTGACTAATATCT-qldh-FCTGGTGTTATTGGTACTGGTAA-qldh-RGGCAGCAGATGGATATGG-ACT1-FGACCGAAGCTCCAATGAATC-ACT1-RAATTGGGACAACGTGGGTAA-
注:下划线部分的碱基序列为酶切位点
1.1.2 主要培养基、试剂和仪器
LB培养基(g/L):蛋白胨10,酵母提取物5,氯化钠10;
MM培养基(g/L):酵母氮源培养基(yeast nitrogen base,YNB) 6.7,葡萄糖20,硫酸铵10;
SM培养基(g/L):YNB 6.7,葡萄糖20,硫酸铵10,尿嘧啶0.06;
SM+5-氟乳清酸(5-fluoroorotic acid,5-FOA)培养基:在SM培养基的基础上添加2 g/L 5-FOA;
YPD培养基(g/L):葡萄糖20,酵母粉10,蛋白胨20;固体培养基需加入20 g/L琼脂粉。
乳酸、丙酮酸标准品,北京索莱宝科技有限公司;乙醇标准品,上海阿拉丁生化科技股份有限公司;YNB、5-FOA,上海生物工程股份有限公司;酵母RNA提取及逆转录试剂盒,宝生物工程(大连)有限公司;荧光定量PCR试剂盒,南京诺唯赞生物科技股份有限公司。
ETC811PCR扩增仪,上海东胜兴业科学仪器有限公司;WD-9403B琼脂糖凝胶电泳仪,北京市六一仪器厂;UV-2100紫外可见分光光度计,上海尤尼科有限公司;1260 Infinity高效液相色谱仪,安捷伦科技有限公司。
1.2 试验方法
1.2.1 敲除框的构建
基于热带假丝酵母ATCC20336的基因组测序结果,找到丙酮酸脱羧酶系PDC1,PDC2。以pdc1的基因敲除框为例,热带假丝酵母ATCC20336的基因组为模板,设计引物PDC1-F1,PDC1-R1,分别带有Mlu I酶切位点,PCR扩增pdc1基因,对PCR产物进行凝胶回收,并与pMD19-T Simple载体进行连接,得到质粒Ts-PDC1;再以质粒Ts-PDC1为模板,设计反向PCR引物rPDC1-F1,rPDC1-R1(分别带有Pst Ⅰ和Xba Ⅰ酶切位点),进行PCR扩增,得到片段PDC1u-Ts-PDC1d后进行凝胶回收,再用限制性内切酶Pst Ⅰ和Xba Ⅰ消化,留存备用。同时对质粒Ts-gda324-URA3用限制性内切酶Pst Ⅰ和Xba Ⅰ消化,将其与凝胶回收且酶切消化后的片段PDC1u-Ts-PDC1d进行连接,获得重组质粒Ts-PDC1-gda324-URA3-PDC1(图1-a)。对上述重组质粒进行限制性内切酶Mlu Ⅰ消化后得到敲除框片段PDC1-gda324-URA3-PDC1。pdc1等位基因第二拷贝的敲除框与上述敲除框存在差异,第二拷贝敲除框的同源臂pdc1n为pdc1内侧基因,第一拷贝敲除框的同源臂为pdc1外侧基因。pdc2基因的敲除框利用与上述相同的方法进行构建。
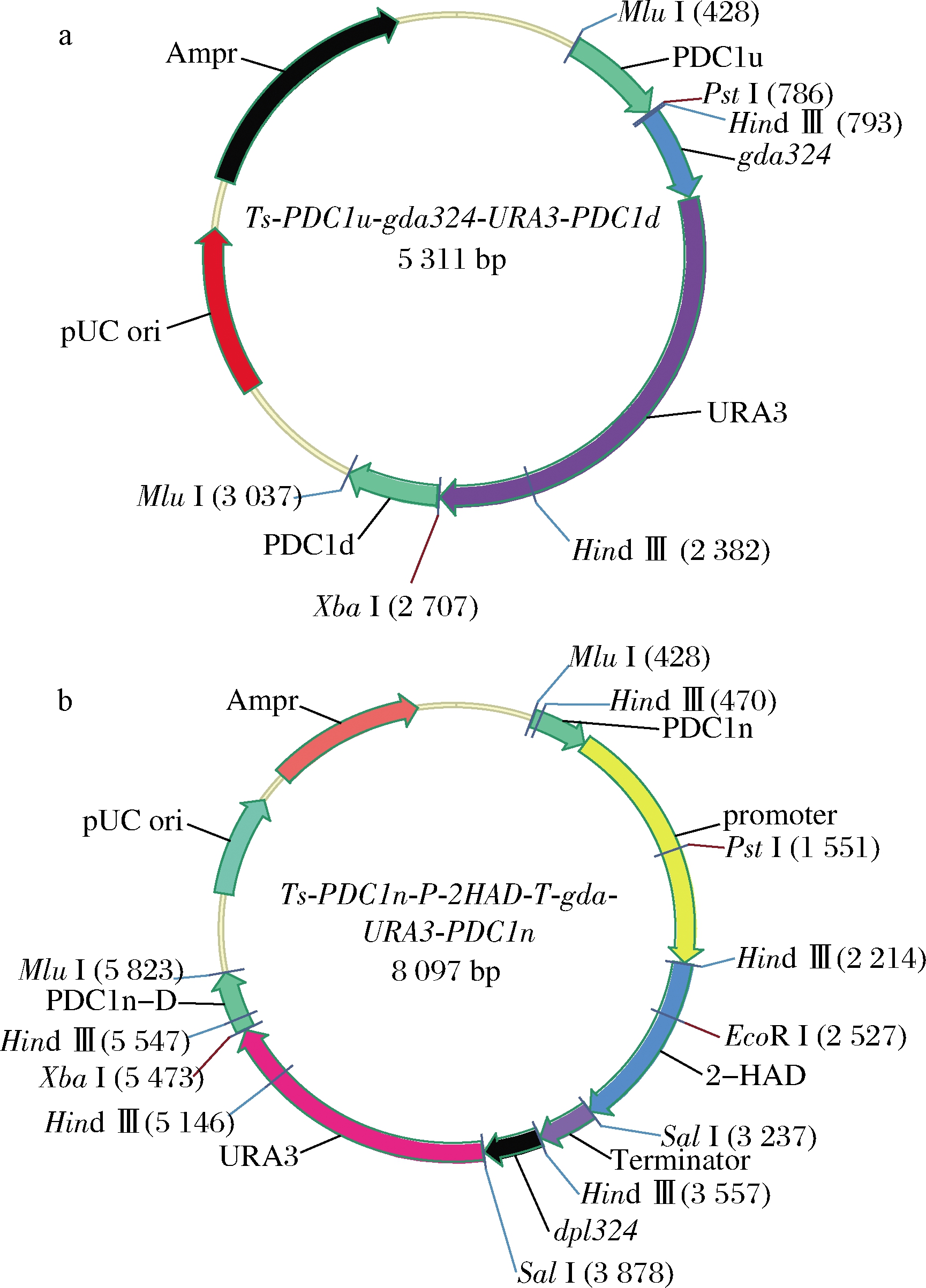
a-pdc1第一拷贝敲除框;b-2-had基因整合框
图1 pdc1基因敲除框和2-had基因整合框的构建
Fig.1 Construction of pdc1 disruption cassette and 2-had integration cassette
1.2.2 整合框的构建
在1.2.1 pdc1敲除框构建的基础上,从pdc1基因第二拷贝的敲除框出发,对质粒Ts-PDC1n-gda-URA3-PDC1n进行Pst I单酶切,对酶切产物进行纯化;基于热带假丝酵母ATCC20336的基因组测序结果,找到2-had基因,以ATCC20336的基因组为模板,利用引物2HAD-F1,2HAD-R1对其进行PCR扩增,对PCR产物进行纯化,将纯化产物与质粒Ts-PGAPDH-GFP-TGAPDH同时进行Hind Ⅲ,Sal Ⅰ双酶切后进行连接,得到质粒Ts-PGAPDH-2HAD-TGAPDH。利用引物P-2HAD-T-f1(1n),P-2HAD-T-r1(1-2n)对质粒Ts-PGAPDH-2HAD-TGAPDH进行分子克隆,下面以pdc1内侧基因pdc1n为整合位点举例,与质粒Ts-PDC1n-gda-URA3-PDC1n的PstⅠ单酶切产物进行一步克隆连接,构建得到质粒Ts-PDC1n-PGAPDH-2HAD-TGAPDH-gda324-URA3-PDC1n,如图1-b利用MluⅠ单酶切,获得PDC1n-PGAPDH-2HAD-TGAPDH-gda324-URA3-PDC1n线性化片段,对所得片段进行纯化。根据文献[16]报道,选取其中来自大肠杆菌的d-ldh基因,并参考热带假丝酵母密码子优化表,由苏州金唯智生物科技有限公司合成基因,D-乳酸脱氢酶基因的整合框构建参考上述2-had的整合框构建。
1.2.3 酵母的氯化锂转化
酵母的氯化锂转化法参考文献[19],以尿嘧啶缺陷型菌株CU-206为出发菌株,在SM固体平板上划线,30 ℃培养2~3 d,挑取单菌落接种于20 mL SM培养基, 在30 ℃ 培养1.5~2 d,OD600值到8~12,转接适量菌液于50 mL SM培养基中,使其初始OD600值为0.1,培养10~12 h,OD600值到1左右时离心(5 000 r/min,5 min)。TE溶液(10 mmol/L Tris-HCl,pH 7.4,1 mmol/L EDTA)清洗1次,瞬时离心,重悬在装有1 mL 100 mmol/L氯化锂保藏管中,30 ℃,200 r/min摇床放置1 h;将鲑鱼精用PCR仪99 ℃加热6 min,加热完毕后迅速置于冰盒中;DNA纯化片段10 μL、细胞88 μL、鲑鱼精2 μL,混匀。置于30 ℃温度下孵育30 min,每隔15 min颠倒1次;加入900 μL 400 g/L PEG3350,含100 mmol/L氯化锂。30 ℃,200 r/min摇床放置1 h;42 ℃热激5 min,快速冷却至室温,插入冰中5 min,瞬时离心后水洗,加100~200 μL无菌水重悬细胞,涂布MM平板,在30 ℃培养箱中培养3 d。
1.2.4 酵母转化子的筛选与URA3的重复使用
挑取上述MM平板上生长的转化子,在新的MM平板上划线,刮取适量菌体提取基因组,利用整合基因位点外侧和内侧的引物进行PCR验证;再将PCR验证正确的转化子涂布到SM+5-FOA平板上,对筛选标记基因URA3弹出,利用PCR初步验证弹出URA3基因后,将PCR产物送公司测序,经序列比对正确后保藏正确菌株,在此基础上重复利用URA3[20],对第二拷贝基因敲除,以及后续基因的整合。以外源d-ldh基因的整合为例,其构建流程见图2。

图2 d-ldh基因整合热带假丝酵母流程图
Fig.2 Gene integration of d-ldh in C.tropicalis
1.2.5 基因表达水平的测定
将菌株ATCC20336、AK04、AK06在YPD平板上划线,30 ℃培养2~3 d,接单菌落到10 mL YPD培养基中30 ℃,200 r/min培养1 d,转接适量菌液到50 mL YPD培养基中,相同的条件下培养7~10 h,待OD600值达到1.5~2.5时,用Yeast RNAiso Kit试剂盒提取各菌株RNA,后使用PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒进行逆转录,再以得到的cDNA为模板,ACT为内参基因,使用ChamQTM Universal SYBR® qPCR Master Mix试剂盒配制体系,应用CFX96Real-Time PCR Detection System扩增仪对目的基因进行qPCR检测,引物如表2所示。
1.2.6 生长曲线与摇瓶发酵
生长曲线:将ATCC20336、AK01、AK02、AK03、AK04、AK05、AK06和AK07分别在MM平板上划线,挑取单菌落转接到20 mL YPD培养基中,在30 ℃,200 r/min培养1.5~2 d;取适量菌液转接到50 mL YPD培养基中,控制它们的初始OD600值为0.1左右,每株菌取3个平行,每隔12 h取1次样,测定OD600值。
摇瓶发酵:将各菌株在YPD平板划线,挑取单菌落转接到20 mL YPD培养基中,200 r/min,30 ℃培养1.5 d;按1 %的接种量转接到50 mL YP-5%D培养基中,每株菌取3个平行,每隔12 h取1次样,测定残糖和pH值,发酵过程中未加入任何中和剂。
1.2.7 乳酸及其副产物的测定
对发酵液样品进行离心[21],12 000 r/min,5 min保留上清液,并稀释相应倍数,用0.22 μm孔径的微孔滤膜过滤后加入到液相样品瓶中;HPLC中使用的是Bio-Rad Aminex HPX-87H色谱柱(300 mm×7.8 mm),保护柱是Bio-rad Carbo-H, 流动相为5 mmol/L H2SO4,流速为0.5 mL/min,柱温为55 ℃,检测器是RID-10A示差折光检测器。
2 结果与分析
2.1 丙酮酸脱羧酶基因的敲除
构建了质粒Ts-PDC1u-gda324-URA3-PDC1d,用Xba I,Pst I双酶切,获得大小为3.4 kbp和1.9 kbp的片段(图3-a);后将该质粒用Mlu I酶切纯化,将线性化片段向出发菌CU-206中转化,获得转化子,提取基因组后用敲除位点外侧引物PDC1wcF1,PDC1wcR1进行PCR验证分别得到2.2 kbp和3.2 kbp的条带(图3-b);将上述正确的转化子涂布到SM+5-FOA平板上,对URA3进行弹出,用PDC1wcF1,PDC1wcR1引物进行PCR验证分别得到1.6 kbp和2.2 kbp的条带;对正确弹出URA3的转化子,向其转化第二拷贝敲除盒PDC1n-gda324-URA3-PDC1n,获得转化子,提取基因组用敲除位点外侧和内侧引物PDC1wcF1,PDC1n-R1进行PCR验证,获得3.4 kbp的条带(图3-c)。以同样的方式构建pdc2基因敲除菌株,电泳验证图此处省略。经测序结果显示成功构建了pdc1基因敲除菌株AK01、pdc2基因敲除菌株AK02和pdc1/pdc2基因敲除菌株AK03。
2.2 内源2-羟基酸脱氢酶与异源D-乳酸脱氢酶的过表达
以ATCC20336的基因组为模板,利用引物2-HAD-F1,2-HAD-R1对其进行PCR扩增,得到1 017 bp的条带;以pdc1内侧基因为整合位点为例,将构建好的质粒Ts-PDC1n-P-2HAD-T-gda-URA3-PDC1n用Mlu I单酶切,分别得到5.4 kpp和2.7 kbp的条带,对酶切产物进行纯化后转化到pdc1单拷贝敲除弹出URA3的菌株中,获得转化子,提取基因组,用整合位点外侧和内侧的引物PDC1wcF1,2HAD-R1,进行PCR验证(图3-d)得到3.7 kbp的条带,对照组中无条带,后送测序,结果显示成功构建并获得pdc1基因敲除且过表达2-had基因的菌株AK04和pdc1/pdc2基因敲除且过表达2-had基因的菌株AK06。
以同样的方法成功构建pdc1基因敲除且过表达异源d-ldh的菌株AK05和pdc1/pdc2基因敲除且过表达d-ldh的菌株AK07,电泳验证图此处省略。

M-DL 5 000 marker
a-1-质粒Ts-PDC1u-gda324-URA3-PDC1d酶切验证;
b-1-ATCC20336对照;b-2-pdc1单拷贝敲除转化子;
c-1-pdc1单拷贝敲除弹出URA3对照;c-2~7-pdc1双拷贝敲除转化子;d-1-pdc1单拷贝敲除弹出URA3对照;
d-2-pdc1双拷贝敲除整合2HAD转化子
图3 pdc1基因敲除与2-had基因整合菌株的PCR验证
Fig.3 PCR identification of pdc1 Gene disruption and 2-had Gene integration strains and plasmid
2.3 基因表达水平的分析
内源2-had过表达菌株AK04和AK06与野生型菌株ATCC20336的mRNA相对丰度如图4所示,AK06的mRNA表达量是野生型菌株的4.85倍,而AK04的mRNA表达量只有野生型的1/10,这一点与AK04的乳酸产量低于野生型菌株相一致。推测其原因可能是pdc1同工酶基因是pdc的关键酶基因,2-had在pdc1基因位点的整合阻碍了2-had的过表达,而在pdc2基因位点的整合能够过表达。
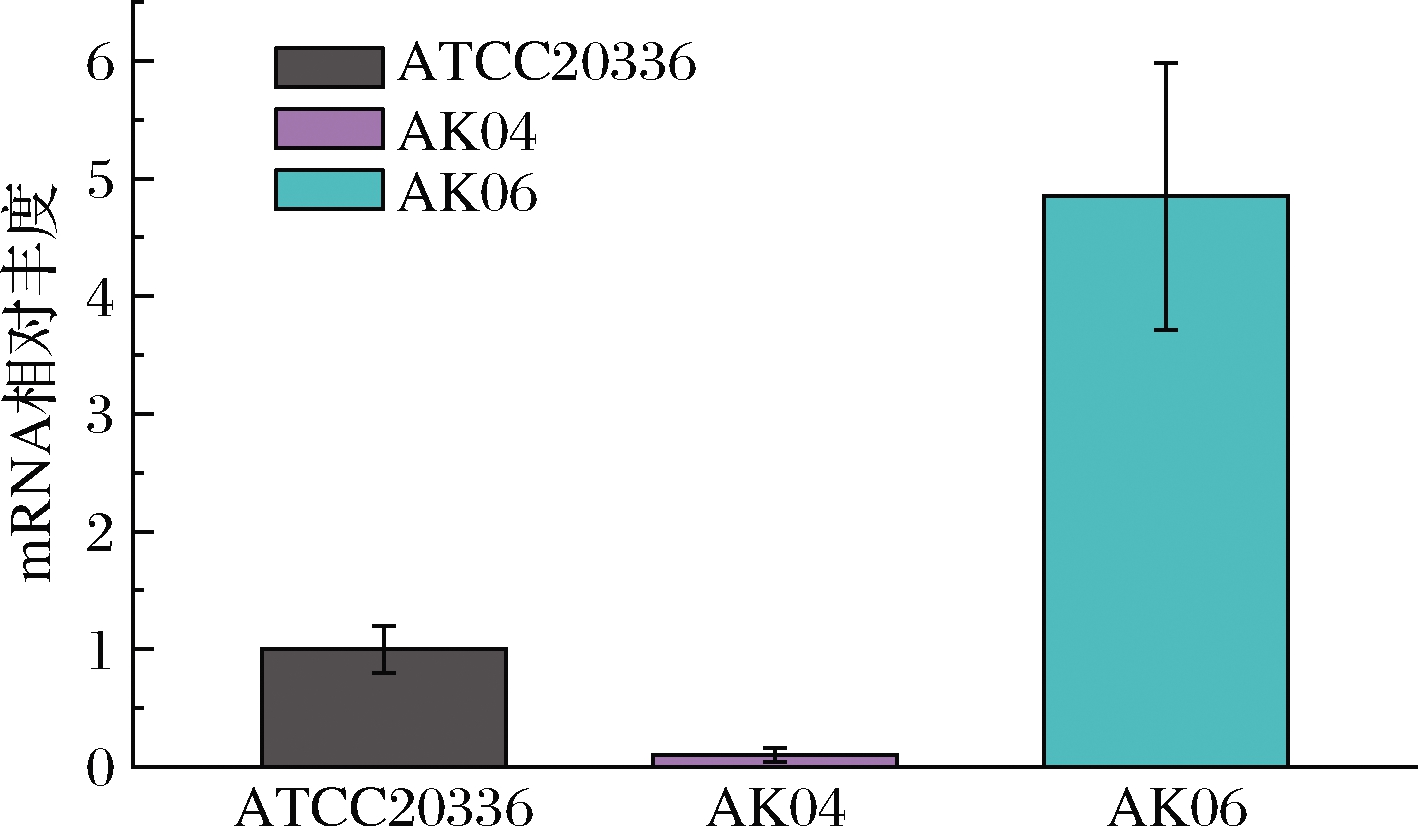
图4 不同菌株2-had基因的表达水平
Fig.4 Gene expression levels of 2-had in different strains
2.4 不同重组菌株的生长情况
野生型菌株ATCC20336与其他7株基因改造菌株在250 mL摇瓶,装液量为50 mL的YPD中30 ℃培养108 h的生长情况如图5所示。相较于野生型菌株ATCC20336和pdc2等位基因敲除菌株AK02,pdc1等位基因敲除的菌株AK01和AK03出现了一定的生长抑制,这一点也可以从各菌株在YP-5%D发酵过程中的残糖消耗得以看出,如图6所示。由此推测丙酮脱酸酶基因pdc的2个同工酶PDC1和PDC2中,pdc1等位基因的敲除对菌株的生长情况影响较大。相较于表达内源2-had的菌株AK04和AK06,表达异源LDH的菌株AK05和AK07也出现了不同程度的生长抑制,推测异源蛋白LDH的表达对菌体产生一定毒性,抑制了菌体的生长。
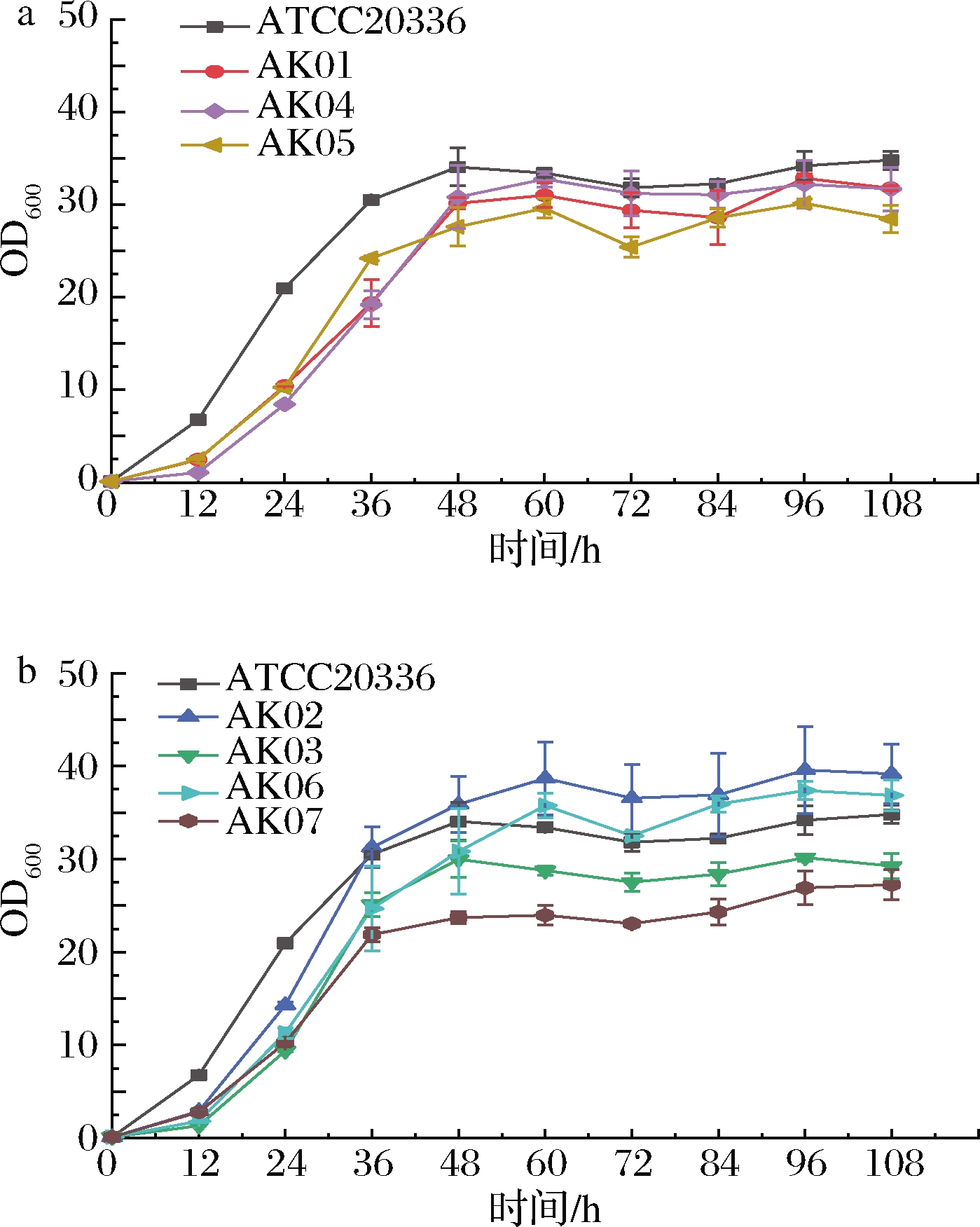
a-ATCC20336与AK01、AK04和AK05的生长情况;
b-ATCC20336与AK02、AK03、AK06和AK07的生长情况
图5 不同菌株的生长曲线
Fig.5 Growth curves of different strains

a-葡萄糖含量;b-pH值;c-甘油含量;d-丙酮酸含量;e-乙醇含量
图6 不同菌株发酵时pH与副产物的变化情况
Fig.6 Variation of pH and by-products during the fermentation of different strains
2.5 重组菌株的乳酸及其副产物的检测
取不同发酵时间的样品,测定pH以及预处理后进行高效液相色谱的检测。残糖和副产物乙醇、丙酮酸、甘油的生产情况如图6所示。图6-a显示了各菌株对于葡萄糖的消耗情况,这也与2.4(图5)中的生长曲线相关联。图6-b反映了各菌株发酵过程中pH的变化情况,野生型菌株ATCC20336的pH一直稳定在6附近,基因重组菌株的pH基本都呈现出先下降后上升的趋势,pH的下降与丙酮酸,乳酸等有机酸的产生有关;在菌体生长平衡期由于菌体的衰亡以及碳源的逐渐耗尽细胞被迫利用有机酸,导致pH有所上升。同时LDH的酶活性也受到pH的影响[22],这也说明后续的发酵优化中对于pH的控制也显得极为重要。图6-c反映了副产物甘油的生产情况,由热带假丝酵母利用葡萄糖生产乳酸的参考代谢路径可知,碳流有相当部分进入甘油生产路径,从而降低了目的产物乳酸的产量,其中AK07在96 h时甘油的产量达到10.85 g/L。图6-d表现出各菌株中间代谢产物丙酮酸的生产情况,其中AK04在72 h时的产量为11.68 g/L。由图6-e各菌株乙醇的生产情况可知,野生型菌株的乙醇产量最大为17.17 g/L,AK01、AK04和AK05基本不产乙醇,可以发现pdc1等位基因的敲除可以显著降低乙醇产量。
不同菌株的乳酸最大产量如图7所示。值得注意的是,作为pdc1和pdc2两同工酶等位基因同时敲除且表达异源LDH的菌株AK07在发酵过程中始终未检测到乳酸的产生,通过提取该菌株的RNA,经逆转录成cDNA,后进行PCR验证发现,异源ldh基因几乎不表达。其中野生型菌株ATCC20336的乳酸产量约为0.21 g/L,AK06在48 h时的产量为0.45 g/L,较野生型菌株的产量提升1倍。AK05在72 h时最大产量为6.10 g/L,是野生型菌株产量的29倍。
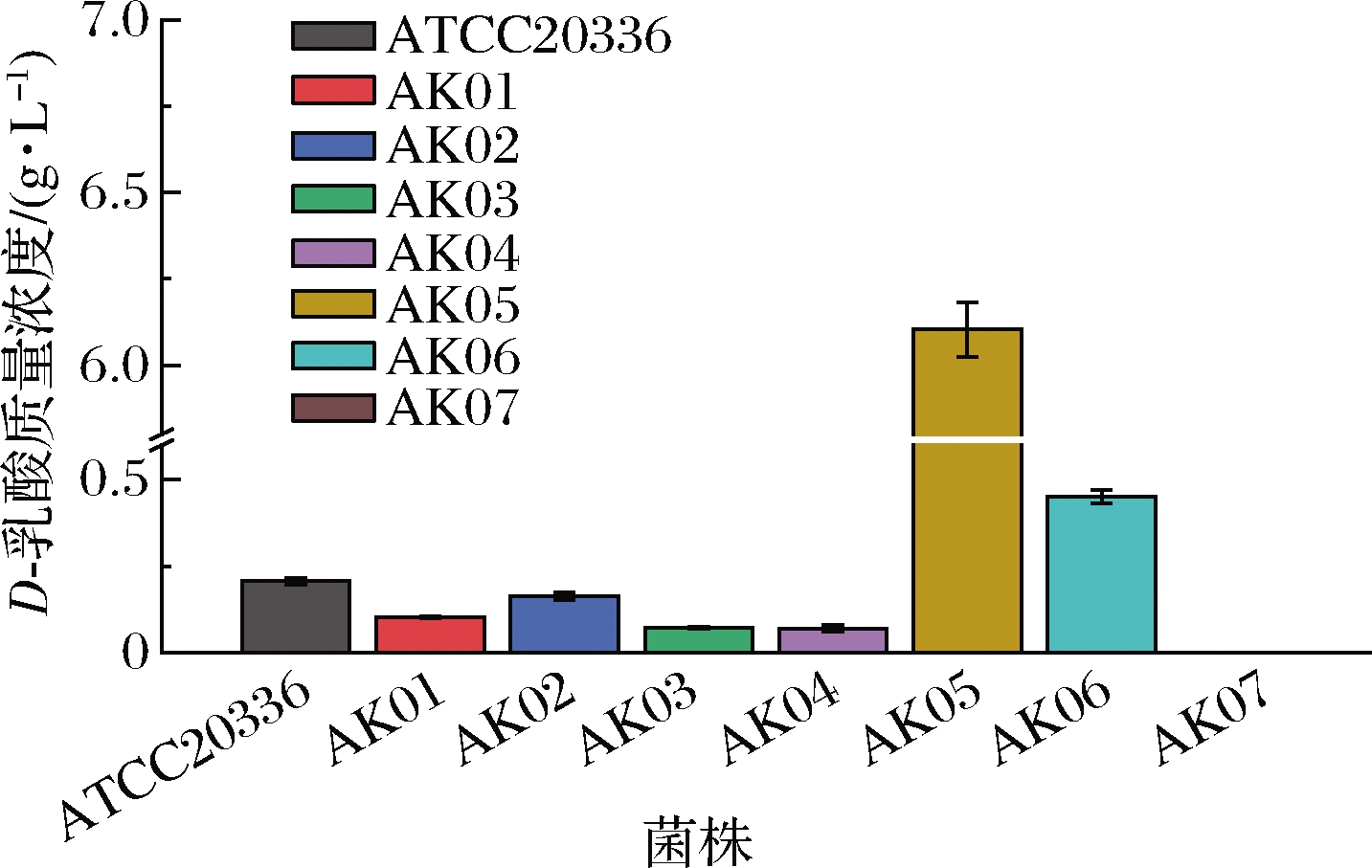
图7 不同菌株的D-乳酸产量
Fig.7 Production of D-LA by different strains
3 结论与讨论
本研究从热带假丝酵母生产乳酸的代谢路径出发,探究了基因工程改造对其乳酸积累的影响。通过对丙酮酸脱羧酶PDC的同工酶基因pdc1,pdc2单独敲除和复合敲除,研究了基因敲除对于菌株生长情况和产物生产性能的作用,发现pdc1基因的敲除能够显著地抑制菌株的生长以及降低菌株乙醇的产量,而pdc2的敲除对于菌株的影响较小;通过过表达热带假丝酵母内源性的2-羟基酸脱氢酶基因2-had,旨在促进丙酮酸向乳酸的转化,结果发现2-had的过表达未对菌株的乳酸产量产生明显的影响;对菌株整合表达来自大肠杆菌且经过密码子优化的D-乳酸脱氢酶基因d-ldh,结果表明d-ldh整合表达菌株AK05的乳酸产量是野生型菌株ATCC20336的29倍,体现出较好的乳酸生产性能,而另一d-ldh整合表达菌株AK07未表现出预期的乳酸生产性能,在其发酵液中未能检测到乳酸。目前也面临着重组菌株在发酵过程中的pH维持较低的水平,不利于乳酸的积累;乳酸生产菌株的中间代谢产物丙酮酸和甘油的产量与出发菌株相比高出许多等问题。
本研究对热带假丝酵母进行了一系列基因的敲除与整合,获得了一株D-乳酸产量最高为6.10 g/L的菌株,为后续进一步代谢改造热带假丝酵母提高D-乳酸产量提供参考。
[1] LIAUD N, ROSSO M N, FABRE N, et al.L-lactic acid production by Aspergillus brasiliensis overexpressing the heterologous ldhA gene from Rhizopus oryzae[J].Microbial Cell Factories, 2015, 14(1):66-75.
[2] RYU A J, KIM T Y, YANG D S, et al.Engineering of Saccharomyces cerevisiae for enhanced production of L-lactic acid by co-expression of acid-stable glycolytic enzymes from Picrophilus torridus[J].Korean Journal of Chemical Engineering, 2018, 35(8):1 673-1 679.
[3] PARK H J, BAE J H, KO H J, et al.Low-pH production of D-lactic acid using newly isolated acid tolerant yeast Pichia kudriavzevii NG7[J].Biotechnology and Bioengineering, 2018, 115(9):2 232-2 242.
[4] WANG Y, TASHIRO Y, SONOMOTO K.Fermentative production of lactic acid from renewable materials:Recent achievements, prospects, and limits[J].Journal of Bioscience & Bioengineering, 2015, 119(1):10-18.
[5] 黄国昌, 熊大维, 张婷, 等.发酵法生产乳酸的研究进展[J].江西科学,2014, 32(5):660-666.
HUANG G C, XIONG D W, ZHANG T, et al.Advances in fermentation methods for lactic acid production[J].Jiangxi Science, 2014, 32(5):660-666.
[6] 钟伟. 酿酒酵母产D-乳酸的代谢工程研究[D].北京:中国科学院大学(中国科学院过程工程研究所),2019.
ZHONG W.Metabolic engineering of Saccharomyces cerevisiae for D-lactic acid production[D].Beijing:University of Chinese Academy of Sciences,2019.
[7] NAKANO S, UGWU C U, TOKIWA Y.Efficient production of D-(-)-lactic acid from broken rice by Lactobacillus delbrueckii using Ca(OH)2 as a neutralizing agent[J].Bioresource Technology, 2012, 104(1):791-794.
[8] BAEK S H, KWON E Y, BAE S J, et al.Improvement of D-lactic acid production in Saccharomyces cerevisiae under acidic conditions by evolutionary and rational metabolic engineering[J].Biotechnology Journal, 2017,12(10):1700015.
[9] ILMÉN M, KOIVURANTA K, RUOHONEN L, et al.Production of L-lactic acid by the yeast Candida sonorensis expressing heterologous bacterial and fungal lactate dehydrogenases[J].Microbial Cell Factories, 2013, 12(1):53.
[10] LEE J W, IN J H, PARK J B, et al.Co-expression of two heterologous lactate dehydrogenases genes in Kluyveromyces marxianus for L-lactic acid production[J].Journal of Biotechnology, 2017, 241(1):81-86.
[11] HOU Q, HE Q, LIU G, et al.Identification and application of novel low pH-inducible promoters for lactic acid production in the tolerant yeast Candida glycerinogenes[J].Journal of Bioscience and Bioengineering, 2019, 128(1):8-12.
[12] KOIVURANTA K T, ILMÉN M, WIEBE M G, et al.L-lactic acid production from D-xylose with Candida sonorensis expressing a heterologous lactate dehydrogenase encoding gene[J].Microbial Cell Factories, 2014, 13(1):1-14.
[13] ZHANG L H, CHEN X Z, CHEN Z, et al.Development of an efficient genetic manipulation strategy for sequential gene disruption and expression of different heterologous GFP genes in Candida tropicalis[J].Applied Microbiology and Biotechnology, 2016, 100(22):9 567-9 580.
[14] 张志禹, 朱涛, 林荣胜,等.热带假丝酵母的二元酸分泌现象及其动力学[J].清华大学学报, 1999, 39(12):27-30.
ZHANG Z Y, ZHU T, LIN R S, et al.Kinetics of secretion and acid production by Candida tropicalis cells in dicarbonxylic acid fermentation system[J].Journal of Tsinghua University, 1999, 39(12):27-30.
[15] 刘树臣, 焦鹏, 曹竹安.热带假丝酵母代谢烷烃过程中的β-氧化和代谢调控[J].微生物学报, 2002, 42(1):125-128.
LIU S C, JIAO P, CAO Z A.Metabolism and β-oxidation of alkane-utilizing Candida tropicalis[J].Acta Microbiologica Sinica, 2002, 42(1):125-128.
[16] ZHONG W, YANG M H, MU T Z, et al.Systematically redesigning and optimizing the expression of D-lactate dehydrogenase efficiently produces high-optical-purity D-lactic acid in Saccharomyces cerevisiae[J].Biochemical Engineering Journal, 2019, 144(1):217-226.
[17] BONETE M J, FERRER J, PIRE C, et al.2-Hydroxyacid dehydrogenase from Haloferax mediterranei, a D-isomer-specific member of the 2-hydroxyacid dehydrogenase family[J].Biochimie, 2000, 82(12):1 143-1 150.
[18] BAEK S H, KWON E Y, KIM Y H, et al.Metabolic engineering and adaptive evolution for efficient production of D-lactic acid in Saccharomyces cerevisiae[J].Applied Microbiology & Biotechnology, 2016, 100(6):2 737-2 748.
[19] RODR GUEZ L, CH
GUEZ L, CH VEZ F P, BASABE L, et al.Development of an integrative DNA transformation system for the yeast Candida utilis[J].FEMS Microbiology Letters, 1998, 165(2):335-340.
VEZ F P, BASABE L, et al.Development of an integrative DNA transformation system for the yeast Candida utilis[J].FEMS Microbiology Letters, 1998, 165(2):335-340.
[20] 项峥, 陈献忠, 张利华,等.利用可重复使用的URA3标记基因建立热带假丝酵母基因敲除系统[J].遗传, 2014, 36(10):1 053-1 061.
XIANG Z, CHEN X Z, ZHANG L H, et al.Development of a genetic transformation system for Candida tropicalis based on a reusable selection marker of URA3 gene[J].Hereditas, 2014, 36(10):1 053-1 061.
[21] 马瑞, 欧阳嘉, 李鑫, 等.高效液相色谱法同时测定生物质乳酸发酵液中有机酸及糖类化合物[J].色谱,2012, 30(1):62-66.
MA R,OUYANG J, LI X, et al.Simultaneous determination of organic acids and saccharides in lactic acid fermentation broth from biomass using high performance liquid chromatography[J].Chinese Journal of Chromatography, 2012,30(1):62-66.
[22] KONG X, ZHANG B, HUA Y, et al.Efficient L-lactic acid production from corncob residue using metabolically engineered thermo-tolerant yeast[J].Bioresource Technology, 2019, 273(2):220-230.