枯草芽孢杆菌被美国食品药品监督管理局认证为食品安全菌株(generally recognized as safe,GRAS),其具有生长周期短、分泌能力强、遗传背景清晰等优点,因此枯草芽孢杆菌作为底盘细胞被广泛应用于发酵工程领域,例如透明质酸[1]、N-乙酰氨基葡萄糖[2]、核黄素[3]、乙偶姻[4-5]、2,3-丁二醇[6]等代谢产品的合成以及蛋白酶[7]、普鲁兰酶[8]、β-环糊精糖基转移酶[9]等工业酶制剂的制造。
与发酵工程领域中使用最广泛的原核生物宿主大肠杆菌和真核生物宿主酿酒酵母相比,枯草芽孢杆菌由于缺乏基因调控元件在生物技术领域受到限制。目前对基因的表达进行调控最常用的策略是在转录水平上进行的,如最常见的启动子、核糖体结合位点(ribosomal binding site,RBS)替换等[10-11]。转录后水平调控是枯草芽孢杆菌自身控制一些基因表达的重要方式,然而目前在枯草芽孢杆菌中转录后水平的调控工具开发却非常少。此前,YANG等[12]基于I型毒素-抗毒素系统bsrG/SR4成功开发了1套转录后调控系统Modulation via the small RNA (sRNA)-dependent operation system(MS-DOS)。在枯草芽孢杆菌I型毒素-抗毒素系统中,毒素基因往往编码1个稳定的有毒短肽,而抗毒素基因在其反义链上编码1个相应的非编码小RNA(sRNA),通过影响翻译和/或降低毒素mRNA稳定性解除毒性。其中bsrE/SR4系统具有双功能,抗毒素小RNA SR4与毒素bsrE mRNA结合,一方面能改变bsrE mRNA的二级结构从而降低毒素蛋白的翻译水平;另一方面两者形成的sRNA-mRNA复合物被RNase Ⅲ切割,稳定性下降从而阻止毒素蛋白的过量表达。将抗毒素SR4与毒素bsrE mRNA配对的区域作为操作区(operation region,OPR)插入到靶基因的终止密码子之后,然后利用质粒过表达SR4。这样SR4就能识别带有OPR的靶基因mRNA链并与之结合形成复合物,然后被RNase Ⅲ识别并切割。通过MS-DOS系统可以轻松的实现单个或多个基因的抑制。相比CRISPR-i,MS-DOS系统不易脱靶,且调控元件是非编码小RNA,相比额外表达dCas9蛋白代谢压力更小[13];同时在构建的MS-DOS系统中,OPR序列插入在目标基因的终止密码子后面,而不会像降解标签ssrA一样,因为额外增加的氨基酸影响蛋白结构[14]。
在工程菌株构建过程中,只要在需进行调控的目的基因后加上OPR4序列,即可利用MS-DOS系统实现单个或多个基因的调控,但是所有加上OPR4序列的基因只能被同时同步调控。在实际生产应用中,在不同阶段往往需要对不同的基因进行不同程度的表达调控,为了实现对多个基因更为精细的调控,更多类似的MS-DOS系统需要被开发。bsrE/SR5系统与bsrG/SR4一样属于枯草芽孢杆菌I型毒素抗毒素系统,能够通过降低mRNA稳定性的方式解除毒性,拥有改造成为MS-DOS系统的潜力。本研究成功构建了以bsrE/SR5为基础的MS-DOS调控系统。通过与SR4的对比,我们对SR5从5′端到3′端进行了截短,确认SR5在仅保留终止子序列的情况下也能保持较高的活性。截短后的SR5不仅进一步减小了菌体的代谢压力,同时因为片段较小(仅37 nt),能够直接设计在引物上,为应用过程中的质粒构建提供了便利。
1 材料与方法
1.1 实验材料
1.1.1 菌种与质粒
本研究所用菌种和质粒信息见表1。
表1 本研究所用菌株和质粒
Table 1 Strains and plasmids used in this study
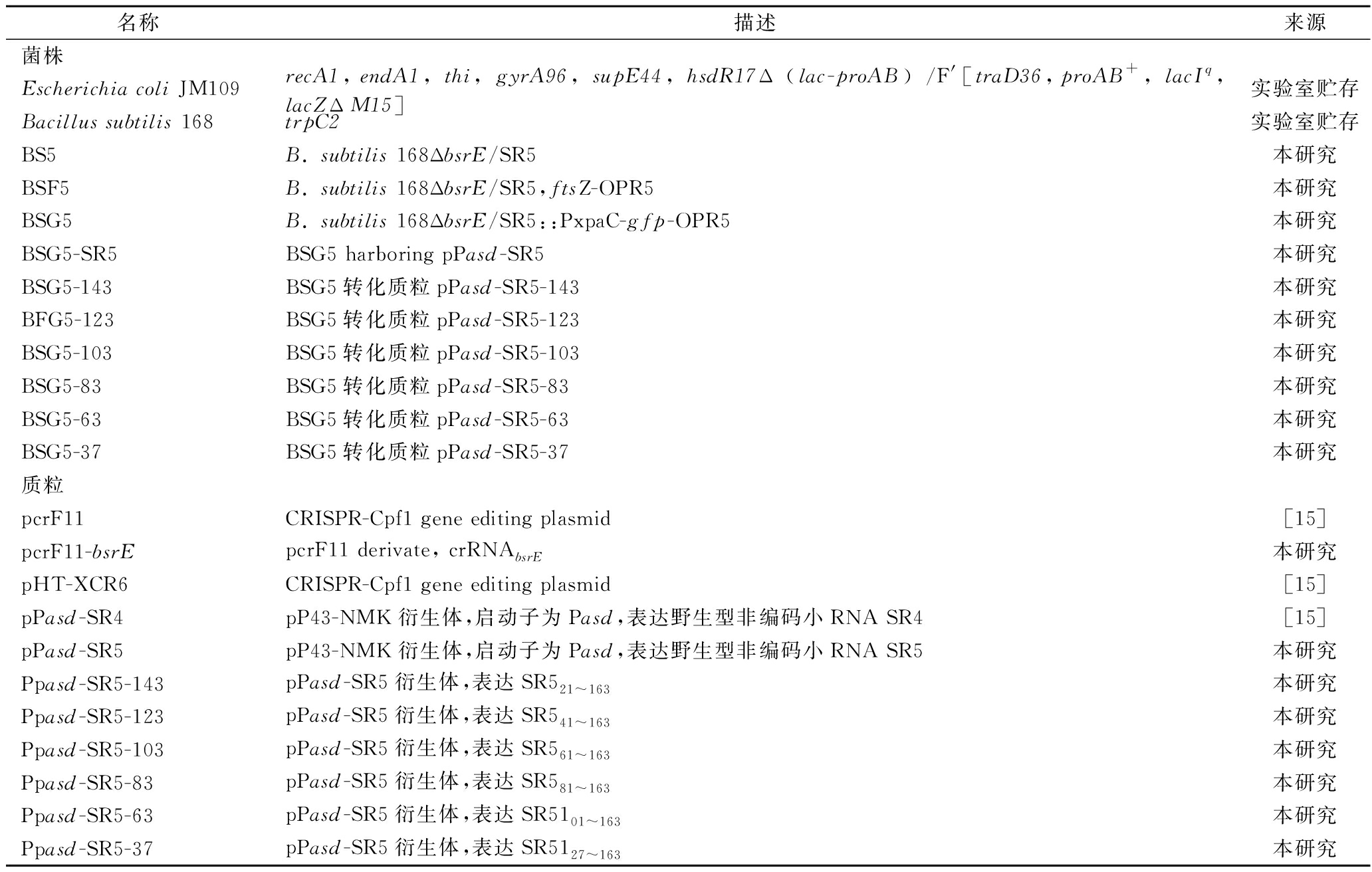
名称描述来源菌株Escherichia coli JM109recA1, endA1, thi, gyrA96, supE44, hsdR17Δ (lac-proAB) /F'[traD36,proAB+, lacIq, lacZΔ M15]实验室贮存Bacillus subtilis 168trpC2实验室贮存BS5B. subtilis 168ΔbsrE/SR5本研究BSF5B. subtilis 168ΔbsrE/SR5,ftsZ-OPR5本研究BSG5B. subtilis 168ΔbsrE/SR5::PxpaC-gfp-OPR5本研究BSG5-SR5BSG5 harboring pPasd-SR5本研究BSG5-143BSG5转化质粒pPasd-SR5-143本研究BFG5-123BSG5转化质粒pPasd-SR5-123本研究BSG5-103BSG5转化质粒pPasd-SR5-103本研究BSG5-83BSG5转化质粒pPasd-SR5-83本研究BSG5-63BSG5转化质粒pPasd-SR5-63本研究BSG5-37BSG5转化质粒pPasd-SR5-37本研究质粒pcrF11CRISPR-Cpf1 gene editing plasmid[15]pcrF11-bsrEpcrF11 derivate, crRNAbsrE本研究pHT-XCR6CRISPR-Cpf1 gene editing plasmid[15]pPasd-SR4pP43-NMK衍生体,启动子为Pasd,表达野生型非编码小RNA SR4[15]pPasd-SR5pP43-NMK衍生体,启动子为Pasd,表达野生型非编码小RNA SR5本研究Ppasd-SR5-143pPasd-SR5衍生体,表达SR521~163本研究Ppasd-SR5-123pPasd-SR5衍生体,表达SR541~163本研究Ppasd-SR5-103pPasd-SR5衍生体,表达SR561~163本研究Ppasd-SR5-83pPasd-SR5衍生体,表达SR581~163本研究Ppasd-SR5-63pPasd-SR5衍生体,表达SR5101~163本研究Ppasd-SR5-37pPasd-SR5衍生体,表达SR5127~163本研究
1.1.2 引物信息
本研究所用引物见表2。
表2 本研究所用引物
Table 2 Primers used in this study.

引物名称引物序列(5'→ 3')G5-F1CAGACCCGTAGGGACCAGTTG5-R1TTTAACTGTTTCGTCCATCTTCTCCACCTGACAACGTGAAAGGAACTTGCG5-F2TGCATTTTGCAAGTTCCTTTCACGTTGTCAGGTGGAGAAGATGGACGAAAG5-R2GTGTACATTCCTCTCTTCTCAGTTTTAATATTATTATCTACTACGTTCAATCCG5-F3ATATTAAAACTGAGAAGAGAGGAATGTACACATGGGTAAGGGAGAAGAACTTTTCAG5-R3CTGAGGCTCAAGGGAGGTCTTATTTGTATAGTTCATCCATGCCATGTGTG5-F4ATGGATGAACTATACAAATAAGACCTCCCTTGAGCCTCAGAACG5-R4CCCCGGGTACACCGAGGTGCTACCAACACCG5-F5TAGCACCTCGGTGTACCCGGGGATCCTCTAGAGATACG5-R5ATGTGTTATAATCAGGCATGCCTGCAGGTCGACGATTCG5-F6ACCTGCAGGCATGCCTGATTATAACACATTTTCCGTGAAATGCG5-R6TGGAATTGGATACAACAAAATGGTF5-F1CGCGAAAGACTTAGGCGCATTAF5-R1GCTCAAGGGAGGTCTTAGCCGCGTTTATTACGGTTTCTF5-F2TAAACGCGGCTAAGACCTCCCTTGAGCCTCAGAACF5-R2GATCCCCGGGTACACCGAGGTGCTACCAACACCF5-F3TAGCACCTCGGTGTACCCGGGGATCCTCTAGAGATACF5-R3CGACCTGCAGGCATGCTGTAAAGGACAAAATCGTTTTCGF5-F4TGTCCTTTACAGCATGCCTGCAGGTCGACGATTCF5-R4CAGATCTTTAAATTTAATCAGAAAAGTCGTTcrbsrE-FAGATTAATCGCCCTATTGACGTATATAcrbsrE-RAATTTATATACGTCAATAGGGCGATTAEud-F1AAACTATATTCCCTGCTGTATTGTTTTTGAAAGEud-R1GTCAATATATTGTGAATTTTTATTATACATCTTCATCATTTCTTACTCTTCTAGCTCEud-F2AGAAATGATGAAGATGTATAATAAAAATTCACAATATATTGACAACGTGAAAGGEud-R2TTGGAGTTATTAGATATTTGGAATTGGATACAACSR5-FCCGCTCGAGCAATACATTACAGTGCATTTCTSR5-RGGCTCAAGGGAGGTCCTGCAGAAGCTTGGGSR5-TRCTCGAGGATTTGACTATGATTTTACGTTTTASR5-143FCTTTAATGAGAAATGCATAAAATAAAAAAGACCGSR5-123FAATAAAAAAGACCGAGGTGCTACCAACSR5-103FTACCAACACCCCGGCTCTGTACSR5-83FACAAAAAGTTGCCCATCAAAGGGSR5-63FGGGCTTGCTCGATGTGGTTCSR5-37FGGCCTATCCCTTCAGGTTCTGAG
1.1.3 主要试剂
PCR引物合成和测序,苏州金唯智生物科技有限公司;2×Taq DNA聚合酶,宝赛生物(杭州)有限公司;胶回收试剂盒,Thermo Scientific公司;PrimeSTAR HS DNA聚合酶、DNA marker、QuickCut限制酶、Blunting Kination Ligation磷酸化连接试剂盒,大连宝生物TaKaRa公司;One Step Cloning Kit一步克隆试剂盒,南京诺唯赞生物科技有限公司;质粒提取试剂盒、卡那霉素、氨苄霉素、博来霉素,生工生物工程(上海)有限公司。
1.2 实验方法
1.2.1 质粒构建与转化
质粒pPasd-SR5及其衍生质粒的构建:以枯草芽孢杆菌B.subtilis 168基因组为模板,使用引物SR5-F/SR5-R获得SR5片段,然后使用一步克隆试剂盒并将得到的片段插入pPasd-SR4质粒的Xho I和Pst I酶切位点之间。截短的SR5由相应的引物进行PCR扩增,并通过磷酸化连接试剂盒连接得到。
质粒pcrF11-bsrE的构建:通过网站http://bioinfolab.miamioh.edu/CRISPR-DT/interface/Cpf1_design.php选择合适的crRNA,设计带有重叠区的1对引物,经过变性退火形成带有黏性末端的引物二聚体,与EcoR31 I酶切后的线性化载体pcrF11连接8 h,得到质粒pcrF11-bsrE。
1.2.2 基因组整合表达的菌株构建
1.2.2.1 利用CRISPR-Cpf1基因组编辑系统敲除bsrE/SR5
构建包含以下2个模块的重组DNA片段bsrEud:bsrE上游1 000 bp(以B.subtilis 168基因组为模板,由引物Eud-F1/R1扩增)和SR5下游1 000 bp(以B.subtilis 168基因组为模板,由引物Eud-F2/R2扩增)通过体外融合PCR连接。在枯草芽孢杆菌B.subtilis 168 中转化质粒pHT-xcR6。再将含有这个质粒的菌株制成感受态,转化质粒pcrF11-bsrE和整合片段bsrEud,在含有氯霉素、卡那霉素和30 g/L木糖的LB培养基中过夜培养,涂布含有氯霉素、卡那霉素和30 g/L木糖的LB平板。挑选单菌落进行菌落PCR验证。挑选测序正确的转化子接种至添加有0.05 g/L SDS的LB培养基中,37 ℃振荡培养后划线于LB平板,通过菌落PCR验证pcrF11-bsrE、pHT-cxR6双质粒丢失的菌株即为菌株BS5。
1.2.2.2 菌株BSF5的构建
枯草芽孢杆菌BSF5是在BS5的基础上于基因ftsZ开放阅读框后紧跟着插入OPR5而产生的。通过融合PCR技术构建包含以下4个模块的重组DNA片段:ftsZ终止密码子上游的500 bp同源臂(以B.subtilis 168基因组为模板,由引物F5-F1/F5-R1扩增),OPR(以B.subtilis 168基因组为模板,由引物F5-F2/F5-R2扩增),lox71-zeo-lox66博来霉素抗性基因(以B.subtilis 168基因组为模板,由引物F5-F3/F5-R3扩增)以及ftsZ终止密码子下游的500 bp DNA同源臂(以B.subtilis 168基因组为模板,由引物F5-F4/F5-R4扩增),并将其转化到BS5中。通过菌落PCR选择突变体,并通过DNA测序确认。根据YAN等[16]报道的方法,从基因组中去除博来霉素抗性基因。
1.2.2.3 菌株BSG5的构建
枯草芽孢杆菌BSG5是在B.subtilis 168的基础上将bsrE/SR5替换为PxpaC-GFP-OPR5而产生的。通过融合PCR技术构建包含以下5个模块的重组DNA片段:bsrE上游的500 bp同源臂(以B.subtilis 168基因组为模板,由引物G5-F1/G5-R1扩增),启动子PxpaC(以B.subtilis 168基因组为模板,由引物G5-F2/G5-R2扩增),绿色荧光蛋白GFP(以GFP为模板,由引物G5-F3/G5-R3扩增),OPR5(以B.subtilis 168基因组为模板,由引物G5-F4/G5-R4扩增),lox71-zeo-lox66博来霉素抗性基因(以B.subtilis 168基因组为模板,由引物G5-F5/G5-R5扩增),SR5下游的500 bp(以B.subtilis 168基因组为模板,由引物G5-F6/G5-R6扩增),并将其转化到B.subtilis 168中。通过菌落PCR选择突变体,并通过DNA测序确认。根据YAN等[16]报道的方法,从基因组中消除博来霉素抗性基因。
1.2.3 荧光强度和生物量测定
将重组菌接种至LB培养基中,培养12 h作为种子液,并以初始OD600为0.1接种至新鲜的LB培养基中;培养容器为96孔黑色板,每个孔的装液量为200 μL,设置3份平行。随后将96孔板在37 ℃下以900 r/min 的转速在孔板摇床振荡培养48 h。每隔2 h,使用多功能酶标仪直接对其进行相对荧光强度和细胞光密度测定。在激发波长为490 nm和发射波长为530 nm,增益值为60的条件下测量样品的相对荧光强度,在600 nm处测定细胞光密度。
2 结果与分析
2.1 SR4与SR5的序列比对与二级结构分析
I型毒素基因座通常编码1个小的疏水性毒性蛋白,反义链转录1个抗毒素RNA,抗毒素RNA通过降低毒素mRNA的稳定性和/或干扰毒素翻译解除毒性。枯草芽孢杆菌中存在多种I型毒素-抗毒素系统[17](图1-a),此前YANG等[12]基于双功能的bsrG/SR4构建了1套转录后调控系统MS-DOS。I型毒素抗毒素系统中,bsrG/SR4和bsrE/SR5研究最为透彻,且两者功能和原理非常相似[18-22]。SR5是一个长度为163 nt的非编码小RNA,能和bsrE mRNA配对并形成长度为128 bp的双链RNA,RNase Ⅲ切割双链RNA,从而降低bsrE mRNA 的稳定性解除毒性。SR5-bsrE mRNA双链复合物的RNase Ⅲ切割位点位于 SR5 3′端起第9个碱基,第10个碱基和第11个碱基之后(图1-b)。所以,SR5理论上的调控功能核心区域也应位于其3′端。对SR4与SR5的序列进行比对分析,发现两者在核酸序列上有着很高的相似度(图1-c)。
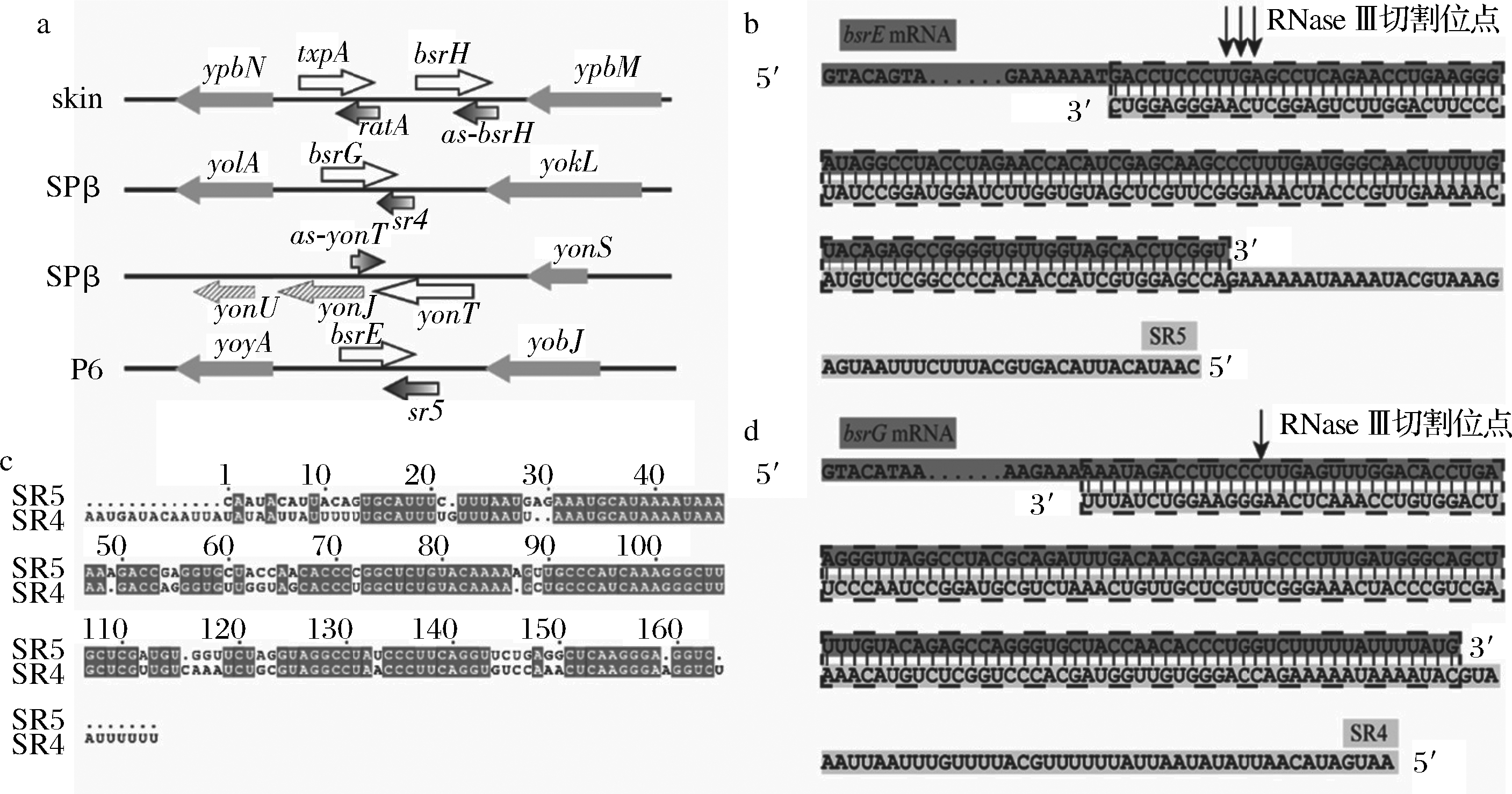
a-枯草芽孢杆菌中存在/潜在的I型毒素抗毒素系统,白色箭头表示毒素,渐变箭头表示抗毒素,条纹箭头表示潜在的毒素;b-bsrE mRNA与SR5的配对情况示意图,bsrE mRNA由深灰色底色表示,SR5由浅灰色底色表示,两者配对序列(即OPR5序列)由虚线方框表示;c-SR4与SR5核酸序列比对图;d-bsrG mRNA与SR4的配对情况示意图,bsrG mRNA由深灰色底色表示,SR5由浅灰色底色表示,两者配对序列(即OPR4序列)由虚线方框表示
图1 SR4与SR5的序列比对分析
Fig.1 Sequence alignment of SR4 and SR5
从RNA二级结构上可以看出SR4和SR5都存在1个rho-不依赖的颈环结构终止子(图2-a、图2-b)。JAHN等[18]在体外分析各种SR4截短体与bsrG mRNA的配对常数时发现,SR4的113~183部分序列对于两者结合非常重要,在SR4113~183基础上从5′~3′进一步截短,SR4与bsrG mRNA的配对常数大大下降。有趣的是,在之前构建的bsrE/SR4调控系统中,SR4在截短到53 nt(SR4131~183,仅保留终止子)时依然对带有OPR的靶基因有强烈的抑制效果,结果表明枯草芽孢杆菌体内可能存在某种机制帮助sRNAs与OPR结合。虽然MEISSNER等[21]在对SR5截短体与bsrE mRNA的体外表征中发现,SR5127~163(仅保留终止子)与bsrE mRNA配对效率极低,我们推测SR5在MS-DOS系统中也只需要更短的配对区即可完成抑制。
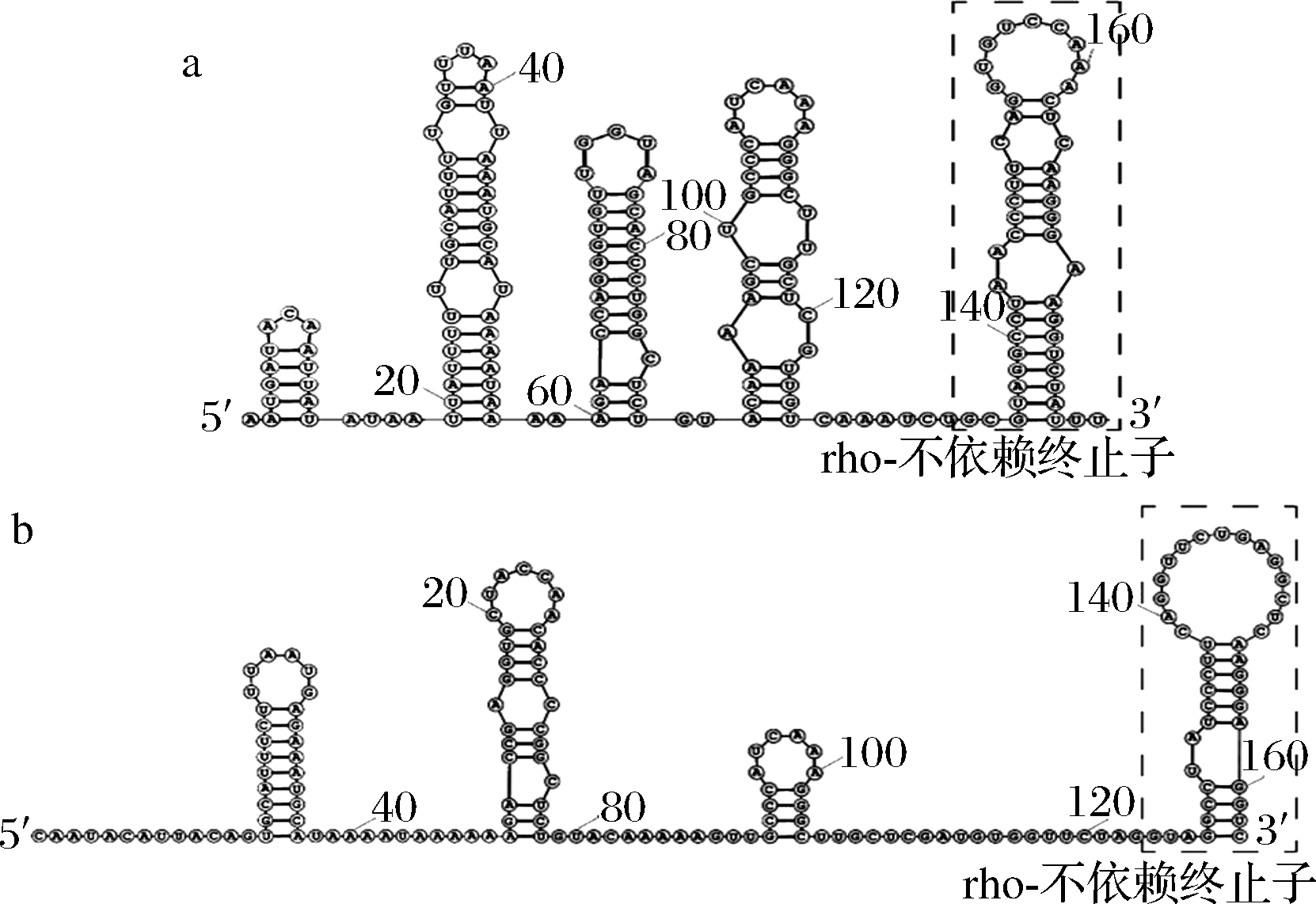
a-SR4二级结构示意图;b-SR5二级结构示意图
图2 SR4与SR5的二级结构
Fig.2 The secondary structures of SR4 and SR5
2.2 基于bsrE/SR5的MS-DOS调控系统的建立与表征
为了证实上述推测,本研究构建了1套以bsrE/SR5为基础的转录后调控系统。首先,利用CRISPR-Cpf1基因组编辑工具将bsrE/SR5基因对从枯草芽孢杆菌B.subtilis 168基因组中敲除,得到菌株BS5,以避免内源表达的非编码小RNA SR5对实验的干扰。然后将bsrE mRNA中与SR5配对的113 bp作为OPR插入到细胞分裂起始蛋白FtsZ基因的终止密码子后面,得到菌株BSF5。经过改造的菌株转录产生的ftsZ mRNA将存在能与SR5配对的OPR区域。随后分别向BS5和BSF5中导入质粒pPasd和pPasd-SR5。FtsZ蛋白在敲除或被严重抑制时,菌体在细胞分裂过程中会表现出明显的缺陷,导致细胞形态由短杆状变为细长型[23]。如图3所示,在不表达SR5时,OPR5的添加并没有影响ftsZ的表达,菌体形态未受到影响;与此同时过表达了SR5的BSF5的菌体分裂受到严重影响,菌体由原来的短杆状变成细长型。通过FtsZ蛋白的验证,我们确定bsrE/SR5系统能够满足MS-DOS调控系统的要求,即OPR5的添加不影响蛋白的正常表达且在过表达SR5的情况下能够强烈抑制带有OPR5靶基因的表达。

a-BS5基因组改造示意图和BS5/pPasd、BS5/pPasd-SR5显微镜观察图;b-BSF5基因组改造示意图和BSF5/pPasd、BSF5/pPasd-SR5显微镜观察图
图3 基于bsrE/SR5的调控系统的构建
Fig.3 Construction of regulation system based on bsrE/SR5
2.3 SR5的截短优化与分析
根据2.1中对SR4与SR5的序列比对与二级结构分析我们可以推测出,在枯草芽孢杆菌中,更短配对区的SR5序列便能够完成抑制。调控小RNA SR5与bsrE mRNA的配对区以及RNase Ⅲ对两者配对形成的双链RNA的切割位点均位于SR5的3′端,因此SR5的核心区应该位于其3′端。为确定SR5的核心序列,我们从5′端对SR5进行不同长度的序列截短。结果如图4所示,设计了6种5′端不同截短长度的SR5序列并将其连接至调控RNA表达质粒上,命名为pPasd-SR5-143、pPasd-SR5-123、pPasd-SR5-103、pPasd-SR5-83、pPasd-SR5-63、pPasd-SR5-37并转化宿主BSG4(图4-c)。SR5和截短后的SR5-37与gfp-OPR5 mRNA的配对示意图如图4-b和图4-d所示。
对含有不同截短长度SR5的重组菌进行荧光强度和生物量测定分析,计算对靶基因GFP表达的抑制效率。结果如图5-a所示,作为调控RNA时,SR5截短至37 nt对GFP的表达抑制效率与使用全长的SR5时没有明显的区别,抑制效率仅从88.8%下降至83.8%,仍然保持较高的抑制水平。如图2所示,SR5是一个非编码小RNA,转录由序列自带的rho-不依赖的终止子终止,SR5基因3′端的37 nt是转录终止子序列,如果对SR5-37进一步的截短会破坏转录终止子的功能,并导致生成长度难以确定的RNA,所以,无法对SR5进行进一步的截短。实验结果表明,OPR5配对区域的延长能够提升对靶基因抑制的效率,但是SR5位于其3′端的37 nt序列足以完成对OPR5的强烈抑制。相比基于bsrG/SR4构建的调控系统,截短后的SR4长度为53 nt,本研究中SR5截短后仅37 nt,且抑制效率仍然保持在83.8%的较高水平,截短后的SR5可以通过PCR轻松的添加到质粒上,简化质粒构建,为其应用提供便利。
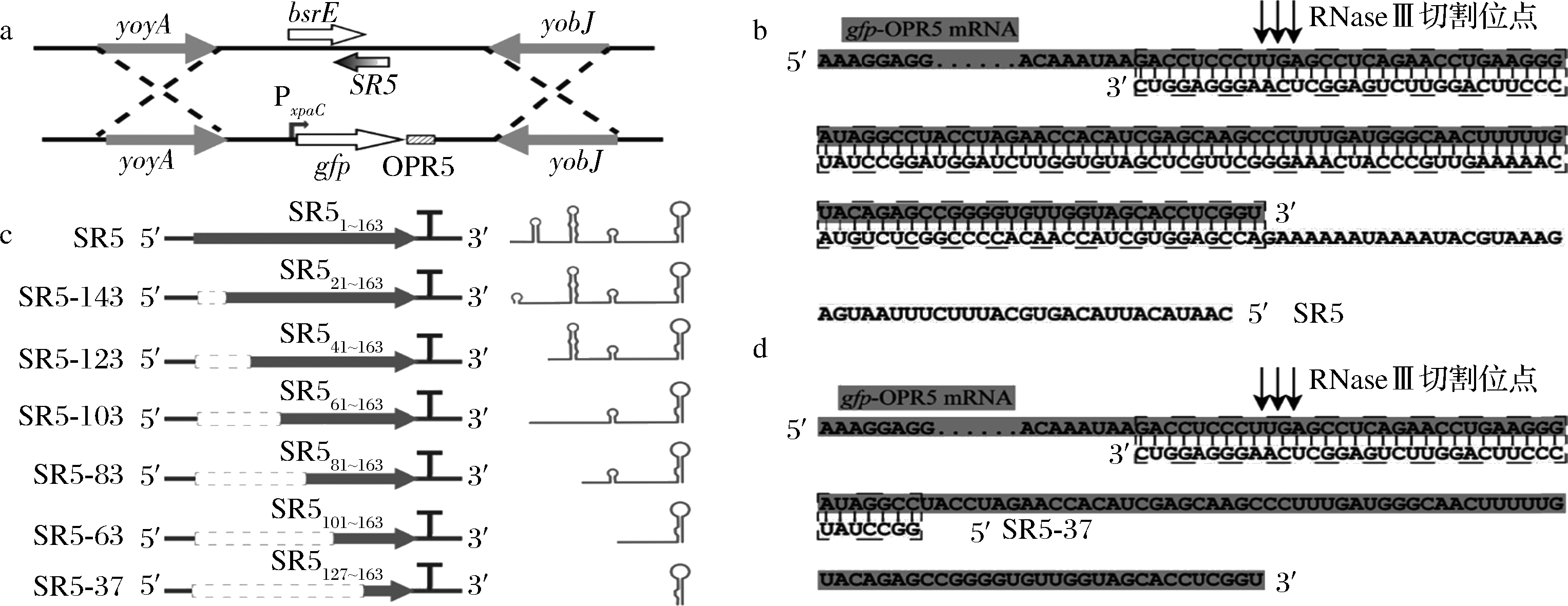
a-BSG5基因组改造示意图;b-SR5与gfp-OPR5的配对示意图;c-SR5截短示意图和二级结构示意图;d-SR5-37与gfp-OPR5的配对示意图
图4 SR5的截短示意图
Fig.4 Schematic diagram of truncation of SR5
注:图4-b中gfp-OPR5 mRNA由深灰色底色表示,SR5由浅灰色底色表示,两者配对序列由虚线方框表示;图4-d中gfp-OPR5 mRNA由深灰色底色表示,SR5-37由浅灰色底色表示,两者配对序列由虚线方框表示
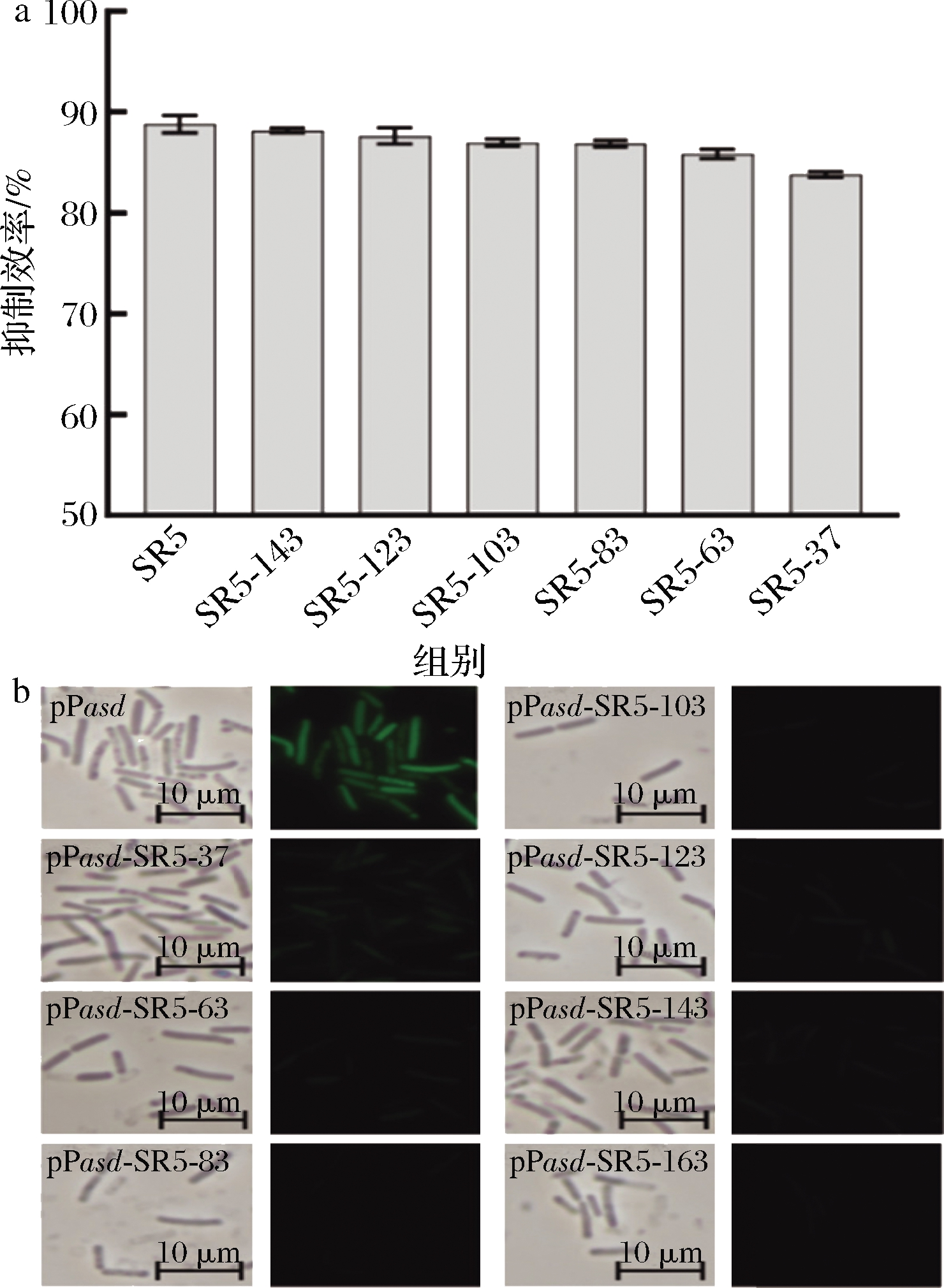
a-SR5不同截短体对GFP的抑制效率;b-BSG5转化不同SR5截短体的荧光显微镜观察图
图5 SR5截短对抑制效率的影响
Fig.5 Effects of truncated SR5 on inhibition efficiency
3 结语
本研究通过对bsrE/SR5和bsrG/SR4进行比对分析,利用与细胞分裂相关的基因ftsZ验证了调控系统的成功构建。然后以绿色荧光蛋白作为表征蛋白,发现基于bsrE/SR5构建的MS-DOS系统抑制效率可以达到88.8%。在此基础上,对非编码小RNA SR5进行截短,发现在仅保留终止子序列(37 nt)的情况下,SR5仍然能保持83.8%的抑制效率。目前,通过SR5的截短,可以实现对基因83.8%~88.8%的抑制。为了满足对基因精确调控的需求,可以利用诱导型启动子调控小RNA SR5的转录水平实现靶基因转录后水平不同抑制效率的可控抑制[12]。在本研究中,SR5在截短至终止子时仍保持较高的抑制效率,而MEISSNER等[21]的体外实验证明仅保留终止子的SR5截短突变体无法在体外与bsrE mRNA结合。在革兰氏阴性菌中,许多sRNAs发挥作用都需要伴侣蛋白Hfq的参与,以确保其稳定性或帮助与靶点的配对,因此推测在枯草芽孢杆菌中可能也存在某种机制帮助sRNAs的配对。
在之前的研究中,我们基于I型毒素-抗毒素系统bsrG/SR4建立了1套通过非编码小RNA进行的转录后调控系统,但是单独的抑制系统无法实现对多基因不同程度的调控。本研究开发的以bsrE/SR5为基础的调控系统丰富了转录后水平调控工具MS-DOS调控系统库。至此,已成功构建了2套MS-DOS系统,并且通过对SR4和SR5的截短对调控系统进行了优化。如图6所示,未来可以通过这2套系统,利用不同的启动子调控小RNA的表达时期和表达水平实现同时对2套基因进行不同时期不同强度的转录后水平抑制。今后可以尝试根据其他的I型毒素-抗毒素系统开发更多的MS-DOS系统,为基因在转录后水平实现更灵活的调控提供技术支持,使枯草芽孢杆菌作为底盘细胞在工业应用上更具潜力和价值。
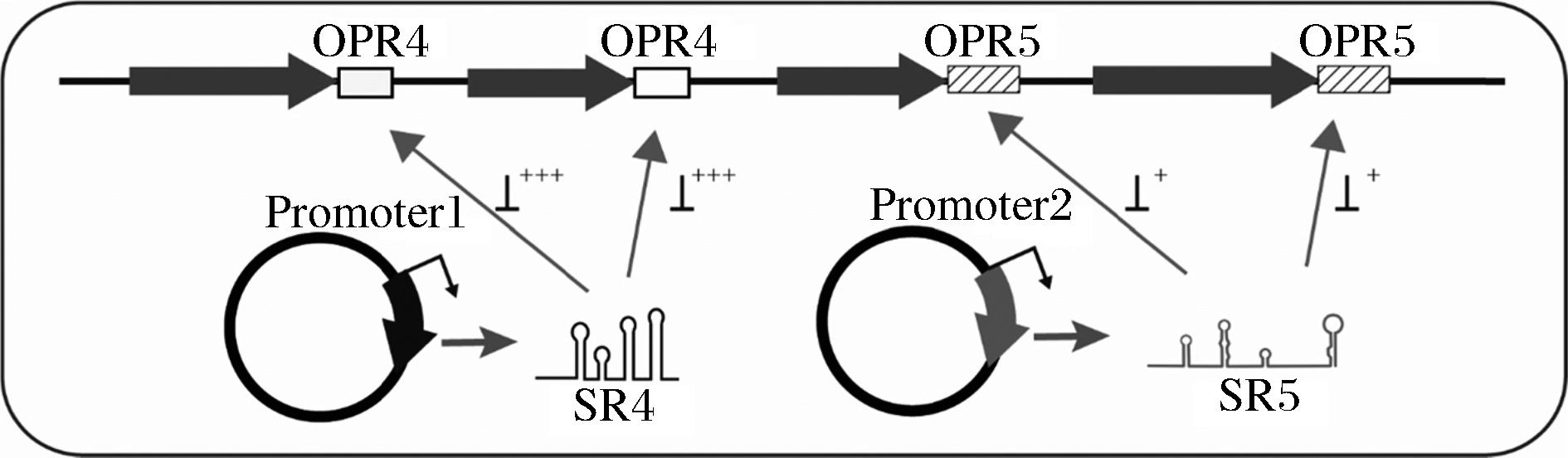
图6 bsrE/SR5与bsrG/SR4双调控系统的设计
Fig.6 Design of bsrE/SR5 and bsrG/SR4 dual-regulation system
[1] JIN P, KANG Z, YUAN P H, et al.Production of specific-molecular-weight hyaluronan by metabolically engineered Bacillus subtilis 168[J].Metabolic Engineering, 2016, 35:21-30.
[2] GU Y, LYU X Q, LIU Y F, et al.Synthetic redesign of central carbon and redox metabolism for high yield production of n-acetylglucosamine in Bacillus subtilis[J].Metabolic Engineering, 2019, 51:59-69.
[3] SUN Y W, LIU C, TANG W Z, et al.Manipulation of purine metabolic networks for riboflavin production in Bacillus subtilis[J].ACS Omega, 2020, 5(45):29 140-29 146.
[4] FAN X G, WU H Y, JIA Z F, et al.Metabolic engineering of Bacillus subtilis for the co-production of uridine and acetoin[J].Applied Microbiology and Biotechnology, 2018, 102(20):8 753-8 762.
[5] YAN P P, WU Y Q, YANG L, et al.Engineering genome-reduced Bacillus subtilis for acetoin production from xylose[J].Biotechnology Letters, 2018, 40(2):393-398.
[6] FU J, HUO G X, FENG L L, et al.Metabolic engineering of Bacillus subtilis for chiral pure meso-2,3-butanediol production[J].Biotechnology for Biofuels, 2016, 9(10):90.
[7] 李洪康, 李由然, 李赢, 等.枯草芽孢杆菌产中性蛋白酶发酵条件优化[J].食品与发酵工业, 2016, 42(5):102-107.
LI H K, LI Y R, LI Y, et al.Optimization of fermentation conditions for the production of neutral protease by Bacillus subtilis[J].Food and Fermentation Industries, 2016, 42(5):102-107.
[8] MENG F Q, ZHU X Y, ZHAO H Z, et al.Improve production of pullulanase of Bacillus subtilis in batch and fed-batch cultures[J].Applied Biochemistry and Biotechnology, 2020, 193(1):296-306.
[9] ZHANG K, DUAN X G, WU J.Multigene disruption in undomesticated Bacillus subtilis ATCC 6051a using the CRISPR/Cas9 system[J].Scientific Reports, 2016, 6(10):6 983-6 995.
[10] SONG Y F, NIKOLOFF J M, FU G, et al.Promoter screening from Bacillus subtilis in various conditions hunting for synthetic biology and industrial applications[J].PloS One, 2016, 11(7):e0158447.
[11] GUIZIOU S, SAUVEPLANE V, CHANG H J, et al.A part toolbox to tune genetic expression in Bacillus subtilis[J].Nucleic Acids Research, 2016, 44(15):7 495-7 508.
[12] YANG S, WANG Y, WEI C B, et al.A new sRNA-mediated posttranscriptional regulation system for Bacillus subtilis[J].Biotechnology and Bioengineering, 2018, 115(12):2 986-2 995.
[13] MORGENS D W, WAINBERG M, BOYLE E A, et al.Genome-scale measurement of off-target activity using Cas9 toxicity in high-throughput screens[J].Nature Communications, 2017, 8(1):299-311.
[14] WIEGERT T, SCHUMANN W.Ssra-mediated tagging in Bacillus subtilis[J].Journal of Bacteriology, 2001, 183(13):3 885-3 889.
[15] WU Y K, LIU Y F, LYU X Q, et al.CAMERS-B:CRISPR/Cpf1 assisted multiple-genes editing and regulation system for Bacillus subtilis[J].Biotechnology and Bioengineering, 2020, 117(6):1 817-1 825.
[16] YAN X, YU H J, HONG Q, et al.Cre/lox system and PCR-based genome engineering in Bacillus subtilis[J].Applied and Environmental Microbiology, 2008, 74(17):5 556-5 562.
[17] DURAND S, JAHN N, CONDON C, et al.Type I toxin-antitoxin systems in Bacillus subtilis[J].RNA Biology, 2012, 9(12):1 491-1 497.
[18] JAHN N, BRANTL S.One antitoxin-two functions:SR4 controls toxin mRNA decay and translation[J].Nucleic Acids Research, 2013, 41(21):9 870-9 880.
[19] JAHN N, BRANTL S.Heat-shock-induced refolding entails rapid degradation of bsrG toxin mRNA by Rnases Y and J1[J].Microbiology, 2016, 162(3):590-599.
[20] JAHN N, PREIS H, WIEDEMANN C, et al.BsrG/SR4 from Bacillus subtilis--the first temperature-dependent type I toxin-antitoxin system[J].Molecular Microbiology, 2012, 83(3):579-598.
[21] MEISSNER C, JAHN N, BRANTL S.In vitro characterization of the type I toxin-antitoxin system bsrE/SR5 from Bacillus subtilis[J].The Journal of Biological Chemistry, 2016, 291(2):560-571.
[22] MULLER P, JAHN N, RING C, et al.A multistress responsive type I toxin-antitoxin system:bsrE/SR5 from the B.subtilis chromosome[J].RNA Biology, 2016, 13(5):511-523.
[23] FEUCHT A, ERRINGTON J.Ftsz mutations affecting cell division frequency, placement and morphology in Bacillus subtilis[J].Microbiology, 2005, 151(6):2 053-2 064.