霉豆腐又称卤腐、腐乳或酱豆腐等,是将大豆按照常规工序制成白豆腐坯后,经过接种或自然发酵、腌制、加配料后发酵等工艺制作而成的一种传统风味发酵食品[1-2],富含氨基酸、蛋白质、大豆异黄酮等成分,且胆固醇和脂肪的含量较低,凭借其细腻的口感和特殊的风味深受消费者的喜爱[3]。霉豆腐在生产过程中由于微生物分泌的酶系的作用,蛋白质被分解成多肽和氨基酸,淀粉转化为糖类,并在多种微生物的共同作用下继续形成有机酸、醛、醇、酮等成分[4]。有研究者利用电子舌技术发现鲜味、咸味、苦味、丰富度是腐乳的主要滋味属性[5]。我国幅员辽阔,不同地区气候差异显著,因而产生了许多具有鲜明地方特色风味的霉豆腐产品,例如绍兴腐乳、桂林腐乳、北京王致和腐乳、克东腐乳等[6]。这些腐乳除克东腐乳以细菌发酵为主形成品质特色外其余产品均以霉菌发酵为主,所以产品特色差异明显。浏阳“霉豆腐”实际上也是一种细菌型发酵的豆腐制品,因为自然发酵过程中并不“长霉”,而是依赖在25~32 ℃将白豆腐置于发酵室中自然发酵48 h,待表面开始出现黏状物后,经过一定时间白酒浸泡滤干,拌入食盐、辣椒粉、香辛料等直接装入玻璃瓶、用植物油覆盖进行发酵1个月左右制成,产品品质风味与其他霉豆腐有较大的差别,但这种差别与形成品质机理存在多少相关性仍缺乏基础研究。
关于霉豆腐及克东腐乳发酵过程中细菌群落及与品质风味之间的关联性已经有很多学者进行了研究,如张雅婷等[7]研究发现细菌和霉菌以及腐乳菌群结构是影响腐乳发酵过程中风味物质变化的主要原因。万红芳等[8]研究认为,来自环境及原材料中其他微生物的侵入与人工接种的发酵菌种混合形成了复杂的微生物系统,对腐乳品质风味的形成产生重要影响。刘井权等[9]、陈曦等[10]研究发现藤黄微球菌(Micrococcus)在腐乳发酵过程中产蛋白酶,可以增加腐乳中游离氨基酸的含量,有益于提高腐乳的风味和品质。曲霉属(Aspergillus)和放线菌属(Actinomucor)是水解酶产生菌,在腐乳中含量丰富[11]。
本文对采取常规方法结合Illumina Miseq测序技术对浏阳霉豆腐生产中关键步骤豆腐制备、发酵24 h、发酵48 h、酒水浸泡、豆腐半成品5个阶段进行细菌群落变化分析,期待为这一传统特色产品创新升级提供技术支持。
1 材料与方法
1.1 实验材料
1.1.1 豆腐样品的采集
供试验材料为5组,包括白豆腐坯、自然发酵24 h、自然发酵48 h、酒水浸泡、豆腐半成品样品,对应编号分别为:DBDF、DF24、DF48、DJS、DBC,样品采集于浏阳某品牌企业。所有试验样品源在4 ℃低温条件下运输至试验室。部分样品直接用于菌落总数测定,部分样品按照每组样品原料捣碎加入适量无菌水混合并使用孔径为0.45 μm的水系滤膜进行过滤,将滤膜及沉淀装入已灭菌的10 mL离心管中,置于-80 ℃冰箱保藏。
1.1.2 材料与试剂
PCA培养基(g/L):胰蛋白胨5.0、酵母浸粉 2.5、葡萄糖1.0、琼脂15.0,pH(7.0±0.2),用于菌落总数的测定。
Pusion Hot start flex 2X Master Mix,用于PCR的扩增,上海仪涛生物仪器有限公司;DL2000 DNA Maker,用于DNA的琼脂糖凝胶电泳,宝日医生物技术(北京)有限公司;Gene colour,用于DNA的琼脂糖凝胶电泳的染色,北京金博益生物技术有限公司;Qubit dsDNA HS Assay Kit,用于DNA的定量检测,美国Invitrogen Life Technologies公司;Biowest Agarose G-10,用于DNA的琼脂糖凝胶电泳,西班牙Biowest公司;50×TAE Buffer,用于DNA的琼脂糖凝胶电泳,上海生工生物工程股份有限公司;AxyPrep PCR Cleanup Kit,用于PCR Cleanup,美国Axygen公司;Stool DNA Kit,用于DNA的提取,美国Omega Bio-Tek公司。
1.1.3 仪器与设备
5424型常温离心机,德国Eppendorf Centrifuge公司;Microfuge 22R型冷冻离心机,美国Beckman Coulter公司;WH-861型旋涡振荡仪,太仓市华利达实验设备有限公司;DK-8D型三温三控恒温水浴锅,上海博迅实业有限公司;A200型PCR仪,用于PCR扩增,杭州朗基科学仪器有限公司;MiSeq pe300测序仪,用于DNA测序,美国Illumina 公司 ;Tanon-2500型电泳仪、凝胶成像仪,用于PCR产物检测,上海天能公司;DW-HL388型超低温冷冻贮存箱,中科美菱低温科技有限责任公司。
1.2 实验方法
1.2.1 样品中菌落总数的测定
采用GB 4789.2—2016[12]进行不同阶段样品菌落总数的检测。
1.2.2 PCR扩增和16S rDNA测序方法
按照Stool DNA Kit(OMEGA bio-tek)说明书提取DNA后,对微生物总DNA进行16S rDNA V3~V4区域扩增。扩增细菌16S rDNA中V3~V4区所选用的引物为:338F(5′-ACTCCTACGGGAGGCAGCAG-3′)、806R(5′-GGACTACHVGGGTWTCTAAT-3′)。PCR体系为25 μL包含DNA模板25 ng、上下游引物各为2.5 μL、12.5 μL PCR预混合物。
PCR反应条件:98 ℃预变性30 s;98 ℃循环35次,变性10 s;54 ℃退火30 s;72 ℃延伸45 s,循环35次,最后72 ℃延伸10 min。取PCR产物10 μL用2%琼脂糖凝胶电泳进行检测,将检测合格样品送至杭州联川生物技术有限公司进行高通量测序[13-14]。
1.2.3 数据分析
对下机数据首先根据样品的条形码(barcode)信息对数据进行拆分,再根据双端序列的重叠(overlap)关系,将序列拼接(merge)成长的tags,并将序列上建库引入的barcode和引物序列去除。使用Vsearch(V2.3.4)软件过滤掉质量值低的序列以及嵌合体序列[15]。再将具有97%以上相似性的序列分配给相同的可操作分类单元(operational taxonomic units,OTU)。每个OTU选择代表性序列,然后使用核糖体数据库项目(ribosomal database project,RDP)分类器将数据归类到每个代表性序列。利用MAFFT(V7.310)软件对不同类群优势种群的差异进行多序列比对,研究不同OTU间的系统发育关系。以Chao 1、Shannon、Simpson和Observed species为指标,用QIIME(1.8.0版)计算了所有样品的各项指标并进行聚类,再通过Alpha及Beta多样性分析单个样本中物种的多样性以及样本间菌群结构之间的差异[16-17]。根据每个OTU代表序列与RDP数据库和Unite数据库的比对,得到各个样本所有OTU的物种注释,对OTU进行物种分类统计后获得门水平和属水平上的物种丰度表。
1.3 样品数量及数据处理
按照生物学样品处理基本要求,菌落总数测定为3个重复,所有生物学重复样品均为4个。
2 结果与分析
2.1 不同发酵阶段菌落总数变化
如表1不同阶段菌落总数计数结果所示,不同阶段样品中的细菌数量均较高,均达到106 CFU/g以上,且在发酵48 h时达到计数最大值。发酵48 h时,菌落总数达到了2.2×109 CFU/g,而随后到酒水浸泡30 s时,菌落总数明显降低,可能是因为乙醇的作用,不仅杀死了样品中部分细菌,也有部分转移到酒水中,导致菌落计数结果显著降低。而半成品阶段菌落总数仍然降低的可能原因是香辛料中加入了大量食盐和植物油。
表1 不同阶段菌落总数变化
Table 1 Changes in the total number of colonies at different stages

阶段豆腐发酵24 h发酵48 h酒水浸泡30 s半成品菌落总数/(CFU·g-1)1.1×1071.2×1092.2×1091.1×1093.2×108
2.2 不同发酵阶段的clean data
为提高后续分析质量和可信度,对原始下机数据进行双端拼接、质量控制、嵌合体过滤后,对本次测序中的全部有效数据进行统计后,结果如表2所示。
表2 不同发酵阶段样品16S rDNA测序获得的有效数据
Table 2 Valid data obtained by 16S rDNA sequencing of samples at different fermentation stages
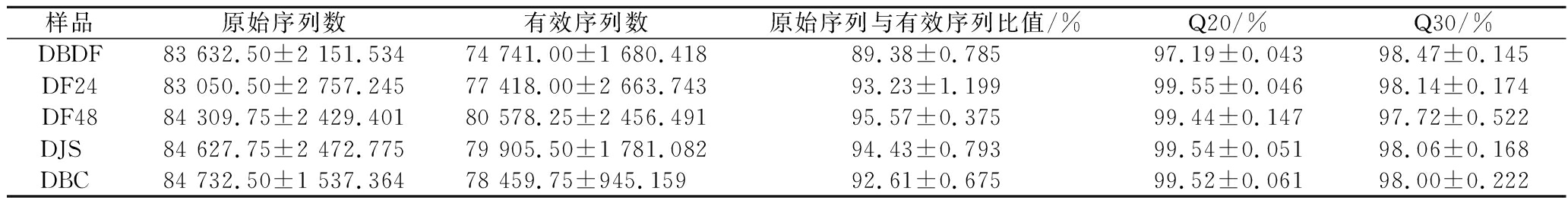
样品原始序列数有效序列数原始序列与有效序列比值/%Q20/%Q30/%DBDF83 632.50±2 151.53474 741.00±1 680.41889.38±0.78597.19±0.04398.47±0.145DF2483 050.50±2 757.24577 418.00±2 663.74393.23±1.19999.55±0.04698.14±0.174DF4884 309.75±2 429.40180 578.25±2 456.49195.57±0.37599.44±0.14797.72±0.522DJS 84 627.75±2 472.77579 905.50±1 781.08294.43±0.79399.54±0.05198.06±0.168DBC 84 732.50±1 537.36478 459.75±945.15992.61±0.67599.52±0.06198.00±0.222
其中有效序列数均在74 000条以上,Q20值均超过97%,并且Q30值均超过了97%,这些数据结果表明本次测序结果较好,测序质量值较高,所有数据都已达到后续试验分析的要求。
2.3 OTU聚类分析
由图1可知,DBDF中仅有367个OTU,而后进入控温发酵阶段,环境中微生物及豆腐自身带入微生物生长增殖,样品中微生物总数在增加,发酵24 h有485个OTU,而DF48样品中则有541个OTU。但在DJS样品中仅测得475个OTU,与酒水浸泡后部分微生物进入酒水中有关,而从DJS样品过渡到DBC样品阶段,样品中的OTU未发生明显变化。DBDF、DF24、DF48、DJS、DBC 5个样品中,独有的OTU数分别为92、122、263、129、115,其变化趋势与不同阶段样品中的总OTU数变化趋势基本吻合,说明在不同的阶段微生物种类和微生物群落结构也随之发生演变。5个样品中共有的OTU数为113,说明这些微生物在霉豆腐形成的各个阶段比较稳定但其他种类的微生物出现交替变化,这种变化应该与不同阶段环境变化有关。
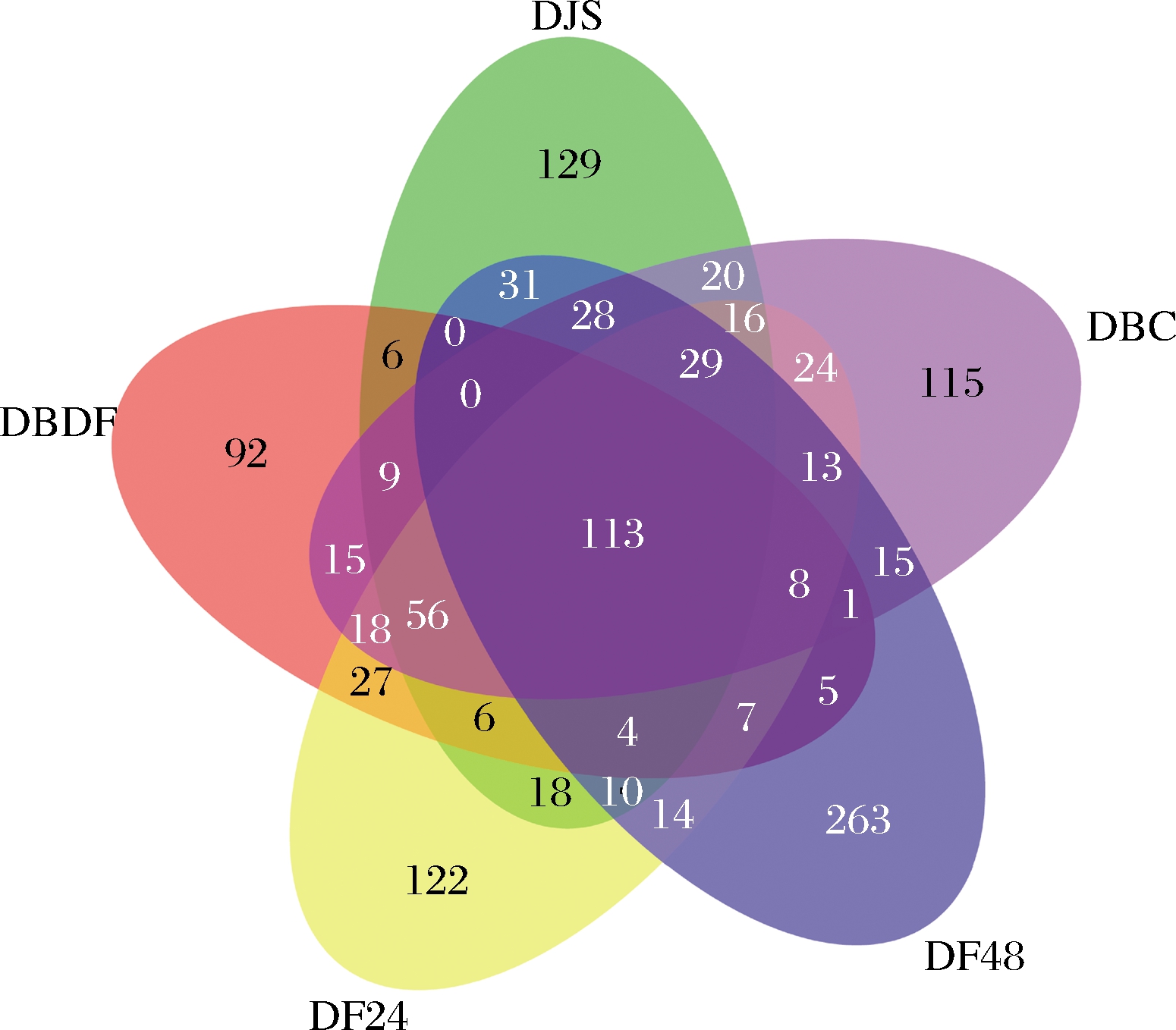
图1 不同发酵阶段中细菌OTU的聚类分析
Fig.1 Cluster analysis of bacterial OTU in different fermentation stages
2.4 不同发酵阶段样品的细菌α-多样性分析
α-多样性是指一个生态系统或者特定区域内微生物的多样性,一般使用包括反应群落丰度的Chao 1、Ace等指数和用来反应群落多样性的Shannon指数、Simpson指数等进行度量分析[18]。由表3可知,白豆腐中微生物的种类是最少的,这是白豆腐本身经过高温热处理后得到的,但进入发酵阶段后,微生物的种类(Chao1指数)呈现先增加后降低再上升的趋势,但微生物的多样性和均匀度(Shannon指数)为先升高再降低的趋势,可能是因为样品在经过酒水浸泡后拌入了辣椒、香辛料等带入新的微生物,导致Chao 1指数有小幅度的上升。在发酵48 h时,样品中的微生物种类、微生物的多样性、均匀度都达到了最高值,此结果与OTU聚类分析结果相似。
表3 不同阶段细菌的α-多样性分析
Table 3 Analysis of α-diversity of bacteria in different stages
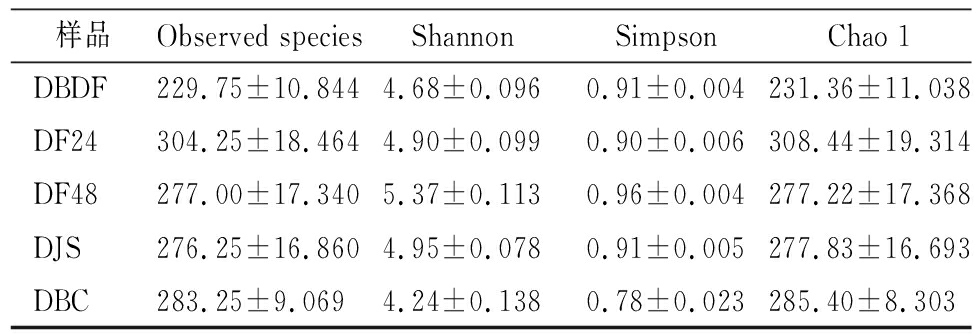
样品Observed speciesShannonSimpsonChao 1DBDF229.75±10.8444.68±0.0960.91±0.004231.36±11.038DF24304.25±18.4644.90±0.0990.90±0.006308.44±19.314DF48277.00±17.3405.37±0.1130.96±0.004277.22±17.368DJS276.25±16.8604.95±0.0780.91±0.005277.83±16.693DBC283.25±9.0694.24±0.1380.78±0.023285.40±8.303
2.5 不同发酵阶段细菌β-多样性分析
由图2可知,在主成分分析(principal component analysis,PCA)中,两坐标轴的贡献率之和为81.06%,说明已经能够反映样品中的大部分信息。发酵48 h的样品(DF48)的微生物菌群与其他样品中的微生物菌群结构差异很大,可能是发酵基本成熟,发酵微生物无论数量种类都是最多的。而DBDF、DF24、DJS 3个样品相距较近,说明三者菌群结构相似度较高,DBC样品与其他样品相距较远,分析原因可能是加入食盐、辣椒面、香辛料、植物油后菌相有些变化开始进入后期厌氧发酵阶段,菌群结构基本趋于稳定。
2.6 不同发酵阶段物种中门水平、属水平差异性
由图2可知,将全部OTU代表序列与RDP数据库和Unite数据库进行比对,得到各个样本所有OTU的物种注释[19]。根据物种注释结果,在门和属2个水平上对物种组成进行统计分析并构建对应的丰度谱[20],得到对应的丰度谱如图3、图4所示。

图2 不同阶段PCA结果图
Fig.2 PCA results of different stages
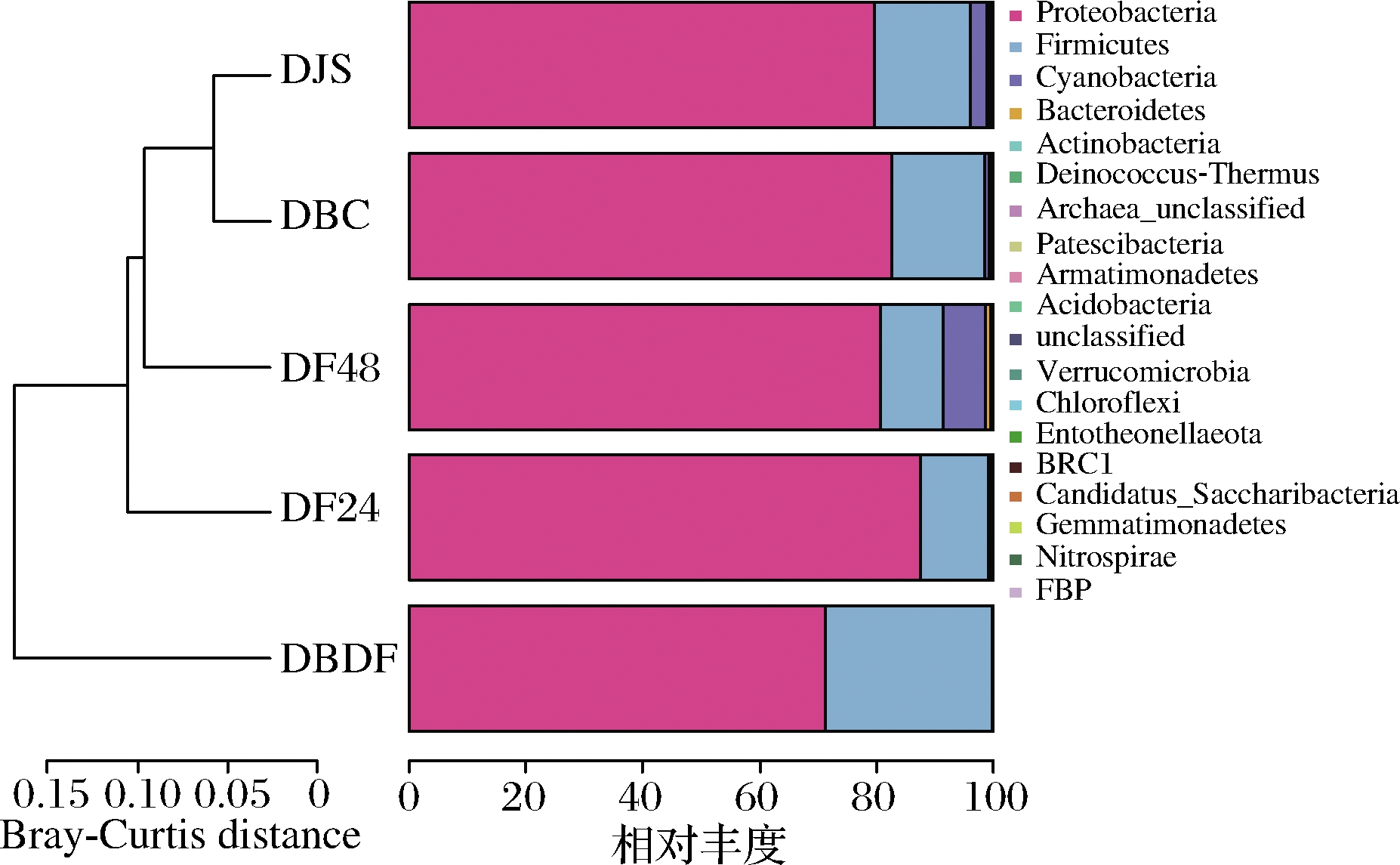
图3 门水平丰度谱及Bray-Curtis聚类分析
Fig.3 Abundance spectrum and Bray-Curtis cluster analysis at the phylum level
由图3可知,在门水平上,所采集的5个样品均是以变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)为主,其中,在DBDF、DF24、DF48、DJS、DBC 5个样品中,变形菌门相对丰度均超过70%,分别达到了71.25%、87.53%、80.64%、79.65%、82.79%,而厚壁菌门也达到了11.64%~28.50%。由此可见,变形菌门和厚壁菌门在样品中占绝对优势。值得注意的是,在DBDF、DF24、DF48、DJS、DBC 5个样品中,拟杆菌门(Bacteroidetes)的丰度占比非常低,仅仅只有0.14%、0.50%、0.93%、0.55%、0.32%,这与石黎琳等[21]的研究存在较大差异,该研究中第2阶段拟杆菌门的丰度占比达到了49%,而浏阳霉豆腐第2阶段中仅有0.32%。这应该是导致浏阳霉豆腐具有特殊风味以及与其他霉豆腐品质存在差异的主要原因,但这仍需进一步的试验研究确定。
由图4可知,本试验在属水平上一共注释了30个属,其中泛菌属(Pantoea)、肠杆菌属(Enterobacter)、明串珠菌属(Leuconostoc)、假单胞菌属(Pseudomonas)为优势菌属。泛菌属D-葡萄糖和其他糖类可产酸,能形成色素。陈卓等[22]发现肠杆菌属、不动杆菌(Acinetobacter)、水栖菌(Enhydrobacter)在腐乳后发酵阶段发挥十分重要的作用,多种菌协同作用使得腐乳发酵前期和中期的蛋白酶活性显著升高。有研究表明,肠杆菌属是腐乳中主要的组胺合成细菌[23-24],而低剂量摄入的组胺是人体中不可或缺的生物活性物质。明串珠菌属在后发酵中后期均有体现,且在后期的比例高于中期,可能是导致霉豆腐后发酵期氨基酸含量不断升高的主要原因,还有研究发现明串珠菌属中的一些菌可以起到一定程度的抑菌作用,对伤寒沙门菌、金黄色葡萄球菌有着一定的拮抗作用。假单胞菌属可以分解蛋白质,在DF24、DF48阶段中检测出的含量相对较高,在发酵过程中分解大豆中的蛋白质,并且在代谢过程中产丙酸、乙酸等,而这些酸是酯和长链脂肪酸合成的前体物质。随着发酵的进行,优势菌属均呈现出先减少后增加的变化趋势。这与后期酒水处理和进入厌氧发酵阶段有关。
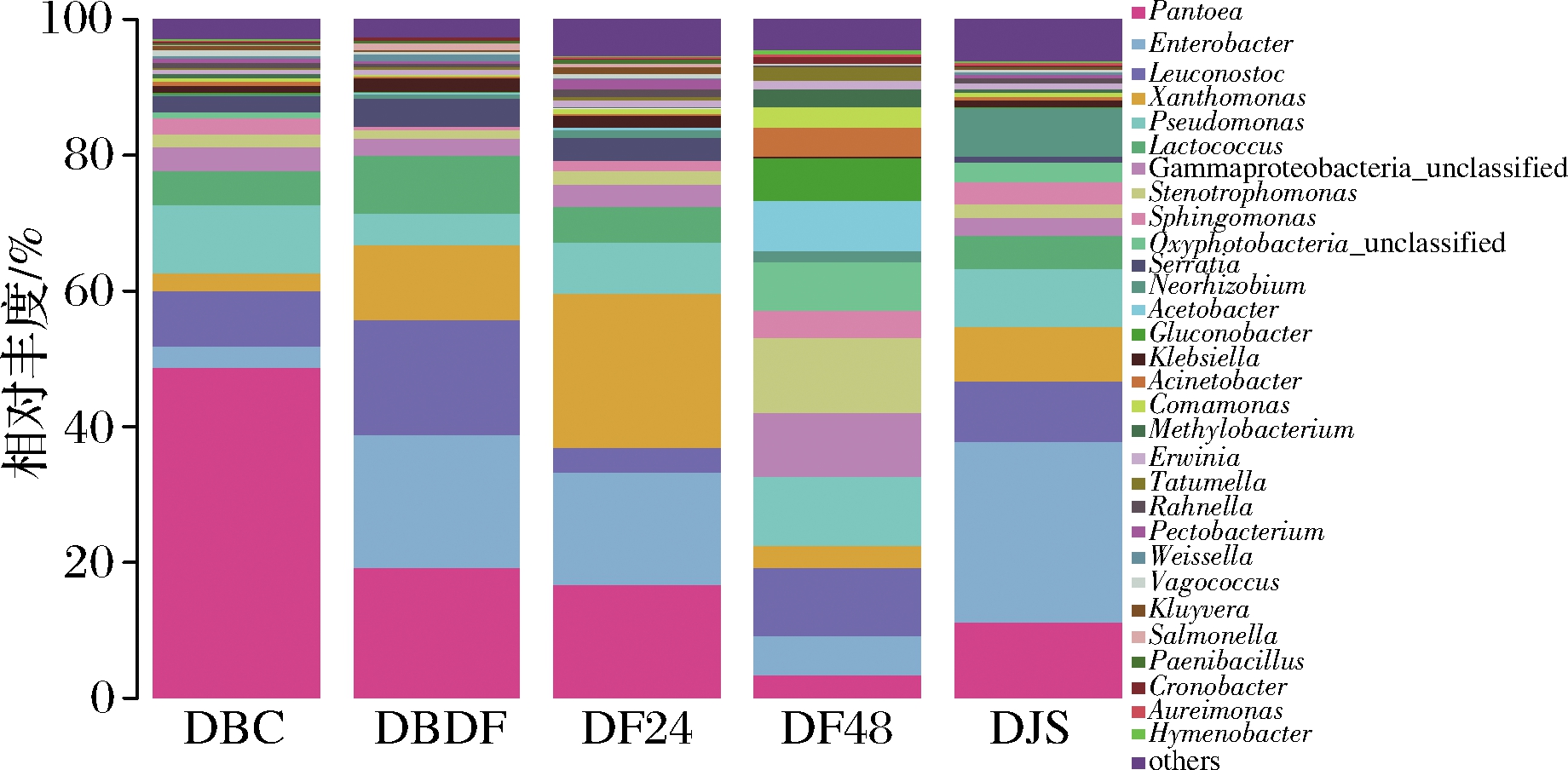
图4 属水平丰度谱
Fig.4 Abundance spectrum at the genus level
3 讨论
在浏阳霉豆腐的形成过程中,有多种微生物参与了这一发酵的过程,随着Illumina Miseq测序技术的不断发展和完善,更易于我们发现和掌握霉豆腐形成过程中微生物菌落的变化规律,本试验对采取常规方法制备的豆腐、发酵24 h、发酵48 h、酒水浸泡、豆腐半成品5个阶段利用Illumina Miseq测序技术进行微生物群落变化分析,从分析结果来看,霉豆腐生产的主要阶段细菌的数量比较高,霉豆腐滋味形成与细菌代谢有着密切的联系,但由于这类特色霉豆腐实际上并不长霉,而是细菌性发酵产品,所以未对其他菌相进行分析,代谢产物研究将在以后的研究中进一步深入。由于浏阳霉豆腐为湖南地方特色发酵豆制品,下一步将进一步研究发酵过程中蛋白质分解规律和风味形成规律,同时结合可培养办法获得关键产蛋白酶的微生物进行纯化鉴定,同时探索发酵第2阶段微生物变化与品质风味相关性,为这一传统特色产品创新升级提供系统化的技术支持。
[1] 张雪梅, 蒲彪.腐乳的研究概况与发展前景[J].食品与发酵工业, 2005, 31(5):94-97.
ZHANG X M, PU B.Developing and prospects of fermented soybean curd[J].Food and Fermentation Industries, 2005, 31(5):94-97.
[2] 李幼筠. 中国腐乳的现代研究[J].中国酿造, 2006,25(1):4-7.
LI Y J.Modern research on Chinese sufu[J].China Brewing, 2006,25(1):4-7.
[3] 李理, 罗立新, 梁世中.腐乳的研究进展[J].食品与发酵工业, 2002, 28(10):70-74.
LI L, LUO L X, LIANG S Z.Advance on sufu[J].Food and Fermentation Industries, 2002, 28(10):70-74.
[4] 哈尔乐哈西·布勒斯别克.改善腐乳品质的加工工艺技术研究[D].杭州:浙江大学, 2015.
BOLESBEK·H.Study on the improvement of the processing technology of sufu quality[D].Hangzhou:Zhejiang university, 2015.
[5] 王鹏, 王文平, 续丹丹,等.红腐乳发酵过程中滋味化合物分析及电子舌鉴别[J].食品科学, 2021, 42(14):170-179.
WANG P, WANG W P, XU D D, et al.Analysis of taste compounds in red sufu during fermentation and discrimination with electronic tongue[J].Food Science, 2021, 42(14):170-179.
[6] 江景泉. 毛霉驯化及微波对腐乳白点抑制效果的研究[D].重庆:西南大学, 2009.
JIANG J Q.Study on the inhibition of furu white flake of the mucor domestication and microwave[D].Chongqing:Southwest University, 2009.
[7] 张雅婷, 孙娜, 于寒松, 等.红腐乳发酵过程中菌群结构与风味相关性研究进展[J].食品与发酵工业, 2020, 46(1):287-294.
ZHANG Y T, SUN N, YU H S, et al.Advances in research on correlation between flora structure and product flavor during red sufu fermentation[J].Food and Fermentation Industries, 2020, 46(1):287-294.
[8] 万红芳, 赵勇, 王正全, 等.生产菌种及环境微生物与腐乳品质关系研究进展[J].食品与发酵工业, 2019, 45(6):255-261.
WAN H F, ZHAO Y, WANG Z Q, et al.Research progress on the relationship between sufu quality and microorganisms from starters and environment[J].Food and Fermentation Industries, 2019, 45(6):255-261.
[9] 刘井权, 孙剑秋, 臧威, 等.QU1.602藤黄微球菌腐乳工艺操作要点[J].中国调味品, 2008, 33(4):62-64.
LIU J Q, SUN J Q, ZANG W, et al.The operation technics of micrococcus luteus QU1.602 sufu[J].China Condiment, 2008, 33(4):62-64.
[10] 陈曦, 陈红, 刘懋, 等.藤黄微球菌(Micrococcus luteus) KDF1胞外蛋白酶酶学性质与水解大豆蛋白产物研究[J].食品与发酵工业, 2013, 39(9):11-17.
CHEN X, CHEN H, LIU M, et al.The extracellular protease from Micrococcus luteus KDF1:Properties and characterization of its hydrolysates from soy protein[J].Food and Fermentation Industries, 2013, 39(9):11-17.
[11] CAI H Y, DUMBA T, SHENG Y H, et al.Microbial diversity and chemical property analyses of sufu products with different producing regions and dressing flavors[J].LWT, 2021, 144:111245.
[12] 国家卫生和计划生育委员会, 国家食品药品监督管理总局. GB 4789.2—2016食品安全国家标准 食品微生物学检验 菌落总数测定[S]. 北京: 中国标准出版社, 2017.
National Health and Family Planning Commission of the People′s Republic of China, China Food and Drug Administration . GB 4789.2—2016. National food safety standard food microbiological inspection determination of aerobic plate count[S]. Beijing: China Standards Press, 2017.
[13] 邹颖玲, 刘姝韵, 王桂瑛, 等.基于PCR-DGGE技术分析宣威火腿中真菌群落结构[J].食品与发酵工业, 2020, 46(6):269-274.
ZOU Y L, LIU S Y, WANG G Y, et al.Analysis of fungal community structure in Xuanwei ham by PCR-DGGE[J].Food and Fermentation Industries, 2020, 46(6):269-274.
[14] 廖新浴, 陈信贤, 刘东红, 等.不同种类腐乳产品中优势菌种的分离与鉴定[J].食品安全质量检测学报, 2018, 9(14):3 755-3 759.
LIAO X Y, CHEN X X, LIU D H, et al.Isolation and identification of dominant bacteria in different types of sufu products[J].Journal of Food Safety & Quality, 2018, 9(14):3 755-3 759.
[15] 杨林. 豆豉发酵过程中菌群分析及功能菌株的筛选[D].南昌:江西师范大学, 2017.
YANG L.Analysis of microbiota and screening of functional microbes in douchi fermentation process[D].Nanchang:Jiangxi Normal University, 2017.
[16] SUL W J, COLE J R, JESUS E D C, et al.Bacterial community comparisons by taxonomy-supervised analysis independent of sequence alignment and clustering[J].Proceedings of the National Academy of Sciences, 2011, 108(35):14 637-14 642.
[17] 陈浩, 何国庆.乳酸菌对发酵豆豉品质的影响[J].食品安全质量检测学报, 2017, 8(12):4 516-4 523.
CHEN H, HE G Q.Effect of lactic acid bacteria on quality of fermented Douchi[J].Journal of Food Safety & Quality, 2017, 8(12):4 516-4 523.
[18] 陶康, 吴凌伟, 金晓芳, 等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学, 2021, 42(8):143-149.
TAO K, WU L W, JIN X F, et al.Analysis of microbial diversity in sufu using high-throughput sequencing[J].Food Science, 2021, 42(8):143-149.
[19] 邓高文, 刘洋, 李跑, 等.基于Illumina MiSeq高通量测序技术分析贵州水豆豉中细菌多样性[J/OL].食品科学, 2021.https://kns.cnki.net/kcms/detail/11.2206.ts.20210306.1503.004.html.
DENG G W, LIU Y, LI P, et al.Analysis of bacterial diversity in Guizhou fermented black bean by Illumina MiSeq high-throughput sequencing technology[J/OL].Food Science, 2021.https://kns.cnki.net/kcms/detail/11.2206.ts.20210306.1503.004.html.
[20] 陈梦娟, 蒋立文, 徐元昊, 等.利用Illumina MiSeq测序分析手筑茯砖茶发酵及干燥阶段真菌群落多样性[J].食品科学, 2020, 41(2):126-132.
CHEN M J, JIANG L W, XU Y H, et al.Analysis of fungal community diversity during fermentation and drying of hand-made fu brick tea by Illumina MiSeq sequencing[J].Food Science, 2020, 41(2):126-132.
[21] 石黎琳, 牟方婷, 李安, 等.基于高通量测序技术分析腐乳自然发酵过程微生物多样性[J].中国酿造, 2021, 40(2):144-149.
SHI L L, MU F T, LI A, et al.Analysis of microbial diversity in sufu during natural fermentation using high-throughput sequencing technology[J].China Brewing, 2021, 40(2):144-149.
[22] 陈卓, 吴学凤, 穆冬冬, 等.红腐乳后酵期风味物质与细菌菌群分析[J].食品科学, 2021, 42(6):118-125.
CHEN Z, WU X F, MU D D, et al.Determination and correlation analysis of flavor components with bacterial community in post-fermented red sufu[J].Food Science, 2021, 42(6):118-125.
[23] HU M, DONG J, TAN G L, et al.Metagenomic insights into the bacteria responsible for producing biogenic amines in sufu[J].Food Microbiology, 2021, 98:103762.
[24] SHI C S, LIU M M, ZHAO H F, et al.Formation and control of biogenic amines in sufu-a traditional Chinese fermented soybean product:A critical review[J].Food Reviews International, 2021.DOI:10.1080/87559129.2021.1936002.