组蛋白赖氨酸去甲基化酶(histone lysine demethylase,KDMs)主要分为两类,其中组蛋白赖氨酸特异性脱甲基酶1(histone lysine specific demethylase 1,Lsd1)是2004年第一个被鉴定出来的组蛋白脱甲基酶,是黄素腺嘌呤二核苷酸依赖性胺氧化酶超家族成员之一[1]。Lsd1的脱甲基反应可被看作底物与黄素辅因子之间的氢化物转移[2],反应过程中同时涉及到与分子氧的反应[3-4]。另一类脱甲基酶则是含有Jumonji-C结构域的组蛋白赖氨酸脱甲基酶(jumonji C,Jmjc),于2006年首次被鉴定出来[5],在Fe2+和O2的参与下直接氧化甲基,以脱甲醛的形式完成去甲基化[6-7]。
KDMs可以催化组蛋白H3K4[1, 8]、H3K9[9-10]的去甲基化反应。此外,KDMs具备丰富的底物特异性[11],可以作用于多种非组蛋白底物,例如肿瘤抑制因子p53[12]、DNA甲基转移酶1[13]。KDMs在多种肿瘤细胞中高表达,已经成为肿瘤治疗药物开发的重要靶蛋白,目前对于该酶的研究主要聚焦于多胺类抑制剂[14-15]以及可逆选择性抑制剂[16]等。在该酶的研究过程中,也有利用大肠杆菌(Escherichia coli)进行原核表达的,LAURENT等[17]在E.coli Rosetta(DE3)-pET15b表达了人源的目的蛋白并进行了生化分析。
近年来,随着食品行业的快速发展,相关的食品加工废水的产生也在快速增长,主要含有氮、磷等化合物。关于食品工业废水的处理已经成为相关领域的热点问题,目前大多采用生物法、物理法和化学法等[18-20],其中N—CH3类化合物很难实现降解,能够对人体的神经系统以及肝肾造成严重损伤,同时对水生生物具有毒害作用。DASH等[21]从土壤中筛选出假单胞菌NCIM5235,能够有效降解咖啡因,但是微生物降解底物单一,不能广泛应用。
本研究选择斑马鱼和咖啡来源的赖氨酸脱甲基酶进行研究,斑马鱼作为模式生物具有清晰的生物学背景,同时通过前期研究,植物中咖啡来源的脱甲基酶表现出与斑马鱼来源相似的底物特异性。本研究首先构建菌株E.coli BL21(DE3)/pET28a-lsD1和E.coli BL21(DE3)/pET28a-jmjC,然后表达纯化获得KDMs。通过发酵条件优化获得体外可溶性KDMs,且首次对其进行酶学性质测定,为KDMs体外抑制剂的研发提供一定基础,同时利用非组蛋白底物进行动力学研究,为后期催化含N—CH3类化合物降解的研究提供依据。
1 材料与方法
1.1 材料
1.1.1 质粒与菌株
大肠杆菌JM109、大肠杆菌BL21(DE3)、质粒载体pET28a均为本实验室保存。
1.1.2 试剂、培养基与仪器
LB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10,121 ℃灭菌20 min。
试剂:Taq DNA聚合酶、质粒小量提取试剂盒、DNA片段纯化试剂盒、胶回收试剂盒,南京诺唯赞生物科技公司;PrimerSTAR Max DNA聚合酶、限制性内切酶Hind Ⅲ、EcoR Ⅰ、Sac Ⅰ,Takara公司;甲醛脱氢酶,SIGMA公司;α-KG、N-甲基-L-亮氨酸,上海麦克林生化科技公司;其他试剂均为国产分析纯。
仪器:SCG蛋白纯化系统,苏州赛谱仪器有限公司;V-1200分光光度计,上海美普达仪器公司;S100D型PCR仪,美国BIO-RAD公司;Spark全自动酶标仪,TECAN公司;CF16RX-Ⅱ冷冻离心机,日本HITACHI公司;HYL-C组合式摇床,江苏太仓市强乐实验设备有限公司。
1.2 实验方法
1.2.1 重组菌的构建
由NCBI数据库中查找得到斑马鱼来源的组蛋白赖氨酸脱甲基酶(Lsd1)的基因序列(GenBanK:NP_001229924.1)以及咖啡来源的组蛋白赖氨酸脱甲基酶(Jmjc)的基因序列(GenBanK:XP_027103441.1),并对两种来源的序列进行大肠杆菌偏好性的密码子优化,由苏州金唯智生物科技公司合成。以合成的目的酶基因序列为模板设计引物,lsD1:P1(5′-CCGGAATTCATGCTGAGCAATAAGAAAAGCG-3′)、P2(5′-CCCAAGCTTTTATT-GGATACTTGGGCTCG-3′),jmjC:P3(5′-CGAGCTCATGAGCATGATGAAACG-3′)、P4(5′-CCCAAGCT-TTTAACGACCGTCCTCAAC-3′),进行PCR扩增。利用EcoR Ⅰ、Hind Ⅲ和Sac Ⅰ、Hind Ⅲ分别对基因片段lsD1和jmjC进行双酶切,连接到质粒pET28a上,构建重组质粒pET28a-lsD1和pET28a-jmjC。,将两个重组质粒以及空载质粒通过热转化法转入到大肠杆菌感受态细胞BL21(DE3)内,37 ℃、200 r/min后培养1 h,离心收集菌体,涂布至30 μg/mL卡那霉素的LB平板,在37 ℃培养箱中倒置培养12 h。从LB平板中随机挑取转化子进行菌落PCR验证,将验证正确的转化子送到生工生物工程(上海)股份有限公司测序,最终获得E.coli BL21(DE3)/pET28a-lsD1和E.coli BL21(DE3)/pET28a-jmjC重组菌株。
1.2.2 Lsd1和Jmjc的诱导表达
将2株重组菌BL21(DE3)/pET28a-lsD1和BL21(DE3)/pET28a-jmjC以及空质粒对照菌平板活化。活化菌株接入装有15 mL LB培养基的50 mL锥形瓶中,在37 ℃,200 r/min的条件下培养12 h,以此作为种子液,以1%的接种量转至装有50 mL LB培养基的250 mL锥形瓶中,于37 ℃,200 r/min的条件下培养至OD600为0.6~0.8,分别加入终浓度为0.1、0.25 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside, IPTG),将添加2种不同浓度IPTG的培养基分别在37 ℃、200 r/min、7 h,25 ℃、200 r/min、12 h,16 ℃、200 r/min、24 h的条件下培养。4 ℃、12 000 r/min离心5 min收集菌体。用Na2HPO4-NaH2PO4缓冲液(20 mmol/L、pH 8.0)洗涤并重悬菌体,用超声仪破碎细胞,4 ℃、12 000 r/min离心10 min,收集上清液即为粗酶液。
1.2.3 目的蛋白纯化以及分析
利用镍柱亲和层析对目的蛋白Lsd1和Jmjc进行纯化。先用A液(20 mmol/L Na2HPO4、20 mmol/L NaH2PO4、500 mmol/L NaCl、20 mmol/L咪唑,pH 8.0)平衡蛋白系统,进样。目的蛋白Lsd1在体积分数80% A液和体积分数20% B液(20 mmol/L Na2HPO4、20 mmol/L NaH2PO4、500 mmol/L NaCl、500 mmol/L咪唑,pH 8.0)条件下被洗脱收集,目的蛋白Jmjc在体积分数60% A液和体积分数40% B液条件下被洗脱收集,之后用20 mmol/L Tris-HCl缓冲液(pH 7.0)透析去除高浓度的盐,采用考马斯亮蓝法测定蛋白浓度,用SDS-PAGE分析检测蛋白。
1.2.4 酶活力测定
2种KDMs组蛋白去甲基化酶反应的一个主要副产物都是甲醛,甲醛脱氢酶在辅酶烟酰胺腺嘌呤二苷酸(nacotinamide adenine dinucleotide,NAD+)存在的条件下,将NAD+还原为NADH,同时将甲醛氧化为甲酸。NADH通过酶标仪检测并定量,激发波长为330 nm,发射波长为460 nm[22-23],从而间接测定活性。反应总体积为400 μL,20 mmol/L Tris-HCl缓冲液(pH 7.0)为整个反应的缓冲液,反应体系如下,Lsd1:10 mg/mL NAD+、0.01 U/μL甲醛脱氢酶、10 mmol/L底物、0.1 mg/mL纯酶(保存于20 mmol/L Tris-HCl缓冲液中,pH 7.0);Jmjc:10 mg/mL NAD+、0.01 U/μL甲醛脱氢酶、10 mmol/L底物、0.1 mg/mL纯酶(保存于20 mmol/L Tris-HCl缓冲液中,pH 7.0)、l mmol/L Fe2+、1 mmol/L α-KG。
酶活力定义:在25 ℃,pH 7.0条件下,1 min内催化底物产生1 μmol 甲醛(HCHO)所需要的酶量,定义为1个酶活力单位(IU)。
1.2.5 酶学性质的研究
后续测定所用的酶液为镍柱亲和层析所收集的纯酶液,测定中的底物均为N-甲基-L-亮氨酸。每个测定条件设置3个平行试验。
(1)目的蛋白Lsd1和Jmjc的最适反应温度和温度稳定性的测定:在pH 7.0,温度10~55 ℃条件下反应6 h,测定酶活力,以酶活力最高值为100%,确定最适反应温度。将2个重组酶在10~70 ℃条件下保存30 min,然后在最适反应温度、pH 7.0条件下反应6 h,测定酶活力,探究相关的温度稳定性。
(2)目的蛋白Lsd1和Jmjc的最适反应pH和pH稳定性的测定:25 ℃条件下,分别在pH 4.0~10.0的缓冲液中反应6 h,测定酶活力,以酶活力最高值为100%,确定最适反应pH。25 ℃条件下,将2个目的蛋白在不同pH缓冲液中保存12 h,之后在最适pH,25 ℃条件下测定酶活力,确定pH稳定性。
(3)金属离子对目的蛋白Lsd1和Jmjc的影响:在反应体系中添加Mn2+、Co2+、Mg2+、Ca2+、Zn2+、Ni2+至终浓度分别为0.1、1.0、5.0 mmol/L,在最适反应条件下测定酶活力,以没有添加金属离子的酶活力为100%。
(4)动力学研究:选择不同浓度N-甲基-L-亮氨酸,在最适温度、最适pH条件下与纯酶液反应6 h。根据米氏方程,使用双倒数法计算Lsd1和Jmjc的Km值以及kcat/Km值。
2 结果与分析
2.1 组蛋白赖氨酸脱甲基酶基因在E.coli中的重组表达
2.1.1 组蛋白赖氨酸脱甲基酶基因的克隆
lsD1和jmjC基因的PCR扩增产物如图1-a和图1-b所示,在2 500、1 800 bp分别有1条单一的扩增片段,与目的片段lsD1(2 514 bp)和jmjC(1 728 bp)的大小一致。
2.1.2 重组菌构建
将PCR扩增的2个目的片段分别用限制性内切酶EcoR Ⅰ、Hind Ⅲ和Sac Ⅰ、Hind Ⅲ双酶切,与相同酶切后的载体pET28a相连接,通过42 ℃热击转化感受态E.coli BL21(DE3)细胞。菌落PCR验证正确,并提取正确转化子的质粒进行双酶切验证。结果如图1-c和图1-d所示,质粒pET28a-lsD1经EcoR Ⅰ、Hind Ⅲ 酶切后出现2个片段,与理论片段大小位置5 000、2 500 bp相符,经过测序验证正确,表明重组菌株BL21(DE3)/pET28a-lsD1构建成功。重组菌株BL21(DE3)/pET28a-jmjC通过同样方法验证正确。
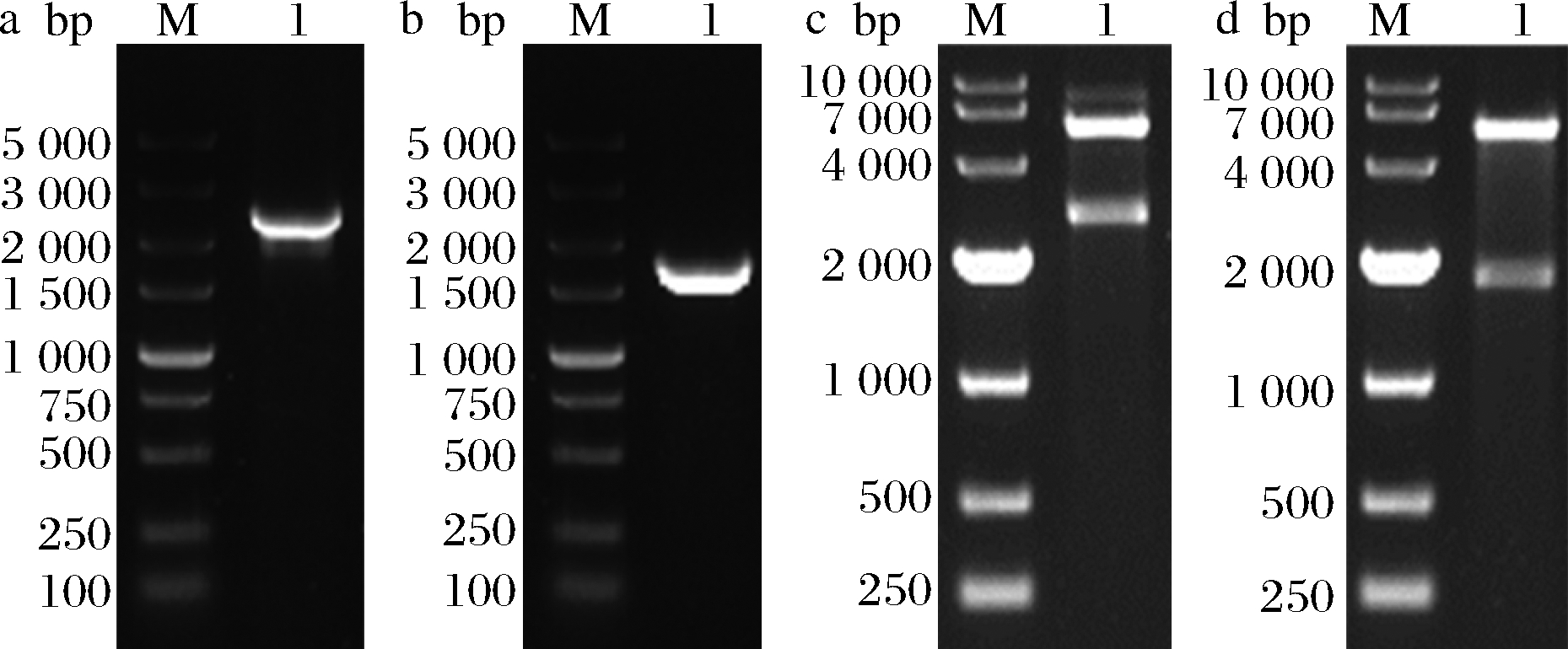
M-DNA marker
a- lsD1基因PCR扩增产物;b- jmjC基因PCR扩增产物;c-重组质粒PET28a-lsD1双酶切验证;d-重组质粒
PET28a- jmjC双酶切验证
图1 lsD1和jmjC基因的PCR扩增产物及双酶切验证
Fig.1 PCR amplification product of lsD1 and jmjC gene, double restriction digestion verification of recombinant vector PET28a-lsD1 and PET28a-jmjC
2.2 组蛋白赖氨酸脱甲基酶诱导表达、纯化
发酵温度过高或者诱导剂浓度过高在蛋白表达过程中会导致目的蛋白的快速、错误折叠,形成包涵体,无法或少量获得可溶性目的蛋白。因此,本文对发酵温度以及诱导剂浓度进行了优化。根据SDS-PAGE分析,在37 ℃条件下,目的蛋白主要以包涵体的形式存在,可溶性蛋白量过低,包涵体复性对于酶活力损失严重;16 ℃条件下,目的蛋白表达量过低;25 ℃条件下,获得了大量的可溶性目的蛋白。为进一步减少包涵体的形成,进行了诱导剂浓度优化,0.10 mmol/L IPTG条件下形成的包涵体明显少于0.25 mmol/L IPTG条件下。因此,本文选择发酵温度25 ℃,0.1 mmol/L IPTG,获得了大量的可溶性目的蛋白。
为了得到高纯度的目的蛋白,选用镍柱亲和层析。通过比较在目的蛋白N端、C端、N端和C端添加6×His标签3种情况的蛋白亲和力,最终选择在目的蛋白C端添加标签。Lsd1(110 kDa)在100 mmol/L咪唑的条件下被洗脱,Jmjc(65 kDa)在200 mmol/L咪唑的条件下被洗脱,结果如图2所示。
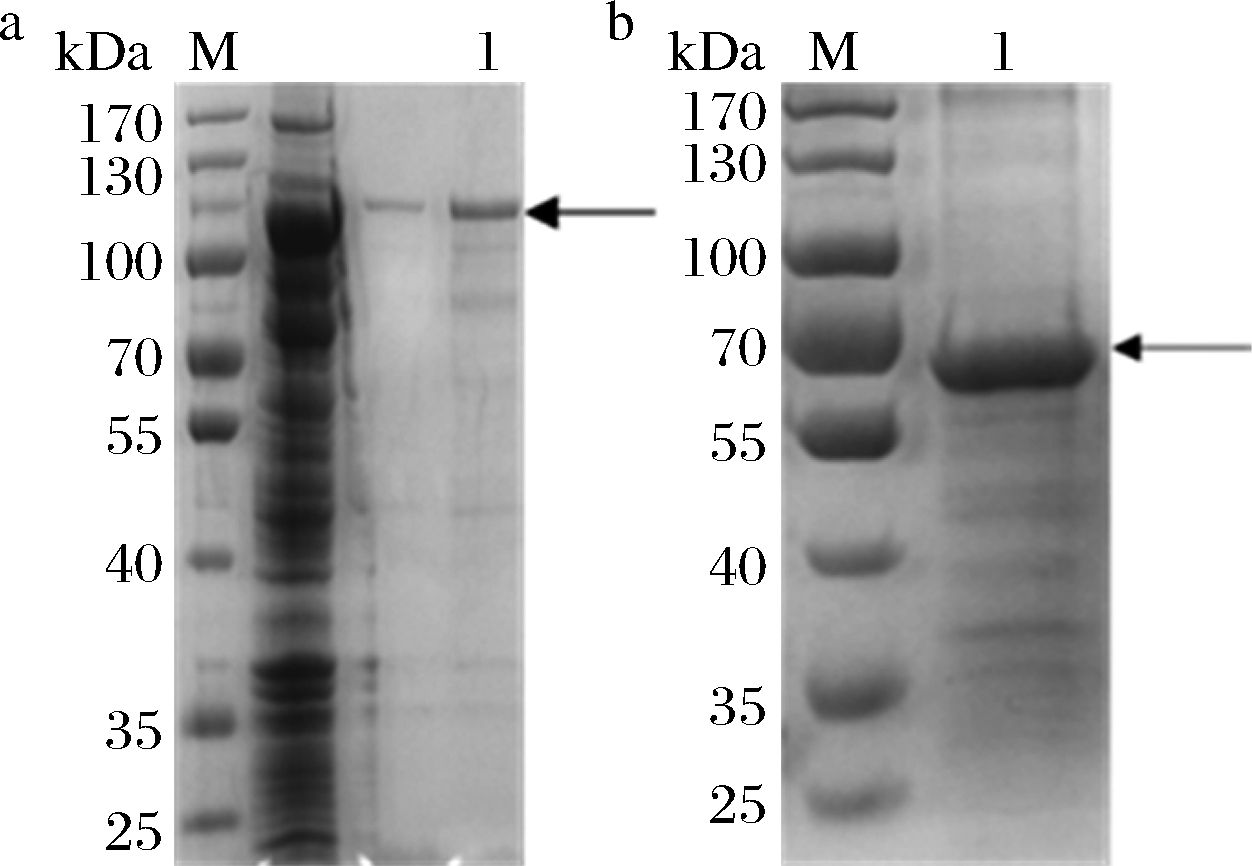
M-蛋白marker
a-Lsd1纯化产物;b-Jmjc纯化产物
图2 重组菌BL21(DE3)/PET28a-lsD1和BL21(DE3)/PET28a-jmjC纯化产物的SDS-PAGE分析
Fig.2 SDS-PAGE analysis of the purified product of recombinant E.coli BL21(DE3)/pET28a-lsD1 and BL21(DE3)/PET28a-jmjC
2.3 重组组蛋白赖氨酸脱甲基酶酶学性质研究
2.3.1 最适反应温度
将酶液分别放置在10、15、20、25、30、35、40、45、55 ℃下进行反应,测定酶活力,以最高酶活力为100%。结果如图3-a所示,Jmjc最适反应温度为20 ℃,Lsd1最适反应温度为35 ℃。Jmjc在温度较低时酶活力依然较高,在达到35 ℃时酶活力快速下降。Lsd1在温度较低时酶活力较低,随着温度升高酶活力逐渐提高,但是在达到45 ℃时酶活力快速下降。
2.3.2 温度稳定性
将酶液分别在10、20、30、40、50、60、70 ℃条件下保存30 min,然后分别在2个酶的最适反应温度下测定剩余酶活力,以最高酶活力为100%。结果如图3-b所示,2个酶在10~40 ℃下比较稳定,几乎没有酶活力损失,酶活力剩余90%以上,高于40 ℃时热稳定性较差,酶活力快速下降,与Jmjc相比,Lsd1具有更好的热稳定性。
2.3.3 最适pH值
2种酶分别在pH 4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0,最适温度条件下反应,以最高酶活力为100%。结果如图3-c所示,Lsd1最适pH值为7.0,在酸性和碱性条件下,酶活力都大量损失,剩余酶活力20%左右。Jmjc最适pH值为8.0,在酸性条件下酶活力很低,pH 4.0~6.0时酶活力剩余30%以下,对于碱性有一定的偏好性,pH 10.0以上酶活力损失较大,与Lsd1相比,Jmjc的酸碱耐受力更强。
2.3.4 pH稳定性
将酶液分别在pH 4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0、12.0,25 ℃条件下保存12 h,并在各自最适温度、最适pH条件下反应测定酶活力,以最高酶活力为100%。结果如图3-d所示,Lsd1和Jmjc在pH 7.0、8.0、9.0剩余酶活力较高,在酸碱环境下耐受性比较差,所以2种酶适合在中性以及弱碱性环境下保存。
2.3.5 金属离子对酶活力的影响
金属离子对Lsd1和Jmjc的作用效果各不相同,结果如图3-e和图3-f所示,Mn2+对Lsd1有明显的促进作用,随着浓度的升高Co2+也表现出一定的促进作用,而Mg2+、Ca2+、Zn2+、Ni2+表现出了明显的抑制作用;同样Mn2+对Jmjc起着明显的促进作用,随着浓度升高Co2+、Ca2+、Zn2+也较小幅度的提高了酶活力,但是随着浓度的持续升高Co2+、Ca2+又表现出了抑制作用,而Mg2+、Ni2+则表现出明显的抑制作用。
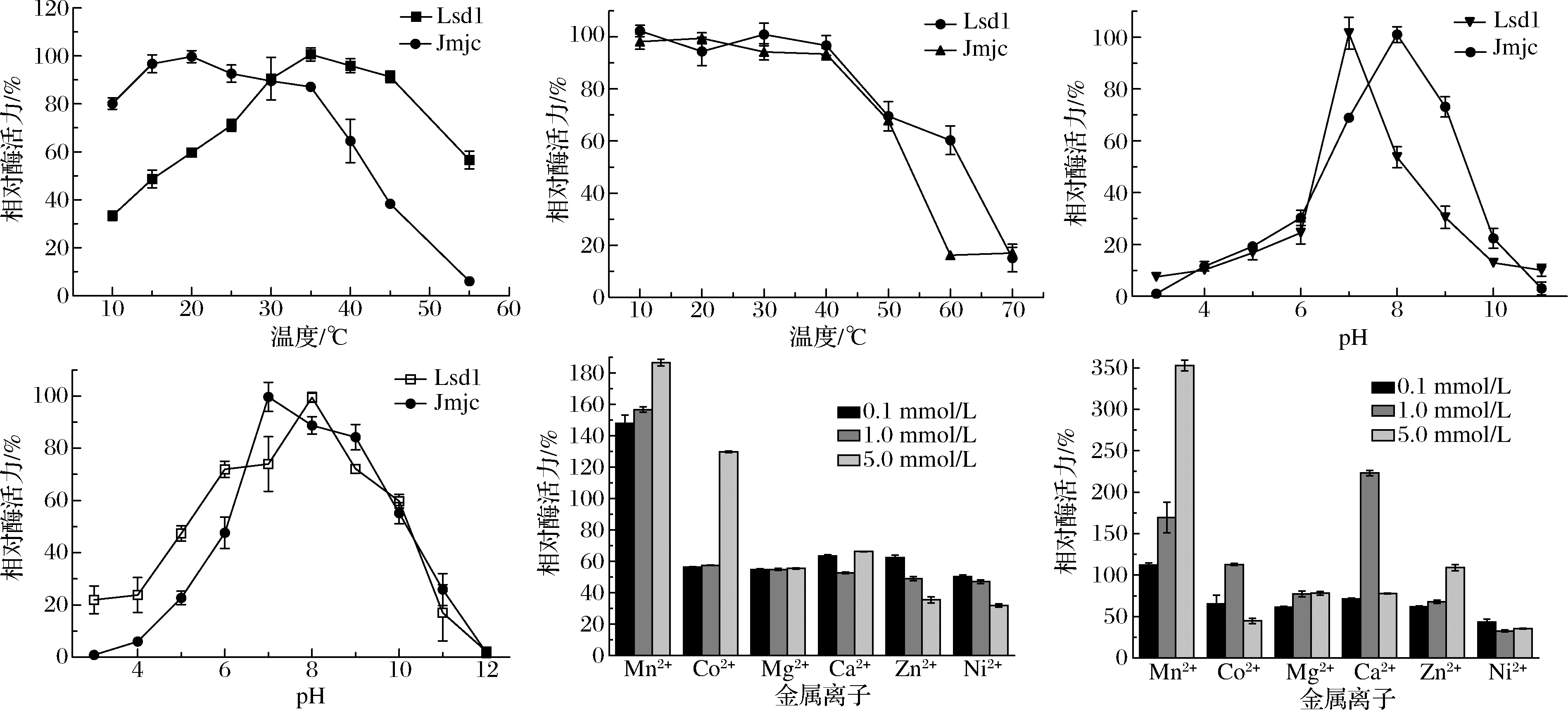
a-最适反应温度;b-温度稳定性;c-最适pH;d-pH稳定性;e-金属离子对Lsd1的影响;f-金属离子对Jmjc的影响
图3 温度、pH、金属离子对酶活力的影响
Fig.3 The influence of temperature, pH and metal ions on enzyme activity
2.3.6 动力学研究
以N-甲基-L-亮氨酸为底物测定反应动力学特性,结果如图4所示,测得Lsd1米氏方程为Y=1 321 480X+1 260 990,米氏常数Km为1.048 mmol/L,Vmax为7.9×10-7 mmol/(L·min),并计算得到kcat/Km分别为8.23×10-6 L/(mol·s);Jmjc米氏方程为Y=1 275 520X+829 909,米氏常数Km为1.537 mmol/L,Vmax为1.2×10-6 mmol/(L·min),并计算得到kcat/Km分别为1.06×10-5 L/(mol·s)。可以看出2个酶对于底物的亲和力较优,但是由于该类酶催化时间较长导致催化效率过低。通过两者比较,Jmjc的催化能力相对优异。
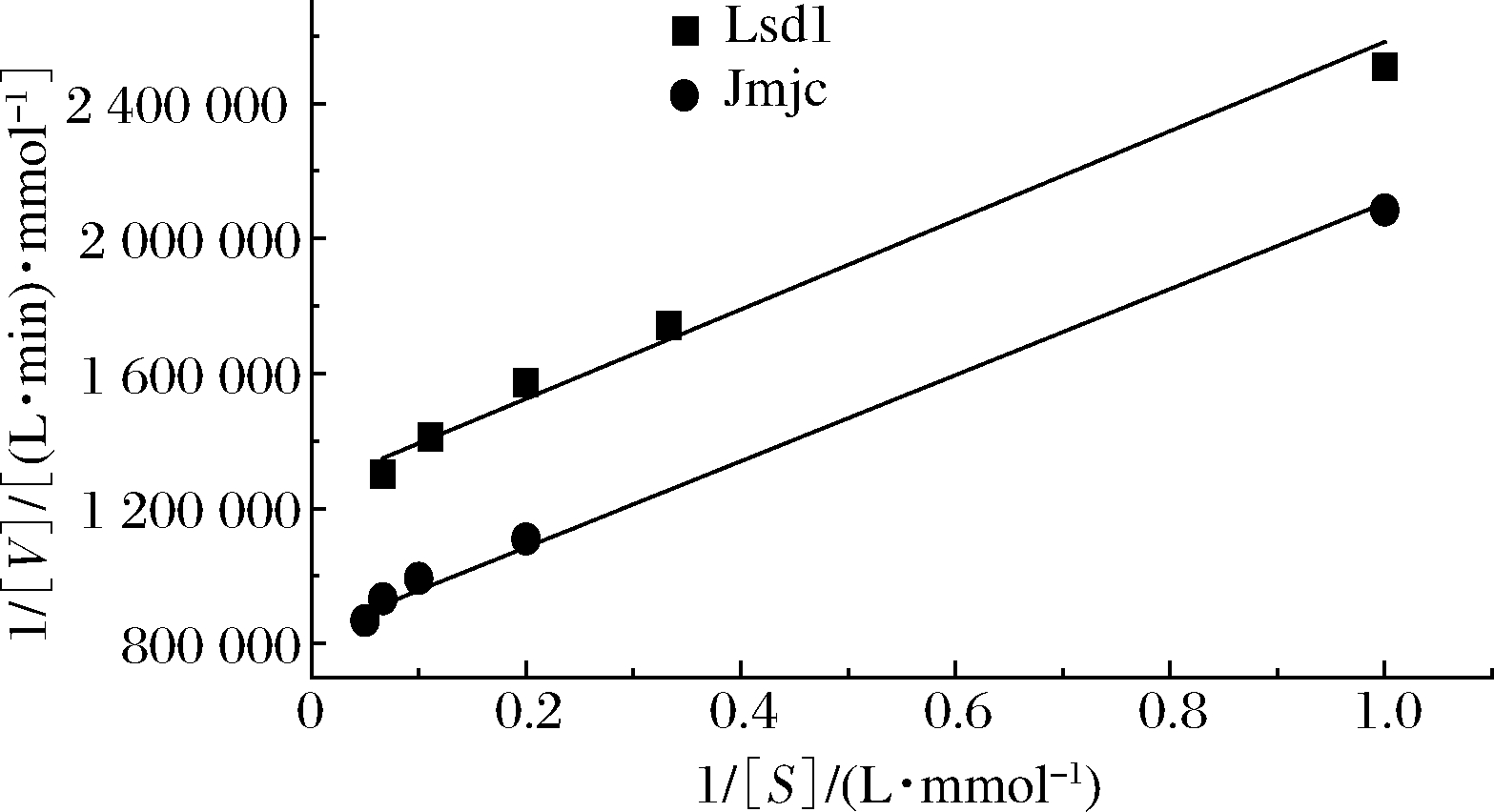
图4 Lsd1和Jmjc降解N-甲基-L-亮氨酸的动力学曲线
Fig.4 Kinetic curve of N-methyl-L-leucine degradation by Lsd1 and Jmjc
3 结论与讨论
本文进行了KDMs的原核表达以及酶学性质的研究。通过异源表达以及镍柱亲和层析成功获得纯化的KDMs,对该酶进行了酶学性质表征,结果显示Lsd1、Jmjc的最适反应温度分别为35、20 ℃,最适反应pH分别为7.0、8.0,Lsd1的温度稳定性相对较高,Jmjc的酸碱耐受力更强;其中Mn2+对Lsd1和Jmjc的酶活力都有明显的促进作用,Co2+、Mg2+、Ca2+、Zn2+、Ni2+对两者有不同程度的抑制作用;动力学研究发现,由于反应时间较长,Lsd1和Jmjc的催化效率较低,kcat/Km分别为8.23×10-6、1.06×10-5 L/(mol·s)。
本研究首次利用非组蛋白底物对组蛋白赖氨酸脱甲基酶的酶学性质进行表征,且根据前期的探索确定,KDMs可以用于含N—CH3化合物的降解。根据报道发现,部分微生物可以直接降解该类化合物,例如假单胞菌GSC 1182[24]可以在48 h后降解80%的咖啡因,但是微生物降解底物单一且菌株种类少,因此具备广谱底物的生物酶法更具优势。与同样可以降解该类物质的Hpo酶[25]相比,该酶的最适反应温度与Jmjc相同为20 ℃,但是Hpo随着温度升高酶活力快速下降,30 ℃时酶活力就损失了20%,在应用方面有很大的局限性,而Lsd1和Jmjc在15~35 ℃酶活力差异较小,45 ℃时酶活力才损失20%,可以用于温度相对较高的降解条件。同时Hpo酶[25]的最适反应pH为6.5,在pH值5.0~7.0,酶活性损失很小,但是碱性条件下酶活力大量损失,降解效率很低,而Lsd1和Jmjc在中性以及碱性的环境中酶活力损失很小,对于N—CH3污染物的中性以及碱性条件降解有很大的应用意义。
同样,两种酶的温度稳定性明显优于已报道的N—CH3降解酶HspB[26],HspB在高于30 ℃时酶活力损失85%以上,而Lsd1和Jmjc在50 ℃时酶活力仅损失30%左右,温度耐受性更好,应用范围更宽泛。此外,与HspB酶[26]相比,2种酶的酸碱耐受性相对优势,酶更加稳定。
但是与Hpo、HspB相比,KDMs的底物亲和力较差,且反应时间较长,因为该酶在体内维持代谢途径平衡对于高催化效率的需求较低,后期可以尝试通过酶的定向改造,提高酶的反应速度以及进一步扩大底物谱, 更有效的降解化学方法难以解决的N—CH3污染物。
[1] SHI Y J, LAN F, MATSON C, et al.Histone demethylation mediated by the nuclear amine oxidase homolog LSD1 [J].Cell, 2004, 119(7):941-953.
[2] CULHANE J C, COLE P A.LSD1 and the chemistry of histone demethylation [J].Current Opinion in Chemical Biology, 2007, 11(5):561-568.
[3] FORNERIS F, BINDA C, VANONI M A, et al.Histone demethylation catalysed by LSD1 is a flavin-dependent oxidative process [J].FEBS Letters, 2005, 579(10):2 203-2 207.
[4] BINDA C, MATTEVI A, EDMONDSON D E.Structure-function relationships in flavoenzyme-dependent amine oxidations:A comparison of polyamine oxidase and monoamine oxidase [J].The Journal of Biological Chemistry, 2002, 277(27):23 973-23 976.
[5] TSUKADA Y I, FANG J, ERDJUMENT-BROMAGE H, et al.Histone demethylation by a family of JmjC domain-containing proteins [J].Nature, 2006, 439(7 078):811-816.
[6] HOU H F, YU H T.Structural insights into histone lysine demethylation [J].Current Opinion in Structural Biology, 2010, 20(6):739-748.
[7] MOSAMMAPARAST N, SHI Y.Reversal of histone methylation:Biochemical and molecular mechanisms of histone demethylases[J].Annual Review of Biochemistry, 2010, 79:155-179.
[8] LEE M G, WYNDER C, COOCH N, et al.An essential role for CoREST in nucleosomal histone 3 lysine 4 demethylation [J].Nature, 2005, 437(7 057):432-435.
[9] LAURENT B, RUITU L, MURN J, et al.A specific LSD1/KDM1A isoform regulates neuronal differentiation through H3K9 demethylation [J].Molecular Cell, 2015, 57(6):957-970.
[10] NICHOLSON T B, CHEN T P.LSD1 demethylates histone and non-histone proteins [J].Epigenetics, 2009, 4(3):129-132.
[11] BURG J M, LINK J E, MORGAN B S, et al.KDM1 class flavin-dependent protein lysine demethylases [J].Peptide Science, 2015, 104(4):213-246.
[12] HUANG J, SENGUPTA R, ESPEJO A B, et al.p53 is regulated by the lysine demethylase LSD1 [J].Nature, 2007, 449(7 158):105-108.
[13] ZHANG J, YUAN B F, ZHANG F, et al.Cyclophosphamide perturbs cytosine methylation in jurkat-T cells through LSD1-mediated stabilization of DNMT1 protein [J].Chemical Research in Toxicology, 2011, 24(11):2 040-2 043.
[14] HUANG Y, STEWART T M, WU Y, et al.Novel oligoamine analogues inhibit lysine-specific demethylase 1 and induce reexpression of epigenetically silenced genes [J].Clinical Cancer Research, 2009, 15(23):7 217-7 228.
[15] HUANG Y, GREENE E, MURRAY STEWART T, et al.Inhibition of lysine-specific demethylase 1 by polyamine analogues results in reexpression of aberrantly silenced genes [J].Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(19):8 023-8 028.
[16] KANOUNI T, SEVERIN C, CHO R W, et al.Discovery of CC-90011:A potent and selective reversible inhibitor of lysine specific demethylase 1 (LSD1) [J].Journal of Medicinal Chemistry, 2020, 63(23):14 522-14 529.
[17] LAURENT B, SHI Y.Expression, purification, and biochemical analysis of the LSD1/KDM1A histone demethylase[J].Methods in Enzymology, 2016, 573:241-259.
[18] 翁新春, 方薇, 陈祎斐.食品工业废水处理工艺及节能探究 [J].资源节约与环保, 2015 (2):63;65.
WENG X C, FANG W, CHEN Y F.Study on wastewater treatment technology and energy saving in food industry[J].Resources Economization & Environmental Protection, 2015 (2):63;65.
[19] 郑元锋. 食品工业废水的生化处理与回用技术 [J].食品界, 2020 (6):104-105.
ZHENG Y F.Biochemical treatment and reuse technology of food industry wastewater [J].Food Industry, 2020 (6):104-105.
[20] 刘梦佳. 食品工业废水处理技术综述 [J].建筑与预算, 2019(4):32-36.
LIU M J.Summary of wastewater treatment technology in food industry [J].Architecture and Budget, 2019(4):32-36.
[21] DASH S S, GUMMADI S N.Enhanced biodegradation of caffeine by Pseudomonas sp.using response surface methodology [J].Biochemical Engineering Journal, 2007, 36(3):288-293.
[22] KOKURA K, SUN L D, FANG J.In vitro histone demethylase assays [J].Methods in Molecular Biology (Clifton, N.J.), 2015, 1 288:109-122.
[23] KING O N F, LI X S, SAKURAI M, et al.Quantitative high-throughput screening identifies 8-hydroxyquinolines as cell-active histone demethylase inhibitors [J].PLoS One, 2010, 5(11):e015535.
[24] GOKULAKRISHNAN S, CHANDRARAJ K, GUMMADI S N.A preliminary study of caffeine degradation by Pseudomonas sp.GSC 1182 [J].International Journal of Food Microbiology, 2007, 113(3):346-350.
[25] TANG H Z, YAO Y X, WANG L J, et al.Genomic analysis of Pseudomonas putida:Genes in a genome island are crucial for nicotine degradation [J].Scientific Reports, 2012, 2:377.
[26] TANG H Z, YAO Y X, ZHANG D K, et al.A novel NADH-dependent and FAD-containing hydroxylase is crucial for nicotine degradation by Pseudomonas putida [J].Journal of Biological Chemistry, 2011, 286(45):39 179-39 187.