非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)感染家猪和野猪引起的一种急性出血性严重传染病。其主要特点是病程短、高热、皮肤及内脏出血、急性感染死亡率高[1]。世界动物卫生组织已将ASF列为A类动物传染病,目前尚无有效的预防疫苗[2]。ASF的持续传播不仅影响到居民的肉类供应,也影响到全球肉类供应的安全[3]。
目前检测ASFV的金标准检测方法是病毒分离,但存在耗时长、成本高等不足[4]。血清学检测也是检测ASFV的主要方法[5],如ELISA和荧光抗体试验,但是不适合早期、快速检测ASFV (抗体在感染后约14 d产生,检测过程存在延迟)[6-7]。胶体金技术虽然快速简单,但是灵敏度相对较低[8]。分子生物学方法比如PCR[9]、实时PCR[10],逆转录PCR[11-12]和线性指数PCR[13]因其高灵敏度被广泛应用于检测ASFV。然而,PCR方法存在耗时、设备昂贵和容易出现假阳性结果等问题。目前也出现了一些用于ASFV检测的等温扩增技术,环介导等温扩增[14-15],重组酶聚合酶扩增[16]和交叉引物扩增[17]方法简单、快速,但是存在假阳性结果和相对复杂的末端检测。以上方法大部分用于猪血、脏器等临床标本。目前有关直接检测食品中ASFV的研究很少,在日常食品中检测出ASFV可以追踪病毒来源,更广泛地监测病毒,进一步减少经济损失。
荧光共振能量转移(fluorescence resonance energy transfer,FRET)是一对光敏分子之间的非辐射能量转移过程。FRET是一种与距离有关的物理现象,能量通过分子内的偶极-偶极相互作用从激发的供体荧光团转移到受体荧光团。能量转移可以发生的距离被限制在10 nm左右。受体分子不一定要以荧光的方式释放能量(比如荧光淬灭)。当受体发色团作为供体的淬灭剂时,供体荧光强度会由于FRET而降低或消失[18]。FRET技术因其检测简便、快速、灵敏等优点,被广泛应用于多种靶标物的检测[19],如小分子[20]、细菌[21]、病毒[22]、细胞[23]等。本研究开发了一种利用量子点(quantum dots,QDs)和纳米金(Au nanoparticles,AuNPs)构建基于FRET的DNA生物传感器,用于ASFV特异性基因序列检测。在靶DNA缺失的情况下,ss-DNA-QDs(探针1)将与ss′-DNA-AuNPs(探针2)杂交,供体QDs与受体AuNPs距离变近,引发FRET效应,QDs的荧光被AuNPs淬灭。然而,在靶DNA存在的情况下,靶DNA与探针2竞争结合探针1,导致FRET被破坏,QDs的荧光恢复。基于FRET的DNA生物传感器为食品安全研究提供了简单、快速和特异的ASFV检测方法。
1 材料与方法
1.1 材料与试剂
猪肉、火腿、猪肉水饺, 中国重庆北碚永辉超市; 病毒核酸提取试剂盒,北京明日达科技发展有限公司。
HAuCl4(分析纯),国药化学试剂有限公司;链霉亲和素-QDs525(分析纯),中国武汉珈源量子点有限公司;非洲猪瘟病毒特异性基因(靶DNA根据标准T/CVMA 5—2018实时荧光PCR检测方法选择ASFV p72基因片段)、随机DNA序列,均为色谱纯,上海生工生物技术有限公司,序列信息如表1所示。所有的缓冲液均用超纯水制备。
表1 序列信息
Table 1 Sequence information

序列名称序列信息靶 DNA5′-CCGATCACATTACCTATTATTAAAAACATT-3′ss-DNA5′-亲和素-TEG-AATGTTTTTAATAATAGGTAATGTGATCGG-3′ss′-DNA5′-SH-C6-CCGATCACATTACCT-3′随机序列5′-GTATGTATGACTAGGAACAGATCGCATGAC-3′
1.2 仪器与设备
FA2004A型电子分析天平,上海精天电子仪器有限公司;F—7000型荧光分光光度计,日本日立公司;UV-2450 型紫外-可见分光光度计,日本岛津公司;Nano ZS ZEN3600型zeta电位及纳米粒度仪,英国 Malvern公司。
1.3 实验方法
1.3.1 ss-DNA-QDs(探针1)的制备
基于链霉亲和素与生物素的特异性亲和力实现ss-DNA与QD的偶联[24],链霉亲和素-QDs525(1 μmol/L,10 μL)和生物素-ss-DNA(10 μmol/L,16 μL)同时加入到硼酸缓冲液(borate buffered saline,BBS)中(50 mmol/L,pH 8.4,174 μL),在37 ℃恒温摇床中(150 r/min)反应1 h。用截留分子质量为50 kDa的超滤管4 ℃ 进行纯化,10 000 r/min离心10 min,重悬于50 mmol/L pH 8.4的BBS中,用超滤管回收,3 400 r/min离心2 min。纯化后的探针1置于4 ℃冰箱中避光保存。
1.3.2 AuNPs的制备
采用柠檬酸还原法[25]制备AuNPs。将三颈烧瓶在食人鱼溶液[V(H2SO4)∶V(30%H2O2)=7∶3]中浸泡1 h,超纯水清洗后备用。将50 mL去离子水和0.5 mL HAuCl4 (质量分数为1%)加入到三颈烧瓶中,用电热套加热搅拌至煮沸,迅速加入1.25 mL柠檬酸三钠(质量分数为1%)溶液,在300 ℃下搅拌反应30 min,关闭热源,继续搅拌直至冷却,最终产品在室温下保存。
1.3.3 ss′-DNA-AuNPs(探针2)的制备
根据之前的报道[26]进行巯基修饰的DNA与AuNPs的偶联,将巯基修饰的ss′-DNA(27 μL,0.1 mmol/L)与醋酸缓冲液(3 μL,500 mmol/L,pH 5.2)混合,然后用三(2-羧乙基)膦(4.5 μL,10 mmol/L)进行活化,反应在微离心管中于室温下进行1 h。将活化的ss′-DNA加入到AuNPs(2 mL,3 nmol/L)中,轻轻摇匀,室温避光孵育16 h。然后逐滴加入Tris-醋酸缓冲液(22 μL,500 mmol/L,pH 8.2)和NaCl (220 μL,0.1 mol/L),轻轻摇匀,室温避光孵育24 h以上。探针2通过离心纯化15 min(12 000 r/min)。收集底部剩余沉淀,用去离子水冲洗2次,去除未反应的ss′-DNA,溶解于Tris-醋酸缓冲液(100 mmol/L NaCl,25 mmol/L 醋酸盐,pH 8.2),最终产品取200 μL浓缩至60 μL,此时ss′-DNA-AuNPs终浓度为10 nmol/L。
1.3.4 探针的表征
制备15 g/L的琼脂糖凝胶。电泳液为5×TBE(54 g Tris,27.5 g硼酸,20 mL 0.5 mol/L EDTA,pH 8.0)稀释成0.5×TBE备用。琼脂糖(0.45 g)和电泳溶液(30 mL)置于锥形瓶中,微波加热1 min,待琼脂糖溶解后,加入1 μL GoldView核酸染料,冷却成凝胶。ss-DNA-QDs(20 nmol/L,8 μL)与2 μL loading buffer混合均匀之后上样,电压120 V,电泳时间60 min,电泳后取出凝胶,用凝胶成像仪进行成像。利用Nano ZS ZEN3600动态光散射测量了AuNPs的粒度和zeta电位。采用紫外可见分光光度计进行AuNPs吸收光谱测量。使用荧光分光光度计进行QDs发射光谱测量。
1.3.5 基于FRET的DNA传感器标准曲线的建立
将8 μL探针1和20 μL探针2在52 μL Tris-HCl(10 mmol/L Tris-HCl,1 mmol/L EDTA,50 mmol/L NaCl,pH 7.4)中于37 ℃孵育1.25 h。然后将不同浓度(1.0、1.25、1.5、1.75、2.0 μmol/L)的ASFV靶DNA(20 μL)加入体系中于37 ℃孵育1.25 h,用荧光分光光度计测定荧光强度,在相同的条件下记录所有荧光强度,每个实验重复3次。
1.3.6 实际样品分析
在猪肉、火腿香肠和猪肉饺子样品中加入不同浓度的靶DNA(1.0、1.5、2.0 μmol/L)制备加标样本,按照病毒核酸提取试剂盒程序进行预处理。按照基于FRET的DNA传感器的步骤进行检测。
1.3.7 数据处理
所得数据使用Origin 8.0和Excel进行数据处理和绘制相关图表,利用 SPSS 19.0 软件通过单因素方差分析法对数据进行分析,每个样品做3个独立平行。
2 结果与分析
2.1 基于FRET的DNA生物传感器的研究策略
本研究开发了一种基于FRET的DNA传感器,用于ASFV特异性基因序列检测(靶DNA,30 nt,表1)原理如图1所示。靶DNA缺失的情况下,ss-DNA-QDs(探针 1)将根据碱基互补配对原则与ss′-DNA-AuNPs(探针2)杂交,使供体QDs与受体AuNPs接近。在单链DNA构象中,长度约为0.15 nm/nt,而在双链DNA构象中长度约为0.31 nm/nt[27]。当探针2与探针1的15个碱基互补结合时,QDs和AuNPs之间只有15个碱基对和15个碱基,距离约为6.59 nm,小于FRET发生的最大距离10 nm[18],因此触发了FRET效应,AuNPs 淬灭了QDs的荧光。然而,靶DNA存在的情况下,根据DNA链位移动力学[28-29],靶DNA与探针2竞争结合探针1,导致FRET被破坏,QDs的荧光发生恢复。

图1 基于FRET的DNA生物传感器检测ASFV
靶DNA的原理图
Fig.1 Schematic illustration of FRET DNA biosensor
for ASFV target DNA detection
2.2 探针的表征
2.2.1 探针1的表征
探针1根据链霉亲和素与生物素的特异性亲和力进行合成。由图2可知,QDs与ss-DNA结合后电泳速度更快,可能与ss-DNA带有大量负电荷有关。在电泳力的作用下,带更多负电荷的探针1向正极方向游动速度更快。因此QDs与ss-DNA成功结合。

1-Marker;2-QDs;3-ss-DNA;4-探针1
图2 琼脂糖凝胶电泳图
Fig.2 Agarose gel electrophoresis digital map
2.2.2 探针2的表征
通过柠檬酸还原法合成AuNPs,由图3-a可知,AuNPs的粒径约为13 nm。由图3-b可知,AuNPs的zeta电位约为-26.5 mV。考察了一周内AuNPs的特征吸收峰波长和吸收峰波长处的吸光度来证明AuNPs的稳定性,由图3-c可知,吸收峰波长在一周内基本没有变化,较为稳定。由图3-d可知,吸收峰波长处的吸光度在一周内基本保持在0.8 左右,比较稳定。ss′-DNA通过巯基与金的化学作用与AuNPs结合,如图3-e所示,探针2的紫外可见吸收光谱相对于AuNPs略向右移。如图3-f所示,在裸的AuNPs中加入100 g/L NaCl后,由于盐浓度的影响屏蔽了纳米粒子间的静电排斥作用[30],探针2保持分散状态。根据以上数据可知,成功合成了探针2。
2.3 基于FRET的DNA传感器检测ASFV的可行性
图4显示了所提策略的可行性。当引入靶DNA时,靶DNA与探针2竞争结合探针1,导致FRET被破坏。因此,荧光强度增加。而加入随机序列时,随机序列不与探针1结合,FRET没有被破坏,因此荧光没有变化。表明该方案能够特异性地检测靶DNA,证实了所提策略的可行性。
2.4 基于FRET的DNA传感器检测性能的优化
为了获得最佳的检测性能,以荧光强度的变化值作为评价指标,优化序列长度、反应缓冲液、探针1和探针2的用量和孵育时间。首先,当ss′-DNA的长度为15个碱基时,荧光恢复程度最高,说明此时检测性能最好。如图5-a所示,当长度>15碱基时,靶DNA不会竞争性地与探针1杂交,这可能是由于碱基之间增加的Watson-Crick结合力,这使得竞争变得困难[28]。图5-b显示了当反应缓冲液为Tris-HCl(10 mmol/L Tris,1 mmol/L EDTA,50 mmol/L NaCl,pH 7.4)时具有最大荧光恢复值ΔF(ΔF=F-F0,其中F和F0分别为靶DNA存在和缺失时的荧光强度)。因此,选择Tris-HCl(10 mmol/L Tris,1 mmol/L EDTA,50 mmol/L NaCl,pH 7.4)作为反应的最适缓冲液。
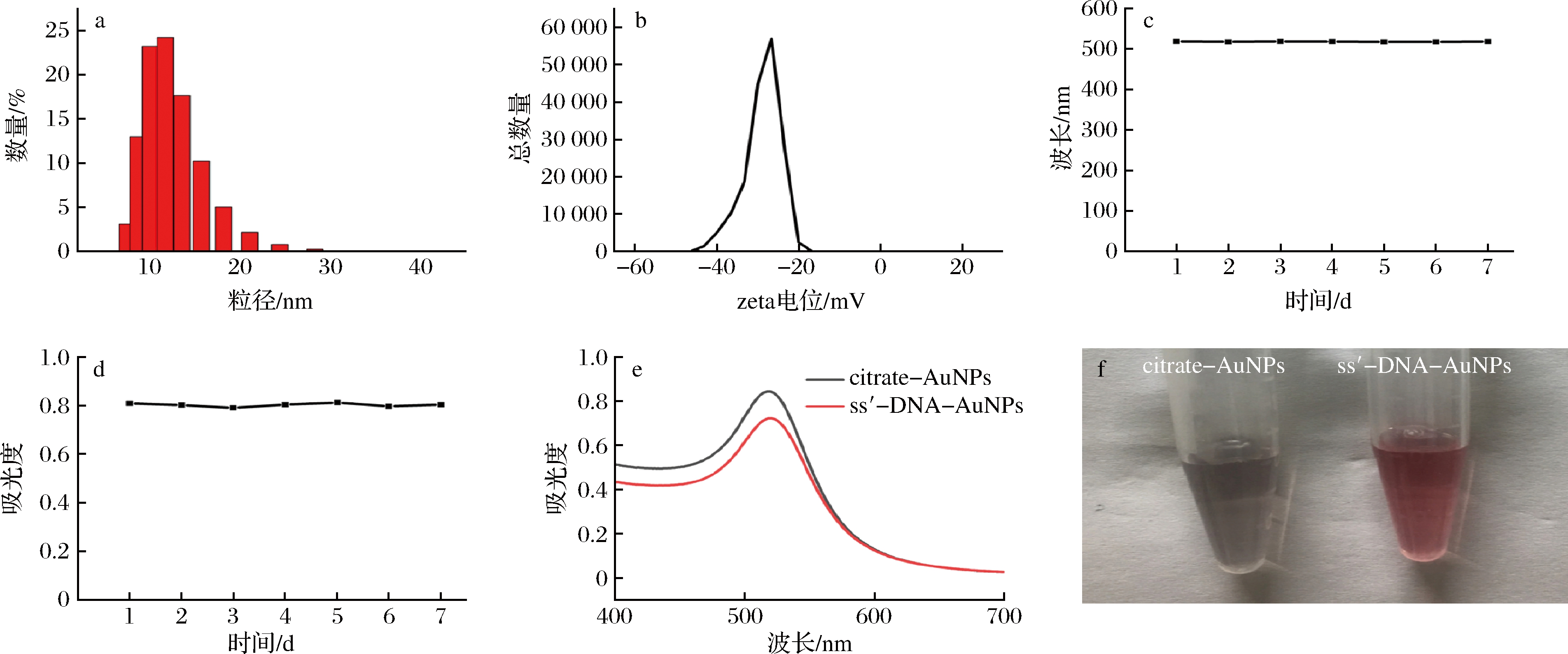
a-AuNPs的粒径分布;b-AuNPs的zeta电位;c-一周内AuNPs的吸收峰波长变化;d-一周内AuNPs吸收峰波长处的吸光度;
e-AuNPs与ss′-DNA-AuNPs的吸收峰波长;f-AuNPs与ss′-DNA-AuNPs的聚集分散状态(添加100 g/L NaCl)
图3 探针2的表征
Fig.3 Characterization of DNA-AuNPs
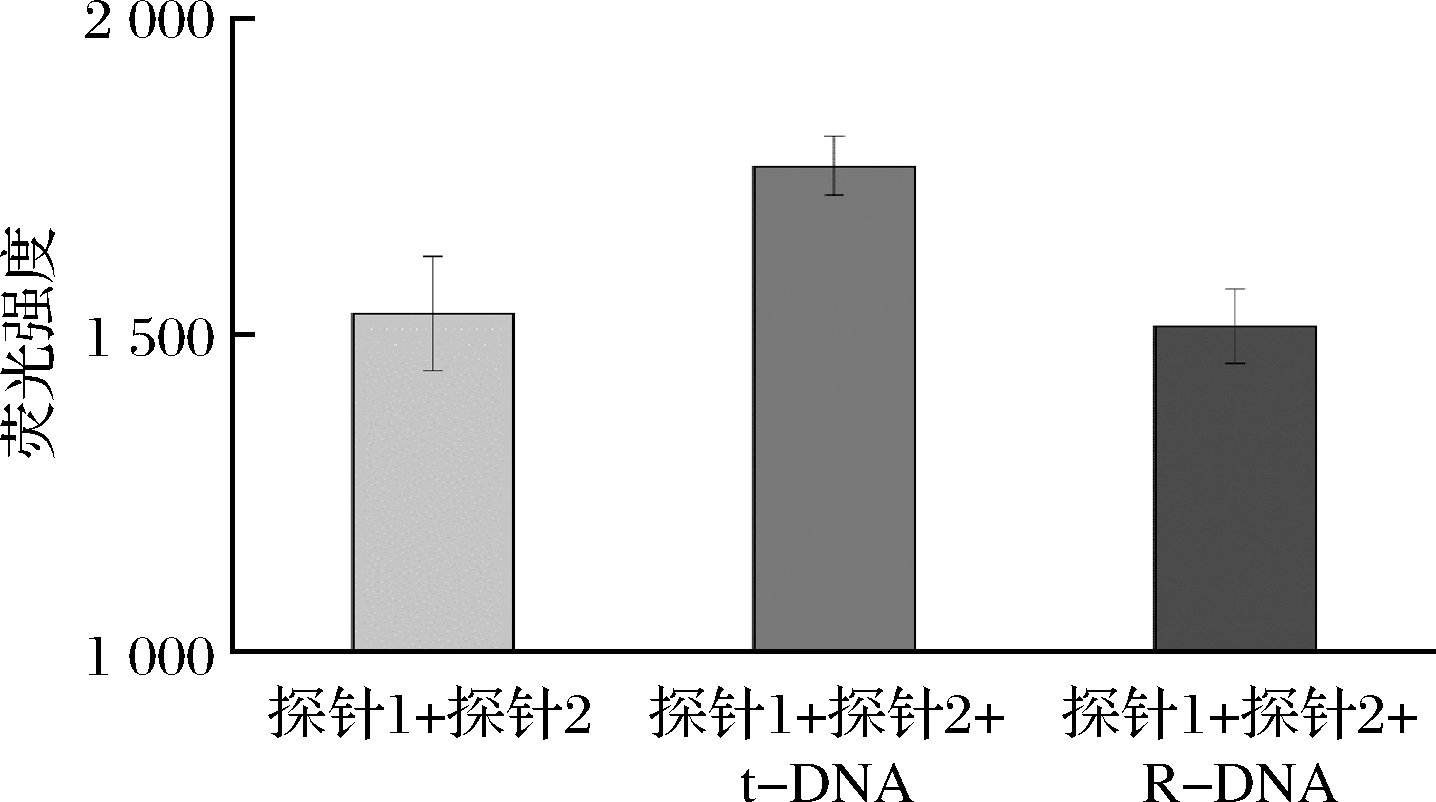
图4 靶DNA(t-DNA)和随机序列(R-DNA)加入探针1
和探针2体系中的的荧光强度
Fig.4 The fluorescence intensity of the system after adding
target DNA (t-DNA) and random sequence (R-DNA)
图5-c中,探针2的浓度为2 nmol/L时,ΔF是最大的。当浓度<2 nmol/L,ΔF较小,这是因为探针1和探针2结合数量少,当浓度>2 nmol/L时,AuNPs浓度升高导致荧光淬灭效率大大提高,但荧光强度恢复不明显。如图5-d所示,当探针1的浓度为4 nmol/L,ΔF最大,浓度<4 nmol/L,QDs数量较少,体系整体荧光强度较低。当浓度>4 nmol/L时,QDs浓度升高导致背景荧光较强,荧光恢复程度较低。最后对孵育时间进行优化,将时间优化分为2个阶段,首先优化探针识别阶段的孵育时间,然后优化加入靶DNA后的竞争时间。如图5-e所示,由于探针1和探针2结合完全,1.25 h后荧光降低值达到最大后保持不变。图5-f中,在1.25 h 后荧光恢复值不再增加,因为靶DNA与探针1达到了最大程度的结合。
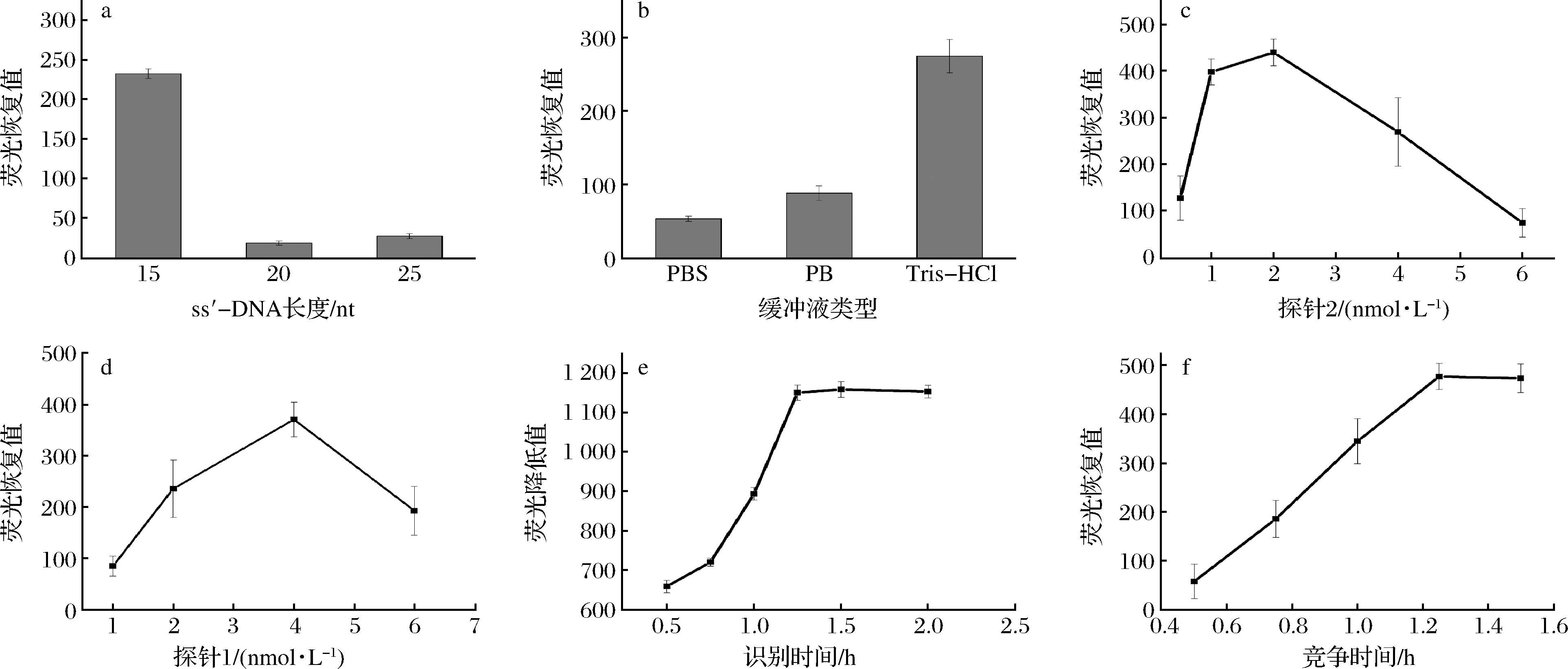
a-ss′-DNA长度优化图;b-缓冲液优化图;c-探针2浓度优化图;d-探针1浓度优化图;e-识别时间优化;f-竞争时间优化
图5 靶DNA检测的优化
Fig.5 Optimization for target DNA detection
2.5 基于FRET的DNA传感器的标准曲线
优化检测条件后,利用基于FRET的DNA传感器检测一系列不同浓度的靶DNA(1.0、1.25、1.5、1.75、2.0 μmol/L),记录荧光变化值。如图6所示,在1~2 μmol/L的范围内,ΔF与靶DNA的浓度之间有良好的线性关系,回归方程为ΔF=180.00Ct-DNA+120.91,回归系数为0.997 9。靶DNA的检出限(LOD)为0.72 μmol/L,由LOD=3S/K计算得出,其中S为空白样品的标准差 (n=10),K为校准曲线的斜率。

图6 基于FRET的DNA传感器的标准曲线
Fig.6 Standard curves of ASFV target DNA
measured by FRET DNA sensor
2.6 实际样品分析
为了验证基于FRET的DNA传感器检测食品中ASFV的适用性和准确性,选择具有代表性的猪肉及其制品,如新鲜猪肉、火腿肠和猪肉饺子等日常食品作为真实样本,在这些常见的食品中实现对非洲猪瘟病毒的检测,有利于追踪病毒来源,更广泛地监测病毒,进一步减少经济损失。在猪肉、火腿肠和猪肉饺子中添加了不同浓度靶DNA(1.0、1.5、2.0 μmol/L),回收率为82.00%~108.00%,变异系数为0.02%~0.15% (表2),说明本文提出的基于FRET的DNA传感器可用于实际食品样品中ASFV的检测。另外,非洲猪瘟病毒是一种较大的双链DNA病毒,通常以稳定的DNA双链形式存在。在样品预处理的过程中,可以通过热处理使双链DNA解旋,然后进行下一步实验。
表2 利用基于FRET的DNA传感器对真实食品样品中
ASFV靶DNA的回收率和准确性研究
Table 2 Recovery and accuracy of ASFV target DNA in
real food samples based on FRET DNA sensor

样品添加量/(μmol·L-1)测得量/(μmol·L-1)回收率/%变异系数(n=3)猪肉11.000.8484.000.14猪肉21.501.3086.670.11猪肉32.002.04102.000.06火腿肠11.000.8282.000.15火腿肠21.501.2986.000.14火腿肠32.002.08104.000.05猪肉水饺11.000.8989.000.02猪肉水饺21.501.2482.670.04猪肉水饺32.002.16108.000.03
3 结论
目前,非洲猪瘟传播形式严峻,且尚无有效的预防疫苗。因此,对非洲猪瘟病毒的检测及防控是非常有必要的。利用QDs和AuNPs构建了一种基于FRET的DNA传感器,用于检测食品样品中的ASFV特异性基因。通过触发和破坏FRET快速检测和定量ASFV靶DNA,可在1.25 h内快速检测ASFV靶DNA,检出限为0.72 μmol/L。在复杂的食品样品(猪肉、火腿肠和猪肉饺子)中验证了基于FRET的DNA传感器的适用性,在猪肉、火腿肠和猪肉饺子中的回收率为82.00%~108.00%,变异系数为0.02%~0.15%。本研究提出一种简单、快速的检测食品中ASFV基因片段的方法,可以有效地用于食品中非洲猪瘟病毒的检测以及监控。在未来的研究当中,可以通过优化条件来提高灵敏度。此外,此策略可以根据不同的靶标物设计不同的探针,可扩展到其他DNA、病毒或蛋白质的传感应用,为食品安全检测方向的进一步研究提供科学依据。
[1] GABRIEL C,BLOME S,MALOGOLOVKIN A,et al.Characterization of African swine fever virus caucasus isolate in European wild boars[J]. Emerging Infectious Diseases, 2011, 17(12):2 342-2 345.
[2] ZAKARYAN H,REVILLA Y.African swine fever virus:Current state and future perspectives in vaccine and antiviral research[J].Veterinary Microbiology,2016,185:15-19.
[3] PENRITH M L,PEREIRA C L,DA SILVA M M R L,et al.African swine fever in Mozambique:Review,risk factors and considerations for control[J].Onderstepoort Journal of Veterinary Research,2007,74(2):149-160.
[4] TUBIASH H S.Quantity production of leukocyte cultures for cultures use in hemadsorption tests with African swine fever virus[J].American Journal of Veterinary Research,1963,24:381-384.
[5] 陈静. 浅谈目前非洲猪瘟常用诊断方法[J].中国畜禽种业,2020,16(10):152-153.
CHEN J.A brief discussion on the common diagnostic methods of African swine fever[J].The Chinese Livestock and Poultry Breeding,2020,16(10):152-153.
[6] GIMENEZ-LIROLA L G,MUR L,RIVERA B,et al.Detection of African swine fever virus antibodies in serum and oral fluid specimens using a recombinant protein 30 (p30) dual matrix indirect ELISA[J].PLoS One,2016,11(9):e0161230.
[7] BERGERON H C,GLAS P S,SCHUMANN K R.Diagnostic specificity of the African swine fever virus antibody detection enzyme-linked immunosorbent assay in feral and domestic pigs in the United States[J].Transboundary and Emerging Diseases,2017,64(6):1 665-1 668.
[8] 张元峰, 刘锡玲,毕路,等.胶体金检测试纸卡在检测非洲猪瘟病毒抗体上的应用[J].养殖与饲料,2020,19(11):15-18.
ZHANG Y F,LIU X L,BI L,et al.Application of colloidal gold test paper card in detection of antibodies against African swine fever virus[J].Animals Breeding and Feed,2020,19(11):15-18.
[9] AGUERO M,FERNANDEZ J,ROMERO L,et al.Highly sensitive PCR assay for routine diagnosis of African swine fever virus in clinical samples[J].Journal of Clinical Microbiology,2003,41(9):4 431-4 434.
[10] FERNANDEZ-PINERO J,GALLARDO C,ELIZALDE M,et al.Molecular diagnosis of African swine fever by a new real-time PCR using universal probe library[J].Transboundary and Emerging Diseases,2013,60(1):48-58.
[11] HAINES F J,HOFMANN M A,KING D P,et al.Development and validation of a multiplex,real-time RT PCR assay for the simultaneous detection of classical and African swine fever viruses[J].PLoS One,2013,8(7):e71019.
[12] AGUERO M,FERNANDEZ J,ROMERO L J,et al.A highly sensitive and specific gel-based multiplex RT-PCR assay for the simultaneous and differential diagnosis of African swine fever and classical swine fever in clinical samples[J].Veterinary Research,2004,35(5):551-563.
[13] RONISH B,HAKHVERDYAN M,STAHL K,et al.Design and verification of a highly reliable Linear-After-The-Exponential PCR (LATE-PCR) assay for the detection of African swine fever virus[J].Journal of Virological Methods,2011,172(1-2):8-15.
[14] JAMES H E,EBERT K,MCGONIGLE R,et al.Detection of African swine fever virus by loop-mediated isothermal amplification[J].Journal of Virological Methods,2010,164(1-2):68-74.
[15] 王林, 高晓龙,吴迪,等.非洲猪瘟病毒实时荧光LAMP检测方法的建立与应用[J].中国兽药杂志,2020,54(8):1-8.
WANG L,GAO X L,WU D,et al.Establishment and application of real-time fluorescent LAMP detection method for African swine fever virus[J].Chinese Journal of Veterinary Drug,2020,54(8):1-8.
[16] WANG J C,WANG J F,GENG Y Y,et al.A recombinase polymerase amplification-based assay for rapid detection of African swine fever virus[J].Canadian Journal of Veterinary Research,2017,81(4):308-312.
[17] FRACZYK M,WOZNIAKOWSKI G,KOWALCZYK A,et al.Development of cross-priming amplification for direct detection of the African swine fever virus,in pig and wild boar blood and sera samples[J].Letters in Applied Microbiology,2016,62(5):386-391.
[18] SINGURU M M R,SUN S C,CHUANG M C.Advances in oligonucleotide-based detection coupled with fluorescence resonance energy transfer[J].Trac-Trends in Analytical Chemistry,2020,123:115 756.
[19] ZHANG X J,HU Y,YANG X T,et al.Förster resonance energy transfer (FRET)-based biosensors for biological applications[J].Biosensors & Bioelectronics,2019,138:111 314.
[20] LI C C,LI Y,ZHANG Y,et al.Single-molecule fluorescence resonance energy transfer and its biomedical applications[J].Trac-Trends in Analytical Chemistry,2020,122:115 753.
[21] JIN B R,WANG S R,LIN M,et al.Upconversion nanoparticles based FRET aptasensor for rapid and ultrasenstive bacteria detection[J].Biosensors & Bioelectronics,2017,90:525-533.
[22] LU M L,MA X C,MOTHES W.Illuminating the virus life cycle with single-molecule FRET imaging[J].Advances in Virus Research,2019,105:239-273.
[23] ZHANG W,LIU X L,LI P,et al.Cellular fluorescence imaging based on resonance energy transfer[J].Trac-Trends in Analytical Chemistry,2020,123:115 742.
[24] HUANG H P,ZHU J J.DNA aptamer-based QDs electrochemiluminescence biosensor for the detection of thrombin[J].Biosensors & Bioelectronics,2009,25(4):927-930.
[25] FRENS G.controlled nucleation for regulation of particle-size in monodisperse gold suspensions[J].Nature Physical Science,1973,241(105):20-22.
[26] LIU J W,LU Y.Preparation of aptamer-linked gold nanoparticle purple aggregates for colorimetric sensing of analytes[J].Nature Protocols,2006,1(1):246-252.
[27] GUO J J,QIU X,MINGOES C,et al.Conformational details of quantum dot-DNA resolved by Förster Resonance Energy Transfer lifetime nanoruler[J].ACS Nano,2019,13(1):505-514.
[28] ZHANG D Y,WINFREE E.Control of DNA strand displacement kinetics using toehold exchange[J].Journal of the American Chemical Society,2009,131(47):17 303-17 314.
[29] WANG D,CHEN H,LI H,et al.Detection of Staphylococcus aureus carrying the gene for toxic shock syndrome toxin 1 by Quantum-Dot-Probe complexes[J].Journal of Fluorescence,2011,21(4):1 525-1 530.
[30] LIU B W,LIU J W.Freezing directed construction of Bio/Nano Interfaces:Rreagentless conjugation,denser spherical nucleic acids,and better nanoflares[J].Journal of the American Chemical Society,2017,139(28):9 471-9 474.