灵菌红素 (prodigiosin,PG)是一类含三吡咯骨架结构的天然红色素,主要由粘质沙雷氏菌(Serratia marcescens)、天蓝色链霉菌等微生物产生,具有抗癌、免疫抑制、抗虫等多种生物活性,可作为免疫抑制剂和抗癌药物进行开发,受到研究者的广泛关注[1-3]。S. marcescens可以在30 ℃下高效生产灵菌红素,但在37 ℃或更高温度下几乎无法生产[4-5]。如图1所示,在S. marcescens中,灵菌红素的合成受分叉路径的控制,2-甲基-3-n-戊基吡咯(2-methyl-3-n-amyl-pyrrole, MAP)和4-甲氧基-2,2′-联吡咯-5-甲醛(4-methoxy-2,2′-bipyrrole-5-carbaldehyde, MBC)在PigC的作用下缩合生成灵菌红素[6]。MBC分支的最后一步是由PigF和PigN催化,将甲基转移至4-羟基-2,2′-联吡咯-5-甲醛(4-hydroxy-2,2′-bipyrrole-5-carbaldehyde, HBC)形成MBC [7]。

图1 粘质沙雷氏菌灵菌红素生物合成途径
Fig.1 Prodigiosin biosynthetic pathway in S.marcescens
聚腺苷酸化是指将未模板化的腺苷残基添加到RNA底物的3′末端的酶促过程[8-9]。尽管早在1962年就在大肠杆菌中发现了第一个poly(A)聚合酶,但多年来细菌中的聚腺苷酸化很少受到关注[10]。聚腺苷酸化广泛用于控制RNA代谢,包括mRNA代谢回转和RNA稳定性监测[11]。在大肠杆菌中,在poly(A)聚合酶的作用下,poly(A)尾巴可在mRNA的3′端形成[12]。先前的研究表明,加工过的转录本和3′末端的稳定RNA结构可以赋予3′-5′核酸外切酶抗降解的能力,因此可以保护邻近的上游区域免受体内核酸外切降解的影响[13]。但是,与真核生物不同的是,在细菌mRNA中只有一小部分是聚腺苷酸化的,而实际的聚腺苷酸化程度只有少数转录本,其水平可低至0.4%~10%[9,14-15]。
在本研究中,选择实验室前期筛选得到并能合成PG的粘质沙雷氏菌JNB5-1为出发菌株,利用发酵培养基发酵产生5.36 g/L的灵菌红素。对粘质沙雷氏菌来源的氧甲基转移酶(O-methyltransferase,PigF)进行表达,并通过在粘质沙雷氏菌JNB5-1灵菌红素合成基因簇pigF 3′端添加poly(A/T)尾来提高pigF转录水平和蛋白表达水平。最终,发酵最高可产生6.21 g/L灵菌红素。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
本研究所用粘质沙雷氏菌JNB5-1、JNB5-1ΔpigF、大肠杆菌(Escherichia coil)BL21 (DE3)、大肠杆菌S17-1 λ pir及质粒pET-28a、pETC-28a、pUT-km均由本实验室保藏。大肠杆菌S17-1 λ pir/pUT-km-pigFP-Apm、JNB5-1F、JNB5-1FP1~JNB5-1FP6,JNB5-1ΔpigFF、JNB5-1ΔpigFFP1~JNB5-1ΔpigFFP6及JNB5-1/pigFFP菌株,为本文构建。
1.1.2 主要实验试剂
限制性内切酶、λ-Hind III digest DNA Marker、DL10000 DNA Marker、DL2000 DNA Marker、蛋白marker,大连宝生物公司;高保真酶、同源重组酶克隆试剂盒、RNA试剂盒、反转录试剂盒及荧光定量PCR试剂盒,南京诺维赞生物科技有限公司;其他试剂均为国产分析纯。
1.1.3 PCR引物设计
本研究中PCR扩增所用引物通过苏州金唯智生物科技有限公司来合成,引物如表1所示。
表1 本研究中所用引物
Table 1 Primers used for PCR in this study

引物名称引物DNA序列(5′-3′)pigF-FatgggtcgcggatccgaattcATGCCTTTAACCAAGCAAGATGCpigF-RctcgagtgcggccgcaagcttTTATTTTTCGCCGACGATCAGpigF-R1ctcgagtgcggccgcaagcttTTTTTATTTTTCGCCGACGATCAGpigF-R2ctcgagtgcggccgcaagcttTTTTTTTTATTTTTCGCCGACGATCAGpigF-R3ctcgagtgcggccgcaagcttTTTTTTTTTTTATTTTTCGCCGACGATCAGpigF-R4ctcgagtgcggccgcaagcttTTTATTTTATTTTTCGCCGACGATCAGpigF-R5ctcgagtgcggccgcaagcttATTATTATTTTTTTATTTTTCGCCGACGATCAGpigF-R6ctcgagtgcggccgcaagcttTTTTTTATTTTATTTTTCGCCGACGATCAGpigF U FacagccggatccccgggtaccATGCCTTTAACCAAGCAAGATGCpigF U RcaaataggggttccgcgTTTTTTATTTTATTTTTCGCCGAC Apm FaaaCGCGGAACCCCTATTTGTTApm RtgctaactggTCAGCCAATCGACTGGCGpigF D FgattggctgaCCAGTTAGCAACGGGAGCTCGpigF D RgcctaggccgaattcgagctcTTTATGGAACAAATCCGGGTGpigF(q)FTTTCTGTTGAGCGACGAA pigF(q)RCCATAGCGGATACAGGAAG16sRNA FGCCCAGGTAAGGTTCTTC16sRNA RGGTGTAGCGGTGAAATGC
注:加粗碱基为终止密码子,斜体碱基为poly(A/T)尾,加下划线
碱基为酶切位点,小写字母是同源臂用于同源重组
1.1.4 培养基及培养条件
LB培养基(g/L):胰蛋白胨10、酵母粉5、NaCl 10;pH 7.0,固体培养基则加1.5%~2%的琼脂粉,121 ℃灭菌20 min。根据实际需要添加适量的抗生素。
发酵培养基(g/L):蔗糖20、牛肉膏15、CaCl210、脯氨酸7.5、MgSO4·7H2O 0.2、FeSO4·7H2O 0.06;pH 7.0,121 ℃灭菌20 min;粘质沙雷氏菌发酵用。
大肠杆菌培养温度37 ℃,旋转式摇床180 r/min;粘质沙雷氏菌培养温度30 ℃,旋转式摇床200 r/min。
1.2 重组菌株的构建与表达
1.2.1 pigF基因的克隆
根据GenBank中粘质沙雷氏菌灵菌红素合成基因簇基因组序列(登录号:AJ833002.1)中pigF基因序列设计其扩增引物(pigF-F/pigF-R),以粘质沙雷氏菌JNB5-1的基因组DNA为模板扩增pigF。利用同源重组试剂盒将所得目的基因片段与线性化pET-28a载体连接,转化E.coli BL21感受态细胞,在50 μg/mL卡那霉素抗性平板上挑取阳性转化子。重组质粒经酶切验证后,送上海生工生物技术有限公司进行测序验证。
1.2.2 poly(A/T)尾的筛选
以粘质沙雷氏菌JNB5-1的基因组为模板,通过PCR扩增分别获得3′端连接(或没有连接)有不同poly(A/T)尾的氧甲基转移酶的pigF、pigF1~pigF6;将pigF、pigF1~pigF6分别与pETC-28a(pET-28a衍生质粒,氯霉素抗性,T7启动子被替换为中等强度启动子P14[16])质粒经BamH I/Sac I双酶切后连接,得到连接产物;将连接产物转化大肠杆菌BL21并涂布在LB固体培养基(含12.5 μg/mL氯霉素)上,于37 ℃恒温培养箱中倒置培养8~12 h,挑取阳性转化子并进行验证(BamH I/Sac I),验证正确的质粒送上海生工测序,确认poly(A/T)尾连接到pigF基因,测序正确即获得大肠杆菌BL21/pETC-28a-pigF、BL21/pETC-28a-pigF1~BL21/pETC-28a-pigF6和重组质粒pETC-28a-pigF、pETC-28a-pigF1~pETC-28a-pigF6。将上述验证成功质粒分别以转化的方式(同大肠杆菌转化法)转化至粘质沙雷氏菌JNB5-1和粘质沙雷氏菌JNB5-1ΔpigF,得到JNB5-1F,JNB5-1FP1~JNB5-1FP6,JNB5-1ΔpigFF及JNB5-1ΔpigFFP1~JNB5-1ΔpigFFP6菌株。
1.2.3 重组粘质沙雷氏菌的构建
以粘质沙雷氏菌JNB5-1的基因组为模板,通过PCR扩增分别获得3′端连接有核苷酸序列如 “AAAAAA”所示的poly(A/T)尾的灵菌红素合成基因簇pigF(引物:pigF U F/pigF U R)、安普霉素抗性基因片段Apm(pigF)(引物:Apm F/Apm R)以及下游片段pigF D(引物:pigF D F/pigF D R)。通过同源重组试剂盒将上述片段与经Kpn I/Sac I双酶切的pUT-km质粒依次相连,得到连接产物;将连接产物转化大肠杆菌S17-1 λ pir并涂布在含有50 μg/mL安普霉素的LB固体培养基上,于37 ℃恒温培养箱中倒置培养8~12 h,挑取转化子并进行验证以及测序验证,验证正确即获得大肠杆菌S17-1 λ pir/pUT-km-pigFP-Apm和敲入质粒pUT-km-pigFP-Apm。将大肠杆菌S17-1 λ pir/pUT-km-pigFP-Apm和粘质沙雷氏菌JNB5-1分别接种至LB液体培养基中,于37 ℃、180 r/min的条件下培养16 h,将培养液离心并收集上述2种菌菌体,在LB固体培养基上进行共培养,通过接合转移的方式将敲入质粒pUT-km-pigFP-Apm转移至粘质沙雷氏菌JNB5-1中。接合转移后涂布在LB固体培养基(含50 μg/mL安普霉素和50 μg/mL克林霉素)上,于37 ℃恒温培养箱中倒置培养12~24 h,得到粘质沙雷氏菌JNB5-1/pigFFP;挑取阳性子接种至LB液体培养基中,于37 ℃、180 r/min的条件下摇瓶培养8~12 h稳定遗传3代,得到培养液;对培养液提基因组并通过PCR扩增(pigF U F/Apm (pigF) R)单菌落中pigF U-Apm基因验证,扩增成功即获得粘质沙雷氏菌JNB5-1/pigFFP。
1.3 PigF诱导表达
将经验证的大肠杆菌阳性转化子活化,然后转接至含50 μg/mL卡那霉素的50 mL LB培养基中,37 ℃ 180 r/min振荡培养,培养至OD约0.6~0.8时进行IPTG(终浓度为0.5 mmol/L)诱导,16 ℃过夜诱导表达。将诱导的菌液于4 ℃ 5 000×g 离心5 min,收集菌体。用PBS缓冲液洗涤菌体3次,重悬细胞后经超声波破碎得粗酶液。
1.4 PigF酶活力的测定
参考ARNOW[17]的方法进行,反应体系包括50 mmol/L Na2HPO4-KH2PO4(pH 7.0)、1 mmol/L DTT、10 mmol/L MgCl2、2 mmol/L SAM、1 mmol/L 3,4-二羟基苯甲酸和适量纯化的酶。将上述反应混合物混匀后在30 ℃水浴中反应25 min,加入1 mL 0.5 mmol/L HCl溶液终止反应。然后分别加入1 mL 10% NaNO2、10% NaMoO4和1 mol/L NaOH溶液,混匀后测定光吸收值A510。以未加酶的反应液作为对照。酶活力单位定义:一个活力单位(U)为标准条件下每1 min甲基化1 μmol 3,4-二羟基苯甲酸所需的酶量。
蛋白含量采用Bradford法[18]测定。
1.5 mRNA半衰期测定
mRNA半衰期测定:菌株在LB培养基中培养至对数中期,先在没有利福平的情况下取样,然后用200 μg/mL利福平处理。加入利福平后0、2、4、6、8和10 min离心收集细胞。提取RNA并进行实时荧光定量PCR。RNA提取和实时荧光定量PCR方法参照试剂盒说明书进行操作。以0 min转录水平为100%,后面每1次取样的转录水平为相对转录水平,取ln值,绘制以时间为横轴的散点图,计算其斜率k值,然后带入公式t1/2=ln2/k计算半衰期t1/2。
1.6 灵菌红素含量的测定
以酸性乙醇为空白对照,取发酵液溶解到酸性乙醇(pH 3.0)中,得到样品;将样品做适度的稀释(50、250、1 000倍),得到稀释样品;将稀释样品密封放置8 h(充分溶解PG)后3 276×g离心10 min,取上清液;测定上清液的A535值,根据标准曲线Y=1.193 6X-0.001计算得到发酵液中PG的含量。其中,Y代表A535值,X代表灵菌红素产量,单位mg/100 mL。
1.7 重组菌株发酵产灵菌红素
菌株经平板活化后,挑取单菌落分别接入至LB培养基(含50 μg/mL安普霉素和50 μg/mL克林霉素)中,于30 ℃、180 r/min的条件下摇床培养16 h,得到种子液;将种子液以体积分数6%接种量接种至50 mL发酵培养基中,于30 ℃、200 r/min的条件下摇床培养96 h,每隔12 h获得培养液,测灵菌红素产量。
2 结果与分析
2.1 温度对S.marcecens JNB5-1合成灵菌红素的影响
粘质沙雷氏菌JNB5-1在30 ℃可以高效合成灵菌红素,在37 ℃甚至更高温度低效甚至无法合成灵菌红素[4]。将粘质沙雷氏菌JNB5-1分别在不同温度(20~40 ℃)条件下发酵培养,结果如图2-a所示。当培养温度低于30 ℃时,菌株JNB5-1合成灵菌红素的产量随着温度的升高而逐渐增加,并在30 ℃时产量达到最大值;随着温度进一步升高,灵菌红素产量迅速下降,当温度高于37 ℃时,菌株JNB5-1基本不再合成灵菌红素 (图2-b)。据文献报道,粘质沙雷氏菌的生存温度范围比较宽泛(20~40 ℃),而灵菌红素的合成却需要相对严格的温度(24~30 ℃)。

a-粘质沙雷氏菌在30 ℃和37 ℃培养条件下摇瓶中产灵菌红素特征;
b-不同温度对灵菌红素产量影响
图2 S.marcecens JNB5-1在不同温度下灵菌红素产量
Fig.2 Production of prodigiosin in S.marcecens JNB5-1
at different temperatures
2.2 poly(A/T)尾对pigF转录水平的影响
本课题组利用双向电泳和基质辅助激光解析电离飞行时间质谱技术分离和鉴定出JNB5-1中涉及灵菌红素合成的关键酶 PigF在28 ℃时蛋白表达水平相较37 ℃提高了7.4倍[19],同时转录组及qPCR得到了一致趋势的结果。温度对酶表达量和酶活力的影响主要包括转录过程和翻译过程的影响,而粘质沙雷氏菌合成灵菌红素受温度调节的机制目前仍不清楚。本研究计划先考察PigF在转录水平上受到温度影响情况。据文献报道,基因聚腺苷酸化可以有效减缓mRNA降解速率,人工添加poly (A)尾聚腺苷酸化基因可以提高其mRNA稳定性,进而提高目标酶的表达量和酶活力[9,14],因此,考虑采用人工添加poly (A)尾策略提高pigF基因mRNA稳定性,进而提高PigF表达量和酶活力,考察其对粘质沙雷氏菌合成灵菌红素的影响。
根据文献报道[20],本研究在pigF 3′端添加6条不同长度poly(A/T)尾(图3-a),连接至pETC-28a质粒分别转化至粘质沙雷氏菌和粘质沙雷氏菌△pigF敲除菌体内,并置于30和37 ℃下培养,观察pigF在转录水平上的变化。由图3-b可知,在粘质沙雷氏菌体内,在添加poly(A/T)尾后,转录水平都有不同程度提升,其中JNB5-1FP5和JNB5-1FP6相对其他菌株提升较为明显,分别提高了69.3%和77.8%,且在37 ℃提升幅度大于较30 ℃,分别提高了1.36倍和1.17倍。在粘质沙雷氏菌△pigF敲除菌体内表达带有3′端带有poly(A/T)尾pigF时,发现了类似的趋势(图3-c)。

a-以pigF为靶基因添加6种不同PNT,黑色序列是pigF基因的最后10个碱基对,斜体字母是PNT;b-在粘质沙雷氏菌中验证poly(A/T)尾的功能在粘质沙雷氏菌中验证poly(A/T)尾的功能;c-在粘质沙雷氏菌△pigF敲除菌中中验证poly(A/T)尾的功能在粘质沙雷氏菌中验证poly(A/T)
尾的功能;d-添加不同poly(A/T)尾对30 ℃培养的粘质沙雷氏菌pigF基因半衰期的影响。
图3 poly(A/T)尾对粘质沙雷氏菌中pigF的影响
Fig.3 Effect of poly(A/T)tails on pigF in S.marcescens
由于聚腺苷酸化可以有效减缓mRNA降解速率,为了确认加尾部分是否在转录的mRNA中得到体现,对JNB5-1、JNB5-1FP1~JNB5-1FP6中pigF基因半衰期进行测定。如图3-d所示,JNB5-1FP1~JNB5-1FP6中的pigF基因半衰期较原始菌株均有所提高,其中JNB5-1FP6中pigF半衰期(4.35 min)较JNB5-1中pigF半衰期(2.82 min)提高了54.6%,表明在添加poly(A/T)尾后可以有效缓解pigF降解,提高pigF转录水平。
结果显示,在pigF 3′端添加6条不同长度poly(A/T)尾可以使得聚腺苷酸化水平得到提升,从而保护mRNA避免被降解,获得了较高的转录本水平。
2.3 PigF诱导表达及酶活测定
将胶回收的pigF片段与线性化的pET-28a连接,构建重组载体pET-28a-pigF,然后转化E.coli BL21(DE3)感受态细胞。获得阳性转化子后提取重组质粒,将重组质粒pET-28a-pigF经单双酶切验证,得到5369 bp和1017 bp大小的片段,分别对应pET-28a和pigF的大小。结果表明,重组质粒pET-28a-pigF构建成功(图4-a)。

a-重组质粒pET-28a-pigF酶切验证;M1-DL2000 DNA Marker;M2-λ-Hind III digest DNA Marker;1-pET-28a-pigF单切验证;2-pET-28a-pigF双切验证;b-重组菌全细胞蛋白及PigF纯化后SDS-PAGE电泳图,M-Protein Marker;1-纯化的PigF;2-重组菌E.coli BL21(DE3)/pET-28a-pigF上清液;3-重组菌E.coli BL21(DE3)/pET-28a上清液
图4 重组pigF构建及其表达
Fig.4 Construction and expression of recombinant pigF
根据1.3小节所述的方法对E.coli BL21(DE3)/pET-28a-pigF进行诱导表达,收集经IPTG过夜诱导的重组菌E.coli BL21(DE3)/pET-28a-pigF菌体,超声波破碎,取上清液进行SDS-PAGE分析(图4-b)。结果表明,重组酶蛋白表达良好,其表观分子质量约为37 kDa。为了检测PigF酶活力,采用比色法体外测定法,在体外酶活力测定结果显示PigF对3,4-二羟基苯甲酸催化活性为48 U/mg(表2)。
表2 重组蛋白PigF比酶活力
Table 2 Specific enzyme activity of recombinant PigF
纯化步骤总蛋白/mg总酶活力/U比酶活力/(U·mg-1)粗酶液8.9427.348
2.4 poly(A/T)尾对PigF酶活力的影响
为了研究poly(A/T)尾对PigF酶活力的影响,将JNB5-1F,JNB5-1FP1~JNB5-1FP6菌株置于30 ℃条件下培养24 h,超声波破碎细胞得粗酶液。根据1.4小节所述的方法测定PigF酶活力。如图5所示,在粘质沙雷氏菌中,当pigF基因3′端添加6条不同长度poly(A/T)尾后,PigF酶活力有不同程度的提高。具体而言,JNB5-1FP6取得最大的比酶活力为3.12 U/mg,相对于对照提高了41.8%(图5)。该结果表明poly(A/T)尾的存在可以提高氧甲基转移酶酶活力。所以后续实验使用“AAAAAA”poly(A/T)尾进行研究。

图5 不同poly(A/T)尾对酶活力的影响
Fig.5 Effect of poly(A/T)tails on enzyme activity
结果表明,当poly(A/T)尾与pigF基因连接时,获得了很高的氧甲基转移酶酶活性水平。当pigF基因过表达时,3′端的poly(A/T)尾部导致pigF转录水平增加,因此pigF在JNB5-1FP1~JNB5-1FP6中的转录丰度得到了提高,暗示由于poly(A/T)尾,pigF基因在粘质沙雷氏菌中的转录和表达得到了增强。结果表明,mRNA上短的poly(A/T)尾的存在可以通过增强mRNA的稳定性来显着增强后续翻译过程。
2.5 poly(A/T)尾整合到粘质沙雷氏菌染色体上的影响
为了克服质粒的不稳定,将3’端带有“AAAAAA”的pigF替换pig基因簇上原有的pigF,得到重组菌JNB5-1/pigFFP。根据GenBank中粘质沙雷氏菌灵菌红素合成基因簇基因组序列(登录号:AJ833002.1)中pigF基因序列(基因大小1 017 bp)。采用pUT-km敲入系统[21],经接合转移和同源重组完成基因敲入。通过PCR扩增单菌落中pigF U-pigF-Apm(pigF)重组基因(约1 940 bp,结果见图6),鉴定与测序结果显示,PCR条带在2 000左右,与理论值相一致(图6)。结果表明3′端带有“AAAAAA”的pigF替换成功,获得重组菌JNB5-1/pigFFP。
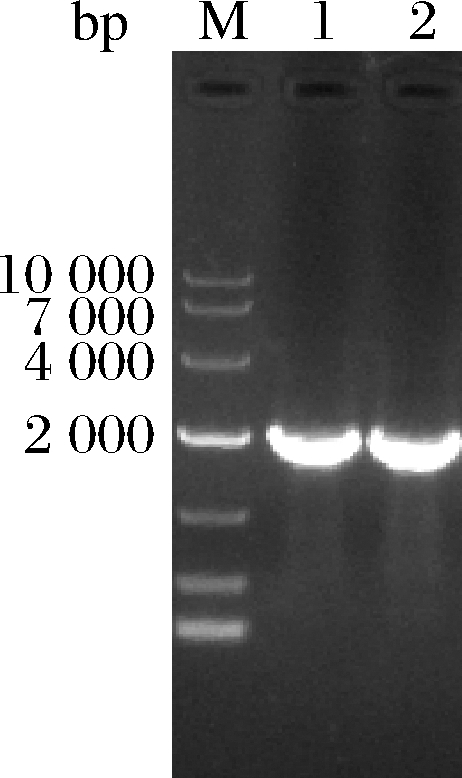
M-DL10000 DNA Marker;1-JNB5-1/pigFFP1 PCR验证结果;
2-JNB5-1/pigFFP2 PCR验证结果
图6 重组粘质沙雷氏菌PCR扩增鉴定
Fig.6 PCR identification of recombinant S.marcescens
2.6 poly(A/T)尾对摇瓶中灵菌红素产量的影响
为了进一步研究重组菌JNB5-1/pigFFP合成灵菌红素的能力,将重组菌JNB5-1/pigFFP菌株进行摇瓶发酵测试,在发酵期间,每隔12 h对发酵液进行取样并检测灵菌红素含量。由图7可知,重组菌JNB5-1/pigFFP合成灵菌红素产量相对于原始菌株具有较大的提高。具体而言,在生长初级阶段,重组菌JNB5-1/pigFFP的生长要滞后于JNB5-1,当进入到对数期后,两者生长差异较小,菌株也开始大量合成灵菌红素;两者在84 h后开始进入衰退期,灵菌红素产量开始下降。菌株发酵至84 h,OD600达到最高,此时灵菌红素含量积累到最高点,JNB5-1中灵菌红素高达(5.36±0.06)g/L,而重组菌JNB5-1/pigFFP中灵菌红素也达到(6.21±0.37)g/L。可见,在重组菌JNB5-1/pigFFP可以实现更高效合成灵菌红素。
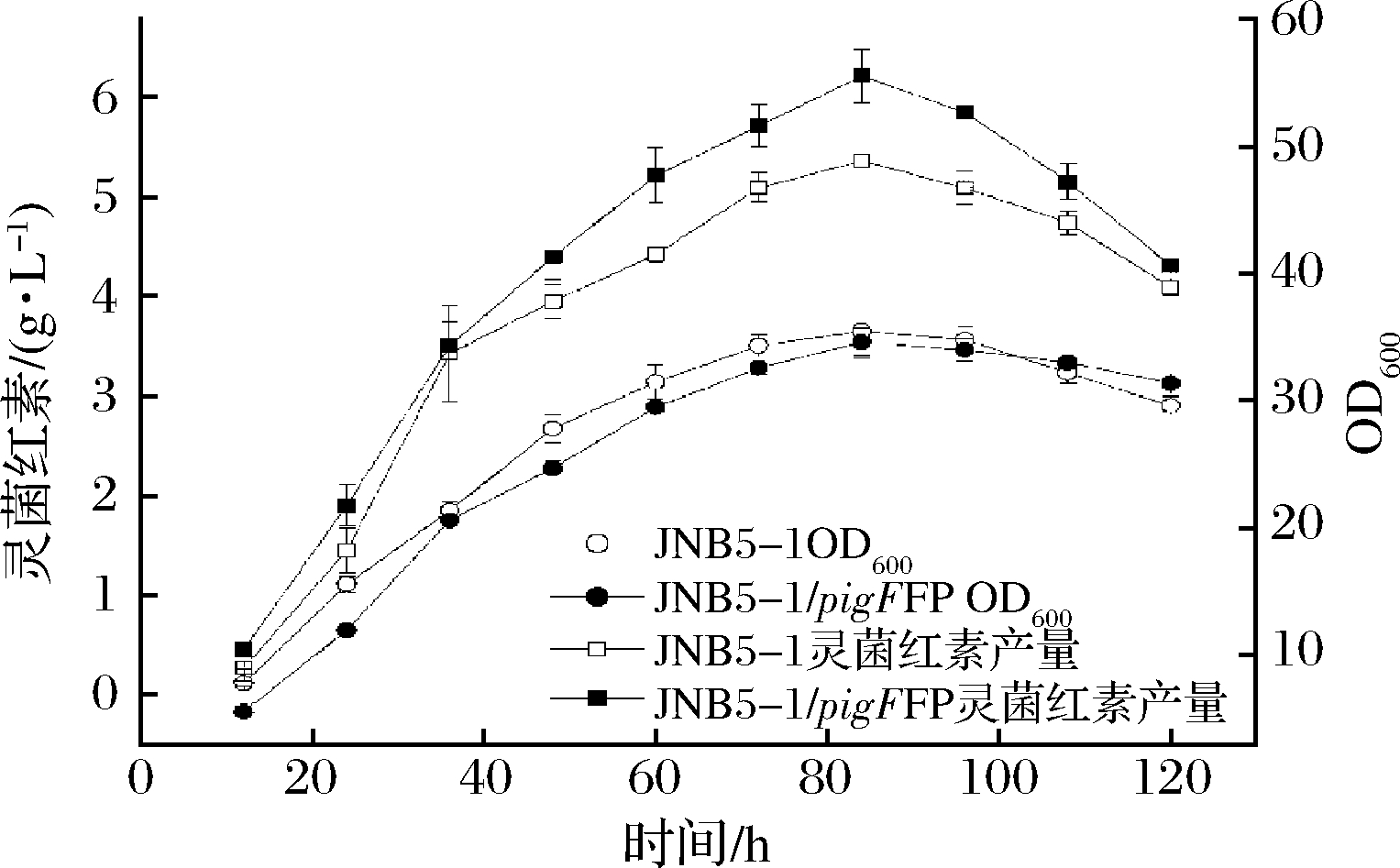
图7 重组粘质沙雷氏菌菌合成灵菌红素摇瓶发酵过程
Fig.7 Shake flask fermentation process of synthetic prodigiosin
in recombinant S.marcescens
3 讨论
WILLIAMSON等[22]发现,ΔpigF突变体中产生了由MAP和HBC合成的一种副产物,使菌落在PGM琼脂上呈现橙色。在前期的研究中,与28 ℃相比,参与灵菌红素合成的PigF在37 ℃时显著下调[19]。以上结果表明,PigF的表达量和酶活力对粘质沙雷氏菌合成灵菌红素的水平具有重要影响。而相对低的温度不但有利于蛋白的稳定性,同时也有利于mRNA的稳定性,而mRNA的稳定性决定了目标酶的表达量。mRNA是一个动态过程,主要包括转录,降解和翻译,在3′-UTR的下游添加poly(A/T)尾巴可以提高目标基因mRNA稳定性,从而提高了目标酶的表达量和酶活力[23]。从这一点来看,粘质沙雷氏菌中pigF表达水平的提高可能是由于poly(A/T)尾巴在维持mRNA稳定性和避免快速降解方面起着重要作用[24](图3-d),最终,通过给pigF添加poly(A/T)尾提高了粘质沙雷氏菌合成灵菌红素的能力。在这项研究中,poly(A/T)尾与其他可用的调控元件(包括启动子[25],核糖体结合位点(RBS)[26]和终止子[27])相比具有许多优势。首先,使用poly(A/T)尾巴作为遗传操纵工具很方便,可以通过合成引物引入,考虑到长poly(A/T)尾的不稳定性,我们设计了最长的poly(A/T)尾仅12 nt[28-29]。其次,poly(A/T)尾巴只能促进靶基因的mRNA稳定性,而对靶基因的上游和下游基因影响很小,因此可以调节基因簇中单个基因的强度而不影响其他基因的表达,对pig基因簇的反应良好[30]。第三,相对于强启动子增强基因的表达,它在代谢工程中以给定的强度增加基因的表达,只要辅以相对弱的多的启动子就能达到相同的效果,因此减轻了细胞负担。总之,在粘质沙雷氏菌中通过添加poly(A/T)尾巴可提高灵菌红素产量,为代谢改造微生物提高灵菌红素产量提供了可借鉴的思路。
[1] GASTMEIER P.Serratia marcescens:An outbreak experience[J].Frontiers in Microbiology,2014,5:81.
[2] GUPTA N,HOCEVAR S N,MOULTON—MEISSNER H A,et al.Outbreak of Serratia marcescens bloodstream infections in patients receiving parenteral nutrition prepared by a compounding pharmacy[J].Clinical Infectious Diseases,2014,59(1):1-8.
[3] 梅建凤,蔡少芬,李靓,等.高产舍雷肽酶的粘质沙雷氏菌诱变选育[J].食品与发酵工业,2018,44(8):177-180.
MEI J F,CAI S F,LI L,et al.Mutation breeding of Serratia marcescens strains for serrapeptase high-yield production[J].Food and Fermentation Industries,2018,44(8):177-180.
[4] TANAKA Y,YUASA J,BABA M,et al.Temperature-dependent bacteriostatic activity of Serratia marcescens[J].Microbes and Environments,2004,19(3):236-240.
[5] WILLIAMS R P,GOTT C L,QADRI S M,et al.Influence of temperature of incubation and type of growth medium on pigmentation in Serratia marcescens[J].Journal of Bacteriology,1971,106(2):438-443.
[6] ROWLEY G,SPECTOR M,KORMANEC J,et al.Pushing the envelope:Extracytoplasmic stress responses in bacterial pathogens[J].Nature Reviews Microbiology,2006,4(5):383-394.
[7] HARRIS A,WILLIAMSON N R,SLATER H,et al.The Serratia gene cluster encoding biosynthesis of the red antibiotic,prodigiosin,shows species-and strain-dependent genome context variation[J].Microbiology,2004,150(Pt 11):3 547-3 560.
[8] EDMONDS M,WINTERS M A.Polyadenylate polymerases[J].Progress in Nucleic Acid Research and Molecular Biology,1976,17:149-179.
[9] MOHANTY B K,KUSHNER S R.Bacterial/archaeal/organellar polyadenylation[J].Wiley Interdisciplinary Reviews:RNA,2011,2(2):256-276.
[10] AUGUST J T,ORTIZ P J,HURWITZ J.Ribonucleic acid-dependent ribonucleotide incorporation.I.Purification and properties of the enzyme[J].Journal of Biological Chemistry,1962,237:3 786-3 793.
[11] HAJNSDORF E,KABERDIN V R.RNA polyadenylation and its consequences in prokaryotes[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2018,373(1 762):20180166.
[12] XU F F,COHEN S N.RNA degradation in Escherichia coli regulated by 3′ adenylation and 5′ phosphorylation[J].Nature,1995,374(6 518):180-183.
[13] GOODRICH A F,STEEGE D A.Roles of polyadenylation and nucleolytic cleavage in the filamentous phage mRNA processing and decay pathways in Escherichia coli[J].RNA,1999,5(7):972-985.
[14] LAALAMI S,ZIG L,PUTZER H.Initiation of mRNA decay in bacteria[J].Cellular and Molecular Life Sciences,2014,71(10):1 799-1 828.
[15] BANDYRA K J,LUISI B F.Licensing and due process in the turnover of bacterial RNA[J].RNA Biology,2013,10(4):627-635.
[16] MUTALIK V K,GUIMARAES J C,CAMBRAY G,et al.Precise and reliable gene expression via standard transcription and translation initiation elements[J].Nature Methods,2013,10(4):354-360.
[17] ARNOW L E.Colorimetric determination of the components of 3,4-dihydroxyphenylalaninetyrosine mixtures[J].Journal of Biological Chemistry,1937,118(2):531-537.
[18] BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].Analytical Biochemistry,1976,72:248-254.
[19] 徐虹,徐美娟,杨套伟,等.温度对粘质沙雷氏菌合成灵菌红素的影响[J].微生物学报,2014,54(5):517-524.
XU H,XU M J,YANG T W,et al.Effect of temperature on prodigiosin synthesis in Serratia marcecens[J].Acta Microbiologica Sinica,2014,54(5):517-524.
[20] YANG T W,IRENE K,LIU H,et al.Enhanced extracellular gamma glutamyl transpeptidase production by overexpressing of PrsA lipoproteins and improving its mRNA stability in Bacillus subtilis and application in biosynthesis of L-theanine[J].Journal of Biotechnology,2019,302:85-91.
[21] HERRERO M,DE LORENZO V,TIMMIS K N.Transposon vectors containing non-antibiotic resistance selection markers for cloning and stable chromosomal insertion of foreign genes in gram-negative bacteria[J].Journal of Bacteriology,1990,172(11):6 557-6 567.
[22] WILLIAMSON N R,SIMONSEN H T,AHMED R A,et al.Biosynthesis of the red antibiotic,prodigiosin,in Serratia:Identification of a novel 2-methyl-3-n-amyl-pyrrole (MAP) assembly pathway,definition of the terminal condensing enzyme,and implications for undecylprodigiosin biosynthesis in Streptomyces[J].Molecular Microbiology,2005,56(4):971-989.
[23] XU S,WANG X,DU G,et al.Enhanced production of L-sorbose from D-sorbitol by improving the mRNA abundance of sorbitol dehydrogenase in Gluconobacter oxydansWSH-003[J].Microbial Cell Factories,2014,13(1):146.
[24] NICHOLSON A L,PASQUINELLI A E.Tales of detailed poly(A) tails[J].Trends in Cell Biology,2019,29(3):191-200.
[25] REDDEN H,ALPER H S.The development and characterization of synthetic minimal yeast promoters[J].Nature Communications,2015,6:7 810.
[26] GUIZIOU S,SAUVEPLANE V,CHANG H J,et al.A part toolbox to tune genetic expression in Bacillus subtilis[J].Nucleic Acids Research,2016,44(15):7 495-7 508.
[27] CURRAN K A,KARIM A S,GUPTA A,et al.Use of expression-enhancing terminators in Saccharomyces cerevisiae to increase mRNA half-life and improve gene expression control for metabolic engineering applications[J].Metabolic Engineering,2013,19:88-97.
[28] GODISKA R,MEAD D,DHODDA V,et al.Linear plasmid vector for cloning of repetitive or unstable sequences in Escherichia coli[J].Nucleic Acids Research,2010,38(6):e88.
[29] TREPOTEC Z,GEIGER J,PLANK C,et al.Segmented poly(A) tails significantly reduce recombination of plasmid DNA without affecting mRNA translation efficiency or half-life[J].RNA,2019,25(4):507-518.
[30] TIAN R,LIU Y,CHEN J,et al.Synthetic N-terminal coding sequences for fine-tuning gene expression and metabolic engineering in Bacillus subtilis[J].Metabolic Engineering,2019,55:131-141.