在全球变暖和化石燃料走向枯竭的背景下,节能减排一直是食品与发酵工业关注的主要问题。在啤酒生产中,高浓酿造作为一种有效的方式,可以减少水资源消耗、废弃物排放、发酵罐数量和人工成本[1]。高浓酿造技术主要应用于拉格啤酒生产,拉格啤酒占全球产量的90%以上[2]。在高浓酿造过程中,酵母细胞暴露于高浓度麦汁的环境中,与常浓酿造相比,酵母的迟滞期延长、发酵周期延长并且残余麦芽三糖显著增多[3]。
自高浓酿造技术出现以来,许多研究者试图从多方面提高酵母发酵性能:提升酵母抗氧化能力以改善发酵[4];筛选抗葡萄糖阻遏菌株来缩短发酵迟滞期[5];通过增强麦芽糖转运来提高发酵末期的糖利用[6];通过添加外源氨基酸来提高酵母活性等[7]。提升酵母对高浓度麦汁的耐受是其中一个重要方向,高浓度麦汁是一个复杂的环境,高浓度的葡萄糖、麦芽糖与α-氨基氮都会影响酵母细胞的基因调控方向。然而目前对高浓度麦汁胁迫酵母细胞的机制没有完善的研究。因此,揭示拉格酵母应对高浓麦汁的应答机制,可以为提升高浓酿造技术水平奠定基础。随着测序技术的发展和生物信息学分析方法的优化[8],转录组和代谢组的交叉分析可以很好地揭示不同条件下菌株的表达与代谢差异[9]。转录组学分析可以筛选不同条件下的显著差异基因与代谢通路[10]。代谢组学分析可以验证基因表达调整后的代谢物变化,并找到核心代谢物。
本研究通过转录组和代谢组分析了拉格酵母M14对常浓与高浓麦汁的应答机制。通过组学关联分析筛选出13个具有显著差异的重要基因。初步分析拉格酵母对高浓麦汁的应答机制,为后续高浓酿造优良酵母的选育提供方向。
1 材料与方法
1.1 菌株与培养基
工业拉格酵母(Saccharomyces pastorianus M14)保藏于本实验室,经过全基因组测序[11](NCBI ID:MVPU00000000)。
活化培养基:葡萄糖20 g/L、蛋白胨20 g/L、酵母粉10 g/L、琼脂粉20 g/L(用于斜面培养基配制)。
麦汁培养基:加拿大麦芽3.2 kg、美国卡斯卡特啤酒花4 g、水9 L、琼脂粉20 g/L(用于平板培养基配制),参照《酿造酒工艺学》[12]中的方法,制备成24 °P麦汁,其他浓度麦汁使用纯净水稀释。
1.2 试剂与仪器
PBS缓冲液,Biosharp公司;标准品葡萄糖、果糖、麦芽糖,国药试剂有限公司;RNA提取试剂盒(UNIQ-10 column Trizol total RNA extraction kit),生工生物工程(上海)股份有限公司;逆转录试剂盒(NEBNext® UltraTM RNA Library Prep Kit),Illumina公司。
SW-CJ-2D双人单面净化工作台,苏州净化有限公司;Compass CX电子天平,Ohaus公司;UV11II紫外-可见分光光度计,上海天美公司;HYL-X恒温摇床,强乐实验设备公司;Research移液器,德国Eppendorf公司;Hve-50全自动高压蒸汽灭菌锅,日本平山制作所株式会社;R5408冷冻离心机,德国Eppendorf公司;Nanodrop2000核酸定量仪,美国Thermo Fisher公司;DMA4500啤酒分析仪,Anton-Paar公司。
1.3 浓度梯度麦汁平板胁迫测定
挑取1环M14细胞于YPD 培养基中,28 ℃,180 r/min,活化12 h。以体积分数10%的接种量进行翻接,28 ℃,180 r/min,培养12 h至稳定期(OD600 nm > 1.2)。7 000 r/min下4 ℃冷冻离心3 min,PBS缓冲液洗涤1次后使用无菌水梯度稀释至10-5,取10 μL菌液在8、12、16、20、24 °P麦汁平板上点板,倒置于11 ℃培养箱静置培养48 h。
1.4 发酵性能验证及指标测定
使用12 °P麦汁分三级对拉格酵母M14进行扩培。第一级25 ℃,180 r/min,扩培18 h;第二级18 ℃,120 r/min,扩培24 h;第三级11 ℃,静置,扩培24 h。取酵母细胞在7 000 r/min下4 ℃冷冻离心3 min,接种于300 mL 12 °P与24 °P麦汁培养基中,接种量为106个/(mL·Brix)。置于11 ℃培养箱发酵,每天记录失重并取1 mL样本分析糖谱。设置3个生物学重复。
发酵度、残留浸出物与酒精度测定采用DMA4500啤酒分析仪,每个样本重复测定2次。
糖谱分析使用高效液相色谱测定,参考ASBC分析方法[13],每个样本测定1次。
1.5 RNA提取与转录组测序
使用1.4小节中方法制备种子液,取酵母细胞在7 000 r/min下4 ℃冷冻离心3 min后迅速接种于预冷至11 ℃的1 000 mL 12 °P与24 °P麦汁培养基中,接种量为106个/(mL·Brix)。11 ℃静置2 h后取酵母细胞,7 000 r/min下4 ℃冷冻离心3 min,使用预冷的PBS缓冲液清洗3次,去除麦汁杂质后存储细胞于1.5 mL EP管内。使用液氮速冻,存于-80 ℃冰箱备用。按照试剂盒说明书对酵母总RNA进行提取,提取后的RNA使用Nanodrop 2000进行核酸定量并进行琼脂糖电泳检测,检测合格的RNA交于北京诺禾致源科技股份有限公司进行Illumina测序。设置3个生物学重复。
高通量测序仪测得的图像数据经CASAVA碱基识别转化为序列数据,然后对原始数据进行过滤,去除接头与低质量数据得到clean data。同时,对clean data进行Q20、Q30和GC含量计算。后续所有分析均是基于clean data进行的高质量分析。使用HISAT2构建参考基因组的索引并将配对末端clean reads与M14基因组比对。featureCounts(1.5.0-p3)用于计算映射到每个基因的读数。然后根据基因的长度计算每个基因的FPKM,并计算映射到该基因的读数。使用DESeq2软件进行2个比较组合之间的差异表达分析[14](每个组3个生物学重复),设置校正后的P<0.05以及|log2fold change|≥1作为显著差异表达的阈值。通过clusterProfiler(3.4.4)软件实现差异表达基因的GO(gene ontology)富集分析与KEGG(Kyoto encyclopedia of genes and genomes)通路中差异表达基因的统计富集,其中修正了基因长度偏差。
1.6 胞内代谢物提取与代谢组检测
代谢组学样本与转录组学样本取样方法相同,采用正离子与负离子模式分别检测[15],在本文的结果与分析中合并分析,设置6个生物学重复。
取100 mg液氮研磨的组织样本,置于EP管中,加入500 μL含1 L/m3甲酸的800 L/m3甲醇水溶液,涡旋振荡,冰浴5 min,4 ℃、15 000 r/min离心10 min,取一定量的上清液使用超纯水稀释至甲醇含量为600 L/m3,并置于带有0.22 μm滤膜的离心管中4 ℃、15 000 r/min离心10 min,收集滤液,进样LC-MS进行分析。从每个实验样本中取等体积样本混匀作为QC样本。空白样本为含1 L/m3甲酸的600 L/m3甲醇水溶液代替实验样本,前处理过程与实验样本相同。
表1 色谱梯度洗脱程序
Table 1 Chromatographic gradient elution program

时间/minA/(L·m-3)B/(L·m-3)0.09821.598212.0010014.0010014.198216.0982
超高效液相色谱使用Hyperil Gold column(C18)色谱柱,色谱条件:柱温40 ℃、流速0.2 mL/min;正模式:流动相A,1 L/m3甲酸、流动相B,甲醇;负模式:流动相A,5 mmol/L醋酸铵,pH 9.0、流动相B,甲醇。
质谱扫描范围选择m/z 100~1 500;ESI源的设置如下:Spray Voltage:3.2 kV;Sheath gas flow rate:35 arb;Aux Gasflow rate:10 arb;Capillary Temp:320 ℃。MS/MS二级扫描为data-dependent scans。
将下机数据文件导入Compound Discoverer 3.0软件中,进行保留时间、质荷比等参数的简单筛选,然后对不同样品根据保留时间偏差0.2 min和质量偏差5 mg/L进行峰对齐,使鉴定更准确,随后根据设置的质量偏差5 mg/L、信号强度偏差30%、信噪比3、最小信号强度100 000、加和离子等信息进行峰提取,同时对峰面积进行定量,再整合目标离子,然后通过分子离子峰和碎片离子进行分子式的预测并与mzCloud和Chemspider数据库进行比对,用blank样本去除背景离子,并对定量结果进行归一化,最后得到数据的鉴定和定量结果。采用PLS-DA模型第一主成分的变量投影重要度(variable importance in the projection,VIP)值,VIP值表示不同分组中代谢物差异的贡献率;并结合T-test的P值来寻找差异性表达代谢物,设置VIP>1.0、校正后的P<0.05以及|log2foldchange|≥1作为显著差异代谢物的阈值。
1.7 数据分析
使用SPSS 20.0软件进行统计学显著性分析。P<0.05视为显著差异。使用GraphPad Prism 8进行图片绘制。
2 结果与分析
2.1 浓度梯度麦汁平板胁迫测定及发酵实验
为了研究高浓度麦汁对拉格酵母的胁迫水平,通过浓度梯度麦汁平板进行了简单的酵母生长情况与耐受性测定。如图1所示,拉格酵母M14在8 °P与12 °P麦汁平板上基本没有生长抑制,在16 °P麦汁平板上10-5浓度的细胞生长量显著降低,而在20 °P与24 °P麦汁平板上10-5浓度的酵母基本无法生长。表明高浓度麦汁给酵母细胞生长带来了较大的压力。

图1 M14菌株的高浓度麦汁胁迫平板实验
Fig.1 Plate experiment of strain M14 under high gravity wort stress
通过小瓶发酵实验比较常浓与高浓酿造发酵指标,并跟踪发酵过程的失重与糖降,有助于了解常浓度与高浓度麦汁造成的发酵性能与糖利用差异。由图2可知,M14的发酵度由12 °P中的58.56%下降到24 °P中的56.01%(图2-a);12 °P麦汁中酵母起酵较快,同时发酵5 d主酵接近结束,麦芽糖被完全利用(图2-c);24 °P麦汁中酵母起酵较慢,同时7 d未能完全消耗麦芽糖(图2-d);实验使用的麦汁糖谱符合ASBC中相关比例标准[13](图2-b)。

a-12 °P与24 °P麦汁的发酵度;b-麦汁的糖谱;c-12 °P下葡萄糖、果糖、麦芽糖与失重变化;d-24 °P下葡萄糖、果糖、麦芽糖与失重变化
图2 M14菌株的常浓与高浓发酵比较
Fig.2 Comparison of normal and high gravity fermentation of strain M14
综上所述,高浓度麦汁对拉格酵母有着较强胁迫,酵母在高浓酿造下迟滞期延长、生长速度下降并且糖分利用不完全,导致发酵周期延长和发酵度下降。因此本研究采用转录组与代谢组手段,进一步分析发酵早期代谢物与表达情况,从分子水平解释高浓度麦汁胁迫下的拉格酵母应答机制。
2.2 转录组分析
2.2.1 样本设置、主成分分析与基因表达热图
起酵阶段的应答对拉格酵母发酵性能至关重要。为了探究发酵早期拉格酵母对高浓麦汁的应答机制,转录组与代谢组样本的取样时机设置在起酵阶段。接种前的酵母参照工业酿造工艺流程进行逐级降温、扩培,确保其接入高浓与常浓发酵培养基时的状态一致、活力良好,并于接种后2 h取样。
转录组样本上机测序后,经过原始数据过滤、测序错误率检查、GC含量分布检查,获得后续分析使用的clean data。使用经过注释的自身参考基因组进行比对,共注释到6 598个基因,然后进行定量分析。采用主成分分析评估组间差异及组内样本重复情况,主成分分析采用线性代数的计算方法,对所有基因变量进行降维处理及主成分提取。对6个样本的基因表达值(FPKM)进行主成分分析(图3-b),组间样本分散,组内样本聚集,表明两组间具有显著差异且测序情况良好,说明拉格酵母M14对高浓麦汁产生了显著的应答调控。
同时绘制了表达基因聚类热图(图3-a)。通过主流的层次聚类对基因的FPKM值进行聚类分析,热图中的基因越靠近其表达模式越相近。因为经过归一化处理,热图中的颜色只能横向比较(同一基因在不同样本中的表达情况),不能纵向比较(同一样本不同基因的表达情况)。由图可知M14在应答12 °P与24 °P麦汁的情况下,组内的表达模式完全一致,组间的总体表达模式接近,但是存在局部的差异(图3-a中红框),有上调的差异区域也有下调的差异区域。这些显著差异的区域是拉格酵母应答高浓麦汁处理的重要基因与途径。

a-表达热图;b-主成分分析;c-差异基因火山图
图3 M14菌株应答常浓与高浓麦汁的表达情况
Fig.3 Gene expression of strain M14 response to normal and high gravity wort
通过宏观分析,验证了实验设计正确和平行性良好,且说明了常浓度与高浓度麦汁处理的拉格酵母差异显著,需要进一步进行显著基因差异分析与通路富集分析。
2.2.2 差异表达基因分析
经过测序结果质检与宏观分析,样本平行性良好,故在差异表达基因与显著差异表达基因的筛选中设置padj阈值为0.05与0.01。如表2所示,经过2组样本间差异表达基因的统计与筛选,一共获得了2 312个差异表达基因,占总基因数的35.04%,其中1 137个上调,1 175个下调,下调基因略多于上调基因(图3-c)。设置|log2fold change| ≥ 1作为显著差异基因筛选的阈值,得到191个显著差异基因,占总差异表达基因的8.26%,其中38个基因显著上调,153个基因显著下调,下调基因多于上调基因。显著差异基因中最大上调倍数为7.15倍,最大下调倍数为11.53倍。
表2 转录组基因统计
Table 2 Transcriptomic gene statistics
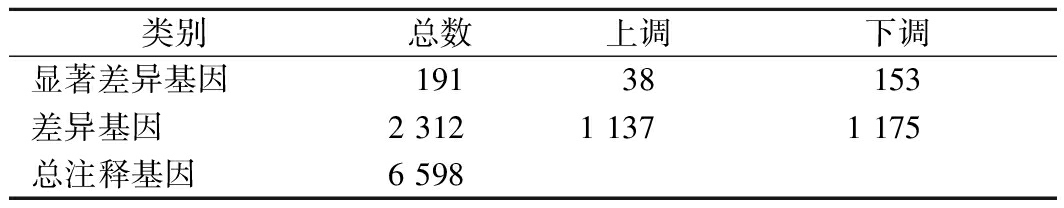
类别总数上调下调显著差异基因19138153差异基因2 3121 1371 175总注释基因6 598
大量基因显著下调可能是高浓度麦汁的胁迫造成的,但也有一定量的基因上调,这可能是拉格酵母M14为了应对麦汁胁迫做出的应答。通过显著差异基因分析,目标基因被锁定到191个以内,后续将通过GO、KEGG富集分析与代谢组数据支撑找到重要差异基因。
2.2.3 差异表达基因的GO富集分析
GO是描述基因功能的综合性数据库,基因被分为生物过程(biological process,BP)、细胞组成(cellular component,CC)和分子功能(molecular function,MF)3个部分。在3个部分中各选择10个以内富集到的显著差异通路进行柱状图绘制,各部分从左往右显著性从高到低如图4所示。高浓度与常浓度的差异基因主要富集到生物过程(BP)的碳水化合物代谢过程(GO:0005975)与氧化还原过程(GO:0055114);富集到细胞组成(CC)的膜的固有成分(GO:0031224)、膜的组成部分(GO:0016021)、蛋白酶体核心复合物(GO:0005839)与蛋白酶体复合体(GO:0000502);富集到细分子功能(MF)的作用于供体的氧化还原酶活性(GO:0016614)、维生素结合(GO:0019842)、作用于受体氧化还原酶活性 (GO:0016616)与辅因子结合(GO:0048037)。

图4 差异基因的GO富集
Fig.4 GO enrichment of DEGs
3个部分富集到的基因功能高度相关,可以将富集到的基因功能概括为碳代谢、氧化还原平衡、膜组成与蛋白酶体4个类别。其中碳代谢与氧化还原平衡与高浓胁迫直接相关,高浓麦汁中的高糖胁迫会直接影响能量代谢;细胞膜和酵母抗逆一直息息相关[16],膜功能的状态决定了酵母生理活性的水平;蛋白酶体功能涉及细胞周期控制、应激反应、基因转录与信号转导多方面[17],可能参与高浓胁迫下的细胞宏观调控。上述结果能够进一步缩小基因范围,加快基因挖掘进程。
2.2.4 差异表达基因的KEGG富集分析
KEGG综合性数据库整合了基因组、化学和系统功能信息。选取最显著的18个通路绘制散点图(图5),点的大小代表了富集到的基因数量,展示在下方的通路显著性较高。

图5 差异基因的KEGG富集
Fig.5 KEGG enrichment of DEGs
具有显著差异且注释到基因数量较多的通路为:碳代谢、氨基酸代谢与蛋白酶体。其中氨基酸代谢普遍上调(40/66),可能由于胁迫刺激细胞内蛋白合成。碳代谢普遍下调(43/69),可能由于高浓胁迫压力,导致糖转运与EMP途径无法快速响应,相比常浓度麦汁更晚进入稳定耗糖阶段,也可能是由于细胞资源被用于应对胁迫压力,影响能量代谢表达水平。KEGG富集与GO富集分析结果基本一致。
2.3 代谢组分析
2.3.1 差异代谢物分析
本研究采用了与转录组一致的样本进行代谢组检测,设置6个生物学平行。下机数据经过图谱处理与数据库搜索,对代谢物进行定性与定量,总共分辨到326种代谢物。质控数据显示代谢组数据可靠、准确。主成分分析显示2组间样本分散且组内样本聚集,表明2组间存在显著差异代谢物且代谢物含量稳定。
设置显著差异代谢物筛选的阈值为VIP>1.0,|log2fold change|≥1且P<0.05,最终筛选到30个显著差异代谢物,占总代谢物数量的9.20%,其中18个代谢物显著上调,12个代谢物显著下调。显著差异代谢物中最大上调倍数为2.75倍,最大下调倍数为2.31倍。代谢物调整具有滞后性,作为对转录组数据的支撑,筛选到的显著代谢物可以论证相关基因通路的表达差异,但是未能表现出显著差异的代谢物不能说明相关基因通路无意义。上述数据表明酵母在短时间内便对高浓麦汁应答,基因表达差异引起了部分代谢物含量变化。
2.3.2 差异代谢物的KEGG富集分析
通过相同的方法对差异代谢物进行KEGG富集分析,选取最显著的18个通路绘制散点图(图6)。其中有10个通路与氨基酸代谢或能量代谢相关,其中最显著的5个通路内有3个为氨基酸代谢。如表3所示,天冬氨酸、丝氨酸、精氨酸与蛋氨酸含量显著上调。说明胞内氨基酸和氨酰化合物在高浓度麦汁刺激下合成加速,与转录组结果一致。而细胞内糖类化合物还没有显著变化,说明了碳代谢普遍下调不是因为胞内糖分过量酵解使得ATP积累导致的反馈抑制,而可能与麦汁胁迫压力相关。
2.4 重要差异基因筛选与分析
转录组分析筛选到显著差异基因并富集到能量代谢、氨基酸代谢与蛋白酶体,代谢组分析验证了氨基酸代谢途径的基因上调,并揭示了碳代谢途径下调的原因。对代谢物和差异基因进行关联分析,并结合酵母SGD数据库(saccharomyces genome database)与

图6 差异代谢物的KEGG富集
Fig.6 KEGG enrichment of differential metabolites
表3 显著差异氨基酸
Table 3 Amino acids with significantly different content
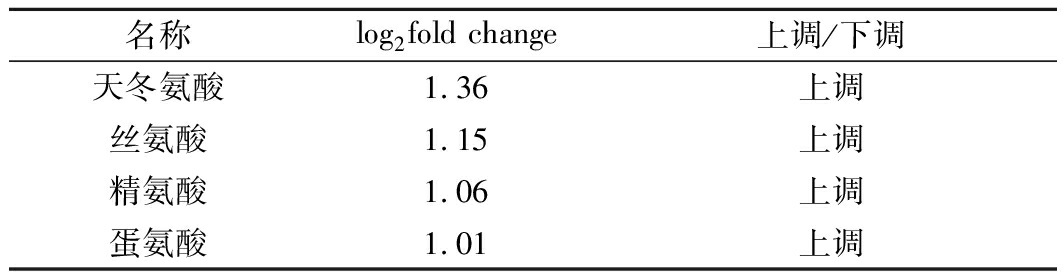
名称log2fold change上调/下调天冬氨酸1.36上调丝氨酸1.15上调精氨酸1.06上调蛋氨酸1.01上调
现有研究文献[18-20],筛选获得了13个重要差异基因。如表4所示,共确定了13个重要差异基因,其中8个基因与碳代谢相关,2个基因与己糖转运相关,3个基因与氨基酸的生物合成相关。
表4 重要差异基因
Table 4 Important DEGs

基因名称log2 fold change上调/下调描述代谢通路HXT12.84上调己糖转运蛋白1/HXT21.08上调己糖转运蛋白2/HXK1-1.05下调己糖激酶糖酵解/糖异生(map00010)PGM2-1.39下调磷酸葡萄糖变位酶糖酵解/糖异生(map00010)PGK1-1.48下调磷酸甘油酸激酶糖酵解/糖异生(map00010)GPM1-1.29下调磷酸甘油酸变位酶糖酵解/糖异生(map00010)GPM2-1.39下调磷酸甘油酸变位酶糖酵解/糖异生(map00010)TSL11.77上调海藻糖6-磷酸合酶/磷酸酶复合亚基淀粉和蔗糖代谢(map00500)TPS31.23上调海藻糖6-磷酸合酶/磷酸酶复合亚基淀粉和蔗糖代谢(map00500)TPS21.80上调海藻糖磷酸酶淀粉和蔗糖代谢(map00500)AAT12.76上调天冬氨酸转氨酶氨基酸的生物合成(map01230)SER22.24上调丝氨酸磷酸酶氨基酸的生物合成(map01230)ARG41.73上调精氨酸裂解酶氨基酸的生物合成(map01230)
代谢组中显著上调的氨基酸与转录组中显著上调的氨基酸相关基因(AAT1、SER2与ARG4)完全一致,除显著差异基因之外,氨基酸生物合成途径普遍上调,表明拉格酵母在应答高浓度麦汁时胞内蛋白质与酶合成活跃。氨基酸的积累对细胞抗逆性能有着积极作用[21],比如带电氨基酸如精氨酸对细胞耐低温有着明显效果[22]。氨基酸的加速合成与积累是拉格酵母细胞适应外界环境的应答策略,有助于细胞维持生长代谢平衡。
高浓度麦汁对拉格酵母细胞最明显的压力是高浓度的葡萄糖与麦芽糖。EMP途径的基因全线下调,包括关键的HXK1与PGK1,同一代谢通路上下游的PGM2、GPM1与GPM2也显著下调。EMP途径的显著下调可能是拉格酵母在高浓麦汁中起酵缓慢的主要原因。受到高浓度麦汁胁迫,己糖激酶HXK1与磷酸甘油酸激酶PGK1等关键基因表达下调,糖酵解速度放缓导致细胞内ATP生产减少,不利于细胞其他代谢活动开展。同时细胞内海藻糖合成相关基因显著上调(TSL1、TPS3与TPS2),海藻糖积累对细胞代谢平衡有重要作用[23]。
同时在重要通路之外观测到了HXT家族基因的显著变化(HXT1与HXT2),HXT家族编码己糖转运蛋白。已有研究表明HXT1的表达和发酵初期的极端高糖相关[24],所以HXT1可能是拉格酵母应答高浓葡萄糖的主要基因。HXT2同时具有低浓与高浓的底物特异性亲和力[25],与HXT1的诱导系统不同,HXT2可能受到环境因素如渗透压、ATP缺乏等调控。
3 结论
本研究通过浓度梯度麦汁平板胁迫测定及发酵实验,比较了常浓度与高浓度下的酵母特性。通过对拉格酵母M14在12 °P与24 °P麦汁处理2 h后的样本进行转录组测序、代谢组检测与关联分析,共获得191个显著差异基因,其中38个上调,153个下调;共获得30个显著差异代谢物,其中18个上调,12个下调。通过GO富集与KEGG富集分析,结合SGD数据库,筛选获得13个重要差异基因(HXT1、HXT2、HXK1、PGM2、PGK1、GPM1、GPM2、TSL1、TPS3、TPS2、AAT1、SER2与ARG4),差异基因主要与糖酵解、海藻糖合成、氨基酸合成和己糖转运相关。本研究初步揭示了拉格酵母应对高浓度麦汁的应答机制,为高浓酿造优良酵母的选育提供技术参考。
[1] LINKO M,HAIKARA A,RITALA A,et al.Recent advances in the malting and brewing industry[J].Journal of Biotechnology,1998,65(2-3):85-98.
[2] SAERENS S,DUONG C,NEVOIGT E.Genetic improvement of brewer’s yeast:Current state,perspectives and limits[J].Applied Microbiology and Biotechnology,2010,86(5):1 195-1 212.
[3] PULIGUNDLA P,SMOGROVICOVA D,MOK C,et al.Recent developments in high gravity beer-brewing[J].Innovative Food Science & Emerging Technologies,2020,64.DOI:10.1016/j.ifsect.2020.102399.
[4] BURPHAN T,TATIP S,LIMCHAROENSUK T,et al.Enhancement of ethanol production in very high gravity fermentation by reducing fermentation-induced oxidative stress in Saccharomyces cerevisiae[J].Scientific Reports,2018,8(1).DOI:10.1038/S41598-018-31558-4.
[5] 郭立芸. 18 °P高浓酿造抗葡萄糖阻遏效应啤酒酵母的选育[J].食品与发酵工业,2015,41(4):91-96.
GUO L.Selection of brewing yeast with glucose repression resistance under the very high gravity brewing (18 °P)[J].Food and Fermentation Industries,2015,41(4):91-96.
[6] VIRVE V,JOHN L.Overexpressed maltose transporters in laboratory and lager yeasts:Localization and competition with endogenous transporters[J].Yeast,2018,35(10):567-576.
[7] LEI H,FENG L,PENG F,et al.Amino acid supplementations enhance the stress resistance and fermentation performance of Lager yeast during high gravity fermentation[J].Applied Biochemistry & Biotechnology,2018,187(1):540-555.
[8] WANG Z,GERSTEIN M,SNYDER M.RNA-Seq:A revolutionary tool for transcriptomics[J].Nature Reviews Genetics,2009,10(1):57-63.
[9] HUBMANN G,FOULQUIE-MORENO M,NEVOIGT E,et al.Quantitative trait analysis of yeast biodiversity yields novel gene tools for metabolic engineering[J].Metabolic Engineering,2013,17(1):68-81.
[10] XU X,WANG J,BAO M,et al.Reverse metabolic engineering in lager yeast:impact of the NADH/NAD+ ratio on acetaldehyde production during the brewing process[J].Applied Microbiology and Biotechnology,2019,103(2):869-880.
[11] LIU C,LI Q,NIU C,et al.Genome sequence of the lager-brewing yeast Saccharomyces sp. strain M14,used in the high-gravity brewing industry in China[J].Genome Announcements,2017,5(43):e01 194-01 117.
[12] 顾国贤. 酿造酒工艺学[M].北京:中国轻工业出版社,1996.
GU G X.Brewing Technology[M].Beijing:China light industry press,1996
[13] 李崎, 刘春凤.ASBC分析方法[M].北京:中国轻工业出版社,2012.
LI Q,LIU C.ASBC Analysis Method[M].Beijing:China Light Industry Press,2012.
[14] ANDERS S,HUBER W.Differential expression analysis for sequence count data[J].Geneome Biology,2010,11:R106.
[15] YUAN M,BREITKOPF S B,YANG X,et al.A positive/negative ion-switching,targeted mass spectrometry-based metabolomics platform for bodily fluids,cells,and fresh and fixed tissue[J].Nature Protocols,2012,7(5):872-881.
[16] SWAN T M,WATSON K.Membrane fatty acid composition and membrane fluidity as parameters of stress tolerance in yeast[J].Canadian Journal of Microbiology,1997,43(1):70-77.
[17] BUBIS J A,SPASSKAYA D S,GORSHKOV V A,et al.Rpn4 and proteasome-mediated yeast resistance to ethanol includes regulation of autophagy[J].Applied Microbiology & Biotechnology,2020,104(9):4 027-4 041.
[18] PIRES E J.Biochemistry of Beer Fermentation[M].Wiley-Blackwell,2015.
[19] LEI H,XU H,FENG L,et al.Fermentation performance of lager yeast in high gravity beer fermentations with different sugar supplementations[J].Journal of Bioence & Bioengineering,2016,122(5):583-588.
[20] PIDDOCKE M P,KREISZ S,HELDT-HANSEN H P,et al.Physiological characterization of brewer′s yeast in high-gravity beer fermentations with glucose or maltose syrups as adjuncts[J].Applied Microbiology and Biotechnology, 2009,84(3):453-464.
[21] NGUYEN T D,WALKER M E,GARDNER J M,et al.Appropriate vacuolar acidification in Saccharomyces cerevisiae is associated with efficient high sugar fermentation[J].Food Microbiology,2018,70(1):262-268.
[22] H TAKAGI F I S N.Isolation of freeze-tolerant laboratory strains of Saccharomyces cerevisiae from proline-analogue-resistant mutants[J].Applied Microbiology & Biotechnology,1997,47(1):405-411.
[23] TREVISOL E T V,PANEK A D,DE MESQUITA J F,et al.Regulation of the yeast trehalose-synthase complex by cyclic AMP-dependent phosphorylation[J].Biochimicanet Biophysica Acta-General Subjects,2014,1 840(6):1 646-1 650.
[24] WIECZORKE R,KRAMPE S,WEIERSTALL T,et al.Concurrent knock-out of at least 20 transporter genes is required to block uptake of hexoses in Saccharomyces cerevisiae[J].Febs Letters,1999,464(3):123-128.
[25] MARC P,KATTIE L,REMY M,et al.Analysis of Saccharomyces cerevisiae hexose carrier expression during wine fermentation:both low-and high-affinity Hxt transporters are expressed[J].Fems Yeast Research,2010,5(4-5):351-361.