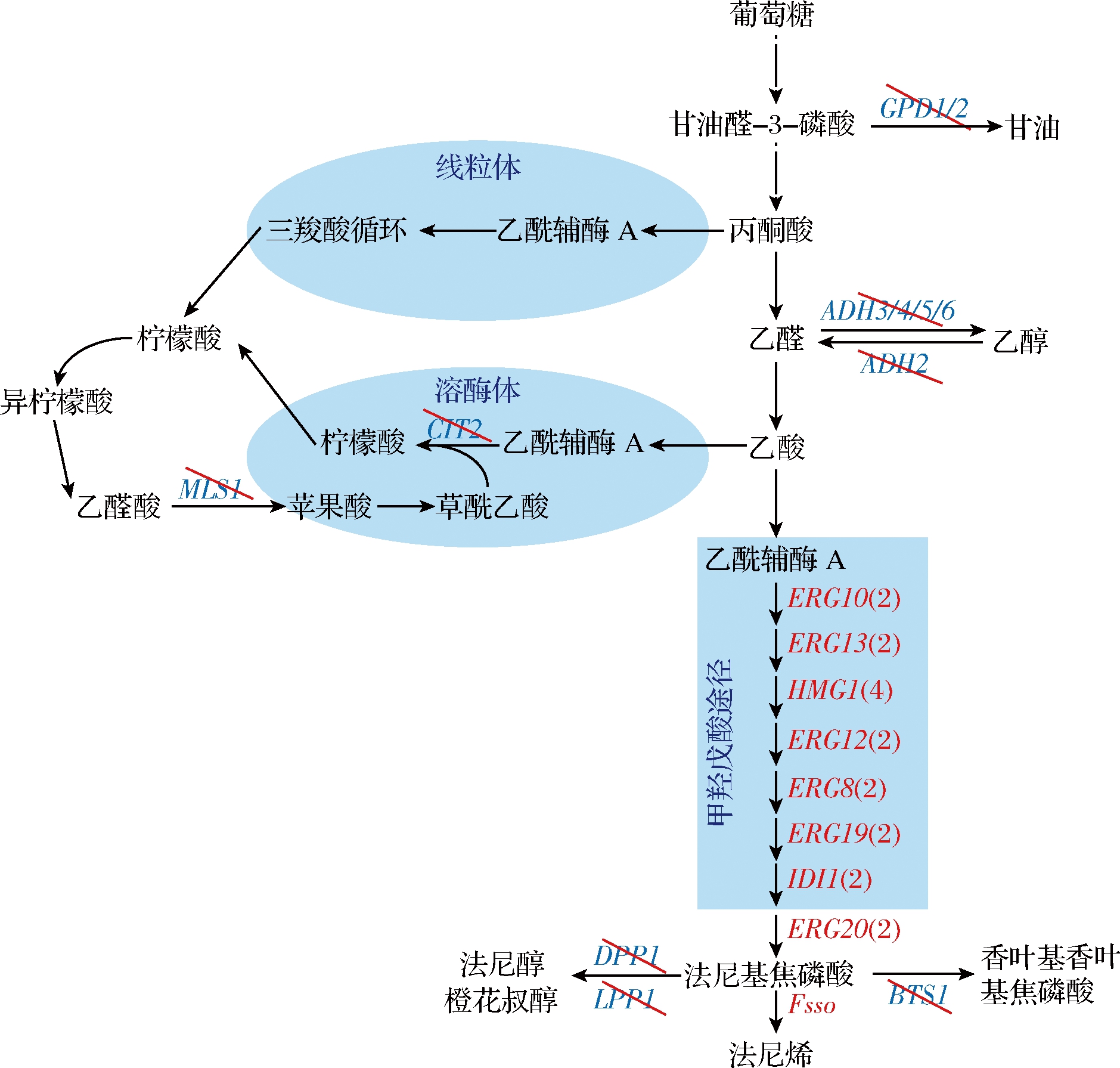
图1 酿酒酵母法尼烯合成途径和关键支路
Fig.1 The biosynthesis pathway and branches of
farnesene production in Saccharomyces cerevisiae
近些年,酿酒酵母因为自身拥有甲羟戊酸代谢途径,被广泛应用在萜类化合物合成的研究中[1-3]。在酿酒酵母中,甲羟戊酸途径通过转化前体胞质乙酰辅酶A提供基本五碳单元异戊二烯基焦磷酸和二甲基丙烯焦磷酸去合成萜类化合物前体香叶基焦磷酸、法尼基焦磷酸(farnesyl diphosphate,FPP)和香叶基香叶基焦磷酸。通过过表达甲羟戊酸途径关键限速酶3-羟基-3-甲基戊二酸单酰辅酶A还原酶(HMGR-CoA reductase,HMGR)编码基因(HMG1和tHMG1)或者过表达甲羟戊酸途径所有基因可以增强甲羟戊酸途径代谢流,继而提高萜类化合物的产量[4-6]。为了增强丙酮酸脱氢酶旁路途径合成前体胞质乙酰辅酶A的能力,通过缺损乙醇脱氢酶(ethanol dehydrogenase,ADH)编码基因(ADH1,3, 4, 5, 6)和甘油三磷酸脱氢酶(glycerol-3-phosphate dehydrogenase,GPD)编码基因(GPD1, 2)可以减少副产物乙醇和甘油的合成,使更多的碳代谢流流向前体胞质乙酰辅酶A的合成,提高酿酒酵母合成萜类化合物的能力[7-9]。同时,有报道通过缺损柠檬酸合酶(citrate synthase)(CIT2)和苹果酸合酶(malate synthase)(MLS1)基因减少胞质乙酰辅酶A流向其他细胞器[10-11]。此外,FPP下游途径也受到众多研究者的关注,通过启动子的替换可以降低甾醇合成途径角鲨烯合酶(squalene synthase)(ERG9)的表达水平,或者通过Erg9P降解的方法去减少FPP流向支路甾醇合成途径[12-15]。二酰基甘油二磷酸磷酸酶(diacylglycerol pyrophosphate phosphatase)(DPP1)和脂磷酸磷酸酶(lipid phosphate phosphatase)(LPP1)基因主要负责类异戊二烯焦磷酸去磷酸化[16],通过缺损DPP1和LPP1基因可以弱化FPP转化合成法尼醇,继而提高萜类化合物的产量[6, 17-18]。
法尼烯(C15H24),包括α和β 2种倍型,是一种挥发性较强且不溶于水的油性物质。有报道表明,法尼烯在植物中具有抵御害虫的能力,在医药行业可以作为维生素E生产的前体,在能源行业可以作为石油的替代品[19]。尽管上面描述的代谢操作策略已经被应用于酿酒酵母合成萜类化合物的研究,但却没有将所有这些代谢操作策略在同一个宿主菌中进行比较[20-21]。本研究以法尼烯为评价产物,利用基因代谢工程操作去构建不同基因缺损法尼烯合成菌株(图1)。首先,结合本实验室前期构建的甲羟戊酸途径强化菌株,通过染色体多拷贝整合法尼烯合成酶编码基因增强FPP转化合成法尼烯的能力。其次,评价缺损乙醇、甘油和法尼醇合成支路关键基因和胞质乙酰辅酶A转运至其他细胞器关键基因对法尼烯合成的影响。然后,评估缺损GAL相关基因对GAL启动子控制基因转录水平的影响。最后,将获得的有效基因缺损操作菌在5 L发酵罐进行补料分批发酵。
1.1.1 菌株、质粒和培养基
本实验所用菌株和质粒信息详见表1。本实验所用培养基115 ℃灭菌20 min。LB培养基(g/L):蛋白胨10,酵母粉5,氯化钠10;YPD培养基(g/L):酵母粉10,蛋白胨20,葡萄糖20;发酵培养基(g/L):酵母粉10,蛋白胨20,葡萄糖20,10%(体积分数)十二烷。固体培养基均加入20 g/L琼脂粉。氨苄青霉素(ampicillin,Amp)添加终质量浓度为100 μg/mL,用于大肠杆菌转化菌株的生长和筛选。遗传霉素(geneticin,G418)、潮霉素B(hygromycin,Hyg B)和硫酸诺尔斯菌素(nourseothricin sulfate,NTC)添加终质量浓度分别为500、500、100 μg/mL,用于酿酒酵母转化菌株的生长和筛选。

图1 酿酒酵母法尼烯合成途径和关键支路
Fig.1 The biosynthesis pathway and branches of
farnesene production in Saccharomyces cerevisiae
表1 本实验涉及菌株和质粒
Table 1 Strains and plasmids used in this study

1.1.2 引物
本研究所用引物见附件1(https://kns.cnki.net/kcms/detail/detail.aspx?dbcode=CAPJ&dbname=CAPJLAST&filename=SPFX20210518004),所有引物由苏州金唯智生物科技有限公司合成。
1.1.3 试验试剂
本实验所用PrimeSTAR DNA polymerase、pMD-19T simple vector、T4 DNA ligase、DNA marker、快速限制性内切酶SacⅡ,大连TAKARA公司;其他快速限制性内切酶,美国Thermo Fisher Scientific公司;2×Taq PCR Master Mix,杭州宝赛公司;G418、Hyg B和NTC,上海生工公司;Amp、醋酸锂、PEG3350、色谱级甲醇、色谱级乙腈、法尼烯标准品(HPLC ≥ 90%),美国Sigma公司;鲑鱼精DNA,北京索莱宝科技有限公司;质粒小提取试剂盒、DNA纯化试剂盒、DNA凝胶回收试剂盒,苏州AXYGEN公司;蛋白胨、酵母粉,OXOID公司;十二烷,上海麦克林生化科技有限公司。
1.1.4 仪器与设备
PCR基因扩增仪和凝胶成像系统,美国Bio-Rad公司;小型高速离心机,美国Thermo Fisher Scientific公司;UV-2100型分光光度计,美国UNICO公司;超低温冰箱,美国Eppendorf公司;超净工作台,苏州净化设备有限公司;生化培养箱,上海博讯医疗生物仪器股份有限公司;恒温摇床,上海知楚仪器有限公司;高压灭菌锅,日本SANYO公司;5 L玻璃发酵罐,迪必尔生物工程上海有限公司。
1.2.1 法尼烯合成酶多拷贝表达质粒的构建
法尼烯合成酶多拷贝表达质粒具体构建过程见附件2(https://kns.cnki.net/kcms/detail/detail.aspx?dbcode=CAPJ&dbname=CAPJLAST&filename=SPFX20210518004)。
1.2.2 缺损基因质粒的构建
缺损质粒具体构建过程见附件3(https://kns.cnki.net/kcms/detail/detail.aspx?dbcode=CAPJ& dbname=CAPJLAST&filename=SPFX20210518004)。
1.2.3 重组菌的筛选
本实验所有线性化的表达质粒均通过醋酸锂转化法整合至酿酒酵母染色体。YPD固体培养基添加终质量浓度500 μg/mL G418用于整合法尼烯合成酶编码基因转化子筛选;选择固体培养基用于筛选氨基酸缺陷型标记转化子筛选。转化平板放置于30 ℃培养箱,转化子通过基因组PCR验证。
1.2.4 重组菌法尼烯合成摇瓶发酵
重组菌于YPD平板划线,30 ℃培养箱活化2 d。挑单菌落接种于20 mL YPD液体培养基,30 ℃,200 r/min培养18 h。按照初始OD600=0.1转接至30 mL发酵培养基,30 ℃,200 r/min培养120 h。
1.2.5 补料分批发酵
重组菌于YPD平板划线,30 ℃培养箱活化2 d。挑单菌落接种于20 mL YPD液体培养基,30 ℃,200 r/min培养18 h作为一级种子液。按照初始1%接种量转接至50 mL YPD培养基,30 ℃,200 r/min培养18 h作为二级种子液。
补料分批发酵方法:发酵初始装液量为2 L YPD培养基,十二烷20% (体积分数),通气量1 L/(L·min),温度30 ℃,添加氨水控制pH=5.5,搅拌转速400 r/min。补料方式为连续补料,葡萄糖溶液质量浓度为600 g/L。
1.2.6 法尼烯HPLC检测
本实验使用ThermoFisher UltiMate 3000高效液相色谱对α-法尼烯进行含量测定。检测条件:Waters C18色谱柱(4.6 mm×250 mm×5 μm),紫外检测波长为232 nm,流动相为90%甲醇、5%乙腈和5%超纯水,流速为0.8 mL/min,柱温为40 ℃,样品检测时间为15 min。
1.2.7 发酵上清液HPLC检测
本实验使用高效液相色谱对乙醇和甘油进行含量测定。检测条件:示差检测器,Shodex糖柱(SC-1011),流动相为5%稀硫酸溶液,流速为0.8 mL/min,柱温为50 ℃,样品检测时间为25 min。
2.1.1 多拷贝法尼烯合成酶表达质粒的构建
利用酶切连接的方法获得Fsso基因多拷贝表达质粒Ts-rso (图2-A)。质粒酶切产物琼脂糖电泳分析如图2-B所示。SacⅡ单酶切质粒Ts-rso,在5.95 kpb和2.71 kbp位置有2条带(泳道1);XbaⅠ单酶切质粒Ts-rso,在4.69 kpb和3.97 kbp位置有2条带(泳道2);NheⅠ和SalⅠ双酶切质粒Ts-rso,在1.71 kpb和6.96 kbp位置有2条带(泳道3)。将质粒Ts-rso送至上海生工测序,序列无碱基突变。说明质粒Ts-rso构建成功。
2.1.2 基因缺损质粒的构建
利用引物605-YAN-U和605-YAN-D菌落PCR筛选(图2-C,目的条带605 bp)获得质粒BTS-ADH3、BTS-ADH4、BTS-ADH5、BTS-ADH6、BTS-GPD1、BTS-GPD2、BTS-GAL1、BTS-GAL7、BTS-GAL10、BTS-CIT2、BTS-MLS1、BTS-BTS1、BTS-LPP1和BTS-DPP1送至上海生工测序,质粒序列无碱基突变。说明上述所有质粒构建成功。
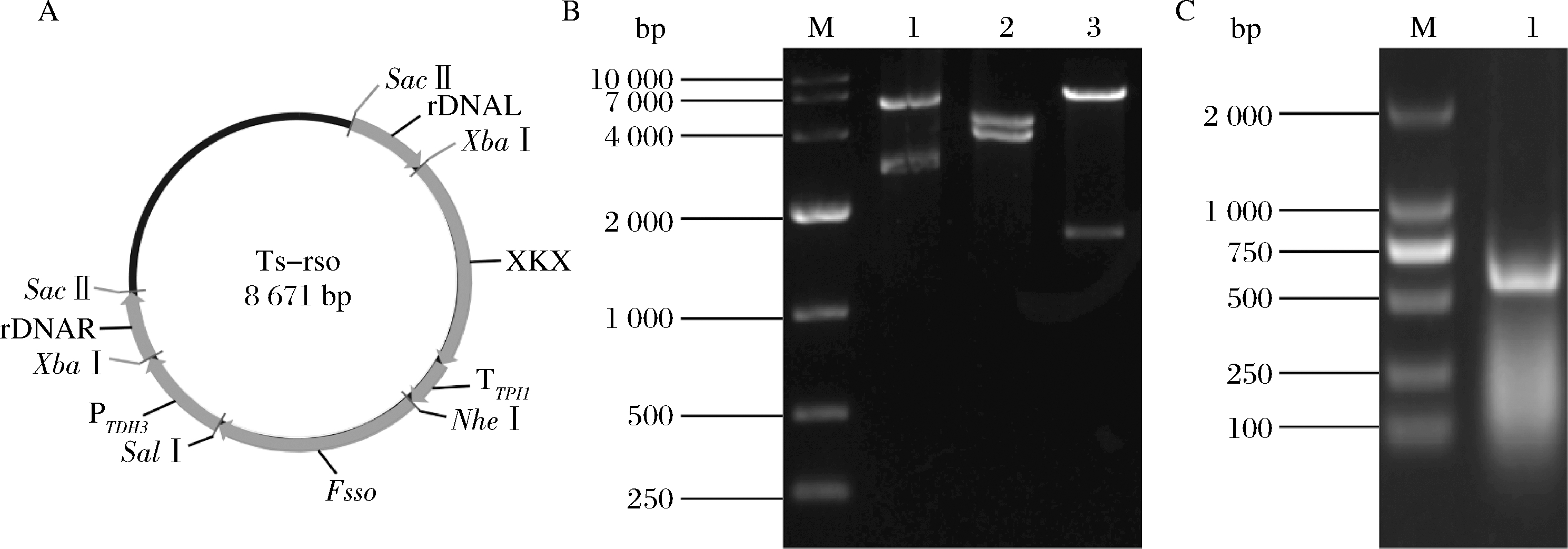
A-质粒Ts-rso物理图谱;B-质粒Ts-rso酶切验证;C-菌落PCR验证
图2 质粒Ts-rso的构建和BTS系列质粒菌落PCR筛选
Fig.2 The physic map and verification of plasmid Ts-rso and colony PCR
2.1.3 正确转化子的筛选
将质粒Ts-rso (限制性内切酶SacⅡ消化)的线性化产物转化酿酒酵母WHE,在G418抗性YPD平板上筛选转化子,提取基因组,用引物Fsso-YAN-U和Fsso-YAN-D进行PCR确认(图3-A),得到大小为1 700 bp左右的条带,说明在rDNA位点成功整合Fsso基因,获得菌株WHE4。
将质粒pHcas9分别与质粒BTS-ADH3、BTS-ADH4、BTS-ADH5、BTS-ADH6、BTS-GPD1、BTS-GPD2、BTS-GAL1、BTS-GAL7、BTS-GAL10、BTS-CIT2、BTS-MLS1、BTS-BTS1、BTS-LPP1和BTS-DPP1转化酿酒酵母WHE4,在HygB和NTC抗性YPD平板上筛选转化子,提取基因组,用基因开放阅读框之外的引物(见表中验证用引物)进行PCR确认(图3-B~图3-G),获得不同组合基因缺失菌株WHE4-10、WHE4-13、WHE4-14、WHE4-15、WHE4-21、WHE4-31、WHE4-32和WHE4-33。
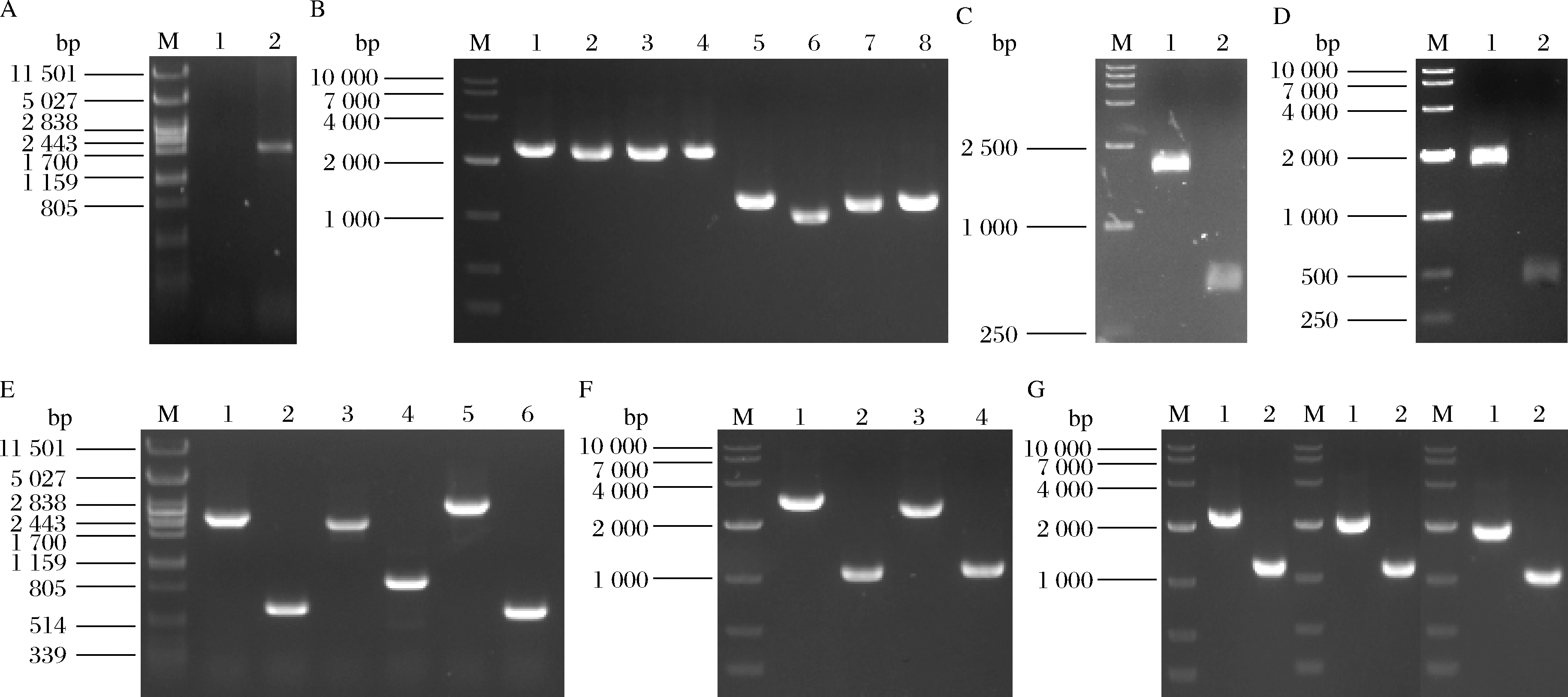
A-Fsso基因整合确认;B-ADH3、4、5、6基因缺损确认;C-GPD1基因缺损确认;D-GPD2基因缺损确认;
E-GAL1、7、10基因缺损确认;F-CIT2和MLS1基因缺损确认;G-BTS1、DPP1和LPP1基因缺损确认
图3 基因整合和缺损确认
Fig.3 PCR verification of integration and deletion
2.2.1 多拷贝整合法尼烯合成酶菌株的发酵
随机挑选转化平板上47个转化子,在30 mL发酵培养基中进行发酵实验并测定法尼烯的产量,结果如图4所示。从图4中可以看出不同转化子法尼烯的产量在235 mg/L左右,选择菌株WHE4(法尼烯产量为253.24 mg/L)作为接下来研究的出发菌。
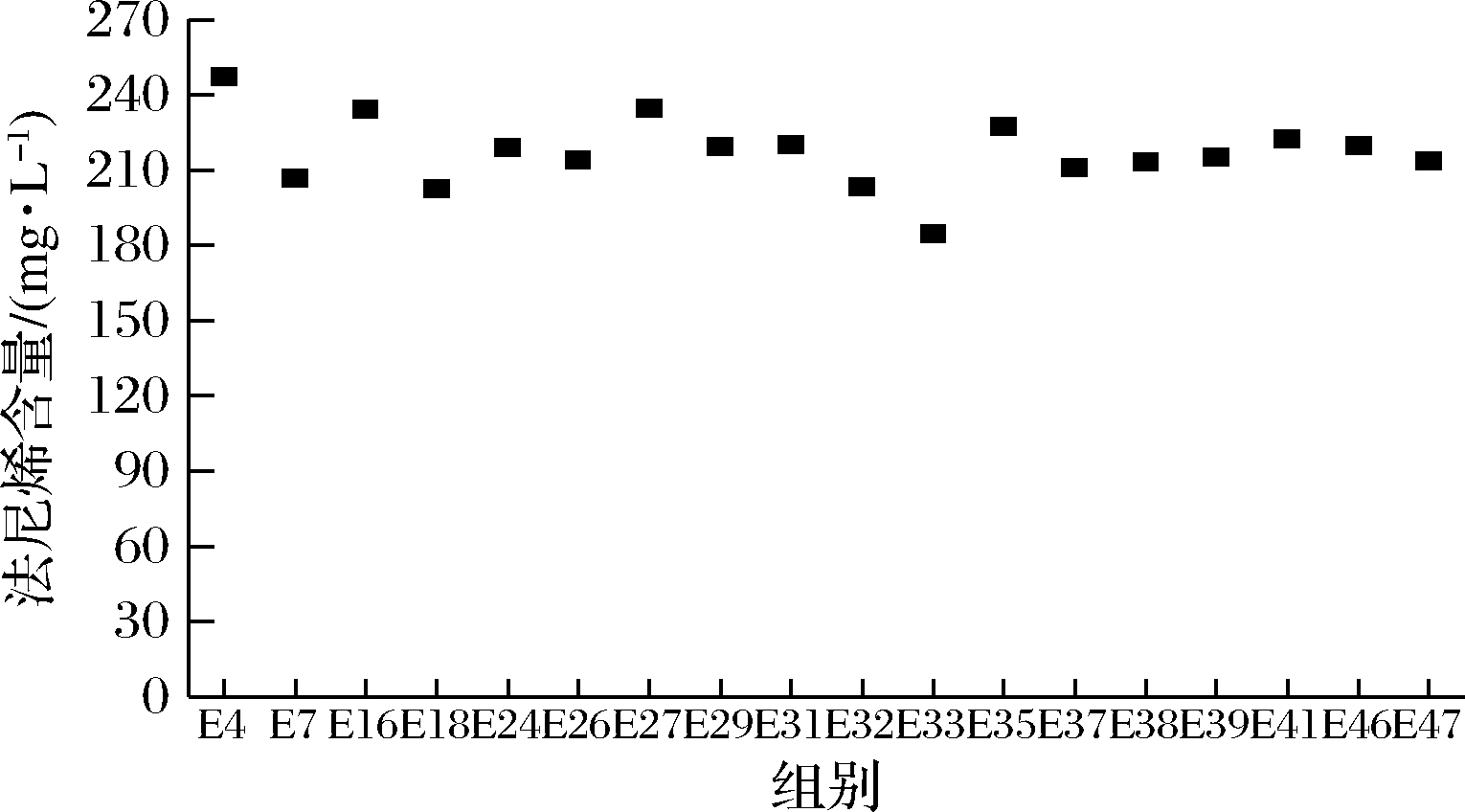
图4 法尼烯合成基因整合菌株法尼烯产量
Fig.4 Farnesene production of engineered strains
integrated with Fsso
2.2.2 乙醇和甘油积累量的比较
将重组菌WHE4-10、WHE4-14和WHE4-15与WHE4在50 mL YPD培养基中进行摇瓶发酵实验并测定乙醇和甘油的积累量和OD600,结果如图5所示,条件1和条件2分别为普通250 mL摇瓶和250 mL挡板摇瓶。菌株WHE4、WHE4-10、WHE4-14和WHE4-15在普通摇瓶中乙醇的最高积累量为8.64、8.77、8.69、8.96 g/L,在挡板摇瓶中乙醇的最高积累量为8.25、8.29、8.36、8.22 g/L,ADH3,ADH4,ADH5和ADH6基因的缺损未能减少乙醇的积累,使用挡板摇瓶进行发酵可以减少乙醇的合成(图5-A)。菌株WHE4、WHE4-10、WHE4-14和WHE4-15在普通摇瓶中甘油的最高积累量为0.99、0.91、0.85、0.66 g/L,在挡板摇瓶中甘油的最高积累量为1.07、0.98、0.92、0.72 g/L,GPD1和GPD2基因的单缺损可以减少甘油的积累,使用挡板摇瓶进行发酵导致甘油的量上升(图5-B)。菌株WHE4、WHE4-10、WHE4-14和WHE4-15在普通摇瓶中最高OD600值为5.15、5.14、5.05和5.47,在挡板摇瓶中最高OD600值为5.36、5.35、5.19和5.55,缺损GPD1、GPD2、ADH3、ADH4、ADH5和ADH6基因对菌体的生长几乎没有影响,使用挡板摇瓶进行发酵OD600有所上升(图5-C)。以上结果表明,通过缺损ADH3、ADH4、ADH5和ADH6基因不能减少乙醇的积累量;缺损GPD1和GPD2基因可以减少甘油的积累。
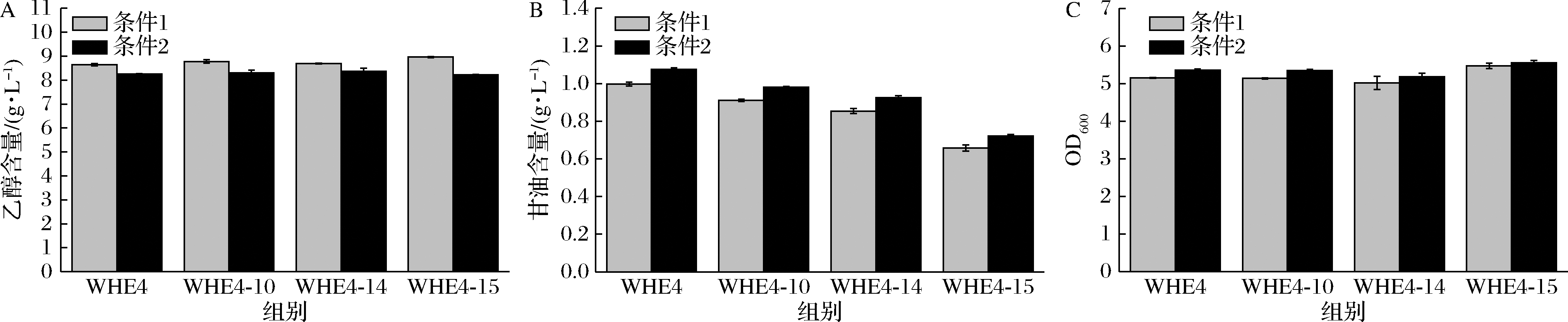
A-乙醇含量的比较;B-甘油含量的比较;C-细胞生长的比较
图5 基因缺损对生长、乙醇含量和甘油含量的影响
Fig.5 Cell growth, ethanol and glycerol production of engineered strains
2.2.3 甲羟戊酸途径转录水平的比较
将重组菌WHE4和WHE4-21在50 mL YPD培养基中进行摇瓶发酵实验并测定甲羟戊酸途径所有基因转录水平变化,结果如图6所示。与菌株WHE4相比,发酵30 h时,甲羟戊酸途径ERG10、ERG13、HMG1、ERG12、ERG8、MVD1、IDI1和ERG20基因的转录水平均处于下调状态。以上结果表明,缺损基因GAL1、GAL7和GAL10不能提高甲羟戊酸途径的强度。与之前研究中报道的缺损GAL1、GAL7和GAL10基因启动子相比[22],可能直接缺损GAL1、GAL7和GAL10基因启动子更加有利于Gal4P与目的GAL启动子的结合,提高目的基因的表达水平。

图6 GAL1、GAL7和GAL10基因缺损对甲羟戊酸途径
基因转录水平的影响
Fig.6 The effects of deletion of GAL1, GAL7 and GAL10 on
transcriptional levels of the mevalonate pathway genes
2.2.4 基因缺损菌株法尼烯产量的比较
将重组菌WHE4-10、WHE4-13、WHE4-14、WHE4-15、WHE4-21、WHE4-31、WHE4-22和WHE4-33在30 mL发酵培养基中进行摇瓶发酵实验并测定法尼烯的产量,结果如图7所示。菌株WHE4-13、WHE4-14、WHE4-15、WHE4-21和WHE4-31法尼烯产量均低于菌株WHE4,菌株WHE4-10、WHE4-32和WHE4-33法尼烯的产量均高于WHE4,分别为257.30、286.49、326.64 mg/L。以上结果表明,单独缺损GPD1、GPD2和LPP1基因、同时缺损CIT2和MLS1基因和同时缺损GAL1、GAL7和GAL10基因对法尼烯的合成无益,同时缺损BTS1和DPP1基因可以弱化FPP的损失,提高法尼烯的产量。
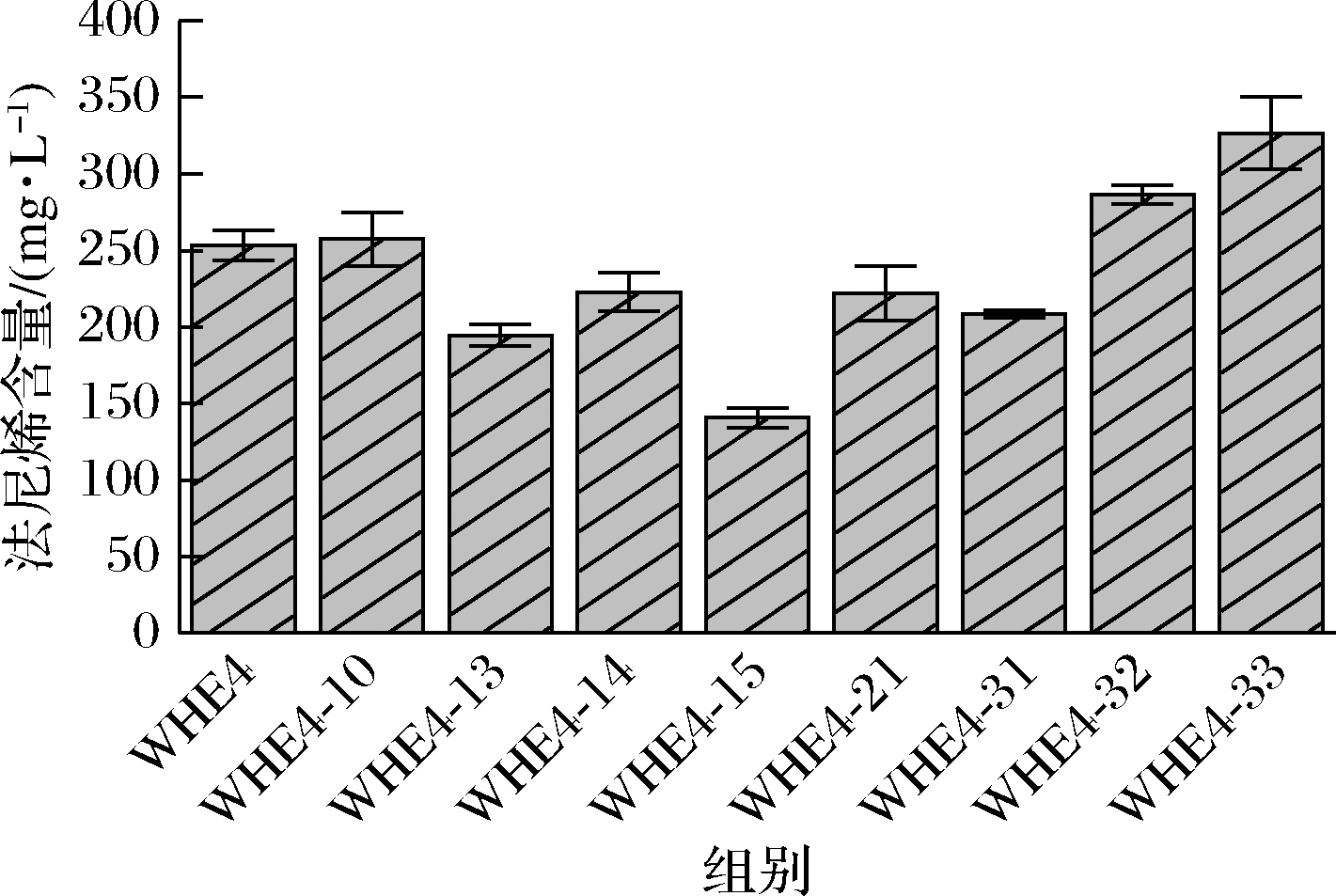
图7 不同菌株法尼烯合成
Fig.7 Farnesene production of engineered strains
2.2.5 5 L发酵罐补料分批发酵
将最佳重组菌WHE4-33在5 L发酵罐水平进行补料分批发酵,结果如图8所示。在初始20 g/L葡萄糖消耗结束后开始流加葡萄糖溶液(600 g/L),发酵液中葡萄糖的质量浓度始终控制在1.0 g/L以下,发酵总时长为144 h,最终乙醇和法尼烯的积累分别为70.06、1 578.91 mg/L。

图8 菌株WHE4-33在5 L发酵罐水平法尼烯的合成
Fig.8 WHE4-33 fed-batch fermentation in a 5 L bioreactor
近些年,酿酒酵母被广泛应用于萜类化合物合成的研究,构建一个酿酒酵母萜类化合物高效合成平台显得极为重要。本研究以酿酒酵母法尼烯合成菌株为研究对象,通过使用CRISPR-cas9基因编辑技术缺损酿酒酵母关键节点基因进行法尼烯合成影响研究。结果发现关键节点基因GPD1、GPD2、CIT2、MLS1、GAL1、GAL7、GAL10、BTS1和DPP1的缺损影响法尼烯的合成;从而说明关键节点基因涉及的甘油合成途径、胞质乙酰辅酶A转运途径、甲羟戊酸途径和FPP消耗分支途径影响法尼烯的合成。然而本研究在补料分批发酵时发现乙醇积累量较高,后续将通过增强乙醇吸收能力提高胞质乙酰辅酶A供给,进一步增强甲羟戊酸途径的代谢流,从而为代谢工程构建酿酒酵母萜类化合物高效合成平台提供参考价值。同时可与已有研究有效策略联合使用,从而构建酿酒酵母萜类化合物高效合成菌株。
[1] NAVALE G R, DHARNE M S, SHINDE S S.Metabolic engineering and synthetic biology for isoprenoid production in Escherichia coli and Saccharomyces cerevisiae [J].Applied Microbiology and Biotechnology, 2021, 105(2):457-475.
[2] WANG C L, LIWEI M, PARK J B, et al.Microbial platform for terpenoid production:Escherichia coli and yeast[J].Frontiers in Microbiology, 2018, 9:2 460.
[3] ZHANG C, HONG K.Production of terpenoids by synthetic biology approaches [J].Frontiers in Bioengineering and Biotechnology, 2020, 8:347.
[4] WESTFALL P J, PITERA D J, LENIHAN J R, et al.Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin [J].Proceedings of the National Academy of Sciences, 2012, 109(3):E111-E118.
[5] PARAMASIVAN K, MUTTURI S.Regeneration of NADPH coupled with HMG-CoA reductase activity increases squalene synthesis in Saccharomyces cerevisiae [J].Journal of Agricultural and Food Chemistry, 2017, 65(37):8 162-8 170.
[6] WANG J, JIANG W, LIANG C, et al.Overproduction of α-farnesene in Saccharomyces cerevisiae by farnesene synthase screening and metabolic engineering [J].Journal of Agricultural and Food Chemistry, 2021, 69(10):3 103-3 113.
[7] SCHADEWEG V, BOLES E.N-Butanol production in Saccharomyces cerevisiae is limited by the availability of coenzyme A and cytosolic acetyl-CoA [J].Biotechnology for Biofuels, 2016, 9:44.
[8] SONG J Y, PARK J S, KANG C D, et al.Introduction of a bacterial acetyl-CoA synthesis pathway improves lactic acid production in Saccharomyces cerevisiae [J].Metabolic Engineering, 2016, 35:38-45.
[9] DE SMIDT O, DU PREEZ J C, ALBERTYN J.Molecular and physiological aspects of alcohol dehydrogenases in the ethanol metabolism of Saccharomyces cerevisiae [J].FEMS Yeast Research, 2012, 12(1):33-47.
[10] KRIVORUCHKO A, SERRANO-AMATRIAIN C, CHEN Y, et al.Improving biobutanol production in engineered Saccharomyces cerevisiae by manipulation of acetyl-CoA metabolism [J].Journal of Industrial Microbiology and Biotechnology, 2013, 40(9):1 051-1 056.
[11] SONG T Q, WU N, WANG C, et al.Crocetin overproduction in engineered Saccharomyces cerevisiae via tuning key enzymes coupled with precursor engineering [J].Frontiers in Bioengineering and Biotechnology, 2020, 8:578005.
[12] PENG B Y, PLAN M R, CHRYSANTHOPOULOS P, et al.A squalene synthase protein degradation method for improved sesquiterpene production in Saccharomyces cerevisiae [J].Metabolic Engineering, 2017, 39:209-219.
[13] CALLARI R, MEIER Y, RAVASIO D, et al.Dynamic control of ERG20 and ERG9 expression for improved casbene production in Saccharomyces cerevisiae [J].Frontiers in Bioengineering and Biotechnology, 2018, 6:160.
[14] ASADOLLAHI M A, MAURY J, MØLLER K, et al.Production of plant sesquiterpenes in Saccharomyces cerevisiae:Effect of ERG9 repression on sesquiterpene biosynthesis [J].Biotechnology and Bioengineering, 2008, 99(3):666-677.
[15] HU T Y, ZHOU J W, TONG Y R, et al.Engineering chimeric diterpene synthases and isoprenoid biosynthetic pathways enables high-level production of miltiradiene in yeast [J].Metabolic Engineering, 2020, 60:87-96.
[16] FAULKNER A, CHEN X M, RUSH J, et al.The LPP1 and DPP1 gene products account for most of the isoprenoid phosphate phosphatase activities [J].The Journal of Biological Chemistry, 1999, 274(21):14 831-14 837.
[17] SCALCINATI G, KNUF C, PARTOW S, et al.Dynamic control of gene expression in Saccharomyces cerevisiae engineered for the production of plant sesquitepene α-santalene in a fed-batch mode [J].Metabolic Engineering, 2012, 14(2):91-103.
[18] HONG J, PARK S H, KIM S, et al.Efficient production of lycopene in Saccharomyces cerevisiae by enzyme engineering and increasing membrane flexibility and NAPDH production [J].Applied Microbiology and Biotechnology, 2019, 103(1):211-223.
[19] BENJAMIN K R, SILVA I R, CHERUBIM J P, et al.Developing commercial production of semi-synthetic artemisinin, and of β-farnesene, an isoprenoid produced by fermentation of Brazilian sugar [J].Journal of the Brazilian Chemical Society, 2016, 27(8):1 339-1 345.
[20] LIU Y H, JIANG X, CUI Z Y, et al.Engineering the oleaginous yeast Yarrowia lipolytica for production of α-farnesene [J].Biotechnology for Biofuels, 2019, 12(1):1-11.
[21] MEADOWS A L, HAWKINS K M, TSEGAYE Y, et al.Rewriting yeast central carbon metabolism for industrial isoprenoid production [J].Nature, 2016, 537(7 622):694-697.
[22] WANG F, LV X, XIE W P, et al.Combining Gal4p-mediated expression enhancement and directed evolution of isoprene synthase to improve isoprene production in Saccharomyces cerevisiae [J].Metabolic Engineering, 2017, 39:257-266.
附件1 本研究所用引物
本实验涉及引物
Primers used in this study

引物名称引物序列(5'-3')酶切位点rDNA-SacⅡ-1TCCCCGCGGGGATTTCCTCTAATCAGGTTCCACCSacⅡrDNA-XbaI-2GCTCTAGAGCTTTTACTGCGGAAGCXbaIrDNA-XbaI-3GCTCTAGAGCGGGCACCTGTCACTTXbaIrDNA-SacⅡ-4TCCCCGCGGGGAATGAGAGTAGCAAACGTAAGTCTAASacⅡXKX-XbaI-1GCTCTAGAGCTTCTATGTTCGGGTTCAGCXbaIXKX-2TATCCAAATTTCAACTGTTATATAGATGACCATGATTACGAACTCTTPI1-1GAGTTCGTAATCATGGTCATCTATATAACAGTTGAAATTTGGATATTPI1-2GAATAAACACACATAAACAAACAAAGATTAATATAATTATATAAAAATATPTDH3-1ATATTTTTATATAATTATATTAATCTTTGTTTGTTTATGTGTGTTTATTCPTDH3-XbaI-2GCTCTAGAGCATACTAGCGTTGAATGTTAGCGTCAXbaIXTP-SalI-7ACGCGTCGACTTTGTTTGTTTATGTGTGTTTATTCSalIXTP-NheI-8CTAGCTAGCTAGGATTAATATAATTATATAAAAATATNheI471-FAAGGAGGGTATTCTGGGCCTCCATGTC471-RTCTGCAGAATTCGTCGACGAGCTCGGTACGAL1-302RAATTGCTTGAAAAAGTCTTCGATCATTTATCTTTCACTGCGGAGGAL1-149FGAAGACTTTTTCAAGCAATTGTTTTAGAGCTAGAAATAGCAAGGAL7-302RTGGTTAGGTCAACAGGAGGCGATCATTTATCTTTCACTGCGGAGGAL7-149FGCCTCCTGTTGACCTAACCAGTTTTAGAGCTAGAAATAGCAAGGAL10-302RTGCAATACCTAGAGGCCTACGATCATTTATCTTTCACTGCGGAGGAL10-149FGTAGGCCTCTAGGTATTGCAGTTTTAGAGCTAGAAATAGCAAGCIT2-302RGGGAGCGTATGGGAAGGTTCGATCATTTATCTTTCACTGCGGAGCIT2-149FGAACCTTCCCATACGCTCCCGTTTTAGAGCTAGAAATAGCAAGMLS1-302RCCAGTAATTTGACGTTATCCGATCATTTATCTTTCACTGCGGAGMLS1-149FGGATAACGTCAAATTACTGGGTTTTAGAGCTAGAAATAGCAAGGPD1-302RTTTCACATTCCCAGGCGTCCCGATCATTTATCTTTCACTGCGGAGGPD1-149FGGACGCCTGGGAATGTGAAAGTTTTAGAGCTAGAAATAGCAAGGPD2-302RTCAGATTTTCGTCGCCGATCCGATCATTTATCTTTCACTGCGGAGGPD2-149FGATCGGCGACGAAAATCTGAGTTTTAGAGCTAGAAATAGCAAGADH3-302RGGTGAACAATGTTGACGTTCGATCATTTATCTTTCACTGCGGAGADH3-149FGAACGTCAACATTGTTCACCGTTTTAGAGCTAGAAATAGCAAGADH4-302RGGATCAGTAACGATCAAAGCGATCATTTATCTTTCACTGCGGAGADH4-149FGCTTTGATCGTTACTGATCCGTTTTAGAGCTAGAAATAGCAAGADH5-302RTCATGTGTGAAGCCAGTACCGATCATTTATCTTTCACTGCGGAGADH5-149FGGTACTGGCTTCACACATGAGTTTTAGAGCTAGAAATAGCAAGADH6-302RTGCGACACATAGCCGTCTTCGATCATTTATCTTTCACTGCGGAGADH6-149FGAAGACGGCTATGTGTCGCAGTTTTAGAGCTAGAAATAGCAAGBTS1-302RCTTCTCAAGGGAGCATTATCGATCATTTATCTTTCACTGCGGAGBTS1-149FGATAATGCTCCCTTGAGAAGGTTTTAGAGCTAGAAATAGCAAGDPP1-302RCCCATTGATGATCCTCTTCCGATCATTTATCTTTCACTGCGGAGDPP1-149FGGAAGAGGATCATCAATGGGGTTTTAGAGCTAGAAATAGCAAGLPP1-302RCTTGGAGTGCTTTTTAAGCCGATCATTTATCTTTCACTGCGGAGLPP1-149FGGCTTAAAAAGCACTCCAAGGTTTTAGAGCTAGAAATAGCAAGGAL1-90CAGTTTGTATTACTTCTTATTCAAATGTCATAAAAGTATCAACAAGTATACTTCTTTTTTTTACTTTGTTCAGAACAACTTCTCATTTTTGAL7-90TAGCAAAGATATAAAAGCAGGTCGGAAATATTTATGGGCATTATTAAAGAAAGTGGAATATTCATTCATATCATATTTTTTCTATTAACTGAL10-90GATATGTATATGGTGGTAATGCCATGTAATATGATTATTAAACTTTTTGCCAGCTTACTATCCTTCTTGAAAATATGCACTCTATATCTTCIT2-90AGGTTCTCAAAACTTTTTGTTTTAATAATACTAGTAACAAGAAAATTGGATTACATCCTACTTTTACACCCCTCTGCATATTTTTCTTTCMLS1-90TTTGAACTAAACAAAGTAGTAAAAGCACATAAAAGAATTAAGAAATCTCCCTTGCCCCAGTGTACACATATATAAAAATATATTCATGCCGPD1-90TGTACACCCCCCCCCTCCACAAACACAAATATTGATAATATAAAGATTTATTGGAGAAAGATAACATATCATACTTTCCCCCACTTTTTTGPD2-90CAATTCTCTTTCCCTTTCCTTTTCCTTCGCTCCCCTTCCTTATCAACACTCTCCCCCCCCCTCCCCCTCTGATCTTTCCTGTTGCCTCTT ADH3-90TCACAGTTAAAACTAGGAATAGTATAGTCATAAGTTAACACCATCTAGCGTGTTACGCACCCAAACTTTTTATGAAAGTCTTTGTTTATAADH4-90AACAACAAGTTTACATTTGCAACAACTAATAGTCAAATAAGAAAAAAAAATCGAACGAACTCATAAACGTCAATTATGCGTGTGCCTTATADH5-90ACCTAAGAAAATTATTTAACTACATATCTACAAAATCAAAGCATCTCTTTTGTAACGAATTTGATGAATATATTTTTACTTTTTATATAAADH6-90CATTCGAGGAAGAAATTCAACACAACAACAAGAAAAGCCAAAATCGTTGTCAAGCTCTTGATAAATGTAGCTCCTTTCTTTTTAACTGCT BTS1-90CTACTAATAGAAAGAGAACAAAGCGTTTACGAGTCTGGAAAATCAAATAAATTGATCAATCAAATTAGTGGAGGAAGATAGTCAGAAATADPP1-90TGTTAAAGGCAAAGAATCAGAATTAAATCATAGCAAACGACCAAAGAATAAAAAAGAATATATACTCCACATGACATACGAAATATACGTLPP1-90ACTCCTACATCAACGCCTAAGGAAACTCGTCATATTCTACCAAGGTAACACTTACAGAGTCCTATCAGGAAAGAATAAAAGCCGATCAAGFsso-YAN-UATGAATCACTCTTATGCTAACCFsso-YAN-DTTATCTCAATGGCTCAACAACC605-YAN-UGCTAAATGTACGGGCGACAGTCAC
续附表

引物名称引物序列(5'-3')酶切位点605-YAN-DCGCGTTGGCCGATTCATTAATGCAGGAL1-YAN-UTTTAGCCTTATTTCTGGGGAL1-YAN-DGGACAAAGGGTTCTCGTAGGAL7-YAN-UGAGGGTAATAACATAGGTGCGAL7-YAN-DCGTTTGGTAAAGTAGAGGGGAL10-YAN-UTCGCAATAGTTGTGAGTGAGAL10-YAN-DCCGGTGAAGACGAGGACCIT2-YAN-UCGTCTATCAGGGCGTCAGCIT2-YAN-DGCGTCATATTCGAGCAACMLS1-YAN-UTTCGTGCTTAGTGATGTCTCMLS1-YAN-DGCTCTGTTCCTTGGGTTTGPD1-YAN-UCAAACGCAACACGAAAGGPD1-YAN-DACAACCACGAGCCAACAGPD2-YAN-UGAAACAATAAGACGACGATGGPD2-YAN-DGAAATAGGTTGCGACAGCADH3-YAN-UATTCGCTCGTTACTACCTATADH3-YAN-DCTTGTACTCCAATGCCTGTADH4-YAN-UTTGTGATGATGGCTGTAGTADH4-YAN-DATAATGCGGCTGGTAAGADH5-YAN-UTAAATGACGCTCATACTCTTADH5-YAN-DAACCAATTTCTACTCCTCCADH6-YAN-UATTGCTGACTAAAGATTCCCADH6-YAN-DGAGATGAGGCAGAACATACCACT1-UTTGGTAACGAAAGATTCAGAGCCCACT1-DAAGATAGAACCACCAATCCAGACGERG10-UTTACTGCCGCTAACGCTTCTCERG10-DGCCTCACCCCAACCTTTGATAATERG13-UTTTTCTTACGGTTCCGGTTTAGCERG13-DTCCTTTGGAGTTTCGGTGATTCHMG1-UGCTGGGTCTGTTGGTGGATTTHMG1-DCGATGGATGGCATGGATACGERG12-UACCGATGACGAGGCTGTAGAAAERG12-DCAGGATGAGAAACACCGATTGAGERG8-UCAAAACAGGGCTGGGCTCCTERG8-DCGCTACATCAAACCCGCTTCCERG19-UCGTGGCAACCTCCGAACTATTERG19-DACAGCATTTGGACCTGCATCAIDI1-UCTACATCGTGCATTCTCCGTCTTIDI1-DCCCCTTGTCTTAGTTTCATCTTCTGERG20-UACTATTCTTTCTACTTGCCTGTCGCERG20-DGTCCTTCTTACCGTAATTTTCGTCT
附件2 法尼烯合成酶多拷贝表达质粒的构建
以质粒PMGKR为模板,PCR扩增loxp-KANMX-loxp,引物为XKX-XbaI-1和XKX-2;以酿酒酵母YPH499基因组DNA为模板,PCR扩增TDH3启动子和TPI1终止子,引物为TTPI1-1,TTPI1-2,PTDH3-1和PTDH3-XbaI-2。以这三段为模板,融合PCR扩增获得loxp-KANMX-loxp-TTPI1-PTDH3,引物为XKX-XbaI-1和PTDH3-XbaI-2。loxp-KANMX-loxp-TTPI1-PTDH3连接至19T simple vector,反向PCR扩增,扩增产物用限制性内切酶SalI和NheI消化获得载体T1,引物分别为XTP-SalI-7和XTP-NheI-8。用限制性内切酶SalI和NheI消化pUC57-Fsso,胶回收获得目的基因Fsso,Fsso与T1连接获得质粒Ts-XTPso。以酿酒酵母YPH499基因组DNA为模板,用引物rDNA-SacⅡ-1和rDNA-SacⅡ-4 PCR扩增获得rDNA片段,连接至19T simple vector,用引物rDNA-XbaI-2和rDNA-XbaI-3反向PCR扩增,扩增产物用限制性内切酶XbaI消化获得载体T2。用限制性内切酶XbaI消化Ts-XTPso,胶回收获得目的片段XTPso,与载体T2连接获得目的质粒Ts-rso。
附件3 缺损质粒的构建
以质粒302和149为模板,引物471-F与GAL1-302R和GAL1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得GAL1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-GAL1。
以质粒302和149为模板,引物471-F与GAL7-302R和GAL7-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得GAL7-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-GAL7。
以质粒302和149为模板,引物471-F与GAL10-302R和GAL10-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得GAL10-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-GAL10。
以质粒302和149为模板,引物471-F与CIT2-302R和CIT2-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得CIT2-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-CIT2。
以质粒302和149为模板,引物471-F与MLS1-302R和MLS1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得MLS1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-MLS1。
以质粒302和149为模板,引物471-F与GPD1-302R和GPD1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得GPD1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-GPD1。
以质粒302和149为模板,引物471-F与GPD2-302R和GPD2-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得GPD2-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-GPD2。
以质粒302和149为模板,引物471-F与ADH3-302R和ADH3-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得ADH3-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-ADH3。
以质粒302和149为模板,引物471-F与ADH4-302R和ADH4-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得ADH4-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-ADH4。
以质粒302和149为模板,引物471-F与ADH5-302R和ADH5-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得ADH5-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-ADH5。
以质粒302和149为模板,引物471-F与ADH6-302R和ADH6-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得ADH6-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-ADH6。
以质粒302和149为模板,引物471-F与BTS1-302R和BTS1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得BTS1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-BTS1。
以质粒302和149为模板,引物471-F与DPP1-302R和DPP1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得DPP1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-DPP1。
以质粒302和149为模板,引物471-F与LPP1-302R和LPP1-149F和471-R分别扩增获得两个片段,引物471-F与471-R融合获得LPP1-471片段。片段和质粒BTS同时用BamH I和BcuI酶切消化,连接获得质粒BTS-LPP1。