基于CRISPR/Cas9系统对乳酸乳球菌进行绿色荧光蛋白标记
宋馨,王慧,袁世豪,黄佳伟,易文涛,艾连中*
(上海理工大学 医疗器械与食品学院,上海食品微生物工程研究中心,上海,200093)
摘 要 乳酸乳球菌是治疗剂在体内运送的良好载体,研究其在体内的真实运送情况需对其进行标记。该实验利用CRISPR/Cas9系统对乳酸乳球菌(Lactococcus lactis)NZ9000进行增强型绿色荧光蛋白(enhanced green fluorescent protein,eGFP)标记,用于研究乳酸乳球菌在体内的运送,评价其作为益生菌的功能。基于该实验室已构建的乳酸乳球菌CRISPR/Cas9编辑质粒pLL25构建重组质粒pYSH,其上携带eGFP及同源臂,电转入乳酸乳球菌NZ9000感受态细胞中,将基因组中的乳酸脱氢酶基因(ldh)替换为绿色荧光蛋白基因,从而使Lactococcus lactis NZ9000获得标记,表达绿色荧光蛋白。对绿色荧光标记的Lactococcus lactis NZ9000突变株,酶标仪定量分析eGFP的表达强度。荧光强度定量分析结果表明,在乳酸乳球菌不同生长阶段,eGFP基因均能稳定表达。
关键词 乳酸乳球菌NZ9000;CRISPR/Cas9系统;绿色荧光蛋白
DOI:10.13995/j.cnki.11-1802/ts.029311
引用格式:宋馨,王慧,袁世豪,等.基于CRISPR/Cas9系统对乳酸乳球菌进行绿色荧光蛋白标记[J].食品与发酵工业,2022,48(2):163-167.SONG Xin,WANG Hui,YUAN Shihao, et al.Green fluorescent protein labeling of Lactococcus lactis based on CRISPR/Cas9 system[J].Food and Fermentation Industries,2022,48(2):163-167.
第一作者:讲师(艾连中教授为通信作者,E-mail:ailianzhong1@126.com)
基金项目:上海市科技兴农重点攻关项目(2019-02-08-00-07-F01152)
收稿日期:2021-09-14,改回日期:2021-10-03
乳酸乳球菌(Lactoccus lactis)是一种公认的益生菌,被应用于发酵乳制品中的历史已达数千年。在食品工业中,乳酸乳球菌作为复合发酵剂的重要组成部分,主要应用于葡萄酒、乳酪及腌制食品中[1-3]。另一方面,乳酸乳球菌作为乳酸菌的代表菌株,其基因组规模相对较小[4],较乳杆菌容易操作,在基因工程及代谢方面也已经作为理想的模型菌株、微生物细胞工厂[5]。由于乳酸乳球菌不含内毒素,现已成为枯草芽孢杆菌及大肠杆菌等更安全的替代菌种[6],并逐渐成为实验室用来异源蛋白表达和各种类型生理研究的主力军[7]。近年来,益生菌被开发用作口服疫苗载体,乳酸乳球菌具有良好的免疫调节特性、公认的安全性、在肠道内极少定植并且是异源蛋白生产的有效细胞工厂[8-9],因此被选作体内运送载体,用以输送治疗剂(如细胞因子)。因此,研究乳酸乳球菌在体内的运送情况具有重要意义。但目前对乳酸菌的运送研究大多通过体外模拟实验和细胞实验,无法真实反映体内情况[10-13]。要研究乳酸乳球菌在体内的运送,需要对其进行标记。增强型绿色荧光蛋白(enhanced green fluorescent protein,eGFP)具有稳定遗传,灵敏度高,对细胞无毒害等特点,并能利用不同启动子调节其表达量,更适合用作乳酸乳球菌体内研究的报告基因[14]。大多数研究通常采用质粒对其进行宿主中的过表达[15-16],但是这种方法稳定性低,且必须引入抗性筛选标记,而将eGFP基因整合到基因组中,可成功避免此缺点。
乳酸乳球菌中已开发多种表达蛋白的工具,但大多应用起来耗时耗力。随着生物技术的不断完善,第三代人工核酸酶(CRISPR-Cas9)被深入研究,人们在不同生物体内陆续构建成功基于CRISPR-Cas的高效基因编辑工具,使研究人员能够对特定的基因或基因组上的特定位点进行精确的靶向修饰,该工具目前已在乳酸菌中得到广泛应用[17]。本实验室已成功构建了适用于乳酸乳球菌,基于CRISPR-Cas9的单质粒基因编辑工具,能够对乳酸乳球菌进行无痕基因编辑[18]。
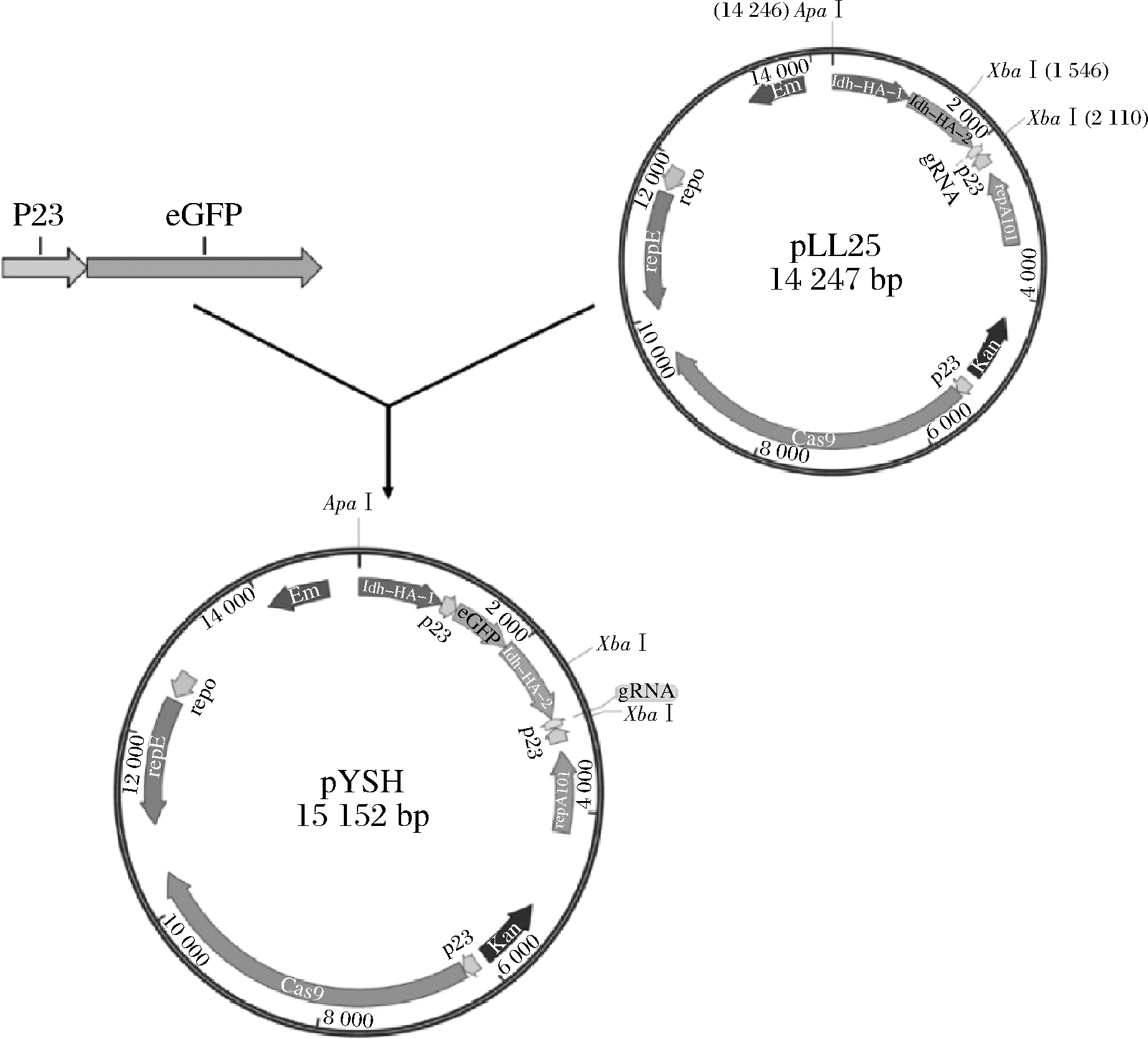
本研究基于实验室已有的乳酸乳球菌基因编辑质粒pLL25,替换目标基因的sgRNA及同源臂,构建重组质粒pYSH,利用CRISPR-Cas9编辑技术,将eGFP基因插入L.lactis NZ9000基因组中,使L.lactis被绿色荧光标记,为L.lactis在体内运送或其他功能的深入研究提供可能。
1 材料与方法
1.1 材料与设备
1.1.1 菌株、质粒及引物
表1和表2列出本研究所用菌株、质粒及引物。Snapgene 4.1.9设计引物,华大基因(上海)股份有限公司合成。
表1 菌株和质粒
Table 1 Strains and plasmids

菌株或质粒特性来源菌株Escherichia coli Top 10商业宿主,用于克隆本实验室L.lactis NZ9000乳酸乳球菌野生型菌株本实验室L.lactis NZ9000△ldh:eGFPeGFP基因替换乳酸乳球菌中ldh基因的突变型菌株本研究 质粒pIB184-eGFP携带eGFP本实验室pLL25敲除L.lactis NZ9000-ldh基因的CRISPR/Cas9编辑质粒本实验室pYSH敲除L.lactis NZ9000-ldh基因并插入eGFP基因的CRISPR/Cas9编辑质粒本研究
表2 引物序列
Table 2 Primer sequences

引物名称序列(5'-3')HA1-FCATCTTTTTCTAAACTAGGGCCCGCGACCGTAACCTTTCAAAC(Apa I)HA1-RGGTTGTCAGGGCTTTTCGTAATCTAGT-TAGGTTTTACAATAGTAAAGGAAAATAAAATP23E-FCCTTTACTATTGTAAAACCTAACTAGATTACGAAAAGCCCTGACAACCP23E-RTCGAAAAGGAGATAAAAATGTTACTTGTACAGCTCGTCCATGHA2-gRNA-FCATGGACGAGCTGTACAAGTAACATTTTTATCTCCTTTTCGATTTTTTATTAATCAATHA2-gRNA-RGAATGACAATGATGTTTCTAGATTAACAATAAATTCAAGAATGTTTTAGAGCTAGAA (Xba I)YZ-FGTTTAGGTTCAGCAACTATTGCAATYZ-RCGGACACGCTGAACTTGTCldh-HA-YZ-FCAGCAACTATTGCAATATTATTAGGCldh-HA-YZ-RGAGACTTTTATCGTTGTTTATG
注:下划线代表酶切位点
1.1.2 菌株及培养条件
大肠杆菌DH5α为克隆宿主,培养基为LB(luria-bertani),所有含有基因编辑质粒的大肠杆菌生长于LB-Kan(卡那霉素质量浓度为50 μg/mL)培养基上,培养温度为30 ℃。GM17培养基用于乳酸乳球菌的培养,配方为(g/L):胰蛋白胨5、大豆蛋白胨5、牛肉膏5、酵母浸出粉2.5、β-磷酸甘油二钠19、MgSO4·7H2O 0.25、葡萄糖5、琼脂粉(Agar)15(液体培养时不添加),必要时,添加红霉素作为筛选标记,工作质量浓度为10 μg/mL。乳酸乳球菌复苏培养基:GM17,CaCl2 2 mmol/L,MgCl2 20 mmol/L。
乳酸乳球菌感受态洗涤缓冲液Ⅰ:体积分数为10%的甘油、0.5 mol/L 蔗糖;洗涤缓冲液Ⅱ:体积分数为10%的甘油、0.5 mol/L蔗糖和0.05 mol/L EDTA;洗涤缓冲液Ⅲ:100 mmol/L 乙酸锂二水、10 mmol/L 二硫苏糖醇(dithiothreitol,DTT)、0.6 mol/L 蔗糖、1 mol/L Tris-HCl(pH 7.5)[19]。
1.1.3 酶及试剂盒
Premix TaqTM酶、DNA限制性内切酶,大连宝生物公司;KOD-plus-neo DNA聚合酶或KOD FX聚合酶,用于PCR扩增及菌落PCR分析,东洋纺生物科技有限公司;无缝克隆试剂盒(ClonExpress Ⅱ One Step Cloning Kit),用于DNA组装及质粒克隆,割胶回收试剂盒(AxyPrepTM DNA Gel Extraction Kit)、质粒小量提取试剂盒(AxyPrepTM Plasmid Miniprep Kit),用于DNA 操作,南京诺唯赞生物科技有限公司。
1.2 实验方法
1.2.1 E.coli Top10感受态细胞制备
-80 ℃冻存的E.coli Top10划线活化,单菌落接种于4 mL LB液体培养基中,37 ℃、200 r/min摇床培养过夜。种子液以1%的接种量转接至50 mL LB液体中(置于250 mL锥形瓶中),37 ℃、200 r/min摇床培养至OD600=0.3~0.5,冰上静置10 min。离心收集菌体,用预冷的0.1 mol/L CaCl2溶液洗涤细胞2次。预冷的含有0.1 mol/L CaCl2的15%甘油溶液重悬细胞。分装,-80 ℃冻藏备用。以上所有离心条件均为4 ℃,4 500 r/min,10 min。
1.2.2 重组质粒pYSH构建
以质粒pLL25为模板,HA1-F、HA1-R、HA2-gRNA-F、HA2-gRNA-R为引物,分别PCR扩增获得上下游同源臂HA-1、HA-2;以pIB184-eGFP为模板,P23E-F、P23E-R为引物,扩增获得插入片段P23-eGFP。PCR反应条件参考说明书。割胶回收上下游同源臂HA-1、HA-2及插入片段P23-eGFP,按摩尔数比为1∶1∶1混合后为模板,以HA1-F/HA2-gRNA-R为引物,overlap 获得片段HA-1+P23+eGFP+HA-2,割胶回收。Apa I和Xba I双酶切质粒pLL25骨架,割胶回收获得线性化载体。将线性化载体骨架与目的片段HA-1+P23+eGFP+HA-2经ClonExpress Ⅱ One Step Cloning Kit无缝克隆连接,构建获得重组质粒pYSH(构建过程如图1所示)。PCR验证引物为TY-F/R。
1.2.3 L.lactis NZ9000感受态制备方法
划线活化L.lactis NZ9000,挑取单菌落,接种于GM17液体培养基中,30 ℃静置培养活化2代。以8%的接种量转接至含有0.5 mol/L蔗糖和8 g/L甘氨酸的GM17液体培养基中,30 ℃静置培养至吸光值OD600≈0.2,离心收集菌体,弃去上清液,溶液Ⅲ重悬细胞,室温静置30 min,离心收集菌体,弃去上清液,溶液Ⅰ、Ⅱ分别洗涤细胞,最后重悬于溶液I中。每100 μL感受态细胞分装于预冷的离心管中,-80 ℃冻藏备用。以上所有离心条件为4 ℃,6 000 r/min,15 min[19]。
1.2.4 L.lactis NZ9000荧光标记
将构建成功的重组质粒pYSH电转至L.lactis NZ9000感受态细胞中,方法如下:取出-80 ℃冰箱冻存的L.lactis NZ9000感受态细胞,冰上解冻;向感受态细胞中加入0.1 μg pYSH质粒并吹打混匀,冰上静置5 min;预冷2 mm电转杯,将混合有质粒的感受态细胞转移至其中,轻轻敲打,使其落于电转杯底部,电击,随后迅速加入900 μL 30 ℃温育的复苏培养基,混匀后转至离心管中,30 ℃ 复苏培养2 h;将复苏液涂布于GM17-Em固体平板,30 ℃静置培养24~36 h,挑取转化子,菌落PCR验证,验证引物为YZ-F/R,L.lactis NZ9000野生型为对照。对PCR产物送测。将测序正确的菌株命名为L.lactis NZ9000Δldh:eGFP。
1.2.5 L.lactis NZ9000Δldh:eGFP荧光强度的测定
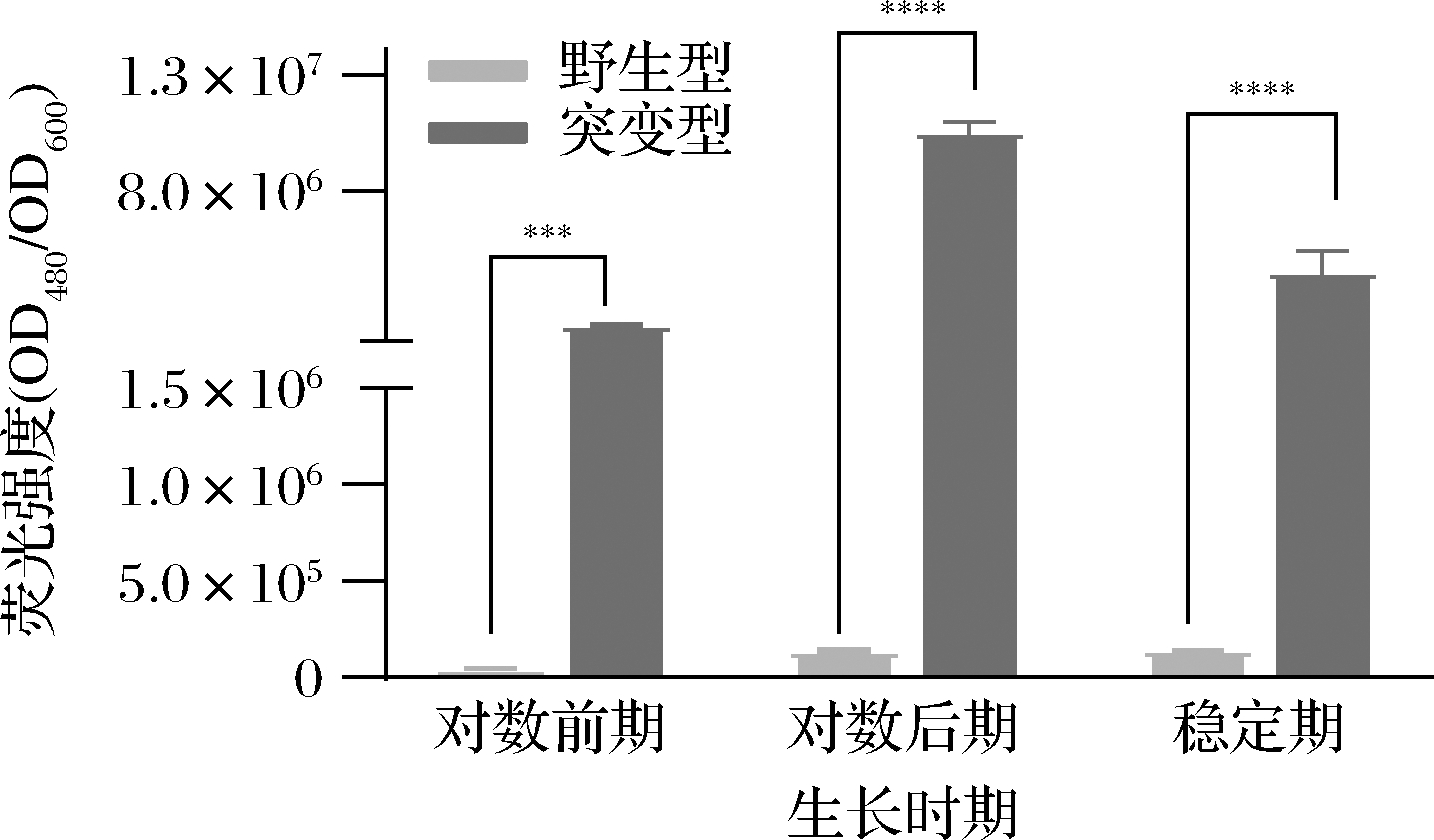
将野生型L.lactis NZ9000和突变型L.lactis NZ9000Δldh:eGFP菌株划线活化,30 ℃静置培养24~36 h,获得单菌落,挑取单菌落分别转接至GM17无抗及含抗的液体培养基。30 ℃静置培养,分别在对数期前期、对数后期、稳定期收集1.5 mL菌液,条件为12 000 r/min、离心1 min;20 mmol/L Tris-HCl(pH 7.4)溶液洗涤菌体2次,重悬,混匀后加入96孔板(每个样品取100 μL)。在激发波长480、发射波长525下,用酶标仪测量其eGFP的表达强度及OD600。每个样品3个平行[20]。使用GraphPad Prism 8.4.0绘图,并对数据进行单因素方差分析(ANOVA),比较不同时期突变型菌株与野生型菌株荧光强度的差异,认为P<0.05有统计学差异(***P<0.001,****P<0.000 1)。
2 结果与分析
2.1 重组质粒pYSH构建
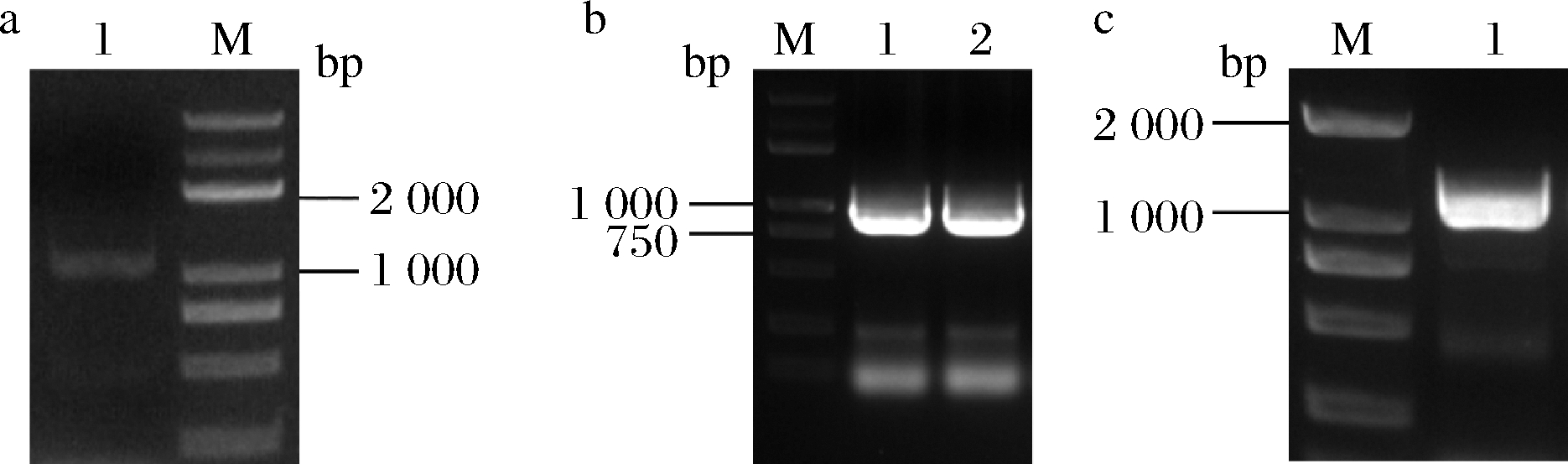
目的片段HA-1、P23+eGFP、HA-2大小分别为1 044、955、1 150 bp,PCR扩增产物进行琼脂糖凝胶电泳分析,结果如图2所示,大小与目的条带一致,说明扩增成功。
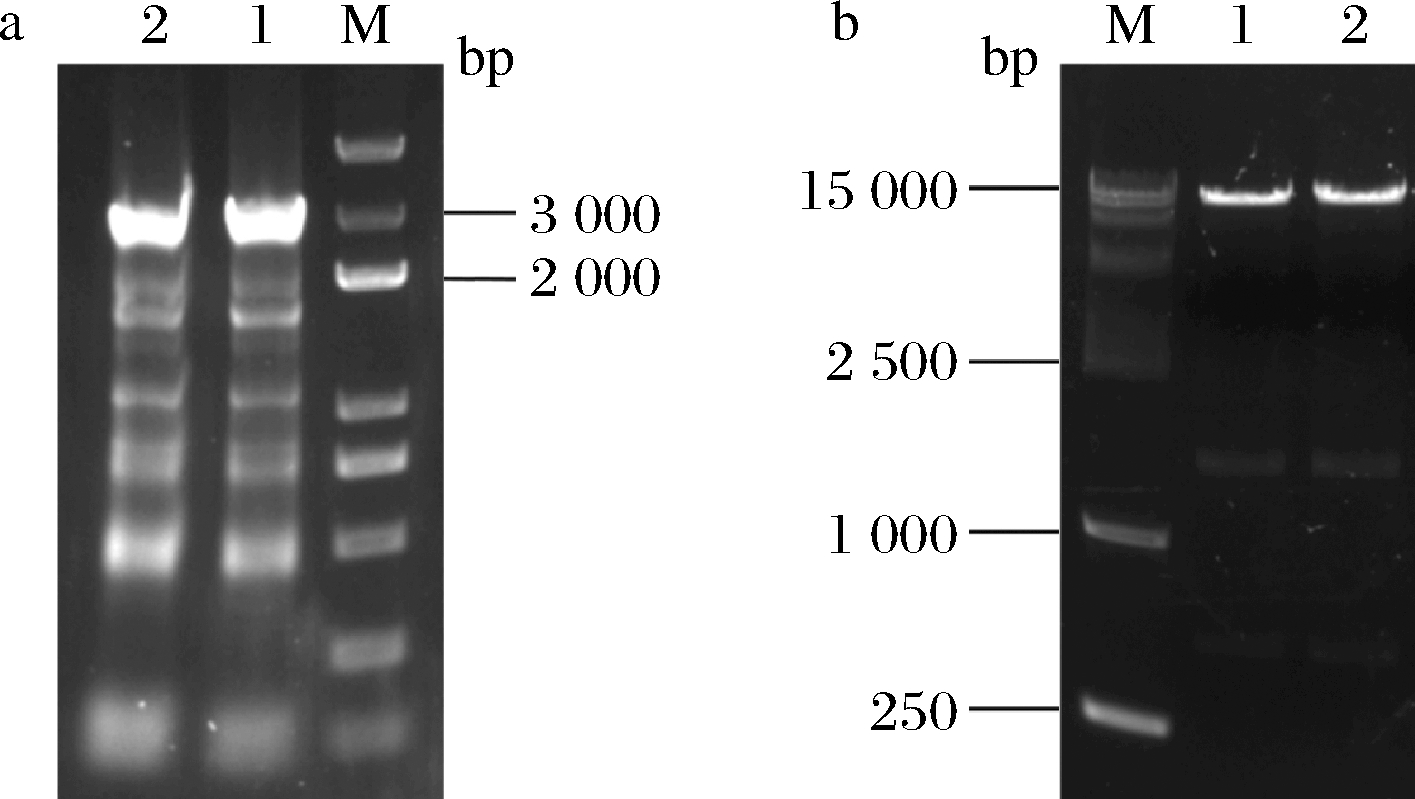
目的片段HA-1+P23+eGFP+HA-2大小为3 016 bp,PCR扩增产物进行琼脂糖凝胶电泳分析,结果如图3所示,大小与目的条带一致,说明overlap扩增成功。
Apa I和Xba I双酶切质粒pLL25,可获得1 547、564、12 136 bp的线性化DNA片段,酶切产物进行1%琼脂糖凝胶电泳,结果见图3,所得条带与理论条带一致,说明酶切正确,可用于后续实验。回收大小为12 136 bp的条带作为载体片段,再与目的片段HA-1+P23+eGFP+HA-2连接,利用E.coli Top 10感受态细胞对连接产物进行转化克隆中,卡那霉素为筛选标记,30 ℃静置培养至长出单菌落,对其进行菌落PCR验证,验证引物为TY-F/R。获得阳性转化子,并对其进行测序鉴定。将测序结果与目的序列进行比较,序列完全一致,说明质粒pYSH构建成功,可用于乳酸乳球菌基因组的编辑。
2.2 L.lactis NZ9000荧光标记
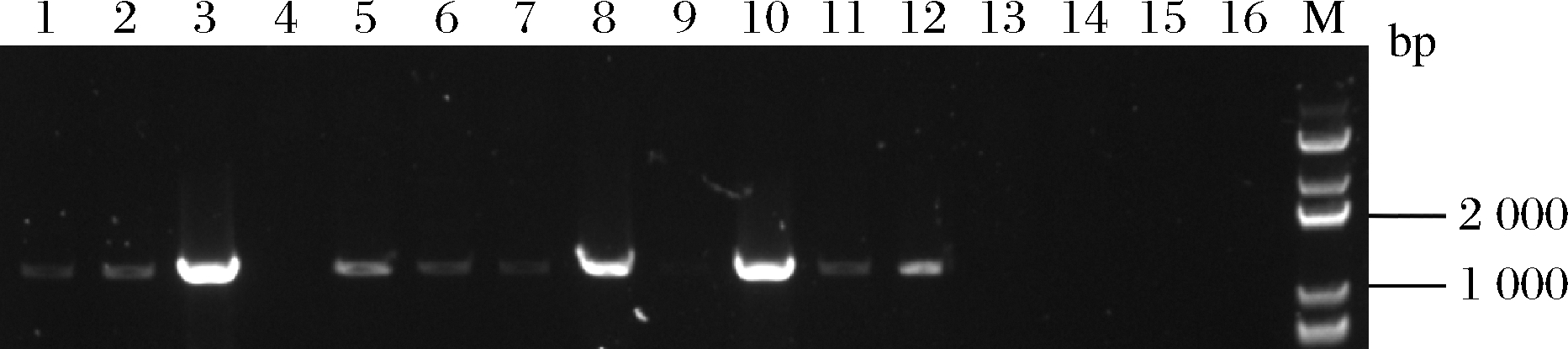
将构建成功的基因编辑质粒pYSH电转入L.lactis NZ9000感受态细胞中,涂布到带有红霉素的筛选平板上,30 ℃培养24~36 h,挑取转化子进行菌落PCR验证,验证结果如图4所示。
将验证有正确条带的阳性转化子挑取到GM17含红霉素液体培养基中,传代培养3代,取菌液为模板,用引物Cldh-HA-YZ-F/R进行PCR验证,PCR产物送测。测序结果显示eGFP成功插入L.lactis NZ9000基因组,获得L.lactis NZ9000Δldh:eGFP突变株。
2.3 L.lactis NZ9000Δldh:eGFP荧光强度的测定
以野生型L.lactis NZ9000为对照,测量不同时期突变菌株L.lactis NZ9000Δldh:eGFP的荧光强度(图5)。结果表明,不同生长时期,突变型菌株的荧光强度与野生型均有极显著性差异。
3 结论与讨论
食品级乳酸菌(lactic acid bacteria,LAB)是开发口服载体的良好候选菌,也是黏膜输送策略中减毒病原体的有吸引力的替代品。其中,乳酸乳球菌不产生内毒素,并且在肠道内极少定植,是一种良好的疫苗及治疗剂等运送载体,具有极大的应用前景。目前,越来越多的研究聚焦于乳酸乳球菌的体内运送情况,但大部分研究都是通过体外模拟或细胞实验进行的,与体内真实运送情况存在一定差异。对乳酸乳球菌进行标记,可以帮助研究其在体内的实际运送情况。在乳酸菌中,近几年常用的标记方法为荧光素类探针标记法,该方法易被样品背景干扰,并且荧光素极易在强光下分解,对pH敏感[21],因此亟需一种更加稳定高效的基因组标记方法。乳酸乳球菌中应用最多的基因插入法是基于质粒的同源重组法,但该方法耗时久、效率低,且重组效率依赖于宿主细胞的电转效率和自身重组酶。本研究利用已构建的CRISPR/Cas9单质粒基因编辑系统将eGFP基因插入L.lactis NZ9000基因组中,利用绿色荧光蛋白标记L.lactis NZ9000,成功获得标记突变株。该突变株性能稳定,无需添加抗性筛选标记,实时定量PCR测定荧光强度,结果表明该突变株中的绿色荧光蛋白在宿主不同生长阶段均能稳定表达荧光,且能随宿主的繁殖稳定遗传,相比传统方法节约前处理时间,省时省力,为进一步深入研究乳酸乳球菌在体内的运送情况提供良好的载体。
参考文献
[1] 陈俊亮, 田芬, 霍贵成, 等.乳酸乳球菌对切达干酪成熟过程中质构和风味的影响[J].食品科学, 2013, 34(21):163-167.
CHEN J L, TIAN F, HUO G C, et al.Effect of Lactococcus lactis on texture and flavor of cheddar cheese during ripening[J].Food Science, 2013, 34(21):163-167.
[2] 赵贵丽, 罗爱平, 黄名正, 等.乳酸乳球菌豆腐生物凝固剂性能评价研究[J].食品工业, 2017, 38(10):146-149.
ZHAO G L, LUO A P, HUANG M Z, et al.Research on the performance evaluation of biological coagulant fermented by Lactococcus lacis[J].The Food Industry, 2017, 38(10):146-149.
[3] 司克辉. 优良乳酸菌菌株在凝固型酸奶中的生产应用[J].农业科技与信息, 2011(10):57-58.
SI K H.Application of good Lactobacillus strains in the production of solidified yoghurt[J].Information of Agricultural Science and Technology, 2011(10):57-58.
[4] YU J, SONG Y Q, REN Y, et al.Genome-level comparisons provide insight into the phylogeny and metabolic diversity of species within the genus Lactococcus[J].BMC Microbiology, 2017, 17(1):213.
[5] SONG A A L, IN L L A, LIM S H E, et al.A review on Lactococcus lactis:From food to factory[J].Microbial Cell Factories, 2017, 16(1):55.
[6] GARCIA-FRUITORS E.Lactic acid bacteria:A promising alternative for recombinant protein production[J].Microbial Cell Factories, 2012, 11(1):157.
[7] LINARES D M, KOK J, POOLMAN B.Genome sequences of Lactococcus lactis MG1363 (revised) and NZ9000 and comparative physiological studies[J].Journal of Bacteriology, 2010, 192(21):5 806-5 812.
[8] MORELLO E,  L G, LLULL D, et al. Lactococcus lactis, an efficient cell factory for recombinant protein production and secretion[J].Journal of Molecular Microbiology & Biotechnology, 2008, 14:48-58.
L G, LLULL D, et al. Lactococcus lactis, an efficient cell factory for recombinant protein production and secretion[J].Journal of Molecular Microbiology & Biotechnology, 2008, 14:48-58.
[9] GU Q, SONG D F, ZHU M Y.Oral vaccination of mice against Helicobacter pylori with recombinant Lactococcus lactis expressing urease subunit B[J].FEMS Immunology & Medical Microbiology, 2009, 56(3):197-203.
[10] MISHRA V, PRASAD D N.Application of in vitro methods for selection of Lactobacillus casei strain as potential probiotics[J].International Journal of Food Microbiology, 2005, 103(1):109-115.
[11] SILVIA B, VALENTINA T, GRETA R, et al.The vaginal isolate Lactobacillus paracasei LPC-S01 (DSM26760) is suitable for oral administration[J].Frontiers in Microbiology, 2015, 6:952.
[12] CHAMPS C D, MARONCLE N, BALESTRINO D, et al.Persistence of colonization of intestinal mucosa by a probiotic strain, Lactobacillus casei subsp. rhamnosus Lcr35, after oral consumption[J].Journal of Clinical Microbiology, 2003, 41(3):1 270-1 273.
[13] 陈佩, 党辉, 贺国旗, 等.一株干酪乳杆菌生物学特性及其发酵豆乳的研究[J].食品研究与开发, 2017, 38(17):17-21.
CHEN P, DANG H, HE G Q, et al.Study on the biological characteristics of Lactobacillus casei and application in the fermented soymilk produce[J].Food Research and Development, 2017, 38(17):17-21.
[14] KONG L H, XIONG Z Q, SONG X, et al.Characterization of a panel of strong constitutive promoters from Streptococcus thermophilus for fine-tuning gene expression[J].Acs Synthetic Biology, 2019, 8(6):1 469-1 472.
[15] MIERAU I, KLEEREBEZEM M.10 years of the nisin-controlled gene expression system (NICE) in Lactococcus lactis[J].Applied Microbiology and Biotechnology, 2005, 68(6):705-717.
[16] ZHU D L, LIU F L, XU H J, et al.Isolation of strong constitutive promoters from Lactococcus lactis subsp.lactis N8[J].FEMS Microbiology Letters, 2015, 362(16):fnv107.
[17] SONG X, ZHANG X Y, XIONG Z Q, et al.CRISPR-Cas-mediated gene editing in lactic acid bacteria[J].Molecular Biology Reports, 2020, 47(10):8 133-8 144.
[18] SONG X, LIU L, LIU X X, et al.Single plasmid systems based on CRISPR-Cas9 for gene editing in Lactococcus lactis[J].Journal of Dairy Science, 2021, 104(10):10 576-10 585.
[19] 韦云莹, 王立峰, 熊智强, 等.响应面法优化乳酸乳球菌电转化效率研究[J].上海理工大学学报, 2018, 40(6):566-571.
WEI Y Y, WANG L F, XIONG Z Q, et al.Optimization of electroporation efficiency of Lactococcus lactis using response surface methodology[J].Journal of University of Shanghai for Science and Technology, 2018, 40(6):566-571.
[20] 刘璐, 艾连中, 夏永军, 等.乳酸乳球菌NZ9000中增强型绿色荧光蛋白报告系统的构建[J].食品与发酵工业, 2020, 46(11):46-51.
LIU L, AI L Z, XIA Y J, et al.A reporter system with the enhanced green fluorescent protein in Lactococcus lactis NZ9000[J].Food and Fermentation Industries, 2020, 46(11):46-51.
[21] 刘璇. 细胞荧光探针的合成与应用[D].长沙:湖南师范大学, 2012.
LIU X.Synthesis and application of cellular fluorescent probes[D].Changsha:Hunan Normal University, 2012.
Green fluorescent protein labeling of Lactococcus lactis based on CRISPR/Cas9 system
SONG Xin,WANG Hui,YUAN Shihao,HUANG Jiawei,YI Wentao,AI Lianzhong*
(Shanghai Engineering Research Center of Food Microbiology, School of Medical Instrument and Food Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China)
ABSTRACT Lactococcus lactis is a good carrier for the delivery of therapeutic agents in vivo. To investigate its actual delivery in vivo, L. lactis needs to be labeled. In this study, L. lactis NZ9000 was labeled with an enhanced green fluorescent protein (eGFP) using the CRISPR/Cas9 system to study its transport in vivo and evaluate its function as a probiotic. Firstly, a recombinant plasmid pYSH carrying eGFP and homologous arms was constructed based on the existing L. lactis CRISPR/Cas9 editing plasmid pLL25 in our laboratory. Secondly, pYSH was electro transferred into L. lactis NZ9000 competent cells, replacing the lactate dehydrogenase (ldh) gene in the genome with the enhanced green fluorescent protein gene, thus enabling L. lactis NZ9000 to express a green fluorescent protein. Finally, we measured the fluorescence intensity of the green fluorescence-labeled L. lactis NZ9000 mutant. The quantitative results of fluorescence intensity showed that eGFP could be stably expressed in different growth stages of L. lactis NZ9000.
Key words Lactococcus lactis NZ9000;CRISPR/Cas9 system;green fluorescent protein