通过敲除聚谷氨酸合成基因提高纳豆杆菌纳豆激酶的生产效率
韩宇星,孟凡强,周立邦,陆兆新*
(南京农业大学 食品科学技术学院,江苏 南京,210095)
摘 要 纳豆激酶是纳豆杆菌(Bacillus natto)产生的具有溶栓作用的酶,纳豆杆菌液体发酵时产生的聚谷氨酸(polygutamic acid,γ-PGA)使发酵液黏度升高,导致纳豆激酶分离纯化困难,成本较高,限制了其规模化生产。该研究采用同源重组双交换技术,敲除纳豆杆菌中合成γ-PGA的关键基因pgsB,降低发酵液中γ-PGA的含量和发酵液黏度,提高后续纳豆激酶的分离纯化效率。结果表明pgsB基因缺失株与野生型菌株相比,发酵液中γ-PGA产量显著下降,在发酵24 h时γ-PGA含量下降了57.9%。此外两者的细胞密度无明显差异,说明pgsB基因的缺失可显著降低发酵液黏度且对菌体的生长没有影响。使用超滤法纯化纳豆激酶的过程中,基因缺失株的纳豆激酶回收率比野生型提高了19.2%。因此,敲除pgsB基因后,纳豆激酶分离纯化的效率得到了提高,为纳豆激酶的工业化生产提供了新方法。
关键词 基因敲除;纳豆激酶;pgsB基因;聚谷氨酸
DOI:10.13995/j.cnki.11-1802/ts.027580
引用格式:韩宇星,孟凡强,周立邦,等.通过敲除聚谷氨酸合成基因提高纳豆杆菌纳豆激酶的生产效率[J].食品与发酵工业,2022,48(2):224-230.HAN Yuxing,MENG Fanqiang,ZHOU Libang, et al.Improve the production efficiency of Bacillus natto nattokinase by knocking out the γ-PGA synthetic gene[J].Food and Fermentation Industries,2022,48(2):224-230.
第一作者:硕士研究生(陆兆新教授为通信作者,E-mail:fmb@njau.edu.cn)
收稿日期:2021-04-07,改回日期:2021-05-17
血浆中不溶性纤维蛋白和血小板凝结形成的血栓导致多种心血管疾病[1],严重威胁着人们的健康,逐渐成为致死率第一的疾病[2]。目前市场上主流的血栓治疗药物有纤维蛋白溶酶原激活剂(tissue-type plasminogenactivator,t-PA)、链激酶(streptokinase,SK)和尿激酶(urokinase,UK)等。但是上述药物存在半衰期短,价格昂贵等缺点。纳豆激酶是从纳豆中提取的碱性丝氨酸蛋白酶[3],它具有良好的溶血栓效果。与市场上的溶栓药物相比,它具有半衰期较长、成本低廉、特异性强、无副作用、可口服、安全性高等优势,是一种优良的溶栓药物替代物。但是,纳豆杆菌液体发酵时产生的聚谷氨酸(polygutamic acid,γ-PGA)升高了发酵液黏度,导致纳豆激酶的分离纯化困难。γ-PGA是由谷氨酸通过肽键聚合而成,分子质量在10~1 000 kDa左右[4],黏度很高,且带有离子状态的羧基基团,使得菌体细胞带有负电荷,使发酵液达到一种稳定状态。发酵液需要稀释多倍才能通过离心或者絮凝的方法去除菌体,因此降低γ-PGA 产量和发酵液黏度是提高纳豆激酶生产效率的关键[5]。
γ-PGA合成酶分为两类,分别是cap和pgs[6]。如炭疽杆菌的cap基因合成的γ-PGA结合在细胞壁上,作为荚膜的主要成分。而芽孢杆菌中pgs合成的γ-PGA游离在微生物周围的环境中。pgs基因簇包含3个基因:pgsB、pgsC、pgsA。ASHIUCHI等[7]将含有pgsB、pgsC、pgsA、pgsAB、pgsAC、pgsBC、pgsABC的质粒分别转化至大肠杆菌,发现只有完整pgsABC基因才能合成出γ-PGA。随后该研究室构建了pgsBCA基因缺失的枯草芽孢杆菌ISW1214(突变体MA41)[8],结果表明该枯草芽孢杆菌不能产生γ-PGA[8],因此pgsB、pgsC和pgs A是γ-PGA合成必要的基因[9]。为提高γ-PGA的产量,研究人员将pgsBCA基因导入至其他的宿主细胞表达γ-PGA,如石峰等[10]、马婕等[11]将枯草芽孢杆菌中的pgsBCA基因转化到大肠杆菌中,重组的大肠杆菌也成功的合成出了γ-PGA。而在pgsBCA 3个基因中,pgsB基因被认为是构成γ-PGA 合成的最主要催化部分[12]。研究人员研究了对pgsB的蛋白序列,并将其蛋白序列与ATP 酶对比,发现二者的相似度极高,说明pgsB可能具有ATP酶的活性并且可以结合ATP[13]。而对pgsB单独催化时发现,仅pgsB也可以聚合产生γ-PGA,不过前提是,pgsB的2种状态需要同时存在[14]。
基于以上的研究,为了提高纳豆激酶的分离纯化效率,本文拟利用双交换重组的方法敲除纳豆杆菌中γ-PGA合成的关键基因pgsB,旨在减少发酵液的黏度,提高纳豆激酶的分离纯化效率,以期得到一种高效、简便的纳豆激酶工业化提纯方法。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
Bacillus natto 400为本实验室筛选的纳豆激酶高产菌,来源于野生菌株B.natto;大肠杆菌JM109(Escherichia coli JM109),北京百斯凯公司;质粒pCBS(温度敏感型复制子、大肠杆菌中氨苄青霉素抗性、芽孢杆菌中红霉素抗性),为本实验室保存;引物,南京金斯瑞生物技术有限公司,引物序列如表1所示。
表1 敲除和鉴定pgsB基因的引物
Table 1 Primers for deletion and identification of pgsB

引物名称 引物序列(5'-3') pgsB-Up-FATGTGGTTACTCATTATAGCCTGTGpgsB-Up-RCTGCAATTTCATCAAGCGTCGpgsB-Dn-FCGCTTGATGAAATTGCAGGAGCAATGAGAATTCTTCCGpgsB-Dn-RCTAGCTTACGAGCTGCTTAACCCBS-BB-RgctataatgagtaaccacatGCATGCATCGATAGATCTGTCCBS-BB-FttaagcagctcgtaagctagTCGACGTCATATGGATCCGATcheck-FCTAGCTAATGTTACGTTACACcheck-RGAAGCGAGAAGAATCATAATG
注:表中小写字母为一步克隆所用的同源序列
1.1.2 培养基与试剂
Taq DNA聚合酶、高保真phanta酶、一步克隆试剂盒、DNA胶回收试剂盒、质粒提取试剂盒,南京诺唯赞公司;氨苄青霉素、红霉素,Sigma公司。
LB培养基(g/L):蛋白胨10、酵母粉5、氯化钠5;一级种子培养基(g/L):葡萄糖10、蛋白胨10、酵母膏5、氯化钠10;二级种子培养基(g/L):葡萄糖20、蛋白胨10、酵母膏5、氯化钠10,pH 7.2。发酵培养基(g/L):葡萄糖10、大豆粉40、磷酸氢二钾1、硫酸镁0.5、磷酸二氢钠4、氯化钙0.2、硫酸锰0.05、消泡剂1;发酵罐补料(g/L):蔗糖500、葡萄糖50;硼砂缓冲液:50 mmol/L硼砂、150 mmol/L氯化钠,pH 8.5;纤维蛋白原溶液:72 mg纤维蛋白原溶于硼砂缓冲液;凝血酶:溶于硼砂缓冲液,浓度1 000 U/mL,使用时用硼砂缓冲液稀释到20 U/mL,-20 ℃保存。磷酸盐缓冲液(g/L):磷酸氢二钠7.2,磷酸二氢钠2.8,pH 7.2。
1.2 实验方法
1.2.1 PCR扩增pgsB上游片段和下游片段
以B.natto 400 DNA为模板,以pgsB-Up-F和pgsB-Up-R为引物,PCR扩增pgsB基因的上游片段(463 bp)。以pgsB-Dn-F和pgsB-Dn-R为引物,PCR扩增获pgsB基因的下游片段(473 bp)。PCR的扩增条件为:预变性95 ℃、5 min;变性95 ℃、20 s,退火55 ℃、20 s,延伸72 ℃、30 s,共35个循环,最后72 ℃延伸2 min。胶回收PCR产物,得到分别带有上下游同源臂的pgsB-up和pgsB-dn。
1.2.2 touchdown PCR 构建pgsB缺失突变基因
采用touchdown PCR的方法将pgsB基因上下游同源臂连接到一起,得到pgsB的缺失突变基因ΔpgsB。以纯化后的pgsB-up和pgsB-dn作为模板,以pgsB-Up-F和pgsB-dn-R为引物,进行touchdown PCR。扩增条件为:预变性95 ℃、2 min,变性95 ℃、15 s,退火60 ℃、15 s,延伸72 ℃、1 min;共5个循环,每次循环退火温度降低1 ℃;随后变性95 ℃、15 s,退火55 ℃、15 s,延伸72 ℃、1 min,共30个循环,最后72 ℃延伸2 min。胶回收PCR产物,得到带有上下游同源臂的pgsB碱基缺失的基因片段ΔpgsB。
1.2.3 构建同源重组双交换的载体
使用诺唯赞质粒提取试剂盒提取pCBS质粒,并以其为模板,以CBS-BB-R和CBS-BB-F为引物反向PCR扩增获得载体(8 098 bp),扩增条件为:预变性95 ℃、1 min,变性95 ℃、20 s,退火55 ℃、20 s,延伸72 ℃、4.5 min,共35个循环;最后72 ℃延伸8 min。胶回收PCR产物,得到pCBS载体。使用诺唯赞一步克隆试剂盒,将ΔpgsB与pCBS载体连接,并转化至E.coli JM109感受态细胞。在含有100 μg/mL氨苄青霉素的LB平板筛选阳性转化子,PCR验证阳性转化子,测序验证后获得用于pgsB敲除的重组质粒pCBS-ΔpgsB。
1.2.4 纳豆杆菌转化
参照李瑞芳等[15]制备枯草芽孢杆菌超级感受态的方法制备纳豆杆菌感受态细胞,参照陆雁等[16]质粒转化的方法,将15 μL质粒pCBS-ΔpgsB加入到500 μL纳豆杆菌感受态中,轻轻摇匀,30 ℃,100 r/min培养30 min,之后再加入600 μL含有5 μg/mL红霉素的LB培养基,30 ℃,200 r/min继续培养1.5 h,菌液10 000×g离心30 s,用100 μL上清液重悬菌体,涂布于含10 μg/mL红霉素的LB固体平板,30 ℃静置培养12 h。以check-F/R为引物,PCR验证质粒是否转化进入纳豆杆菌。
1.2.5 蓝白斑筛选
将测序验证后的阳性转化子在42 ℃下转接LB培养基培养2次,在含10 μg/mL红霉素的固体LB平板上涂布40 μL质量浓度为20 mg/mL的5-溴-4-氯-3-吲哚-β-D-半乳糖苷(5-Bromo-4-chloro-3-indolyl-β-D-galactopyranoside,X-Gal)溶液,再涂布100 μL的菌液。在42 ℃下培养12 h,4 ℃冷藏8 h后,挑取蓝色菌落(单个同源臂重组)。在30 ℃培养12 h后涂布至含X-Gal的LB固体平板上,30 ℃培养12 h,4 ℃冷藏8 h后,挑取白色菌落(2个同源臂重组),进行PCR验证并测序。
1.2.6 pgsB基因敲除菌株发酵生产纳豆激酶
将测序验证后的B.natto 400 ΔpgsB株在LB平板上划线,37 ℃下培养12 h。将单菌落接种于一级种子培养基中,37 ℃、180 r/min摇床中培养12 h。以5%接种量接种于二级种子液中,培养4 h(对数生长中期)。将50 L通气搅拌式发酵罐灭菌,配制20 L的发酵培养基,采用火焰接种法接种二级种子液(5%接种量)。调节发酵参数:温度37 ℃、通风比为1∶1、搅拌转速400 r/min、溶解氧(dissolved oxygen,DO)>20%。通过氨水和10 %磷酸控制发酵液pH 7.2,通过连续补料(补料培养基)的方法控制培养基糖含量在0.5%~1.0%(质量分数)。在该条件下培养24 h之后放罐。
1.2.7 纳豆激酶酶活力的测定
在1.4 mL硼砂缓冲液中加入0.4 mL纤维蛋白原溶液,37 ℃孵育5 min。再加入0.1 mL凝血酶,颠倒混匀,37 ℃水浴10 min,使纤维蛋白凝固(模拟血栓)。随后加入0.1 mL离心后的发酵上清液,颠倒混匀,37 ℃水浴60 min,每20 min颠倒混匀几次。最后加入0.2 mol/L三氯乙酸,颠倒混匀,终止反应并静置20 min。12 000×g离心10 min,上清液通过紫外分光光度计测定A275。对照样品先加入三氯乙酸,再加入待测菌液,酶活力计算如公式(1)所示:
酶活力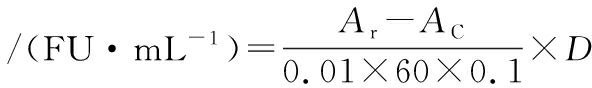
(1)
式中:Ar为待测样品的吸光度,AC为对照样品的吸光度,D为稀释倍数。
1.2.8 γ-PGA含量的测定
发酵液10 000×g离心20 min,取上清液,加入等体积的无水乙醇,10 000×g离心10 min,弃上清液,烘干后称量沉淀的重量,该重量即为γ-PGA产量。
1.2.9 发酵液黏度的测定
采用NDJ-9SB型旋转黏度计对发酵液黏度进行测定。
2 结果与分析
2.1 ΔpgsB片段的构建
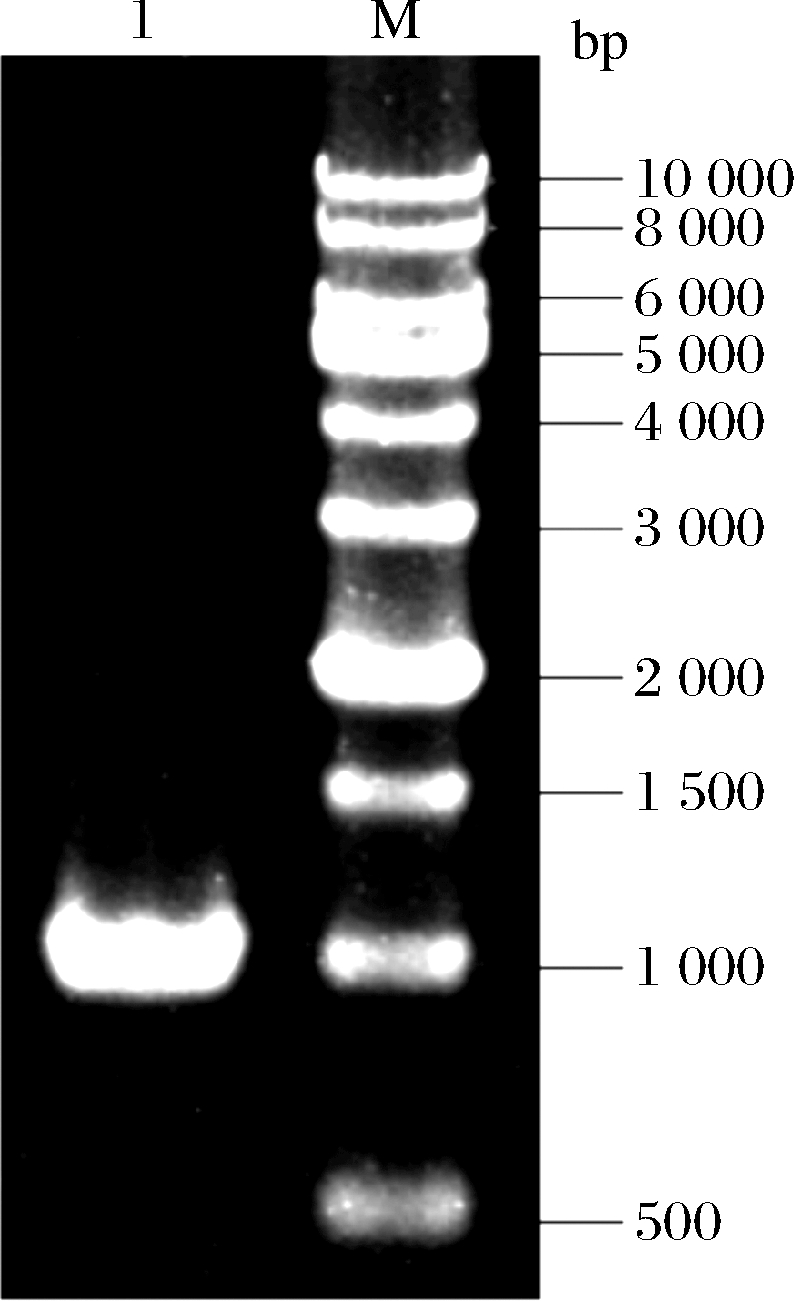
以B.natto 400的DNA为模板,以pgsB-Up-F、pgsB-Up-R、pgsB-Dn-F、pgsB-Dn-R为引物扩增pgsB上下游同源臂,凝胶电泳检测结果如图1所示,在500 bp左右处出现特异性条带,与理论长度463、473 bp相符。以纯化后的上下游同源臂为模板,以pgsB-Up-F、pgsB-Dn-R为引物,采用touchdown PCR的方法构建缺失突变的pgsB片段(ΔpgsB),结果如图2所示,在1 000 bp左右处出现特异性条带,与理论长度918 bp相符,表明ΔpgsB片段构建成功。
2.2 重组质粒的构建与鉴定
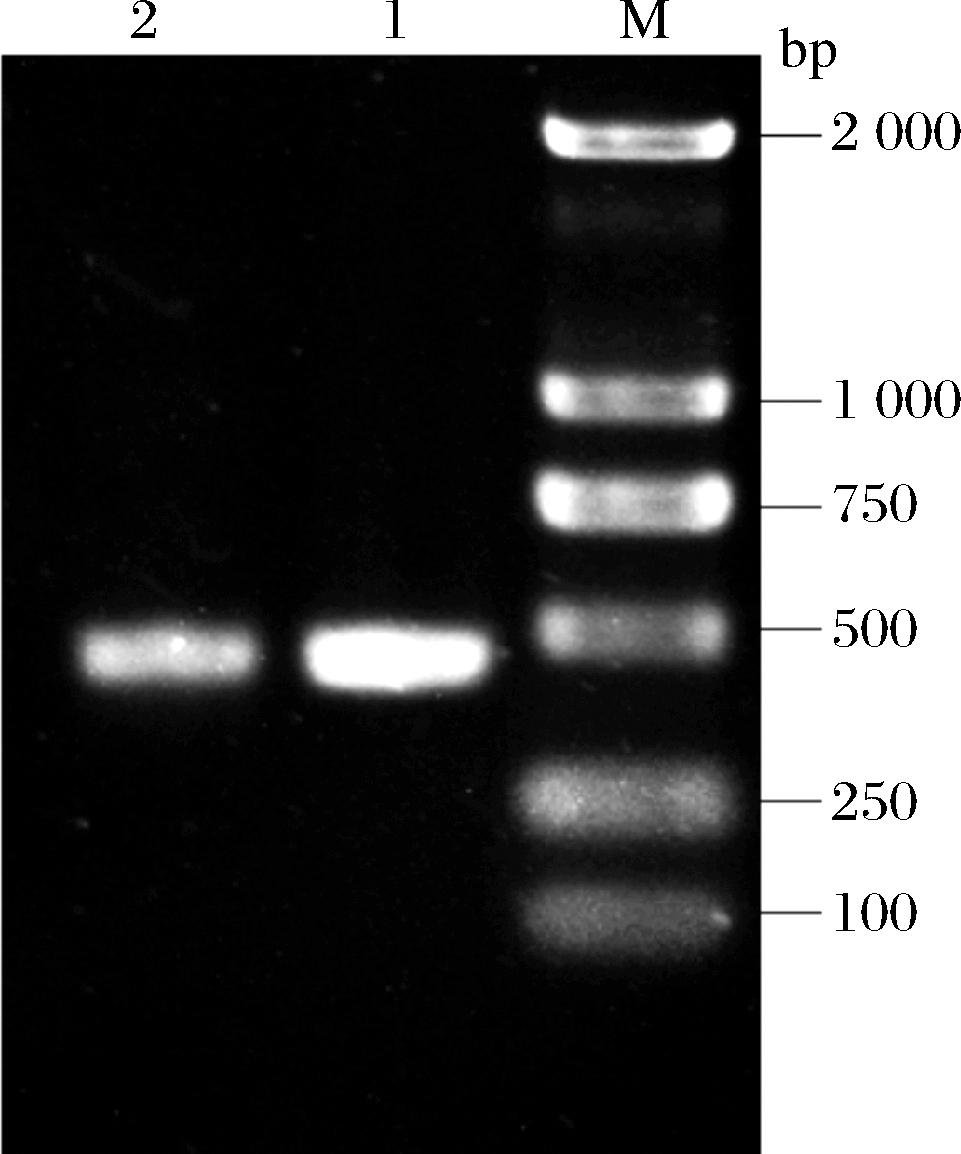
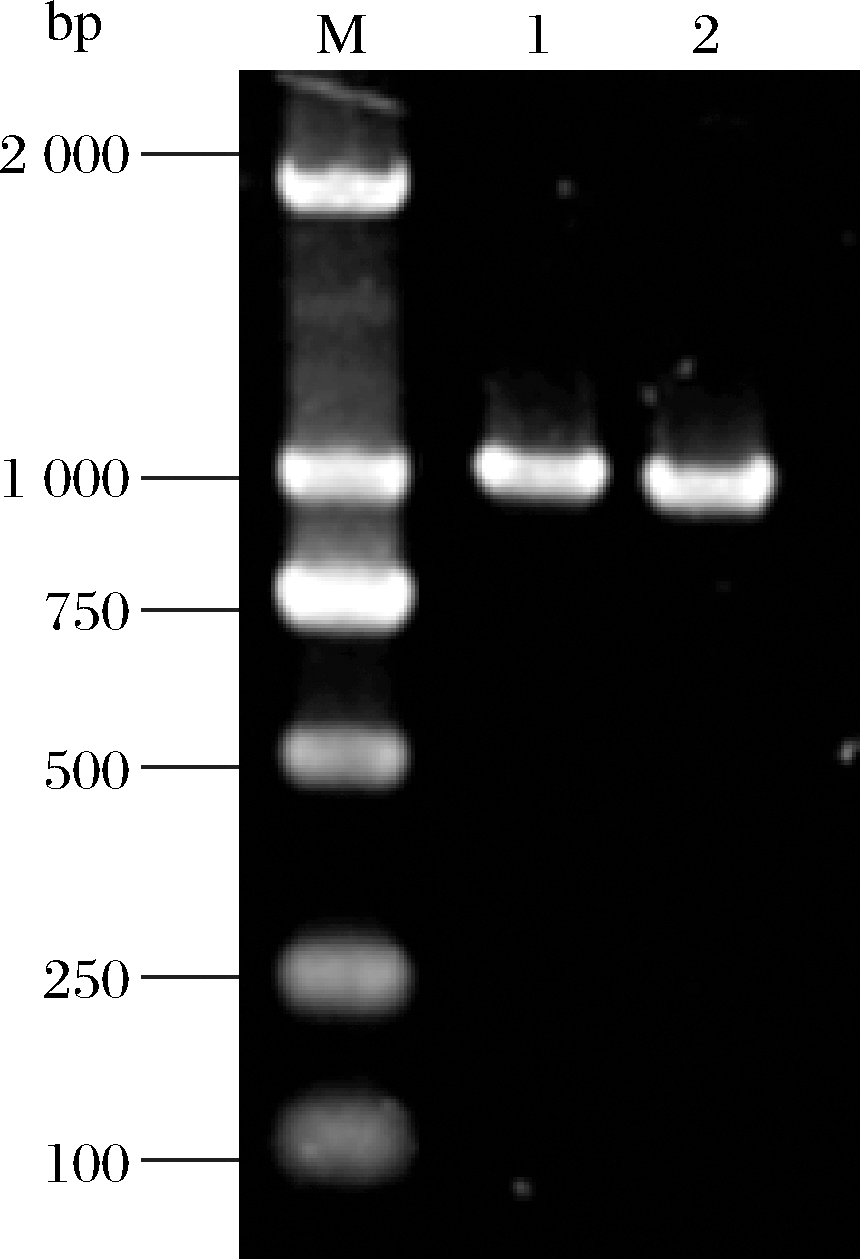
ΔpgsB基因和载体pCBS通过一步克隆试剂盒连接到一起,随后转化至JM109感受态细胞中,涂布至含氨苄青霉素的LB固体培养基上,用引物check-F/R对氨苄青霉素平板上生长的菌落进行菌液PCR筛选,结果如图3所示。
2.3 同源重组双交换
按照诺唯赞质粒提取试剂盒提取重组质粒pCBS-ΔpgsB。并将pCBS-ΔpgsB转入纳豆杆菌感受态细胞内,在含红霉素的LB固体培养基上筛选,PCR验证条带。接着按照1.2.5进行蓝白斑筛选,将阳性转化子在42 ℃条件下转接2次,在含红霉素的LB平板筛选蓝色菌,在30 ℃培养后,挑取白色菌落,用引物check-F/R进行PCR验证,结果如图4所示。在1 000 bp左右处出现特异性条带,说明ΔpgsB基因已经成功整合到纳豆杆菌基因组上,pgsB基因敲除完成。
2.4 pgsB基因的敲除对γ-PGA产量的影响
2.4.1 在锥形瓶中pgsB基因的敲除对γ-PGA产量及纳豆激酶活力的影响
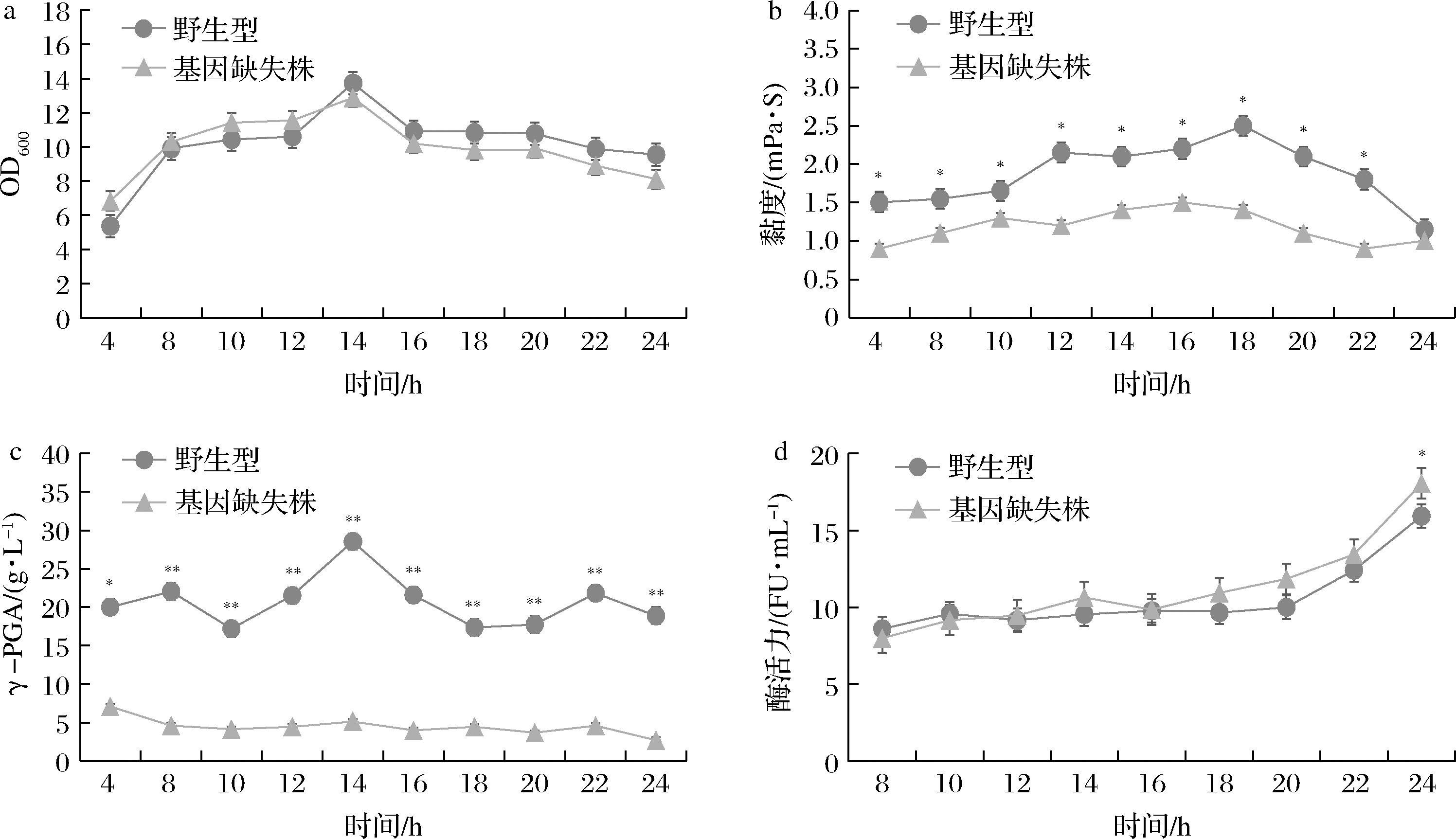
将ΔpgsB株与野生型菌株活化后在摇瓶中发酵,每隔2 h取出1瓶测定其黏度、OD600、γ-PGA含量以及发酵液酶活力。结果表明野生型菌株与ΔpgsB株均在培养10 h左右进入稳定期(图5-a)。野生型菌株发酵液的黏度在18 h前呈上升趋势,在18 h左右达到顶峰,随后快速下降。而ΔpgsB株发酵液的黏度在16 h前呈上升趋势,在16 h左右达到顶峰,之后缓慢下降。在培养22 h之前,两者的发酵液在黏度上存在显著性差异(图5-b)。野生型菌株发酵液中γ-PGA的含量均显著的高于ΔpgsB株,在14 h左右野生型菌株γ-PGA的含量达到最大值,为28.536 g/L。而pgsB基因敲除后,γ-PGA含量显著下降。在14 h 左右γ-PGA含量仅为5.157 g/L,是野生型的18.07%(图5-c)。两者的发酵液中纳豆激酶活力在24 h之前没有显著的差异(图5-d)。该结果说明,pgsB基因的敲除对菌体生长和纳豆激酶的产生并没有显著影响,但是γ-PGA含量以及发酵液黏度显著降低。
2.4.2 在50 L发酵罐中pgsB基因的敲除对γ-PGA产量及纳豆激酶活力的影响
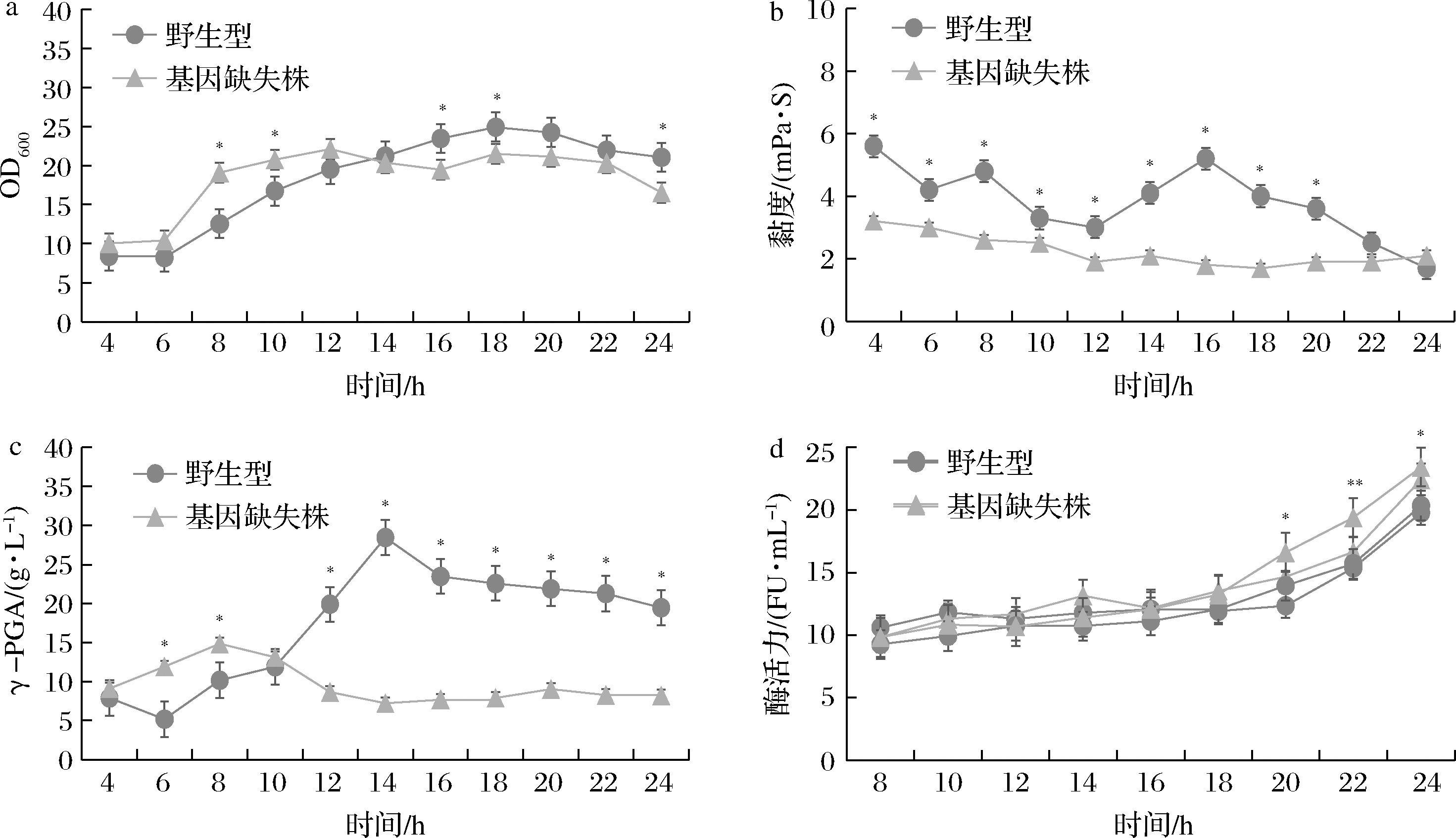
在50 L发酵罐中,野生型菌株在培养18 h时菌体密度达到最高(OD600=24),随后缓慢下降。而ΔpgsB株在10 h达到稳定期(OD600=20),生长速度显著高于野生菌(图6-a)。而从黏度曲线来看,ΔpgsB株的发酵液黏度显著低于野生型菌株发酵液的黏度(图6-b)。发酵罐中γ-PGA含量的变化趋势与其在摇瓶中趋势大致相同,野生型菌株的γ-PGA产量在14 h前不断上升,在14 h达到最大值28.46 g/L,随后缓慢下降,而ΔpgsB株的γ-PGA产量较低,在8 h达到最高值15 g/L。发酵24 h后,ΔpgsB株γ-PGA的产量为8.19 g/L,仅为野生型菌株的42.1%(图6-c)。根据1.2.7的方法,测试ΔpgsB株及野生型纳豆杆菌发酵24 h的酶活力,ΔpgsB株的酶活力为23.4 FU/mL,野生型菌株的酶活力为20.3 FU/mL,酶活力提高了15.2 %(图6-d),综上所述,pgsB基因敲除后,纳豆杆菌发酵液黏度降低,纳豆激酶酶活力提高。
2.5 pgsB基因的敲除对纳豆激酶分离纯化的影响
发酵液8 000×g离心20 min后获得纳豆激酶粗酶液,用30 kDa与10 kDa的超滤管对粗酶液超滤,超滤时间为30 min。由于野生型发酵液经过离心后黏度仍然大于ΔpgsB株的发酵液,故超滤效率较低,野生型发酵液的超滤通量为0.1 mL/min,而ΔpgsB株的超滤通量为0.12 mL/min,较野生型提高了20%。此外,野生型经超滤后,酶活力回收率为48.75%,而ΔpgsB株的酶活力回收率为58.97%,比野生株提高了10.22%。所以,pgsB基因的敲除提高了纳豆激酶分离纯化的效率。
3 讨论
纳豆激酶的分离纯化方法包括超滤法[17]、双水相分离[18]、柱层析柱[19]、反相胶束萃取法[20]、磁性微球吸附法[21]、集成化分离技术[22]等,但尚未有从分子层面减少发酵液黏度以提高纳豆激酶分离效率的报道。本文利用双交换基因重组技术,敲除了纳豆杆菌中γ-PGA的合成基因pgsB。在50 L发酵罐发酵中,相对于野生型纳豆芽孢杆菌,ΔpgsB株产生的γ-PGA显著减少,在发酵24 h时,野生型菌株γ-PGA 的产量为19.46 g/L,而ΔpgsB株的产量仅为8.19 g/L,下降了57.9%。相应的ΔpgsB株发酵液的整体黏度也低于野生型菌株,而纳豆激酶酶活力相对于野生型菌株提高了3 FU/mL。原因可能是敲除pgsB基因后,发酵液黏度降低,改善了溶氧条件和营养物质传递。此外发酵液黏度下降后,纳豆激酶的分离纯化也更加的便捷,降低了生产成本,提高了纳豆激酶的分离纯化效率。
纳豆杆菌pgsB基因被敲除后,γ-PGA产量下降,但仍然有γ-PGA合成。研究表明,控制枯草芽孢杆菌合成γ-PGA的基因主要是pgsA、pgsB及pgsC。虽然pgsB的基因被敲除了,但pgsA和pgsC也可能合成γ-PGA。因此后续实验中将敲除pgsA和pgsC,进一步降低γ-PGA的产量和发酵液的黏度。此外,除了pgsB、pgsA及pgsC,γ-PGA的产生也受多种基因的调控,研究表明在枯草芽孢杆菌中,高浓度磷酸化的DegU对pgsB、pgsC、pgsA的转录有促进作用[23]。除了DegU外,高浓度的DegQ也可以调节pgsB的转录[24]。所以,要减少γ-PGA的产生,提高分离纯化效果,还需要进一步的研究。
参考文献
[1] 孙宇婷, 高春艳.微粒在血栓性疾病中的研究进展[J].医学综述, 2021, 27(2):275-279.
SUN Y T, GAO C Y.Research progress of microparticles in thrombotic diseases[J].Medical Recapitulate, 2021, 27(2):275-279.
[2] 倪庆仁, 戴进前, 张迪, 等.血栓预防策略及研究进展[J].实用心脑肺血管病杂志, 2021, 29(1):6-9.
NI Q R, DAI J Q, ZHANG D, et al.Strategy and research progress of thrombosis prevention[J].Practical Journal of Cardiac Cerebral Pneumal and Vascular Disease, 2021, 29(1):6-9.
[3] WENG Y Q, YAO J, SPARKS S, et al.Nattokinase:An oral antithrombotic agent for the prevention of cardiovascular disease[J].International Journal of Molecular Sciences, 2017, 18(3):523.
[4] 彭英云.γ-聚谷氨酸生产,合成机制和抗冷冻性的研究[D].无锡:江南大学, 2015.
PENG Y Y.Bioproduction, synthesis mechanism and cryoprotective studies of poly-γ-glutamic acid[D].Wuxi:Jiangnan University, 2015.
[5] BAJAJ I, SINGHAL R.Poly (glutamic acid)-an emerging biopolymer of commercial interest[J].Bioresource Technology, 2011, 102(10):5 551-5 561.
[6] ZHONG Z.Study progress on poly-γ-glutamate synthetase and synthesis mechanism[J].Biotechnology Bulletin, 2010, 23(2-6):101-106.
[7] ASHIUCHI M, SHIMANOUCHI K,HORIUCHI T, et al.Genetically engineered poly-gamma-glutamate producer from Bacillus subtilis ISW1214[J].Journal of the Agricultural Chemical Society of Japan, 2006, 70(7):1 794-1 797.
[8] ASHIUCHI M, NAWA C, KAMEI T, et al.Physiological and biochemical characteristics of poly gamma-glutamate synthetase complex of Bacillus subtilis[J].European Journal of Biochemistry, 2001, 268(20):5 321-5 328.
[9] 郑重, 吴剑光, 邱乐泉, 等.微生物聚谷氨酸(γ-PGA)合成酶及合成机理的研究进展 [J].生物技术通报, 2010(6):52-56.
ZHENG Z, WU J G, QIU L Q, et al.Research progress of microbial polyglutamic(γ-PGA) acid synthase and its synthesis mechanism [J].Biotechnology Bulletin, 2010(6):52-56.
[10] 石峰, 徐志南, 岑沛霖.利用枯草芽孢杆菌制备γ-聚谷氨酸[C].中国资源生物技术与糖工程学术研讨会论文集,山东:山东微生物学会, 2005.
SHI F, XU Z N, CEN P L.Preparation of γ - polyglutamic acid by Bacillus subtilis[C].Proceedings of the Symposium on Resource Biotechnology and Sugar Engineering in China,Shandong:Shandong society of Microbiology, 2005.
[11] 马婕, 王丹, 李强, 等.基因工程大肠杆菌合成γ-聚谷氨酸[J].过程工程学报, 2009, 9(4):791-795.
MA J, WANG D, LI Q, et al.Biosynthesis of poly-γ-glutamate acid by Escherichia coli[J].The Chinese Journal of Process Engineering, 2009, 9(4):791-795.
[12] VETTING M W, DE CARVALHO L P S, YU M, et al.Structure and functions of the GNAT superfamily of acetyltransferases[J].Archives of Biochemistry & Biophysics, 2005, 433(1):212-226.
[13] EVELAND S S, POMPLIANO D L, ANDERSON M S, et al.Conditionally lethal Escherichia coli murein mutants contain point defects that map to regions conserved among murein and folyl poly-gamma-glutamate ligases:Identification of a ligase superfamily[J].Biochemistry, 1997, 36(20):6 223-6 229.
[14] URUSHIBATA Y, TOKUYAMA S, TAHARA Y, et al.Characterization of the Bacillus subtilis ywsC Gene, involved in γ-polyglumatic acid prodution[J].Journal of Bacteriology, 2002, 184(2):337-343.
[15] 李瑞芳, 薛雯雯, 黄亮, 等.枯草芽孢杆菌感受态细胞的制备及质粒转化方法研究[J].生物技术通报,2011(5):227-230.
LI R F, XUE W W, HUANG L, et al.Competent preparation and plasmid transformation of of Bacillus subtilis[J].Biotechnology Bulletin, 2011(5):227-230.
[16] 陆雁, 王青艳, 朱绮霞, 等.枯草芽孢杆菌高效转化及其转化子验证方法[J].广西科学院学报, 2012, 28(2):117-119.
LU Y, WANG Q Y, ZHU Q X, et al.Method for enhancing the transformation efficiency in Bacillus subtilis[J].Journal of Guangxi Academy of Sciences, 2012, 28(2):117-119.
[17] 陈景鑫, 刘妍妍, 沙维, 等.超滤法提取纳豆激酶的技术参数优化[J].食品科技, 2010, 35(5):225-229.
CHEN J X, LIU Y Y, SHA W, et al.Study on technology for extracting nattokinase by ultrafiltration[J].Food Science and Technology, 2010, 35(5):225-229.
[18] 宋文超. 高产纳豆激酶菌株筛选和纳豆激酶分离纯化及药效学研究[D].武汉:华中农业大学, 2013.
SONG W C.Screening strains producing high nattokinase activity, separation and purification of nattokinase fibrinolytic effect on thrombotic mice[D].Wuhan:Huazhong Agricultural University, 2013.
[19] 刘柳, 李南薇, 郭勇, 等.亲和层析法分离纯化纳豆激酶[J].食品科学, 2011, 32(16):58-61.
LIU L, LI N W, GUO Y, et al.Affinity chromatographic purification of nattokinase[J].Food Science, 2011, 32 (16):58-61.
[20] 刘俊果, 邢建民, 畅天狮, 等.反胶团萃取分离纯化纳豆激酶[J].科学通报, 2006, 51(2):133-137.
LIU J G, XING J M, CHANG T S, et al.Extraction and purification of nattokinase by reverse micelles[J].Scientific Bulletin, 2006, 51(2):133-137.
[21] 法芸, 张金玲, 赵海杰, 等.纳豆激酶分离纯化和酶活性测定的研究进展 [J].色谱, 2019, 37(3):274-278.
FA Y, ZHANG J L, ZHAO H J, et al.Progress in separation and purification of nattokinase and its enzyme activity determination[J].Chinese Journal of Chromatography, 2019, 37(3):274-278.
[22] 苟金霞, 高栋.纳豆激酶集成化分离技术[J].中国生物工程杂志, 2008, 28(1):119-123.
GOU J X, GAO D.Novel progress in separation and purification technology of nattokinase[J].China Biotechnology, 2008, 28(1):119-123.
[23] M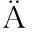 DER U, ANTELMANN H, BUDER T, et al.Bacillus subtilis functional genomics:Genome-wide analysis of the DegS-DegU regulon by transcriptomics and proteomics[J].Molecular Genetics & Genomics Mgg, 2002, 268(4):455-467.
DER U, ANTELMANN H, BUDER T, et al.Bacillus subtilis functional genomics:Genome-wide analysis of the DegS-DegU regulon by transcriptomics and proteomics[J].Molecular Genetics & Genomics Mgg, 2002, 268(4):455-467.
[24] STANLEY N R, LAZAZZERA B A.Defining the genetic differences between wild and domestic strains of Bacillus subtilis that affect poly-gamma-dl-glutamic acid production and biofilm formation[J].Molecular Microbiology, 2010, 57(4):24-27.
Improve the production efficiency of Bacillus natto nattokinase by knocking out the γ-PGA synthetic gene
HAN Yuxing,MENG Fanqiang,ZHOU Libang,LU Zhaoxin*
(College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095,China)
ABSTRACT Nattokinase is a thrombolytic enzyme produced by Bacillus natto. The polyglutamic acid (γ-PGA) produced during the cultivation of B. natto increases the viscosity of the fermentation broth, which makes the separation and purification of nattokinase difficult. It increases the cost and limits the large-scale production of nattokinase. In this study, the γ-PGA synthetic gene pgsB was knocked out by homologous recombination, to reduce the content of γ-PGA and the viscosity of the fermentation broth, which might improve the efficiency of separation and purification of nattokinase. The result showed that the yield of γ-PGA of pgsB-deleted strain reduced significantly compared to the wild strain. At the 24th hour of fermentation, the yield of γ-PGA of pgsB-deleted strain reduced 57.9%. Besides, there was no significant difference in cell density, indicating that the deletion of pgsB gene reduced the viscosity of the fermentation broth and had no effect on the growth of the bacteria. The recovery rate of nattokinase of the pgsB-deleted strain was increased by 19.2% in the process of purifying nattokinase by ultrafiltration. Therefore, the separation and purification efficiency of nattokinase had been improved after knocking out the pgsB gene, which might provide a new method for the industrial production of nattokinase.
Key words double knockout;nattokinase;pgsB gene;γ-PGA

 DER U, ANTELMANN H, BUDER T, et al.
DER U, ANTELMANN H, BUDER T, et al.