不同窖龄及位置窖泥微生物群落和代谢组分的差异
毕天然1,黄钧1,张宿义2,陈晓茹1,陈苏祺1,母雨1,蔡晓波2,邱川峰2,周荣清1*
1(四川大学 轻工科学与工程学院,四川 成都,610065)2(泸州老窖股份有限公司,四川 泸州,646000)
摘 要 基于多相检测技术探究了不同窖龄窖泥微生物群落及主要代谢组分的时空特点。结果表明,窖池微环境和代谢物间相互影响,胁迫微生物群落定向进化。Caproiciproducens和Clostridium sensu stricto 12等7种细菌与窖龄相关,产甲烷古菌则与窖泥的位置有关。此外,不锈钢代替窖泥盖,降低了窖池中Lactobacillus和Nitrosopumilaceae的丰度。窖池微环境和群落间互作共同影响群落组成,Caproiciproducens、Clostridium sensu stricto 12、Methanobacterium和Methanoculleus与己酸酯呈正相关。荧光原位杂交定量检测的结果表明,梭菌属等功能菌群随窖龄的增长而增高,且在窖底泥中含量更高。
关键词 窖泥;微生物群落;风味代谢;高通量测序;荧光原位杂交
DOI:10.13995/j.cnki.11-1802/ts.027756
引用格式:毕天然,黄钧,张宿义,等.不同窖龄及位置窖泥微生物群落和代谢组分的差异[J].食品与发酵工业,2022,48(2):231-237.BI Tianran,HUANG Jun,ZHANG Suyi, et al.Difference of the microbial community and metabolite in pit mud with different age and position[J].Food and Fermentation Industries,2022,48(2):231-237.
第一作者:硕士研究生(周荣清教授为通信作者,E-mail:zhourqing@scu.edu.cn)
收稿日期:2021-04-21,改回日期:2021-05-11
浓香型白酒是复杂的微生物种属协同作用的结果。在其漫长的重复生产过程中,环境胁迫作用驱动窖泥微生物群落的定向进化是生产高质量基酒的必要条件[1],尤其是发酵后期,窖泥中栖息的功能菌群对独特风味的形成具有重要作用[2]。已完成的研究结果表明,窖池中所栖息的Clostridiaceae、Ruminococcaceae、Methanosaeta、Methanobacterium、Methanocorpusculum等功能菌群随着窖龄的增长而增多,Lactobacillus则降低,小生境中氧、酸及乙醇胁迫作用是驱动其变化的主要因素[3-5]。窖池的不同位置窖泥菌群多样性差异显著,DING等[6]在窖壁泥中检出Acinetobacter,而窖底泥未检出,WANG等[7]报道Lactobacillus丰度随窖池深度增加而减小,而Clostridium与之相反。近年来高通量测序技术已成为研究群落的重要工具之一,广泛应用于白酒的研究[5, 8]。荧光原位杂交(fluorescence in situ hybridization,FISH)技术是细胞水平表征活细菌群落结构轮廓的有效手段之一,曾与PCR-DGGE组合,从分子和细胞水平表征浓香型白酒酿造过程的群落特征[9]。
本文基于Illumina Miseq测序等多相技术,探寻不同窖龄窖池窖壁和窖底泥微生物群落及主要代谢组分的差异,应用生物信息学方法解析了群落中种属间互作关系及其与主要代谢组分的相关性。旨在多尺度研究窖泥中核心功能菌群的代谢调控规律,为诠释浓香白酒酿造过程微生态理论奠定基础,为优化生产过程提供技术支撑。
1 材料与方法
1.1 样品收集
样品:窖泥样取自泸州老窖股份有限公司(四川泸州)的30、100、200年窖龄窖池。参考DING等[3]所述方法分别从窖壁和窖底采样,每样取200 g,无菌PE袋密封后保存于-20 ℃冰箱。30、100、200年窖池的窖壁(JB)和窖底(JD)泥的编号分别是JB30、JD30、JB100、JD100、JB200和JD200。
1.2 主要理化性质检测
pH测量:参考HU等[8]所述方法,准确称取2.00 g窖泥,加6 mL超纯水,涡旋振荡5 min,超声30 min,离心(12 000 r/min,4 ℃)10 min,上清液用于pH测量。
总酸和总酯测定:准确称取10.00 g窖泥,加入到100 mL 30%乙醇溶液中,涡旋振荡5 min,超声30 min,4 ℃过夜浸提,滤纸过滤,滤清液按照GB/T 10345—2007所述的方法测定总酸和总酯。
1.3 代谢组分分析
有机酸测定:准确称取5.00 g窖泥, 加20 mL 9 mmol/L H2SO4溶液,超声60 min,其间每隔15 min涡旋振荡5 min,离心(12 000 r/min,4 ℃)10 min收集上清液,上清液经活化后的C18 SPE小柱纯化,再经0.22 μm滤膜过滤,滤液参考DUBER等[10]所述HPLC法检测有机酸含量。以标准品乳酸、乙酸、丙酸、丁酸和己酸的保留时间确定样品中有机酸种类,并通过外标法计算含量。
色谱条件:色谱柱为Alltech OA-1000(300 mm×7.8 mm)有机酸柱,流动相为9 mmol/L H2SO4溶液,流速0.3 mL/min,紫外检测器,检测波长210 nm,柱温75 ℃,进样量10 μL,Agilent 1260高效液相色谱仪(Agilent Technology,美国)。
挥发性组分测定:参考DING等[11]所述的顶空固相微萃取-气相色谱-质谱联用法(headspace solid phase microextraction combined with gas chromatography-mass spectrometry,HS-SPME-GC-MS)测定样品的挥发性组分。准确称取0.50 g窖泥于顶空瓶中,加入10 μL 5.1 mg/L辛酸甲酯内标物质,60 ℃水浴中平衡15 min、50/30 μm DVB/CAR/PDMS纤维萃取头吸附50 min,取出后插入进样口,250 ℃解离5 min进行GC-MS分析。样品质谱数据与标准谱库(NIST2005)比对,匹配度>800(最大为 1 000)的物质予以报道,采用内标法对样品中各检出物质进行半定量。
色谱-质谱条件:色谱柱为HP-INNOWAX毛细管色谱柱(30.0 m×0.32 mm×0.25 um, Agilent, USA);升温程序:40 ℃保持5 min,然后以 4 ℃/min 升至 100 ℃,再以6 ℃/min升至 230 ℃,保持10 min,进样口温度250 ℃;高纯氦作为载气,流速 1.0 mL/min;离子源温度和连接线温度分别为230、250 ℃;EI 电子能量为70 eV;质谱扫描范围为 35~400 amu,Trace GC Ultra-DSQ II气相色谱-单四级杆质谱联用仪(Thermo Fisher Electron,美国)。
1.4 高通量测序检测微生物群落组成
按照HE等[12]所述步骤,采用Fast DNA SPIN试剂盒(MP Biomedicals,Santa Ana,CA,USA)提取样品DNA,NanoDrop ND-1000光度计(Thermo Fisher Scientific,Waltham,MA,USA)和1%琼脂糖电泳检测提取的DNA含量、纯度和完整性。338F/806R和A RC787F/ARC1059R分别用于细菌和古菌16S rDNA 基因V3~V4高变区和ARC区的扩增,而ITS5/ITS1扩增真菌ITS1区。PCR流程参考LI等[13],PCR产物切胶获得目的片段并纯化回收,分光光度计测定后,等分子质量混合。通过试剂盒MiSeq ReagentKit v3构建文库后,送上海派森诺生物科技有限公司Illumina MiSeq平台测序。
测序数据删除碱基平均质量≤Q20及模糊序列,FLASH 软件(V1.2.7,http://ccb.jbu.edu/software/FLASH)拼接初步筛选的双端序列(碱基重叠长度>10 bp,且无碱基错配),将拼接后的序列分配到相应的样本中,获得有效序列。QIIME 软件(Quantitative Insights Into Microbial Ecology,v1.8.0,http://qiime.org/)删除低质量序列(长度≤150 bp,5′端碱基错配>1和相同碱基>8的序列)获得高质量序列。使用UCLUST把高质量序列按照97%的序列相似度聚成不同的可操作分类单元(operational taxonomic units,OTU)。分别采用Greengenes数据库(Releasese 13.8,http://greengenes secondgenome.com/)和 UNIT 数据库(Release 5.0,http://unite.ut.ee/)作为OTU分类鉴定的模板序列,获得每个OTU的微生物分类信息。基于每个 OTU在各样本中所含序列数构建OTU矩阵,确定微生物在各样品中的相对丰度。
1.5 荧光原位杂交检测群落组成轮廓
准确称取1.00 g窖泥,加25 mL 10 mmol/L PBS(pH 7.2)缓冲液,涡旋振荡5 min,离心(800 r/min,4 ℃)10 min收集上清液,上清液离心(12 000 r/min,4 ℃)10 min收集沉淀,相同缓冲液重复3次,获得的菌体沉淀为FISH检测样品,参照何翠容等[14]所述条件与步骤进行FISH检测,靶向探针见表1。
表1 荧光原位杂交靶向探针
Table 1 Target probes of fluorescence in situ hybridization
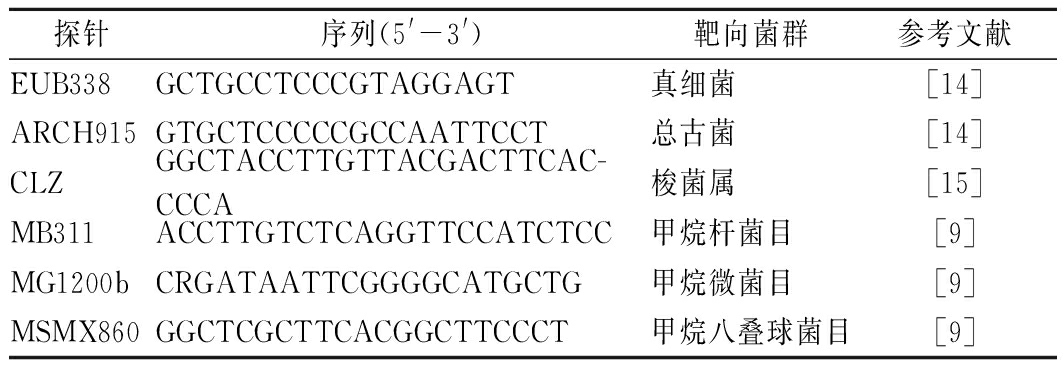
探针序列(5'-3')靶向菌群参考文献EUB338GCTGCCTCCCGTAGGAGT真细菌[14]ARCH915GTGCTCCCCCGCCAATTCCT总古菌[14]CLZGGCTACCTTGTTACGACTTCAC-CCCA梭菌属[15]MB311ACCTTGTCTCAGGTTCCATCTCC甲烷杆菌目[9]MG1200bCRGATAATTCGGGGCATGCTG甲烷微菌目[9]MSMX860GGCTCGCTTCACGGCTTCCCT甲烷八叠球菌目[9]
1.6 数据处理
热图分析、非加权组平均聚类分析(unweighted pair-group method with arithmetic means, UPMGA)和冗余分析(redundancy analysis,RDA)使用R语言软件(R x64 3.6.1,https://r-project.org/)完成。SPSS 19.0软件(SPSS Inc.Chicago,IL,USA)用于Spearman′s相关系数分析和单因素显著性分析,并根据Spearman′s相关系数绘制群落关联网络图。每组检测重复3次,所得数值结果以平均值±标准偏差表征。
2 结果与分析
2.1 窖龄及位置对窖泥理化和主要代谢组分的影响
如表2所示,不同窖龄窖底泥pH和总酸差异不大,总酯的含量则是JD200>JD30>JD100。以不锈钢盖代替泥封窖的100年窖池,窖壁泥pH高于其余2种窖龄,总酸和总酯含量相反。窖底泥的总酸和总酯含量显著高于窖壁泥,JB100的pH高于JD100,其余2种窖龄的窖底和窖壁泥pH略有差异,类似曾报道的结果[16]。
表2 理化及主要代谢组分的差异
Table 2 Differences of physiochemical properties and main metabolic components
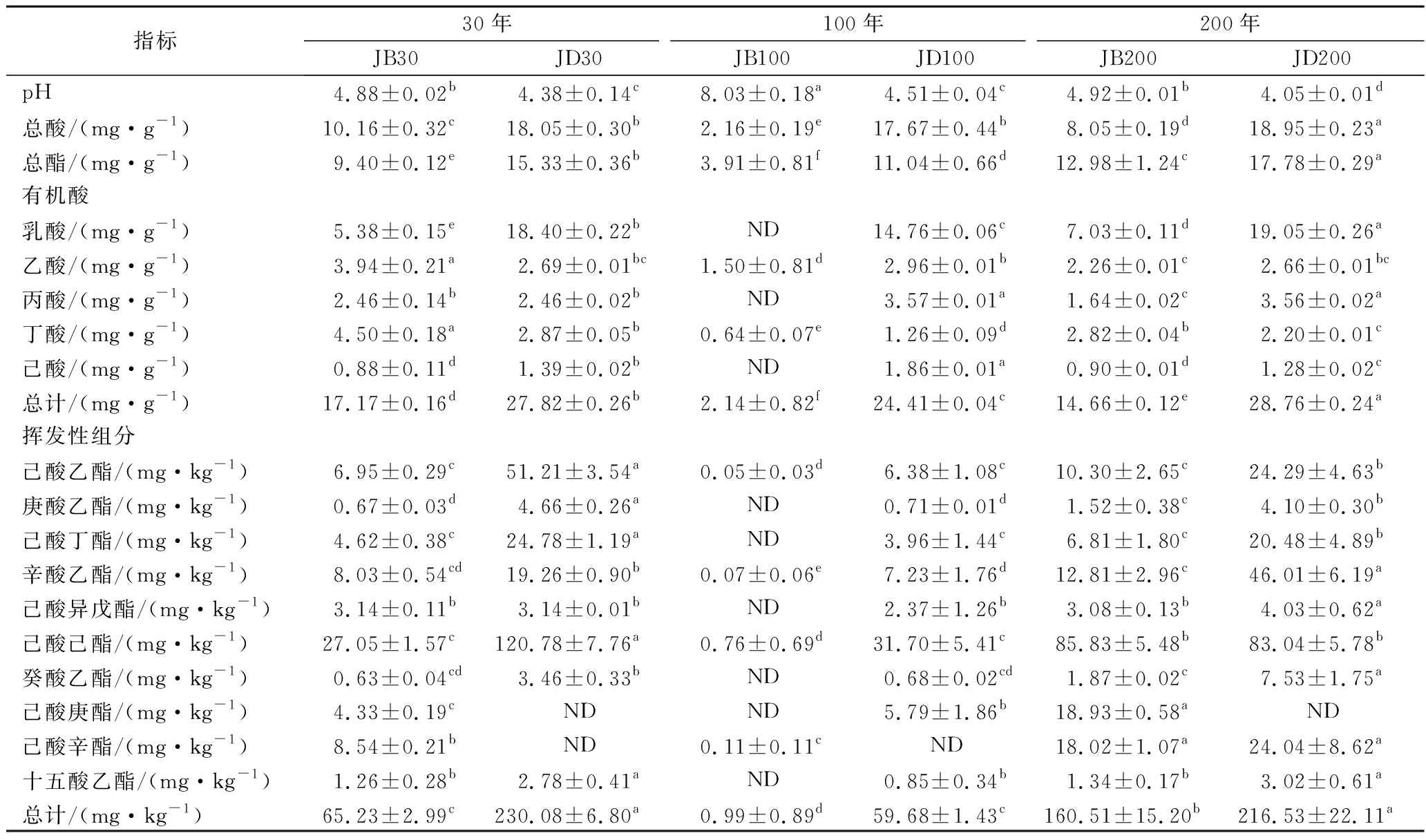
指标30年100年200年JB30JD30JB100JD100JB200JD200pH4.88±0.02b4.38±0.14c8.03±0.18a4.51±0.04c4.92±0.01b4.05±0.01d总酸/(mg·g-1)10.16±0.32c18.05±0.30b2.16±0.19e17.67±0.44b8.05±0.19d18.95±0.23a总酯/(mg·g-1)9.40±0.12e15.33±0.36b3.91±0.81f11.04±0.66d12.98±1.24c17.78±0.29a有机酸乳酸/(mg·g-1)5.38±0.15e18.40±0.22bND14.76±0.06c7.03±0.11d19.05±0.26a乙酸/(mg·g-1)3.94±0.21a2.69±0.01bc1.50±0.81d2.96±0.01b2.26±0.01c2.66±0.01bc丙酸/(mg·g-1)2.46±0.14b2.46±0.02bND3.57±0.01a1.64±0.02c3.56±0.02a丁酸/(mg·g-1)4.50±0.18a2.87±0.05b0.64±0.07e1.26±0.09d2.82±0.04b2.20±0.01c己酸/(mg·g-1)0.88±0.11d1.39±0.02bND1.86±0.01a0.90±0.01d1.28±0.02c总计/(mg·g-1)17.17±0.16d27.82±0.26b2.14±0.82f24.41±0.04c14.66±0.12e28.76±0.24a挥发性组分己酸乙酯/(mg·kg-1)6.95±0.29c51.21±3.54a0.05±0.03d6.38±1.08c10.30±2.65c24.29±4.63b庚酸乙酯/(mg·kg-1)0.67±0.03d4.66±0.26aND0.71±0.01d1.52±0.38c4.10±0.30b己酸丁酯/(mg·kg-1)4.62±0.38c24.78±1.19aND3.96±1.44c6.81±1.80c20.48±4.89b辛酸乙酯/(mg·kg-1)8.03±0.54cd19.26±0.90b0.07±0.06e7.23±1.76d12.81±2.96c46.01±6.19a己酸异戊酯/(mg·kg-1)3.14±0.11b3.14±0.01bND2.37±1.26b3.08±0.13b4.03±0.62a己酸己酯/(mg·kg-1)27.05±1.57c120.78±7.76a0.76±0.69d31.70±5.41c85.83±5.48b83.04±5.78b癸酸乙酯/(mg·kg-1)0.63±0.04cd3.46±0.33bND0.68±0.02cd1.87±0.02c7.53±1.75a己酸庚酯/(mg·kg-1)4.33±0.19cNDND5.79±1.86b18.93±0.58aND己酸辛酯/(mg·kg-1)8.54±0.21bND0.11±0.11cND18.02±1.07a24.04±8.62a十五酸乙酯/(mg·kg-1)1.26±0.28b2.78±0.41aND0.85±0.34b1.34±0.17b3.02±0.61a总计/(mg·kg-1)65.23±2.99c230.08±6.80a0.99±0.89d59.68±1.43c160.51±15.20b216.53±22.11a
注:表中不同小写字母代表差异显著(P<0.05);ND代表未检出
各样品共检出了5种有机酸和31种酯类成分,有机酸及10种优势酯类组分(丰度排名前10),含量如表2所示。窖底泥有机酸及这些酯类组分总含量显著高于窖壁泥,窖底泥间有机酸含量略有差异,JD100酯类组分最低。JB100有机酸和酯类组分含量显著低于JB30和JB200,后两者间也略有差异。JB30中乳酸、乙酸和丁酸是优势有机酸,JB200乳酸是优势有机酸,乙酸和丁酸次之。JB100仅检出己酸乙酯、辛酸乙酯、己酸己酯和己酸辛酯,其中己酸己酯的丰度是76.77%,JD100己酸己酯丰度是53.13%。30年和200年窖池泥中的己酸乙酯、辛酸乙酯、己酸已酯和己酸辛酯是优势组分,JB30中己酸乙酯和己酸己酯的丰度<JD30,辛酸乙酯和己酸辛酯则相反,且在JD30未检出己酸辛酯,200和30年窖池的结果则相反。
2.2 窖龄及位置对微生物群落α-多样性的影响
如表3所示,100年窖池的细菌α-多样性指数中的Chao1及Observed species高于30年和200年窖池。可能因封窖方式不同,增大了细菌的物种数,且窖壁和窖底物种数量差异显著。200年窖池中细菌物种数略低于30年窖池,无论是稀有还是丰富物种,30年和100年窖池细菌菌群易被外源物种扰动,尤其是100年窖池。30年和100年窖池泥中的古菌群的物种数量低于200年的,且随着窖龄的增长,窖壁和窖底泥差异减小。真菌的物种数量仅略有差异。
表3 不同窖泥微生物群落α-多样性的差异
Table 3 Differences of microbial communities′
α-diversity among pit muds
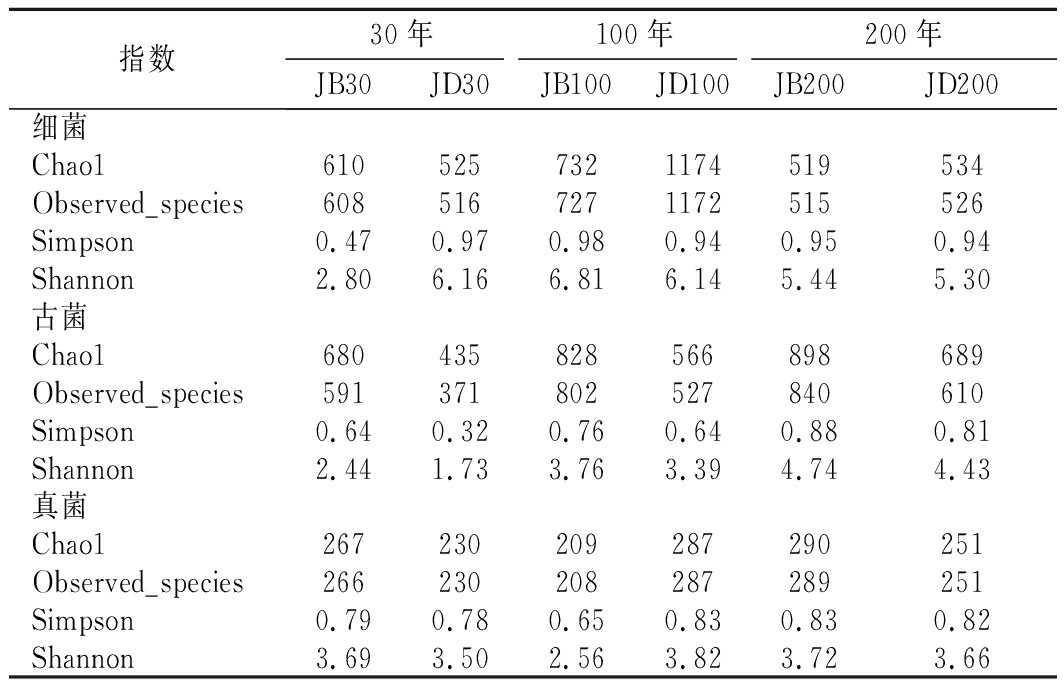
指数30年100年200年JB30JD30JB100JD100JB200JD200细菌Chao16105257321174519534Observed_species6085167271172515526Simpson0.470.970.980.940.950.94Shannon2.806.166.816.145.445.30古菌Chao1680435828566898689Observed_species591371802527840610Simpson0.640.320.760.640.880.81Shannon2.441.733.763.394.744.43真菌Chao1267230209287290251Observed_species266230208287289251Simpson0.790.780.650.830.830.82Shannon3.693.502.563.823.723.66
基于Bray-Curtis距离的微生物群落UPMGA结果如图1所示。JB200和JD200的细菌群落结构类似,100年窖池的也仅略有差异,JB30和JD30差异显著,类似DENG等[17]的结果。每个窖池古菌群落均未聚到一簇,与α-多样性所呈现的规律一致。JB200真菌结构与其余窖泥差异显著。
2.3 窖龄及位置对微生物群落组成的影响
如图2所示,群落中的优势种属(相对丰度>1%)分别由44种细菌、10种古菌及13种真菌组成,相对丰度总和分别为88.4%~92.8%、98.8%~99.7%和92.1%~97.4%。优势细菌主要是Christensenellaceae R-7 group等12个种属。热图分析表明Clostridium sensu stricto 12、uncultured_Clostridiaceae 1、Caproiciproducen、unclassified_Ruminococcaceae 和Christensenellaceae R-7 group等5个种属的丰度与窖龄呈正相关,类似WANG等[4]报道的结果。Clostridium和Caproiciproducens分别能将乙醇和乳酸转化为己酸[18-19]等浓香型白酒呈香组分的前体物质。窖底泥中Christensenellaceae R-7 、group Aminobacterium、Petrimonas和uncultured_Clostridiaceae 1的丰度高于窖壁泥,可能与窖底厌氧程度有关[20-22]。7个隶属于梭菌纲的属丰度为36.4%,与窖龄有关,4个隶属于梭菌纲、拟杆菌纲和互养菌纲的属丰度为16.6%,与位置有关。30年和200年窖池中Lactobacillus的平均丰度(36.9%和8.8%)显著高于100年窖池(0.9%)。
Methanobacterium、Methanoculleus、Methanosarcina和unclassified_Nitrosopumilaceae是主要优势古菌,与位置有关。窖底泥甲烷菌及Methanobacterium的丰度较窖壁泥高,而Methanosarcina相反。unclassified_Nitrosopumilaceae是好氧菌[24],在窖壁泥中的丰度高于窖底泥,此外在100年窖池中丰度最低。
Dipodascus、Candida、Apiotrichum、unclassified_Dipodascacea、unidentified_Dipodascaceae和unclassified_Saccharomycetales是主要优势真菌。隶属于Dipodascaceae的3种属在所有窖泥中均被检出,丰度在24.9%~90.2%,Dipodascus在Dipodascaceae中的比例>52.2%。Candida在JB200中的丰度是53.6%,在其余5个窖泥中则< 5.4%。Candida能代谢乙醇和乳酸转化为风味组分[24]。
应用FISH检测3种不同窖龄窖泥中的细菌群落结构结果如表4所示。JB100真细菌的数量高于JB30的,JD100真细菌数量高于JD30和JD200的。梭菌属的数量在窖底高于窖壁,且随着窖龄延长而增高,100年窖池梭菌属的比例略高于其余2种窖龄。产甲烷古菌的总量随窖龄增长而增高,窖壁氢营养型古菌的比例随窖龄增加而增高,窖底泥中则是乙酸营养型古菌的比例增高。值得注意的是FISH检测的结果与高通量测序的结果显著不同,前者检出的甲烷杆菌目的比例>36%,而后者的结果<3%,主要是因为FISH是活细胞水平检出,而后者在OTU水平,样品既包括活细胞又有死亡细胞,引物和扩增速率误差等干扰也难避免。2种方法的趋势相同则从细胞和分子水平反映窖龄及位置的影响规律。
表4 窖泥微生物群落的荧光原位杂交检测结果 单位:109cell/g
Table 4 Fluorescence in situ hybridization detection′s
results of microbial communities in pit mud

种类30年100年200年JB30JD30JB100JD100JB200JD200真细菌3.68±0.356.03±0.766.36±0.559.55±0.806.62±0.557.87±0.75梭菌属1.76±0.553.52±0.233.68±0.357.29±0.643.77±0.665.02±0.42总古菌2.34±0.236.03±0.372.93±0.429.21±0.515.95±0.359.38±0.48甲烷杆菌目0.84±0.302.34±0.371.34±0.353.94±1.172.93±0.513.01±0.35甲烷微菌目0.34±0.191.84±0.480.67±0.371.76±0.191.76±0.553.94±0.37甲烷八叠球菌目0.59±0.231.34±0.350.67±0.233.60±0.761.42±0.562.18±0.35
综合以上分析,窖池的环境,尤其是酸度/pH显著影响群落结构及代谢,群落及代谢又影响窖泥小生境,这些因素互作是窖泥群落定向进化的关键环境胁迫作用[25],封窖方式则可能是影响定向进化另一个关键的环境胁迫因素。
2.4 优势菌的相关性分析
基于斯皮尔曼相关性 (|ρ|>0.6,P<0.05)分析丰度> 1.0%属水平的微生物的结果如图3所示。这些微生物包含54对共现、26对排斥关系。模块化分析结果表明关联网络主要由模块内和模块间互作等2种关联模式构成。在模块内关联的3个模块中,module_1的Caproiciproducens和Clostridium sensu stricto 12呈正相关,这2个属随窖龄的变化趋势相同,它们是窖泥中主要的己酸菌,能够通过逆向β-氧化合成乙酸、丁酸和己酸,其中乙酸和丁酸是己酸合成中的电子受体[26],两者的一些产物互为彼此的二次发酵底物。module_6则是Methanosarcina与Anaerocella呈正相关,丰度与位置的关系相同。module_3中Methanoculleus和Methanofollis呈正相关,它们同为甲烷微菌科古菌,生理特性和应激能力相似,在窖池微环境变化的过程中具有相同的变化趋势。这类网络结构简单,节点数较少,窖池微环境的变化如乳酸、含氧量等可能是引起微生物群落发生变化的主要因素。模块间关联共有4个模块,又分为多对一和多对多的2类互作关系。在多对一的互作关系中,如module_7和module_8,Sedimentibacter和Syntrophomonas等为中心的共现群落与Lactobacillus和Candida共现对间呈负相关,而与Dipodascus呈正相关。在多对多的互作关系中,如module_4和module_5,Petrimonas与Methanobacterium等组成的共现群落与由unclassified_Nitrosopumilaceae等组成的共现群落以及由uncultured_Clostridiaceae 1等组成的互作群落之间呈负相关。这类互作关系,除自身复杂外,群落丰度还与位置有关,可能是小生境胁迫与群落互作共同致其改变。Lactobacillus的产物乳酸的积累,降低了窖泥pH,可能改变群落结构,Lactobacillus与Candida呈正相关可能是因为二者之间存在协同共生关系[27]。为了维持群落的动态平衡,种间氢转移,与甲烷菌互养,提高碳利用率及促进碳循环效率[21-22,28]是浓香型白酒窖池发酵重要调控机制。
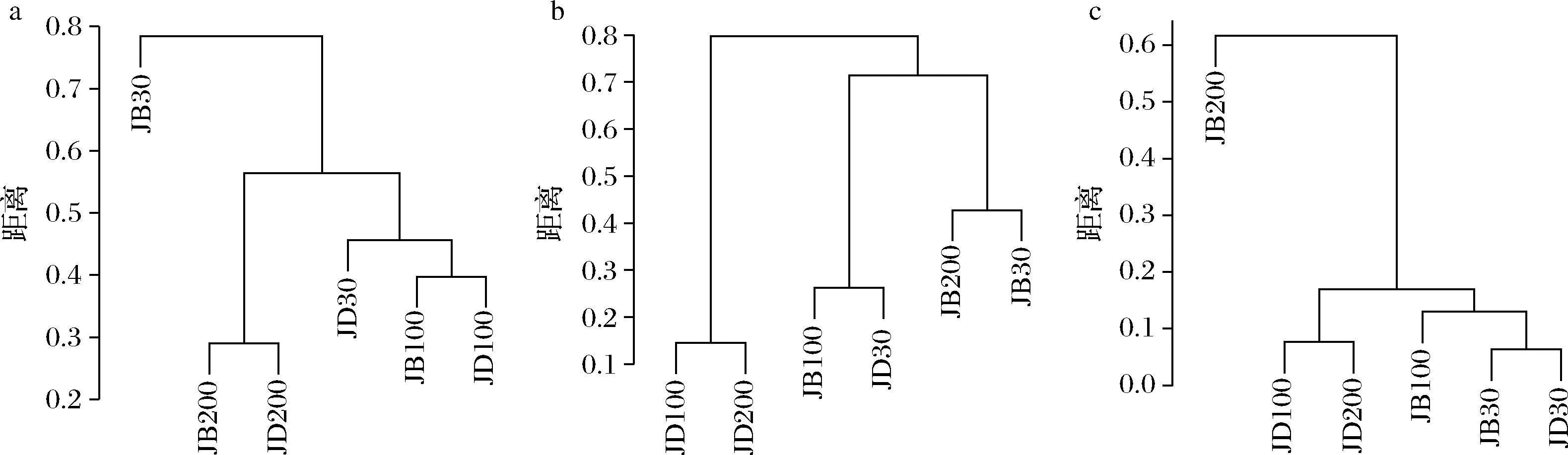
2.5 核心微生物与窖泥风味之间的RDA
基于高通量测序所得窖泥核心菌群与主要风味组分间的RDA结果见图4。Lactobacillus与乙酸和丁酸呈正相关,但与乳酸和己酸呈负相关。发酵过程中乳酸随黄水传递至窖底,窖底泥乳酸的含量高于窖壁,而乳酸菌丰度随窖池的深度增加而降低[7]。Lactobacillus与己酸乙酯、己酸己酯和辛酸乙酯呈负相关。乳酸是Caproiciproducens和Clostridium sensu stricto 12的底物[19],与之正相关,乙酸、丁酸作为合成己酸的电子受体被转化,所以呈负相关。己酸与醇缩合为己酸酯,虽然Caproiciproducens和Clostridium sensu stricto 12与己酸仅是弱相关性,但与己酸酯正相关。Petrimonas等与乳酸、己酸和己酸乙酯呈正相关。Methanobacterium与己酸呈强烈的正相关,unclassified_Nitrosopumilaceae与己酸呈负相关,Methanoculleus与己酸酯呈正相关。Methanobacterium和Methanoculleus是氢营养型古菌[29],能够降低生物合成己酸过程中的生物氢气[18],促进己酸和己酸酯的合成。自养型古菌Nitrosopumilaceae[23]和Clostridium[30]都以CO2作为底物,二者底物的竞争降低了己酸的合成速率。Dipodascus和Candida仅与丁酸和己酸己酯等相关。
3 结论
应用高通量测序及FISH研究窖龄及位置对微生物群落结构的影响,百年老窖泥的细菌群落中Caproiciproducens和Clostridium sensu stricto 12的丰度高,窖壁和窖底的古菌群落结构差异显著,前者Nitrosopumilaceae显著高于后者的,产甲烷古菌的丰度则在后者中更高。除了窖龄和位置以外,封窖材料可能也对微生物群落结构产生影响,不锈钢盖窖池中Lactobacillus和Nitrosopumilaceae的丰度显著低于泥窖,其具体的影响机制还需要进一步研究。群落结构的差异导致理化性质及酯代谢组分组成及含量差异显著。RDA的结果表明Caproiciproducens和Clostridium sensu stricto 12与乳酸和己酸酯呈正相关,产甲烷古菌与己酸和己酸酯呈正相关。FISH检出的菌群轮廓差异与高通量测序检测的结果是相似的。
参考文献
[1] ZHAO J S, ZHENG J, ZHOU R Q, et al.Microbial community structure of pit mud in a Chinese strong aromatic liquor fermentation pit[J].Journal of the Institute of Brewing, 2012, 118:356-360.
[2] WANG X S, DU H, XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor[J].International Journal of Food Microbiology, 2017, 244:27-35.
[3] DING X F, WU C D, HUANG J, et al.Interphase microbial community characteristics in the fermentation cellar of Chinese Luzhou-flavor liquor determined by PLFA and DGGE profiles[J].Food Research International, 2015, 72:16-24.
[4] WANG X J, ZHU H M, REN Z Q, et al.Characterization of microbial diversity and community structure in fermentation pit mud of different ages for production of strong-aroma Baijiu[J].Polish Journal of Microbiology, 2020, 69(2):151-164.
[5] TAO Y, LI J, RUI J, et al.Prokaryotic communities in pit mud from different-aged cellars used for the production of Chinese strong-flavored liquor[J].Applied & Environmental Microbiology, 2014, 80(7):2 254-2 260.
[6] DING X F, WU C D, ZHANG L Q, et al.Characterization of eubacterial and archaeal community diversity in the pit mud of Chinese Luzhou-flavor liquor by nested PCR-DGGE[J].World Journal of Microbiology and Biotechnology, 2014, 30(2):605-612.
[7] WANG C D, CHEN Q, WANG Q, et al.Long-term batch brewing accumulates adaptive microbes, which comprehensively produce more flavorful Chinese liquors[J].Food Research International, 2014, 62:894-901.
[8] HU X L, DU H, REN C, et al.Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J].Applied & Environmental Microbiology, 2016, 82(8):2 506-2 515.
[9] WU C D, DING X F, HUANG J, et al.Characterization of archaeal community in Luzhou-flavor pit mud[J].Journal of the Institute of Brewing, 2015, 121(4):597-602.
[10] DUBER A, JAROSZYNSKI L, ZAGRODNIK R, et al.Exploiting the real wastewater potential for resource recovery-n-caproate production from acid whey[J].Green Chemistry, 2018, 20(16):3 790-3 803.
[11] DING X F, HUANG J, WU C D, et al.Effects of different distillation patterns on main compounds of Chinese Luzhou-flavor raw liquors[J].Journal of the Institute of Brewing, 2017, 123(3):442-451.
[12] HE G Q, HUANG J, WU C D, et al.Bioturbation effect of fortified Daqu on microbial community and flavor metabolite in Chinese strong-flavor liquor brewing microecosystem[J].Food Research International, 2020, 129:108851.
[13] LI X Z, RUI J P, MAO Y J, et al.Dynamics of the bacterial community structure in the rhizosphere of a maize cultivar[J].Soil Biology & Biochemistry, 2014, 68:392-401.
[14] 何翠容, 吴冬梅, 郑佳, 等.浓香型白酒窖池细菌与古菌随窖龄变化的特征[J].酿酒科技, 2013(1):27-30;38.
HE C R, WU D M, ZHENG J, et al.Investigation on bacterial and archaea communities in the fermentation pits of Nong-flavor liquor as pit age goes by[J].Liquor-Making Science & Technology, 2013(1):27-30;38.
[15] HU L T, HUANG J, LI H, et al.Design, optimization and verification of 16S rRNA oligonucleotide probes of fluorescence in-situ hybridization for targeting Clostridium spp. and Clostridium kluyveri[J].Journal of microbiology and biotechnology, 2018, 28(11):1 823-1 833.
[16] 胡晓龙, 余苗, 王康丽, 等.浓香型白酒窖泥微生物群落多样性及理化因素对其影响[J].食品研究与开发, 2021, 42(2):178-185;204.
HU X L, YU M, WANG K L, et al. Diversity of microbial community and its correlation with physicochemical factors in Luzhou-flavor liquor pit mud[J]. Food Research and Development, 2021, 42(2):178-185; 204.
[17] DENG B, SHEN C H, SHAN X H, et al.PCR-DGGE analysis on microbial communities in pit mud of cellars used for different periods of time[J].Journal of the Institute of Brewing, 2012, 118(1):120-126.
[18] DING H B, TAN G, WANG J Y.Caproate formation in mixed-culture fermentative hydrogen production[J].Bioresource Technology, 2010, 101(24):9 550-9 559.
[19] CANDRY P,  L, FAVERE J, et al.Mildly acidic pH selects for chain elongation to caproic acid over alternative pathways during lactic acid fermentation[J].Water Research, 2020, 186:116396.
L, FAVERE J, et al.Mildly acidic pH selects for chain elongation to caproic acid over alternative pathways during lactic acid fermentation[J].Water Research, 2020, 186:116396.
[20] HE J, HAI L, ORGOLDOL K, et al.High-throughput sequencing reveals the gut microbiome of the bactrian camel in different ages[J].Current Microbiology, 2019, 76(7):810-817.
[21] HAMDI O, BEN HANIA W, POSTEC A, et al.Aminobacterium thunnarium sp.nov., a mesophilic, amino acid-degrading bacterium isolated from an anaerobic sludge digester, pertaining to the phylum Synergistetes[J].International Journal of Systematic and Evolutionary Microbiology, 2015, 65:609-614.
[22] GRABOWSKI A, TINDALL B J, BARDIN V, et al.Petrimonas sulfuriphila gen.nov., sp.nov., a mesophilic fermentative bacterium isolated from a biodegraded oil reservoir[J].International Journal of Systematic and Evolutionary Microbiology, 2005, 55(3):1 113-1 121.
[23] QIN W, HEAL K R, RAMDASI R, et al.Nitrosopumilus maritimus gen.nov.sp.nov., Nitrosopumilus cobalaminigenes sp.nov., Nitrosopumilus oxyclinae sp.nov. and Nitrosopumilus ureiphilus sp.nov., four marine ammonia-oxidizing archaea of the phylum Thaumarchaeota[J].International Journal of Systematic and Evolutionary Microbiology, 2017, 67(12):5 067-5 079.
[24] XING X, WANG Y, HUO N, et al.Candida ethanolica strain Y18 enhances aroma of Shanxi aged-vinegar[J].Food Science and Technology Research, 2018, 24(6):1 069-1 081.
[25] ZHANG M Z, WU X F, MU D D, et al.Profiling the effects of physicochemical indexes on the microbial diversity and its aroma substances in pit mud[J].Letters in Applied Microbiology, 2020, 71:667-678.
[26] SAN-VALERO P, FERN NDEZ-NAVEIRA
NDEZ-NAVEIRA 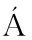 , VEIGA M C, et al.Influence of electron acceptors on hexanoic acid production by Clostridium kluyveri[J].Journal of Environmental Management, 2019, 242:515-521.
, VEIGA M C, et al.Influence of electron acceptors on hexanoic acid production by Clostridium kluyveri[J].Journal of Environmental Management, 2019, 242:515-521.
[27] 谢凤行, 周可, 张峰峰, 等.产朊假丝酵母、植物乳杆菌混合发酵及净水效果[J].食品与发酵工业, 2019, 45(6):185-192.
XIE F X, ZHOU K, ZHANG F F, et al.Mixed fermentation of Candida utilis and Lactobacillus plantarum and its water purification effect[J].Food and Fermentation Industries, 2019, 45(6):185-192.
[28] ZHANG C Y, LIU X L, DONG X Z.Syntrophomonas curvata sp.nov., an anaerobe that degrades fatty acids in co-culture with methanogens[J].International Journal of Systematic and Evolutionary Microbiology, 2004, 54(3):969-973.
[29] KATO S, TAKASHINO M, IGARASHI K, et al.An iron corrosion-assisted H2-supplying system:A culture method for methanogens and acetogens under low H2 pressures[J].Scientifc Reports, 2020, 10:19124.
[30] YOUNESI H, NAJAFPOUR G, MOHAMED A R.Ethanol and acetate production from synthesis gas via fermentation processes using anaerobic bacterium, Clostridium jungdahlii[J].Biochemical Engineering Journal, 2005, 27(2):110-119.
Difference of the microbial community and metabolite in pit mud with different age and position
BI Tianran1,HUANG Jun1,ZHANG Suyi2,CHEN Xiaoru1,CHEN Suqi1,MU Yu1,CAI Xiaobo2,QIU Chuanfeng2,ZHOU Rongqing1*
1(College of Biomass Science & Engineering, Sichuan University, Chengdu 610065, China)2(Luzhoulaojiao Co.Ltd., Luzhou 646000, China)
ABSTRACT The time-space characteristics of microbial communities and major metabolic components in pit mud were explored by multiphase detection technology. The results suggested that pits′ microenvironment and flavored metabolites influenced each other, which led to the directed evolution of microbial communities. Seven of genera, including Caproiciproducens, Clostridium sensu stricto 12, were related with age, while methanogen was associated with location. Besides, the abundance of Lactobacillus and Nitrosopumilaceae was decreased as sealing the cellar by stainless steel instead of pit mud. Community composition was not only affected by the microenvironment of pits, but also by community interaction. Caproiciproducens, Clostridium sensu stricto 12, Methanobacterium and Methanoculleus were positively correlated with caproate ester. The quantitative detection results of fluorescence in situ hybridization uncovered that the functional flora such as Clostridium were increased with the increasing of pit age, and was higher in the bottom of the cellar.
Key words pit mud;microbial community;flavoured metabolites;high-throughput sequencing;fluorescence in situ hybridization






 NDEZ-NAVEIRA
NDEZ-NAVEIRA