表1 嗜酸乳杆菌对照组、氯化锂处理组自聚集率变化
及生长情况变化
Table 1 The self-aggregation rate and growth of
Lactobacillus acidophilus in control, treated
with lithium chloride

组别生长(OD600nm)自聚集率/%对照组 0.881±0.007a80.58±2.50a氯化锂处理组0.706±0.004b48.52±4.08b
注:不同小写字母表示差异显著(P<0.05)
许多乳杆菌的表面都包裹着一层蛋白质,即表层蛋白。携带此类蛋白的乳杆菌有嗜酸乳杆菌、发酵乳杆菌、卷曲乳杆菌、干酪乳杆菌、格氏乳杆菌、短乳杆菌、约氏乳杆菌等[1]。嗜酸乳杆菌是一类重要的益生菌,能缓解轮状病毒腹泻、腹泻型肠易激综合征及牛乳过敏等,主要通过定殖于宿主肠道、与致病菌竞争黏附位点、调节机体免疫等机制实现其益生功能[2-3]。
表层蛋白不仅作为黏附因子帮助乳杆菌定殖于肠道,其对菌株的本身特性有很多影响,ABRAMOV等[4]构建了4个嗜酸乳杆菌表层蛋白突变体,在存在酒精、胆盐等环境胁迫的条件下4个突变体的生长速度有显著差异,这表明表层蛋白的存在影响了嗜酸乳杆菌在不良环境中的生长。同时,表层蛋白的剥离使嗜酸乳杆菌的黏附性降低,且剥离后的乳杆菌菌量降低1~2个对数级[5],表明其生长状态受到表层蛋白剥离的影响,而菌体的生长状态与代谢水平息息相关。
近年来,为更加深入地了解表层蛋白的特性以推动其在生物技术领域的应用,许多研究从基因水平[6]、蛋白质水平[7]等方面阐述了乳杆菌表层蛋白特性,而在代谢水平上,表层蛋白对嗜酸乳杆菌的影响尚不清楚。非靶向代谢组学广泛应用于分析生物个体的代谢状态,它可以对生物系统的各种生化代谢物进行轮廓分析,其主要基于核磁共振波谱仪(nuclear magnetic resonance spectrometer,NMR)、GC-MS、液相色谱-质谱联用仪(liquid chromatograph-mass spectrometer,LC-MS)等平台[8]。目前,对于嗜酸乳杆菌NCFM表层蛋白的研究较为广泛且深入,在了解其氨基酸组成的基础上,本研究拟通过基于GC-MS的非靶向代谢组学揭示嗜酸乳杆菌NCFM表层蛋白对嗜酸乳杆菌的自聚集能力、菌体生长、代谢产物等特性的影响。
嗜酸乳杆菌NCFM,美国菌种保藏中心,由江南大学食品生物技术中心保藏。
甲醇(HPLC级)、乙腈(HPLC级),Merck KGaA,吡啶溶液(99%)、N-甲基-N-(三甲基甲硅烷基)三氟乙酰胺+1%三甲基氯硅烷[N-methyl-N-(trimethylsilyl) trifluoroacetamide+1% trimethylchlorosilane, MSTFA+1%TMCS]、甲氧胺盐酸盐(methoxyamine hydrochloride,MeOX),Sigma,LiCl(分析纯),国药沪试。
Trace 1310-TSQ8000气质联用仪、真空干燥机,Thermo公司;高通量组织破碎仪,新芝;酶标仪,ThermoFisher;干式恒温金属浴,杭州瑞诚仪器有限公司。
1.3.1 乳杆菌的培养
取出-80 ℃保藏的菌种,以2%的接种量接种于 5 mL MRS液体培养基中,37 ℃静置培养 18 h。活化3代后可用于实验。
1.3.2 表层蛋白的去除
参考JOHNSON等[9]的方法并做适当改动。培养好的菌液于室温下6 000×g离心15 min,收集菌体。向菌体中加入原乳杆菌培养液1/20体积的5 mol/L LiCl溶液并置于恒温摇床(37 ℃,220 r/min)处理2 h后,于室温下12 000 ×g离心15 min。弃上清液,得到的菌体即为去除表层蛋白的乳杆菌,空白对照组以生理盐水代替。
1.3.3 乳杆菌的生长及自聚集能力测定
将去除表层蛋白和对照组的乳杆菌以2%的接种量接种于 5 mL MRS 培养基中,37 ℃静置培养18 h。调节菌悬液的浓度为108 CFU/mL,记录其OD值为A0,将菌悬液涡旋振荡约10 s,室温静置4 h,上清液的OD600nm记为A4,按如下公式计算自聚集率[10]。
1.3.4 胞内代谢物的提取
将1.3.2收集的菌体置于1.5 mL EP管中,迅速放入液氮中淬灭,置于-80 ℃冰箱备用。加入400 μL预冷的提取剂[V(甲醇)∶V(乙腈)∶V(水)=2∶2∶1],再加入100 μL的小磁珠,放在预冷的模块中,以高通量组织破碎仪破碎。高通量组织破碎仪参数设置为破碎45 s,停15 s,循环10次。破碎后于4 ℃以12 000×g离心15 min,收集提取液,并转移至新的离心管中。每个样品取300 μL上清液放入真空浓缩仪,于45 ℃,0.1 kPa干燥1.5 h,得到干燥不含水的代谢组样品[11]。为确保试验的准确性,每组设置6个平行。
1.3.5 代谢物的衍生化
加入100 μL的0.1 mg/mL甲氧胺盐酸吡啶溶液,涡旋振荡30 s,于37 ℃干式恒温金属浴90 min,然后加入40 μL的MSTFA试剂(含1% TMCS),37 ℃金属浴30 min[12]。室温12 000×g离心15 min,吸取100 μL上清液至内衬管中,进行GC-MS分析。
1.3.6 GC-MS条件
扫描的质量数为33~600 m/z,离子源类型为EI源,程序升温:起始温度为70 ℃,以5 ℃/min 的升温速率提高至 230 ℃,后以90 ℃/min的升温速率提高至 320 ℃,在此条件下维持5 min;载气(He)流速1 mL/min;电子电离能量:70 eV;柱子型号为 RTX-5MS(30 m×0.25 mm,0.25 μm)[11]。
1.3.7 数据采集与分析
使用气质联用仪对胞内代谢物进行检测分析,使用AbfConverter.4.0.0软件进行格式转换,用MS-DIAL 4.3.8 软件对所产生的原始数据文件进行峰提取、保留时间校正、峰对齐等处理得到包括胞内代谢物的质谱信息、峰面积和保留时间等相关数据,用MS-DIAL4.3.8软件配置的DB_FiehnBinbase-FiehnRI数据库[13]进行化合物的鉴定。将得到的归一化后的峰面积数据矩阵导入metaboanalyst 4.0[14]进行数据分析及代谢通路分析,使用R包PCAtools,DESeq2,ggplot2等进行可视化。结合T检验寻找差异代谢物和计算显著性(P<0.05)。
5 mol/L的氯化锂溶液可以去除菌体表面超过90%的表层蛋白,且不会导致菌体失活[15],故本研究采取该方法以去除表层蛋白。乳杆菌的生长和自聚集率变化如表1所示。嗜酸乳杆菌NCFM自聚集率变化则随表层蛋白的去除有显著下降,表明表层蛋白在嗜酸乳杆菌NCFM的黏附性上起重要作用,这也与文献报道相符合[16]。LiCl处理后嗜酸乳杆菌生长终浓度比对照组低,我们推测表层蛋白的去除会造成乳酸菌胞内氨基酸含量的降低,而氨基酸的重新合成,需要糖酵解和三羧酸循环途径过程中产生的能量,故影响菌体的正常生长和代谢,导致生长速率降低[17]。
表1 嗜酸乳杆菌对照组、氯化锂处理组自聚集率变化
及生长情况变化
Table 1 The self-aggregation rate and growth of
Lactobacillus acidophilus in control, treated
with lithium chloride

组别生长(OD600nm)自聚集率/%对照组 0.881±0.007a80.58±2.50a氯化锂处理组0.706±0.004b48.52±4.08b
注:不同小写字母表示差异显著(P<0.05)
采用基于GC-MS的代谢组学分析技术,测定表层蛋白去除与否对乳酸菌胞内代谢物的影响,并用主成分分析(principal component analysis,PCA)对胞内代谢物进行轮廓分析,结果如图1-A所示。本次试验中,使用 PCA 模型可以取得较好的拟合效果,模型的PC1和PC2分别为53%和26.9%,满足数据的拟合要求。从图1-A可看出,表层蛋白的去除对乳酸菌胞内代谢有显著影响,处理组与对照组的样本在得分图上可以较好的分开,并且组内的6个样本点具有较好的统一性。
与PCA相似,偏最小二乘法判别分析(partial least squares discriminant analysis,PLS-DA)也是目前代谢组学数据分析中最常使用的一种分类方法,为有监督的分析方法,它在降维的同时结合了回归模型[18],可以结合判别阈值VIP(variable important in projection)值化合物进行差异分析,便于寻找差异代谢物。PLS-DA的结果如图1-B所示。2组的样品各居一侧,区分效果明显,说明表层蛋白的去除对乳杆菌细胞内代谢产生影响,胞内的小分子代谢物存在差异。
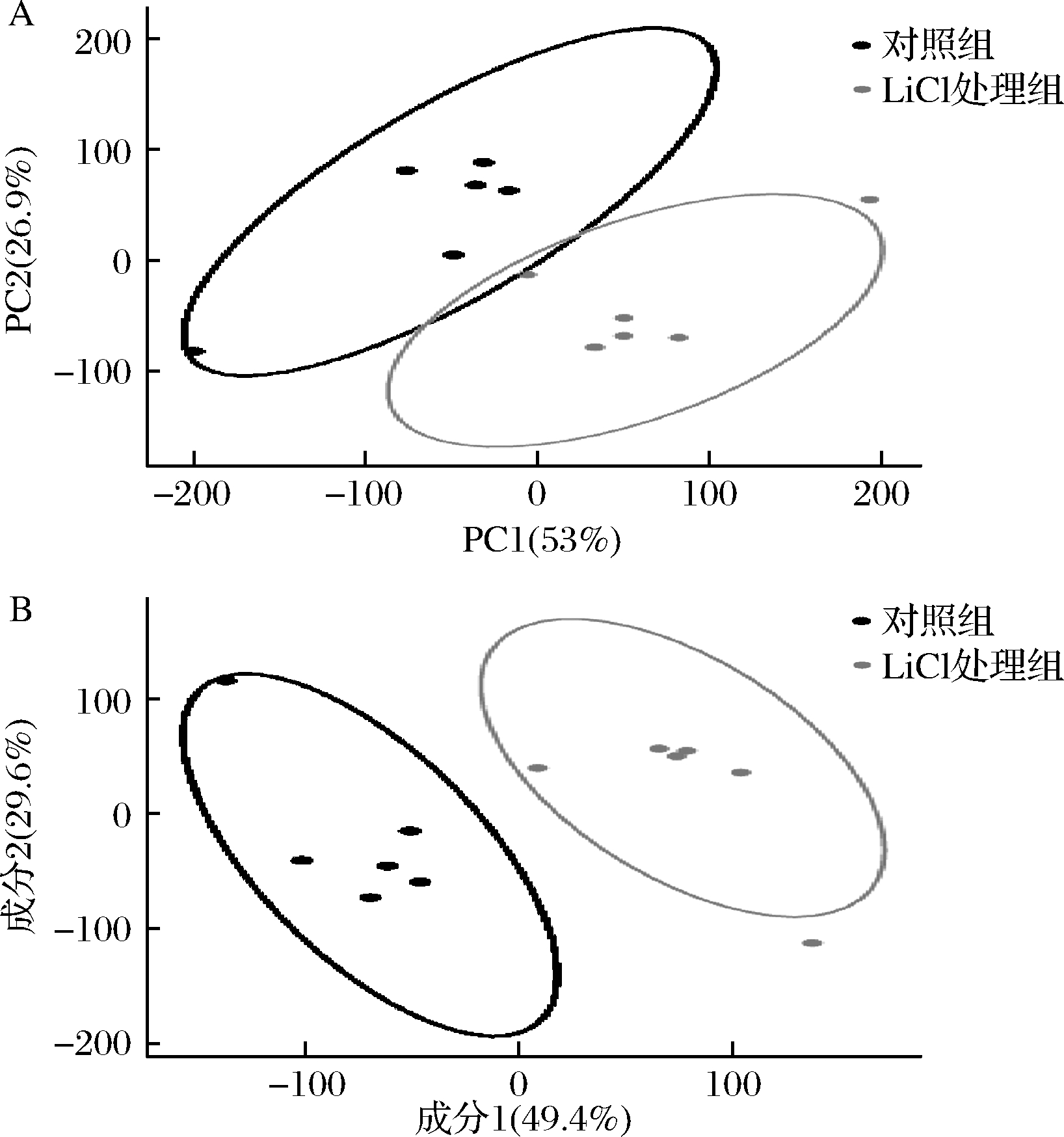
A-PCA得分图;B-PLS-DA结果图
图1 GC-MS分析嗜酸乳杆菌NCFM对照组和氯化锂
处理组2组样品的PCA得分图和PLS-DA结果图
Fig.1 The analysis results in GC-MS of the PCA and
PLS-DA between two groups of samples of Lactobacillus
acidophilus control and treated with lithium chloride
PLS-DA分析显示有组分类和成对判别,模型中的VIP值大于1的化合物表示对2组的分离具有显著贡献[18],在该PLS-DA模型中鉴别出13个投影值(VIP值)大于1的差异代谢物,如图2-A所示。结合VIP值、Fold Change和P值进行差异代谢物的筛选,为直观地表现出代谢物的差异情况,以及显示2组样品之间的总体模式特征,对差异代谢物绘制了火山图(图2-B)和聚类热图(图2-C)。由图2-A可知,乳酸的VIP值很高,表明乳酸含量在两组间差异显著(图2-A)。同时,多种有机酸、部分糖类物质的含量随表层蛋白的缺乏而表现出显著的变化。乳杆菌降解葡萄糖,发生糖酵解过程产生丙酮酸,乳酸脱氢酶会将丙酮酸还原为乳酸,乳酸作为乳杆菌糖代谢过程中的重要终产物[19],在去除表层蛋白后乳酸积累增加。这与苹果酸、烯醇式丙酮酸等物质表现出明显的上调趋势相一致,说明表层蛋白的缺乏可能使乳酸菌需要更加旺盛的糖代谢,以满足氨基酸合成及菌体自身生长代谢所需要的能量,此时氨基酸合成会竞争生长所需要的能量[17]。从火山图(图2-B)可以看出,GC-MS鉴定到129种已知的化合物,其中15种代谢物|log2FoldChange|>1,为显著差异代谢物。在聚类热图(图2-C)中,多种氨基酸、有机酸、糖有显著差异,这可能与氨基酸合成和能量代谢密切相关。
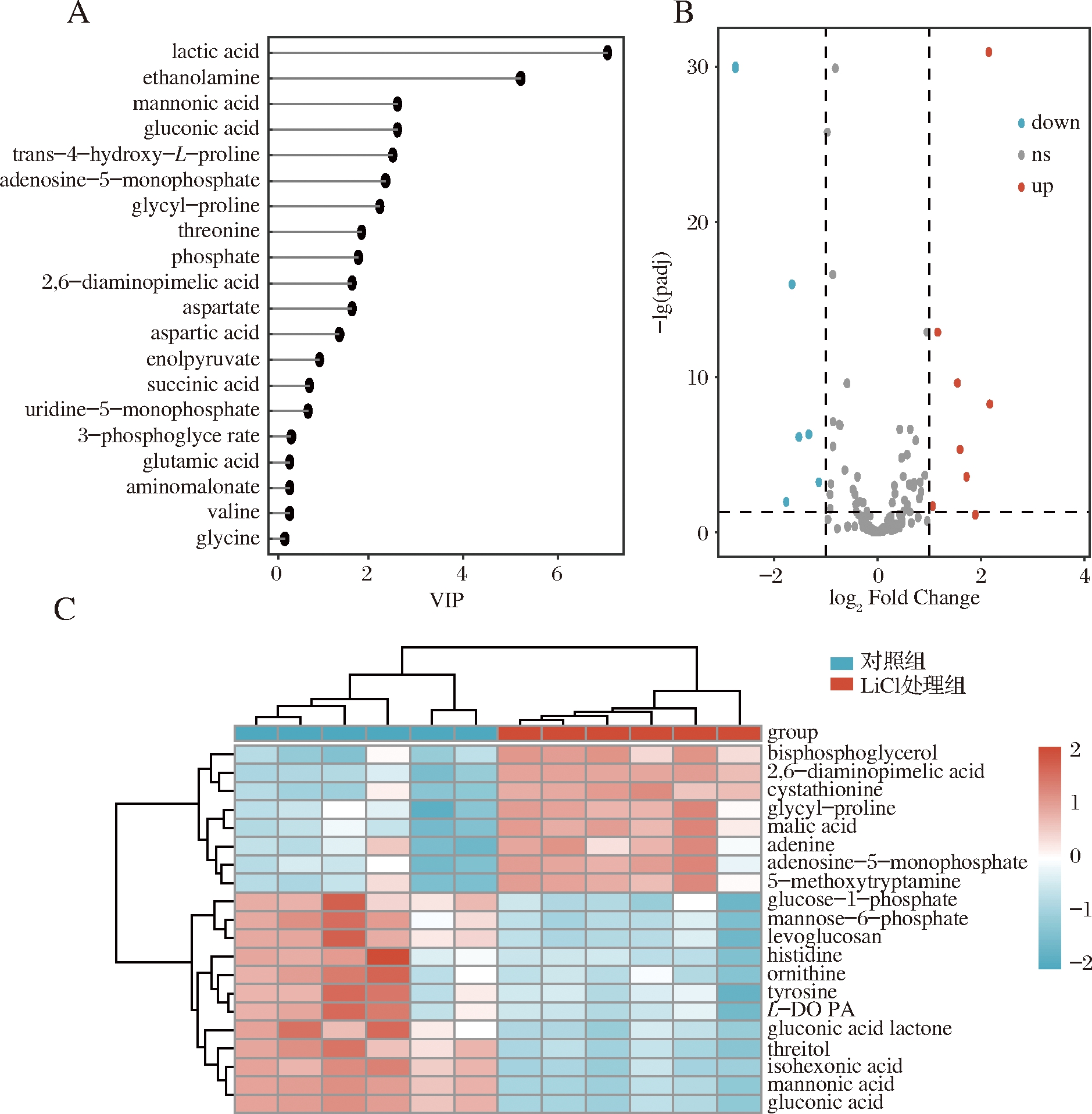
A-VIP得分图;B-差异代谢物火山图;C-差异代谢物热图
图2 嗜酸乳杆菌NCFM对照组和氯化锂处理组2组样品差
异代谢物,VIP得分图,差异代谢物火山图,差异代谢物热图
Fig.2 Different metabolites between two groups of Lactobacillus
acidophilus control and lithium chloride treatment, VIP score plot,
volcano of different metabolites, heatmap of different metabolites
乳杆菌的表层蛋白等电点偏碱性,其原因是带正电荷的氨基酸远高于带负电荷的氨基酸。同时表层蛋白中带羟基的氨基酸含量也比较高,占比为 23.0%~33.0%[20]。从聚类热图(图2-C)可以看出,其中的鸟氨酸、组氨酸、酪氨酸呈下调趋势,以其峰面积表示相对含量,对其进一步进行T检验,如图3-B~图3-D所示,分析2组样品这3种氨基酸含量具有显著差异。鸟氨酸为一种碱性氨基酸,为精氨酸分解而成的产物,而组氨酸为带正电的氨基酸,嗜酸乳杆菌fb116, 嗜酸乳杆菌fb214的表层蛋白分别含有3.99、4.82 mg/g的组氨酸[21]。酪氨酸是一种含有酚羟基的芳香族极性α-氨基酸,嗜酸乳杆菌fb116,嗜酸乳杆菌fb214分别含有4.26、6.12 mg/g的酪氨酸[21]。它们为组成乳杆菌表层蛋白的氨基酸,在5 mol/L的氯化锂溶液处理后含量降低,而重新合成氨基酸需要时间积累,故含量显著低于对照组。
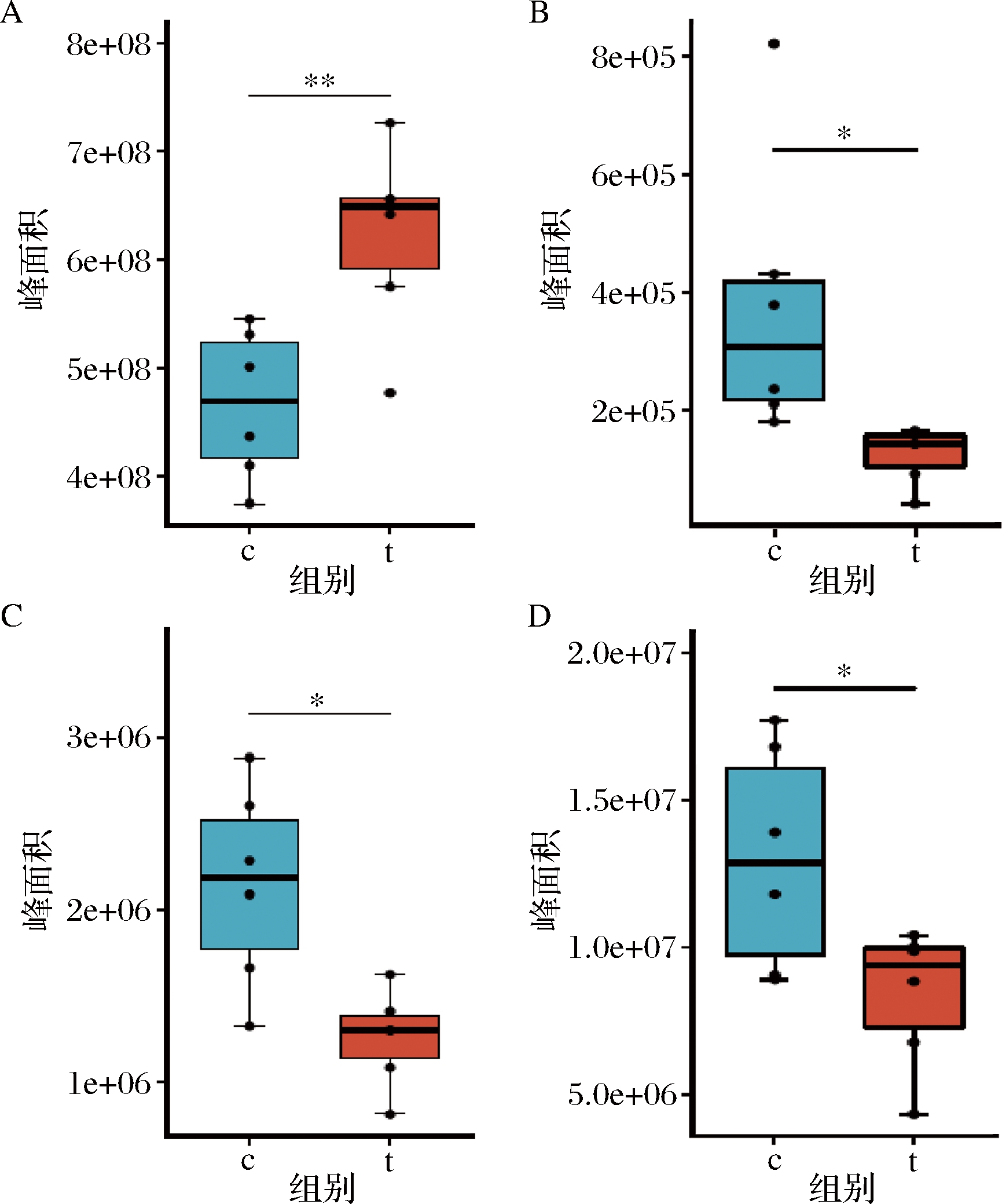
c-对照组;t-氯化锂处理组
A-乳酸;B-组氨酸;C-鸟氨酸;D-酪氨酸
图3 嗜酸乳杆菌NCFM对照组、氯化锂处理组
2组样品部分差异代谢物
Fig.3 Different metabolites between two groups of Lactobacillus
acidophilus control and lithium chloride treatment
对VIP>1或|log2FoldChange|>1的差异代谢物进行代谢通路富集分析,如图4所示,结果表明精氨酸和脯氨酸代谢、赖氨酸的生物合成、精氨酸生物合成、氨基糖和核苷酸糖代谢、丙氨酸,天冬氨酸和谷氨酸代谢这5条代谢通路得到了显著富集,其P<0.1,这表明去除表层蛋白后的一段时间内,氨基酸相关代谢十分活跃,这也暗示菌体的表层蛋白又在重新合成。这与先前文献报道的表层蛋白去除后过一段时间又可以在菌体上检测到表层蛋白的现象一致[22]。乳杆菌的表层蛋白总氨基酸含量约为36%,其中,酸性氨基酸含量约为15%,稍低于碱性氨基酸含量[20]。赖氨酸最为常见合成表层蛋白的碱性氨基酸,约占10%。组氨酸、精氨酸含量则相对较低。精氨酸和脯氨酸代谢、赖氨酸的生物合成、精氨酸生物合成与表层蛋白组成的氨基酸合成密切相关,同时,丙氨酸,天冬氨酸和谷氨酸代谢通路也被富集到了,这与文献报道表层蛋白中的酸性氨基酸包括丙氨酸,天冬氨酸和谷氨酸[23]相符合。
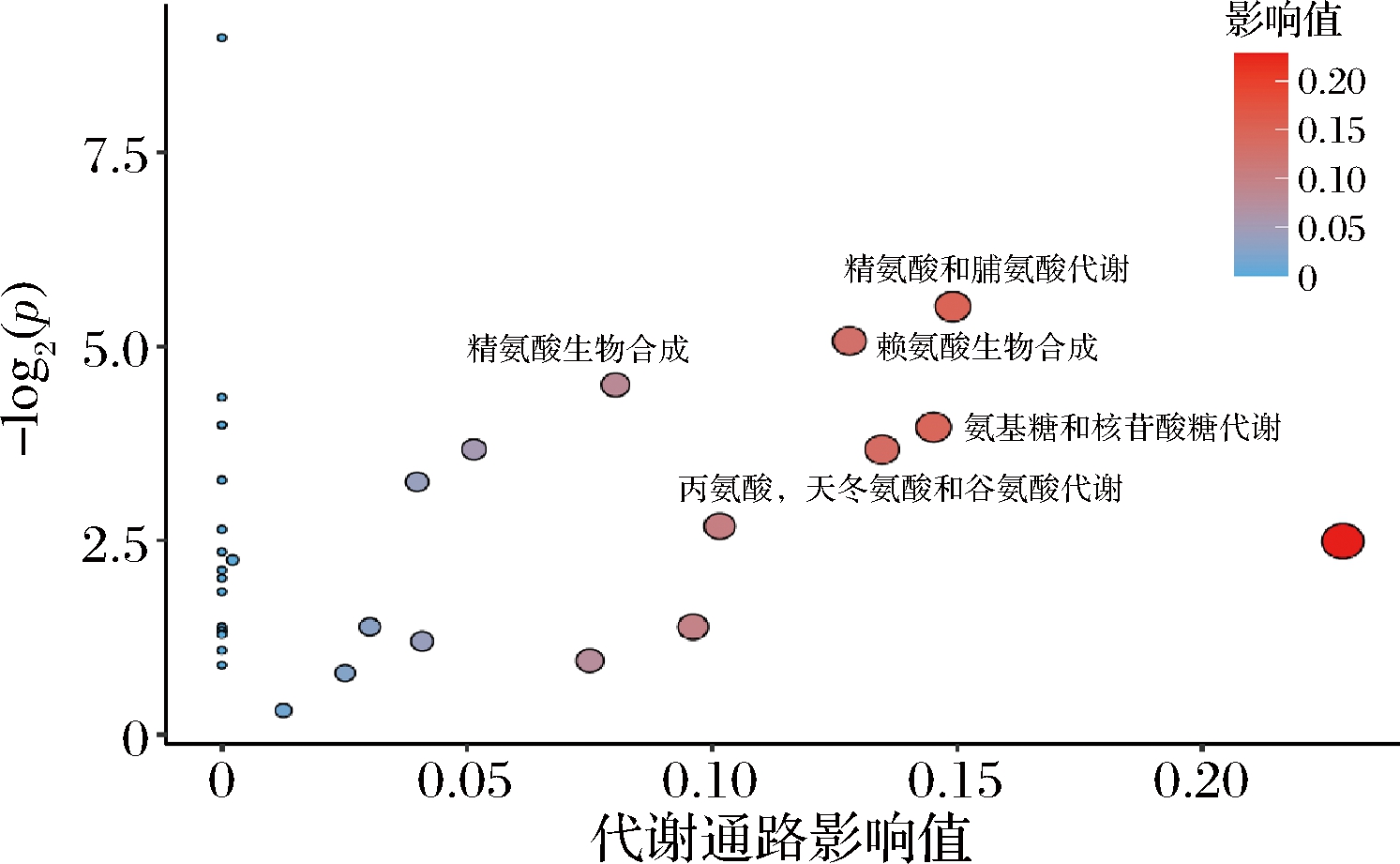
图4 两组样品差异代谢物代谢通路富集分析
Fig.4 Enrichment analysis of metabolic pathways of
different metabolites between two groups
本研究采用代谢组学分析技术研究表层蛋白对乳酸菌胞内代谢的影响,结果表明表层蛋白缺乏对乳酸菌胞内代谢轮廓有显著影响。表层蛋白的去除对乳酸菌氨基酸代谢有显著影响,胞内氨基酸浓度的低水平使得菌株需要更多的能量以恢复表层蛋白,并有可能通过竞争能量和中间产物使三羧酸循环的效率下降,对乳酸菌的生长代谢产生影响,本研究结果为表层蛋白在菌体生长代谢过程中的作用提供理论指导。对于非靶向代谢组学鉴定到的化合物,具体功能和含量后续仍需通过化合物鉴定和HPLC等设备进行更加精确的定量。
[1] 王慧芳.乳杆菌S-层蛋白的肠道免疫调节功能及作用机制研究[D].无锡:江南大学, 2020.
WANG H F.Study on the intestinal immunomodulatory function and mechanism of Lactobacillus surface-layer proteins[D].Wuxi:Jiangnan University, 2020.
[2] WANG H F, ZHANG L, LI Q P, et al.Surface-layer protein produced by Lactobacillus crispatus JCM 2009 ameliorates lipopolysaccharide-induced inflammation through autophagy cross-talk with the NF-κ B signaling pathway[J].International Journal of Biological Macromolecules, 2021, 166:633-640.
[3] ZHANG Y C, XIANG X L, LU Q H, et al.Adhesions of extracellular surface-layer associated proteins in Lactobacillus M5-L and Q8-L[J].Journal of Dairy Science, 2016, 99(2):1 011-1 018.
[4] ABRAMOV V M, KOSAREV I V, PRIPUTENEVICH T V, et al.S-layer protein 2 of Lactobacillus crispatus 2029, its structural and immunomodulatory characteristics and roles in protective potential of the whole bacteria against foodborne pathogens[J].International Journal of Biological Macromolecules, 2020, 150:400-412.
[5] 牛钰涵.表层蛋白对乳杆菌益生性质的影响及其抑菌功能[D]:无锡:江南大学, 2019.
NIU Y H.Effect of surface layer proteins from Lactobacillus on the strains′ probiotic properties and their antibacterial function[D].Wuxi:Jiangnan University, 2019.
[6] PALOMINO M M, WAECHNER P M, MARTIN J F, et al.Influence of osmotic stress on the profile and gene expression of surface layer proteins in Lactobacillus acidophilus ATCC 4356[J].Applied Microbiology and Biotechnology, 2016, 100(19):8 475-8 484.
[7] KLOTZ C, GOH Y J, O'FLAHERTY S, et al.Deletion of S-layer associated Ig-like domain protein disrupts the Lactobacillus acidophilus cell surface[J].Frontiers in Microbiology, 2020, 11(14):1-14.
[8] PATTI G J, YANES O, SIUZDAK G.Metabolomics:The apogee of the omics trilogy[J].Nature Reviews Molecular Cell Biology, 2012, 13(4):263-269.
[9] JOHNSON B, SELLE K, O'FLAHERTY S, et al.Identification of extracellular surface-layer associated proteins in Lactobacillus acidophilus NCFM[J].Microbiology-Sgm, 2013, 159:2 269-2 282.
[10] CHEN X Y, XU J J, SHUAI J B, et al.The S-layer proteins of Lactobacillus crispatus strain ZJ001 is responsible for competitive exclusion against Escherichia coli O157:H7 and Salmonella typhimurium[J].International Journal of Food Microbiology, 2007, 115(3):307-312.
[11] LU H Q, CHEN H Q, TANG X, et al.Evaluation of metabolome sample preparation and extraction methodologies for oleaginous filamentous fungi Mortierella alpina[J].Metabolomics, 2019, 15(4):50.
[12] DUNN W B, BROADHURST D, BEGLEY P, et al.Procedures for large-scale metabolic profiling of serum and plasma using gas chromatography and liquid chromatography coupled to mass spectrometry[J].Nature Protocols, 2011, 6(7):1 060-1 083.
[13] KIND T, WOHLGEMUTH G, LEE D Y, et al.FiehnLib:Mass spectral and retention index libraries for metabolomics based on quadrupole and time-of-flight gas chromatography/mass spectrometry[J].Analytical Chemistry, 2009, 81(24):10 038-10 048.
[14] CHONG J, SOUFAN O, LI C, et al.MetaboAnalyst 4.0:Towards more transparent and integrative metabolomics analysis[J].Nucleic Acids Research, 2018, 46(1):486-494.
[15] LORTAL S, VANHEIJENOORT J, GRUBER K, et al.S-Layer of Lactobacillus-Helveticus ATCC-12046 - isolation, chemical characterization and re-formation after extraction with lithium-chloride[J].Journal of General Microbiology, 1992, 138:611-618.
[16] HYMES J P, JOHNSON B R, BARRANGOU R, et al.Functional analysis of an S-layer-associated fibronectin-binding protein in Lactobacillus acidophilus NCFM[J].Applied and Environmental Microbiology, 2016, 82(9):2 676-2 685.
[17] 程新, 赵延胜, 董英, 等.Mn2+对植物乳杆菌影响的代谢组学分析[J].中国食品学报, 2019, 19(2):258-265.
CHENG X, ZHAO Y S, DONG Y, et al.Metabolomics analysis of Mn2+ effect on the metabolic pathways in Lactobacillus plantarum[J].Journal of Chinese Institute of Food Science and Technology, 2019, 19(2):258-265.
[18] BOULESTEIX A L, STRIMMER K.Partial least squares:A versatile tool for the analysis of high-dimensional genomic data[J].Briefings in Bioinformatics, 2007, 8(1):32-44.
[19] 石轩峰.植物乳杆菌乳酸脱氢酶的发酵优化、制备及酶促反应研究[D]:无锡:江南大学, 2005.
SHI X F.Studies on optimization, preparation and enzyme catalytic reaction of lactate dehydrogenase by Lactobacillus plantarum[D].Wuxi:Jiangnan University, 2005.
[20] 孟珺, 王艳阳, 康媛媛, 等.乳杆菌表层蛋白理化性质与生物功能的研究进展[J].河南工业大学学报(自然科学版), 2020, 41(4):129-138.
MENG J, WANG Y Y, KANG Y Y, et al.Advances in research on physicochemical properties and biological functions of Lactobacillus surface layer proteins[J].Journal of Henan University of Technology(Natural Science Edition), 2020, 41(4):129-138.
[21] MENG J, ZHU X, GAO S M, et al.Characterization of surface layer proteins and its role in probiotic properties of three Lactobacillus strains[J].International Journal of Biological Macromolecules, 2014, 65:110-114.
[22] 胡斌, 田丰伟, 赵建新, 等.嗜酸乳杆菌NCFM表层蛋白的提取及其与菌体酸和胆盐耐受及粘附性能的关系[J].食品工业科技, 2014, 35(5):118-121.
HU B, TIAN F W, ZHAO J X, et al.Extraction of surface layer protein from Lactobacillus acidophilus NCFM and the relationship among SLP and strain acid,bile resistance and cell adhesion[J].Science and Technology of Food Industry, 2014, 35(5):118-121.
[23] 孟珺.乳杆菌表层蛋白的理化性质和生物学功能研究[D]:无锡:江南大学, 2016.
MENG J.Study of the physicochemical properties and biological functions of Lactobacillus surface layer proteins[D].Wuxi:Jiangnan University, 2016.