奶酪作为发酵型乳制品蕴藏丰富具有益生特性的乳酸菌。据报道[1]微生物发酵法能够降解食物中的过敏原,并且利用不同的菌种(乳酸菌、酵母菌、芽孢杆菌)对食物蛋白进行发酵,其过敏原降解的程度也有所不同。FRIAS等[2]利用植物乳杆菌发酵豆粕48 h,SDS-PAGE结果显示豆粕中的致敏蛋白被全部降解,ELISA结果显示免疫球蛋白E结合能力降低了96%以上。肖叶等[3]利用植物乳杆菌水解对虾中的原肌球蛋白得到了类似的结果。此外,酪蛋白或脱脂牛乳经乳酸菌发酵后能够使β-酪蛋白[4]、β-乳球蛋白[5]显著降解,且免疫活性下降。根据上述应用研究发现乳酸菌能够降低牛乳,大豆,虾类过敏原的免疫活性,具有效果显著、安全性高、特异性强和应用价值高等优势,但利用乳酸菌消减鱼类过敏原鲜有研究。
鱼类是引起食物过敏的常见水产品之一,其中淡水鱼相比海水鱼更易致敏[6]。据显示,人们对鱼类产生过敏反应主要是由小清蛋白(parvalbumin,PV)引起的[7]。其分子质量约为10~14 kDa,具有极高的热稳定性。KUBOTA等[8]研究表明使用140 ℃ 以上的高温处理鱼组织,才能使PV的免疫活性下降。
因此,本研究通过分离奶酪中的乳酸菌,利用脱脂牛乳平板透明圈法作为产蛋白酶乳酸菌的初步筛选标准,并对菌株进行16S rDNA测序鉴定。通过提取草鱼中的PV与筛选的乳酸菌进行孵育,评价孵育过程中PV含量和免疫活性的变化,筛选有效降低PV免疫活性的乳酸菌,以期为乳酸菌发酵鱼肉开发低致敏性鱼肉制品提供参考。
奶酪,新疆乌鲁木齐王家梁农贸市场;新鲜草鱼,上海市浦东新区芦潮港。
MRS培养基,青岛海博生物有限公司;预染和非预染蛋白质标记物、BCA蛋白定量试剂盒,北京天根生化科技有限公司;小清蛋白单克隆抗体EG8,实验室自制;R-250考马斯亮蓝染色液、2×上样缓冲液、十二烷基硫酸钠、Tris分析纯,上海生工生物工程有限公司;聚偏二氟乙烯膜(polyvinylidene fluoride,PVDF膜)(0.45 μm),美国Millipore公司;脱脂奶粉,美国BD公司;DAB显色液(D0426-50SET),美国Sigma公司。
GHP-9080隔水式恒温培养箱,上海一恒科学仪器有限公司;YXQ-LS-100S11立式压力蒸气灭菌器,上海博讯实业有限公司;SW-CJ-ZF可调式垂直单向洁净工作台,上海天恒医疗器械有限公司;JA1003电子天平,常州市宏恒电子仪器厂;ZQWY-200振荡培养箱,上海知楚仪器有限公司;高速冷冻离心机,日本日立公司;电泳仪,美国伯乐公司;TS-8脱色摇床,江苏海门其林贝尔仪器制造有限公司;Synergy2多功能酶标仪,德国 Hermle 公司。
1.3.1 乳酸菌的分离筛选
无菌条件称取25.0 g奶酪放入含225 mL生理盐水的无菌均质袋中,用拍击式均质器制成样品匀液,进行10倍梯度稀释并涂布MRS固体培养基,37 ℃厌氧培养48 h[9]。观察菌落生长情况(颜色、形状、隆起、边缘等),挑取特征菌落于MRS平板划线获得单菌落纯培养物。通过过氧化氢酶反应、生理生化鉴定筛选目的菌。
1.3.2 产蛋白酶乳酸菌的筛选
参照胡玲萍等[10]的方法略作修改,将菌株接种于MRS肉汤培养基置于37 ℃培养24 h,菌液离心(8 000 r/min,10 min)取50 μL上清液添加至脱脂牛乳琼脂平板孔内,37 ℃放置过夜,观察平板上产生的透明圈并测量记录。每个菌株进行3个平行实验。
1.3.3 16S rDNA基因的PCR扩增
参考郭艳荣等[11]的方法,将MRS培养18 h菌液离心收集菌体(8 000 r/min,15 min),用DNA快速抽提试剂盒提取DNA,使用16S rDNA 通用引物进行PCR扩增。
1.3.4 PCR扩增结果和系统发育树的构建
将PCR产物送至上海生工生物工程有限公司进行测序。采用BioEdit 7.0软件拼接序列,于NCBI数据库的GenBank中与已知菌株的16S rDNA基因序列进行比对[11-12]寻找同源性最高的菌种。使用MEGA 7.0软件中的邻接法对8株乳酸菌作系统发育进化树[13]。
1.3.5 草鱼PV的提取与富集
参照ZHANG等[14]的方法略作修改,新鲜草鱼取白色肌肉200 g,加入5倍体积的抽提液进行匀浆,离心(4 ℃,10 000 r/min,20 min)取上清液加入三氯乙酸(trichloroacetic acid solution,TCA)溶液,冰浴搅拌10 min,调节pH至5.2,继续搅拌1 h。离心取上清液再次加入TCA溶液,搅拌10 min,离心(4 ℃,12 000 r/min,20 min)收集沉淀并加入少量去离子水溶解,调节pH至中性。所得溶液透析24 h后进行冷冻干燥。采用SDS-PAGE和蛋白免疫印迹(Western blot)对提取的PV进行验证。
1.3.5.1 SDS-PAGE
参照ZHANG等[14]的方法,浓缩胶和分离胶浓度分别为4%和14%,样品上样量为8 μL。
1.3.5.2 Western blot
参照SONG等[15]的方法略作修改。用半干式碳板转印仪将蛋白条带转印至PVDF膜上,条件为恒流180 mA,20 min。用封闭液覆盖于PVDF膜,室温封闭1 h。用含有0.05% 吐温-20磷酸缓冲液(phosphate buffered saline-tween 20,PBST)洗涤。单克隆抗体EG8(体积比1∶10 000)作为一抗,室温孵育1 h,PBST洗涤。HRP-羊抗小鼠IgG(体积比1∶2 500)作为二抗,室温孵育1 h,PBST洗涤。DAB显色液显色后拍照保存。
1.3.6 乳酸菌和PV溶液的孵育反应
参照PESCUMA等[16]的方法略作修改,将8株乳酸菌分别接入MRS液体培养基活化2次,菌液10 000 r/min离心,用PBS洗涤菌体3次,将菌体悬浮于PBS,浓度为1×108CFU/mL。按体积比1∶1加入2 mg/mL PV蛋白溶液于恒温摇床37 ℃,200 r/min孵育24 h。对照为未加菌体的PV溶液。孵育结束后离心(8 000 r/min,10 min)收集上清液,采用SDS-PAGE分析PV含量的变化。
1.3.7 孵育时间对PV含量和免疫活性的影响
菌株活化及处理参照1.3.6方法进行。孵育时间梯度设为18、20、22、24、26、36 h。孵育后离心收集上清液用SDS-PAGE分析PV含量的变化,采用竞争性ELISA评价PV免疫活性的变化。
1.3.8 竞争性ELISA
参照丁婕等[17]的方法。PV用碳酸盐缓冲液(pH 9.6)稀释至5 μg/mL,4 ℃包被过夜,PBST洗涤3次,每次15 s。加入封闭液于37 ℃封闭2 h,PBST洗涤。每孔加入预先与EG8孵育1 h的样品100 μL,37 ℃孵育1 h,PBST洗涤。加入HRP-羊抗小鼠免疫球蛋白G(体积比1∶2 500)100 μL,37 ℃ 孵育 1 h,洗涤后加入邻苯二胺底物显色液100 μL,显色后加入2 mol/L硫酸终止反应,用酶标仪测定450 nm处的吸光度值(OD值)。以碳酸盐缓冲液(pH 9.6)作为阴性对照,以抗体稀释液稀释的EG8作为阳性对照,每个样品3个平行。样品中PV免疫活性的消减率的计算如公式(1)和公式(2)所示:
抑制率![]()
(1)
消减率![]()
(2)
式中:阳性OD450为阳性对照孔的OD值;样品OD450为样品孔的OD值;阴性OD450为阴性对照孔的 OD值。
实验结果采用平均值±标准偏差表示,由SPSS Statistics 19.0软件进行显著性分析,由Origin 2018 64 Bit软件进行制图。
通过形态学观察和部分生理生化试验,从奶酪中分离筛选出45株分离株,进一步利用透明圈法筛选得到8株符合乳酸菌特征的产蛋白酶菌株,编号标记为A-5~B-19,其菌落形态特征及部分生理生化特性如表1所示,产透明圈大小见表2。
表1 菌株的形态特征及生理生化特性
Table 1 Morphological and biochemical characteristics of the strain

菌株编号菌落形态革兰氏染色菌体形态过氧化氢酶乳糖棉子糖麦芽糖硫化氢葡萄糖产酸葡萄糖产气A-5圆形隆起,乳白色,边缘整齐,表面光滑湿润G+杆状-+++-+-A-9微小圆形隆起,白色,边缘整齐,表面光滑湿润G+杆状-+++-+-A-16圆形微隆起,白色,边缘不整齐,表面光滑较湿润G+杆状-+-+-+-B-2圆形扁平,乳白色,边缘整齐,表面光滑G+杆状-+-+-+-B-6圆形隆起,灰白半透明,边缘不整齐,表面光滑G+球状-+++-+-B-14微小圆形微隆起,白色,边缘整齐,表面较光滑G+杆状-+++-+-B-17圆形隆起,乳白色,边缘整齐,表面光滑湿润G+杆状-+++-+-B-19圆形隆起,白色半透明,边缘整齐,表面光滑湿润G+球状-+++-+-
注:“+”为阳性反应;“-”为阴性反应;G+为革兰氏阳性菌
表2 菌株产透明圈大小情况
Table 2 The size of the clear zone produced by the strains

菌株编号透明圈大小/mmA-56.50±0.87aA-91.17±0.29dA-163.67±0.58cB-25.83±0.28abB-65.50±0.50abB-143.83±0.76cB-175.16±1.04bB-194.52±1.68bc
注:组间不同小写字母代表存在显著性差异(P <0.05)
提取8株分离株的DNA并进行PCR扩增,经1.0%琼脂糖凝胶电泳检测如图1所示,8株待测菌株在约1 500 bp处条带清晰明亮,无杂带,表明PCR扩增成功,能满足后续菌株16S rDNA序列的测定。
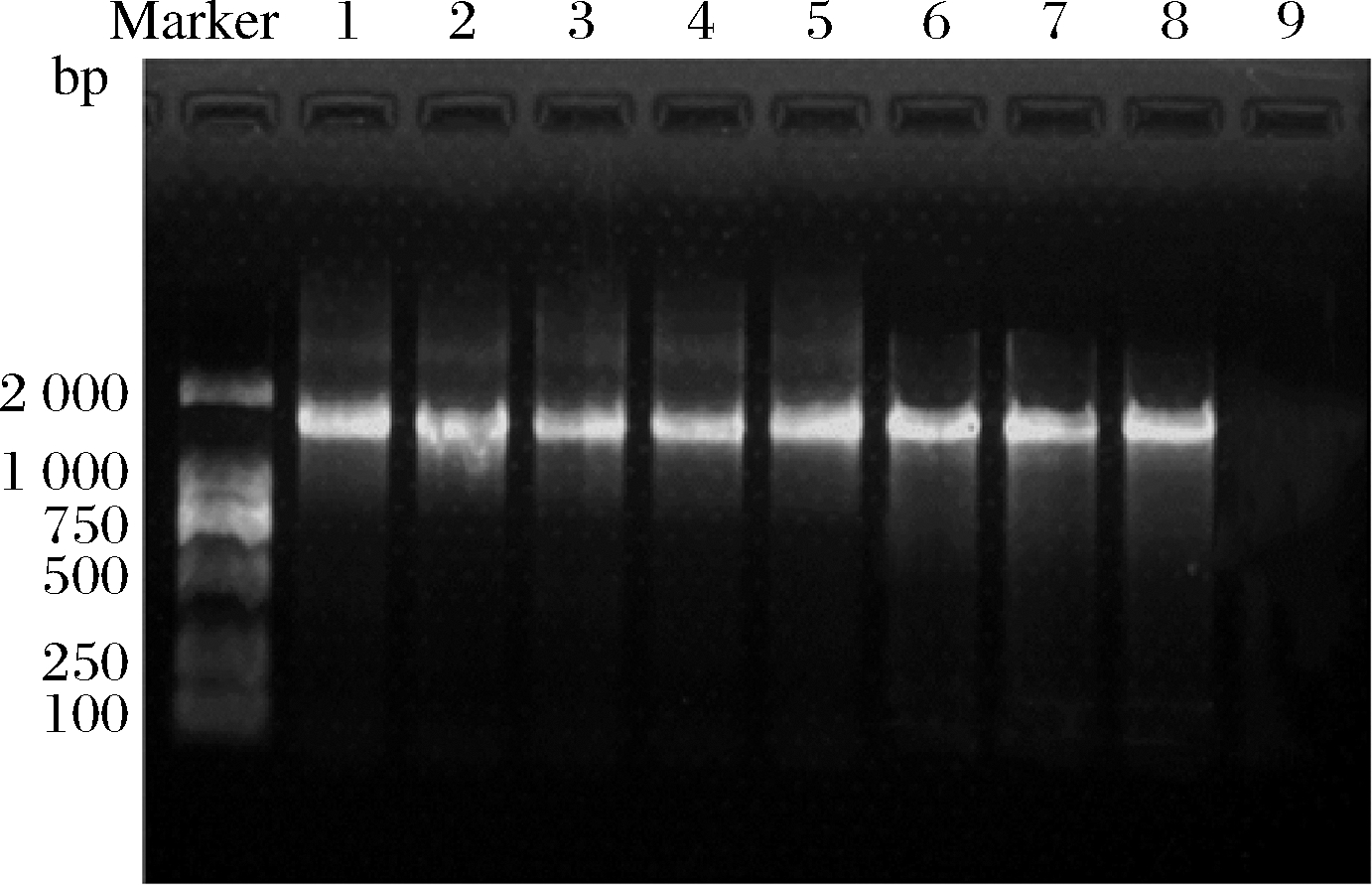
1~8-A-5~B-19;9-阴性对照
图1 16S rDNA PCR 扩增后的琼脂糖凝胶电泳
Fig.1 Agarose gel electrophoresis of 16S rDNA amplification products
测序结果通过NCBI中的BLAST进行序列相似性比对,将8株分离株鉴定为乳酸菌的2个属,3个种。利用MEGA 7.0软件进行系统发育关系研究和系统进化树构建,结果见图2。Lactobacillus plantarum、Lactobacillus brevis、Lactobacillus casei、Enterococcus faecium各自聚为一类。其中A-5、A-9、B-14和B-17与Lactobacillus plantarum IDCC 3502,Lactobacillus plantarum MG5211在同一分支,且同源性为100%,故将其鉴定为Lactobacillus plantarum;B-2和A-16与Lactobacillus casei M8,Lactobacillus casei MT.ZH493聚为一类,且序列同源性达100%,因此将其鉴定为Lactobacillus casei;B-6,B-19和Enterococcus faecium IDCC 2103同源性达到100%,故鉴定其为Enterococcus faecium。
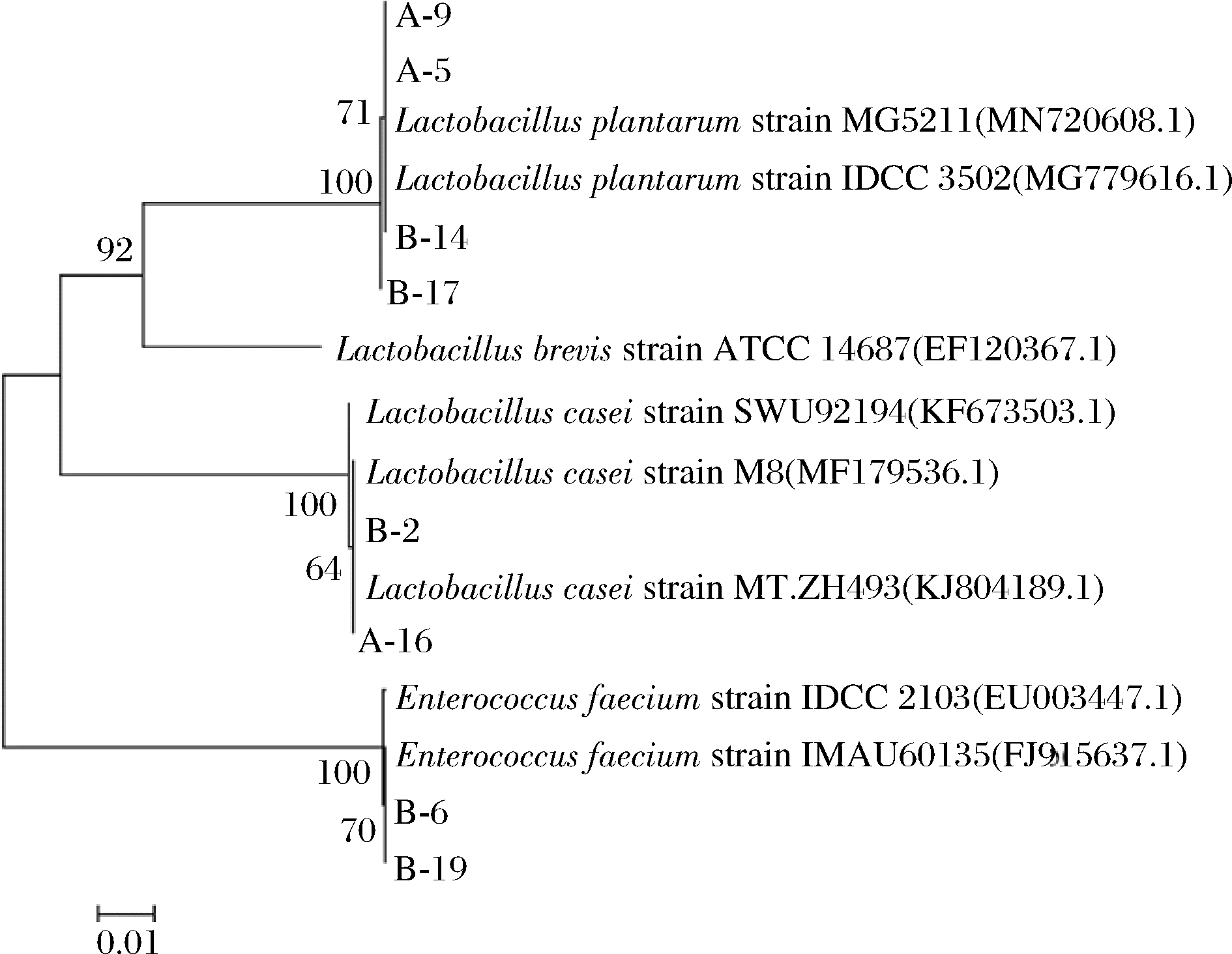
图2 菌株的系统发育树
Fig.2 Phylogenetic tree of strains
草鱼肌肉提取液和TCA处理样品的电泳分析结果如图3所示。SDS-PAGE的14.4 kDa附近有2个蛋白质条带(图3-a),其分子质量与报道的PV分子质量接近[7,18]。此外,免疫印迹结果显示11 kDa附近的条带与EG8单抗有特异性反应,因EG8单抗对鱼类过敏原PV有特异性反应[19],故该条带被鉴定为草鱼的PV。对比TCA处理前后的电泳条带,发现第2次TCA处理后的样品条带(图3-a)显著变粗,表明2次TCA处理可以有效富集草鱼中的PV,且含量较多,纯度较高。
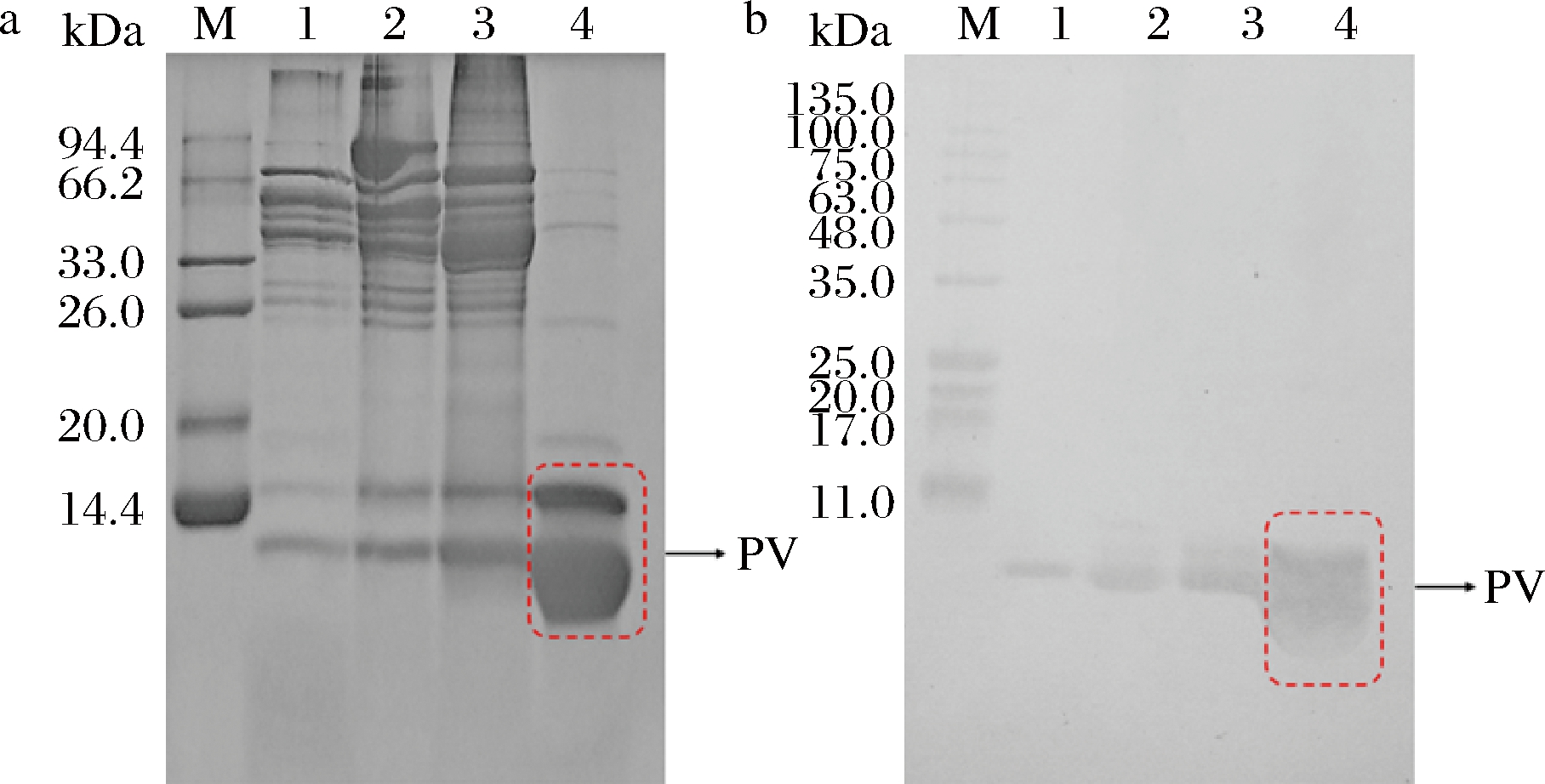
M-蛋白Marker;1-粗蛋白上清液;2-粗蛋白沉淀;
3-1次TCA沉淀;4-2次TCA沉淀透析后样品
a-SDS-PAGE;b-Western blot
图3 草鱼PV提取过程样品的SDS-PAGE和Western blot
Fig.3 SDS-PAGE and Western blot of PV extract samples of
Ctenopharyngodon idella
图4中1~8依次为菌株A-5~B-19与PV孵育24 h的样品条带。由图4可知,Lactobacillus casei B-2和Lactobacillus plantarum B-17孵育PV 24 h后,PV条带发生了明显的变化。经Lactobacillus casei B-2孵育后位于14.4 kDa左右的条带消失,而在14.4 kDa下方出现了新的条带,颜色变浅且出现多个条带,最下方的条带与对照样品相比明显变细,表明PV经这2株乳酸菌处理后发生了部分降解。经菌株Lactobacillus plantarum B-17孵育后的PV条带颜色显著变浅,说明PV含量明显减少。其他菌株对PV条带无较大影响,因此挑选Lactobacillus casei B-2和Lactobacillus plantarum B-17进一步研究孵育时间对PV含量和免疫活性的影响。

M-蛋白Marker;C-PV对照;1~8-菌株A-5~B-19孵育PV样品
图4 PV孵育24 h的 SDS-PAGE
Fig.4 SDS-PAGE of PV incubation for 24 h
Lactobacillus casei B-2和Lactobacillus plantarum B-17对PV溶液不同孵育时间的电泳分析结果如图5所示,这2株菌孵育36 h的PV条带相对于对照样品几乎不可见,表明此时PV蛋白质基本被全部降解了。而Lactobacillus casei B-2与PV孵育的18~24 h间PV含量无明显变化,26 h时14.4 kDa上方的条带消失,而在下方出现新条带,位于12 kDa左右的条带明显变窄,颜色变浅,可见PV出现了部分降解。

M-蛋白Marker;C-PV对照
a-B-2;b-B-17
图5 孵育PV后的含量变化
Fig.5 Changes in the content of PV after incubation
Lactobacillus casei B-2和Lactobacillus plantarum B-17孵育PV后,通过计算消减率表示不同时间段PV免疫活性的变化情况。消减率越高说明对PV免疫活性的消减效果越好,反之亦然。结果如图6所示,随着反应时间的延长,消减率逐步增加。反应36 h 时,Lactobacillus casei B-2和Lactobacillus plantarum B-17对PV免疫活性消减率最高,分别为96.5% 和84.8%。
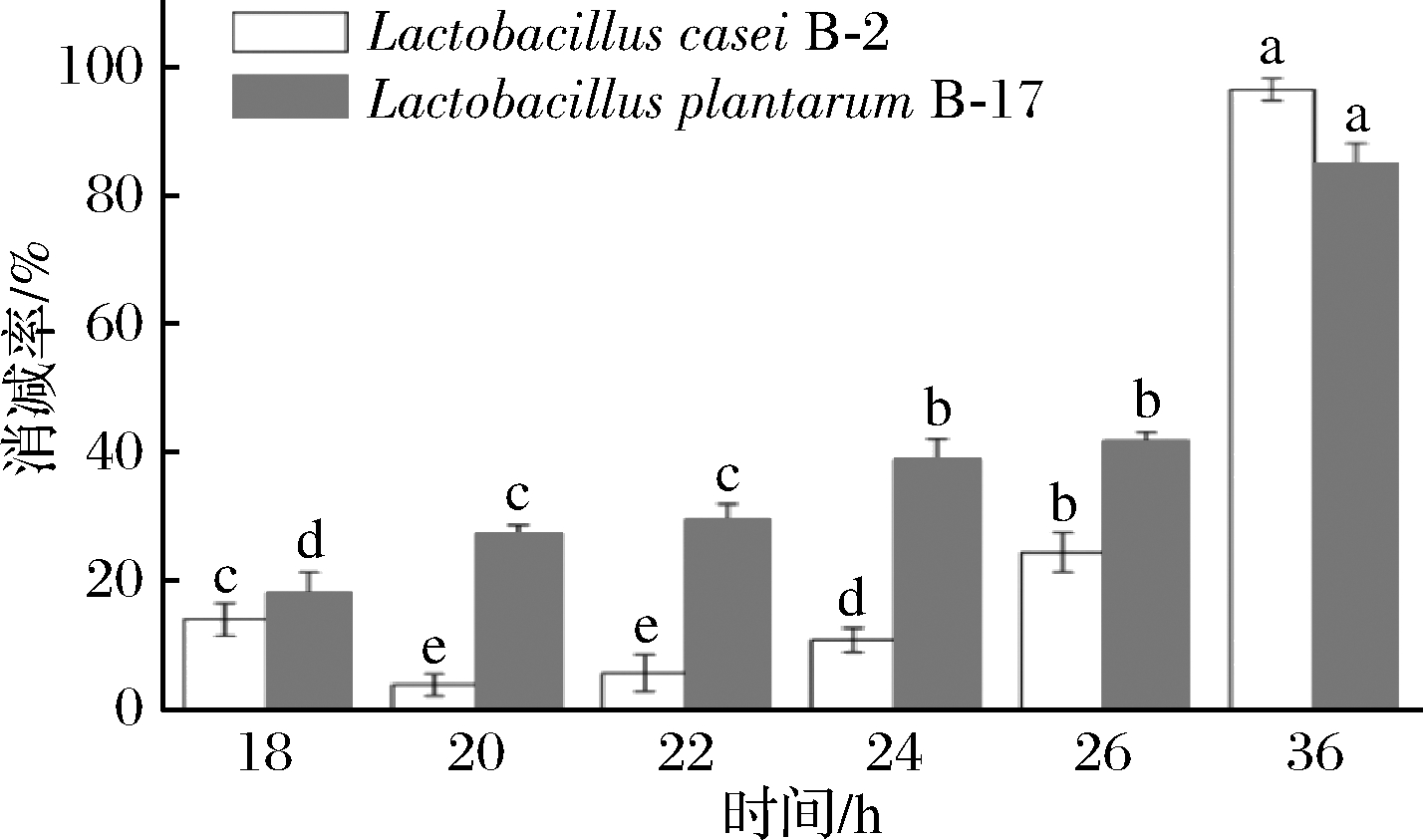
图6 B-2、B-17孵育PV的免疫活性变化
Fig.6 changes of PV Antigenicity treated by
B-2 and B-17 strains
注:组间不同小写字母代表存在显著性差异(P<0.05)
据报道乳酸菌有1个高效的蛋白水解系统,由1个细胞膜蛋白酶(启动蛋白质降解)、1个转运系统和几个胞内肽酶组成[20-21],且不同乳酸菌菌株对不同种类的过敏原蛋白具有不同的水解效果[22]。本研究从奶酪中筛选获得8株产蛋白酶的乳酸菌,其产透明圈大小显示了一定的差异性,说明具有不同的分解牛乳蛋白的能力,透明圈越大,分解牛乳蛋白的能力则越强,产生的蛋白酶活性越高。其中Lactobacillus casei B-2和Lactobacillus plantarum B-17产透明圈平均值为5.83、5.16 mm,与PV溶液孵育36 h使PV显著降解,且免疫活性分别降低96.5% 和84.8%。虽然Enterococcus faecalis B-6, Lactobacillus plantarum A-5 显示了相似甚至更好的产蛋白酶能力,但对PV几乎无影响。相关研究[23]发现L.delbrueckii subsp.bulgaricus 92059水解β-酪蛋白24 h时蛋白完全降解,而L.helveticus 92201对β-酪蛋白的水解效果并不明显。说明不同的乳酸菌菌株对致敏蛋白的降解效果不同,在本研究中也得到了类似的结果。LAW等[24]研究指出乳酸菌产生的蛋白酶将乳蛋白裂解成多肽,多肽酶将多肽裂解成更小的多肽和氨基酸,从而抗原表位可能被裂解。因此,我们推测孵育过程中PV发生降解是由于Lactobacillus casei B-2和Lactobacillus plantarum B-17产生蛋白酶将PV水解为多肽,进而引起PV免疫活性下降。
目前,乳酸菌发酵法降解食物中的过敏原多研究报道于牛乳和大豆制品,在降解过敏原的同时还能产生风味物质,赢得广大消费者的青睐。因此,利用乳酸菌发酵鱼肉制品具有良好的应用前景,以期在降解过敏原的同时提升产品风味和口感。本研究筛选到的Lactobacillus casei B-2和Lactobacillus plantarum B-17 能够显著降低PV的免疫活性,从而显示出了降低PV致敏性的潜力,今后可进一步探究不同发酵条件对鱼肉品质和风味的影响,将乳酸菌发酵应用于低致敏性鱼类制品的开发。
[1] 伍恒, 钱和, 汪何雅, 等.发酵法对降低食品过敏原的作用[J].食品工业科技, 2011, 32(8):453-457.
WU H, QIAN H, WANG H Y, et al.A summary on removing the allergen from food by fermentation[J].Science and Technology of Food Industry, 2011, 32(8):453-457.
[2] FRIAS J, SONG Y S, MART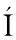 NEZ-VILLALUENGA C, et al.Immunoreactivity and amino acid content of fermented soybean products[J].Journal of Agricultural & Food Chemistry, 2008, 56(1):99-105.
NEZ-VILLALUENGA C, et al.Immunoreactivity and amino acid content of fermented soybean products[J].Journal of Agricultural & Food Chemistry, 2008, 56(1):99-105.
[3] 肖叶, 叶精勤, 李晓晨, 等.植物乳杆菌(Lactobacillus plantarum)对过敏原原肌球蛋白免疫活性的消减作用[J].食品科学, 2021,42(18):95-102.
XIAO Y, YE J Q, LI X C, et al.Study on the reduction effect of Lactobacillus plantarum on the immunological activity of tropomyosin[J].Food Science, 2021,42(18):95-102.
[4] TZVETKOVA I, DALGALARRONDO M, DANOVA S, et al.Hydrolysis of major dairy proteins by lactic acid bacteria from Bulgarian yogurts[J].Journal of Food Biochemistry, 2007, 31(5):680-702.
[5] KLEBER N, WEYRICH U, HINRICHS J.Screening for lactic acid bacteria with potential to reduce antigenic response of β-lactoglobulin in bovine skim milk and sweet whey[J].Innovative Food Science&Emerging Technologies, 2006, 7(3):233-238.
[6] 杨华, 刘卫兵, 伯慧敏.慢性荨麻疹370例体外变应原检测分析[J].中国医学文摘(皮肤科学), 2009, 26(5):273-275.
YANG H, LIU W B, BO H M.Investigation of allergens in 370 patients with chronic urticaria[J].China Medical Abstract of Dermatology, 2009, 26(5):273-275.
[7] KOBAYASHI Y, YANG T, YU C T, et al.Quantification of major allergen parvalbumin in 22 species of fish by SDS-PAGE[J].Food Chemistry, 2016, 194:345-353.
[8] KUBOTA H, KOBAYASHI A, KOBAYASHI Y, et al.Reduction in IgE reactivity of Pacific mackerel parvalbumin by heat treatment[J].Food Chemistry, 2016, 206:78-84.
[9] 国家卫生和计划生育委员会, 国家食品药品监督管理总局.GB 4789.35—2016食品微生物学检验 乳酸菌检验[S].北京:中国标准出版社, 2017.
National Health and Family Planning Commission, China Food and Drug Administration.GB 4789.35—2016 Food microbiological inspection lactic acid bacteria test[S].Beijing:Standards Press of China, 2017.
[10] 胡玲萍, 裘迪红, 王鑫钰.泡菜中高产蛋白酶乳酸菌的筛选鉴定及其酶学特性初步研究[J].食品与发酵工业, 2015, 41(6):35-40.
HU L P, QIU D H, WANG X Y.Screening and identification of lactic acid bacteria with high productivity of protease in pickled vegetable and studies on the enzymology characteristics of the protease[J].Food and Fermentation Industries, 2015, 41(6):35-40.
[11] 郭艳荣, 党娜, 苏馨, 等.埃塞俄比亚发酵食品中乳酸菌的分离鉴定[J].中国乳品工业, 2020, 48(5):15-19.
GUO Y R, DANG N, SU X, et al.Isolation and identification of lactic acid bacteria from fermented foods in Ethiopian[J].China Dairy Industry, 2020, 48(5):15-19.
[12] 呼斯楞, 刘红新, 于洁, 等.内蒙古呼伦贝尔地区传统发酵乳中乳酸菌的多样性分析[J].微生物学通报, 2016, 43(5):984-990.
HU S L, LIU H X, YU J, et al.Diversity of lactic acid bacteria associated with traditional fermented milk from Hulun Buir in Inner Mongolia[J].Microbiology, 2016, 43(5):984-990.
[13] 周滟晴, 刘婷, 周婉婷, 等.高产酸果醋醋酸菌的筛选鉴定及其耐醇和耐温性探究[J].食品与发酵工业, 2021,47(10):72-78.
ZHOU Y Q, LIU T, ZHOU W T, et al.Isolation and identification of an acetic acid bacteria with high acid yield and research on tolerance to ethanol and temperature[J].Food and Fermentation Industries, 2021,47(10):72-78.
[14] ZHANG X P, LI Y R, TAO Y, et al.A novel method based on infrared spectroscopic inception-resnet networks for the detection of the major fish allergen parvalbumin[J].Food Chemistry, 2021, 337:127986.
[15] SONG Y N, LI Z X, LIN H, et al.Effect of malondialdehyde treatment on the IgE binding capacity and conformational structure of shrimp tropomyosin[J].Food Chemistry, 2015, 175:374-380.
[16] PESCUMA M, HÉBERT E M, HAERTLÉ T, et al.Lactobacillus delbrueckii sub sp.bulgaricus CRL 454 cleaves allergenic peptides of β-lactoglobulin[J].Food Chemistry, 2015, 170:407-414.
[17] 丁婕, 王梦梦, 汪立平, 等.鼠李糖乳酸杆菌对河豚肝脏中河豚毒素毒性的影响[J].食品科学, 2019, 40(19):237-241.
DING J, WANG M M, WANG L P, et al.Effect of Lactobacillus rhamnosus on the toxicity of tetrodotoxin in the liver of puffer fish[J].Food Science, 2019, 40(19):237-241.
[18] SHARP M F, STEPHEN J N, KRAFT L, et al.Immunological cross-reactivity between four distant parvalbumins-impact on allergen detection and diagnostics[J].Molecular Immunology, 2015, 63(2):437-448.
[19] KAWASE S, USHIO H, OHSHIMA T, et al.Preparation of monoclonal antibodies against tuna parvalbumin[J].Fisheries Science, 2001, 67(3):559-561.
[20] DO CARMO A, DA SILVA D, DE OLIVEIRA M, et al.Genes involved in protein metabolism of the probiotic lactic acid bacterium Lactobacillus delbrueckii UFV H2b20[J].Beneficial Microbes, 2011, 2(3):209-220.
[21] HEBERT E M, MAMONE G, PICARIELLO G, et al.Characterization of the pattern of α s1-and β-casein breakdown and release of a bioactive peptide by a cell envelope proteinase from Lactobacillus delbrueckii subsp.lactis CRL 581[J].Applied and Environmental Microbiology, 2008, 74(12):3 682-3 689.
[22] ANGGRAINI H, TONGKHAO K, CHANPUT W.Reducing milk allergenicity of cow, buffalo, and goat milk using lactic acid bacteria fermentation[R].Malang:8th Annual Basic Science International Conference (BaSIC)-Coverage of Basic Sciences toward the World′s Sustainability Challanges, 2018.
[23] KLICHE T, LI B, BOCKELMANN W, et al.Screening for proteolytically active lactic acid bacteria and bioactivity of peptide hydrolysates obtained with selected strains[J].Applied Microbiology and Biotechnology, 2017, 101(20):7 621-7 633.
[24] LAW J, HAANDRIKMAN A.Proteolytic enzymes of lactic acid bacteria[J].International Dairy Journal, 1997, 7(1):1-11.