酸粥是内蒙古、山西、陕西和广西等地的一种传统发酵食品[1-2],主要以糜米、大米为原料,部分地区也会加入玉米糁和面粉等发酵而成。因其维生素、有机酸等营养成分的含量较高而成为一种老少皆宜的谷物发酵食品[3]。近年来,越来越多的学者对酸粥开展研究,发现微生物在酸粥发酵过程当中起着至关重要的作用。王玉荣等[4]、张青等[5]使用MiSeq高通量测序技术对酸粥样品的细菌多样性分析结果发现酸粥中细菌主要是乳杆菌(Lactobacillus)和醋酸杆菌(Acetobacter)。折米娜[6]采用高通量测序技术对酸粥样品进行真菌多样性分析,结果发现16份样品中的子囊菌门(Ascomycota)平均相对含量高达97.54%。秦慧彬等[7]使用超高效液相色谱技术测定山西酸粥游离氨基酸含量发现甜味和苦味氨基酸含量较高。薛建岗等[2]、李文亚[8]在酸粥中检测到了氨基酸、钙、磷,并发现发酵后的酸粥总氨基酸含量高于未发酵的糜米。早在2001年陈忠军[9]已发现酸粥的独特风味与乳杆菌、酵母菌具有相关性。郭昊翔[10]结合高通量测序和高效液相色谱技术对酸粥的细菌群落演替和代谢产物进行相关性分析发现乳杆菌属(Lactobacillus)、梭菌属(Clostridium)和肠球菌属(Enterococcus)等均与有机酸、维生素的形成有关。
高通量测序作为研究食品发酵过程最关键的技术已经得到了快速发展。Illumina公司的MiSeq测序技术现已应用到包括辣椒酱[11]、酒窖泥[12]、腐乳[13]、酸马奶[14]和米酸汤[15]等传统发酵食品的核心细菌类群研究。虽然已有酸粥微生物多样性与营养成分相关研究[6-10],但是关于酸粥发酵过程中微生物演替与营养成分形成的相关性研究鲜见报道。
本研究为了探究酸粥发酵过程中的微生物群落变化,采用Illumina MiSeq测序技术对不同发酵时间段的酸粥进行细菌16S rRNA V3-V4区与真菌ITS序列测序予以探究,同时测定不同发酵时间段氨基酸含量,并监测其各发酵时间点酸度、可溶性固形物及蛋白质含量变化,使用相关性分析微生物与氨基酸形成规律,以期深度了解酸粥自然发酵过程。就目前酸粥生产规模小、产量少等情况[16],为现代工业化生产高品质产品提供理论依据与数据支持。
1 材料与方法
1.1 材料与试剂
糜米(Panicum miliaceum L.),市售;发酵引子:2020年8月采集自内蒙古鄂尔多斯市农户家中以糜米和大米为原料自然发酵24~30 h的酸粥;NaOH、甲基红乙醇溶液、溴甲酚绿乙醇溶液、HCI、硼酸溶液(20 g/L)、H2SO4(优级纯)、CuSO4(分析纯)、K2SO4(分析纯)、Na2CO3(分析纯),天津福晨化学试剂有限公司。
1.2 仪器与设备
SX-500蒸汽灭菌锅,日本TOMY公司;HH-4数显恒温水浴锅,江苏荣华仪器制造有限公司;UB-7 pH计,美国Denver Instrument公司;SW-CJ-2D双人单面垂直净化工作台,苏州安泰空气技术公司;JY1002电子天平,上海蒲春计量仪器有限公司;L-8900型氨基酸分析仪,日本日立公司;K9860全自动凯氏定氮仪,中国海能公司;GeneAmp® 9700型PCR仪,美国ABI公司。
1.3 实验方法
1.3.1 酸粥的制备与样本收集
将糜米淘洗沥干,按照糜米∶水=1∶3(g∶mL)加入蒸馏水,65 ℃水浴30 min。以采集的家庭自制酸粥为引子,按10%(体积分数)接种量接入冷却的米汤中,置于30 ℃静置发酵48 h,分别在发酵0、6、12、18、24、30、36、42和48 h取样,用于测定各项指标。
1.3.2 理化指标测定
参照GB 5009.237—2016使用pH计测定样品的pH值;使用全自动折光仪测定样品的可溶性固形物含量;参照GB 5009.5—2016使用全自动凯氏定氮仪测定样品的蛋白质含量;参照GB 5009.5—2016 (第一法)测定样品的氨基酸含量。以上指标每个样品平行3次。
1.3.3 酸粥细菌和真菌Illumina MiSeq测序
完成酸粥发酵液中基因组DNA提取并利用1%(质量分数,下同)琼脂糖凝胶电泳检测,细菌与真菌分别以338F/806R,ITS3F/ITS4R为引物进行PCR扩增。将PCR产物用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒切胶回收PCR产物。测序由上海美吉生物科技有限公司完成。
测序得到双端序列数据后,将成对的reads 拼接成一条序列,同时对reads的质量和拼接的效果进行质控过滤,根据序列首尾两端的标签序列和引物序列区分样品得到有效序列,并校正序列方向。参照崔梦君等[17]的方法,采用QIIME(v1.70)平台,将有效序列以97%的相似度进行分类操作单元(operational taxonomic units,OTU)划分后进行同源性比对,从而确定细菌和真菌的从门至属水平的分类学地位,并且进行α多样性分析。
1.4 数据分析
使用SPSS 26进行数据分析,数据以平均值±标准误差表示,当P<0.05时组间差异显著,当P<0.01时组间差异极显著。使用Origin 2021进行结果可视化。相关性分析选择Spearman系数,利用微科盟生科云平台(https://www.bioincloud.tech/)绘制相关性网络图。
2 结果与分析
2.1 理化特性分析
如图1-a所示,发酵过程中pH值整体呈下降趋势,在发酵0~24 h由4.09快速降至3.26,达到自然发酵酸粥酸度[18],随后下降趋势放缓,发酵前期pH值迅速下降的原因可能是产酸微生物在此阶段较为活跃,代谢旺盛,产酸能力较强。整个发酵过程中的pH值在3.12~4.09,能够抑制有害菌的生长,保证酸粥的品质。酸粥蛋白质含量的整体变化趋势较小(图1-b),从发酵开始就快速积累,直至48 h时达到峰值0.36 g/100 g,与郭昊翔等[19]的结果相似,微生物生长代谢过程中分泌大量的蛋白质可能是其含量增加的原因。发酵过程中可溶性固形物含量变化呈现出与蛋白质含量相同的变化规律,在检测时间内整体上随着发酵时间的延长其含量逐渐升高。

a-pH;b-蛋白质和可溶性固形物
图1 酸粥发酵过程中理化指标的变化
Fig.1 Changes of physicochemical characteristics of congee during fermentation
氨基酸在发酵过程中可被酵母利用,参与酸粥中的风味物质的合成。从酸粥9个采样点样品中共检测到16种氨基酸,其中谷氨酸与天冬氨酸含量最高,且2种氨基酸是鲜味氨基酸,使食品具有更优良的感官特性。酸粥中乳酸菌的含量较高[8-9],并且已知乳酸菌可以促进谷氨酸和天冬氨酸的释放[20],所以导致样品中2个氨基酸含量最高的原因可能是乳酸菌。从图2可以看出,在酸粥发酵前42 h随着发酵时间的延长多数氨基酸逐渐增加,精氨酸、亮氨酸、丙氨酸、谷氨酸和天冬氨酸在发酵过程中的变化较为显著。各氨基酸的含量在发酵前期与中期不断增加,在42 h时减少,但在发酵终点时达到最高值,48 h时总氨基酸含量增加至发酵前(0 h)的2倍。发酵48 h时的必需氨基酸与总氨基酸的比例为39%,接近于联合国粮食及农业组织/世界卫生组织推荐的蛋白质中必需氨基酸占总氨基酸的百分比[8],表明酸粥发酵可改善原料营养结构,提升其利用率。

图2 发酵过程中酸粥氨基酸含量的变化
Fig.2 Changes of amino acid content in congee during fermentation
2.2 酸粥发酵过程中微生物群落结构变化
2.2.1 酸粥微生物α-多样性分析
不同发酵时间段的酸粥样品16S rRNA序列测序结果共产生738 355条有效序列,ITS序列测序后共产生1 271 953条有效序列,进一步对有效序列进行α多样性分析,结果见表1。发酵前6 h细菌的Chao 1指数显著增加,真菌的却略有下降,可能是由于细菌在生长繁殖过程中释放出乳酸、乙酸等物质抑制了真菌的生长。发酵30 h开始,Chao 1指数和香农指数均有上升趋势,可能是一些真菌随着发酵逐渐适应环境,能够利用残留物质进行繁殖。在发酵48 h时微生物多样性与丰富度指数达到最高值,表明通过发酵细菌和真菌多样性与丰富度均得到提升。另外可以发现,整个发酵过程中的细菌Chao 1指数、香农指数和辛普森指数大于真菌,说明酸粥的细菌物种多样性与丰富度均大于真菌。
表1 发酵过程中酸粥细菌和真菌α-多样性指数
Table 1 α-Diversity index of bacteria and fungi in fermented congee
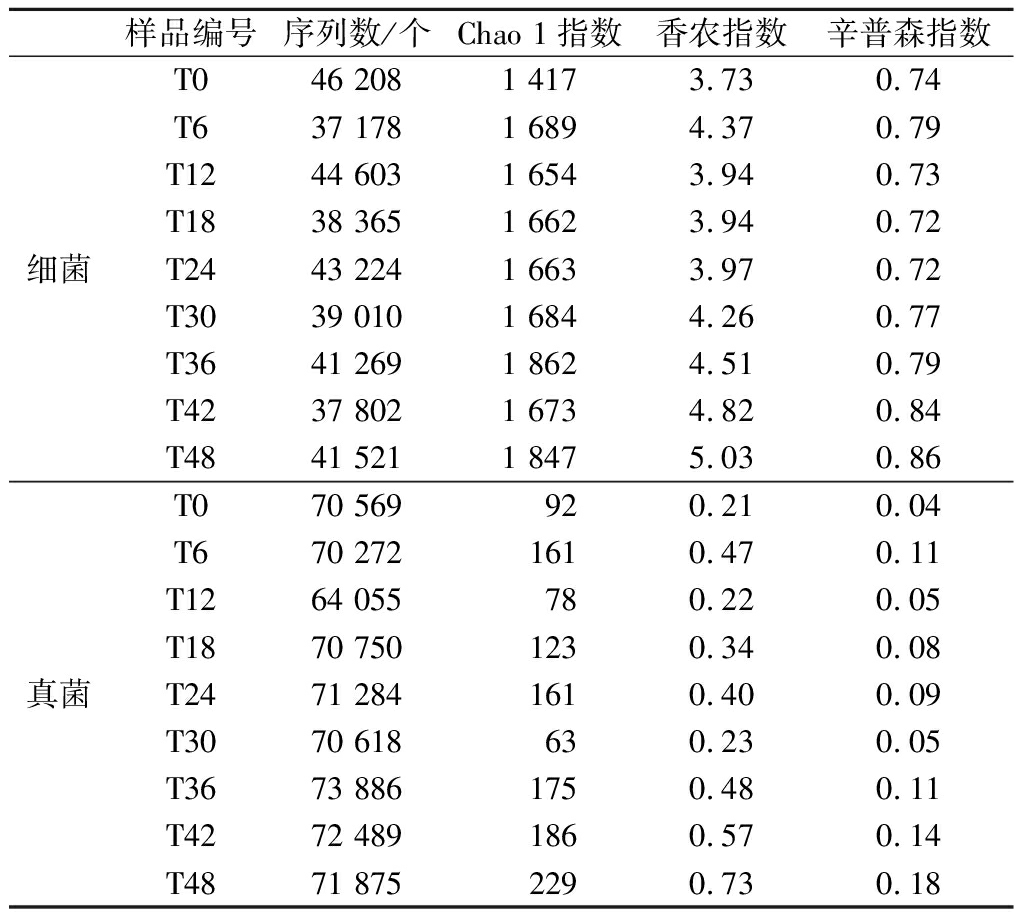
样品编号序列数/个Chao 1指数香农指数辛普森指数细菌T046 2081 4173.730.74T637 1781 6894.370.79T1244 6031 6543.940.73T1838 3651 6623.940.72T2443 2241 6633.970.72T3039 0101 6844.260.77T3641 2691 8624.510.79T4237 8021 6734.820.84T4841 5211 8475.030.86真菌T070 569 920.210.04T670 2721610.470.11T1264 055780.220.05T1870 7501230.340.08T2471 2841610.400.09T3070 618630.230.05T3673 8861750.480.11T4272 4891860.570.14T4871 8752290.730.18
注:测序量为32 010条序列时计算每个样品的Chao 1、香农指数与辛普森指数
2.2.2 细菌菌群结构特性分析
经过与物种数据库比对,分别获得了从门至属水平的物种分类信息。由图3-a可知,酸粥细菌主要由厚壁菌门(Firmicutes,91.56%)、变形菌门(Proteobacteria,8.14%)与放线菌门(Actinobacteria,0.03%)构成。随着发酵的进行,Firmicutes相对丰度虽然有所浮动,但在48 h时的含量仍为最高,相对丰度为93.24%。Proteobacteria在发酵前12 h时达到峰值23.82%,随后呈现较小浮动,在发酵终点时的丰度为6.49%。由此可知,Firmicutes为酸粥的优势菌门,郭昊翔[10]使用MiSeq测序技术对酸粥细菌菌群的变化进行分析发现Firmicutes为发酵后期的优势菌门。
在属水平下检测到的相对丰度大于0.1%的细菌属有隶属于Firmicutes的乳杆菌属(Lactobacillus)和肠球菌属(Enterococcus),隶属于Proteobacteria的小坂菌属(Kosakonia)、劳尔氏菌属(Ralstonia)和欧文氏菌属(Erwinia)等。在发酵6~12 h时细菌多样性最高,与α-多样性分析结果一致。酸粥发酵过程中的Lactobacillus平均相对丰度为91.53%,为绝对优势菌属,与王玉荣等[21]、王成等[22]的结果一致。Lactobacillus在发酵过程中产生以乳酸为主的有机酸,以降低发酵液的pH值来抑制其他微生物生长[23]。Kosakonia、Enterococcus和Erwinia均有先增加后减少的趋势,可能是由于Lactobacillus所产生的乳酸杆菌素抑制其生长[24]。由此可知,Lactobacillus在保证酸粥品质方面起着重要的作用。

a-门水平;b-属水平
图3 细菌群落结构组成
Fig.3 Structure of bacterial community on phylum level and genus level
2.2.3 真菌菌群结构特性分析
酸粥发酵过程中的真菌群落结构变化如图4-a所示,优势真菌门为子囊菌门(Ascomycota)和Anthophyta。Ascomycota在发酵初期的相对丰度为98.53%,随着发酵时间的增加其含量呈较小浮动,在发酵48 h时下降至91.36%。Anthophyta初始丰度为1.41%,48 h显著增加至8.39%。虽然随着发酵时间的延长Ascomycota的丰度有所下降,但是其丰度均大于90%,为酸粥发酵过程中的主要真菌门,与折米娜[6]的结果一致。
在属水平上,双足囊菌属(Dipodascus)与耶氏酵母属(Yarrowia)为优势菌属,在各发酵时间的平均相对丰度分别为47.12%和48.22%,随着发酵的进行其相对丰度略有下降。虽然毕赤酵母属(Pichia)含量微弱,但随着发酵时间的延长其相对丰度却有所增加,从0.47%增加至1.63%。王玉荣等[4]从内蒙古鄂尔多斯酸粥中分离鉴定出Yarrowia与Pichia。MENDONÇA等[25]发现Yarrowia与蛋白水解活性有关。童敏江等[26]对汾酒主要功能微生物进行单菌株发酵实验,结果发现Pichia具有产乙酸乙酯作用。Dipodascus尚未在酸粥中被发现,鲍奕达等[27]在郫县豆瓣样品中检测到该菌属并发现其与氨基酸转胺后的产物具有相关性,因此推测该菌属可能与氨基酸分解代谢有关,可能是潜在的风味物质产生菌。
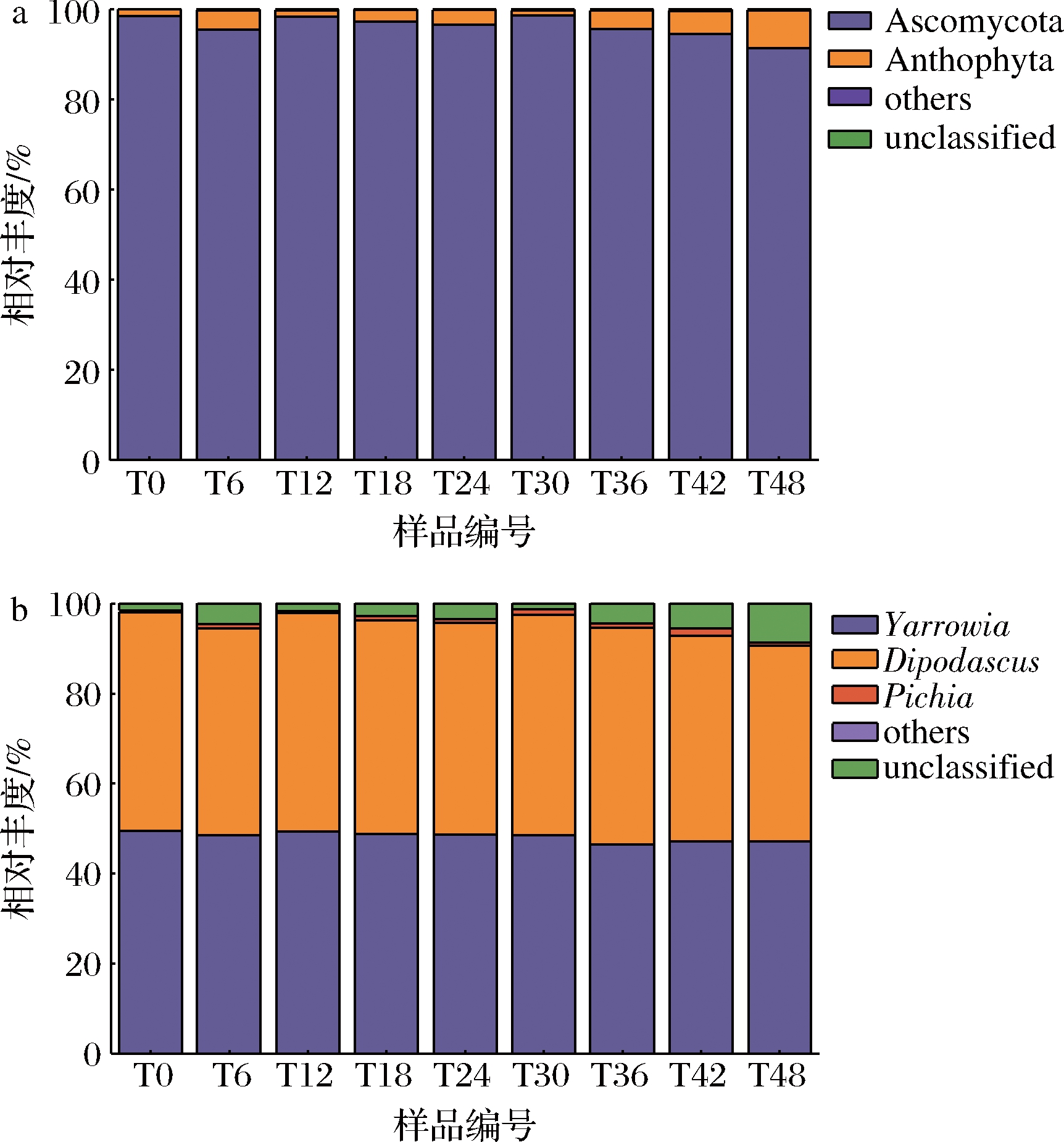
a-门水平;b-属水平
图4 真菌群落结构组成
Fig.4 Structure of fungal community on phylum level and genus level
2.3 微生物与理化指标之间的关联性分析
理化因子与核心微生物属间的Spearman相关系数的结果表明(图5),Lactobacillus、Klebsiella、Kosakonia、Ralstonia、Erwinia、Yarrowia和Dipodascus与pH正相关,与氨基酸负相关,可见酸性环境抑制部分微生物的生长。Pichia与大多数理化特性呈正相关,包括苏氨酸、丝氨酸、甘氨酸、丙氨酸、缬氨酸、蛋氨酸、异亮氨酸、亮氨酸、苯丙氨酸、组氨酸、赖氨酸、精氨酸、脯氨酸、蛋白质和可溶性固形物。任宇婷等[28]通过对广西地区酸粥细菌菌群功能进行预测,发现酸粥中存在的Pichia与氨基酸代谢有关,因此推测总氨基酸在42 h时突然下降的原因也可能是Pichia在此发酵阶段的丰度最高。Pichia与pH呈负相关,可能是由于其生长pH值比较宽(pH 3~7),且较适宜酸性环境,因此在一定范围内pH值越小、酸度越大,越适宜Pichia的生长。

注:正相关和负相关分别用红色和蓝色表示
图5 酸粥核心微生物与理化特性的相关性
Fig.5 Correlation between physicochemical indicators and microbial diversity in fermented congee
3 结论
本研究基于高通量测序方法分析不同发酵时间段酸粥的微生物变化,结果发现整个发酵过程以隶属于Firmicutes的Lactobacillus为绝对优势细菌属,优势真菌属为Yarrowia、Dipodascus和Pichia。对酸粥理化指标进行测定发现,通过发酵酸粥pH下降,蛋白质、可溶性固形物均有所上升,pH值的下降有效抑制了部分条件致病菌的生长。氨基酸作为挥发性风味化合物的前体物质在发酵过程中亦呈上升趋势,且氨基酸组成更为合理。对酸粥微生物群落与理化指标进行相关性分析,结果发现pH和氨基酸对酸粥微生物群落有较大影响,因此控制酸粥发酵酸度对产品安全性与风味有着至关重要的作用。
[1] 秦慧彬, 黄志伟, 张志强, 等.传统食品酸粥的发酵工艺[J].食品与发酵工业, 2019, 45(17):111-116.
QIN H B, HUANG Z W, ZHANG Z Q, et al.Fermentation process of preparing traditional food Suanzhou[J].Food and Fermentation Industries, 2019, 45(17):111-116.
[2] 薛建岗, 陈永福, 于海静, 等.内蒙古西部地区自然发酵酸粥化学成分及微生物组成分析[J].食品科技, 2013, 38(7):10-14.
XUE J G, CHEN Y F, YU H J, et al.Analysis of chemical and microbiological composition in naturally fermented acidic-gruel from western areas of Inner Mongolia[J].Food Science and Technology, 2013, 38(7):10-14.
[3] 张青, 夏美茹.酸粥的营养价值、功能特性及其研究进展[J].现代食品, 2020(9):119-120.
ZHANG Q, XIA M R.Research progress of nutritive value, functional characteristics and products of acidic-gruel[J].Modern Food, 2020(9):119-120.
[4] 王玉荣, 折米娜, 刘康玲, 等.内蒙古鄂尔多斯地区酸粥真菌多样性研究[J].中国酿造, 2018, 37(8):50-55.
WANG Y R, SHE M N, LIU K L, et al.Fungal diversity in acidic-gruel from Ordos area of Inner Mongolia[J].China Brewing, 2018, 37(8):50-55.
[5] 张青, 郭淑文, 苏靖, 等.基于高通量测序技术巴彦淖尔市酸粥中细菌多样性分析[J].中国酿造, 2020, 39(9):101-105.
ZHANG Q, GUO S W, SU J, et al.Bacterial diversity in sour porridge from Bayannur based on high-throughput sequencing[J].China Brewing, 2020, 39(9):101-105.
[6] 折米娜. 内蒙古西部地区酸粥真菌群落结构研究及分离菌株对品质影响的评价[D].呼和浩特:内蒙古农业大学, 2019.
SHE M N.Diversity of fungal microflora of acidic-gruel in western Inner Mongolia and evaluation of the effect of isolated strains on quality[D].Hohhot:Inner Mongolia Agricultural University, 2019.
[7] 秦慧彬, 田翔, 王海岗, 等.超高效液相色谱法测定传统发酵酸粥的游离氨基酸含量[J].食品科技, 2021, 46(4):273-280.
QIN H B, TIAN X, WANG H G, et al.Determination of free amino acids in traditional fermented acidic-gruel by ultra-high performance liquid chromatography[J].Food Science and Technology, 2021, 46(4):273-280.
[8] 李文亚. 晋西北酸粥营养成分的分析及其发酵工艺的研究[D].太原:山西大学, 2017.
LI W Y.Analysis of nutritional components and fermentation technology of northwestern Shanxi sour porridge[D].Taiyuan:Shanxi University, 2017.
[9] 陈忠军. 内蒙古河套地区酸粥中发酵菌生物学特性研究[D].呼和浩特:内蒙古农业大学, 2001.
CHEN Z J.Studies on biological properties of fermentogen from acidic-gruel from the HETAO area in Inner Mongolia[D].Hohhot:Inner Mongolia Agricultural University, 2001.
[10] 郭昊翔. 基于细菌群落结构变化的内蒙古自然发酵酸粥代谢产物及风味的研究[D].呼和浩特:内蒙古农业大学, 2020.
GUO H X.Research on the metabolites and flavor of naturally fermented sour porridge in Inner Mongolia based on the change of bacterial community structure[D].Hohhot:Inner Mongolia Agricultural University, 2020.
[11] 沈馨, 王艳, 代凯文, 等.基于Miseq高通量测序技术的辣椒酱核心细菌类群研究[J].食品研究与开发, 2018, 39(10):151-157.
SHEN X, WANG Y, DAI K W, et al.Characterization of core bacterial microflora in chilli sauce by miseq high throughput sequencing technologies[J].Food Research and Development, 2018, 39(10):151-157.
[12] 杨小丽, 尚雪娇, 余海忠, 等.基于Miseq高通量测序技术的古襄阳酒窖泥细菌多样性评价[J].中国酿造, 2018, 37(7):26-30.
YANG X L, SHANG X J, YU H Z, et al.Bacterial diversity in pit mud of Guxiangyang Baijiu analyzed by Miseq high-throughput sequencing[J].China Brewing, 2018, 37(7):26-30.
[13] XU D D, WANG P, ZHANG X, et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu, a traditional Chinese fermented soybean food[J].Food Microbiology, 2020, 86:103340.
[14] XIA Y N, YU J Q, LIU H, et al.Novel insight into physicochemical and flavor formation in koumiss based on microbial metabolic network[J].Food Research International, 2021, 149:110659.
[15] 田亚. 贵州米酸汤优势微生物与品质特性的相关性研究[D].贵阳:贵州大学, 2020.
TIAN Y.Study on the correlation between the dominant microorganisms and quality characteristics of Guizhou rice sour soup[D].Guiyang:Guizhou University, 2020.
[16] 陈美标, 廖富迎, 郑文辉.乳酸菌谷物发酵食品的发酵工艺研究[J].食品安全导刊, 2018(36):118-119.
CHEN M B, LIAO F Y, ZHENG W H.Study on fermentation technology of lactic acid bacteria fermented cereal food [J].China Food Safety Magazine, 2018(36):118-119.
[17] 崔梦君, 王玉荣, 葛东颖, 等.遵义地区莽椒细菌多样性及PICRUSt基因功能预测分析[J].食品与发酵工业, 2020, 46(4):106-112.
CUI M J, WANG Y R, GE D Y, et al.Study on bacterial diversity of Mangjiao in Zunyi region and PICRUSt-based predicted metagenomic analysis[J].Food and Fermentation Industries, 2020, 46(4):106-112.
[18] 王志伟, 陈忠军.不同乳酸菌菌株对酸粥品质影响的研究[J].食品科技, 2010, 35(8):61-64.
WANG Z W, CHEN Z J.Research on influence of different Lactobacillus strains on the quality of the acidic-gruel[J].Food Science and Technology, 2010, 35(8):61-64.
[19] 郭昊翔, 满都拉, 任宇婷, 等.传统酸粥发酵过程中营养成分及风味的变化规律[J].食品与发酵工业, 2020, 46(12):166-172.
GUO H X, Mandlaa, REN Y T, et al.Variation of nutrients and flavors in the fermentation process of traditional sour porridge[J].Food and Fermentation Industries, 2020, 46(12):166-172.
[20] 单玉鑫, 徐文欢, 李采婵, 等.酸鱼产品中微生物群落结构与品质之间的关系研究[J].食品工业科技, 2020, 41(6):277-283.
SHAN Y X, XU W H, LI C C, et al.Relationship between microbial community structure and quality of Suanyu products[J].Science and Technology of Food Industry, 2020, 41(6):277-283.
[21] 王玉荣, 折米娜, 刘康玲, 等.基于MiSeq高通量测序技术内蒙古地区酸粥细菌多样性研究[J].食品工业科技, 2018, 39(19):124-129.
WANG Y R, SHE M N, LIU K L, et al.Diversity of bacteria microflora in acidic-gruel of Inner Mongolia area by MiSeq high throughput sequencing[J].Science and Technology of Food Industry, 2018, 39(19):124-129.
[22] 王成, 肖晗, 郭浩楠, 等.自然发酵酸粥中细菌多样性分析及优势乳酸菌的分离与鉴定[J].食品科技, 2020, 45(10):46-52.
WANG C, XIAO H, GUO H N, et al.Bacterial diversity analysis, isolation and identification of dominant lactic acid bacteria in natural fermented sour porridge[J].Food Science and Technology, 2020, 45(10):46-52.
[23] HOLZAPFEL W H, GEISEN R, SCHILLINGER U.Biological preservation of foods with reference to protective cultures, bacteriocins and food-grade enzymes[J].International Journal of Food Microbiology, 1995, 24(3):343-362.
[24] 谢玲, 兰林, 李宇航, 等.米浆发酵过程中的微生物群落变化[J].食品与发酵工业, 2021, 47(21):67-72.
XIE L, LAN L, LI Y H, et al.Microbial community changes in rice slurry fermentation[J].Food and Fermentation Industries, 2021, 47(21):67-72.
[25] MENDONÇA R C S, GOUV A D M, HUNGARO H M, et al.Dynamics of the yeast flora in artisanal country style and industrial dry cured sausage (yeast in fermented sausage)[J].Food Control, 2013, 29(1):143-148.
A D M, HUNGARO H M, et al.Dynamics of the yeast flora in artisanal country style and industrial dry cured sausage (yeast in fermented sausage)[J].Food Control, 2013, 29(1):143-148.
[26] 童敏江, 雷振河, 李琦, 等.汾酒酿造过程中主要功能微生物的筛选和研究[J].食品与发酵工业, 2010, 36(2):17-21.
TONG M J, LEI Z H, LI Q, et al.Screening and study of the main functional microbes during the brewing process of the Fen liquor[J].Food and Fermentation Industries, 2010, 36(2):17-21.
[27] 鲍奕达, 张丽杰, 郑鹏飞, 等.郫县豆瓣发酵过程的微生物多样性及溯源分析[J].微生物学报, 2020, 60(11):2 555-2 571.
BAO Y D, ZHANG L J, ZHENG P F, et al.Diversity and source tracking of microbial community in Pixian broad bean paste[J].Acta Microbiologica Sinica, 2020, 60(11):2 555-2 571.
[28] 任宇婷, 陈春利, 朱永亮, 等.广西扶绥酸粥中微生物组成及营养成分分析[J].食品与发酵工业, 2021, 47(20):37-43.
REN Y T, CHEN C L, ZHU Y L, et al.The microbial and nutritional composition of sour porridge from Fusui, Guangxi[J].Food and Fermentation Industries, 2021, 47(20):37-43.