鲊,作为我国流传千年的一种食物保存方法,通常是用米粉或面粉等加入其他佐料经过密封发酵,在我国云贵川湘鄂赣等地常见[1]。鄂西地区将辣椒称为广椒,则有了鲊广椒这一特色菜肴,其滋味酸辣咸香,烹饪方式多种多样。近年来,国内针对鲊广椒的品质、工艺和微生物类群等方面均有开展研究。尚雪娇等[2]采用仿生技术对当阳地区鲊广椒的感官品质及有机酸进行了分析,结果发现鲊广椒中芳香类挥发性物质较少,且在酸味指标上差异较大,而酸味的来源主要为乳酸。王玉荣等[3]采用第二代测序技术分别对当阳地区鲊广椒中的真菌和细菌多样性进行了解析,结果显示念珠菌属(Candida)、曲霉菌属(Eurotium)和毕赤酵母属(Pichia)为鲊广椒中主要的真菌属;乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)和片球菌属(Pediococcus)等乳酸菌为鲊广椒中主要的细菌属[4]。此外,CAI等[5]针对恩施土家族苗族自治州鲊广椒细菌类群展开了研究,结果亦表明鲊广椒中蕴含了大量的Lactobacillus等乳酸菌类群。不同地域和制作工艺等因素可能对鲊广椒中的微生物类群有着一定的影响[6],因此解析不同地区鲊广椒的微生物类群对全面掌握鲊广椒中的微生物种类具有重要意义。
作为湖北省直接管辖的县级市之一,天门市坐落于长江中游城市群,当地亦有鲊广椒的制作与食用习俗,而其较为特殊的地理环境可能使天门地区鲊广椒与其他地区鲊广椒的微生物类群存在一定差异。本研究采用MiSeq高通量测序技术对天门地区鲊广椒中细菌多样性进行解析的同时,对菌属之间的相关性亦展开探讨,并进一步预测菌群功能,此外还采用纯培养方式对其中的乳酸菌进行鉴定和保藏,以期在后续鲊广椒产业化加工和产品创新中提供理论参考和菌株支持。
1 材料与方法
1.1 材料与试剂
样品采集:15份鲊广椒样品分别于2019年10月采集自湖北省天门市人民路市场、官路农贸市场和南湖临时菜市场,样品编号为TM01~15。所有样品均为不同农户家中自制,样品选择标准:发酵时间超过30 d,且未经过熟制;色泽呈玉米黄或略带红色;不存在除鲊广椒以外的异味。
基因组提取试剂盒,德国QIAGEN公司;DNA聚合酶、rTaq酶和10×Buffer、dNTP,宝生物工程(大连)有限公司;MRS、LB培养基,青岛海博生物技术有限公司;正向引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)、反向引物806R(5′-GGACTACHVGGGTWTCTAAT-3′),武汉天一辉远生物科技有限公司。
1.2 主要仪器设备
Veriti FAST梯度PCR仪,美国ABI公司;UVPCDS8000凝胶成像分析系统,美国Protein Simple公司;Illumina MiSeq PE250高通量测序平台,美国Illumina公司;R930型机架式服务器,美国DELL公司;DG250型厌氧工作站,英国Don Whitley公司。
1.3 实验方法
1.3.1 宏基因组DNA的提取、PCR扩增和高通量测序
鲊广椒宏基因组DNA提取:每份样品取2 g,参照基因组提取试剂盒使用方法提取宏基因组DNA。
PCR扩增和高通量测序:在正向引物前段添加8个碱基对作为核苷酸标签以便进行序列配对。针对提取合格的总DNA的16S rRNA V3~V4区域进行PCR扩增,扩增条件均参照李娜等[7]的方法,检测合格的扩增产物寄至上海美吉生物医药科技有限公司完成测序。
1.3.2 序列质控和生物信息学分析
参照郭壮等[8]的方法对下机序列进行配对和质控,下机后的序列采用QIIME平台(v1.9.0)进行生物信息学分析[9]。采用UCLUST两步法按照100%和97%相似度构建分类操作单元(operational taxonomic units,OTU)[10]矩阵,使用ChimeraSlayer软件将含有嵌合体序列的OTU删除[11];余下OTU分别选出1 条代表性序列在数据库[12-13]中进行比对,将OTU的分类学地位分别鉴定到门、纲、目、科和属水平上;使用主坐标分析和围绕中心点的划分(partitioning around medoid,PAM)聚类分析对鲊广椒样品进行β多样性分析;通过计算所有样品的Chao 1指数、发现物种数、香农指数和辛普森指数对样品进行α多样性分析;采用PICRUSt软件预测鲊广椒的菌群功能[14]。
1.3.3 乳酸菌的分离鉴定
乳酸菌的分离:参照向凡舒等[15]的方法对样品进行预处理和倍比稀释涂布后,将MRS平板于30 ℃厌氧培养36~48 h后取出。选取形状、颜色、表面光滑程度和大小均有不同,且周围产生透明圈的疑似乳酸菌菌落,采用平板划线法进行纯化,同时对纯化后的菌株进行革兰氏染色和过氧化氢酶试验,最后采用甘油保藏法将革兰氏阳性且过氧化氢酶阴性的菌株保藏于-80 ℃备用。
乳酸菌的鉴定:首先富集菌体并提取疑似乳酸菌菌株DNA,继而参照1.3.1中的方法进行PCR扩增,对合格产物进行连接转化,最后挑取阳性克隆子寄至上海桑尼生物科技有限公司测序,返回的序列经过手动去引物后在BLAST网站上进行比对[16]。
1.3.4 数据处理
使用past3软件进行Mann-Whitnay检验,分析不同聚类间的显著差异;使用Origin2017软件绘制主坐标分析图和柱形图;使用R(v4.1.0)软件绘制PAM聚类图[17]和α指数小提琴图;使用GraphPad Prism 9绘制不同聚类间菌属差异柱形图;使用Cytoscape(v3.7.2)绘制菌属相关性网络图;使用STAMP(2.1.3)绘制COG功能差异图;使用MEGA7.0和R(v4.1.0)软件共同构建系统发育树。
2 结果与分析
2.1 鲊广椒样品的β多样性分析
本研究首先采用主坐标分析法和PAM聚类分析法来探讨不同鲊广椒样品的细菌群落结构特征,结果如图1所示。由图1-a可知,第一主成分和第二主成分的占比分别为86.38%和4.74%,且所有样品在空间排布上呈现出了明显的聚类和分离趋势。其中10个样品(TM02、TM03、TM04、TM05、TM07、TM08、TM09、TM010、TM13和TM15)较为集中的排布在第一主成分的负轴周围,将其视为聚类Ⅰ。而另外5个样品(TM01、TM06、TM11、TM12和TM14)则较为分散的排布在第一、三和四象限中,将其视为聚类Ⅱ。
经PAM聚类分析发现,当15份样品被分为2个聚类时,CH指数最高,为39,而当样品被分为3~5个聚类时,CH指数分别为27、10和5,这表明最佳聚类数为2。由图1-b可知,利用主坐标分析将K-Means聚类分析结果可视化后的2个聚类分离趋势亦非常明显,其中10 个样品全部分布在第二象限和第三象限,而另外5个样品则全部分布在第一象限和第四象限。K-Means聚类趋势同主坐标分析得到的聚类其样品组成完全相同。值得注意的是,聚类Ⅰ中的样品与聚类Ⅱ相比分布更为集中,这说明聚类Ⅰ中样品的菌群结构更为相近。
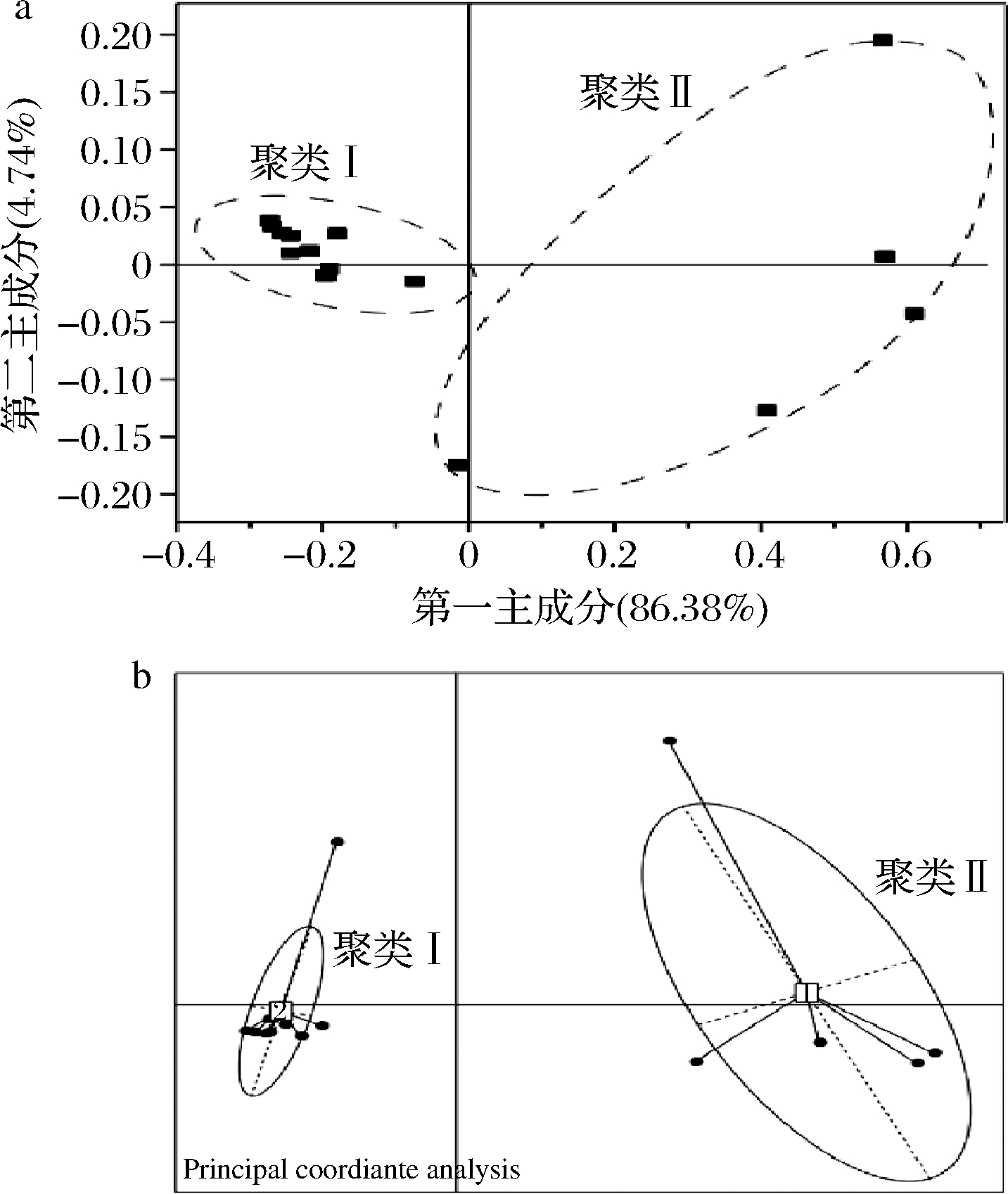
a-基于加权UniFrac距离的主坐标分析; b-基于OTU水平的PAM聚类分析
图1 基于加权UniFrac距离的主坐标分析和基于 OTU水平的PAM聚类分析
Fig.1 Principal coordinate analysis based on weighted UniFrac distance and PAM cluster analysis based on OTU level
2.2 α多样性分析
在对样品进行β多样性分析的基础上,本研究进一步通过α指数来分析2个聚类中的物种丰富度和均匀度。本研究对所有样品序列均进行了抽平处理,2个聚类的Chao 1指数、发现物种数、香农指数和辛普森指数如图2所示。
由图2可知,聚类Ⅰ的4项α多样性指数均小于聚类Ⅱ,且两个聚类在Chao 1指数(图2-a)和发现物种数(图2-b)上的差异非常显著(P<0.01),在香农指数(图2-c)上差异显著(P<0.05),而在辛普森指数(图2-d)上差异不显著(P>0.05)。这表明聚类Ⅰ的细菌类群在丰富度和多样性上均要显著低于聚类Ⅱ。

图2 两个聚类的α多样性指数比较分析
Fig.2 Comparative analysis of α diversity index of the two clusters
注:*表示差异显著,P<0.05;**表示差异非常显著,P<0.01;NS表示差异不显著,P>0.05
2.3 基于细菌门和细菌属水平的细菌多样性分析
所有样品经过测序共得到359 236条高质量序列,经过97%相似度划分得到11 582个OTU,经比对共鉴定到18个门和260个属。本研究将平均相对含量>1.0%的门和属作为鲊广椒中的优势门和属[18],2个聚类中的优势门相对含量分布及差异分析如图3所示。聚类Ⅰ中的优势门为Firmicutes(硬壁菌门,96.99%)和Proteobacteria(变形菌门,2.30%),聚类Ⅱ中的优势门为Proteobacteria(62.94%)、Firmicutes(22.86%)和Actinobacteria(放线菌门,9.00%)。经过Mann-Whitnay检验发现2个聚类在3个门上均差异显著,且在Firmicutes(图3-b)和Proteobacteria(图3-c)上的差异极显著(P<0.001)。
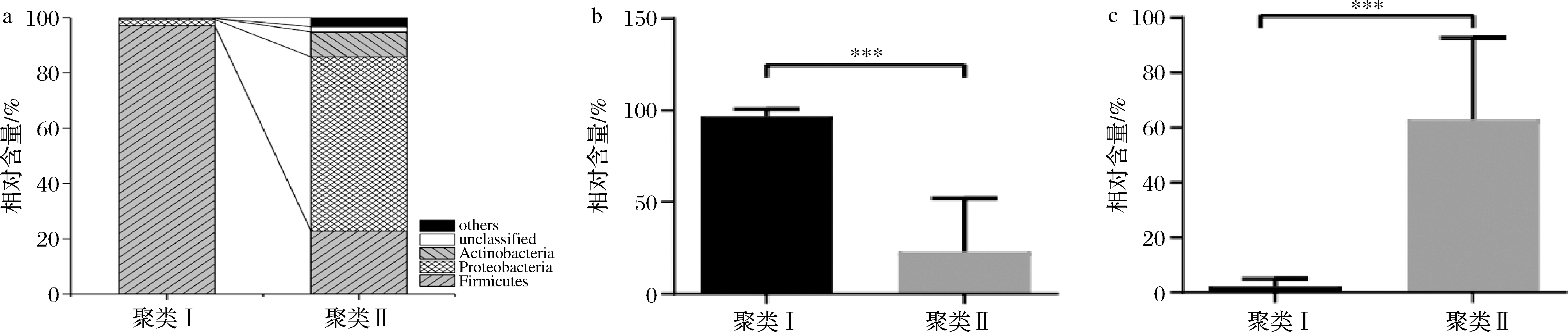
a-优势细菌门分布;b-硬壁菌门;c-变形菌门
图3 两个聚类中优势细菌门分布及硬壁菌门和变形菌门相对含量差异分析
Fig.3 Analysis on the distribution of dominant bacteria and the relative content difference of Firmicutes and Proteobacteria in the two clusters
注:***表示差异极显著,P<0.001(下同)
2个聚类中的优势属相对含量分布及差异分析如图4所示。由图4可知,聚类Ⅰ中有4个优势属,分别为Lactobacillus(乳杆菌属,89.77%)、Tetragenococcus(四联球菌属,3.29%)、Weissella(魏斯氏菌属,2.20%)和Staphylococcus(葡萄球菌属,1.52%),其中乳酸菌含量高达95.26%;聚类Ⅱ中有8 个优势属,分别为Klebsiella(克雷伯氏菌属,19.81%)、Kosakonia(小坂菌属,12.24%)、Tetragenococcus(10.89%)、Lactobacillus(8.17%)、Pseudomonas(假单胞菌属,5.5%)、Xanthomonas(黄单胞菌属,4.08%)、Rhizobium(根瘤菌属,4.08%)和Corynebacterium(棒状杆菌属,3.35%),乳酸菌含量仅为19.06%。经Mann-Whitnay检验发现,2个聚类间在Lactobacillus、Klebsiella、Kosakonia和Pseudomonas上均存在极显著差异(P<0.001)。由此可见,聚类Ⅱ中的优势菌属数量要明显多于聚类Ⅰ,但聚类Ⅰ中乳酸菌含量要远高于聚类Ⅱ。王玉荣等[4]前期在解析湖北省当阳地区鲊广椒细菌类群时得到的结论与本研究的聚类Ⅰ相似,而与聚类Ⅱ中的菌群结构差异较大。
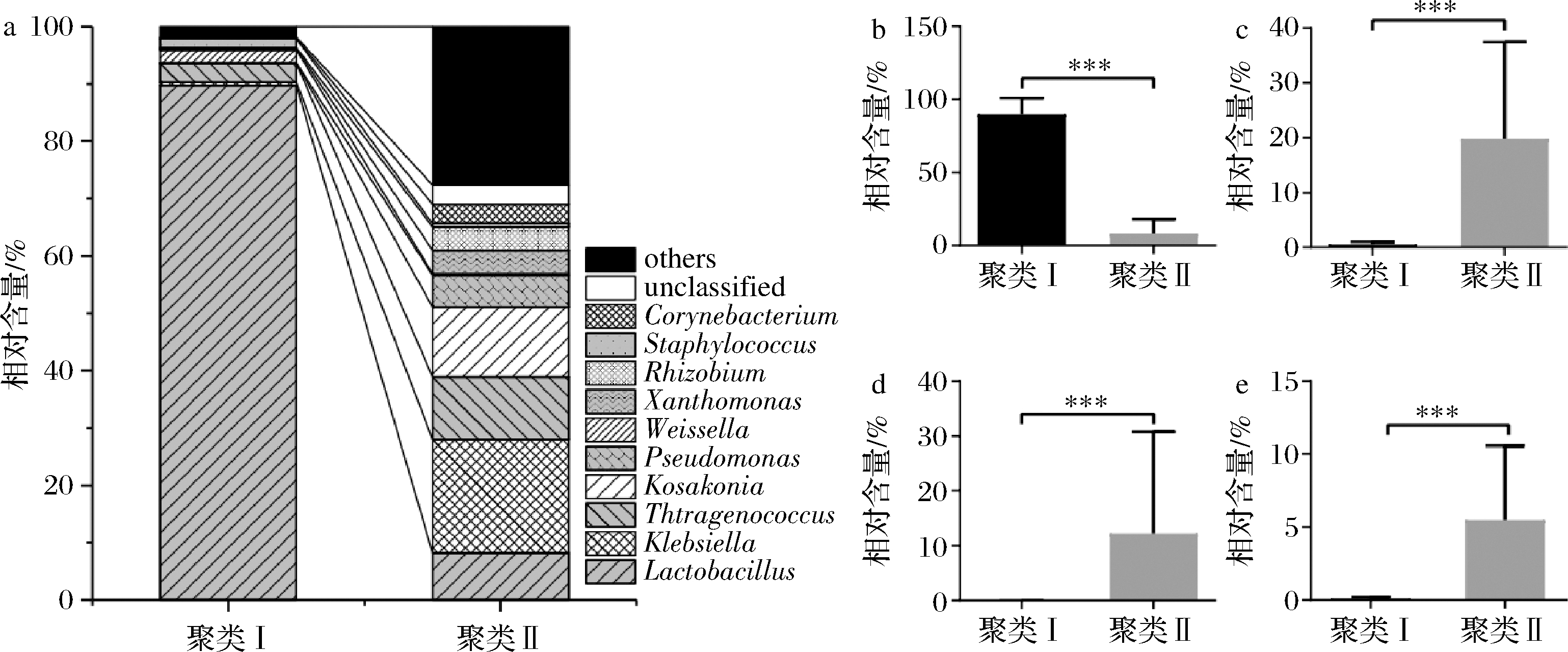
a-优势细菌属;b-乳杆菌属;c-克雷伯氏菌属;d-小坂菌属e-假单胞菌属
图4 两个聚类中优势细菌属分布及乳杆菌属、克雷伯氏菌属、小坂菌属和假单胞菌属相对含量差异分析
Fig.4 Analysis on the distribution of dominant bacteria and the relative content difference of Lactobacillus, Klebsiella, Kosakonia and Pseudomonas in the two clusters
聚类Ⅱ中的部分菌属包含着一些具有条件致病性的菌种,例如隶属于Klebsiella的Klebsiella pneumoniae被认为是全球性的病原菌,可导致化脓性肝脓肿、肺炎和软组织感染等[19];隶属于Pseudomonas的Pseudomonas aeruginosa对人体健康具有急慢性感染性[20];而Xanthomonas spp.是一类广泛的植物病原菌[21]。由此可见,2个聚类样品在安全品质上可能亦存在差异,这或许是不同农户选择的制作原料在品质上存在较大偏差,亦或是制作环境差异所导致的。
2.4 优势细菌属的相关性分析
为探讨鲊广椒中菌群间的相关关系,本研究进一步对优势菌属之间的相关性进行了分析,结果如图5所示。Xanthomonas与Rhizobium显著正相关(R=0.540,P<0.05),与Kosakonia极显著正相关(R=0.879,P<0.001);Pseudomonas与Klebsiella极显著正相关(R=0.820,P<0.001);而Lactobacillus与Xanthomonas(R=-0.596,P<0.05)和Kosakonia(R=-0.552,P<0.05)显著负相关,与Pseudomonas(R=-0.698,P<0.01)和Klebsiella呈非常显著负相关关系(R=-0.738,P<0.01)。王玉荣等[22]采用第三代测序技术解析当阳地区鲊广椒中细菌多样性,结果表明L.plantarum是其中最主要的菌种,而MAO等[23]的研究表明L.plantarum产生的乳酸等成分可起到抑菌作用。因而从理论上推断,Lactobacillus对Xanthomonas等具有条件致病性菌属的生长繁殖可能具有一定的抑制作用。

图5 鲊广椒中优势细菌属的相关性网络图
Fig.5 Correlation network of dominant genus in Zha-chili
注:实线表示正相关,虚线表示负相关, 线条的粗细代表相关性的大小。
2.5 不同聚类的细菌类群基因功能差异分析
本研究进一步对不同聚类的菌群功能以及功能差异进行了分析。所有样品共注释4 166个COG,分别属于23个功能大类。2个聚类在功能上存在的差异如图6所示,聚类Ⅰ在D、L、J、R和F功能上显著大于聚类Ⅱ(P<0.05),而聚类Ⅱ在U、A、Q、C、T、O、P、K、G和E功能上显著大于聚类Ⅰ(P<0.05),但2个聚类在R、G和E功能上的表达均较高。玉米面作为天门鲊广椒的主要原料,其营养成分主要为淀粉和蛋白质[24-25],而这两者通常是为微生物提供营养物质的良好来源。因而,在菌群吸收营养物质维持生存的同时,必将会引起一些产物的代谢反应,这或许是鲊广椒中细菌类群在碳水化合物和氨基酸的转运与代谢功能上具有较高表达的原因。
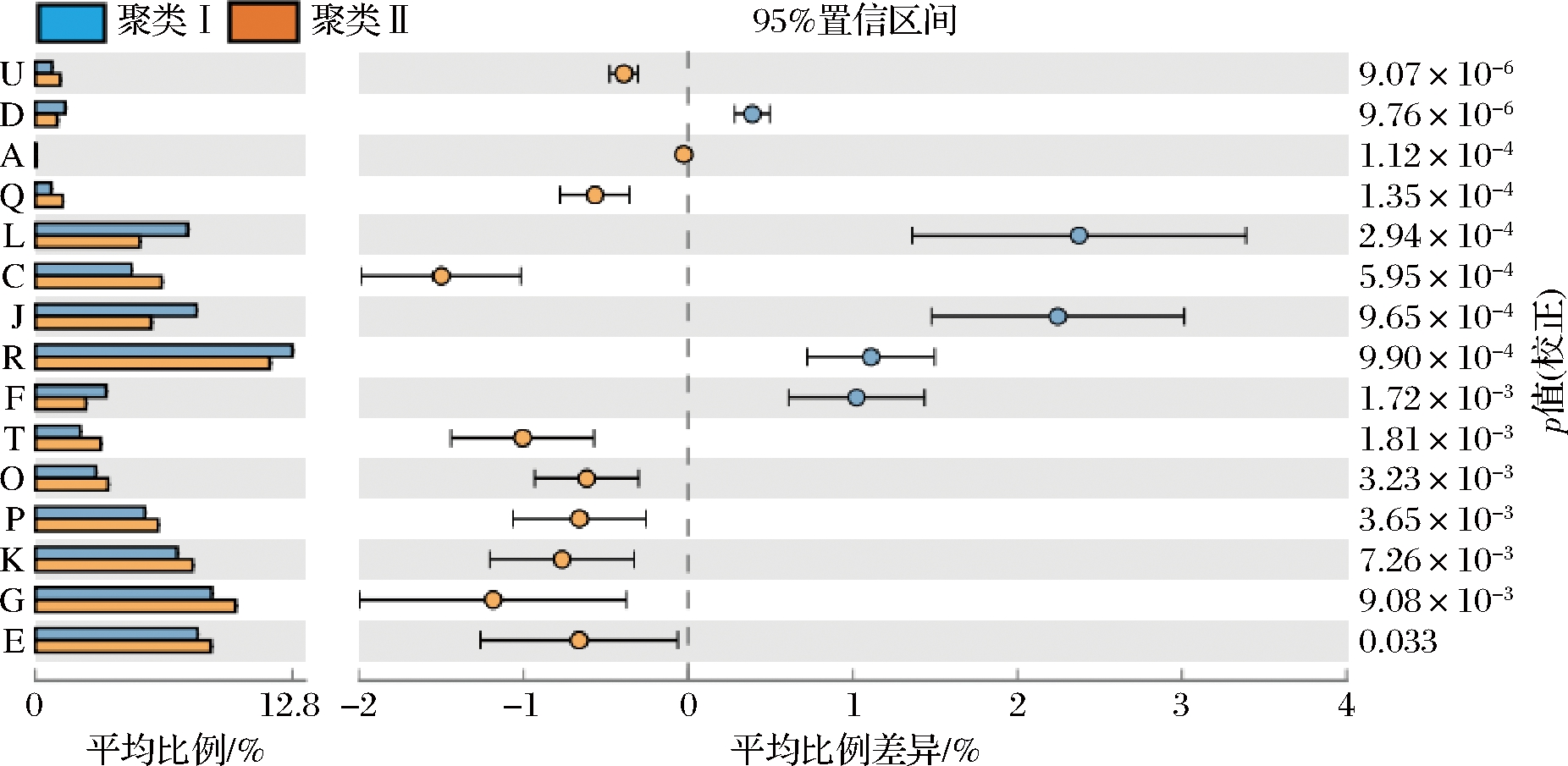
A-RNA加工与修饰;C-能量生产和转换;D-细胞周期控制,细胞 分裂,染色体分割;E-氨基酸转运和代谢;F-核苷酸转运和代谢; G-碳水化合物运输和代谢;J-翻译、核糖体结构与生物合成; K-转录;L-复制、重组和修复;O-翻译后饰,蛋白质周转,伴侣; P-无机离子及代谢;Q-次生代谢物的生物合成、运输和分解代谢; R-一般功能预测预测;T-信号转导机制;U-细胞内运输、 分泌和囊泡运输
图6 两个聚类中细菌基因功能预测差异分析
Fig.6 Differential analysis of predicted bacterial gene functions in the two clusters
2.6 乳酸菌多样性分析
通过纯培养,本研究所采集的15份样品共分离得到34株疑似乳酸菌分离株,经序列比对后鉴定结果如图7所示。
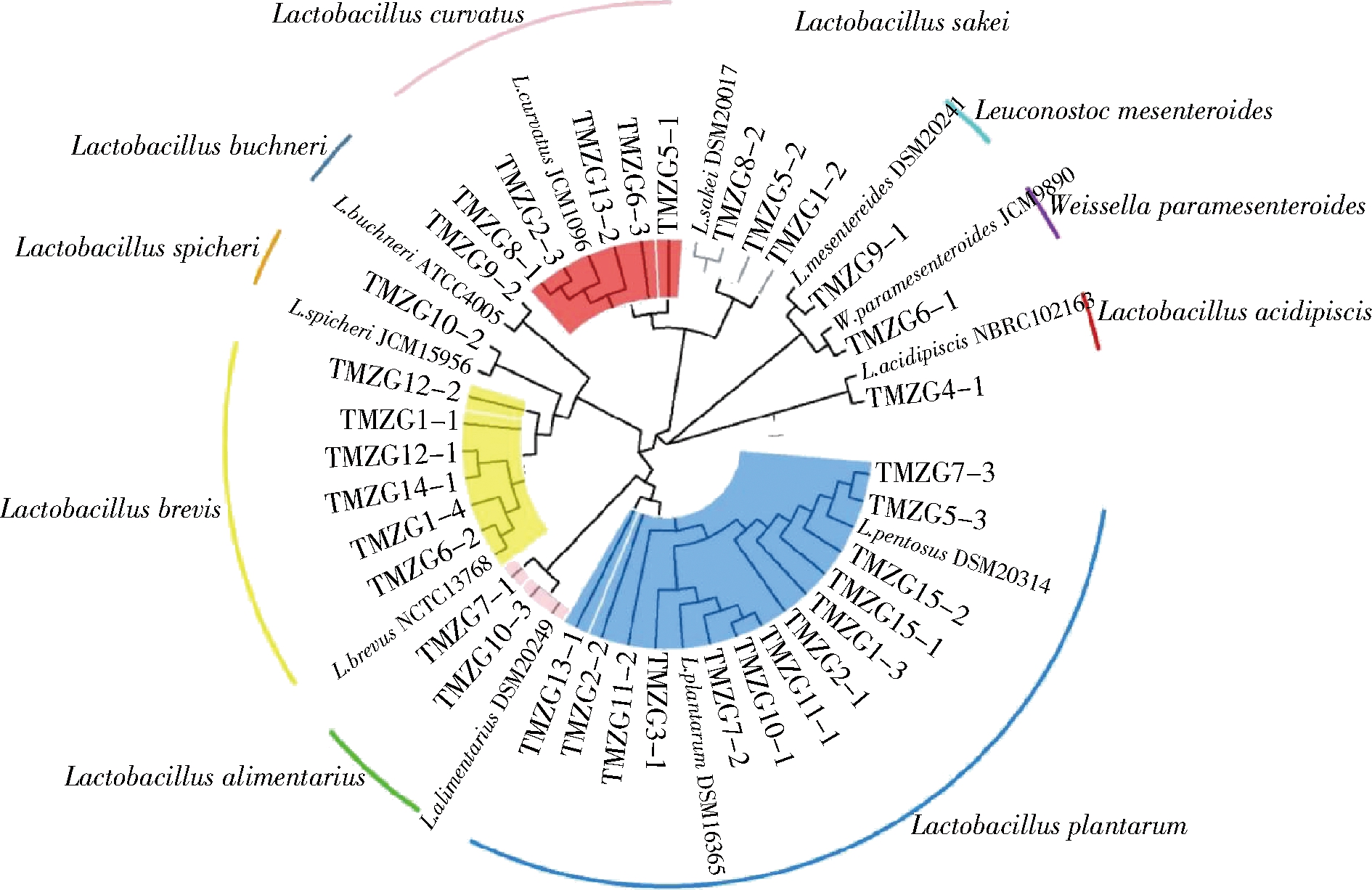
图7 鲊广椒中乳酸菌分离菌株系统发育树
Fig.7 Phylogenetic tree of lactic acid bacteria isolates from Zha-chili
由图7可知,所有分离株共被鉴定到10个种,其中有13株鉴定为L.plantarum(植物乳杆菌,38.24%),6株鉴定为L.brevis(短乳杆菌,17.65%),5株鉴定为L.curvatus(弯曲乳杆菌,14.7%),3株鉴定为L.sakei(清酒乳杆菌,8.82%),2株鉴定为L.alimentarius(食品乳杆菌,5.88%),各有1株鉴定为L.spicheri(辣味乳杆菌,2.94%)、L.acidipiscis(嗜酸乳杆菌,2.94%)、W.paramesenteroides(类肠膜魏斯氏菌,2.94%)、L.buchneri(布氏乳杆菌,2.94%)和Leuconostoc mesenteroides(肠膜明串珠菌,2.94%)。由此可见,L.plantarum为天门地区鲊广椒中的优势乳杆菌。
3 结论
本研究采用高通量测序和传统微生物学相结合的手段对天门地区鲊广椒细菌群落结构进行了解析,对其蕴含的乳酸菌类群进行了分离鉴定。结果发现,15份天门鲊广椒样品被划分为2个聚类,聚类Ⅰ中的细菌主要为隶属于Firmicutes的Lactobacillus,而聚类Ⅱ中的细菌主要为隶属于Proteobacteria的Klebsiella和Kosakonia以及隶属于Firmicutes的Tetragenococcus,其中L. plantarum、L. brevis和L. curvatus为鲊广椒中的优势乳酸菌。由此可见,天门地区鲊广椒的细菌群落结构复杂多样,乳酸菌类群丰富,通过本研究的开展可为后续鲊广椒产业化的实现和产品的创新提供一定的理论参考和菌种支持。
[1] 李娜, 张苗苗, 舒娜, 等.咸丰和当阳地区鲊广椒细菌群落结构差异性研究[J].中国酿造, 2020, 39(10):48-53.
LI N, ZHANG M M, SHU N, et al.Difference of bacterial community structure of Zhaguangjiao in Xianfeng and Dangyang region[J].China Brewing, 2020, 39(10):48-53.
[2] 尚雪娇, 雷炎, 代凯文, 等.当阳地区鲊广椒品质的评价[J].食品研究与开发, 2018, 39(24):33-38.
SHANG X J, LEI Y, DAI K W, et al.Quality evaluation of Zhaguangjiao samples collected from Dangyang[J].Food Research and Development, 2018, 39(24):33-38.
[3] 王玉荣, 代凯文, 沈馨, 等.鲊广椒真菌多样性及其对滋味品质影响的评价[J].食品科学, 2018, 39(18):173-178.
WANG Y R, DAI K W, SHEN X, et al.Characterization of fungal microflora and its influence on taste quality of Zhaguangjiao, a Chinese traditional fermented chili product[J].Food Science, 2018, 39(18):173-178.
[4] 王玉荣, 沈馨, 董蕴, 等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械, 2018, 34(4):25-30.
WANG Y R, SHEN X, DONG Y, et al.Characterization of bacterial microflora and their functions on flavor quality[J].Food & Machinery, 2018, 34(4):25-30.
[5] CAI W C, WANG Y R, HOU Q C, et al.PacBio sequencing combined with metagenomic shotgun sequencing provides insight into the microbial diversity of Zha-chili[J].Food Bioscience, 2021, 40:100884.
[6] 崔梦君, 王玉荣, 葛东颖, 等.遵义地区莽椒细菌多样性及PICRUSt基因功能预测分析[J].食品与发酵工业, 2020, 46(4):106-112.
CUI M J, WANG Y R, GE D Y, et al.Study on bacterial diversity of Mangjiao in Zunyi region and PICRUSt-based predicted metagenomic analysis[J].Food and Fermentation Industries, 2020, 46(4):106-112.
[7] 李娜, 崔梦君, 马佳佳, 等.基于Illumina MiSeq测序和传统可培养方法的洪湖鲊广椒乳酸菌多样性研究[J].食品与发酵工业, 2021, 47(4):110-115;122.
LI N, CUI M J, MA J J, et al.Lactic acid bacterial diversity of Zha-chili in Honghu County based on Illumina Miseq sequencing and traditional culture method[J].Food and Fermentation Industries, 2021, 47(4):110-115;122.
[8] 郭壮, 葛东颖, 尚雪娇, 等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技, 2018, 39(22):93-98;106.
GUO Z, GE D Y, SHANG X J, et al.Comparative analysis on the diversity of bacterial microflora in degenerated and normal pit mud[J].Science and Technology of Food Industry, 2018, 39(22):93-98;106.
[9] CAPORASO J G, LAUBER C L, WALTERS W A, et al.Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J].The ISME Journal, 2012, 6(8):1 621-1 624.
[10] CALLAHAN B J, MCMURDIE P J, HOLMES S P.Exact sequence variants should replace operational taxonomic units in marker-gene data analysis[J].The ISME Journal, 2017, 11(12):2 639-2 643.
[11] HAAS B J, GEVERS D, EARL A M, et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Research, 2011, 21(3):494-504.
[12] ANUKAM K.Effects of ampicillin on the gut microbiome of an adult male as determined by 16S rRNA V4 metagenomics sequencing and greengenes bioinformatics suite[J].Journal of Advances in Microbiology, 2018, 7(4):1-18.
[13] COLE J R, WANG Q, FISH J A, et al.Ribosomal Database Project:Data and tools for high throughput rRNA analysis[J].Nucleic Acids Research, 2014, 42(Database issue):D633-D642.
[14] AGRAWAL S, KINH C T, SCHWARTZ T, et al.Determining uncertainties in PICRUSt analysis:An easy approach for autotrophic nitrogen removal[J].Biochemical Engineering Journal, 2019, 152:107328.
[15] 向凡舒, 黄怡, 陈小艳, 等.建始地区酸豇豆盐水细菌多样性及乳酸菌分离鉴定[J].中国酿造, 2020, 39(10):61-65.
XIANG F S, HUANG Y, CHEN X Y, et al.Bacterial diversity of fermented cowpea brine collected from Jianshi area and identification of lactic acid bacteria isolates[J].China Brewing, 2020, 39(10):61-65.
[16] 雷炎, 马佳佳, 雷敏, 等.恩施鲊广椒乳酸菌的分离鉴定及其对挥发性风味物质的影响[J].中国酿造, 2019, 38(7):126-130.
LEI Y, MA J J, LEI M, et al.Isolation and identification of lactic acid bacteria from Zhaguangjiao of Enshi and its effect on volatile flavor substances[J].China Brewing, 2019, 38(7):126-130.
[17] ARUMUGAM M, RAES J, PELLETIER E, et al.Enterotypes of the human gut microbiome[J].Nature, 2011, 473(7346):174-180.
[18] ZHANG J C, GUO Z, XUE Z S, et al.A Phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles, geography and ethnicities[J].The ISME Journal, 2015, 9(9):1 979-1 990.
[19] CHOBY J E, HOWARD-ANDERSON J, WEISS D S.Hypervirulent Klebsiella pneumoniae:Clinical and molecular perspectives[J].Journal of Internal Medicine, 2020, 287(3):283-300.
[20] VALENTINI M, GONZALEZ D, MAVRIDOU D A, et al.Lifestyle transitions and adaptive pathogenesis of Pseudomonas aeruginosa[J].Current Opinion in Microbiology, 2018, 41:15-20.
[21] TIMILSINA S, POTNIS N, NEWBERRY E A, et al.Xanthomonas diversity, virulence and plant-pathogen interactions[J].Nature Reviews Microbiology, 2020, 18(8):415-427.
[22] 王玉荣, 孙永坤, 代凯文, 等.基于单分子实时测序技术的3个当阳广椒样品细菌多样性研究[J].食品工业科技, 2018, 39(2):108-112;118.
WANG Y R, SUN Y K, DAI K W, et al.Study on the diversity of bacteria microflora in 3 Zhaguangjiao samples of Dangyang area by single molecule real-time sequencing technique[J].Science and Technology of Food Industry, 2018, 39(2):108-112;118.
[23] MAO Y, ZHANG X J, XU Z H.Identification of antibacterial substances of Lactobacillus plantarum DY-6 for bacteriostatic action[J].Food Science & Nutrition, 2020, 8(6):2 854-2 863.
[24] 杨爽, 肖瑜.蛹虫草固态发酵对玉米粉加工性质及消化性的影响[J].食品工业科技, 2021, 42(11):122-127.
YANG S, XIAO Y.Effect of solid-state fermentation with Cordyceps militaris on processing and digestive properties of corn flour[J].Science and Technology of Food Industry, 2021, 42(11):122-127.
[25] 张大力, 冯艳鹤, 刘炳利, 等.自然发酵糯玉米中细菌多样性分析及纯菌种对其加工特性的影响[J].食品科学, 2018, 39(18):109-114.
ZHANG D L, FENG Y H, LIU B L, et al.Analysis of bacterial diversity in naturally fermented waxy corn and effects of pure fermentation on its processing characteristics[J].Food Science, 2018, 39(18):109-114.