安全性是微生物菌种应用于工业生产的重要前提,抗生素耐药性、产毒与致病性是评价菌种安全性的重要方面。2002年,联合国粮食及农业组织(Food and Agriculture Organization of the United Nations,FAO)/世界卫生组织(World Health Organization,WHO)发布的适用于食品用益生菌的评估指南要求:对于食品用益生菌,应考虑菌种在抗生素耐药性、特定代谢活性物质、人体副作用、有害事件的流行病监测以及毒素产生等方面的安全性;任何给定的益生菌在抗生素耐药性转移、机会性毒力方面不应存在重大风险[1]。欧盟“安全资格认证(Qualified Presumption of Safety,QPS)”评价程序[2]和美国“一般认为安全(Generally Recognized as Safe,GRAS)”认证[3]同样强调了针对微生物菌种应开展抗生素耐药性、产毒与致病性分析,以评估其安全性。随着基因测序技术的不断更迭,人们对菌种安全性的理解也在不断发生变化和趋于完善。2020年益生菌的科学共识中指出,对于新属种的益生菌菌株,应通过全基因组测序生物信息分析阐述评估其所携带的耐药基因、致病基因等特征[4]。基于基因测序技术的生物信息学分析能够筛查菌种中与抗生素耐药、毒力和致病性等相关的基因,挖掘菌种潜在安全性风险的遗传机制,为评价微生物菌种的安全性提供一种便捷手段。
目前,芽胞杆菌属(Bacillus)内已有多个物种被列入欧盟QPS名单[5]和国际乳品联合会IDF 495—2018《发酵食品用微生物菌种调查及安全性声称》公报[6],被证明是安全的。在我国,尚无芽胞杆菌物种被列入《可用于食品的菌种名单》,但该属内多个物种曾被报道用于我国传统发酵食品中[7],芽胞杆菌新菌种在使用前需进行全面的安全性评价。黄水芽胞杆菌Bacillus aquiflavi 3H-10是从我国四川省宜宾地区传统发酵浓香型白酒黄水样品中分离的1株新种菌株[8],可产生蛋白酶以及苯甲醛、2,5-二甲基吡嗪、己酸乙酯等多种风味物质(暂未发表),在白酒发酵实际生产中具有应用潜力。本文通过测定黄水芽胞杆菌3H-10的基因组完成图序列,针对抗生素耐药性、产毒与致病性开展相关安全性基因分析,并结合抗生素敏感性试验和小鼠毒力试验,对黄水芽胞杆菌3H-10的安全性进行综合评价。
1 材料与方法
1.1 实验材料
1.1.1 实验菌种及培养基
本研究所用菌种黄水芽胞杆菌3H-10(=CICC 24755T),中国工业微生物菌种保藏管理中心,前期由本实验室分离自我国四川省宜宾地区传统发酵浓香型白酒的黄水样品[8];所用培养基为大豆酪蛋白TSB培养基、大豆酪蛋白琼脂TSA培养基。
1.1.2 仪器与设备
隔水式培养箱,上海一恒科学仪器有限公司;恒温培养振荡器,上海智城分析仪器制造有限公司;Eppendorf微量可调移液器、5424R小型台式冷冻离心机,德国Eppendorf公司;HG-50高压灭菌锅,日本 HIRAYAMA公司;AC2-4S1生物安全柜,新加坡Esco公司;BioDrop核酸蛋白分析仪,豪沃生物科技(上海)有限公司;Qubit 3.0核酸荧光定量仪,美国ThermoFisher Scientific公司;Hiseq2500基因测序仪,美国Illumina公司;PacBio Sequel基因测序仪,美国PacBio公司。
1.1.3 试剂
TSA琼脂,美国BD公司;溴代十六烷基三四胺(cetyl trimethyl ammonicum bromide,CTAB)、Tris-饱和酚,美国Sigma公司;氯仿,北京市通广精细化工公司;异戊醇,西陇化工股份有限公司;异丙醇,北京化工厂;λDNA/Hind Ⅲ分子量标记、Qubit dsDNA HS Assay Kit试剂盒,美国Thermo Fisher Scientific公司;GoldView、琼脂糖,北京全式金生物技术有限公司;抗生素E-test药敏纸片,法国生物梅里埃公司;SPF级ICR小鼠(许可证编号SCXK(京)2016-0006),北京维通利华实验动物技术有限公司。
1.2 实验方法
1.2.1 菌株DNA提取及质检
取TSB于37 ℃ 200 r/min培养3 d的菌悬液,8 000 r/min离心收集约1 g湿菌体至1.5 mL离心管中,加入600 μL CTAB裂解液,65 ℃水浴45 min,13 000 r/min离心10 min;取上清液于一新离心管中,加入等体积的V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1,充分混匀,12 000 r/min离心10 min;取上清液,加等体积的V(氯仿)∶V(异戊醇)=24∶1,充分混匀,12 000 r/min离心10 min;取上清液,加等体积的预冷异丙醇试剂,-20 ℃沉淀30 min,12 000 r/min离心20 min;收集沉淀,75%乙醇冲洗,室温放置使DNA干燥;使用100 μL TE溶解DNA,-20 ℃保存备用。使用Qubit 3.0核酸荧光定量仪测定DNA样本浓度;使用BioDrop核酸蛋白分析仪测定DNA样品的A260/A280和A260/A230值,判断DNA纯度。
1.2.2 DNA测序、数据质控与组装
采用Illumina Hiseq 2500基因测序仪和Pacbio Sequel基因测序仪分别进行小片断和大片断文库测序。对原始下机数据进行如下质控过滤:去除质量值连续≤20的碱基数达到40%的reads、去除含N碱基比例≥10%的reads、去除接头和重复污染。采用proovread v2.12软件对下机数据进行纠错,并采用Falcon v0.3.0和GATK v6-13软件分别进行序列组装和单碱基纠错,得到高质量的基因组完成图。
1.2.3 基因预测与注释
采用Glimmer v3.02软件对组装结果进行基因预测,预测基因序列与COG(Cluster of Orthologous Groups of proteins)和KEGG(Kyoto Encyclopedia of Genes and Genomes)等数据库进行比对,获得相应基因的注释信息。
1.2.4 耐药基因检测
通过与Comprehensive Antibiotic Resistance Database (CARD)[9]和ResFinder[10]数据库比对,检测菌株基因组中的潜在耐药基因。通过RGI软件将预测基因的氨基酸序列与CARD数据库的比对,以“perfect”和“strict”算法输出结果作为耐药基因的检测结果;与ResFinder数据库比对时,设置序列相似性≥90%,序列覆盖度≥60%。
1.2.5 毒力基因检测
通过与Virulence Factors of Pathogenic Bacteria (VFDB)[11]和VirulenceFinder[12]数据库比对,检测菌株基因组中的潜在毒力基因。与VFDB数据库比对时,使用的软件为diamond,E值≤e-5,序列相似性≥70%,序列覆盖度≥70%;与VirulenceFinder数据库比对时,设置序列相似性≥90%,序列覆盖度≥60%。
1.2.6 抗生素敏感性测定
使用生理盐水制备并调整菌悬液至0.5麦氏浊度,使用无菌拭子沾取菌悬液并均匀涂布于CAMHB固体平板上,待琼脂表面干燥后,使用无菌镊子取抗生素E-test试剂条置于平板表面,37 ℃培养72 h,观察结果。椭圆形抑菌环与E-test试条的交界点药物浓度即为最低抑菌浓度(minimum inhibitory concentration,MIC)值。根据美国临床和实验室标准协会CLSI M45—2016《Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria:approved guideline》[13]共选取10种、9类常见抗生素用于药物敏感性实验。菌种耐药性的判断标准参照欧盟指南《Guidance on the characterisation of microorganisms used as feed additives or as production organisms》[14]与CLSI M45—2016[13]。质控菌株为金黄色葡萄球菌CICC 10473T(=ATCC 29213T)。
1.2.7 小鼠毒力试验
将菌种转接于TSB培养基中,置于37 ℃,好氧培养72 h至计数约108 CFU/mL,作为培养物原液备用。取部分培养物原液浓缩5倍,作为培养物5倍浓缩液备用。选用SPF级ICR小鼠80只,雌雄各半,在北京维通利华实验动物技术有限公司动物房[许可证编号为SYXK(京)2017-0022]饲养。雄性小鼠体重16.0~25.0 g,雌性小鼠体重16.0~25.0 g。ICR小鼠80只,雌雄各半,分别随机分为4组,每组10只。剂量组设置为:培养基原液对照组,培养物原液组,培养基5倍浓缩对照组,培养物5倍浓缩组。将受试物以20.0 mL/kg BW剂量给小鼠连续灌胃3 d,观察7 d,记录实验过程中小鼠的体重变化、中毒表现及死亡情况。使用SPSS 18.0软件进行数据分析,培养物组与对照组间定量资料的比较用两样本t检验。
2 结果与分析
2.1 黄水芽胞杆菌3H-10全基因组概况
通过Illumina和PacBio平台分别测序获得569×和788×原始下机数据,经数据质控过滤与拼接,获得菌株3H-10的基因组信息。其基因组为一条共价闭合环状的DNA(图1-a),大小3 806 094 bp,G+C含量为35.66%(摩尔百分含量)。基因组中共预测到4 031个蛋白质编码基因、81个tRNA基因、各9个5S/16S/23S rRNA基因、16个sRNA基因。通过与多个数据库进行比对,最终注释基因3 324个。黄水芽胞杆菌3H-10的基因组大小与G+C含量符合芽胞杆菌属物种基因组的一般特征[15]。与近缘物种基因组相比,黄水芽胞杆菌3H-10在基因组大小和G+C含量等方面与Weizamannia ginsengihumi Gsoil 114T较相似(表1)。

a-菌株3H-10基因组圈图,从外到内8圈依次代基因组大小、预测基因、正链基因、负链基因、tRNA、rRNA、sRNA、GC含量和GC偏移; b-菌株3H-10预测基因COG功能分类
图1 黄水芽胞杆菌3H-10的基因组完成图与预测基因COG功能分类
Fig.1 Circos plot for the complete genome of B.aquiflavi 3H-10 and the COG function classification of the predicted genes
对黄水芽胞杆菌3H-10基因功能进行分析,利用COG进行基因注释与功能分类,共注释基因2 385个(59.16%),可分为24个功能组(图1-b),其中预测为一般功能基因分组最多。黄水芽胞杆菌3H-10基因组中涉及氨基酸运输与代谢,翻译、核糖体结构与生物合成,转录、辅酶转运与翻译、脂转运与代谢,能量产生与转换等的注释基因数相对较多,在150~250。通过与KEGG数据库进行比对发现,菌株3H-10中存在较完整的氨基酸、脂类、碳水化合物、辅因子与维生素的代谢途径,并包含对芳香族、萜类和聚酮等化合物的降解途径,推测菌株3H-10具有产生多种次级代谢产物的能力,这可能与白酒酿造中丰富的风味物质形成相关。
表1 黄水芽胞杆菌3H-10及其近缘物种基因组的基本特征
Table 1 Basic information of the assembled genomes of B.aquiflavi 3H-10 and closely relatives

菌株基因组大小/bpScaffold数量/个G+C含量/%蛋白质编码基因/个rRNA基因个数/个tRNA基因个数/个NCBI登录号分离源B.aquiflavi 3H-10T3 806 094135.66 4 0312781CP082780中国浓香型白酒黄水样品[8]Bacillus mesophilus SA4T4 268 4312636.80 4 0901594JAAIWM000000000废弃海洋太阳能盐田土壤[16]Weizmannia ginsengihumi Gsoil 114T3 738 5586336.00 3 4021986JAAIWK000000000人参田土壤[17]Margalitia shackletonii LMG 18435T5 154 1429636.60 4 84523102JAAIWL000000000火山土[17]Niallia circulans NBRC 13626T5 097 2465935.60 4 8511164BCVE00000000土壤[17]
2.2 耐药性分析
2.2.1 耐药基因分析
抗生素依据其化学结构可归为多种类别,包括常见的氨基糖苷类、β-内酰胺类、氟喹诺酮类、叶酸途径拮抗剂、磷霉素类、糖肽类、林可酰胺类、大环内酯类、硝基咪唑类、多粘菌素类、假单胞菌酸类、利福霉素、链阳霉素类、四环素类、苯丙醇类和磺胺类等。目前,针对常见抗生素已有大量抗生素耐药基因被报道(表2),并收录到相关耐药基因数据库中。CARD和ResFinder数据库是关于耐药基因及其产物和相关表现型的生物信息学数据库,涵盖了当前已发现的多数抗生素耐药基因[9-10]。通过将黄水芽胞杆菌3H-10的基因组序列与这两个数据库比对,均未检测到上述耐药基因(表2)。
表2 常见抗生素、耐药基因及黄水芽胞杆菌3H-10在耐药基因数据库的比对结果
Table 2 Common antibiotics, antimicrobial resistance genes and the analysis result of the genome of B.aquiflavi 3H-10 against related antimicrobial resistance databases

抗生素类别常见抗生素常见耐药基因CARD数据库比对结果ResFinder数据库比对结果氨基糖苷类链霉素、卡那霉素、庆大霉素ant(4)-Ib,aph(3)-IV,aac(6′)-aph(2″), aph(3′)-III, strA, strB--β-内酰胺类青霉素、头孢类、头霉素类、碳青霉烯类blaZ, TEM-1, mexAB-oprM, NDM-1--氟喹诺酮类环丙沙星、左氧氟沙星norA, norB, pmrA, mexAB-oprM--叶酸途径拮抗剂甲氧苄啶dfrA18, mexPQ-opmE--磷霉素类磷霉素fomA, fosC--多肽类万古霉素、替考拉宁、多粘菌素vanA, murG, vanX, bacA, bcrA--林可酰胺类克林霉素lsaA, ermC, lmrC, lnuA--大环内酯红霉素ermA, ermB,ermC,ermD,ermK, erm34--硝基咪唑类甲硝唑msbA--假单胞菌酸类假单胞菌酸mupA, ileS--利福霉素利福平rpoB, efrAB--链阳霉素类链阳霉素lmrP--四环素类四环素tetK, tetL, tetM, tetW, tet42--苯丙醇类氯霉素cat86, cat(pC194), fexA--磺胺类甲氧苄啶mexPQ-opmE, oqxAB--
2020年CHINET中国细菌耐药监测显示,临床分离菌株对常见抗菌药物的耐药率呈现增长趋势。芽胞杆菌在自然界中广泛分布,其生存环境包括土壤、水、食物及临床等。该属中常见的耐药基因包括氨基糖苷类耐药基因ant(4′)-Ib、aph(3′)-III和aph(3′)-IV,氯霉素耐药基因cat86和fexA,红霉素耐药基因ermD、ermK和erm34,四环素耐药基因tetK、tetL、tetM、tetW和tet42、杆菌肽耐药基因bacA和bcrA等[18-19]。通过基于序列比对的靶向分析,在黄水芽胞杆菌3H-10基因组中未检测到这些基因。
2.2.2 抗生素敏感性测定
检测结果显示,菌株黄水芽胞杆菌3H-10对所选全部抗生素,即青霉素、美罗培南、万古霉素、庆大霉素、红霉素、四环素、环丙沙星、甲氧苄啶-磺胺甲噁唑、氯霉素、利福平,均敏感(表3)。实验所用9类、10种抗生素依据CLSI M45—2016[13]选取,基本覆盖世界卫生组织所发布《对人类医学至关重要的抗微生物药物》中重要抗生素大类,其中头孢菌素类(第三代、四代和五代)、糖肽类、大环内酯类和酮内酯类、多粘菌素类、喹诺酮类被列为最高优先至关重要抗微生物药物[20]。芽胞杆菌目前未被列入世界卫生组织[21]和我国CHINET对细菌耐药性监控的微生物之列;有研究报道,部分芽胞杆菌对链霉素、克林霉素、氯霉素、卡那霉素、红霉素、四环素具有耐药性[18-19]。抗生素敏感性试验显示,菌株3H-10对常见重要抗生素无耐药性,这与该菌株基因组中未检测到相关耐药基因的结果一致。综合耐药基因型与表型结果可见,黄水芽胞杆菌3H-10自身无横向传播耐药性或耐药基因的风险。
表3 黄水芽胞杆菌3H-10对常见抗生素的敏感性检测结果
Table 3 Antimicrobial susceptibility test result of B.aquiflavi 3H-10 to common antibiotics
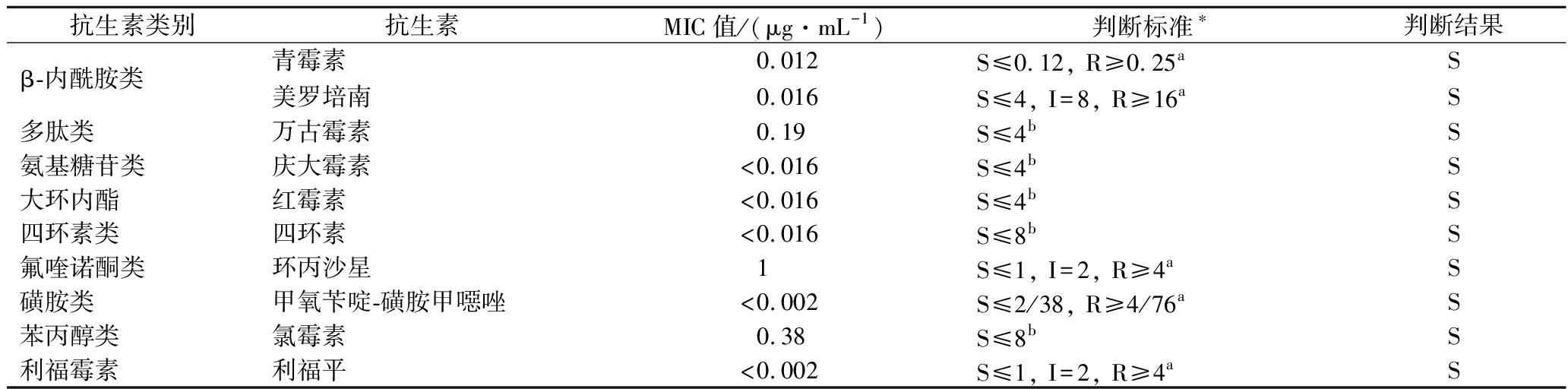
抗生素类别抗生素MIC值/(μg·mL-1)判断标准∗判断结果β-内酰胺类青霉素 0.012S≤0.12, R≥0.25aS美罗培南0.016S≤4, I=8, R≥16aS多肽类万古霉素0.19S≤4bS氨基糖苷类庆大霉素<0.016S≤4bS大环内酯红霉素<0.016S≤4bS四环素类四环素<0.016S≤8bS氟喹诺酮类环丙沙星1S≤1, I=2, R≥4aS磺胺类甲氧苄啶-磺胺甲噁唑<0.002S≤2/38, R≥4/76aS苯丙醇类氯霉素0.38S≤8bS利福霉素利福平<0.002S≤1, I=2, R≥4aS
注*:S,susceptible,敏感;I,intermediate,中介;R,resistant, 耐药;a判断标准来自CLSI M45—2016[13];b判断标准来自欧盟指南[14]
2.3 毒力分析分析
2.3.1 毒力基因分析
毒力是菌种安全性评价的重要方面。细菌的毒力因子主要可分为4大类:与宿主黏附和入侵相关、细菌分泌系统和效应物、毒素以及铁离子系统[11]。VFDB和VirulenceFinder数据库收录了当前已报道的大量细菌毒力因子基因序列[11-12],被国内外与菌种安全评价相关的多个标准和指南所推荐。通过将黄水芽胞杆菌3H-10的基因组序列与这两个数据库进行比对,未检测到上述毒力基因(表4)。
表4 黄水芽胞杆菌3H-10基因组中毒力基因的检测结果
Table 4 Detection of virulence genes in the genome of B.aquiflavi 3H-10

毒力因子芽胞杆菌属常见毒力因子相关毒力基因VFDB数据库比对结果VirulenceFinder数据库比对结果侵袭性毒力因子1) 黏附炭疽杆菌S层蛋白质AbslA/yuaB, badA--2) 入侵//--3) 产生毒素炭疽毒素、呕吐毒素、溶血素BL、非溶血性肠毒素、细胞毒素KCya,lef, pagA, cesA, cesB, hblA,hblC, hblD等--4) 基于朊纤蛋白丝的运动//--5) 分泌系统Ⅶ型分泌系统BAS2037, BAS2042, essC;esxB, esxL等--防御性毒力因子1) 抗吞噬荚膜capA, capB, capC, capE, dep/capD等--2) 抗蛋白质水解//--3) 细胞代谢//--4) 相转变//--5) 血清抗性//--7) 免疫球蛋白酶//--8) 应激蛋白//--9) 补充蛋白酶//--非特异性毒力因子/1) 铁吸收系统//--2) 镁吸收系统//--3) 胞外酶//--毒力相关基因的调控1) 调控子炭疽毒素激活子AtxAatxA--
在芽胞杆菌中,蜡样芽胞杆菌常被报道携带毒力基因,包括与呕吐毒素相关的非核糖体多肽合成酶系(NRPS)基因、与腹泻毒素相关的溶血素BL基因、非溶血性的肠毒素Nhe基因(nheA、nheB、nheC)、肠毒素FM基因(entFM)、肠毒素T基因(bceT)和细胞毒素K基因(cytK)[22-23]。通过基于序列比对的靶向分析,在3H-10基因组中未检测到这些毒力基因。
2.3.2 小鼠毒力试验
小鼠毒力试验是目前检测菌株是否存在安全风险最直接、有效的生理学证据,在我国食品、保健食品、饲料等领域微生物安全性评价中占有重要地位。我国《保健食品原料用菌种安全性检验与评价技术指导原则》(2020年版)、《食品安全国家标准食品用菌种安全性评价程序(征求意见稿)》、《TCSWSL 016—2019 水产动物饲用乳酸菌筛选标准》、《团体标准TCSWSL 017—2019 水产动物饲用芽孢杆菌筛选标准》等均要求针对菌种开展小鼠毒力试验。本文利用菌株黄水芽胞杆菌3H-10培养原液和5倍浓缩液对SPF级ICR小鼠进行灌喂,观察菌种对小鼠体重、毒性反应的影响。表5和表6分别显示3H-10培养物对雄、雌小鼠体重的影响,可见雄、雌性小鼠的初始体重和终体重在培养物组与其相应对照组间比较均无显著性差异(P>0.05),即在试验期间菌株3H-10培养物对雄、雌性小鼠的体重无影响。
表5 黄水芽胞杆菌3H-10培养物对雄性小鼠体重的影响
Table 5 Effect of B.aquiflavi 3H-10 culture to weight of male mice
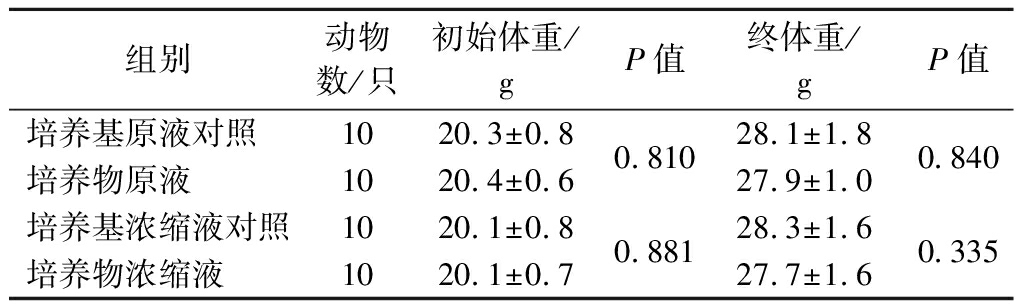
组别动物数/只初始体重/gP值终体重/gP值培养基原液对照1020.3±0.8培养物原液1020.4±0.60.81028.1±1.827.9±1.00.840培养基浓缩液对照1020.1±0.8 培养物浓缩液1020.1±0.70.881 28.3±1.627.7±1.60.335
表6 黄水芽胞杆菌3H-10培养物对雌性小鼠体重的影响
Table 6 Effect of B.aquaflavi 3H-10 culture to weight of female mice

组别动物数/只初始体重/gP值终体重/gP值培养基原液对照1019.9±0.8培养物原液1019.8±0.70.70825.2±1.224.7±1.70.513培养基浓缩液对照1019.7±0.5培养物浓缩液1019.8±0.50.66724.8±1.625.7±1.70.247
表7为菌株3H-10培养物对小鼠急性毒性的影响。结果显示,将菌株3H-10培养物原液和5倍浓缩液,以20.0 mL/kg BW的剂量给小鼠连续灌胃3 d,观察7 d,未观察到受试小鼠有毒性反应或死亡。结合毒力基因与小鼠毒力试验结果可见,菌株3H-10自身无毒力或致病性风险。
表7 黄水芽胞杆菌3H-10培养物对小鼠急性毒性作用
Table 7 Acute toxicity effect of B.aquiflavi 3H-10 culture to mice

组别雄性动物中毒反应雌性动物中毒反应动物数/只反应数/只死亡数/只动物数/只反应数/只死亡数/只培养基原液对照10001000培养物原液10001000培养基浓缩液对照10001000培养物浓缩液10001000
3 结论
近年来,高通量测序技术飞速发展,基因组测序逐渐成为微生物鉴定、分型溯源和功能挖掘等分析中的常规手段,也使得从基因层面评估菌种的安全性、解析风险因素的遗传机制成为可能。目前,国内外已有多个关于抗生素耐药基因、毒力与致病基因的数据库。其中,CARD是关于耐药基因及其产物和相关表现型的生物信息学数据库,于2013年由加拿大McMaster大学开发[9]。目前包含263种病原菌,14 795个WGS拼接序列,175 753条等位序列,覆盖与28类、336种抗生素相关的3 014条耐药基因序列。ResFinder数据库于2012年由丹麦技术大学开发与维护,目前包括与27类、127种抗生素相关的2 817条耐药序列[10]。VFDB数据库由中国医学科学院和中国协和医科大学于2005年建立,目前包括74种病原菌属、951株细菌、1 284种毒力因子、32 827条毒力相关基因序列[11]。VirulenceFinder由丹麦技术大学开发,覆盖4种常见致病菌(李斯特菌、金黄色葡萄球菌、大肠杆菌和肠球菌)中与7种主要毒力因子相关的502种毒力基因[12]。这些与安全性相关数据库为筛查菌种全基因组序列中的耐药、毒力与致病性等风险基因提供了重要的数据基础,具有重要应用价值。SULTHANA等[24]对枯草芽胞杆菌Bacillus subtilis UBBS-14进行全基因组测序和基因安全性评价,发现该菌株不携带任何含抗生素耐药性基因的质粒,不包含任何推定的有害毒力因子编码基因,从而在基因组水平上证实了该菌株作为益生菌的安全性。SAROJ等[25]对凝结芽胞杆菌Bacillus coagulans LBSC (DSM 17654)进行全基因组测序,并对耐药基因、产生物胺、毒力因子基因等安全相关基因开展分析,从分子水平上证明该菌株的安全性。基因安全性分析是菌种安全性评价的重要方面,与抗生素敏感性、小鼠毒力试验等传统分析方法有机结合、互为补充,可实现对工业用菌种全面而系统的安全性评价。
本研究对分离自我国四川省宜宾地区的黄水芽胞杆菌3H-10进行全基因组测序,并开展基于基因型与表型的抗生素耐药性、毒力与致病性分析,全面评价菌种的安全性。黄水芽胞杆菌3H-10基因组中不含有耐药基因,对常见抗生素敏感;基因组中无毒力相关基因,受试小鼠不产生毒性反应或死亡反应。因此推测,菌株3H-10无横向转移耐药基因的风险,无毒力或人体致病性。黄水芽胞杆菌3H-10分离自我国传统发酵浓香型白酒黄水样品,前期研究发现其具有较好的产蛋白酶活性,并可产生多种次级代谢风味物质,在浓香型白酒发酵中具有一定应用潜力。本研究对黄水芽胞杆菌3H-10在抗生素耐药性、毒力与致病性方面的安全性评估,为该菌株在浓香型白酒发酵中的实际应用奠定了良好基础。
[1] FAO/WHO, Joint FAO/WHO Working group report on drafting guidelines for the evaluation of probiotics in food[R].London, Ontario, Canada,2002, May.
[2] EUROPEAN FOOD SAFETY AUTHORITY (EFSA).Introduction of a Qualified Presumption of Safety (QPS) approach for assessment of selected microorganisms referred to EFSA - Opinion of the Scientific Committee[J].EFSA Journal, 2007, 5(12):587.
[3] United States Food and Drug Administration (FDA).Guidance for industry:enzyme preparations:recommendations for submission of chemical and technological data for food additive petitions and GRAS notices[A].2010.
[4] 益生菌的科学共识(2020年版)[J].中国食品学报, 2020, 20(5):303-307.
Scientific consensus on probiotics(2020)[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(5):303-307.
[5] KOUTSOUMANIS K, ALLENDE A, ALVAREZ-ORDONE A, et al.Scientific opinion on the update of the list of QPS recommended biological agents intentionally added to food or feed as notified to EFSA (2017—2019)[J].EFSA Journal, 2019, 15(3):e04664.
[6] BOURDICHON F, ALPER I, BIBILONI R, et al.Inventory of microbial food cultures with safety demonstration in fermented food products[R].Update of the Bulletin the International Dairy Federation No 455-2012,2018.
[7] 姚粟, 于学健, 白飞荣, 等.中国传统发酵食品用微生物菌种名单的研究[J].食品与发酵工业, 2017, 43(9):238-258.
YAO S, YU X J, BAI F R, et al.Research on the inventory of microbial species in Chinese traditional fermented foods[J].Food and Fermentation Industries, 2017, 43(9):238-258.
[8] XIE J Y, CHENG K, ZHAO D, et al.Bacillus aquiflavi sp.nov., isolated from yellow water of strongly flavored Chinese Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(5):3 406-3 412.
[9] ALCOCK B P, RAPHENYA A R, LAU T T Y, et al.CARD 2020:Antibiotic resistome surveillance with the comprehensive antibiotic resistance database[J].Nucleic Acids Research, 2019, 48(D1):D517-D525.
[10] BORTOLAIA V, KAAS R S, RUPPE E, et al.ResFinder 4.0 for predictions of phenotypes from genotypes[J].Journal of Antimicrobial Chemotherapy, 2020, 75(12):3 491-3 500.
[11] LIU B, ZHENG D D, JIN Q, et al.VFDB 2019:A comparative pathogenomic platform with an interactive web interface[J].Nucleic Acids Research, 2018, 47(D1):D687-D692.
[12] MALBERG TETZSCHNER A M, JOHNSON J R, JOHNSTON B D, et al.In silico genotyping of Escherichia coli isolates for extraintestinal virulence genes by use of whole-genome sequencing data[J].Journal of Clinical Microbiology, 2020, 58(10):e01269-e01220.
[13] CLSI document M45-2 016.Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria:approved guideline-second edition[S].Clinical and Laboratory Standards Institute (CLSI), 2016.
[14] RYCHEN G, AQUILINA G, AZIMONTI G, et al.Guidance on the characterisation of microorganisms used as feed additives or as production organisms[J].EFSA Journal, 2018, 16(3):e05206.
[15] LOGAN N, VOS P.Genus I.Bacillus.Bergey’s Manual of Systematic Bacteriology.Second Edition.Volume three, the Firmicutes[M].2nd ed.Springer, 2009:21.
[16] ZHOU Y X, LIU G H, LIU B, et al.Bacillus mesophilus sp.nov., an alginate-degrading bacterium isolated from a soil sample collected from an abandoned marine solar saltern[J].Antonie Van Leeuwenhoek, 2016, 109(7):937-943.
[17] GUPTA R S, PATEL S, SAINI N, et al.Robust demarcation of 17 distinct Bacillus species clades, proposed as novel Bacillaceae genera, by phylogenomics and comparative genomic analyses:Description of Robertmurraya kyonggiensis sp.nov.and proposal for an emended genus Bacillus limiting it only to the members of the subtilis and cereus clades of species[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(11):5 753-5 798.
[18] ADIMPONG D B, SØRENSEN K I, THORSEN L, et al.Antimicrobial susceptibility of Bacillus strains isolated from primary starters for African traditional bread production and characterization of the bacitracin operon and bacitracin biosynthesis[J].Applied and Environmental Microbiology, 2012, 78(22):7 903-7 914.
[19] VAN HOEK A H A M, MEVIUS D, GUERRA B, et al.Acquired antibiotic resistance genes:An overview[J].Frontiers in Microbiology, 2011, 2:203.
[20] WHO.Critically important antimicrobials for human medicine 6threvision 2018.Ranking of medically important antimicrobials for risk management of antimicrobial resistance due to non-human use[A].2018.
[21] WHO.Global antimicrobial resistance surveillance system (GLASS) report:early implementation,2 017-2 018.
[22] 庄子慧, 何丽, 郭云昌, 等.我国食源性蜡样芽孢杆菌毒力基因和药物敏感性研究[J].中国食品卫生杂志, 2013, 25(3):198-201.
ZHUANG Z H, HE L, GUO Y C, et al.Virulent gene profiles and antibiotic susceptibility of foodborne Bacillus cereus in China[J].Chinese Journal of Food Hygiene, 2013, 25(3):198-201.
[23] 刘秀峰, 潘洁茹, 林萍.应用多重PCR技术检测120株蜡样芽胞杆菌毒力基因[J].现代预防医学, 2018, 45(21):3 972-3 976.
LIU X F, PAN J R, LIN P.Detection of 120 strains of Bacillus cereus virulence genes by multiplex PCR[J].Modern Preventive Medicine, 2018, 45(21):3 972-3 976.
[24] SULTHANA A, LAKSHMI S G, MADEMPUDI R S.Genome sequencing and annotation of Bacillus subtilis UBBS-14 to ensure probiotic safety[J].Journal of Genomics, 2019, 7:14-17.
[25] SAROJ D B, GUPTA A K.Genome based safety assessment for Bacillus coagulans strain LBSC (DSM 17654) for probiotic application[J].International Journal of Food Microbiology, 2020, 318:108523.