食源性致病菌引起的食品安全问题严重威胁人们的生命健康。长期以来滥用抗生素已使多种细菌产生耐药性,因此,开发新型抗菌剂迫在眉睫。抗菌肽是由核糖体合成的,可抑制微生物生长或杀死微生物的一类小分子多肽或蛋白,通常具有良好的稳定性和广谱抑菌活性[1]。抗菌肽的主要抑菌方式是增强膜的通透性,破坏膜质子动力势,并引起膜穿孔,导致细菌裂解死亡[2-3]。因此,抗菌肽不易使细菌产生广泛的耐药性,被认为是最具潜力的抗菌剂之一。环肽是抗菌肽中较特殊的一类,肽在核糖体合成后,前导肽会被酶切,碳链主链头尾氨基酸残基脱水缩合形成一个闭合的环[4]。与线性肽相比,环肽表现出更强的热稳定性和抑菌活性[5]。目前报道的产环肽菌株有粪肠球菌S-48(Enterocin AS-48)[6]、结核链球菌(Uberolysin)[7]、拜氏梭菌ATCC 25752(Circularin A)[8]以及枯草芽孢杆菌(Subtilosin A)[9]等,尚未见有产自凝结芽孢杆菌的环肽报道。
随着测序技术的发展,利用泛基因组和次级代谢产物挖掘软件,更多的抗菌肽被挖掘和应用。钱文江等[10]对从NCBI数据库中获得的33个凝结芽孢杆菌的基因组进行次级代谢产物合成基因簇分析,发现与已知基因簇有同源性的43个基因簇中有11个基因簇与Amylocyclicin高度同源,因此,凝结芽孢杆菌极可能具有Amylocyclicin代谢合成途径。Amylocyclicin最初发现于解淀粉芽孢杆菌(Bacillus amyloliquefaciens)FZB42,是一类对革兰氏阳性菌表现出较高抑菌活性的新型环状细菌素[11]。
本实验室前期研究发现,凝结芽孢杆菌(Bacillus coagulans)CGMCC 9951对志贺氏菌等食源性致病菌表现出良好的抑菌活性,主要抑菌物质是有机酸和抗菌肽,并对次级代谢产物中的有机酸的抑菌特性进行了研究[12],而抗菌肽尚待深入研究。基于此,开展凝结芽孢杆菌CGMCC 9951的全基因组测序,与次级代谢产物数据库进行比对,挖掘抗菌肽合成基因簇,并将其中一个抗菌肽基因在大肠杆菌中进行表达,对表达产物进行质谱和抑菌活性验证,以期为凝结芽孢杆菌CGMCC 9951抗菌肽的进一步研究和应用奠定基础。
1 材料与方法
1.1 材料与试剂
凝结芽孢杆菌(Bacillus coagulans)CGMCC 9951,单核细胞增生李斯特氏菌(Listeria monocytogenes) ATCC 19115,实验室分离保藏菌株,均在LB(Luria-Bertani)肉汤培养基中培养(pH 8.0)。
DNA提取试剂盒、BL21(DE3)感受态细胞、pGEX-4T-1表达载体、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG)、3′, 3′, 5′, 5′, -四甲基联苯胺(tetramethylbenzidine,TMB)显色试剂盒、蛋白免疫印迹(Western blot)一抗和二抗,生工生物工程(上海)股份有限公司。
1.2 仪器与设备
Pacbio RS Ⅱ测序仪,Pacific Biosciences公司;JY88-II超声波细胞破碎仪,天津津立仪器设备科技发展有限公司;QE-OrbitrapTM高场静电场轨道阱质谱仪、Nanodrop 2000超微量紫外分光光度计,Thermo Fisher公司。
1.3 实验方法
1.3.1 总DNA提取
凝结芽孢杆菌CGMCC 9951在37 ℃振荡培养16 h至生长对数期,8 000 r/min离心10 min收集菌体,按DNA提取试剂盒步骤提取细菌基因组DNA。
采用Qubit 4.0和Nanodrop 2000对基因组DNA进行定量和纯度检测。将高质量的DNA(OD260/280为1.8~2.0;OD260/230为2.0~2.2,DNA总量≥15 μg,质量浓度≥50 ng/μL)用于物种鉴定和后续建库测序。
1.3.2 文库构建与基因注释
采用第三代测序平台PacBio RS Ⅱ对凝结芽孢杆菌CGMCC 9951进行完成图测序(北京奥维森基因科技有限公司)。使用SMRT Link v5.0.1工具(PacBio公司)进行reads组装,GeneMarks(http://topaz.gatech.edu/)进行细菌的编码基因预测,将编码基因在蛋白相邻类的聚簇数据库(Cluster of Orthologous Groups of Proteins,COG)、京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)、基因本体论数据库(Gene Ontology,GO)进行功能注释。
1.3.3 抗菌肽基因簇挖掘
用antiSMASH 5.0[13]软件(http://antismash.secondarymetabolites.org/)对次级代谢产物和RiPPPs进行挖掘。将全基因组序列的FAST格式上传后,选择“strict”检测标准,勾选“Known Cluster Blast”、“Active Site Finder”“Sub Cluster Blast”和“RREF indeer”后提交,对获得的核心肽氨基酸序列与NCBI(http://www.ncbi.nlm.nih.gov/)数据库进行Protein BLAST比对,采用MEGA 5.0软件运用Neighbor-Joining方法建立基于氨基酸序列和相关环肽的系统发育树。
1.3.4 抗菌肽结构预测
用Expasy(http://web.expasy.org/protparam/)对抗菌肽的分子质量、等电点、电荷数和不稳定系数进行预测[14];用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)对跨膜结构域进行预测,用SignalP(http://www.cbs.dtu.dk/services/SignalP)对信号肽进行预测,用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对二级结构进行分析[15];用SWISS-MODEL(https://swissmodel.expasy.org/)对三级结构进行模拟[16]。
1.3.5 Region 3抗菌肽质粒构建及表达
质粒构建与验证:以Region 3成熟肽氨基酸序列为模板,由生工生物工程(上海)股份有限公司设计引物,进行PCR扩增目的产物。通过连接酶将PCR产物和克隆载体进行连接,对克隆载体进行酶切,将目的基因酶连至载体,连接液转入TOP 10感受态中,并对筛选出的阳性克隆菌株进行测序验证。
重组蛋白的诱导表达:将阳性克隆转化感受态细胞大肠杆菌BL21(DE3)于42 ℃热激后涂布在含50 μg/mL氨苄青霉素的平板上,37 ℃培养过夜;挑取单克隆菌落到含50 μg/mL氨苄青霉素的液体培养基中,37 ℃培养至OD值为0.6时,分别添加0.5和1.0 mmol/L IPTG于16 ℃条件下振荡培养过夜。
表达检测:离心收集菌体并重悬于PBS(0.01 mol/L,pH 7.4),用超声波细胞破碎仪裂解细胞(300 W,工作5 s,间歇5 s)至悬浮液透亮。离心后收集上清液和细胞碎片,细胞碎片用8 mol/L尿素溶解,再用2 mol/L尿素复性。分别对上清液和细胞碎片制样,进行SDS-PAGE电泳(12%分离胶,5%浓缩胶)检测蛋白分子质量,并进行蛋白免疫印迹检测[一抗为兔抗谷胱甘肽巯基转移酶(glutathione S-transferase,GST)标签,二抗为羊抗兔],采用TMB显色后观察试验结果。
1.3.6 重组蛋白的质谱验证
将SDS-PAGE凝胶电泳的目标条带切胶,按照质谱检测要求处理后送卡梅德生物科技(天津)有限公司鉴定。质谱参数:质谱一级谱扫描范围350~ 1600 m/z;二级质谱模式CID(Elite)。数据使用Proteome Discoverer 2.0(Thermo Fisher公司)进行处理。
1.3.7 抗菌活性验证
按照1.3.5重组蛋白的诱导表达和检测方法,每600 mL重组大肠杆菌发酵液菌体超声波处理后细胞碎片重悬至6 mL为制备样品。以终浓度为106 CFU/mL(涂板计数)的单核细胞增生李斯特氏菌为指示菌,取200 μL涂板,采用牛津杯法[17]检测融合蛋白的抑菌活性,每个杯中添加200 μL样品,37 ℃培养24 h后,采用十字交叉法测量透明圈直径。
2 结果与分析
2.1 总DNA提取的质量检测
对提取的凝结芽孢杆菌CGMCC 9951的总DNA进行质量检测。Nanodrop检测其OD260/280为1.98,OD260/230为2.17;Qubit检测其质量浓度为208 ng/μL,体积290 μL,总量60.32 μg,其质量和浓度达到建库要求,进行建库。
2.2 凝结芽孢杆菌CGMCC 9951全基因组分析
通过第三代测序平台PacBio RS II对凝结芽孢杆菌CGMCC 9951进行完成图测序。该染色体基因组为环状,染色体的总数为1,没有发现质粒存在,基因组大小为3 608 370 bp,其GC含量为46.18%。
针对测序样品的组装基因组序列,结合编码基因的预测结果,使用Circos软件对样品基因组进行展示。如图1所示,环形图谱最外圈是基因组序列位置坐标,由外到里分别是编码基因和基因功能注释结果(COG、KEGG、GO)。其中COG注释的基因共有2 434个,占总注释基因的57.19 %,分为信息存储与处理、细胞过程和信号、新陈代谢3大类,复制、重组和修复、能量生产和转换、氨基酸运输和代谢等24个分类。KEGG注释的基因有3 358个,占总注释基因的78.90%,分别从细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢、有机系统6大部分,细胞生长与死亡、跨膜运输、信号转导、复制和修复等27个分类进行基因注释。GO注释的基因有2 399个,分别从生物学途径、细胞学组件和分子功能3大类,生物调节、酶调节活动等47个小分类进行基因注释。

图1 凝结芽孢杆菌CGMCC 9951基因组环形图谱及分类注释
Fig.1 Ring map and taxonomic annotation of the genome of Bacillus coagulans CGMCC 9951
2.3 antiSMASH对凝结芽孢杆菌CGMCC 9951抗菌肽的挖掘
利用antiSMASH软件对凝结芽孢杆菌CGMCC 9951全基因组次级代谢产物进行分析,共得到6个结果,见表1。其中Region 3和Region 6为核糖体合成和翻译修饰后的肽产物(RiPP)簇,即潜在的目的产物抗菌肽。Region 1是T3PKS合成酶(Ⅲ聚酮化合物),Region 2是硫肽类抗生素,Region 4是β-内酯含蛋白酶抑制剂,Region 5是萜烯类化合物。
表1 antiSMASH对次级代谢产物的预测
Table 1 Prediction of secondary metabolites by antiSMASH

基因簇标签描述基因位置Region 1T3PKSⅢ型聚酮合酶270 136~311 308Region 2硫肽硫肽类1 117 119~1 141 983Region 3抗菌肽核糖体合成和翻译修饰后的肽产物簇2 716 030~2 725 501Region 4β-内酯含β-内酯的蛋白酶抑制剂2 958 193~2 986 574Region 5萜烯萜烯类2 999 892~3 021 757Region 6抗菌肽核糖体合成和翻译修饰后的肽产物簇3 469 990~3 480 217
Region 3和Region 6的合成基因簇及核心肽氨基酸序列见图2。

图2 Region 3和Region 6的合成基因簇及 核心肽氨基酸序列
Fig.2 Synthesis of Region 3 and Region 6 gene clusters and amino acid sequences of core peptides
将2个氨基酸序列在NCBI的Protein BLAST数据库进行同源性比对,基于氨基酸序列的系统发育树分析结果(图3),发现Region 3与Amylocyclicin的相似度较高,而Region 6在数据库无比对结果。Amylocyclicin是由解淀粉芽孢杆菌(Bacillus amyloliquefaciens)FZB42的核糖体产生的新型环状细菌素,对革兰氏阳性菌有较高的抑菌活性,其在N端Leu和C端Try之间脱水缩合形成环化,成熟环肽的分子质量为6 381 Da(未环化肽的分子质量为6 399 Da,相差1个H2O分子)[11]。最终,选择Region 3进行表达,开展后续实验。
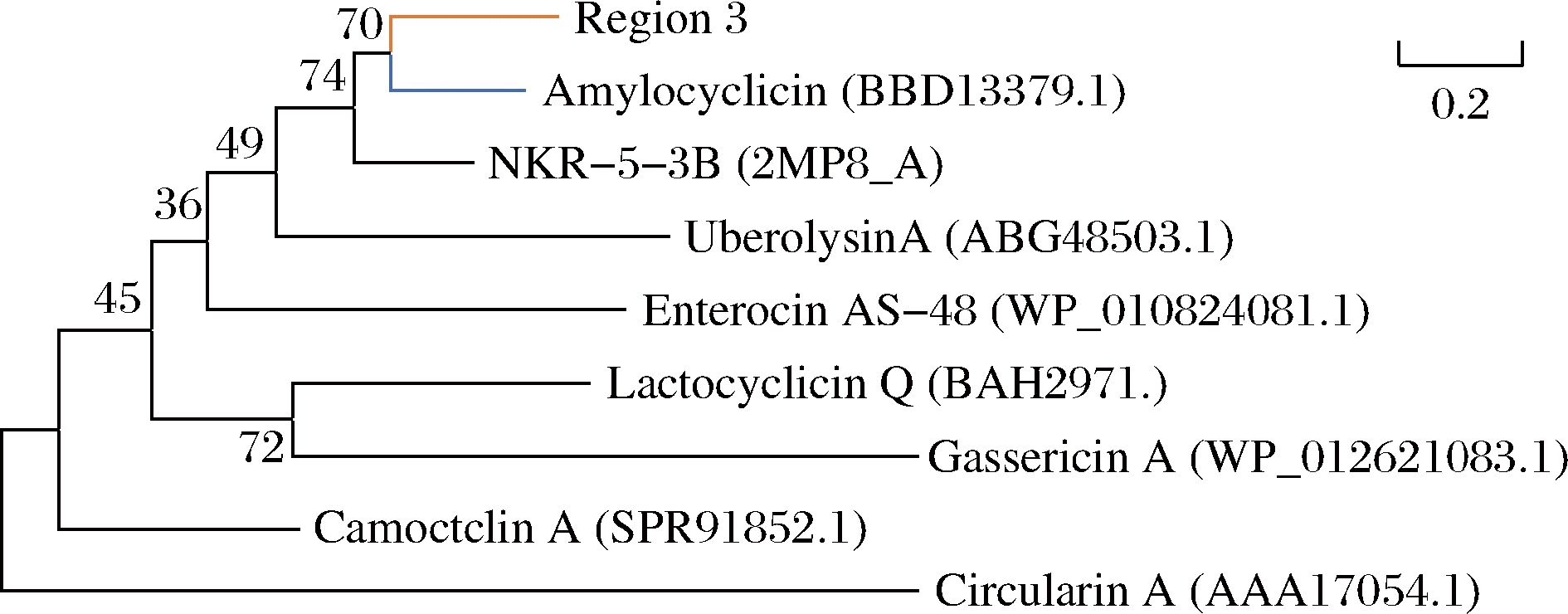
图3 Region 3基于氨基酸序列和相关环肽的系统发育树
Fig.3 Phylogenetic tree of Region 3 based on amino acid sequences and related cyclic peptides
2.4 Region 3抗菌肽的结构预测
通过Expasy分析一级结构,Region 3成熟肽由64个氨基酸残基组成,分子式为C291H488N72O84S1,6 371.57 Da,氨基酸分布见图4-a。此外,预测到负电荷残基总数为2,正电荷残基总数为7,正电荷数目的增加可提高抗菌肽与细菌细胞膜的结合能力,降低抑菌浓度[18]。其次,预测不稳定系数为4.28,远小于40,归为稳定蛋白,良好的稳定性有助于抗菌肽的加工和保存。

a-氨基酸分布;b-疏水性;c-信号肽分析; d-跨膜区分析
图4 Region 3抗菌肽理化性质预测
Fig.4 Prediction of physicochemical properties of Region 3 antimicrobial peptide
疏水性如图4-b所示,多肽链呈两亲性,平均系数为0.841,归为疏水蛋白。疏水性是影响抗菌活性的重要因素之一,疏水性太低会导致抗菌肽与细菌细胞膜的亲和力过低而不易结合,太高易导致抗菌肽自我聚集而降低溶解度[19]。一般细胞内部大多为亲水性蛋白,膜蛋白亲水性通常较差,故推测该抗菌肽可能是结合在细菌细胞膜上的一个小分子蛋白。信号肽分析结果见图4-c,表示剪切位点存在可能性的C值最大值在第41位氨基酸残基,在此区域有可能出现剪切位点,其次为第52位氨基酸残基。信号肽存在可能性S值和综合分析值Y值均未超过阈值0.5,表明该抗菌肽不存在信号肽区域。使用TMHMM进行跨膜区分析结果见图4-d。该肽含有一个跨膜螺旋域,肽链一端在膜外,一端在膜内。综上分析,该肽为一个带正电荷、疏水的、结构稳定的膜上蛋白,根据预测的理化性质进行下一步表达载体的选择和构建。
如图5-a所示,Region 3抗菌肽二级结构主要由α螺旋、β转角、延伸链和无规卷曲构成,其中有43个氨基酸参与α螺旋的形成,占67.19%;5个氨基酸参与β转角的形成;有10个氨基酸参与延伸链的形成;6个氨基酸参与无规卷曲的形成。SWISS-MODEL同源建模三级结构预测如图5-b所示,以NKP-5-3B为模板(氨基酸序列见图5-c),两者氨基酸序列重复率为60.9%,三级结构高度相似,都是由4个紧密堆积的α螺旋结构组成。研究表明,α螺旋可能是膜渗透和抗菌活性所必需的[20],大多数抗菌肽在水溶液中是线性无结构的,当与目标膜结合时,才折叠成螺旋结构,并且只有当α螺旋抗菌肽序列超过一定长度,才足以跨越整个细菌膜,形成通道而杀死细菌[21-22]。

a-抗菌肽二级结构;b-抗菌肽三级结构;c-样品和模板氨基酸序列
图5 Region 3抗菌肽结构预测
Fig.5 Prediction of Region 3 antimicrobial peptide structure
因此,以α螺旋为主要结构的Region 3抗菌肽可能具有良好的抑菌活性。
2.5 Region 3抗菌肽的表达
为了方便后期制备和纯化抗菌肽,采用带有GST标签的载体和大肠杆菌原核表达系统。目的氨基酸序列为LASTLGISTAAAKKAIDIIDTASTIASIISLIGVV-TGAGAISYAVVATAKAMIKKYGKKYAAAW(载体信息见图6-a),表达的融合蛋白总长度为290个氨基酸,分子质量32 658.4 Da,等电点为8.27。对重组质粒进行酶切,电泳结果如图6-b所示,条带位于5 000 bp的是载体,小于500 bp的是目的基因(192 bp),说明重组质粒构建成功。将重组质粒转化到TOP 10感受态细胞中,检测筛选出阳性克隆的质粒,测序验证无误后再转化到E.coli BL21(DE3)感受态细胞中,所得菌株即为构建好的表达菌株。

a-载体信息;b-重组质粒酶切
图6 载体及重组质粒酶切
Fig.6 Vector and recombinant plasmid digestion
通常情况下,低温环境会降低细菌的蛋白表达速度,减少包涵体形成。例如黄夏冰等[23]在37 ℃用不同浓度IPTG诱导表达的蛋白均为包涵体,将温度降低至15 ℃时,诱导产物绝大部分为可溶蛋白,且在0.5 mmol/L IPTG条件下可溶性蛋白表达量最高。因此,将重组大肠杆菌先在37 ℃培养至OD值为0.6后,再添加IPTG于16 ℃低温培养过夜,然后将离心菌体超声波破碎后的上清液和破碎细胞进行SDS-PAGE,结果见图7-a。添加0.5和1.0 mmol/L IPTG诱导剂的细胞沉淀在32 kDa处都有表达条带,且0.5 mmol/L的IPTG表达量更大;而加入0.5和1.0 mmol/L IPTG诱导的细胞上清液在32 kDa位置条带都很浅,说明表达蛋白主要是在细胞碎片中。此外,蛋白免疫印迹检测图谱显示,在32 kDa出现印记条带(见图7-b箭头所指),证明成功表达了融合蛋白。

图7 抗菌肽的SDS-PAGE(a)及Western blot(b)分析图
Fig.7 SDS-PAGE(a) and Western blot(b) analysis of antimicrobial peptides
注:M-Protein Marker;1-未诱导表达总蛋白;2-16 ℃上清液, 0.5 mmol/L IPTG;3-16 ℃沉淀, 0.5 mmol/L IPTG; 4-16 ℃上清液, 1.0 mmol/L IPTG;5-16 ℃沉淀, 1.0 mmol/L IPTG
2.6 融合蛋白的质谱验证分析
通过蛋白质谱覆盖试验检测到一条肽段,氨基酸序列为ERAEISMLEGAVLDIR,是GST标签蛋白第88位到103位氨基酸序列,总离子流图和目标肽段离子流图见图8。GST标签蛋白可以增大目标蛋白的溶解性,也为进一步分离纯化目的蛋白奠定基础。通过质谱分析成功检测到了GST标签蛋白的多肽片段,表明重组大肠杆菌成功表达了融合蛋白。

a-总离子流图;b-目标肽段离子流图
图8 总离子流图和目标肽段离子流图
Fig.8 Total ion flow diagram and target peptide ion flow diagram
2.7 融合蛋白活性验证
融合蛋白活性验证结果见图9,添加0.5 mmol/L IPTG于16 ℃诱导表达的大肠杆菌的超声波处理后细胞碎片对单核细胞增生李斯特氏菌有良好的抑菌活性,抑菌圈直径为(15.2±1.4)mm,而IPTG浓度增至1.0 mmol/L时对指示菌抑制效果稍弱,这与诱导表达的SDS-PAGE结果相一致,而未诱导表达的大肠杆菌则没有抑菌活性。因此,融合蛋白具有抑菌活性证实Region 3是编码抗菌肽的基因,且利用原核表达体系表达的融合蛋白具有较好的抗菌活性。相似的,丁静等[24]利用大肠杆菌表达的人源抗菌肽LL-37对多种常见的细菌均有较好的抑制作用。此外,王莲哲等[25]利用毕赤酵母GS115表达的树蛙抗菌肽Cathelicidin对金黄色葡萄球菌和大肠杆菌都有良好的抑菌活性。因此,通过异源表达可以获得大量抑菌性能优良的抗菌肽,为进一步的机理研究和应用奠定基础。

a-0.5 mmol/L IPTG诱导细胞碎片;b-1.0 mmol/L IPTG 诱导细胞碎片;c-未诱导细胞碎片
图9 重组蛋白抑菌活性
Fig.9 Antibacterial activity of recombinant protein
3 结论
凝结芽孢杆菌CGMCC 9951具有广谱抑菌活性和优良的热稳定性[12, 26],应用前景良好。但目前从微生物发酵液中大量获取抗菌肽纯品较为困难,不利于研究应用。因此,本研究通过对凝结芽孢杆菌CGMCC 9951的次级代谢产物进行挖掘,并与NCBI数据库比对,选择与Amylocyclin具有较高相似性的Region 3核心肽氨基酸序列进行表达。通过一系列结构分析软件,对Region 3核心肽氨基酸序列进行分析,预测其成熟肽分子质量为6 371.57 Da,带正电荷,呈疏水性稳定蛋白。选择大肠杆菌原核表达系统对Region 3核心肽进行表达,采用0.5 mmol/L IPTG和16 ℃低温过夜培养对目的蛋白进行诱导表达以减少包涵体的形成。SDS-PAGE、蛋白免疫印迹和质谱验证结果表明成功构建了Region 3蛋白原核表达系统,且表达的蛋白主要位于细胞碎片中,对单核细胞增生李斯特氏菌表现出良好的抑菌活性。融合蛋白的成功表达为后续的纯化、性质、抑菌机理和应用研究奠定基础。
[1] ZANGENEH M, KHORRAMI S, KHALEGHI M.Bacteriostatic activity and partial characterization of the bacteriocin produced by L.plantarum sp.isolated from traditional sourdough[J].Food Science & Nutrition, 2020, 8(11):6 023-6 030.
[2] 杨昆, 王欢, 高洁, 等.抗菌肽BCp12对大肠杆菌壁膜及DNA损伤的作用机制[J].食品科学, 2021, 42(19):114-121.
YANG K, WANG H, GAO J, et al.Mechanism by which antimicrobial peptide BCp12 acts on the cell wall and membrane of Escherichia coli cells and induces DNA damage[J].Food Science, 2021, 42(19):114-121.
[3] WANG Y, QIN Y X, ZHANG Y, et al.Antibacterial mechanism of plantaricin LPL-1, a novel class IIa bacteriocin against Listeria monocytogenes[J].Food Control, 2019, 97:87-93.
[4] MAQUEDA M, S NCHEZ-HIDALGO M, FERN
NCHEZ-HIDALGO M, FERN NDEZ M, et al.Genetic features of circular bacteriocins produced by Gram-positive bacteria[J].FEMS Microbiology Reviews, 2008, 32(1):2-22.
NDEZ M, et al.Genetic features of circular bacteriocins produced by Gram-positive bacteria[J].FEMS Microbiology Reviews, 2008, 32(1):2-22.
[5] MONTALB N-L
N-L PEZ M, SPOLAORE B, PINATO O, et al.Characterization of linear forms of the circular enterocin AS-48 obtained by limited proteolysis[J].FEBS Letters, 2008, 582(21-22):3 237-3 242.
PEZ M, SPOLAORE B, PINATO O, et al.Characterization of linear forms of the circular enterocin AS-48 obtained by limited proteolysis[J].FEBS Letters, 2008, 582(21-22):3 237-3 242.
[6] BA OS A, ARIZA J J, NU
OS A, ARIZA J J, NU EZ C, et al.Effects of Enterococcus faecalis UGRA10 and the enterocin AS-48 against the fish pathogen Lactococcus garvieae.studies in vitro and in vivo[J].Food Microbiology, 2019, 77:69-77.
EZ C, et al.Effects of Enterococcus faecalis UGRA10 and the enterocin AS-48 against the fish pathogen Lactococcus garvieae.studies in vitro and in vivo[J].Food Microbiology, 2019, 77:69-77.
[7] WIRAWAN R E, SWANSON K M, KLEFFMANN T, et al.Uberolysin:a novel cyclic bacteriocin produced by Streptococcus uberis[J].Microbiology (Reading, England), 2007, 153(Pt 5):1 619-1 630.
[8] KEMPERMAN R, KUIPERS A, KARSENS H, et al.Identification and characterization of two novel clostridial bacteriocins, circularin A and closticin 574[J].Applied and Environmental Microbiology, 2003, 69(3):1 589-1 597.
[9] VENKATASAMY V, DURAIRAJ R, KARUPPAIAH P, et al.An in silico evaluation of molecular interaction between antimicrobial peptide subtilosin A of Bacillus subtilis with virulent proteins of Aeromonas hydrophila[J].International Journal of Peptide Research and Therapeutics, 2021, 27(3):1 709-1 718.
[10] 钱文江, 汪步青, 李葳茜, 等.凝结芽孢杆菌次级代谢挖掘与泛基因组分析[J].生物技术通报, 2020, 36(10):88-98.
QIAN W J, WANG B Q, LI W X, et al.Secondary metabolic pathway mining and pan-genome analysis of Bacillus coagulans[J].Biotechnology Bulletin, 2020, 36(10):88-98.
[11] SCHOLZ R, VATER J, BUDIHARJO A, et al.Amylocyclicin, a novel circular bacteriocin produced by Bacillus amyloliquefaciens FZB42[J].Journal of Bacteriology, 2014, 196(10):1 842-1 852.
[12] 周子吕, 古绍彬, 吴影, 等.凝结芽孢杆菌CGMCC 9951抑菌特性研究[J].河南科技大学学报(自然科学版), 2021, 42(4):77-82;9.
ZHOU Z L, GU S B, WU Y, et al.Studies of antibacterial properties of Bacillus coagulans CGMCC 9951[J].Journal of Henan University of Science and Technology (Natural Science), 2021, 42(4):77-82;9.
[13] LIU L W, HAO T T, XIE Z J, et al.Genome mining unveils widespread natural product biosynthetic capacity in human oral microbe Streptococcus mutans[J].Scientific Reports, 2016, 6:37479.
[14] GUO X, CHEN J X, SUN H M, et al.Mining, heterologous expression, purification and characterization of 14 novel bacteriocins from Lactobacillus rhamnosus LS-8[J].International Journal of Biological Macromolecules, 2020, 164:2 162-2 176.
[15] 刘悦, 邵学超, 王天添, 等.东北林蛙抗菌肽dybowskin-1ST的结构预测及生物学活性分析[J].生物工程学报, 2021, 37(8):2 890-2 902.
LIU Y, SHAO X C, WANG T T, et al.Structure prediction and biological activity analysis of dybowskin-1ST antimicrobial peptide in Rana dybowskii[J].Chinese Journal of Biotechnology, 2021, 37(8):2 890-2 902.
[16] SARKAR A, SEN S.3D structure prediction of VAPC1 and identification of dual natural inhibitors for VPAC1 and EGFR[J].Journal of Bioenergetics and Biomembranes, 2019, 51(2):89-102.
[17] ASGHAR M A, ASGHAR M A.Green synthesized and characterized copper nanoparticles using various new plants extracts aggravate microbial cell membrane damage after interaction with lipopolysaccharide[J].International Journal of Biological Macromolecules, 2020, 160:1 168-1 176.
[18] HANCOCK R E.Cationic peptides:Effectors in innate immunity and novel antimicrobials[J].The Lancet Infectious Diseases, 2001, 1(3):156-164.
[19] 孟德梅, 刘庆艳, 郭雅君, 等.天然抗菌肽的分子结构与功能特性研究进展[J].中国食品学报, 2019, 19(10):342-350.
MENG D M, LIU Q Y, GUO Y J, et al.Research progress on molecular structure and functional characteristics of natural antimicrobial peptides[J].Journal of Chinese Institute of Food Science and Technology, 2019, 19(10):342-350.
[20] TSENG T S, TSAI K C, CHEN C.Characterizing the structure-function relationship reveals the mode of action of a novel antimicrobial peptide, P1, from jumper ant Myrmecia pilosula[J].Molecular BioSystems, 2017, 13(6):1 193-1 201.
[21] FILLION M, VALOIS-PAILLARD G, LORIN A, et al.Membrane interactions of synthetic peptides with antimicrobial potential:Effect of electrostatic interactions and amphiphilicity[J].Probiotics and Antimicrobial Proteins, 2015, 7(1):66-74.
[22] GRAU-CAMPISTANY A, STRANDBERG E, WADHWANI P, et al.Hydrophobic mismatch demonstrated for membranolytic peptides and their use as molecular rulers to measure bilayer thickness in native cells[J].Scientific Reports, 2015, 5:9388.
[23] 黄夏冰, 康巧珍, 傅国, 等.GST-NAP融合蛋白可溶性表达及柱上切割GST标签[J].郑州大学学报(理学版), 2015, 47(4):94-98.
HUANG X B, KANG Q Z, FU G, et al.Expression of soluble GST-NAP fusion protein and GST-tag-cleavage on column[J].Journal of Zhengzhou University (Natural Science Edition), 2015, 47(4):94-98.
[24] 丁静, 沈娟, 朱家勇, 等.人源抗菌肽LL-37的原核表达及活性鉴定[J].生物技术, 2012, 22(1):18-22.
DING J, SHEN J, ZHU J Y, et al.Prokaryotic expression and activities of human antimicrobial peptide LL-37[J].Biotechnology, 2012, 22(1):18-22.
[25] 王莲哲, 刘士俊, 刘佳乐, 等.树蛙抗菌肽Cathelicidin在毕赤酵母中的表达及抑菌活性分析[J].农业生物技术学报, 2021, 29(1):67-72.
WANG L Z, LIU S J, LIU J L, et al.Expression of tree frog(Rhacophorus) cathelicidin peptide in Pichia pastoris and its antibacterial activity analysis[J].Journal of Agricultural Biotechnology, 2021, 29(1):67-72.
[26] GU S B, ZHAO L N, WU Y, et al.Potential probiotic attributes of a new strain of Bacillus coagulans CGMCC 9951 isolated from healthy piglet feces[J].World Journal of Microbiology & Biotechnology, 2015, 31(6):851-863.