酒精发酵是酵母将糖分代谢成酒精和CO2的过程,可赋予葡萄酒浓郁的香气和独特的口感。酿酒酵母具有快速的糖消耗能力,因而常被用作酒精发酵过程中主要的发酵剂[1],由于葡萄酒的味道、颜色和香气与微生物的生长和代谢密切相关,且只有活性的微生物才会在发酵过程中起作用。因此,有必要建立一种快速和可靠的方法来检测和识别葡萄酒酿造中的活性微生物。
平板计数法作为一种依赖培养的计数方法,已被广泛应用于葡萄酒酿造过程中的微生物计数[2-3]。然而,其耗时的生长过程和缺乏对可生存但不可培养(viable but non culturable,VBNC)微生物的计数能力,可能造成测得的微生物群体密度偏低[4]。据报道,在葡萄酒发酵过程中,死亡的微生物和VBNC状态的微生物可能占微生物总数量的60%以上[5]。
最近,人们研究和开发了以微生物菌株DNA为模板的分子计数方法,以取代依赖培养的计数方法[6-7]。实时定量PCR(real-time quantitative PCR,qPCR)已经作为一种特定的区别于依赖培养的检测方法,应用于测量各种培养基条件下的微生物数量[8]。然而,qPCR技术缺乏区分死菌和活菌DNA的能力。这可能造成死菌DNA的错误扩增,并进一步导致对微生物数量的高估[9]。据报道,叠氮溴化丙啶(propidium monoazide,PMA)的引入可提高qPCR技术对微生物计数的准确性[10]。PMA作为一种光敏的DNA配体染料,可以穿透死菌的细胞膜,在强光下进一步交联死菌细胞的DNA,来自死菌细胞DNA的qPCR扩增被抑制[11-12]。同时,活菌细胞拥有完整的细胞膜结构,这使得PMA无法进入细胞膜与DNA发生作用[7]。
针对活菌的细胞实时定量PCR(CELL-qPCR)方法在计算葡萄酒中的微生物数量方面受到了广泛关注。这种技术省去了DNA提取过程,直接利用细胞作为qPCR扩增的模板,缩短了反应时间,降低了反应成本[13-14]。目前尚无PMA与CELL-qPCR方法结合监控酿酒过程中微生物动力学的研究。本研究拟开发和优化PMA-CELL-qPCR检测方法,以准确测定葡萄酒中的活菌,并进一步应用于葡萄酒酿造过程中,以测量微生物的动力学。研究结果有助于阐明葡萄酒发酵过程中的微生物生长机制,并进一步为葡萄酒酿造过程的质量改进提供有益的启示。
1 材料与方法
1.1 主要试剂与仪器设备
酵母浸粉、葡萄糖、蛋白胨、氯霉素、荧光染料叠氮溴化丙啶(propidium monoazide,PMA),二甲基亚砜、溶壁酶、酵母DNA提取试剂盒,北京天根生化科技有限公司;商业化酿酒酵母菌株Red Fruit®,意大利Enartis公司;酵母提取物-蛋白胨-葡萄糖(yeast extract peptone dextrose, YPD)培养基,美国Difco公司;2x FastStart Essential DNA Green Master,美国 Roche公司, qPCR Mix Plus (ROX), 爱沙尼亚Solis BioDyne公司。
YXQ-LS-50型立式压力蒸汽灭菌器、HPX-9082MBE电热恒温培养箱,上海博讯实业有限公司医疗设备厂;FA204电子分析天平,上海舜宇恒平科学仪器有限公司;VORTEX-5漩涡混合器,海门市其林贝尔仪器制造有限公司;SW-CJ-1D型单人净化工作台,浙江孚夏医疗科技有限公司;WD-9413B凝胶成像分析仪、HB150-S2金属浴、DYY-6C型电泳仪,北京六一生物科技有限公司;5425高速离心机,德国艾本德公司;FQD-96A实时荧光定量PCR仪,杭州博日科技有限公司;Q-bit检测仪,浙江蓝景科技有限公司;UV-3300紫外可见分光光度计,上海美普达仪器有限公司。
1.2 菌种和培养基
YPD固体培养基:加入20 g/L琼脂于液体培养基内,同时加入100 mg/L氯霉素。
葡萄酒培养基:怀来、平谷两地赤霞珠葡萄汁。
商业化酵母菌株在YPD培养基中28 ℃下培养24小时。为模拟葡萄酒发酵过程环境,将YPD培养基中培养至对数末期的酵母菌,转移至相同体积的赤霞珠葡萄汁中培养2 h后,用于后续实验检测计数。
1.3 细胞悬浮液处理
对于每个微生物菌株(酿酒酵母),制备4个细胞悬液样品,分别为:无PMA处理的活菌细胞(对照)、PMA处理的活菌细胞、PMA处理的死菌细胞、PMA处理的活菌和死菌细胞混合物。为了验证PMA处理对死菌细胞的功效,将细胞悬液在85 ℃金属浴中处理20 min,以破坏细胞膜。通过YPD固体培养基培养,确认热处理后的细胞已经死亡。
1.4 PMA处理的优化
参考LYU等[15]的方法并稍加修改,将PMA溶解在20%(体积分数)的二甲基亚砜中,制备PMA储备溶液(20 mmol/L)。PMA储备液在-20 ℃下避光储存。将不同浓度的PMA溶液加入到1 mL细胞悬液中,产生不同浓度(0、5、10、25、50 μmol/L)的PMA溶液。之后,将PMA处理的细胞悬液在避光条件下200 r/min、4 ℃搅拌20 min。暗反应孵育结束后,将细胞悬液暴露在距离7 cm的450 nm的蓝色LED灯下(5 mm,3.7 V,20 mA,2 600 MCD)光活化10 min,完成PMA和细胞DNA的交联[15]。未经PMA处理的细胞悬浮液被用作阴性对照。
1.4 酵母细胞酶处理条件的优化
使用溶壁酶分解酵母细胞壁。在PMA处理过的细胞悬液中加入一定量的溶壁酶30 ℃培养,具体优化条件如表1所示。随后,将细胞悬液在4 000 r/min离心10 min,收集酵母细胞。细胞颗粒用无菌蒸馏水洗涤2次。
表1 溶壁酶优化条件
Table 1 Optimised conditions for lyticase

优化条件实验组处理时间/min0304560处理浓度/U05075100
1.6 DNA提取
从培养基和葡萄酒中各取1 mL细胞悬液,用酵母DNA提取试剂盒提取酵母DNA。取细胞悬液12 000 r/min离心1 min收集菌体,去上清液,然后加入山梨醇,溶壁酶进行破壁处理,4 000 r/min离心10 min收集沉淀,后续提取过程遵循酵母DNA提取试剂盒步骤。
1.7 引物
实验最初选用3对引物对酵母进行扩增,并最终选用引物(S.c-f:5′-ACATATGAAGTATGTTTCTATATAACGGTG-3′;S.c-r:5′-TGGTGCTGGTGCGGATCTA-3′)对酿酒酵母进行定量,引物的序列和特征列于表2。
表2 酿酒酵母引物序列
Table 2 Primer sequences of S.cerevisiae

菌株名称目的基因长度/bp正向引物(5′→3′)反向引物(5′→3′)参考文献酿酒酵母35S rDNA222GCCCAGTGCTCTGAATGTCGCTCAACAGGGTCTTCTTTCC[16]28S rDNA124GAGTCGAGTTGTTTGGGAATGCTCTCTTTCCAAAGTTCTTTTCATCTTT[15]RAPD bands175ACATATGAAGTATGTTTCTATATAACGGTGTGGTGCTGGTGCGGATCTA[15]
1.8 qPCR条件
PCR扩增反应体系为25 μL,包括:2× FastStart Essential DNA Green Master、qPCR Mix Plus (ROX) 12.5 μL,正反引物各1 μL, DNA模板2 μL或细胞模板10 μL,剩余体积无菌水补充。
qPCR扩增条件:95 ℃预变性15 min,40个循环:95 ℃变性15 s,然后60 ℃退火/延伸20 s。熔解曲线从60 ℃缓慢加热到95 ℃,每隔5 s加热0.5 ℃,连续收集荧光得到。对扩增的DNA进行熔解曲线分析以确定反应的特异性。所有的分析都是在C100TM热循环仪、FQD-96A实时系统中进行。通过将qPCR的循环阈值(cycle threshold,Ct)与不同细胞浓度(102~109 CFU/mL)作图得到标准曲线。
1.9 PMA-CELL-qPCR方法应用于葡萄酒
将2个不同产地的葡萄去梗并挤压成葡萄汁。然后对葡萄汁进行膜过滤杀菌,将生长至106 CFU/mL的酿酒酵母接入葡萄汁以启动酒精发酵。在发酵期间,每2~3 d对葡萄酒进行采样。使用CELL-qPCR、PMA-CELL-qPCR和平板计数法测量葡萄酒样品中的酿酒酵母细胞量。
1.10 数据统计分析
实验均平行3次,数据均使用GraphPad Prism软件分析。采用5%显著性水平的t检验来确定不同方法间的差异。对PMA-CELL-qPCR和CELL-qPCR标准曲线进行统计分析,在Tukey多重比较检验下采用单因素方差分析,以确定基质对细胞定量能力的影响。使用Duncan多重比较检验下的单因素方差分析,以确定PMA-CELL-qPCR检测的特异性,P<0.05表示差异显著。
2 结果与分析
2.1 最佳PMA浓度的确定
使用不同浓度的PMA分别处理细胞浓度为107 CFU/mL的活菌细胞和热致死细胞,然后用CELL-qPCR技术进行计数,将其Ct与未经PMA处理的活菌细胞进一步比较,以确定最佳的ΔCt值。ΔCt按公式(1)计算:
ΔCt=Ct处理-Ct未处理
(1)
由图1所示,热致死细胞的ΔCt值随着PMA处理浓度的增加而增加。5 μmol/L PMA处理组活酿酒酵母细胞与未经PMA处理的热致死细胞相比,ΔCt只增加了0.34(P>0.05),这表明PMA可以穿过热致死细胞的细胞膜与DNA相互作用,并抑制qPCR中的DNA扩增。在进行CELL-qPCR扩增之前,PMA处理过的热致死细胞还要用溶壁酶处理。用5 μmol/L PMA处理的热致死细胞与未经PMA处理的活菌细胞相比,ΔCt差异显著(P<0.05),这表明酿酒酵母的细胞壁结构并没有阻止PMA分子进入细胞。在PMA浓度为25 μmol/L和50 μmol/L的情况下,对热致死细胞的抑制作用最高。然而,PMA浓度为50 μmol/L时,活菌细胞的DNA扩增也受到显著抑制(P<0.05)。浓度在5~25 μmol/L的PMA处理对活菌细胞的DNA扩增无负面作用。

图1 PMA浓度对酿酒酵母活/死细胞的 PMA-CELL-qPCR信号的影响
Fig.1 Effect of PMA concentration on PMA-CELL-qPCR signals of live/dead cells of S.cerevisiae
注:不同大写字母表示不同组间死菌细胞差异显著,不同小写 字母表示不同组间活菌细胞差异显著(P<0.05)
实验结果表明,高浓度的PMA通过与热致死细胞的DNA相互作用,能够完全抑制死菌DNA扩增。然而,过量的PMA分子在CELL-qPCR中抑制了活菌细胞DNA扩增。类似的研究也有报道,PAN等[17]发现50 μmol/L 的PMA对李斯特菌活菌有轻微的细胞毒性效应,SHAO等[18]使用高浓度的PMA处理保加利亚乳杆菌时,其细胞计数明显减少,但目前这一现象的机制还未被阐明。本研究中,酿酒酵母的最佳PMA处理浓度为25 μmol/L,此浓度下,死菌细胞的DNA扩增被完全抑制,且对活菌细胞DNA扩增无负面作用。
2.2 酵母CELL-qPCR样品预处理的优化
本研究中直接以细胞为模板与从细胞中提取的DNA为模板进行qPCR扩增,两者Ct值有明显差异。ANDORR 等[19]研究表明,酿酒酵母的细胞壁可能是造成Ct值差异的原因。在本研究中,对酿酒酵母进行PMA处理后,进一步对其进行溶壁酶破壁处理。溶壁酶处理有助于降低Ct值,提高检测灵敏度和限度。
等[19]研究表明,酿酒酵母的细胞壁可能是造成Ct值差异的原因。在本研究中,对酿酒酵母进行PMA处理后,进一步对其进行溶壁酶破壁处理。溶壁酶处理有助于降低Ct值,提高检测灵敏度和限度。
由图2-a所示,对照组(DNA模板)的Ct值与未经溶壁酶处理的酿酒酵母细胞相比,存在显著差异(P<0.05)。在溶壁酶处理30 min后,细胞的Ct值明显下降(P<0.05)。表明酿酒酵母的细胞壁结构抑制了CELL-qPCR检测下的DNA扩增。在进行45 min的溶壁酶处理后,以细胞为模板与从细胞中提取的DNA为模板进行qPCR扩增,两者Ct值差异明显缩小。而将溶壁酶处理时间延长到60 min并没有明显改变Ct值(P>0.05)。对于不同溶壁酶浓度的影响,如图2-b所示,与对照组相比,溶壁酶处理后的样品表现出较低的Ct值。使用CELL-qPCR在75 U的溶壁酶处理下,酿酒酵母细胞的Ct值与qPCR的量化结果相似。与75 U的溶壁酶浓度相比,100 U溶壁酶浓度的Ct值无明显变化(P>0.05)。因此,酿酒酵母溶壁酶预处理的最佳条件为溶壁酶75 U处理45 min。这也是第一项为CELL-qPCR定量而对酿酒酵母进行的溶壁酶处理的优化研究。
2.3 培养基环境下的标准曲线
为了验证CELL-qPCR和PMA-CELL-qPCR方法对细胞的计数,将这些方法的扩增量与qPCR在相同细胞悬浮液下的DNA扩增量进行了比较。表3显示了Ct值与细胞浓度之间的相关性。

a-溶壁酶处理时间;b-溶壁酶浓度
图2 酿酒酵母PMA-CELL-qPCR预处理条件优化
Fig.2 Optimization of the pretreatment conditions of S.cerevisiae PMA-CELL-qPCR
注:不同字母表示不同组间差异显著(P<0.05)
表3 在培养基和葡萄酒中使用特异性引物构建的 qPCR、CELL-qPCR、PMA-CELL-qPCR的 标准曲线的相关系数、效率(平板计数值与循环阈值的对数)和检测限
Table 3 Correlation coefficients, efficiencies of standard curves (log of plate count value vs.cycle threshold) and detection limit constructed by qPCR, CELL-qPCR, PMA-CELL-qPCR using specific primers in culture and wine
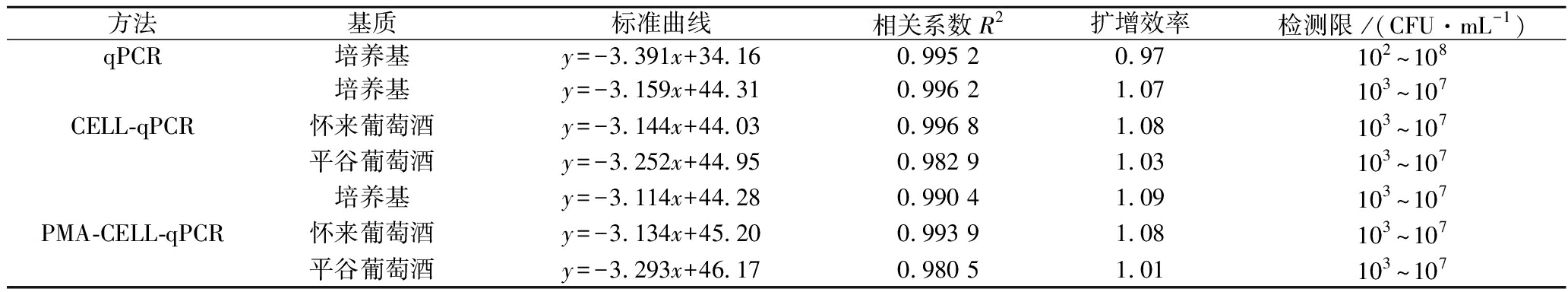
方法基质标准曲线相关系数R2扩增效率检测限 /(CFU·mL-1)qPCR培养基y=-3.391x+34.160.995 20.97102~108CELL-qPCR培养基y=-3.159x+44.310.996 21.07103~107怀来葡萄酒y=-3.144x+44.030.996 81.08103~107平谷葡萄酒y=-3.252x+44.950.982 91.03103~107PMA-CELL-qPCR培养基y=-3.114x+44.280.990 41.09103~107怀来葡萄酒y=-3.134x+45.200.993 91.08103~107平谷葡萄酒y=-3.293x+46.170.980 51.01103~107
如图3所示,qPCR方法在102~108 CFU/mL的细胞浓度范围内具有良好的线性关系,其线性相关系数高于0.99(R2>0.99)。与qPCR方法相比,CELL-qPCR和PMA-CELL-qPCR方法在检测限上表现出明显的差异。在CELL-qPCR和PMA-CELL-qPCR方法下,酿酒酵母在细胞浓度为103~107 CFU/mL建立了线性相关关系。在本研究中,所有的扩增方法都显示出良好的扩增效率(0.9~1.1),qPCR的表现略胜于其他2种检测方法(表3)。然而,CELL-qPCR的相关系数、扩增效率都与qPCR方法相当,且省去了DNA提取过程,加快了检测速度,减少了检测成本。因此,在对菌株进行定量分析时,CELL-qPCR可以作为qPCR的替代方法。此外,使用PMA-CELL-qPCR方法绘制的酿酒酵母标准曲线在检测限、相关系数和扩增效率等方面都与CELL-qPCR方法下的标准曲线相似。这表明PMA-CELL-qPCR方法同样具有良好的稳定性,可以作为量化活性微生物的方法。
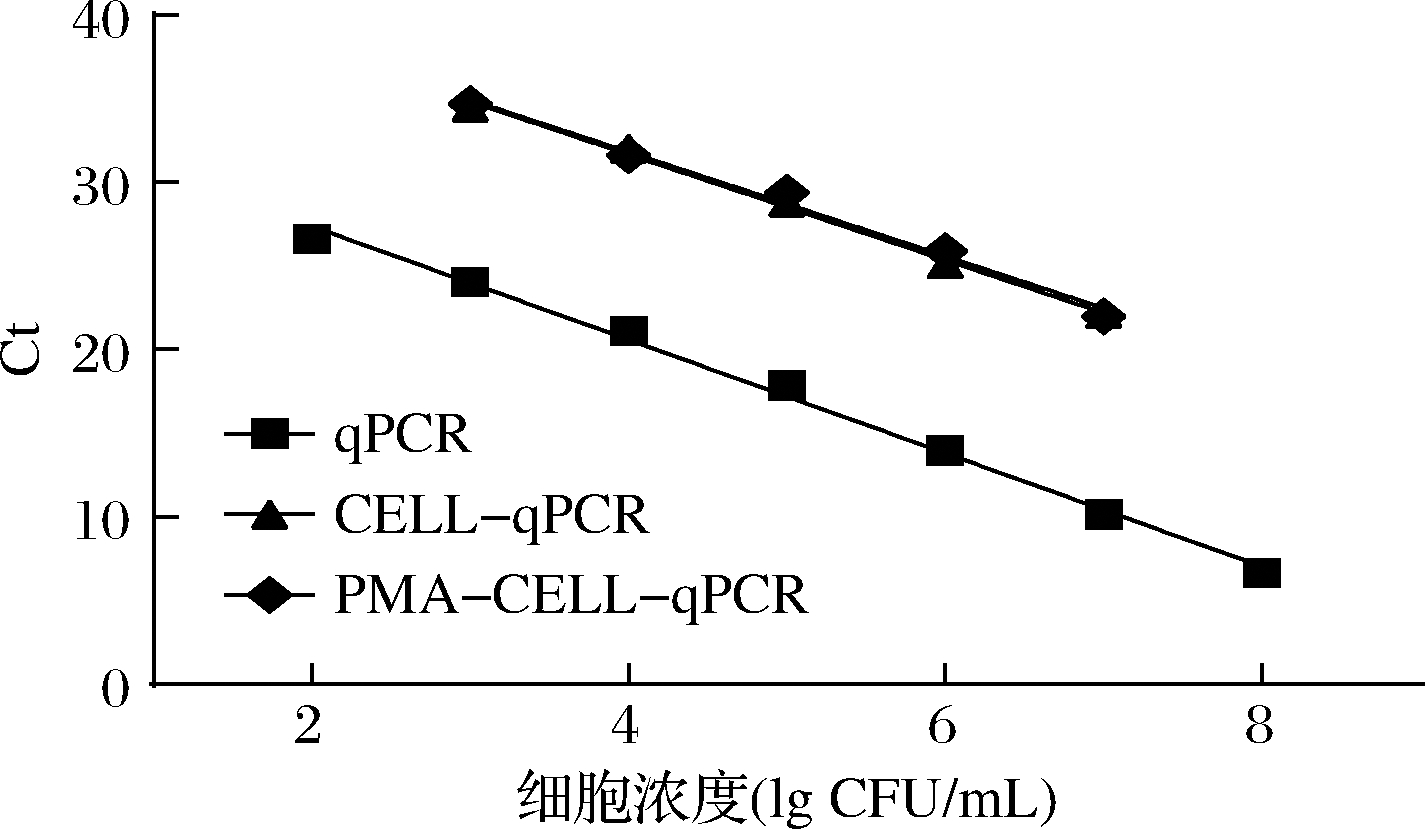
图3 酿酒酵母在培养基中标准曲线
Fig.3 Standard curve of S.cerevisiae in the medium matrix
2.4 死菌对PMA处理效率的影响
本研究中的PMA-CELL-qPCR方法是为了监测葡萄酒发酵过程中的活菌数量而提出的。因此,有必要评估葡萄酒发酵过程中的死菌细胞是否会影响该方法的准确性。为了评估死菌如何影响PMA的处理效率,取3个浓度的热致死细胞(103、105、107 CFU/mL)与活菌细胞(103、107 CFU/mL)按1∶1(体积比)混合。比较使用和不使用PMA处理的混合细胞样品的Ct值。ΔCt按照公式(2)计算:
ΔCt=Ct活菌-Ct活死菌混合
(2)
当使用CELL-qPCR对微生物种群进行定量时,随着活菌细胞中热致死细胞浓度的增加,样品Ct值也明显增加(图4),这表明CELL-qPCR方法不能区分死菌和活菌。
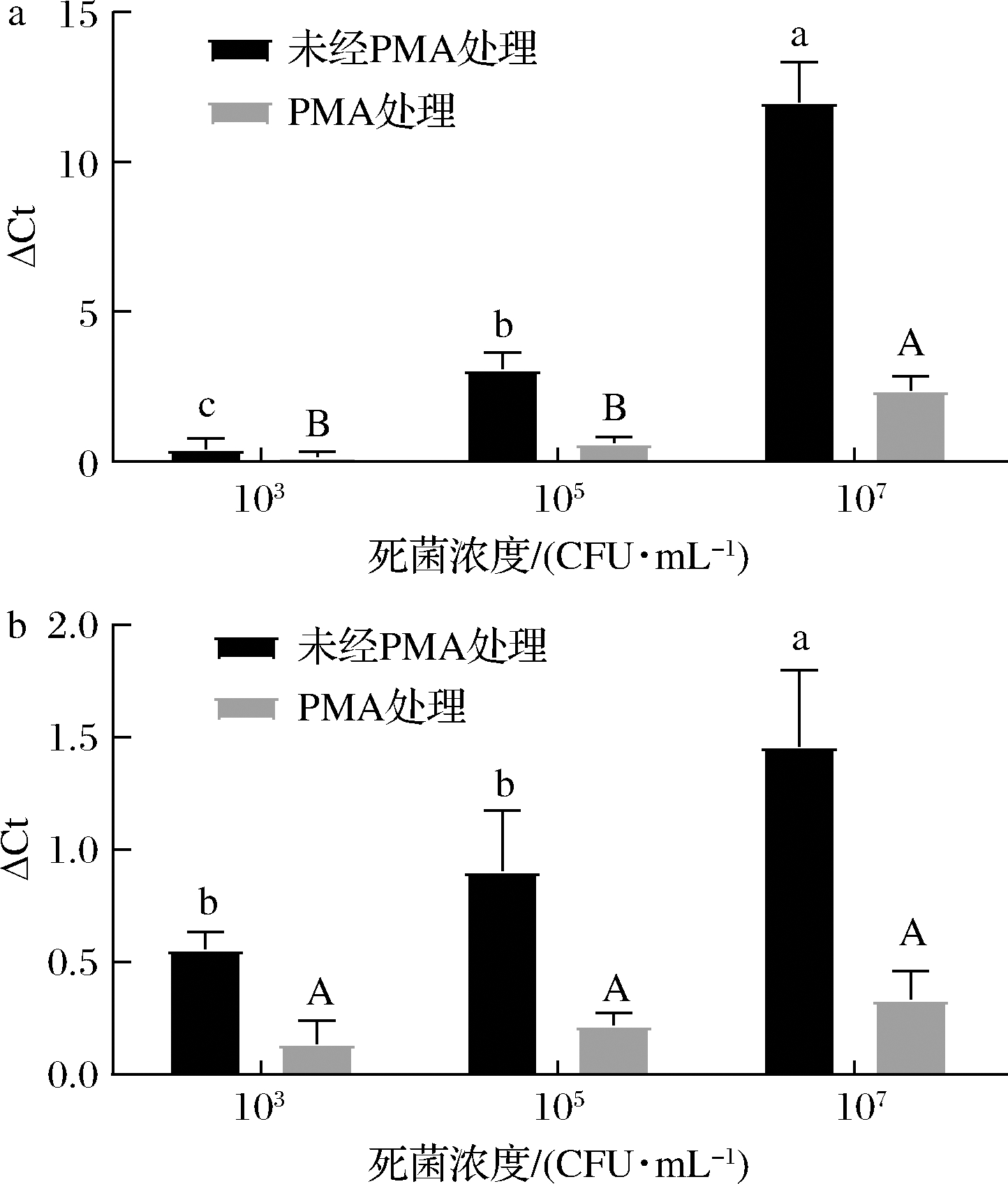
a-103 CFU/mL活菌;b-107 CFU/mL活菌
图4 不同浓度死细胞对PMA-CELL-qPCR检测 103和107 CFU/mL酿酒酵母的影响
Fig.4 Effects of concentrations of dead cells on the detection of 103 and 107CFU/mL S.cerevisiae by PMA-CELL-qPCR
注:不同大写字母表示不同组未经PMA处理差异显著,不同 小写字母表示不同组间PMA处理差异显著(P<0.05)
如图4-a所示,PMA处理使CELL-qPCR的活菌细胞定量得到明显改善,活菌细胞(103 CFU/mL)与103和105 CFU/mL热致死细胞混合,与单纯的活菌细胞相比,Ct值差异不显著(P>0.05)。在与107 CFU/mL热致死细胞混合的活菌细胞样品中,ΔCt值明显增加(P<0.05)。表明过量的死菌会降低PMA分子与DNA分子相互作用的效率[20]。当活菌数量≤死菌数量时,PMA-CELL-qPCR方法可以有效抑制死菌的扩增。TAKAHASHI等[21]研究发现,当活菌与死菌的比例高于1∶1 000时,PMA-qPCR可以准确地量化活的金黄色葡萄球菌的数量,与本研究结果一致。
对于浓度为107 CFU/mL的活菌细胞,加入3个浓度水平的热致死细胞并没有改变ΔCt值(P>0.05)(图4-b),表明PMA-CELL-qPCR定量分析可以在高水平活菌条件下取得更可靠的结果,死菌产生的干扰可被PMA有效地消除。值得注意的是,当死菌与活菌比例高于10 000∶1时,PMA-CELL-qPCR测定可能产生0.5 lg CFU/mL的细胞定量检测误差。当死菌与活菌比例低于100∶1时,PMA-CELL-qPCR方法可以消除死活菌的错误判断,提供准确可靠的活菌群体定量。
2.5 葡萄酒中标准曲线
由图5所示,在葡萄酒基质中,使用CELL-qPCR和PMA-CELL-qPCR方法在细胞浓度为103~107 CFU/mL时观察到线性标准曲线。这与YPD培养基中观察到的酿酒酵母群体的标准曲线相似(图3)。同时,2种方法的标准曲线都表现出良好的重复性(R2>0.98)(表3)。对于PMA-CELL-qPCR的标准曲线,平谷葡萄酒和怀来葡萄酒中酿酒酵母的扩增效率分别为1.012和1.085,与CELL-qPCR技术下的扩增效率相当,表明葡萄酒环境中的PMA预处理并不影响酿酒酵母的扩增准确性。PMA-CELL-qPCR和CELL-qPCR在监测葡萄酒基质中的酿酒酵母种群方面可以发挥类似的作用,PMA-CELL-qPCR方法可以替代CELL-qPCR方法检测葡萄酒发酵过程中的酿酒酵母菌群动态变化。

a-平谷葡萄酒;b-怀来葡萄酒
图5 酿酒酵母在葡萄酒基质中标准曲线
Fig.5 Standard curve of S.cerevisiae in wine matrix
2.6 PMA-CELL-qPCR在葡萄酒中的应用
选取平谷和怀来2个地区的赤霞珠葡萄榨汁灭菌(不含酵母),研究是否可以利用PMA-CELL-qPCR方法来监测葡萄酒酿造过程中的活菌群动力学。平谷和怀来葡萄汁的初始还原糖含量如表4所示。
表4 两个产地葡萄酒在发酵前后的还原糖含量 单位:g/L
Table 4 Reducing sugar content of wines of two origins before and after fermentation

葡萄酒产地还原糖含量酒精发酵开始酒精发酵结束平谷147.31±2.561.58±0.62怀来142.64±4.512.79±0.7
在葡萄汁中接种浓度为106 CFU/mL的酿酒酵母,以启动酒精发酵(图6)。整个酒精发酵过程持续了16 d。采用平板计数、CELL-qPCR和PMA-CELL-qPCR方法测定2种葡萄酒样品中的酿酒酵母活菌数。
如图6-a所示,在平谷葡萄酒中,由于葡萄汁中的营养物质充足,在酒精发酵的初始阶段,酿酒酵母迅速生长。在酒精发酵的第6天,酿酒酵母的数量快速增加,并达到了最高浓度(2.57×107 CFU/mL),3种检测方法显示了相似的酿酒酵母数量。3种检测方法显示了相似的酿酒酵母数量。可能是因为葡萄酒中的高营养水平,大部分的酿酒酵母是有活力的。在酒精发酵的中期阶段(第6~12天),葡萄酒样品中的酿酒酵母数量保持在107 CFU/mL的水平。使用CELL-qPCR检测方法计算的酿酒酵母数量明显高于(10倍)实际存活的菌株数量,酿酒酵母的变化随着酒精发酵过程的进行而增加,通过CELL-qPCR测定的酿酒酵母菌浓度比实际活菌浓度高104倍。可能是因为酿酒酵母的细胞壁结构可以延迟细胞的降解,从死亡的酿酒酵母中释放的DNA分子可以在葡萄酒中保持一定的时间[22]。酒精发酵后期(第12~16天),酿酒酵母的数量明显减少,可能是由于葡萄酒中营养水平降低以及还原糖含量的减少导致的。在发酵结束后的一段时间内,有活力的酿酒酵母数量不断减少。
如图6-b所示,在怀来葡萄酒的发酵过程中,经平板计数、CELL-qPCR和PMA-CELL-qPCR方法证实,在酒精发酵的初始阶段,酿酒酵母细胞浓度快速增至107 CFU/mL。然而,与平板计数法和PMA-CELL-qPCR方法相比,CELL-qPCR方法对菌株的计数结果在葡萄酒的其余酿造期(第6~20天)均表现出差异。用PMA-CELL-qPCR方法测定的葡萄酒中的活菌细胞数与用平板计数法测定的结果相似。酒精发酵结束后,由于还原糖含量减少,酿酒酵母数量呈下降趋势。表明不同的葡萄酒基质对死菌的降解速度以及死菌释放的DNA可能起到不同的作用。

a-平谷葡萄酒发酵;b-怀来葡萄酒发酵
图6 通过不同方法对葡萄酒酿造过程中 酿酒酵母进行定量分析
Fig.6 Quantitative analysis of S.cerevisiae during wine brewing determined by different methods
由于2种葡萄酒样品都存在大量的死菌,CELL-qPCR方法显示死菌与活菌数量存在巨大差异。然而,在PMA-CELL-qPCR和平板计数法之间发现细胞群计数误差仅为0.5 lg CFU/mL,可能是由于发酵过程葡萄酒体系中存在大量VBNC状态的微生物。与本研究结果类似,使用qPCR、PMA-qPCR和平板法计数Majiapo葡萄酒中酿酒酵母动态变化时,qPCR与平板计数相差一个数量级,而PMA-qPCR与平板结果拟合较好[19]。与本实验不同的是,以DNA为模板的扩增在提取DNA过程中会去除环境DNA干扰因素,相比CELL-qPCR,qPCR计数方法与平板计数误差小于本研究中结果。
图6中,PMA-CELL-qPCR方法确定的酿酒酵母细胞计数结果与平板计数法测量的结果相当一致,表明PMA-CELL-qPCR可以准确、可靠地测定葡萄酒中的活体微生物。更重要的是,这种技术可以在不考虑微生物浓度水平的情况下直接检测和量化葡萄酒中的活性微生物群体。
3 结论
本研究通过将PMA与CELL-qPCR相结合,优化了PMA处理浓度、CELL-qPCR前处理方法,并最终利用PMA-CELL-qPCR准确定量,开发的PMA-CELL-qPCR计数方法能够准确量化葡萄酒相关基质中的酿酒酵母的活菌数量。且这种检测方法省略了DNA提取过程,可以将死菌与活菌区分开来,同时降低假阳性结果出现的可能。此外,与葡萄酒酿造过程中的传统平板计数法相比,PMA-CELL-qPCR表现出了一致的计数结果,极大提升了工作效率。该方法在食品领域具有潜在的应用前景,为葡萄酒及果酒产品发酵过程中活菌快速检测提供了新的参考方法。
[1] ALBERGARIA H, ARNEBORG N.Dominance of Saccharomyces cerevisiae in alcoholic fermentation processes:Role of physiological fitness and microbial interactions[J].Applied Microbiology and Biotechnology, 2016, 100(5):2 035-2 046.
[2] BARTLE L, MITCHELL J G, PATERSON J S.Evaluating the cytometric detection and enumeration of the wine bacterium, Oenococcus oeni[J].Cytometry Part A, 2021, 99(4):399-406.
[3] NUNES DE LIMA A, MAGALH ES R, CAMPOS F M, et al.Survival and metabolism of hydroxycinnamic acids by Dekkera bruxellensis in monovarietal wines[J].Food Microbiology, 2021, 93:103617.
ES R, CAMPOS F M, et al.Survival and metabolism of hydroxycinnamic acids by Dekkera bruxellensis in monovarietal wines[J].Food Microbiology, 2021, 93:103617.
[4] ROUSSEL C, GALIA W, LERICHE F, et al.Comparison of conventional plating, PMA-qPCR, and flow cytometry for the determination of viable enterotoxigenic Escherichia coli along a gastrointestinal in vitro model[J].Applied Microbiology and Biotechnology, 2018, 102(22):9 793-9 802.
[5] QUIR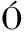 S C, HERRERO M, GARC
S C, HERRERO M, GARC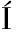 A L A, et al.Quantitative approach to determining the contribution of viable-but-nonculturable subpopulations to malolactic fermentation processes[J].Applied and Environmental Microbiology, 2009, 75(9):2 977-2 981.
A L A, et al.Quantitative approach to determining the contribution of viable-but-nonculturable subpopulations to malolactic fermentation processes[J].Applied and Environmental Microbiology, 2009, 75(9):2 977-2 981.
[6] FITTIPALDI M, NOCKER A, CODONY F.Progress in understanding preferential detection of live cells using viability dyes in combination with DNA amplification[J].Journal of Microbiological Methods, 2012, 91(2):276-289.
[7] NOCKER A, CHEUNG C-Y, CAMPER A K.Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs.dead bacteria by selective removal of DNA from dead cells[J].Journal of Microbiological Methods, 2006, 67(2):310-320.
[8] LI T T, WANG J S, WANG Z Y, et al.Quantitative determination of mutton adulteration with single-copy nuclear genes by real-time PCR[J].Food Chemistry, 2021, 344:128622.
[9] COCOLIN L, MILLS D.Wine yeast inhibition by sulfur dioxide:A comparison of culture-dependent and independent methods[J].American Journal of Enology and Viticulture, 2003, 54:125-130.
[10] ZHANG S J, WANG L L, LU S Y, et al.A novel, rapid, and simple PMA-qPCR method for detection and counting of viable Brucella organisms[J].Journal of Veterinary Research, 2020, 64(2):253-261.
[11] TANTIKACHORNKIAT M, SAKAKIBARA S, NEUNER M, et al.The use of propidium monoazide in conjunction with qPCR and Illumina sequencing to identify and quantify live yeasts and bacteria[J].International Journal of Food Microbiology, 2016, 234:53-59.
[12] VAN FRANKENHUYZEN J K, TREVORS J T, LEE H, et al.Molecular pathogen detection in biosolids with a focus on quantitative PCR using propidium monoazide for viable cell enumeration[J].Journal of Microbiological Methods, 2011, 87(3):263-272.
[13] SOARES-SANTOS V, PARDO I, FERRER S.Cells-qPCR as a direct quantitative PCR method to avoid microbial DNA extractions in grape musts and wines[J].International Journal of Food Microbiology, 2017, 261:25-34.
[14] SOARES-SANTOS V, PARDO I, FERRER S.Improved detection and enumeration of yeasts in wine by Cells-qPCR[J].LWT, 2018, 90:90-97.
[15] LYU X C, LI Y, QIU W W, et al.Development of propidium monoazide combined with real-time quantitative PCR (PMA-qPCR) assays to quantify viable dominant microorganisms responsible for the traditional brewing of Hong Qu glutinous rice wine[J].Food Control, 2016, 66:69-78.
[16] XIONG T, CHEN J K, HUANG T, et al.Fast evaluation by quantitative PCR of microbial diversity and safety of Chinese Paocai inoculated with Lactobacillus plantarum NCU116 as the culture starter[J].LWT, 2019, 101:201-206.
[17] PAN Y, BREIDT F.Enumeration of viable Listeria monocytogenes cells by real-time PCR with propidium monoazide and ethidium monoazide in the presence of dead cells[J].Applied and Environmental Microbiology, 2007, 73(24):8 028-8 031.
[18] SHAO Y Y, WANG Z X, BAO Q H, et al.Application of propidium monoazide quantitative real-time PCR to quantify the viability of Lactobacillus delbrueckii ssp.bulgaricus[J].Journal of Dairy Science, 2016, 99(12):9 570-9 580.
[19] ANDORR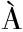 I, ESTEVE-ZARZOSO B, GUILLAM
I, ESTEVE-ZARZOSO B, GUILLAM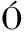 N J M, et al.Determination of viable wine yeast using DNA binding dyes and quantitative PCR[J].International Journal of Food Microbiology, 2010, 144(2):257-262.
N J M, et al.Determination of viable wine yeast using DNA binding dyes and quantitative PCR[J].International Journal of Food Microbiology, 2010, 144(2):257-262.
[20] ZHAO Y K, CHEN H, LIU H M, et al.Quantitative polymerase chain reaction coupled with sodium dodecyl sulfate and propidium monoazide for detection of viable Streptococcus agalactiae in milk[J].Frontiers in Microbiology, 2019, 10:661.
[21] TAKAHASHI H, KASUGA R, MIYA S, et al.Efficacy of propidium monoazide on quantitative real-time PCR based enumeration of Staphylococcus aureus live cells treated with various sanitizers[J].Journal of Food Protection, 2018, 81(11):1 815-1 820.
[22] CAMPBELL G R, PROSSER J, GLOVER A, et al.Detection of Escherichia coli O157:H7 in soil and water using multiplex PCR[J].Journal of Applied Microbiology, 2001, 91(6):1 004-1 010.